Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Modulación reversible mediada por luz de la vía de la proteína quinasa activada por mitógeno durante la diferenciación celular y
* Estos autores han contribuido por igual
En este artículo
Resumen
Este protocolo describe una estrategia optogenética para modular la actividad de la proteína quinasa activada por mitógeno (MAPK) durante la diferenciación celular y el desarrollo embrionario de Xenopus . Este método permite la activación reversible de la vía de señalización MAPK en el cultivo de células de mamíferos y en organismos vivos multicelulares, como los embriones Xenopus , con alta resolución espacial y temporal.
Resumen
La actividad quinasa es crucial para una plétora de funciones celulares, incluyendo proliferación celular, diferenciación, migración y apoptosis. Durante el desarrollo embrionario temprano, la actividad de la quinasa es altamente dinámica y generalizada a través del embrión. Los enfoques farmacológicos y genéticos se utilizan comúnmente para investigar las actividades de la quinasa. Desafortunadamente, es difícil lograr una resolución espacial y temporal superior usando estas estrategias. Además, no es factible controlar la actividad quinasa de una manera reversible en células vivas y organismos multicelulares. Tal limitación sigue siendo un cuello de botella para lograr una comprensión cuantitativa de la actividad de quinasa durante el desarrollo y la diferenciación. Este trabajo presenta una estrategia optogenética que aprovecha un sistema bicistrónico que contiene proteínas fotoactivables Arabidopsis thaliana criptocromo 2 (CRY2) y el dominio N-terminal de la hélice bucle-hélice-base que interactúa con criptocromo. ReversiBle de la ruta de señalización de la proteína quinasa activada por mitógenos (MAPK) se logra a través de la translocación de proteínas mediada por luz en células vivas. Este enfoque puede aplicarse a cultivos de células de mamíferos y embriones de vertebrados vivos. Este sistema bicistrónico puede generalizarse para controlar la actividad de otras quinasas con mecanismos de activación similares y puede aplicarse a otros sistemas modelo.
Introducción
Los factores de crecimiento están involucrados en un amplio espectro de funciones celulares, incluyendo proliferación, diferenciación, migración y apoptosis, y juegan papeles cruciales en muchos eventos biológicos, incluyendo el desarrollo embrionario, el envejecimiento y la regulación del estado mental 1 , 2 , 3 , 4 , 5 . Muchos factores de crecimiento señalan a través de complejas cascadas de señalización intracelular. Estos eventos de señalización son a menudo operados por la fosforilación de proteínas reversible de una manera regulada con precisión 6 , 7 . Por lo tanto, una comprensión de los resultados de señalización de las proteínas quinasas, que son responsables de la fosforilación de proteínas, es fundamentalmente importante.
Diferentes factores de crecimiento actúan a través de una red de señalización intracelular bastante común, aunque estimulan distInct respuestas celulares [ 8 , 9] . Los mediadores intracelulares comunes de las tirosina quinasas receptoras incluyen Ras, Raf, quinasa regulada por señal extracelular (ERK), proteína quinasa activada por mitógeno (MAPK) / ERK quinasa (MEK), phosphoinositide 3-quinasa (PI3K), Akt y fosfolipasa C gamma (PLC $ γ $) 10 , 11 . La acumulación de pruebas sugiere que la señalización de la diversidad y la especificidad dependen de la regulación espacial y temporal de la actividad de señalización [ 12] . Por ejemplo, en las células de feocromocitoma de rata (PC12), estimulación del factor de crecimiento epidérmico (EGF), lo que resulta en la proliferación celular, transitoriamente activa la vía ERK [ 9] . Por otra parte, la estimulación con factor de crecimiento nervioso (NGF), que conduce a la diferenciación celular, activa la vía ERK de una manera sostenida [ 9 , 13] . En cultivo rEn las neuronas del hipocampo, la señalización transitoria por el factor neurotrófico derivado del cerebro (BDNF) promueve el crecimiento de las neuritas primarias, mientras que la señalización sostenida conduce a una mayor ramificación de neuritas 14 . Durante el desarrollo embrionario temprano, phosphorylated ERK actividad es temporalmente dinámica y está extendida a través del embrión [ 6] . Una reciente pantalla genética durante la embriogénesis Xenopus temprana mostró que ERK y Akt cascadas de señalización, dos vías del factor de crecimiento primario aguas abajo, mostrar la etapa específica de activación perfiles [ 7] . Por lo tanto, la comprensión de los resultados de la señalización quinasa requiere de herramientas que pueden sondear las características espaciales y temporales de la actividad quinasa con suficiente resolución.
Los enfoques experimentales convencionales para investigar la naturaleza dinámica de la transducción de señales durante el desarrollo carecen de la resolución espacial y temporal deseable. Por ejemplo, los enfoques farmacológicos utilizanO biológicas para estimular o suprimir la transducción de señales en células y tejidos. La naturaleza difusiva de estas pequeñas moléculas hace que sea difícil restringir su acción a una región específica de interés [ 15] . Los enfoques genéticos ( por ejemplo, la transgénesis, el sistema Cre-Lox o mutagénesis) a menudo conducen a la activación o represión irreversible de la expresión del gen diana o la actividad proteica 16 , 17 , 18 . El sistema Tet-On / Tet-Off 19 ofrece un control temporal mejorado de la transcripción de genes, pero carece de un control espacial estricto porque se basa en la difusión de la tetraciclina. Los desarrollos recientes en la dimerización de la proteína inducida químicamente 20 o la foto-descoloración 21 , 22 , 23 , 24 han mejorado enormementeControl temporal de las redes de señalización. El control espacial, sin embargo, sigue siendo un desafío debido a la naturaleza difusiva de los productos químicos enjaulados.
Los recientes enfoques optogenéticos emergentes, que aprovechan la potencia de la luz para controlar las interacciones proteína-proteína, permiten la modulación de vías de señalización con una alta precisión espaciotemporal y reversibilidad. Poco después de su éxito inicial en el control de disparo neuronal 25 , 26 , 27 , optogenetics se ha extendido para controlar otros procesos celulares, tales como la transcripción de genes, la traducción, la migración celular, la diferenciación y la apoptosis 28 , 29 , 30 , 31 , 32 , 33 , 34 . Una estrategia con la pHotoactivatable proteína de parche Arabidopsis thaliana criptocromo 2 (CRY2) de proteínas y el dominio N-terminal de criptocromo de interacción de hélice-hélice-hélice (CIBN) se ha desarrollado recientemente para controlar Raf1 kinasa actividad en células de mamíferos y Xenopus embriones [ 35] . CRY2 se une a CIBN a la luz azul de estimulación, y el complejo de proteínas CRY2 / CIBN se disocia espontáneamente en la oscuridad [ 34] . La luz azul excita el cofactor CRY2, flavin adenine dinucleotide (FAD), que conduce a un cambio conformacional en CRY2 y su posterior unión a CIBN. Pueden producirse mutantes constitutivamente activos (W374A) y deficientes en flavina (D387A) de CRY2 a través de mutaciones en la bolsa de unión a FAD: el mutante W374A de CRY2 se une a CIBN independiente de la luz, mientras que el mutante CRY2 D387A no se une a CIBN bajo azul - estimulación de luz 36 , 37 . El sistema optogenético descrito iN este protocolo utiliza CRY2 de tipo salvaje y CIBN para inducir la activación de Raf1 mediada por translocación de proteínas en células vivas. Se sabe que el reclutamiento de membrana de Raf1 mejora su actividad 38 . En este sistema, un tándem CIBN módulo está anclado a la membrana plasmática y CRY2-mCherry se fusiona con el N-terminal de Raf1 35 . En ausencia de luz azul, CRY2-mCherry-Raf1 permanece en el citoplasma, y Raf1 es inactivo. La estimulación con luz azul induce la unión de CRY2-CIBN y recluta Raf1 en la membrana plasmática, donde Raf1 se activa. La activación de Raf estimula una cascada de señalización Raf / MEK / ERK. Las proteínas de fusión CRY2 y CIBN están codificadas en un sistema genético bicistrónico. Esta estrategia puede ser generalizada para controlar otras quinasas, como Akt, cuyo estado de activación también puede ser activado por la translocación de proteínas en las células [ 39] . Este trabajo presenta protocolos detallados para la implementación de esta estrategia optogenética en cultu de células de mamíferosRes y organismos multicelulares.
Protocolo
La investigación en animales se realizó de acuerdo con las pautas establecidas por el Comité de Cuidado y Uso de Animales Institucionales de Illinois (IACUC) y el Departamento de Recursos Animales de la Universidad de Illinois (DAR).
1. Inducción optogenética de la localización de proteínas en BHK21 Cultura de células de mamíferos
NOTA: Los pasos 1.1-1.3 proporcionan un método para ensamblar una cámara de cultivo de células para la obtención de imágenes con objetivos de alta ampliación ( por ejemplo, 63X o 100X), que típicamente tienen distancias de trabajo cortas. Estos objetivos requieren un cubreobjetos de vidrio fino ( por ejemplo, # 1.5, 170 μm de grosor) como sustrato de formación de imágenes. Alternativamente, se puede usar un plato de cultivo de células de fondo de vidrio / portaobjetos. En tal caso, los pasos 1.1-1.3 pueden omitirse.
- Limpieza de cubreobjetos de vidrio
- Coloque 30 cubreobjetos de vidrio en un soporte de cubreobjetos.
- En un vaso de precipitados de plástico de 600 ml, agregue 20 g de detergente y 400 ml de agua caliente del grifo. PlacEl vaso en un agitador magnético y revuelva hasta que se disuelva todo el detergente. Retire la espuma con papel de seda.
- Utilice el fondo de una placa de Petri de 100 mm x 15 mm como separador de la barra de agitación y como superficie para el soporte del cubreobjetos. Para mantener un flujo adecuado durante el proceso de limpieza, hacer 3-4 orificios de drenaje en la placa de Petri.
- Para hacer los orificios de drenaje, calentar un soldador y quemar a través del plástico en una campana ventilada.
PRECAUCIÓN: No toque el soldador caliente con las manos desnudas. - Coloque el cubreobjetos en la placa de Petri en el vaso de precipitados. Se agita durante 2 ha una noche a temperatura ambiente.
- Lavar tres veces con 500 ml de agua DI en un vaso de precipitados de 1.000 ml. La solución detergente puede reutilizarse.
- Recoger los cubreobjetos individuales con fórceps. Sumerja los cubreobjetos en etanol a prueba de 190% (95%) durante 1 minuto.
- Seque los cubreobjetos dentro de una capucha estéril. Guarde los cubreobjetos en un recipiente estéril de plástico hasta su uso.
- Fabricación de cubreobjetos recubiertos de poli-L-lisina (PLL)
- Hacer tampón borato 0,1 M disolviendo 1,24 g de ácido bórico y 1,9 g de tetraborato disódico en 400 ml de agua. Ajustar el pH a 8,5 con NaOH 10 M.
- Disolver PLL en el tampón de borato hasta una concentración final de 1 mg / mL. Alícuota de la solución PLL en tubos de centrífuga estériles de 50 ml.
- Añadir 50 ml de solución de revestimiento de PLL a un plato de cultivo de tejidos de 10 cm. Colocar el plato en una incubadora a 37 ° C para calentar la solución PLL.
- Abra el contenedor de cubreobjetos (paso 1.1.8) en una campana esterilizada.
- Sumerja los cubreobjetos limpiados uno por uno en la solución de PLL precalentada en el plato de cultivo de tejidos. Evite la formación de burbujas para sumergir todas las superficies.
- Coloque el plato con el cubreobjetos en una incubadora de CO 2 a 37 ° C durante la noche. Saque cada cubreobjetos y colóquelo en un porta vasos estéril. Enjuague tres veces con agua ultrapura esterilizada en autoclave (18,2 MΩ & #183 mu m de resistividad) en un vaso de precipitados de 1.000 ml en una cubierta estéril.
- Seque los cubreobjetos del porta-cubreobjetos de la cubierta estéril. Guarde los cubreobjetos en un recipiente estéril de plástico hasta su uso.
- Ensamblaje de cámaras de cultivo celular de polidimetilsiloxano (PDMS)
- En un recipiente de plástico, se pesan 40 g de PDMS base y se añaden 4 g de agente de curado para conseguir una relación de 10: 1 en peso / peso de PDMS y agente de curado. Mezclar el PDMS / agente de curado agitando con una punta de pipeta durante 2 min. Alternativamente, utilice un mezclador centrífugo.
- Utilice un vaso de precipitados con un diámetro de 4 pulgadas como plantilla para hacer un soporte con un trozo de papel de aluminio. Coloque el soporte de papel de aluminio en una placa de Petri de 15 cm. Coloque una oblea de silicio de 4 pulgadas dentro de este soporte de aluminio.
- Vierta la solución mixta de PDMS en el soporte. Utilice una vara de cristal delgada para tocar el borde de la oblea suavemente. Retire las burbujas de aire atrapadas debajo de la oblea.
- Introducir la placa de Petri de 15 cm en un chambe de vacíoR para desgasear la solución PDMS. Continuar la desgasificación durante unos 20 minutos, hasta que no se generen burbujas. Mientras tanto, precaliente un horno de convección a 65 ° C.
- Coloque cuidadosamente la placa Petri de 15 cm en el horno. Incubar durante 2 h.
- Retire la placa del horno. Utilice una varilla de vidrio para tocar suavemente el borde del PDMS y asegúrese de reticulación completa. Si no, incubar más, hasta que el PDMS es sólido y no pegajoso.
- Retire la hoja de aluminio de la placa y descárguela de la oblea de silicio. Comenzando desde el borde, retire cuidadosamente el PDMS curado de la oblea de silicio.
PRECAUCIÓN: Evite aplicar demasiada fuerza, ya que puede romper la oblea. - Recorte el PDMS en piezas rectangulares de 20 mm x 30 mm para colocar el cubreobjetos de 24 mm x 40 mm con una cuchilla de afeitar.
- Utilice una cuchilla en forma de cincel para cortar una abertura rectangular de 10 mm x 20 mm desde el centro de cada pieza PDMS.
- Limpie las cámaras PDMS utilizando el protocolo de detergente descrito en el paso 1.1.
- Secar las cámaras PDMS en una campana limpia. Coloque las cámaras secas en un recipiente autoclavable cubierto con papel de aluminio.
- Autoclavear el recipiente por gravedad (121 ° C, 15 psi) durante 30 min y almacenar el recipiente a temperatura ambiente.
- Cultivo y transfección de células BHK21
- Hacer 500 ml de medio para cultivo de células BHK21 mezclando 445 ml de DMEM con 50 ml de FBS y 5 ml de 100 × Penn-Strep-Glutamina (10.000 U / mL de penicilina, 10 mg / ml de estreptomicina y 29,2 mg / mL de L Glutamina). Asegúrese de que la concentración final de FBS es del 10%.
- Alícuota de 10 ml de medio no dependiente de HEPES CO 2 para la obtención de imágenes de células vivas.
NOTA: El medio independiente de CO 2 soporta el crecimiento celular sin un incubador de CO 2 y es ideal para la obtención de imágenes de las células en condiciones atmosféricas. Consulte la lista de materiales para obtener información detallada. Además de la línea celular BHK21, otros tipos de células de mamíferos también deben darResultados similares. - Ensamble un dispositivo de cultivo celular colocando una cámara PDMS esterilizada esterilizada (paso 1.3) en un cubreobjetos estéril revestido con PLL (paso 1.2) en una cubierta estéril.
- Coloque cada dispositivo en una placa de Petri estéril de 60 mm.
- Utilice 0,5 ml de tripsina al 0,25% para separar las células de un pocillo de una placa de cultivo de tejidos de 12 pocillos. Contar la densidad celular con un hemocitómetro. Se plaquean 20.000 células BHK21 en la cámara (aproximadamente 10.000 células / cm2) con 200 μl de medio de cultivo celular.
- Prepare el plásmido de ADN con un kit de preparación de plásmidos (consulte la Lista de materiales).
NOTA: El diseño y la funcionalidad del CRY2-mCherry-Raf1-2A-2CIBN-GFP-CaaX se muestran en la Figura 1A - 1B . - Veinticuatro (24) h después del recubrimiento celular, transfectan las células con 50-100 ng de plásmido CRY2-mCherry-Raf1-2A-2CIBN-GFP-CaaX, de acuerdo con el protocolo del fabricante.
- Tres (3) horas después de la transfección, cambie el mEdium a 200 μl de medio de cultivo fresco (etapa 1.4.1) y permitir que las células se recuperen durante la noche incubando el cultivo en una incubadora a 37ºC, 5% de CO 2 .
- Fluorescencia de imágenes de células vivas
- Encienda el ordenador, la fuente de luz, el microscopio y la cámara. Utilice un microscopio confocal de fluorescencia (equipado con un objetivo de inmersión de aceite 100X) para el resto del protocolo.
NOTA: También se puede usar un microscopio de epifluorescencia invertida. - Sustituir el medio de cultivo celular por 200 μl de medio independiente de CO 2 .
- Configurar el protocolo de adquisición de datos antes de colocar las células en el microscopio. Utilice el canal FITC de excitación de 488 nm para la estimulación optogenética. Utilice el canal de TRITC de excitación de 561 nm para localizar las células transfectadas y rastrear la localización celular de la proteína marcada con mCherry.
- Establezca la ganancia de los canales FITC y TRITC como 120 y 200, respectivamente. Utilice un tiempo de píxel de 2,21; s y tamaño de 512 x 512 píxeles.
- Mida la potencia de la luz de 488 nm colocando un medidor de potencia cerca de la ventana de objetivo. Una potencia total de 2 μW (aproximadamente 10.000 W / cm2 en el foco) es suficiente para inducir la asociación de CIBN-CRY2PHR.
- Configure una adquisición de marca de tiempo con un intervalo de 5 s y un tiempo de adquisición total de 2 min.
- Aplique el material apropiado para el índice (aceite de inmersión) en la ventana del objetivo. Utilice el modo de contraste de fase para enfocar las células en la superficie de la cubreobjetos.
- Localice una célula transfectada bajo una luz de 561 nm moviendo la etapa del microscopio.
PRECAUCIÓN: Evite usar luz azul durante este paso, ya que activará la asociación entre proteínas fotoactivables. - Una vez que una célula transfectada se encuentra, iniciar la adquisición de datos.
NOTA: Las células exitosamente transfectadas deben mostrar fluorescencia tanto en los canales FITC (de 2CIBN-GFP-CaaX) como en los de TRITC (de CRY2-mCherry-Raf1) ( Figura 1C-D ). - Grabe una serie de imágenes con fecha y hora en los canales FITC y TRITC ( Figura 1D ).
- Para investigar la disociación espontánea del complejo de proteína CRY2-CIBN, registrar otra marca de tiempo con el canal de Txred solo durante 20 min, con un intervalo de 30 s.
- Guarde la imagen con hora para el análisis de datos.
- Encienda el ordenador, la fuente de luz, el microscopio y la cámara. Utilice un microscopio confocal de fluorescencia (equipado con un objetivo de inmersión de aceite 100X) para el resto del protocolo.
- Análisis de imagen
- Abra las imágenes con un software de análisis de imágenes que pueda extraer la intensidad de una imagen 40 .
- Seleccione dos instantáneas representativas: una antes de la exposición a la luz azul y la otra exposición posterior a la luz azul.
- Dibujar una línea a través de la célula que abarca el fondo, la membrana plasmática y el citoplasma. Proyecte la intensidad a lo largo de esta línea. Guarde los valores y ploteelos para comparar la diferencia antes y después de la exposición a la luz ( Figura 1D ).
- Para analizar la cinética de asociación de proteínas,T una región representativa de interés (ROI) en la membrana plasmática, un ROI en el citoplasma y un ROI en el fondo.
- Adquirir las intensidades medias de la membrana plasmática (I PM ), el citoplasma (I CYT ) y el fondo (I BKD ) para la pila de imágenes.
- Calcular la relación de intensidad membrana / citosólica para cada imagen utilizando la siguiente ecuación:
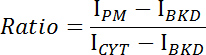
- Trazar la relación en función del tiempo y determinar la cinética de unión o disociación de CRY2-CIBN ( Figura 1E- F ).
2. Construcción de una matriz de LED para la estimulación de luz a largo plazo en una incubadora de CO 2
NOTA: El esquema general de la configuración experimental se muestra en la Figura 2A .
- Haga el arreglo del LED insertando 12 LED azules en dos breadboarDs y conectando resistencias de limitación de corriente.
NOTA: Con una fuente de alimentación de 30 V, se pueden conectar cuatro LED en serie y su brillo puede ser controlado por una resistencia limitadora de corriente. - Coloque los paneles en una caja de aluminio.
NOTA: La altura de la caja de aluminio debe ser de 2 pulg. Esta altura es óptima para iluminar una placa de cultivo de tejidos de 12 pocillos, porque el tamaño de la mancha de luz divergente es el mismo que el de un solo pozo. - Utilice dos alambres metálicos para conectarse a la fuente de alimentación. Asegúrese de que la longitud del cable es suficiente cuando la caja de luz se coloca dentro de una incubadora de CO 2 .
- Utilice un difusor de luz transparente como la tapa de la caja de luz ( Figura 2C ).
- Calibre la potencia de salida de cada LED en un rango de entradas de tensión. Utilice una potencia de 0,2 mW / cm2 para el ensayo de diferenciación de células 24 h PC12. Utilice una potencia de 5 mW / cm 2 para los embriones o explantes vivos de XenopusEnsayos
3. Inducción Optogenética de Diferenciación de Células PC12
- Cultivo celular y transfección
- Hacer 500 ml de medio para un cultivo celular de feocromocitoma de rata (PC12) mezclando 407,5 ml de F12K con 75 ml de suero de caballo + 12,5 ml de FBS + 5 ml de 100 × Penn-Strep-Glutamina (concentraciones finales de suero de caballo y FBS : 15% y 2,5%, respectivamente). Hacer medio de suero bajo mezclando 1 volumen de medio completo en 99 volúmenes de medio F12K.
- Placa de células PC12 en una placa de 12 pocillos a una densidad de 300.000 células / pocillo o 75.000 células / cm 2 .
- Transfectar las células con CRY2-mCherry-Raf1-2A-2CIBN-GFP-CaaX 24 h después del recubrimiento celular (similar al paso 1.4.7). Utilice 1,2 μg de ADN para cada pocillo. Dejar que las células se recuperen durante la noche en medio de cultivo en un incubador a 37ºC suplementado con 5% de CO2. Compruebe la eficacia de la transfección 16 h después de la transfección.
NOTA: Un 30-50% de ef transfecciónDebe lograrse la eficiencia necesaria para asegurar un conteo de células suficiente. - Cambiar el medio a medio de suero bajo 24 h después de la transfección. Utilizar 1 ml de medio por pocillo de una placa de 12 pocillos.
- Diferenciación de células PC12 inducida por luz
- Coloque el conjunto LED en una incubadora de CO 2 . Conéctelo a la fuente de alimentación utilizando un par de cables. Ajuste la potencia del LED a 0,2 mW / cm 2 . Colocar la placa de 12 pocillos que contiene células PC12 transfectadas (paso 3.1.3) en la ventana de la matriz LED. Aplique iluminación continua durante 24 h en un incubador a 37 ° C suplementado con 5% de CO 2 .
- Fluorescencia de imágenes de células vivas
- Siga los pasos 1.5.1-1.5.4 para el microscopio y la preparación de la muestra.
- Configure la adquisición de datos de una sola instantánea. Utilice 200 ms para el canal GFP y Txred.
- Captura imágenes de las células transfectadas en los canales GFP y Txred. Registro de aproximadamente 200 celLs para cada condición. Guarde los archivos para el análisis de datos.
- Análisis de imagen
- Contar el porcentaje de células diferenciadas sobre el número total de células transfectadas.
- Utilice cualquier módulo de conteo de células en el software de análisis de imágenes para contar las células.
- Contar manualmente las células diferenciadas.
NOTA: Las células diferenciadas se definen como aquéllas en las que al menos una neurita es discerniblemente más larga que el cuerpo celular ( Figura 3A -3B ). - Repita el paso 3.4.3 con células indiferenciadas.
- Calcular la relación de diferenciación usando la siguiente ecuación:
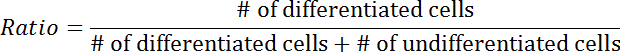
4. Control Optogenético de la Actividad Quinasa en Embriones Xenopus
- Preparación de tampones
- Preparar 1 l de 1x Marc Marcador modificado (MMR): 100 mMNaCl, KCl 2 mM, MgCl _ { 2 } 1 mM, CaCl _ { 2 } 2 mM y HEPES 5 mM; PH 7,5.
- Preparación de mRNA
- Digerir el ADN del plásmido CRY2-mCherry-Raf1-2A-2CIBN-GFP-CaaX (2 μg) con 1 μl de ApaI (50 unidades) a 37 ° C durante 2 h.
- Precipitar el ADN linealizado en 60 μl de etanol al 100%. Girar a 12.000 × g durante 5 min a temperatura ambiente para sedimentar el ADN. Retire con cuidado el sobrenadante y lave el pellet de nuevo con 60 μl de etanol al 70%. Girar a 12.000 × g durante otros 5 min a temperatura ambiente. Retire cuidadosamente el sobrenadante y resuspenda el sedimento de ADN en 6 μl de H 2 O libre de ARNasa.
- Realizar la transcripción in vitro mediante la incubación de 1 μ g de ADN linealizado con 2 μ l de SP6 suministrado RNA polimerasa, una mezcla de ribonucleótidos (10 mM ATP, CTP y UTP, 2 mM GTP y 8 mM cap analógico), y 20 μ l de nuclease- Agua libre a temperatura ambiente durante 1 h.
- Quite el DNA por digestión con DNasa I a 37ºC durante al menos 15 minutos y purificar el ARN sintetizado usando una columna de rotación de membrana de sílice.
- Detener la reacción y precipitar el ARN mediante la adición de 30 μL de agua libre de nucleasa y 30 μL de solución de precipitación de LiCl. Mezclar bien y dejar enfriar durante 30 minutos a -20 ° C.
- Centrifugar a 4 ° C durante 15 min a 12.000 × g para sedimentar el ARN.
- Retire cuidadosamente el sobrenadante. Lavar el ARN una vez con 1 ml de etanol al 70% y resuspender en 40 μl de agua libre de nucleasa.
- Cargar 40 μL de ARN en una columna de espín. Centrifugar a 4 ° C durante 1 min a 12.000 × g para eliminar los nucleótidos no incorporados y tapas.
- Cosecha de los embriones Xenopus
- Siga el protocolo descrito por Sive et al. 41 para obtener los embriones de Xenopus .
- Microinyección de mRNA
- FabricarLas agujas para la microinyección tirando de los capilares de cristal con un extractor capilar. Ajuste el programa de tracción a: calor = 355, fuerza de tracción = 80, velocidad = 50 y tiempo = 100.
- Retire la capa de gelatina de los embriones tratándolos con 3% de cisteína (diluida en 0,2x MMR).
- Transferir los embriones a 3% de polisacáridos y 0.5x MMR solución para microinyección. Inyectar 500 pg a 1 ng de ARN CRY2-mCherry-Raf1-2A-2CIBN-GFP-CaaX en cada embrión.
NOTA: La inyección de una dosis superior a 1 ng da como resultado una activación independiente de la luz azul de la señalización MAPK.
- Estimulación optogenética de la actividad quinasa
- Cultura microinyectó embriones en polisacárido al 3%, solución MMR 0,5x a temperatura ambiente hasta que alcanzaron la etapa gastrula media (etapa 12). A continuación, cultivar los embriones en una solución de 0,2x MMR.
- Transferir los embriones o explantes a una placa de 12 pocillos. Coloque la placa de 12 pocillos en la matriz de LED construida en el hogar (paso 2.5) para la luz azulT (475 nm).
- Coloque un espejo en la parte superior de la placa de 12 pocillos para asegurar la iluminación de luz azul de los embriones o explantes.
- Ajuste la potencia de la luz azul a 5 mW / cm 2 .
NOTA: El tratamiento con luz azul puede realizarse en cualquier momento deseado, ya sea en polisacárido al 3%, solución MMR 0,5x o solución MMR 0,2x. - Cosecha de los embriones en cualquier momento deseado. Utilícelos para el análisis histológico, Western-blot o de expresión génica.
Resultados
Expresión Ratiométrica de pares de proteínas fotoactivables: La Figura 1A muestra el diseño de una construcción optogenética bicistrónica, CRY2-mCherry-Raf1-P2A-CIBN-CIBN-GFP-CaaX (denominada CRY2-2A-2CIBN), basada en la proteína teschovirum- 1 2A (P2A), que muestra la más alta eficiencia de omisión de ribosomas entre las líneas celulares de mamíferos [ 42] . En trabajos previos, se ha determinado que la relación óptima ...
Discusión
Cuando se construye la caja de luz, se debe medir la potencia de los LED individuales. Según la experiencia anterior, la potencia de salida puede variar entre LED individuales debido a la variación de fabricación. Seleccione un conjunto de LED que tengan una potencia de salida de 10% entre sí. El número de LEDs, la resistencia de limitación de corriente y la entrada de potencia se pueden modificar para diferentes tipos de recipientes de cultivo celular ( por ejemplo, una placa de 6 pocillos o de 24 pocill...
Divulgaciones
Los autores declaran que no tienen intereses financieros en competencia.
Agradecimientos
Este trabajo fue apoyado por la Universidad de Illinois en Urbana-Champaign (UIUC) y los Institutos Nacionales de Salud (NIGMS R01GM111816).
Materiales
| Name | Company | Catalog Number | Comments |
| Glass coverslip | VWR | 48393 230 | Substrate for live cell imaging |
| Coverslip holder | Newcomer Supply | 6817B | Holder for coverslips |
| Detergent | ThermoFisher | 16 000 104 | For cleaning coverslips |
| Boric acid | Sigma-Aldrich | B6768-500G | For making PLL buffer |
| Disodium tetraborate | Sigma-Aldrich | 71996-250G | For making PLL buffer |
| Plastic beaker | Nalgene | 1201-1000 | For cleaning coverslips |
| Sodium hydroxide | Sigma-Aldrich | 221465-2.5KG | For adjust pH |
| Poly-L-lysine hydrobromide | Sigma-Aldrich | P1274-500MG | For coating coverslip |
| Diethylpyrocarbonate (DEPC)-Treated Water | ThermoFisher Scientific | 750024 | For DNA preparation |
| Cover Glass Forceps | Ted Pella | 5645 | Cover glass handling |
| Tissue cutlure dish | Thermofisher | 12565321 | Cell culture dish |
| Sterile centrifuge tubes | ThermoFisher | 12-565-271 | Buffer storage |
| Transfection Reagent | ThermoFisher | R0534 | Transfection |
| CO2-independent medium | ThermoFisher | 18045088 | For live cell imaging |
| Polydimethylsiloxane (PDMS) | Ellsworth Adhesives | 184 SIL ELAST KIT 0.5KG | Form make cell chamber |
| Plasmid Maxiprep kit | Qiagen | 12965 | Plasmid preparation |
| DMEM medium | ThermoFisher | 11965-084 | Cell culturing medium component |
| F12K medium | ThermoFisher | 21127022 | Cell culturing medium component |
| Horse serum | ThermoFisher | 16050122 | Cell culturing medium component |
| Fetal Bovine Serum | Signa-Aldrich | 12303C-500 mL | Cell culturing medium component |
| Penicillin-Streptomycin-Glutamine | ThermoFisher | 10378016 | Cell culturing medium component |
| Trypsin (0.25%), phenol red | ThermoFisher Scientific | 15050065 | For mammalian cell dissociation |
| Agarose | Fisher Scientific | BP1356-100 | For DNA preparation |
| Ficoll PM400 | GE Heathcare Life Sciences | 17-5442-02 | For embryo buffer |
| L-Cysteine hydrochloride monohydrate | Sigma-Aldrich | 1.02839.0025 | Oocyte preparation |
| ApaI | ThermoFisher | FD1414 | For linearization of plasmids |
| Dnase I | ThermoFisher | AM2222 | For removing DNA template in the in vitro transcription assay |
| Index-match materials (immersion oil) | Thorlabs | MOIL-20LN | For matching the index between sample substrate and objective |
| Blue LED | Adafruit | 301 | Light source for optogenetic stimulation |
| Resistor kit | Amazon | EPC-103 | current-limiting resistor |
| Aluminum boxes | BUD Industries | AC-401 | light box |
| BreadBoard | Jekewin | 837654333686 | For making LED array |
| Hook up Wire | Electronix Express | 27WK22SLD25 | For making LED array |
| Relay Module | Jbtek | SRD-05VDC-SL-C | For intermittent light control |
| DC Power Supply | TMS | DCPowerSupply-LW-(PS-305D) | Power supply for LED |
| Silicon Power Head | Thorlabs | S121C | For light intensity measurement |
| Power meter | Thorlabs | PM100D | For light intensity measurement |
| Microscope | Leica Biosystems | DMI8 | For live cell imaging |
| BioSafety Cabinet | ThermoFisher | 1300 Series A2 | For mammalian cell handling |
| CO2 incubator | ThermoFisher | Isotemp | For mammalian cell culturing |
| Stereo microscope | Leica | M60 | For embryo micro-manipulation |
| Microinjector | Narishige | IM300 | For embryo microinjection |
| Micropipette puller | Sutter Instruments | P87 | Needle puller |
| in vitro transcription kit | ThermoFisher | AM1340 | For in vitro transcription. The kit includes nuclease-free water, SP6 RNA Polymerase, ribonucleotide mixture, cap analog, lithium choride precipitation solution, and spin column |
| RNA purfication kit | Qiagen | 74104 | Silica-membrane spin column for purification of synthesized RNA |
| Convection oven | MTI corporation | EQ-DHG-9015 | PDMS curing |
| Centrifugal mixer and teflon container | THINKY | AR310 | For mixing PDMS |
| Silicon wafer | UniversityWafer | 452 | Base for making PDMS devices |
| Blade | Techni Edge | 01-801 | For cutting PDMS |
| Capillary glass | Sutter Instruments | BF100-58-10 | For fabrication of injecting needles. |
Referencias
- Schlessinger, J., Ullrich, A. Growth factor signaling by receptor tyrosine kinases. Neuron. 9 (3), 383-391 (1992).
- Thisse, B., Thisse, C. Functions and regulations of fibroblast growth factor signaling during embryonic development. Dev Biol. 287 (2), 390-402 (2005).
- Perrimon, N., Pitsouli, C., Shilo, B. Z. Signaling mechanisms controlling cell fate and embryonic patterning. Cold Spring Harb Perspect Biol. 4 (8), a005975 (2012).
- Salles, F. H., et al. Mental disorders, functional impairment, and nerve growth factor. Psychol Res Behav Manag. 10, 9-15 (2017).
- Basson, M. A. Signaling in cell differentiation and morphogenesis. Cold Spring Harb Perspect Biol. 4 (6), (2012).
- Schohl, A., Fagotto, F. beta-catenin, MAPK and Smad signaling during early Xenopus development. Development. 129 (1), 37-52 (2002).
- Zhang, S. W., Li, J. J., Lea, R., Amaya, E., Dorey, K. A Functional Genome-Wide In Vivo Screen Identifies New Regulators of Signalling Pathways during Early Xenopus Embryogenesis. PloS one. 8 (11), e79469 (2013).
- Sweeney, C., et al. Growth factor-specific signaling pathway stimulation and gene expression mediated by ErbB receptors. J Biol Chem. 276 (25), 22685-22698 (2001).
- Marshall, C. J. Specificity of receptor tyrosine kinase signaling: transient versus sustained extracellular signal-regulated kinase activation. Cell. 80 (2), 179-185 (1995).
- Vandergeer, P., Hunter, T., Lindberg, R. A. Receptor Protein-Tyrosine Kinases and Their Signal-Transduction Pathways. Annu Rev Cell Biol. 10, 251-337 (1994).
- Lemmon, M. A., Schlessinger, J. Cell signaling by receptor tyrosine kinases. Cell. 141 (7), 1117-1134 (2010).
- Hunter, T. Signaling--2000 and beyond. Cell. 100 (1), 113-127 (2000).
- Qiu, M. S., Green, S. H. PC12 cell neuronal differentiation is associated with prolonged p21ras activity and consequent prolonged ERK activity. Neuron. 9 (4), 705-717 (1992).
- Ji, Y., et al. Acute and gradual increases in BDNF concentration elicit distinct signaling and functions in neurons. Nat Neurosci. 13 (3), 302-309 (2010).
- Luby-Phelps, K. Cytoarchitecture and physical properties of cytoplasm: volume, viscosity, diffusion, intracellular surface area. Int Rev Cytol. 192, 189-221 (2000).
- Sauer, B. Inducible gene targeting in mice using the Cre/lox system. Methods-a Companion to Methods in Enzymology. 14 (4), 381-392 (1998).
- Ling, M. M., Robinson, B. H. Approaches to DNA mutagenesis: an overview. Anal Biochem. 254 (2), 157-178 (1997).
- Gama Sosa, M. A., De Gasperi, R., Elder, G. A. Animal transgenesis: an overview. Brain Struct Funct. 214 (2-3), 91-109 (2010).
- Wanka, F., et al. Tet-on, or Tet-off, that is the question: Advanced conditional gene expression in Aspergillus. Fungal Genet Biol. 89, 72-83 (2016).
- Karginov, A. V., Ding, F., Kota, P., Dokholyan, N. V., Hahn, K. M. Engineered allosteric activation of kinases in living cells. Nat Biotechnol. 28 (7), 743-747 (2010).
- Liu, Q. Y., Deiters, A. Optochemical Control of Deoxyoligonucleotide Function via a Nucleobase-Caging Approach. Accounts of Chemical Research. 47 (1), 45-55 (2014).
- Arbely, E., Torres-Kolbus, J., Deiters, A., Chin, J. W. Photocontrol of tyrosine phosphorylation in mammalian cells via genetic encoding of photocaged tyrosine. J Am Chem Soc. 134 (29), 11912-11915 (2012).
- Gautier, A., Deiters, A., Chin, J. W. Light-activated kinases enable temporal dissection of signaling networks in living cells. J Am Chem Soc. 133 (7), 2124-2127 (2011).
- Nguyen, D. P., et al. Genetic Encoding of Photocaged Cysteine Allows Photoactivation of TEV Protease in Live Mammalian Cells. J Am Chem Soc. 136 (6), 2240-2243 (2014).
- Deisseroth, K. Optogenetics. Nat Methods. 8 (1), 26-29 (2011).
- Boyden, E. S., Zhang, F., Bamberg, E., Nagel, G., Deisseroth, K. Millisecond-timescale, genetically targeted optical control of neural activity. Nat Neurosci. 8 (9), 1263-1268 (2005).
- Banghart, M., Borges, K., Isacoff, E., Trauner, D., Kramer, R. H. Light-activated ion channels for remote control of neuronal firing. Nat Neurosci. 7 (12), 1381-1386 (2004).
- Zhang, K., Cui, B. Optogenetic control of intracellular signaling pathways. Trends in Biotechnology. 33 (2), 92-100 (2015).
- Tischer, D., Weiner, O. D. Illuminating cell signalling with optogenetic tools. Nat Rev Mol Cell Bio. 15 (8), 551-558 (2014).
- Kim, B., Lin, M. Z. Optobiology: optical control of biological processes via protein engineering. Biochemical Society Transactions. 41 (5), 1183-1188 (2013).
- Tucker, C. L. Manipulating cellular processes using optical control of protein-protein interactions. Prog Brain Res. 196, 95-117 (2012).
- Toettcher, J. E., Gong, D. Q., Lim, W. A., Weiner, O. D. Light Control of Plasma Membrane Recruitment Using the Phy-Pif System. Method Enzymol. 497, 409-423 (2011).
- Zoltowski, B. D., Gardner, K. H. Tripping the light fantastic: blue-light photoreceptors as examples of environmentally modulated protein-protein interactions. Biochemistry. 50 (1), 4-16 (2011).
- Kennedy, M. J., et al. Rapid blue-light-mediated induction of protein interactions in living cells. Nat Methods. 7 (12), 973-975 (2010).
- Krishnamurthy, V. V., et al. Reversible optogenetic control of kinase activity during differentiation and embryonic development. Development. 143 (21), 4085-4094 (2016).
- Liu, H., et al. Photoexcited CRY2 Interacts with CIB1 to Regulate Transcription and Floral Initiation in Arabidopsis. Science. 322 (5907), 1535-1539 (2008).
- Li, X., et al. Arabidopsis cryptochrome 2 (CRY2) functions by the photoactivation mechanism distinct from the tryptophan (trp) triad-dependent photoreduction. Proc Natl Acad Sci U S A. 108 (51), 20844-20849 (2011).
- Leevers, S. J., Paterson, H. F., Marshall, C. J. Requirement for Ras in Raf Activation Is Overcome by Targeting Raf to the Plasma-Membrane. Nature. 369 (6479), 411-414 (1994).
- Kohn, A. D., Takeuchi, F., Roth, R. A. Akt, a pleckstrin homology domain containing kinase, is activated primarily by phosphorylation. J Biol Chem. 271 (36), 21920-21926 (1996).
- Schindelin, J., et al. Fiji: an open-source platform for biological-image analysis. Nature Methods. 9 (7), 676-682 (2012).
- Sive, H. L., Grainger, R. M., Harland, R. M. . Early Development of Xenopus laevis: A Laboratory Manual. , (2000).
- Kim, J. H., et al. High cleavage efficiency of a 2A peptide derived from porcine teschovirus-1 in human cell lines, zebrafish and mice. PloS one. 6 (4), e18556 (2011).
- Ishimura, A., et al. Oncogenic Met receptor induces ectopic structures in Xenopus embryos. Oncogene. 25 (31), 4286-4299 (2006).
- Zhang, K., et al. Light-Mediated Kinetic Control Reveals the Temporal Effect of the Raf/MEK/ERK Pathway in PC12 Cell Neurite Outgrowth. PloS one. 9 (3), e92917 (2014).
- Mohanty, S. K., Lakshminarayananan, V. Optical Techniques in Optogenetics. J Mod Opt. 62 (12), 949-970 (2015).
- Taslimi, A., et al. Optimized second-generation CRY2-CIB dimerizers and photoactivatable Cre recombinase. Nature chemical biology. 12 (6), 425-430 (2016).
- Kawano, F., Suzuki, H., Furuya, A., Sato, M. Engineered pairs of distinct photoswitches for optogenetic control of cellular proteins. Nat Commun. 6, 6256 (2015).
- Wang, H., et al. LOVTRAP: an optogenetic system for photoinduced protein dissociation. Nat Methods. 13 (9), 755-758 (2016).
- Chang, K. Y., et al. Light-inducible receptor tyrosine kinases that regulate neurotrophin signalling. Nat Commun. 5, 4057 (2014).
- Boulina, M., Samarajeewa, H., Baker, J. D., Kim, M. D., Chiba, A. Live imaging of multicolor-labeled cells in Drosophila. Development. 140 (7), 1605-1613 (2013).
- Liu, H., Gomez, G., Lin, S., Lin, C. Optogenetic control of transcription in zebrafish. PLoS One. 7 (11), e50738 (2012).
- Buckley, C. E., et al. Reversible Optogenetic Control of Subcellular Protein Localization in a Live Vertebrate Embryo. Dev Cell. 36 (1), 117-126 (2016).
- Motta-Mena, L. B., et al. An optogenetic gene expression system with rapid activation and deactivation kinetics. Nature chemical biology. 10 (3), 196-202 (2014).
- Beyer, H. M., et al. Red Light-Regulated Reversible Nuclear Localization of Proteins in Mammalian Cells and Zebrafish. ACS Synth Biol. 4 (9), 951-958 (2015).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados