A subscription to JoVE is required to view this content. Sign in or start your free trial.
Method Article
אור בתיווך אפנון הפוך של מיטוגן המופעל חלבון קינאז נתיב במהלך הבחנה Cell ו
* These authors contributed equally
In This Article
Summary
פרוטוקול זה מתאר אסטרטגיה optogenetic כדי לווסת מיטוגן המופעל חלבון קינאז (MAPK) פעילות במהלך התמיינות תאים והתפתחות עובריים Xenopus . שיטה זו מאפשרת הפעלה הפיכה של המסלול איתות MAPK בתרבית תאים יונקים ועל אורגניזמים חיים תאיים, כמו עוברי Xenopus , עם רזולוציה מרחבית וזמנית גבוהה.
Abstract
פעילות קינאז היא קריטית עבור שפע של פונקציות הסלולר, כולל התפשטות תאים, בידול, הגירה, אפופטוזיס. במהלך התפתחות עובריים מוקדם, פעילות קינאז הוא דינמי מאוד נפוץ על פני העובר. גישות פרמקולוגיות וגנטיות משמשים בדרך כלל לחקר פעילות קינאז. למרבה הצער, זה מאתגר להשיג רזולוציה מרחבית וטמפורלית מעולה באמצעות אסטרטגיות אלה. יתר על כן, אין זה אפשרי לשלוט בפעילות קינאז באופן הפיך בתאים חיים ואורגניזמים רב תאיים. מגבלה כזו נותרה צוואר בקבוק להשגת הבנה כמותית של פעילות קינאז במהלך הפיתוח וההבחנה. עבודה זו מציגה אסטרטגיה optogenetic כי מנצל מערכת bicistronic המכיל חלבונים photoactivatable ארבידופסיס thaliana cryptochrome 2 (CRY2) ו N- מסוף מושלם של cryptochrome אינטראקציה סליל בסיסי לולאה סליל (CIBN). רברסיהפעלת BL של מיגוגן המופעל קינאז חלבון (MAPK) נתיב איתות מושגת באמצעות טרנספורמציה חלבון אור בתיווך תאים חיים. גישה זו יכולה להיות מיושמת על תרבויות תאים יונקים ועוברי חוליות לחיות. מערכת bicistronic זו יכולה להיות כללית כדי לשלוט בפעילות של קינאזות אחרות עם מנגנוני הפעלה דומים ניתן להחיל על מערכות מודל אחרים.
Introduction
גורמי גדילה מעורבים במגוון רחב של תפקודי תאים, כולל התפשטות, הבחנה, הגירה ואפופטוזיס, וכן תפקידים מרכזיים באירועים ביולוגיים רבים, כולל התפתחות עובריים, הזדקנות, והסדרת המעמד הנפשי 1 , 2 , 3 , 4 , 5 . גורמי גדילה רבים אותות דרך מפלי איתות תאיים מורכבים. אירועים אלה איתות מופעלים לעתים קרובות על ידי זירזון הפיך החלבון בצורה מסודרת במדויק 6 , 7 . לכן, הבנה של תוצאות איתות של קינאזות חלבון, אשר אחראים על זרחון חלבון, הוא חשוב ביסודו.
גורמי גדילה שונים פועלים באמצעות רשת איתות תאית שכיחה למדי, למרות שהם מעוררים את הדיסטתגובות סלולר Inct 8 , 9 . מתווכים תאיים נפוצים של קינאזות קולטן טירוזין כוללים ראס, רף, קינאז של תאית (ERK), מינוגנים המופעלים על ידי מיטרוגן (MAPK) / ERK Kinase (MEK), phosphoinositide 3-kinase (PI3K), Akt ו- phospholipase C (PLCγ) 10 , 11 . צבירת עדויות מצביעה על כך שהסימן לגיוון ולספציפיות תלוי בהסדרה המרחבית והזמנית של פעילות איתות. לדוגמה, בתאי pheochromocytoma חולדה (PC12), גורם לגדילה של גורם האפידרמיס (EGF), אשר גורם להתפשטות תאים, מפעיל באופן זמני את מסלול ERK 9 . מצד שני, גירוי עם גורם הגדילה העצבי (NGF), אשר מוביל התמיינות תאים, מפעיל את מסלול ERK באופן מתמשך 9 , 13 . ב תרבותי rעל נוירונים בהיפוקמפוס, איתות חולף על ידי גורם נוירוטרופי המופק מהמוח (BDNF) מקדם תולדה נייוריטית ראשונית, בעוד סיגנלי מתמשכת מוביל לעלייה ניורית הסתעפות 14 . במהלך התפתחות עובריים מוקדם, פעילות ERK phosphorylated הוא דינמי זמנית הוא נפוץ על פני העובר 6 . מסך גנטי עדכני במהלך אמבריוגנזה מוקדמת של Xenopus הראה ש- ERK ו- Akt cascades איתותים, שני מסלולי גורם עיקרי לצמיחה במורד הזרם, הצגת פרופילי הפעלה ספציפיים לשלב. לכן, הבנה של תוצאות איתות kinase קורא כלים שיכולים לחקור את המאפיינים המרחביים והזמן של פעילות קינאז עם רזולוציה מספקת.
גישות ניסיוניות קונבנציונאלי לחקור את הטבע הדינמי של התמרה האות במהלך הפיתוח חוסר ברזולוציה מרחבית וזמני רצוי. לדוגמה, גישות תרופתי לנצל כימית קטנהאו מולקולות ביולוגיות כדי לעורר או לדכא את התמרת האותות בתאים וברקמות. האופי המפוזר של מולקולות קטנות אלה מקשה על הגבלת פעולתן לאזור מסוים של עניין. גישות גנטיות ( למשל, transgenesis, מערכת Cre-Lox, או mutagenesis) לעתים קרובות להוביל הפעלה בלתי הפיכה או הדחקה של ביטוי הגן היעד או פעילות חלבון 16 , 17 , 18 . מערכת Tet-On / Tet-Off 19 מציעה בקרה משופרת בזמן של תעתיק גנים, אך חסרת שליטה מרחבית קפדנית משום שהיא מסתמכת על דיפוזיה של טטרציקלין. ההתפתחויות האחרונות ב dimerization חלבון המושרה כימית 20 או תמונה-unaging 21 , 22 , 23 , 24 יש לשפר מאודאד שליטה זמנית של רשתות איתות. השליטה המרחבית, לעומת זאת, נותרה מאתגרת בשל אופים המפוזר של הכימיקלים הכלובים.
גישות optogenetic המתעוררים לאחרונה, אשר לרתום את כוחו של האור כדי לשלוט אינטראקציות חלבון חלבונים, לאפשר אפנון של מסלולי איתות עם דיוק גבוהה spatiotemporal וכן הפיכות. זמן קצר לאחר הצלחתו הראשונית בשליטה על הירי העצבני 25 , 26 , 27 , אופטוגנטיקה הורחבה כדי לשלוט בתהליכים תאיים אחרים, כגון תעתיק גנים, תרגום, נדידת תאים, הבחנה ואפופטוזיס 28 , 29 , 30 , 31 , 32 , 33 , 34 . אסטרטגיה באמצעות pצמד חלבון של חלבון חלבון (CIBN), שפותח לאחרונה על מנת לשלוט בפעילות רפא 1 קינאז בתאי יונקים ועוברים של קסנופוס 35 . CRY2 נקשר ל- CIBN עם גירוי כחול-בהיר, ומרכיב החלבון CRY2 / CIBN מתנתק באופן ספונטני בחשיכה. אור כחול מרגש את cofactor CRY2, flavin adenine dinucleotide (FAD), אשר מוביל לשינוי קונפורמיטיבי CRY2 ואת מחייב לאחר מכן CIBN. מוטציות אקטיביות (W374A) ו flavin לקוי (D387A) מוטציות CRY2 ניתן לייצר באמצעות מוטציות בכיס FAD מחייב: מוטציה CRY2 W374A נקשר CIBN עצמאית של אור, ואילו מוטציה CRY2 D387A לא להיקשר CIBN תחת כחול גירוי אור 36 , 37 . המערכת האופטו-גנטית המתוארת iN זה פרוטוקול משתמש wild-type CRY2 ו CIBN כדי לגרום להפעלת טרשת נפוצה transocal בתיווך פעולה RF1 בתאים חיים. זה ידוע כי גיוס הממברנה של Raf1 משפר את פעילותה 38 . במערכת זו, מודול CIBN טנדם מעוגן לקרום הפלזמה ו- CRY2-mCherry מתמזג למסוף N של רף 35 . בהיעדר אור כחול, CRY2-mCherry-Raf1 נשאר בציטופלזמה, ו- Raf1 אינו פעיל. גירוי כחול-אור גורם לכריכה של CRY2-CIBN ומגייס את ה- RF1 לממברנת הפלזמה, שם מופעל RF1. הפעלת רף מגרה מפל איתות של RF / MEK / ERK. שני חלבונים CRY2- ו CIBN- היתוך מקודדים במערכת גנטית bicistronic. אסטרטגיה זו יכולה להיות generalized לשלוט קינאזות אחרות, כגון Akt, אשר מצב ההפעלה יכול גם להיות מופעל על ידי טרנסלוקציה חלבון בתאים 39 . עבודה זו מציגה פרוטוקולים מפורטים ליישום אסטרטגיה זו optogenetic ב cultu תא יונקיםרס ואורגניזמים תאיים.
Protocol
מחקר בבעלי חיים נערך על פי הנחיות שנקבעו על ידי טיפול בבעלי חיים מוסדיים של אילינוי ושימוש ועדת (IACUC) ואת אוניברסיטת אילינוי המחלקה של בעלי חיים משאבים (DAR).
1. אינדוקציה Optogenetic של לוקליזציה חלבון ב BHK21 תרבית תאים יונקים
הערה: צעדים 1.1-1.3 לספק שיטה להרכיב תא תרבות תא הדמיה עם מטרות הגדלה גבוהה ( למשל, 63X או 100X), אשר בדרך כלל יש מרחקים עבודה קצר. יעדים אלה דורשים coverslip זכוכית דקה ( למשל, # 1.5, 170 עובי מיקרומטר) כמו מצע הדמיה. לחלופין, זכוכית בתחתית לתא התרבות / שקופית ניתן להשתמש. במקרה כזה, צעדים 1.1-1.3 ניתן לדלג.
- ניקוי זכוכית coverslips
- מקום 30 coverslips זכוכית בעלים coverslip.
- בתוך כוס פלסטיק 600 מ"ל, מוסיפים 20 גרם של דטרגנט ו 400 מ"ל של מי ברז חמים. פלאקאת הכוס על מגש מגנטי ומערבבים עד שכל חומר הניקוי מתמוסס. הסר את הקצף באמצעות נייר טישו.
- השתמש בתחתית של 100 מ"מ x 15 מ"מ צלחת פטרי כמו spacer מן המערב בר וכמשטח עבור מחזיק coverslip. כדי לשמור על זרימה נאותה במהלך תהליך הניקוי, לעשות 3-4 חורים ניקוז בצלחת פטרי.
- כדי להפוך את חורי ניקוז, לחמם ברזל הלחמה ולשרוף דרך פלסטיק במכסה המנוע.
זהירות: אין לגעת בברזל המחומם בידיים חשופות. - מניחים את בעל coverslip על צלחת פטרי בכוס. מערבבים במשך 2 שעות עד הלילה בטמפרטורת החדר.
- לשטוף שלוש פעמים עם 500 מ"ל של מים DI במארז 1000 מ"ל. ניתן להשתמש מחדש בפתרון הדטרגנט.
- לאסוף coverslips בודדים באמצעות מלקחיים. לטבול את coverslips 190-הוכחה (95%) אתנול במשך 1 דקות.
- יבש את coverslips בתוך ברדס סטרילית. חנות coverslips במיכל פלסטיק סטרילי עד לשימוש.
- ביצוע פולי- L- ליזין (PLL) מצופה coverslips
- בצע 0.1 חיץ borate B על ידי המסת 1.24 גרם של חומצה בורית ו 1.9 גרם של tetraborate דיסול ב 400 מ"ל של מים. התאם את ה- pH ל 8.5 עם 10 M NaOH.
- ממיסים PLL במאגר borate לריכוז סופי של 1 מ"ג / מ"ל. Aliquot הפתרון PLL ב 50 מ"ל צינורות צנטריפוגה סטרילית.
- הוסף 50 מ"ל של תמיסת ציפוי PLL לצלחת 10 ס"מ רקמה תרבות. מניחים את המנה 37 ° C חממה כדי לחמם את הפתרון PLL.
- פתח את מיכל coverslip (שלב 1.1.8) ברדס סטרילית.
- לטבול את coverslips ניקה אחד אחד בפתרון מראש PLL מחומם בצלחת תרבות רקמות. הימנע היווצרות בועה כדי לטבול את כל המשטחים.
- מניחים את המנה עם coverslip ב 37 ° C CO 2 חממה לילה. להוציא את כל coverslip ולמקם אותו מחזיק coverslip סטרילית. לשטוף שלוש פעמים עם מים ultrapure autoclaved (18.2 MΩ & #183; התנגדות ס"מ) בכוס 1,000 מ"ל במכסה סטרילי.
- יבש coverslips ב מחזיק coverslip במכסה המנוע סטרילי. חנות coverslips במיכל פלסטיק סטרילי עד לשימוש.
- הרכבה polydimethylsiloxane (PDMS) תא בתרבות תאים
- במיכל פלסטיק, לשקול 40 גרם של בסיס PDMS ולהוסיף 4 גרם של סוכן ריפוי להשיג 10: 1 w / w יחס של PDMS סוכן ריפוי. מערבבים את PDMS / ריפוי סוכן על ידי ערבוב עם קצה פיפטה עבור 2 דקות. לחלופין, להשתמש במערבל צנטריפוגלי.
- השתמש כוס עם קוטר 4 אינץ 'כתבנית כדי להפוך את בעל עם חתיכת רדיד אלומיניום. מניחים את מחזיק רדיד אלומיניום בצלחת פטרי 15 ס"מ. מניחים פרוסות סיליקון 4 אינץ 'בתוך זה מחזיק רדיד אלומיניום.
- יוצקים את הפתרון PDMS מעורב לתוך בעל. השתמש מוט זכוכית דקה לגעת בקצה של רקיק בעדינות. הסר כל בועות אוויר לכודים מתחת לפרוסות.
- הכנס את צלחת פטרי 15 ס"מ לתוך chambe ואקוםR כדי degas הפתרון PDMS. המשך degassing במשך כ 20 דקות, עד לא בועות נוצרים. בינתיים, מחממים תנור הסעה ל 65 מעלות צלזיוס.
- מניחים בזהירות את צלחת פטרי 15 ס"מ לתוך התנור. דגירה של 2 שעות.
- מוציאים את הצלחת מהתנור. השתמש מוט זכוכית לגעת בעדינות את קצה PDMS ו להבטיח crosslinking מלאה. אם לא, דגירה ארוכה יותר, עד PDMS הוא מוצק ולא דביק.
- הסר את רדיד אלומיניום מהצלחת לקלף אותו פרוסות סיליקון. החל מן הקצה, בזהירות לקלף את PDMS נרפא את פרוסות סיליקון.
זהירות: הימנע החלת כוח רב מדי, כפי שהוא יכול לשבור את רקיק. - לחתוך את PDMS לתוך 20 מ"מ x 30 מ"מ מלבני חתיכות כדי להתאים את 24 מ"מ x 40 מ"מ coverslip עם סכין גילוח.
- השתמש להב בצורת אזמל לחתוך פתח מלבני של 10 מ"מ x 20 מ"מ ממרכז כל חתיכת PDMS.
- נקה את חדרי PDMS באמצעות פרוטוקול דטרגנט המתואר בשלב 1.1.
- יבש את חדרי PDMS במכסה נקי. מניחים את התאים היבשים במיכל autoclavable מכוסה בנייר אלומיניום.
- החיטוי מיכל באמצעות כוח הכבידה (121 מעלות צלזיוס, 15 psi) למשך 30 דקות ולאחסן את המיכל בטמפרטורת החדר.
- BHK21 תאים culturing ו transfection
- הפוך 500 מ"ל של בינוני לתרבות תאים BHK21 על ידי ערבוב 445 מ"ל של DMEM עם 50 מ"ל של FBS ו 5 מ"ל של 100 × פן סטר גלוטמין (10,000 U / מ"ל פניצילין, 10 מ"ג / מ"ל סטרפטומיצין, ו 29.2 מ"ג / מ"ל L - גלוטמין). ודא כי הריכוז הסופי של FBS הוא 10%.
- Aliquot 10 מ"ל של HEPES שאינם CO 2- בינוני עצמאית עבור הדמיה תא חי.
הערה: CO 2- בינונית עצמאית תומך בתא הגידול ללא חממה CO 2 והוא אידיאלי עבור תאים הדמיה תחת תנאים אטמוספריים. לקבלת מידע מפורט, עיין ברשימת החומרים. בנוסף לקו התא BHK21, סוגים אחרים של תאים יונקים צריך גם לתתתוצאות דומות. - להרכיב מכשיר תרבות התא על ידי הצבת אחד autoclaved, סטרילי PDMS קאמרית (שלב 1.3) על coverslip סטרילי PLL מצופה (שלב 1.2) במכסה סטרילי.
- מניחים כל מכשיר בצלחת פטרי סטרילית 60 מ"מ.
- השתמש 0.5 מ"ל של טריפסין 0.25% לנתק את התאים מבאר אחת של 12 גם צלחת רקמה תרבות. ספירת צפיפות התאים עם hemocytometer. צלחת 20,000 תאים BHK21 בתא (כ - 10,000 תאים / ס"מ 2 ) עם 200 μL של המדיום תרבית תאים.
- הכן פלסמיד DNA עם ערכת הכנה פלסמיד (ראה את רשימת החומרים).
הערה: העיצוב והפונקציונליות של CRY2-mCherry-Raf1-2A-2CIBN-GFP-CaaX מוצגים בתרשים 1A - 1B . - עשרים וארבע (24) h לאחר ציפוי התא, transfect התאים עם 50-100 ng של CRY2-mCherry-Raf1-2A-2CIBN-GFP-CaaX פלסמיד, על פי פרוטוקול של היצרן.
- שלוש (3) שעות לאחר transfection, לשנות את mאדיום μL 200 של המדיום תרבות טרי (שלב 1.4.1) ולאפשר לתאים לשחזר לילה על ידי דוגרים את התרבות ב 37 מעלות צלזיוס, 5% CO 2 חממה.
- פלואורסצנטי לחיות תא הדמיה
- הפעל מחשב, מקור אור, מיקרוסקופ ומצלמה. השתמש מיקרוסקופ פלואורסצנטי confocal (מצויד מטרה 100X שמן טבילה) עבור שאר הפרוטוקול.
הערה: מיקרוסקופ הפוך epifluorescence יכול לשמש גם. - החלף את המדיום תרבית תאים עם 200 μL של CO 2 בינוני בינונית.
- הגדרת פרוטוקול רכישת נתונים לפני הצבת התאים על המיקרוסקופ. השתמש 488 ננומטר עירור FITC ערוץ לגירוי optogenetic. השתמש 561 ננומטר עירור עירור TRITC לאתר תאים transfected ולעקוב אחר לוקליזציה הסלולר של חלבון שכותרתו mCherry.
- הגדר את הרווח של ערוצי FITC ו TRITC כמו 120 ו 200, בהתאמה. השתמש זמן מגורים פיקסל של 2.21; S וגודל של 512 x 512 פיקסלים.
- מדוד את עוצמת האור 488 ננומטר על ידי הנחת מד צריכת חשמל קרוב לחלון המטרה. כוח כולל של 2 μW (כ -10,000 W / cm 2 במוקד) מספיק כדי לעורר CIBN-CRY2PHR העמותה.
- הגדרת רכישת חותמת עם מרווח 5 s ו סך זמן הרכישה של 2 דקות.
- החל המתאים חומר ההתאמה לאינדקס (שמן טבילה) על החלון אובייקטיבי. השתמש במצב בניגוד שלב להתמקד התאים על משטח coverslip.
- אתר תא transfected תחת 561 ננומטר אור על ידי הזזת הבמה מיקרוסקופ.
זהירות: הימנע משימוש באור כחול במהלך שלב זה, שכן הוא יפעיל את הקשר בין חלבונים photoactivatable. - לאחר תא transfected נמצא, ליזום רכישת נתונים.
הערה: תאים transfected בהצלחה צריך להראות הקרינה בשני FITC (מ 2CIBN-GFP-CaaX) ו TRITC (מ CRY2-mCherry-Raf1) ערוצים ( איור 1C-D ). - הקלט סדרה של זמן חותמת תמונות בשני ערוצי FITC ו TRITC ( איור 1D ).
- כדי לבדוק את הדיסוציאציה הספונטנית של CRY2-CIBN מורכבות חלבון, להקליט עוד חותמת עם ערוץ Txred לבד למשך 20 דקות, עם מרווח של 30 שניות.
- שמור את הזמן חותמת התמונה לניתוח נתונים.
- הפעל מחשב, מקור אור, מיקרוסקופ ומצלמה. השתמש מיקרוסקופ פלואורסצנטי confocal (מצויד מטרה 100X שמן טבילה) עבור שאר הפרוטוקול.
- ניתוח תמונה
- פתח את התמונות עם תוכנת ניתוח תמונות שיכולים לחלץ את האינטנסיביות מתמונה 40 .
- בחרו שתי תמונות מייצגות: אחת לפני החשיפה לאור כחול וחשיפה אחרת לאור כחול-כחול.
- צייר קו על פני התא פורש על הרקע, קרום הפלזמה, ואת הציטופלסמה. פרויקט את העוצמה לאורך הקו הזה. שמור את הערכים ואת העלילה אותם כדי להשוות את ההבדל לפני ואחרי החשיפה לאור ( איור 1D ).
- כדי לנתח את הקינטיקה של התאחדות חלבון, selecT נציג אחד של האזור עניין (ROI) של קרום פלזמה, החזר ROI אחד בציטופלסמה, ואת ההחזר על ההשקעה אחד ברקע.
- לרכוש את עוצמות הממוצע של קרום הפלזמה (אני PM ), הציטופלסמה (I CYT ), ואת הרקע (אני BKD ) עבור מחסנית התמונה.
- לחשב את היחס בין עוצמת הממברנה / cytosolic עבור כל תמונה באמצעות המשוואה הבאה:
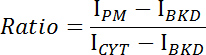
- מגרש היחס לעומת הזמן ולקבוע את קינטיקה מחייב או דיסוציאציה של CRY2-CIBN ( איור 1E- F ).
2. בניית מערך LED עבור גירוי אור ארוך טווח בחממה CO 2
הערה: סכמטי הכולל של תוכנית הניסוי מוצג באיור 2 א .
- הפוך את מערך LED על ידי הוספת 12 נוריות כחולות לתוך שתי סירות הלחםDs וחיבור נגדים מגבילים הנוכחי.
הערה: באמצעות ספק כוח של 30 וולט, ניתן לחבר ארבע נוריות בסדרה, וניתן לשלוט על בהירותן על ידי נגד מגביל. - מניחים את לוחות הלחם לתוך קופסת אלומיניום.
הערה: גובה תיבת אלומיניום צריך להיות 2 פנימה גובה זה הוא אופטימלי להארת צלחת 12-רקמה תרבות, כי הגודל של נקודת אור מתגלגל זהה לזה של באר אחת. - השתמש בשני חוטי מתכת כדי להתחבר לאספקת החשמל. ודא כי אורך חוט מספיק כאשר תיבת האור ממוקם בתוך חממה CO 2 .
- השתמש מפזר אור שקוף כמו כיסוי של תיבת האור ( איור 2 ג ).
- כייל את יציאת החשמל של כל LED בטווח של תשומות מתח. השתמש בכוח של 0.2 mW / cm 2 עבור 24 שעות PC12 התא הבדל assay. השתמש בכוח של 5 mW / cm 2 עבור עוברי Xenopus חיים או explantsמבחני.
3. אינדוקציה Optogenetic של PC12 התא התמיינות
- Cell culturing ו transfection
- הפוך 500 מ"ל של בינוני עבור pheochromocytoma חולדה (PC12) תרבות התא על ידי ערבוב 407.5 מ"ל של F12K עם 75 מ"ל של סרום סוס + 12.5 מ"ל של FBS + 5 מ"ל של 100 × Penn-Strep- גלוטמין (ריכוז סופי של הסרום סוס FBS : 15% ו -2.5%, בהתאמה). הפוך בינוני נמוך בסרום על ידי ערבוב 1 נפח בינוני מלא ב 99 כרכים של מדיום F12K.
- פלייט PC12 תאים בצלחת 12 גם בצפיפות של 300,000 תאים / טוב או 75,000 תאים / ס"מ 2 .
- Transfect התאים עם CRY2-mCherry-Raf1-2A-2CIBN-GFP-CaaX 24 שעות לאחר ציפוי התא (בדומה צעד 1.4.7). השתמש 1.2 מיקרוגרם של DNA עבור כל טוב. אפשר לתאים להתאושש לילה במדיום תרבות 37 ° C חממה בתוספת 5% CO 2 . בדוק את יעילות transfection 16 שעות לאחר transfection.
הערה: transfection 30-50% effIciency צריך להיות מושגת על מנת להבטיח ספירת תא מספיק. - שינוי בינוני בינוני נמוך 24 שעות לאחר transfection. השתמש 1 מ"ל של מדיום לכל טוב של צלחת 12 גם.
- אור המושרה PC12 התמיינות תאים
- מניחים את מערך LED בחממה CO 2 . חבר אותו לאספקת החשמל באמצעות זוג חוטים. הגדר את העוצמה של LED ל 0.2 mW / cm 2 . מניחים את צלחת 12 היטב המכיל תאים transfected PC12 (שלב 3.1.3) על החלון של מערך LED. החל תאורה אור רציפה במשך 24 שעות 37 ° C חממה בתוספת 5% CO 2 .
- פלואורסצנטי לחיות הדמיה תא
- בצע את שלבים 1.5.1-1.5.4 עבור מיקרוסקופ הכנה המדגם.
- הגדר רכישה של נתוני מצב יחיד. השתמש 200 ms עבור שניהם GFP ערוץ Txred.
- לכידת תמונות של תאים transfected בשני ערוצי GFP ו Txred. שיא בערך 200 celלכל תנאי. שמור את הקבצים לניתוח נתונים.
- ניתוח תמונה
- ספירת אחוז תאים מובחנים על המספר הכולל של תאים transfected.
- השתמש בכל תא ספירת מודול בתוכנת ניתוח התמונה לספור את התאים.
- ספור את התאים המבודדים באופן ידני.
הערה: תאים מובחנים מוגדרים כאלו שבהם לפחות neurite אחד הוא ניכר יותר מאשר גוף התא ( איור 3 א -3 ב ). - חזור על שלב 3.4.3 עם תאים לא מובחנים.
- חישוב יחס בידול באמצעות המשוואה הבאה:
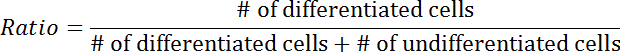
4. Optogenetic שליטה של פעילות קינאז Xenopus עוברי
- הכנת מאגרים
- הכן 1 L של 1x מארק השתנה הצלצול (MMR): 100 מ"מNaCl, 2 KM מ"מ, 1 מ"מ MgCl 2 , 2 מ"מ CaCl 2 , ו 5 מ"מ HEPES; PH 7.5.
- הכנת mRNA
- לעכל את CRY2-mCherry-Raf1-2A-2CIBN-GFP-CaaX DNA פלסמיד (2 מיקרוגרם) עם μL 1 של ApaI (50 יחידה) ב 37 מעלות צלזיוס למשך 2 שעות.
- דליפה ליניארית לזרז μL 60 של אתנול 100%. ספין ב 12,000 × גרם במשך 5 דקות בטמפרטורת החדר כדי pellet ה- DNA. בזהירות להסיר את supernatant ולשטוף את גלולה שוב עם 60 μL של 70% אתנול. ספין ב 12,000 × גרם עוד 5 דקות בטמפרטורת החדר. בזהירות להסיר את supernatant ו resuspend גלולה DNA ב μL 6 של RNase ללא H 2 O.
- בצע שעתוק במבחנה על ידי דוגרים 1 מיקרוגרם של DNA לינארית עם μL 2 של פולימרז SP6 RNA מסופק, תערובת ribonucleotide (10 מ"מ ATP, CTP, ו UTP, 2 GTP מ"מ, ו 8 מ"מ אנלוגי שווי), ו 20 μL של nuclease- מים חינם בטמפרטורת החדר למשך 1 שעות.
- הסר את Dתבנית NA על ידי עיכול DNase אני ב 37 מעלות צלזיוס למשך לפחות 15 דקות לטהר את RNA מסונתז באמצעות טור ספיקה סיליקה קרום.
- עצור את התגובה להאיץ את RNA ידי הוספת 30 μL של מים nuclease ללא 30 μL 30 של תמיסת משקעים LiCl. מערבבים היטב צינה במשך 30 דקות ב -20 מעלות צלזיוס.
- צנטריפוגה ב 4 מעלות צלזיוס למשך 15 דקות ב 12,000 × גרם כדי גלולה RNA.
- הסר בזהירות את supernatant. לשטוף את רנ"א פעם עם 1 מ"ל של אתנול 70% ו resuspend ב 40 μL של מים ללא nuclease.
- טען 40 μL של רנ"א בעמודה ספין. צנטריפוגה ב 4 מעלות צלזיוס למשך דקה 1 ב 12,000 × גרם כדי להסיר את נוקליאוטידים מאוגדים וכובעים.
- קציר קסנופוס עוברי
- בצע את הפרוטוקול המתואר על ידי Sive et al. 41 כדי לקבל את העוברים קסנופוס .
- MRNA microinjection
- לְפַבְּרֵקאת המחטים עבור microinjection על ידי משיכת נימי זכוכית עם חולץ נימי. הגדר את משיכת התוכנית: חום = 355, למשוך כוח = 80, מהירות = 50, שעה = 100.
- הסר את המעיל ג 'לי מן העוברים על ידי טיפול בהם עם ציסטאין 3% (בדילול של MMR 0.2x).
- מעבירים את העוברים כדי polysucrose 3% ו 0.5x פתרון MMR עבור microinjection. להזריק 500 pg ל 1 ng של CRY2-mCherry-Raf1-2A-2CIBN-GFP-CaaX RNA לתוך כל עוברי.
הערה: הזרקה של מינון גבוה מ 1 ng תוצאות ההפעלה כחול עצמאית אור של איתות MAPK.
- גירוי אופטוגנטי של פעילות קינאז
- תרבות microinjected עוברים ב polysucrose 3%, תמיסת MMR 0.5x בטמפרטורת החדר עד שהם מגיעים לשלב gastrula באמצע (שלב 12). ואז, תרבות העוברים בפתרון MMR 0.2x.
- מעבירים את העוברים או explants לצלחת 12 גם. מניחים את הצלחת 12-היטב על מערך LED בנוי הביתה (שלב 2.5) עבור כחול lighT (475 ננומטר) טיפול.
- מניחים מראה על החלק העליון של צלחת 12 היטב כדי להבטיח תאורה מלאה אור כחול של העוברים או explants.
- כוונן את העוצמה של האור הכחול ל 5 mW / cm 2 .
הערה: טיפול באור כחול יכול להתבצע בכל זמן הרצוי, ב 3% polysucrose, פתרון MMR 0.5x, או תמיסת MMR 0.2x. - קציר את העוברים בכל זמן הרצוי. השתמש בהם עבור ניתוח היסטולוגית, כתם המערבי, או ביטוי גנים.
תוצאות
ביטוי Ratiometric של זוגות חלבון Photoactivatable: איור 1A מראה את העיצוב של מבנה optogenetic bicistronic, CRY2-mCherry-Raf1-P2A-CIBN-CIBN-GFP-CaaX (המכונה CRY2-2A-2CIBN), המבוססת על teschovirum חזירי- 1 2A (P2A) פפטיד, אשר מראה את היעילות הגבוהה ביותר ריבוזום-דילוג בין שורות תאים יונקים
Discussion
בעת בניית תיבת האור, את הכוח של נוריות LED בודדות יש למדוד. בהתבסס על ניסיון קודם, תפוקת החשמל יכולה להשתנות בין נוריות LED בודדות עקב שונות בייצור. בחר קבוצה של נוריות כי יש פלט כוח בתוך 10% אחד מהשני. מספר נוריות, הנגד המגביל הנוכחי, ואת קלט הכוח יכול להיות שונה עבור סוגים ?...
Disclosures
המחברים מצהירים כי אין להם אינטרסים פיננסיים מתחרים.
Acknowledgements
עבודה זו נתמכה על ידי אוניברסיטת אילינוי ב Urbana-Champaign (UIUC) ואת המכונים הלאומיים לבריאות (NIGMS R01GM111816).
Materials
| Name | Company | Catalog Number | Comments |
| Glass coverslip | VWR | 48393 230 | Substrate for live cell imaging |
| Coverslip holder | Newcomer Supply | 6817B | Holder for coverslips |
| Detergent | ThermoFisher | 16 000 104 | For cleaning coverslips |
| Boric acid | Sigma-Aldrich | B6768-500G | For making PLL buffer |
| Disodium tetraborate | Sigma-Aldrich | 71996-250G | For making PLL buffer |
| Plastic beaker | Nalgene | 1201-1000 | For cleaning coverslips |
| Sodium hydroxide | Sigma-Aldrich | 221465-2.5KG | For adjust pH |
| Poly-L-lysine hydrobromide | Sigma-Aldrich | P1274-500MG | For coating coverslip |
| Diethylpyrocarbonate (DEPC)-Treated Water | ThermoFisher Scientific | 750024 | For DNA preparation |
| Cover Glass Forceps | Ted Pella | 5645 | Cover glass handling |
| Tissue cutlure dish | Thermofisher | 12565321 | Cell culture dish |
| Sterile centrifuge tubes | ThermoFisher | 12-565-271 | Buffer storage |
| Transfection Reagent | ThermoFisher | R0534 | Transfection |
| CO2-independent medium | ThermoFisher | 18045088 | For live cell imaging |
| Polydimethylsiloxane (PDMS) | Ellsworth Adhesives | 184 SIL ELAST KIT 0.5KG | Form make cell chamber |
| Plasmid Maxiprep kit | Qiagen | 12965 | Plasmid preparation |
| DMEM medium | ThermoFisher | 11965-084 | Cell culturing medium component |
| F12K medium | ThermoFisher | 21127022 | Cell culturing medium component |
| Horse serum | ThermoFisher | 16050122 | Cell culturing medium component |
| Fetal Bovine Serum | Signa-Aldrich | 12303C-500 mL | Cell culturing medium component |
| Penicillin-Streptomycin-Glutamine | ThermoFisher | 10378016 | Cell culturing medium component |
| Trypsin (0.25%), phenol red | ThermoFisher Scientific | 15050065 | For mammalian cell dissociation |
| Agarose | Fisher Scientific | BP1356-100 | For DNA preparation |
| Ficoll PM400 | GE Heathcare Life Sciences | 17-5442-02 | For embryo buffer |
| L-Cysteine hydrochloride monohydrate | Sigma-Aldrich | 1.02839.0025 | Oocyte preparation |
| ApaI | ThermoFisher | FD1414 | For linearization of plasmids |
| Dnase I | ThermoFisher | AM2222 | For removing DNA template in the in vitro transcription assay |
| Index-match materials (immersion oil) | Thorlabs | MOIL-20LN | For matching the index between sample substrate and objective |
| Blue LED | Adafruit | 301 | Light source for optogenetic stimulation |
| Resistor kit | Amazon | EPC-103 | current-limiting resistor |
| Aluminum boxes | BUD Industries | AC-401 | light box |
| BreadBoard | Jekewin | 837654333686 | For making LED array |
| Hook up Wire | Electronix Express | 27WK22SLD25 | For making LED array |
| Relay Module | Jbtek | SRD-05VDC-SL-C | For intermittent light control |
| DC Power Supply | TMS | DCPowerSupply-LW-(PS-305D) | Power supply for LED |
| Silicon Power Head | Thorlabs | S121C | For light intensity measurement |
| Power meter | Thorlabs | PM100D | For light intensity measurement |
| Microscope | Leica Biosystems | DMI8 | For live cell imaging |
| BioSafety Cabinet | ThermoFisher | 1300 Series A2 | For mammalian cell handling |
| CO2 incubator | ThermoFisher | Isotemp | For mammalian cell culturing |
| Stereo microscope | Leica | M60 | For embryo micro-manipulation |
| Microinjector | Narishige | IM300 | For embryo microinjection |
| Micropipette puller | Sutter Instruments | P87 | Needle puller |
| in vitro transcription kit | ThermoFisher | AM1340 | For in vitro transcription. The kit includes nuclease-free water, SP6 RNA Polymerase, ribonucleotide mixture, cap analog, lithium choride precipitation solution, and spin column |
| RNA purfication kit | Qiagen | 74104 | Silica-membrane spin column for purification of synthesized RNA |
| Convection oven | MTI corporation | EQ-DHG-9015 | PDMS curing |
| Centrifugal mixer and teflon container | THINKY | AR310 | For mixing PDMS |
| Silicon wafer | UniversityWafer | 452 | Base for making PDMS devices |
| Blade | Techni Edge | 01-801 | For cutting PDMS |
| Capillary glass | Sutter Instruments | BF100-58-10 | For fabrication of injecting needles. |
References
- Schlessinger, J., Ullrich, A. Growth factor signaling by receptor tyrosine kinases. Neuron. 9 (3), 383-391 (1992).
- Thisse, B., Thisse, C. Functions and regulations of fibroblast growth factor signaling during embryonic development. Dev Biol. 287 (2), 390-402 (2005).
- Perrimon, N., Pitsouli, C., Shilo, B. Z. Signaling mechanisms controlling cell fate and embryonic patterning. Cold Spring Harb Perspect Biol. 4 (8), a005975 (2012).
- Salles, F. H., et al. Mental disorders, functional impairment, and nerve growth factor. Psychol Res Behav Manag. 10, 9-15 (2017).
- Basson, M. A. Signaling in cell differentiation and morphogenesis. Cold Spring Harb Perspect Biol. 4 (6), (2012).
- Schohl, A., Fagotto, F. beta-catenin, MAPK and Smad signaling during early Xenopus development. Development. 129 (1), 37-52 (2002).
- Zhang, S. W., Li, J. J., Lea, R., Amaya, E., Dorey, K. A Functional Genome-Wide In Vivo Screen Identifies New Regulators of Signalling Pathways during Early Xenopus Embryogenesis. PloS one. 8 (11), e79469 (2013).
- Sweeney, C., et al. Growth factor-specific signaling pathway stimulation and gene expression mediated by ErbB receptors. J Biol Chem. 276 (25), 22685-22698 (2001).
- Marshall, C. J. Specificity of receptor tyrosine kinase signaling: transient versus sustained extracellular signal-regulated kinase activation. Cell. 80 (2), 179-185 (1995).
- Vandergeer, P., Hunter, T., Lindberg, R. A. Receptor Protein-Tyrosine Kinases and Their Signal-Transduction Pathways. Annu Rev Cell Biol. 10, 251-337 (1994).
- Lemmon, M. A., Schlessinger, J. Cell signaling by receptor tyrosine kinases. Cell. 141 (7), 1117-1134 (2010).
- Hunter, T. Signaling--2000 and beyond. Cell. 100 (1), 113-127 (2000).
- Qiu, M. S., Green, S. H. PC12 cell neuronal differentiation is associated with prolonged p21ras activity and consequent prolonged ERK activity. Neuron. 9 (4), 705-717 (1992).
- Ji, Y., et al. Acute and gradual increases in BDNF concentration elicit distinct signaling and functions in neurons. Nat Neurosci. 13 (3), 302-309 (2010).
- Luby-Phelps, K. Cytoarchitecture and physical properties of cytoplasm: volume, viscosity, diffusion, intracellular surface area. Int Rev Cytol. 192, 189-221 (2000).
- Sauer, B. Inducible gene targeting in mice using the Cre/lox system. Methods-a Companion to Methods in Enzymology. 14 (4), 381-392 (1998).
- Ling, M. M., Robinson, B. H. Approaches to DNA mutagenesis: an overview. Anal Biochem. 254 (2), 157-178 (1997).
- Gama Sosa, M. A., De Gasperi, R., Elder, G. A. Animal transgenesis: an overview. Brain Struct Funct. 214 (2-3), 91-109 (2010).
- Wanka, F., et al. Tet-on, or Tet-off, that is the question: Advanced conditional gene expression in Aspergillus. Fungal Genet Biol. 89, 72-83 (2016).
- Karginov, A. V., Ding, F., Kota, P., Dokholyan, N. V., Hahn, K. M. Engineered allosteric activation of kinases in living cells. Nat Biotechnol. 28 (7), 743-747 (2010).
- Liu, Q. Y., Deiters, A. Optochemical Control of Deoxyoligonucleotide Function via a Nucleobase-Caging Approach. Accounts of Chemical Research. 47 (1), 45-55 (2014).
- Arbely, E., Torres-Kolbus, J., Deiters, A., Chin, J. W. Photocontrol of tyrosine phosphorylation in mammalian cells via genetic encoding of photocaged tyrosine. J Am Chem Soc. 134 (29), 11912-11915 (2012).
- Gautier, A., Deiters, A., Chin, J. W. Light-activated kinases enable temporal dissection of signaling networks in living cells. J Am Chem Soc. 133 (7), 2124-2127 (2011).
- Nguyen, D. P., et al. Genetic Encoding of Photocaged Cysteine Allows Photoactivation of TEV Protease in Live Mammalian Cells. J Am Chem Soc. 136 (6), 2240-2243 (2014).
- Deisseroth, K. Optogenetics. Nat Methods. 8 (1), 26-29 (2011).
- Boyden, E. S., Zhang, F., Bamberg, E., Nagel, G., Deisseroth, K. Millisecond-timescale, genetically targeted optical control of neural activity. Nat Neurosci. 8 (9), 1263-1268 (2005).
- Banghart, M., Borges, K., Isacoff, E., Trauner, D., Kramer, R. H. Light-activated ion channels for remote control of neuronal firing. Nat Neurosci. 7 (12), 1381-1386 (2004).
- Zhang, K., Cui, B. Optogenetic control of intracellular signaling pathways. Trends in Biotechnology. 33 (2), 92-100 (2015).
- Tischer, D., Weiner, O. D. Illuminating cell signalling with optogenetic tools. Nat Rev Mol Cell Bio. 15 (8), 551-558 (2014).
- Kim, B., Lin, M. Z. Optobiology: optical control of biological processes via protein engineering. Biochemical Society Transactions. 41 (5), 1183-1188 (2013).
- Tucker, C. L. Manipulating cellular processes using optical control of protein-protein interactions. Prog Brain Res. 196, 95-117 (2012).
- Toettcher, J. E., Gong, D. Q., Lim, W. A., Weiner, O. D. Light Control of Plasma Membrane Recruitment Using the Phy-Pif System. Method Enzymol. 497, 409-423 (2011).
- Zoltowski, B. D., Gardner, K. H. Tripping the light fantastic: blue-light photoreceptors as examples of environmentally modulated protein-protein interactions. Biochemistry. 50 (1), 4-16 (2011).
- Kennedy, M. J., et al. Rapid blue-light-mediated induction of protein interactions in living cells. Nat Methods. 7 (12), 973-975 (2010).
- Krishnamurthy, V. V., et al. Reversible optogenetic control of kinase activity during differentiation and embryonic development. Development. 143 (21), 4085-4094 (2016).
- Liu, H., et al. Photoexcited CRY2 Interacts with CIB1 to Regulate Transcription and Floral Initiation in Arabidopsis. Science. 322 (5907), 1535-1539 (2008).
- Li, X., et al. Arabidopsis cryptochrome 2 (CRY2) functions by the photoactivation mechanism distinct from the tryptophan (trp) triad-dependent photoreduction. Proc Natl Acad Sci U S A. 108 (51), 20844-20849 (2011).
- Leevers, S. J., Paterson, H. F., Marshall, C. J. Requirement for Ras in Raf Activation Is Overcome by Targeting Raf to the Plasma-Membrane. Nature. 369 (6479), 411-414 (1994).
- Kohn, A. D., Takeuchi, F., Roth, R. A. Akt, a pleckstrin homology domain containing kinase, is activated primarily by phosphorylation. J Biol Chem. 271 (36), 21920-21926 (1996).
- Schindelin, J., et al. Fiji: an open-source platform for biological-image analysis. Nature Methods. 9 (7), 676-682 (2012).
- Sive, H. L., Grainger, R. M., Harland, R. M. . Early Development of Xenopus laevis: A Laboratory Manual. , (2000).
- Kim, J. H., et al. High cleavage efficiency of a 2A peptide derived from porcine teschovirus-1 in human cell lines, zebrafish and mice. PloS one. 6 (4), e18556 (2011).
- Ishimura, A., et al. Oncogenic Met receptor induces ectopic structures in Xenopus embryos. Oncogene. 25 (31), 4286-4299 (2006).
- Zhang, K., et al. Light-Mediated Kinetic Control Reveals the Temporal Effect of the Raf/MEK/ERK Pathway in PC12 Cell Neurite Outgrowth. PloS one. 9 (3), e92917 (2014).
- Mohanty, S. K., Lakshminarayananan, V. Optical Techniques in Optogenetics. J Mod Opt. 62 (12), 949-970 (2015).
- Taslimi, A., et al. Optimized second-generation CRY2-CIB dimerizers and photoactivatable Cre recombinase. Nature chemical biology. 12 (6), 425-430 (2016).
- Kawano, F., Suzuki, H., Furuya, A., Sato, M. Engineered pairs of distinct photoswitches for optogenetic control of cellular proteins. Nat Commun. 6, 6256 (2015).
- Wang, H., et al. LOVTRAP: an optogenetic system for photoinduced protein dissociation. Nat Methods. 13 (9), 755-758 (2016).
- Chang, K. Y., et al. Light-inducible receptor tyrosine kinases that regulate neurotrophin signalling. Nat Commun. 5, 4057 (2014).
- Boulina, M., Samarajeewa, H., Baker, J. D., Kim, M. D., Chiba, A. Live imaging of multicolor-labeled cells in Drosophila. Development. 140 (7), 1605-1613 (2013).
- Liu, H., Gomez, G., Lin, S., Lin, C. Optogenetic control of transcription in zebrafish. PLoS One. 7 (11), e50738 (2012).
- Buckley, C. E., et al. Reversible Optogenetic Control of Subcellular Protein Localization in a Live Vertebrate Embryo. Dev Cell. 36 (1), 117-126 (2016).
- Motta-Mena, L. B., et al. An optogenetic gene expression system with rapid activation and deactivation kinetics. Nature chemical biology. 10 (3), 196-202 (2014).
- Beyer, H. M., et al. Red Light-Regulated Reversible Nuclear Localization of Proteins in Mammalian Cells and Zebrafish. ACS Synth Biol. 4 (9), 951-958 (2015).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved