Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Modulation réversible médiée par la lumière du pathogène protéinique Kinase activé par Mitogen pendant la différenciation cellulaire et
* Ces auteurs ont contribué à parts égales
Dans cet article
Résumé
Ce protocole décrit une stratégie opto-génétique pour moduler l'activité de la protéine kinase activée par mitogène (MAPK) pendant la différenciation cellulaire et le développement embryonnaire de Xenopus . Cette méthode permet l'activation réversible de la voie de signalisation MAPK dans la culture de cellules de mammifères et dans les organismes vivants multicellulaires, comme les embryons de Xenopus , avec une résolution spatiale et temporelle élevée.
Résumé
L'activité de la kinase est cruciale pour une pléthore de fonctions cellulaires, y compris la prolifération cellulaire, la différenciation, la migration et l'apoptose. Au début du développement embryonnaire, l'activité kinase est très dynamique et répandue dans l'embryon. Les approches pharmacologiques et génétiques sont couramment utilisées pour sondage des activités kinase. Malheureusement, il est difficile d'obtenir une résolution spatiale et temporelle supérieure en utilisant ces stratégies. En outre, il n'est pas possible de contrôler l'activité kinase de manière réversible dans les cellules vivantes et les organismes multicellulaires. Une telle limitation reste un goulet d'étranglement pour obtenir une compréhension quantitative de l'activité kinase pendant le développement et la différenciation. Ce travail présente une stratégie opto-génétique qui profite d'un système bicistronique contenant des protéines photoactivables Arabidopsis thaliana cryptochrome 2 (CRY2) et le domaine N-terminal de l'hélice-hélice-hélice basique interagissant avec le cryptochrome (CIBN). ReversiL'activation de la voie de signalisation de la protéine kinase activée par mitogène (MAPK) est obtenue grâce à une translocation de protéines à médiation légère dans des cellules vivantes. Cette approche peut être appliquée aux cultures cellulaires de mammifères et aux embryons de vertébrés vivants. Ce système bicistronique peut être généralisé pour contrôler l'activité d'autres kinases avec des mécanismes d'activation similaires et peut être appliqué à d'autres systèmes modèles.
Introduction
Les facteurs de croissance sont impliqués dans un large éventail de fonctions cellulaires, y compris la prolifération, la différenciation, la migration et l'apoptose, et jouent un rôle central dans de nombreux événements biologiques, y compris le développement embryonnaire, le vieillissement et la régulation de l'état mental 1 , 2 , 3 , 4 , 5 . De nombreux facteurs de croissance signalent des cascades de signalisation intracellulaire complexes. Ces événements de signalisation sont souvent opérés par la phosphorylation réversible des protéines de manière réglementée 6 , 7 . Ainsi, une compréhension des résultats de signalisation des protéines kinases, qui sont responsables de la phosphorylation des protéines, est fondamentalement importante.
Différents facteurs de croissance agissent à travers un réseau de signalisation intracellulaire assez fréquent, bien qu'ils stimulent distRéactions cellulaires internes 8 , 9 . Les médiateurs intracellulaires communs des tyrosine kinases réceptrices comprennent Ras, Raf, la kinase extracellulaire régulée par le signal (ERK), la protéine kinase activée par un mitogène (MAPK) / ERK kinase (MEK), la phosphoinositide 3-kinase (PI3K), l'Akt et la phospholipase C gamma (PLCγ) 10 , 11 . Les données recueillies suggèrent que la diversité et la spécificité de la signalisation dépendent de la régulation spatiale et temporelle de l'activité de signalisation 12 . Par exemple, dans les cellules de phéochromocytome de rat (PC12), la stimulation du facteur de croissance épidermique (EGF), qui entraîne une prolifération cellulaire, active de manière transitoire la voie ERK 9 . D'autre part, la stimulation avec le facteur de croissance nerveuse (NGF), qui conduit à la différenciation cellulaire, active la voie ERK de manière soutenue 9 , 13 . En cultureChez les neurones de l'hippocampe, la signalisation transitoire par le facteur neurotrophique dérivé du cerveau (BDNF) favorise l'essor primaire des neurites, tandis que la signalisation prolongée conduit à une augmentation de la dérivation des neurites 14 . Au début du développement embryonnaire, l'activité ERK phosphorylée est temporairement dynamique et est répandue dans l'embryon 6 . Un écran génétique récent lors de l'embryogenèse précoce de Xenopus a montré que les cascades de signalisation ERK et Akt, deux voies du facteur de croissance primaire en aval, affichent des profils d'activation spécifiques à l'étape 7 . Ainsi, une compréhension des résultats de la signalisation kinase nécessite des outils qui peuvent sonder les caractéristiques spatiales et temporelles de l'activité kinase avec une résolution suffisante.
Les approches expérimentales conventionnelles pour sonder la nature dynamique de la transduction du signal pendant le développement n'ont pas la résolution spatiale et temporelle souhaitable. Par exemple, les approches pharmacologiques utilisent une petite chemMolécules biologiques ou biologiques pour stimuler ou supprimer la transduction du signal dans les cellules et les tissus. La nature diffusive de ces petites molécules rend difficile de restreindre leur action à une région d'intérêt spécifique 15 . Les approches génétiques ( par exemple, la transgénèse, le système Cre-Lox ou la mutagenèse) conduisent souvent à une activation ou à une répression irréversible de l'expression du gène cible ou de l'activité protéique 16 , 17 , 18 . Le système Tet-On / Tet-Off 19 offre un contrôle temporel amélioré de la transcription des gènes mais manque de contrôle spatial strict car il repose sur la diffusion de la tetracycline. Les développements récents de la dimérisation des protéines chimiquement induites 20 ou des photos 21 , 22 , 23 , 24 ont considérablement amélioréLe contrôle temporel des réseaux de signalisation. Le contrôle spatial, cependant, reste difficile en raison de la nature diffusive des produits chimiques en cage.
Les approches optogénétiques émergentes récentes, qui exploitent la puissance de la lumière pour contrôler les interactions protéine-protéine, permettent la modulation des voies de signalisation avec une haute précision spatio-temporelle ainsi qu'une réversibilité. Peu après son succès initial dans le contrôle des tirs neuronaux 25 , 26 , 27 , l'optogenèse a été étendue pour contrôler d'autres processus cellulaires, tels que la transcription des gènes, la traduction, la migration cellulaire, la différenciation et l'apoptose 28 , 29 , 30 , 31 , 32 , 33 , 34 . Une stratégie utilisant la pLa combinaison de protéines hotoactivables La protéine de cryptochrome 2 d' Arabidopsis thaliana (CRY2) et le domaine N-terminal de l'hélice-boucle de base-hélice basique (CIBN) qui coopère avec le cryptochrome ont récemment été développés pour contrôler l'activité de Raf1 kinase dans des cellules de mammifères et des embryons de Xenopus 35 . CRY2 se lie à CIBN lors de la stimulation de la lumière bleue, et le complexe de protéines CRY2 / CIBN se dissocie spontanément dans l'obscurité 34 . La lumière bleue excite le cofacteur CRY2, le dinucléotide d'adénine de flavine (FAD), ce qui conduit à une modification conformationnelle de CRY2 et à sa liaison ultérieure à CIBN. Des mutants actifs actifs (W374A) et des déficients en flavine (D387A) de CRY2 peuvent être produits par des mutations dans la poche de liaison au FAD: le mutant CRY2 W374A se lie à CIBN indépendamment de la lumière, alors que le mutant CRY2 D387A ne se lie pas à la CIBN sous le bleu - stimulation lumineuse 36 , 37 . Le système opto-génétique décrit iN ce protocole utilise CRY2 et CIBN de type sauvage pour induire une activation de Raf1 médiée par translocation de protéines dans des cellules vivantes. On sait que le recrutement membranaire de Raf1 améliore son activité 38 . Dans ce système, un module tandem CIBN est ancré à la membrane plasma et CRY2-mCherry est fusionné à la N-terminal de Raf1 35 . En l'absence de lumière bleue, CRY2-mCherry-Raf1 reste dans le cytoplasme et Raf1 est inactif. La stimulation à la lumière bleue induit la liaison CRY2-CIBN et recrute Raf1 à la membrane plasmatique, où Raf1 est activé. L'activation de Raf stimule une cascade de signalisation Raf / MEK / ERK. Les protéines de fusion CRY2 et CIBN sont codées dans un système génétique bicistronique. Cette stratégie peut être généralisée pour contrôler d'autres kinases, telles que Akt, dont l'état d'activation peut également être activé par la translocation des protéines dans les cellules 39 . Ce travail présente des protocoles détaillés pour la mise en œuvre de cette stratégie optogénétique dans la culture de cellules de mammifèresRes et des organismes multicellulaires.
Protocole
La recherche sur les animaux a été menée conformément aux lignes directrices établies par le Comité de l'établissement et de l'alimentation en établissement des animaux de l'Illinois (IACUC) et le Département des ressources animales de l'Université de l'Illinois (DAR).
1. Induction optogénétique de la localisation des protéines dans la culture cellulaire de mammifères BHK21
REMARQUE: Les étapes 1.1-1.3 fournissent une méthode pour assembler une cellule de culture cellulaire pour l'imagerie avec des objectifs de grossissement élevé ( par exemple, 63X ou 100X), qui ont généralement de courtes distances de travail. Ces objectifs nécessitent une fine lamelle de verre ( par exemple, 1,5 pouce d'épaisseur de 170 μm) en tant que substrat d'imagerie. Alternativement, on peut utiliser un plateau / glissière de culture de fond en verre. Dans un tel cas, les étapes 1.1-1.3 peuvent être ignorées.
- Nettoyage des lamelles de verre
- Placez des lamelles en verre dans un porte-lamelle.
- Dans un bécher en plastique de 600 ml, ajouter 20 g de détergent et 400 ml d'eau chaude au robinet. PlacE le bécher sur un agitateur magnétique et remuer jusqu'à ce que tout le détergent soit dissous. Retirer la mousse à l'aide de papier de soie.
- Utilisez le fond d'une boîte de Petri de 100 mm x 15 mm comme espaceur de la barre d'agitation et comme surface pour le porte-lamelle. Pour maintenir un débit adéquat pendant le processus de nettoyage, faites 3-4 trous de vidange dans la boîte de Petri.
- Pour faire les trous de vidange, chauffer un fer à souder et brûler à travers le plastique dans un capot ventilé.
ATTENTION: Ne pas toucher le fer à souder chauffé à mains nues. - Placez le porte-lamelle sur la boîte de Petri dans le bécher. Mélanger pendant 2 heures jusqu'à la nuit à température ambiante.
- Laver trois fois avec 500 mL d'eau DI dans un bécher de 1000 mL. La solution de détergent peut être réutilisée.
- Ramassez les lamelles individuelles à l'aide d'une pince. Immergez les lamelles dans l'éthanol à 190 épreuves (95%) pendant 1 min.
- Séchez les lamelles à l'intérieur d'un capuchon stérile. Rangez les lamelles dans un récipient en plastique stérile jusqu'à l'utilisation.
- Fabriquer des lamelles revêtues de poly-L-lysine (PLL)
- Faire un tampon de borate 0,1 M en dissolvant 1,24 g d'acide borique et 1,9 g de tétraborate disodique dans 400 ml d'eau. Ajuster le pH à 8,5 avec du NaOH 10 M.
- Dissoudre PLL dans le tampon de borate jusqu'à une concentration finale de 1 mg / mL. Aliquotez la solution de PLL dans des tubes centrifuges stériles de 50 ml.
- Ajouter 50 ml de solution de revêtement PLL dans un plat de culture de tissu de 10 cm. Placer le plat dans un incubateur à 37 ° C pour réchauffer la solution PLL.
- Ouvrez le conteneur de lamelle (étape 1.1.8) dans un capot stérile.
- Immergez les lamelles nettoyées une par une dans la solution de PLL préchauffée dans le plat de culture de tissus. Évitez la formation de bulles afin d'immerger toutes les surfaces.
- Placez le plat avec la lamelle coulissante dans un incubateur de CO 2 de 37 ° C toute la nuit. Retirez chaque lamelle et placez-le dans un porte-lamelle stérile. Rincer trois fois avec de l'eau ultrapure autoclavée (18,2 MΩ et #183; cm de résistivité) dans un bécher de 1000 ml dans une hotte stérile.
- Séchez les lamelles dans le porte-lamelle dans le capot stérile. Rangez les lamelles dans un récipient en plastique stérile jusqu'à l'utilisation.
- Assemblage de cellules de culture cellulaire de polydiméthylsiloxane (PDMS)
- Dans un récipient en plastique, peser 40 g de base PDMS et ajouter 4 g d'agent de durcissement pour obtenir un rapport 10: 1 p / p de PDMS et agent de durcissement. Mélanger le PDMS / agent de durcissement en remuant avec une pointe de pipette pendant 2 min. Alternativement, utilisez un mélangeur centrifuge.
- Utilisez un bécher avec un diamètre de 4 pouces en tant que modèle pour fabriquer un support avec une feuille d'aluminium. Placez le support en aluminium dans une boîte de Petri de 15 cm. Placez une plaquette de silicium de 4 pouces dans ce support en aluminium.
- Verser la solution PDMS mélangée dans le support. Utilisez une fine tige en verre pour toucher doucement le bord de la plaquette. Retirez les bulles d'air piégées sous la plaquette.
- Insérez la boîte de Petri de 15 cm dans un chambe sous videR pour dégager la solution PDMS. Continuer le dégazage pendant environ 20 minutes, jusqu'à ce qu'aucune bulle ne soit générée. En attendant, préchauffer un four à convection à 65 ° C.
- Placez soigneusement la boîte de Petri de 15 cm dans le four. Incuber pendant 2 h.
- Retirez la plaque du four. Utilisez une barre de verre pour toucher doucement le bord du PDMS et assurez-vous une réticulation complète. Sinon, incuber plus longtemps, jusqu'à ce que le PDMS soit solide et non collant.
- Retirez le film d'aluminium de la plaque et éloignez-la de la plaquette de silicium. À partir du bord, éplucher soigneusement le PDMS duré de la plaquette de silicium.
ATTENTION: Évitez d'appliquer trop de force, puisqu'il peut casser la gaufrette. - Coupez le PDMS dans des pièces rectangulaires de 20 mm x 30 mm pour ajuster la lamelle de 24 mm x 40 mm avec une lame de rasoir.
- Utilisez une lame en forme de ciseau pour couper une ouverture rectangulaire de 10 mm x 20 mm du centre de chaque pièce PDMS.
- Nettoyer les chambres PDMS en utilisant le protocole détergent décrit à l'étape 1.1.
- Séchez les chambres PDMS dans un capot propre. Placez les chambres sèches dans un récipient autoclavable recouvert de papier d'aluminium.
- Enclenchez le récipient à l'aide de la gravité (121 ° C, 15 psi) pendant 30 minutes et rangez le récipient à température ambiante.
- Culture et transfection des cellules BHK21
- Faire 500 mL de milieu pour la culture de cellules BHK21 en mélangeant 445 ml de DMEM avec 50 mL de FBS et 5 mL de Penn-Strep-Glutamine 100 × (10 000 U / mL de pénicilline, 10 mg / ml de streptomycine et 29,2 mg / mL de L -glutamine). Assurez-vous que la concentration finale de FBS est de 10%.
- Aliquote 10 ml de milieu indépendant de HEPES CO 2 pour l'imagerie des cellules vivantes.
NOTE: Le milieu indépendant de CO 2 supporte la croissance cellulaire sans incubateur de CO 2 et est idéal pour imager des cellules dans des conditions atmosphériques. Reportez-vous à la liste des matériaux pour obtenir des informations détaillées. En plus de la lignée cellulaire BHK21, d'autres types de cellules de mammifères devraient également donnerDes résultats similaires. - Assembler un dispositif de culture cellulaire en plaçant une chambre PDMS stérilisée autoclavée (étape 1.3) sur une lamelle stérile revêtue de PLL (étape 1.2) dans une hotte stérile.
- Placez chaque appareil dans une boîte de Petri stérile de 60 mm.
- Utiliser 0,5 ml de trypsine à 0,25% pour détacher les cellules d'un puits d'une plaque de culture tissulaire à 12 puits. Compter la densité cellulaire avec un hémocytomètre. Placer 20 000 cellules BHK21 dans la chambre (environ 10 000 cellules / cm 2 ) avec 200 μL de milieu de culture cellulaire.
- Préparer un plasmide d'ADN avec un kit de préparation de plasmide (voir la Liste des matériaux).
REMARQUE: La conception et la fonctionnalité du CRY2-mCherry-Raf1-2A-2CIBN-GFP-CaaX sont illustrées à la Figure 1A - 1B . - Vingt-quatre (24) h après le placage cellulaire, transfectent les cellules avec 50-100 ng de plasmide CRY2-mCherry-Raf1-2A-2CIBN-GFP-CaaX, selon le protocole du fabricant.
- Trois (3) heures après la transfection, changez le mÀ 200 μL de milieu de culture frais (étape 1.4.1) et permettent aux cellules de se rétablir pendant la nuit en incubant la culture dans un incubateur à 37 ° C, 5% de CO 2 .
- L'imagerie par cellule vive en fluorescence
- Allumez un ordinateur, une source lumineuse, un microscope et une caméra. Utilisez un microscope à fluorescence confocale (équipé d'un objectif d'immersion en huile 100X) pour le reste du protocole.
NOTE: Un microscope à épifluorescence inversée peut également être utilisé. - Remplacer le milieu de culture cellulaire avec 200 μL de milieu indépendant de CO 2 .
- Configurez le protocole d'acquisition de données avant de placer les cellules sur le microscope. Utilisez un canal FITC d'excitation de 488 nm pour la stimulation optogénétique. Utilisez un canal TRITC d'excitation de 561 nm pour localiser les cellules transfectées et suivre la localisation cellulaire des protéines marquées par mCherry.
- Réglez le gain des canaux FITC et TRITC comme 120 et 200, respectivement. Utilisez un temps de pixel de 2.21; s et une taille de 512 x 512 pixels.
- Mesurez la puissance de la lumière de 488 nm en plaçant un compteur de puissance à proximité de la fenêtre d'objectif. Une puissance totale de 2 μW (environ 10 000 W / cm 2 au foyer) est suffisante pour induire une association CIBN-CRY2PHR.
- Mettre en place une acquisition d'horodatage avec un intervalle de 5 s et un temps d'acquisition total de 2 min.
- Appliquez un matériau d'appariement approprié (huile d'immersion) sur la fenêtre d'objectif. Utilisez le mode de contraste de phase pour se concentrer sur les cellules sur la surface de la lamelle.
- Localiser une cellule transfectée sous une lumière de 561 nm en déplaçant le stade du microscope.
ATTENTION: Évitez d'utiliser la lumière bleue pendant cette étape, car elle active l'association entre les protéines photoactivables. - Une fois qu'une cellule transfectée est située, lancez l'acquisition de données.
NOTE: Les cellules transfectées avec succès doivent présenter une fluorescence dans les canaux FITC (à partir de 2CIBN-GFP-CaaX) et TRITC (à partir de CRY2-mCherry-Raf1) ( Figure 1C-D ). - Enregistrez une série d'images estampillées dans les deux canaux FITC et TRITC ( Figure 1D ).
- Pour sonder la dissociation spontanée du complexe de protéines CRY2-CIBN, enregistrez un autre timestamp avec le canal Txred seul pendant 20 min, avec un intervalle de 30 s.
- Enregistrez l'image timbrée pour l'analyse des données.
- Allumez un ordinateur, une source lumineuse, un microscope et une caméra. Utilisez un microscope à fluorescence confocale (équipé d'un objectif d'immersion en huile 100X) pour le reste du protocole.
- L'analyse d'image
- Ouvrez les images avec un logiciel d'analyse d'image qui peut extraire l'intensité d'une image 40 .
- Sélectionnez deux instantanés représentatifs: un avant l'exposition à la lumière bleue et l'autre exposition post-lumière bleue.
- Dessinez une ligne à travers la cellule couvrant l'arrière-plan, la membrane plasmique et le cytoplasme. Expliquez l'intensité sur cette ligne. Enregistrez les valeurs et traitez-les pour comparer la différence avant et après l'exposition à la lumière ( Figure 1D ).
- Pour analyser la cinétique de l'association des protéines, sélectionnezT une région représentative d'intérêt (ROI) dans la membrane plasmatique, un ROI dans le cytoplasme et un ROI en arrière-plan.
- Acquérir les intensités moyennes de la membrane plasmatique (I PM ), du cytoplasme (I CYT ) et de l'arrière-plan (I BKD ) pour la pile d'images.
- Calculer le rapport de l'intensité membranaire / cytosolique pour chaque image en utilisant l'équation suivante:
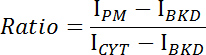
- Tracer le rapport par rapport au temps et déterminer la cinétique de liaison ou de dissociation de CRY2-CIBN ( Figure 1E- F ).
2. Construction d'une matrice LED pour la stimulation de la lumière à long terme dans un incubateur de CO 2
REMARQUE: Le schéma global de l'installation expérimentale est illustré à la figure 2A .
- Faites le tableau LED en insérant 12 LED bleues en deux panneauxDs et les résistances de limitation de courant de connexion.
REMARQUE: avec une alimentation de 30 V, quatre LED peuvent être connectées en série et leur luminosité peut être contrôlée par une résistance de limitation de courant. - Placez les panneaux dans une boîte en aluminium.
REMARQUE: La hauteur de la boîte en aluminium doit être de 2 po. Cette hauteur est optimale pour éclairer une plaque de culture de tissus à 12 puits, car la taille du spot lumineux divergent est identique à celle d'un seul puits. - Utilisez deux fils métalliques pour vous connecter à l'alimentation électrique. Assurez-vous que la longueur du fil est suffisante lorsque la boîte à lumière est placée dans un incubateur de CO 2 .
- Utilisez un diffuseur de lumière transparent comme le couvercle de la boîte à lumière ( Figure 2C ).
- Calibrez la puissance de chaque LED à une gamme d'entrées de tension. Utilisez une puissance de 0,2 mW / cm 2 pour le test de différenciation cellulaire PC12 24 h. Utiliser une puissance de 5 mW / cm 2 pour les embryons ou les explants de Xenopus en directTests.
3. Induction optogénétique de la différenciation cellulaire PC12
- Culture cellulaire et transfection
- Faire 500 ml de milieu pour une culture cellulaire de phéochromocytome de rat (PC12) en mélangeant 407,5 ml de F12K avec 75 ml de sérum de cheval + 12,5 ml de FBS + 5 ml de 100 × Penn-Strep-Glutamine (concentrations finales de sérum de cheval et FBS : 15% et 2,5%, respectivement). Faire un faible milieu sérique en mélangeant 1 volume de milieu complet dans 99 volumes de milieu F12K.
- Placer les cellules PC12 dans une plaque de 12 puits à une densité de 300 000 cellules / puits ou 75 000 cellules / cm 2 .
- Transférez les cellules avec CRY2-mCherry-Raf1-2A-2CIBN-GFP-CaaX 24 h après le placage cellulaire (similaire à l'étape 1.4.7). Utiliser 1,2 μg d'ADN pour chaque puits. Autoriser les cellules à se rétablir pendant une nuit dans un milieu de culture dans un incubateur à 37 ° C complété par 5% de CO 2 . Vérifier l'efficacité de transfection 16 h après la transfection.
REMARQUE: une transfection de 30 à 50%Il conviendrait d'assurer un nombre suffisant de cellules. - Changer le milieu à faible teneur en sérum 24 h après la transfection. Utiliser 1 ml de milieu par puits d'une assiette à 12 puits.
- Différenciation cellulaire PC12 induite par la lumière
- Placez le tableau LED dans un incubateur de CO 2 . Connectez-le à l'alimentation en utilisant une paire de fils. Réglez la puissance de la LED à 0,2 mW / cm 2 . Placez la plaque de 12 puits contenant des cellules PC12 transfectées (étape 3.1.3) sur la fenêtre de la matrice LED. Appliquer l'éclairage continu de la lumière pendant 24 h dans un incubateur à 37 ° C complété par 5% de CO 2 .
- Imagerie par cellule vive en fluorescence
- Suivez les étapes 1.5.1-1.5.4 pour le microscope et la préparation de l'échantillon.
- Configurer l'acquisition de données à une seule capture. Utilisez 200 ms pour le canal GFP et Txred.
- Capturez les images des cellules transfectées dans les deux canaux GFP et Txred. Enregistrez environ 200 celC'est pour chaque condition. Enregistrez les fichiers pour l'analyse des données.
- L'analyse d'image
- Comptez le pourcentage de cellules différenciées sur le nombre total de cellules transfectées.
- Utilisez le module de comptage de cellules dans le logiciel d'analyse d'image pour compter les cellules.
- Comptez manuellement les cellules différenciées.
NOTE: Les cellules différenciées sont définies comme celles dans lesquelles au moins un neurite est discernablement plus long que le corps de la cellule ( Figure 3A- 3B ). - Répétez l'étape 3.4.3 avec des cellules indifférenciées.
- Calculez le taux de différenciation en utilisant l'équation suivante:
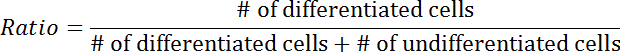
4. Contrôle optogénétique de l'activité de la kinase chez les embryons de Xenopus
- Préparation des tampons
- Préparez 1 L de 1x Timer Modifié Marc (MMR): 100 mMNaCl, 2 mM de KCl, 1 mM de MgCl2, 2 mM de CaCl2 et 5 mM de HEPES; PH 7,5.
- Préparation de l'ARNm
- Recréer l'ADN plasmidique CRY2-mCherry-Raf1-2A-2CIBN-GFP-CaaX (2 μg) avec 1 μL d'ApaI (50 unités) à 37 ° C pendant 2 h.
- Précipiter l'ADN linéarisé dans 60 μL d'éthanol à 100%. Tourner à 12 000 × g pendant 5 minutes à température ambiante pour granuler l'ADN. Retirez soigneusement le surnageant et lavez encore le culot avec 60 μL d'éthanol à 70%. Faire tourner à 12 000 × g pendant encore 5 minutes à température ambiante. Retirez avec précaution le surnageant et remettez à nouveau le culot d'ADN dans 6 μl de H20 libre de RNase.
- Effectuer la transcription in vitro en incubant 1 μg d'ADN linéarisé avec 2 μl d'ARN polymérase SP6 fournie, un mélange de ribonucléotides (ATP 10 mM, CTP et UTP, GTP 2 mM et analogue de capuchon 8 mM) et 20 μL de nuclease- Eau libre à température ambiante pendant 1 h.
- Supprimer le DModèle NA par digestion par DNase I à 37 ° C pendant au moins 15 min et purification de l'ARN synthétisé à l'aide d'une colonne de spin de silice-membrane.
- Arrêter la réaction et précipiter l'ARN en ajoutant 30 μL d'eau exempte de nuclease et 30 μL de solution de précipitation au LiCl. Bien mélanger et refroidir pendant 30 min à -20 ° C.
- Centrifuger à 4 ° C pendant 15 min à 12 000 × g pour granuler l'ARN.
- Retirez soigneusement le surnageant. Laver l'ARN une fois avec 1 mL d'éthanol à 70% et ré-endiguer dans 40 μL d'eau exempte de nuclease.
- Chargez 40 μL d'ARN dans une colonne de spin. Centrifuger à 4 ° C pendant 1 min à 12 000 × g pour éliminer les nucléotides et capsules non incorporés.
- Récolter des embryons de Xenopus
- Suivez le protocole décrit par Sive et al. 41 pour obtenir les embryons de Xenopus .
- Micro-injection d'ARNm
- FabriquerLes aiguilles pour la micro-injection en tirant des capillaires en verre avec un extracteur capillaire. Réglez le programme de traction sur: chaleur = 355, force de tirage = 80, vitesse = 50 et temps = 100.
- Retirez la couche de gelée des embryons en les traitant avec 3% de cystéine (diluée dans 0,2x MMR).
- Transférer les embryons à 3% de polysucrose et à solution de MMX 0.5x pour microinjection. Injecter 500 pg à 1 ng d'ARN CRY2-mCherry-Raf1-2A-2CIBN-GFP-CaaX dans chaque embryon.
REMARQUE: l'injection d'une dose supérieure à 1 ng entraîne une activation de la signalisation MAPK indépendante de la lumière bleue.
- La stimulation optogénétique de l'activité kinase
- Culture des embryons micro-injectés dans 3% de polysucrose, solution de MMR 0,5x à température ambiante jusqu'à ce qu'elles atteignent le stade de la moyenne des gaztrules (étape 12). Ensuite, culturez les embryons dans une solution de MMR 0,2x.
- Transférer les embryons ou les explants dans une assiette de 12 puits. Placez la plaque à 12 puits sur la matrice LED à la maison (étape 2.5) pour la lumière bleueT (475 nm) de traitement.
- Placez un miroir sur le dessus de la plaque à 12 puits pour assurer un éclairage bleu clair des embryons ou des explants.
- Réglez la puissance de la lumière bleue à 5 mW / cm 2 .
REMARQUE: Le traitement à la lumière bleue peut être effectué à n'importe quel moment souhaité, soit en polysucrose à 3%, en solution de MMX 0,5x, soit en solution de MMR 0,2x. - Récolte les embryons à tout moment souhaité. Utilisez-les pour une analyse histologique, Western-blot ou d'expression de gènes.
Résultats
L'expression ratiométrique des paires de protéines photoactivables: la figure 1A montre la conception d'une construction optogénétique bicistronique, CRY2-mCherry-Raf1-P2A-CIBN-CIBN-GFP-CaaX (appelée CRY2-2A-2CIBN), basée sur le teschovirum porcine- 1 2A (P2A) peptide, qui montre l'efficacité de saut de ribosome le plus élevé parmi les lignées de cellules de mammifères 42 . Dans les travaux antérieurs, il a ét...
Discussion
Lors de la construction de la boîte à lumière, la puissance des LED individuelles doit être mesurée. Sur la base de l'expérience précédente, la puissance de sortie peut varier entre les LED individuelles en raison de la variance de fabrication. Sélectionnez un ensemble de LEDs ayant une puissance inférieure à 10% l'une de l'autre. Le nombre de diodes électroluminescentes, la résistance de limitation de courant et l'entrée de puissance peuvent être modifiés pour différents types de conten...
Déclarations de divulgation
Les auteurs déclarent qu'ils n'ont pas d'intérêts financiers concurrents.
Remerciements
Ce travail a été soutenu par l'Université de l'Illinois à Urbana-Champaign (UIUC) et les National Institutes of Health (NIGMS R01GM111816).
matériels
| Name | Company | Catalog Number | Comments |
| Glass coverslip | VWR | 48393 230 | Substrate for live cell imaging |
| Coverslip holder | Newcomer Supply | 6817B | Holder for coverslips |
| Detergent | ThermoFisher | 16 000 104 | For cleaning coverslips |
| Boric acid | Sigma-Aldrich | B6768-500G | For making PLL buffer |
| Disodium tetraborate | Sigma-Aldrich | 71996-250G | For making PLL buffer |
| Plastic beaker | Nalgene | 1201-1000 | For cleaning coverslips |
| Sodium hydroxide | Sigma-Aldrich | 221465-2.5KG | For adjust pH |
| Poly-L-lysine hydrobromide | Sigma-Aldrich | P1274-500MG | For coating coverslip |
| Diethylpyrocarbonate (DEPC)-Treated Water | ThermoFisher Scientific | 750024 | For DNA preparation |
| Cover Glass Forceps | Ted Pella | 5645 | Cover glass handling |
| Tissue cutlure dish | Thermofisher | 12565321 | Cell culture dish |
| Sterile centrifuge tubes | ThermoFisher | 12-565-271 | Buffer storage |
| Transfection Reagent | ThermoFisher | R0534 | Transfection |
| CO2-independent medium | ThermoFisher | 18045088 | For live cell imaging |
| Polydimethylsiloxane (PDMS) | Ellsworth Adhesives | 184 SIL ELAST KIT 0.5KG | Form make cell chamber |
| Plasmid Maxiprep kit | Qiagen | 12965 | Plasmid preparation |
| DMEM medium | ThermoFisher | 11965-084 | Cell culturing medium component |
| F12K medium | ThermoFisher | 21127022 | Cell culturing medium component |
| Horse serum | ThermoFisher | 16050122 | Cell culturing medium component |
| Fetal Bovine Serum | Signa-Aldrich | 12303C-500 mL | Cell culturing medium component |
| Penicillin-Streptomycin-Glutamine | ThermoFisher | 10378016 | Cell culturing medium component |
| Trypsin (0.25%), phenol red | ThermoFisher Scientific | 15050065 | For mammalian cell dissociation |
| Agarose | Fisher Scientific | BP1356-100 | For DNA preparation |
| Ficoll PM400 | GE Heathcare Life Sciences | 17-5442-02 | For embryo buffer |
| L-Cysteine hydrochloride monohydrate | Sigma-Aldrich | 1.02839.0025 | Oocyte preparation |
| ApaI | ThermoFisher | FD1414 | For linearization of plasmids |
| Dnase I | ThermoFisher | AM2222 | For removing DNA template in the in vitro transcription assay |
| Index-match materials (immersion oil) | Thorlabs | MOIL-20LN | For matching the index between sample substrate and objective |
| Blue LED | Adafruit | 301 | Light source for optogenetic stimulation |
| Resistor kit | Amazon | EPC-103 | current-limiting resistor |
| Aluminum boxes | BUD Industries | AC-401 | light box |
| BreadBoard | Jekewin | 837654333686 | For making LED array |
| Hook up Wire | Electronix Express | 27WK22SLD25 | For making LED array |
| Relay Module | Jbtek | SRD-05VDC-SL-C | For intermittent light control |
| DC Power Supply | TMS | DCPowerSupply-LW-(PS-305D) | Power supply for LED |
| Silicon Power Head | Thorlabs | S121C | For light intensity measurement |
| Power meter | Thorlabs | PM100D | For light intensity measurement |
| Microscope | Leica Biosystems | DMI8 | For live cell imaging |
| BioSafety Cabinet | ThermoFisher | 1300 Series A2 | For mammalian cell handling |
| CO2 incubator | ThermoFisher | Isotemp | For mammalian cell culturing |
| Stereo microscope | Leica | M60 | For embryo micro-manipulation |
| Microinjector | Narishige | IM300 | For embryo microinjection |
| Micropipette puller | Sutter Instruments | P87 | Needle puller |
| in vitro transcription kit | ThermoFisher | AM1340 | For in vitro transcription. The kit includes nuclease-free water, SP6 RNA Polymerase, ribonucleotide mixture, cap analog, lithium choride precipitation solution, and spin column |
| RNA purfication kit | Qiagen | 74104 | Silica-membrane spin column for purification of synthesized RNA |
| Convection oven | MTI corporation | EQ-DHG-9015 | PDMS curing |
| Centrifugal mixer and teflon container | THINKY | AR310 | For mixing PDMS |
| Silicon wafer | UniversityWafer | 452 | Base for making PDMS devices |
| Blade | Techni Edge | 01-801 | For cutting PDMS |
| Capillary glass | Sutter Instruments | BF100-58-10 | For fabrication of injecting needles. |
Références
- Schlessinger, J., Ullrich, A. Growth factor signaling by receptor tyrosine kinases. Neuron. 9 (3), 383-391 (1992).
- Thisse, B., Thisse, C. Functions and regulations of fibroblast growth factor signaling during embryonic development. Dev Biol. 287 (2), 390-402 (2005).
- Perrimon, N., Pitsouli, C., Shilo, B. Z. Signaling mechanisms controlling cell fate and embryonic patterning. Cold Spring Harb Perspect Biol. 4 (8), a005975 (2012).
- Salles, F. H., et al. Mental disorders, functional impairment, and nerve growth factor. Psychol Res Behav Manag. 10, 9-15 (2017).
- Basson, M. A. Signaling in cell differentiation and morphogenesis. Cold Spring Harb Perspect Biol. 4 (6), (2012).
- Schohl, A., Fagotto, F. beta-catenin, MAPK and Smad signaling during early Xenopus development. Development. 129 (1), 37-52 (2002).
- Zhang, S. W., Li, J. J., Lea, R., Amaya, E., Dorey, K. A Functional Genome-Wide In Vivo Screen Identifies New Regulators of Signalling Pathways during Early Xenopus Embryogenesis. PloS one. 8 (11), e79469 (2013).
- Sweeney, C., et al. Growth factor-specific signaling pathway stimulation and gene expression mediated by ErbB receptors. J Biol Chem. 276 (25), 22685-22698 (2001).
- Marshall, C. J. Specificity of receptor tyrosine kinase signaling: transient versus sustained extracellular signal-regulated kinase activation. Cell. 80 (2), 179-185 (1995).
- Vandergeer, P., Hunter, T., Lindberg, R. A. Receptor Protein-Tyrosine Kinases and Their Signal-Transduction Pathways. Annu Rev Cell Biol. 10, 251-337 (1994).
- Lemmon, M. A., Schlessinger, J. Cell signaling by receptor tyrosine kinases. Cell. 141 (7), 1117-1134 (2010).
- Hunter, T. Signaling--2000 and beyond. Cell. 100 (1), 113-127 (2000).
- Qiu, M. S., Green, S. H. PC12 cell neuronal differentiation is associated with prolonged p21ras activity and consequent prolonged ERK activity. Neuron. 9 (4), 705-717 (1992).
- Ji, Y., et al. Acute and gradual increases in BDNF concentration elicit distinct signaling and functions in neurons. Nat Neurosci. 13 (3), 302-309 (2010).
- Luby-Phelps, K. Cytoarchitecture and physical properties of cytoplasm: volume, viscosity, diffusion, intracellular surface area. Int Rev Cytol. 192, 189-221 (2000).
- Sauer, B. Inducible gene targeting in mice using the Cre/lox system. Methods-a Companion to Methods in Enzymology. 14 (4), 381-392 (1998).
- Ling, M. M., Robinson, B. H. Approaches to DNA mutagenesis: an overview. Anal Biochem. 254 (2), 157-178 (1997).
- Gama Sosa, M. A., De Gasperi, R., Elder, G. A. Animal transgenesis: an overview. Brain Struct Funct. 214 (2-3), 91-109 (2010).
- Wanka, F., et al. Tet-on, or Tet-off, that is the question: Advanced conditional gene expression in Aspergillus. Fungal Genet Biol. 89, 72-83 (2016).
- Karginov, A. V., Ding, F., Kota, P., Dokholyan, N. V., Hahn, K. M. Engineered allosteric activation of kinases in living cells. Nat Biotechnol. 28 (7), 743-747 (2010).
- Liu, Q. Y., Deiters, A. Optochemical Control of Deoxyoligonucleotide Function via a Nucleobase-Caging Approach. Accounts of Chemical Research. 47 (1), 45-55 (2014).
- Arbely, E., Torres-Kolbus, J., Deiters, A., Chin, J. W. Photocontrol of tyrosine phosphorylation in mammalian cells via genetic encoding of photocaged tyrosine. J Am Chem Soc. 134 (29), 11912-11915 (2012).
- Gautier, A., Deiters, A., Chin, J. W. Light-activated kinases enable temporal dissection of signaling networks in living cells. J Am Chem Soc. 133 (7), 2124-2127 (2011).
- Nguyen, D. P., et al. Genetic Encoding of Photocaged Cysteine Allows Photoactivation of TEV Protease in Live Mammalian Cells. J Am Chem Soc. 136 (6), 2240-2243 (2014).
- Deisseroth, K. Optogenetics. Nat Methods. 8 (1), 26-29 (2011).
- Boyden, E. S., Zhang, F., Bamberg, E., Nagel, G., Deisseroth, K. Millisecond-timescale, genetically targeted optical control of neural activity. Nat Neurosci. 8 (9), 1263-1268 (2005).
- Banghart, M., Borges, K., Isacoff, E., Trauner, D., Kramer, R. H. Light-activated ion channels for remote control of neuronal firing. Nat Neurosci. 7 (12), 1381-1386 (2004).
- Zhang, K., Cui, B. Optogenetic control of intracellular signaling pathways. Trends in Biotechnology. 33 (2), 92-100 (2015).
- Tischer, D., Weiner, O. D. Illuminating cell signalling with optogenetic tools. Nat Rev Mol Cell Bio. 15 (8), 551-558 (2014).
- Kim, B., Lin, M. Z. Optobiology: optical control of biological processes via protein engineering. Biochemical Society Transactions. 41 (5), 1183-1188 (2013).
- Tucker, C. L. Manipulating cellular processes using optical control of protein-protein interactions. Prog Brain Res. 196, 95-117 (2012).
- Toettcher, J. E., Gong, D. Q., Lim, W. A., Weiner, O. D. Light Control of Plasma Membrane Recruitment Using the Phy-Pif System. Method Enzymol. 497, 409-423 (2011).
- Zoltowski, B. D., Gardner, K. H. Tripping the light fantastic: blue-light photoreceptors as examples of environmentally modulated protein-protein interactions. Biochemistry. 50 (1), 4-16 (2011).
- Kennedy, M. J., et al. Rapid blue-light-mediated induction of protein interactions in living cells. Nat Methods. 7 (12), 973-975 (2010).
- Krishnamurthy, V. V., et al. Reversible optogenetic control of kinase activity during differentiation and embryonic development. Development. 143 (21), 4085-4094 (2016).
- Liu, H., et al. Photoexcited CRY2 Interacts with CIB1 to Regulate Transcription and Floral Initiation in Arabidopsis. Science. 322 (5907), 1535-1539 (2008).
- Li, X., et al. Arabidopsis cryptochrome 2 (CRY2) functions by the photoactivation mechanism distinct from the tryptophan (trp) triad-dependent photoreduction. Proc Natl Acad Sci U S A. 108 (51), 20844-20849 (2011).
- Leevers, S. J., Paterson, H. F., Marshall, C. J. Requirement for Ras in Raf Activation Is Overcome by Targeting Raf to the Plasma-Membrane. Nature. 369 (6479), 411-414 (1994).
- Kohn, A. D., Takeuchi, F., Roth, R. A. Akt, a pleckstrin homology domain containing kinase, is activated primarily by phosphorylation. J Biol Chem. 271 (36), 21920-21926 (1996).
- Schindelin, J., et al. Fiji: an open-source platform for biological-image analysis. Nature Methods. 9 (7), 676-682 (2012).
- Sive, H. L., Grainger, R. M., Harland, R. M. . Early Development of Xenopus laevis: A Laboratory Manual. , (2000).
- Kim, J. H., et al. High cleavage efficiency of a 2A peptide derived from porcine teschovirus-1 in human cell lines, zebrafish and mice. PloS one. 6 (4), e18556 (2011).
- Ishimura, A., et al. Oncogenic Met receptor induces ectopic structures in Xenopus embryos. Oncogene. 25 (31), 4286-4299 (2006).
- Zhang, K., et al. Light-Mediated Kinetic Control Reveals the Temporal Effect of the Raf/MEK/ERK Pathway in PC12 Cell Neurite Outgrowth. PloS one. 9 (3), e92917 (2014).
- Mohanty, S. K., Lakshminarayananan, V. Optical Techniques in Optogenetics. J Mod Opt. 62 (12), 949-970 (2015).
- Taslimi, A., et al. Optimized second-generation CRY2-CIB dimerizers and photoactivatable Cre recombinase. Nature chemical biology. 12 (6), 425-430 (2016).
- Kawano, F., Suzuki, H., Furuya, A., Sato, M. Engineered pairs of distinct photoswitches for optogenetic control of cellular proteins. Nat Commun. 6, 6256 (2015).
- Wang, H., et al. LOVTRAP: an optogenetic system for photoinduced protein dissociation. Nat Methods. 13 (9), 755-758 (2016).
- Chang, K. Y., et al. Light-inducible receptor tyrosine kinases that regulate neurotrophin signalling. Nat Commun. 5, 4057 (2014).
- Boulina, M., Samarajeewa, H., Baker, J. D., Kim, M. D., Chiba, A. Live imaging of multicolor-labeled cells in Drosophila. Development. 140 (7), 1605-1613 (2013).
- Liu, H., Gomez, G., Lin, S., Lin, C. Optogenetic control of transcription in zebrafish. PLoS One. 7 (11), e50738 (2012).
- Buckley, C. E., et al. Reversible Optogenetic Control of Subcellular Protein Localization in a Live Vertebrate Embryo. Dev Cell. 36 (1), 117-126 (2016).
- Motta-Mena, L. B., et al. An optogenetic gene expression system with rapid activation and deactivation kinetics. Nature chemical biology. 10 (3), 196-202 (2014).
- Beyer, H. M., et al. Red Light-Regulated Reversible Nuclear Localization of Proteins in Mammalian Cells and Zebrafish. ACS Synth Biol. 4 (9), 951-958 (2015).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon