Method Article
Múltiplex del Cytokine Perfil de esplenocitos de ratón estimulados utilizando una plataforma basada en grano de cytometric del inmunoensayo
En este artículo
Resumen
Este protocolo describe la cuantificación de múltiples citoquinas objetivos simultáneamente en sobrenadantes de cultivo de tejidos de esplenocitos de ratón estimulados usando grano multiplex inmunoensayo basado en la plataforma y un citómetro de flujo.
Resumen
Los inmunoensayos basados en grano emplean el mismo principio básico como inmunoensayo de sándwich. Captura de cuentas, que se pueden distinguir por el tamaño y la intensidad de fluorescencia interna allophycocyanin (APC), se conjugan para anticuerpos específicos para un analito determinado. A continuación, un panel seleccionado de captura definido conjuntos de grano se incuba con una muestra biológica que contenga los analitos específicos de los anticuerpos de captura. Se añade un anticuerpo de detección biotinilado cóctel, que conduce a la formación de bocadillos de grano-analito-detección anticuerpo de captura.
Por último, se añade estreptavidina-ficoeritrina (PE-SA), que se une a los anticuerpos de detección biotinilado, proporcionando intensidades de la señal fluorescente en proporción a la cantidad de analito límite. El PE fluorescente señal de regiones específicas del analito granos se cuantifica mediante citometría de flujo y las concentraciones de analitos particular se determinan usando software de análisis de datos y la curva estándar generada en el ensayo.
En este experimento, utilizamos un panel ratón T helper citoquinas simultáneamente cuantificar la concentración de 13 objetivos separados del cytokine en sobrenadantes de cultivo de tejidos de esplenocitos de ratón cultivadas bajo diferentes condiciones de estimulación.
Introducción
En su papel como moléculas de señalización solubles, citocinas median los procesos altamente coordinados y multifactoriales que regulan las respuestas inmunológicas de host. Se expresan en todas las fases del proceso inflamatorio, de iniciación a la resolución y regular una red de interacción compleja que incluye su propia síntesis y la de sus receptores celulares1. Esta red de comunicación se acoda con una complejidad que va más allá de las relaciones sinérgicas o antagónicas que puedan existir entre los componentes individuales. De hecho, muchas citocinas son conocidos a compartir funciones redundantes o por lo menos parcialmente superpuestos2,3.
Enfoques de Biología de sistemas integrados que cuantifican simultáneamente múltiples analitos del cytokine actualmente ofrecen una comprensión cada vez más amplia de la cytokinomes único que orquestar la respuesta inmune subyacente enfermedad de múltiples Estados4,5. Estos Estados de enfermedad van desde inflamación generalizada y cáncer, las afecciones neuro-degenerativas, enfermedades cardiovasculares6,7,8,9.
Tales redes de citoquinas pueden ser interrogados con eficacia usando inmunoensayos basados en grano LEGENDplex. Estos ensayos se basan en el mismo principio que el sándwich genitals del ensayo (ELISA) y utilizan microesferas codificada de la fluorescencia con anticuerpos de captura covalentemente Unidos a su superficie. Estos anticuerpos se inmovilizan en superficies mucho menores que los requeridos por los formatos tradicionales de ELISA. Esto permite que dichos ensayos se realizará con mucho menos volumen de la muestra, mientras que al mismo tiempo reducir no vinculante y proporcionando multiplexado análisis de varios analitos.
El ensayo descrito en el presente Protocolo utiliza esta tecnología para cuantificar hasta 13 objetivos simultáneamente. Datos de este ensayo pueden obtenerse usando una amplia variedad de citómetros de flujo comúnmente disponibles y a diferencia de otros ensayos disponibles no requieren el uso de instrumentación para análisis específicos dedicado. Con un creciente catálogo de analito validado paneles, estos ensayos se han utilizado en varios proyectos de investigación biomédica10,11,de12,13.
Para demostrar la facilidad y utilidad del formato de ensayo, en este experimento que utilizamos un panel de citoquinas helper T de ratón para cuantificar 13 separar las concentraciones de citoquinas de sobrenadantes de cultivo de tejidos obtenidos de esplenocitos de ratón cultivadas bajo múltiples condiciones estimulantes. Además de sobrenadantes de cultivo de tejidos, este ensayo también se puede realizar utilizando muestras de suero o plasma.
Protocolo
todos los experimentos con animales se realizaron según las recomendaciones de NIH contenidas dentro de la guía para el cuidado y uso de animales de laboratorio y fue aprobados por el IACUC en BioLegend.
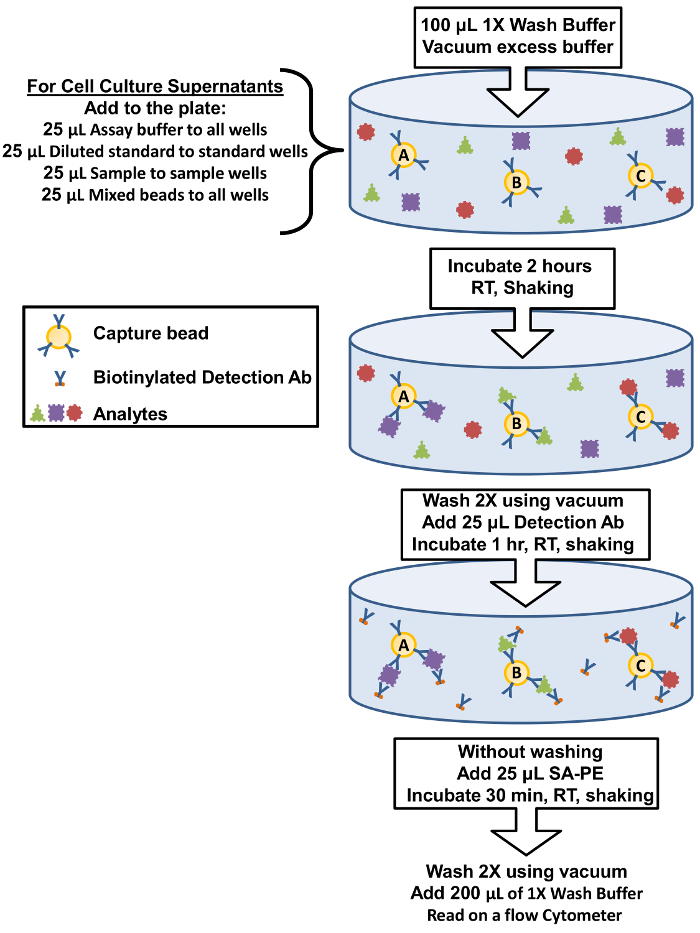
figura 1: Resumen de procedimiento de ensayo de placa de filtro. El protocolo para realizar el análisis utilizando placas de filtro se representa como un diagrama que representa los componentes del ensayo clave y pasos de incubación. haga clic aquí para ver una versión más grande de esta figura.
1. preparación de muestras biológicas
Nota: el sitio anatómico elegido y el volumen de la colección de la sangre queda a criterio de cada investigador individual y no afectará el resultado del análisis. Sin embargo, una vez que se obtienen, las muestras deben ser manipuladas según los pasos descritos a continuación.
- Preparación de las muestras de suero
- recoger el volumen de sangre en tubo de la colección preferida y dejar que coagule durante al menos 30 min.
- Centrifugar la muestra durante 10 min a 1.000 x g a temperatura ambiente.
- Quitar el suero y análisis inmediato o alícuota en tubos de microcentrífuga de polipropileno y almacenar las muestras en ≤ -20 ° C.
- Preparación de las muestras de Plasma
- recoger el volumen de la sangre con ácido etilendiaminotetracético (EDTA) revestido de tubos de sangre.
- Centrifugar por 10 min a 1.000 x g a temperatura ambiente dentro de 30 minutos de colección de.
- Eliminar plasma y ensayo inmediatamente o alícuota en tubos de microcentrífuga de polipropileno y almacenar las muestras en ≤ -20 ° C.
- Preparación de muestras sobrenadante de cultivo de tejidos
- Centrifugue la muestra durante 10 min a 1.500 x g a 4 ° C para eliminar las células y desechos.
- Recoger sobrenadante y análisis inmediato o alícuota en tubos de microcentrífuga de polipropileno y almacenar en el ≤ -20 ° C.
- Deshielo de congelar muestras biológicas
- descongelar completamente cualquier muestras biológicas previamente congeladas en hielo y agitar brevemente antes de su uso. Evitar congelaciones y descongelaciones más de 2.
Nota: Las muestras utilizadas en este protocolo se obtuvieron mediante cultivo de esplenocitos de ratón BALB/c (1 x 10 6 células a 37 ° C + 5% CO 2 en diversas condiciones: sin estimular, LPS (100 ng/mL), αCD3 (1 μg/mL revestido de placa) + αCD28 (1 μg/mL solubilidad), PMA (20 ng/mL) + Ionomycin (500 ng/mL). Sobrenadantes de cultivo fueron recogidos después de 48 h y procesado como se describe en la sección 1.3.
- descongelar completamente cualquier muestras biológicas previamente congeladas en hielo y agitar brevemente antes de su uso. Evitar congelaciones y descongelaciones más de 2.
2. Preparación del reactivo
- Preparation of Pre-mixed Antibody-Immobilized perlas
- someter a ultrasonido premezclados granos botella por 1 min en un baño sonicador a temperatura ambiente y luego vortex 30 antes de s a utilizar. Si no se dispone de ningún baño sonicador, aumentar el tiempo de vórtice a 1 min
- Preparación del tampón de lavado
- traer 20 x Wash Buffer (x 20 PBS con 1% Tween-20) suministrado con el kit a temperatura ambiente y agitar para poner todas las sales en solución.
- Diluir 25 mL de 20 x Wash Buffer con 475 mL de agua desionizada. Porción no utilizada de almacén entre 2 ° C y 8 ° C hasta por un mes.
- Preparación de la matriz B (para el uso con muestras de Plasma y suero)
- añadir 5,0 mL de la solución de ensayo (PBS con 1% BSA) el juego de la botella que contiene el liofilizado matriz B. permitir por lo menos 15 min para reconst completa itution, entonces vortex para mezclar bien. B restos de matriz reconstituido puede almacenarse en ≤-70 ° C hasta por un mes.
3. Preparación estándar
Nota: cada analito en este panel tiene una superior concentración estándar de 10.000 pg/mL.
- Añadir 250 μl de tampón de ensayo para reconstituir el liofilizado ratón de Cytokine estándar cóctel. Mezclar por Vortex brevemente y dejar el frasco permanezca a temperatura ambiente durante 10 minutos
- Transferir el cóctel estándar a un tubo de microcentrífuga de polipropileno etiquetado " C7 ". Esto se utilizará como estándar arriba.
- Etiqueta de 6 tubos de microcentrífuga de polipropileno como C6, C5, C4, C3, C2 y C1.
- Añadir 75 μl de tampón de ensayo para cada uno de estos tubos.
- De transferencia 25 μl del C7 estándar superior al tubo de C6 y mezcla bien con un vórtex. Este será el estándar C6.
- Continuar realizando serie punta de 1:4 diluciones utilizando una nueva pipeta para que cada tubo añadir 25 μl del estándar anterior a la 75 μl de tampón de ensayo en el tubo estándar más bajo siguiente seguido de Vortex para obtener normas C5, C4, C3 , C2 y C1. Usar el tampón de ensayo como el estándar de 0 pg/mL (C0).
4. Dilución de la muestra
Nota: experiencias piloto preliminares con este ensayo de diluciones múltiples pueden ser necesarias para determinar el factor de dilución más apropiado para un conjunto particular de muestras biológicas. Un factor de dilución adecuado produce cálculos de concentración de la muestra que se encuentran dentro de los límites de la curva estándar. Los pasos siguientes se significan como directrices y tenga que determinarse empíricamente según tipo de muestra.
- Diluir suero o plasma de 2 muestras con tampón de ensayo (e.g. diluir 50 μl de muestra con 50 μl de tampón de ensayo) en tubos de microcentrífuga de polipropileno.
Nota: Si necesita más dilución de la muestra, diluciones deben hacerse con matriz B en vez de tampón de ensayo para asegurar una medición precisa. Agregar las muestras de suero o plasma sin dilución resultarán en la precisión de análisis bajo y pueden obstruir la placa del filtro. Matriz B se compone de suero de ratón combinados agotado de objetivos análisis endógeno. Se utiliza como una muestra de suero o plasma diluyente para evitar efectos de matriz, que se saben que la sensibilidad analítica del efecto de inmunoensayos. - Prueba de muestras sobrenadante de cultivo sin dilución de la célula.
Nota: Los niveles del analito pueden variar mucho de muestra a muestra. Si es necesario, diluir las muestras sobrenadante usando una preparación fresca de su correspondiente medio de cultivo celular o tampón de ensayo.
5. Procedimiento de ensayo
Nota: el ensayo puede realizarse en placas de filtro de polipropileno, tubos de micro FACS o microplacas de fondo V. El procedimiento de ensayo de placa de filtro se recomienda debido a muestras de buena consistencia, ensayo de robustez y facilidad de manejo. Este procedimiento requiere una unidad de filtración de vacío para el lavado (proporcionado por el usuario final).
- Permita que todos los reactivos a temperatura ambiente (20-25 ° C) antes de uso.
- Conjunto de la placa del filtro en una cubierta de placa invertida en todo momento durante los pasos de la instalación e incubación de la prueba, para que la parte inferior de la placa no toque ninguna superficie ya que puede ocasionar fugas.
- Mantener la placa en posición vertical durante el procedimiento de ensayo completo, incluyendo los pasos de lavado, para evitar perder granos.
- Mantener la placa en el oscuro o envuelto con papel de aluminio para todos los pasos de incubación.
- Ejecutar todos los estándares y las muestras como duplicados, dispuestos en la placa en un orden secuencial.
- Humedezca previamente la placa del filtro mediante la adición de 100 μl de 1 x de tampón de lavado para cada bien y dejar reposar 1 min a temperatura ambiente (si está usando microplacas filtro de fondo).
- Volumen de buffer de quitar mediante el uso de los colectores de vacío (5-10 s). No exceda 10 " Hg de vacío. Lavado de exceso de Blot Almacenador intermediario de la parte inferior de la placa presionando la placa en una pila de toallas de papel limpias. Coloque el plato encima de la cubierta de la placa invertida.
Nota: Los pasos 5.1-5.2 pueden omitirse si se realiza el ensayo con un V-microplacas o micro FACS tubos. - Para muestras sobrenadante de cultivo de células, añadir 25 μl de tampón de ensayo a todos los pocillos. Añadir 25 μl de cada estándar a los pozos estándar. Añada 25 μl de cada muestra en los pocillos de la muestra.
- Para la medición de muestras de suero o plasma, añada 25 μl de la matriz B en los pozos estándar. Añada 25 μl de tampón de ensayo a los pocillos de la muestra. Añadir 25 μl de cada estándar a los pozos estándar. Añada 25 μl de cada suero diluido o muestra de plasma a los pocillos de la muestra.
- Vortex mezclados granos para 30 s. Añadir 25 μl de granos mezclados en cada pocillo, sacudiendo la botella grano intermitentemente para evitar el asentamiento del grano. El volumen final debe ser 75 μL en cada pocillo después de la adición de granos de.
- Selle la placa con un sellador de placas. Envuelva toda la placa, incluyendo la cubierta de la placa invertida, con papel de aluminio. Colocar la placa sobre un agitador de placa, fijarlo y agitar a aproximadamente 500 rpm durante 2 h a temperatura ambiente. Para evitar fugas, no aplique presión positiva el sellador de placa.
- Sin invertir, colocar la placa en el colector de vacío y aplique vacío como antes y añadir 200 μL de 1 x de tampón de lavado para cada bien.
- Sacar el contenido de los pocillos de la placa de ensayo de filtración de vacío. Secar el tampón de lavado de exceso de la parte inferior de la placa con una almohadilla absorbente o toallas de papel. Repita este paso una vez más.
Nota: Si el ensayo se realiza utilizando un FACS V fondo microplacas o micro tubos saltar pasos 5.12 y 5.13. En su lugar centrifugar la placa a 1.000 x g durante 5 min a temperatura ambiente, luego retire el sobrenadante con una pipeta multicanal. - Añadir 25 μl de anticuerpos de detección (consulte la tabla de materiales) a cada pocillo.
- Selle la placa con un sellador de placas frescas. Envolver toda la placa, incluyendo la cubierta de la placa invertida, con papel de aluminio.
- Colocar la placa sobre un agitador de placa y agitar a aproximadamente 500 rpm durante 1 h a temperatura ambiente.
- Sin aspirar, añadir 25 μl del reactivo de SA-PE directamente a cada pocillo. No dilución del reactivo es necesario.
- Selle la placa con un sellador de placas frescas. Envolver toda la placa, incluyendo la cubierta de la placa invertida, con papel de aluminio.
- Colocar la placa sobre un agitador de placa y agitar a aproximadamente 500 rpm por 30 min a temperatura ambiente.
- Repita el paso anterior 5.13.
- Añadir 200 μL de 1 x de tampón de lavado para cada pozo. Vuelva a suspender las cuentas en un agitador de placa durante 1 minuto
- Utilizando una pipeta multicanal, transferir muestras de la placa de filtro a los tubos para leer las muestras en un citómetro de flujo FACS.
Nota: Volumen de muestra puede aumentarse de 200 μl a 300 μL añadiendo un extra de 100 μl de 1 x de tampón de lavado a cada tubo para evitar que la muestra de funcionamiento en seco. Si es necesarias muestras pueden ser almacenadas a 4 ° C protegido de la luz y analizados al día siguiente. Sin embargo, almacenamiento prolongado de la muestra puede llevar a la menor señal.
6. Configuración de citómetro de flujo
Nota: para generar datos fiables, el citómetro de flujo debe configurarse correctamente antes de adquisición de datos. Este proceso varía para los usuarios individuales en algunos relación dependiendo de la configuración del instrumento particular que el ensayo se realiza en. Un proceso de instalación generalizada para un citómetro capaz de medir PE y APC y equipado con ambos un 488 y 633 nm láser se analiza más adelante.
- Poner en marcha el flujo citómetro y adquisición de software según el fabricante ' instrucciones suministradas con el instrumento de.
- Crear una trama de punto con FSC (forward scatter) en el eje x y el SSC (dispersión de lado) para el eje y. Establece FSC y SSC en modo linear.
- Crear una segunda trama de punto con el PE en el eje x y el APC para el eje y. Este terreno debe configurarse como un modo de visualización basado en el registro de.
- Vortex el frasco de granos crudos incluidos en el kit de 30 s para volver a suspender las cuentas. Estos granos contienen un colorante interno de APC y se componen de dos poblaciones de tamaño.
- Transfiera 400 μL de los granos crudos a un tubo nuevo de FACS.
- Ajustar la velocidad de flujo citómetro de flujo a baja.
- Ejecutar los granos crudos, cuidadosamente ajustar la ganancia y la tensión de FSC y SSC para ambas poblaciones de tamaño de estos granos son visiblemente separadas y fáciles de puerta.
- Ajustar el umbral de la FSC para excluir los eventos no deseados (es decir, burbujas de aire o desechos).
- Parcela en el FSC y SSC, dibujar una puerta que incluye todas las poblaciones de grano.
Nota: Los granos crudos contienen dos poblaciones de tamaño de los granos, el más pequeño " un granos de " y el más grande " grano B " regiones. - Mostrar las poblaciones de grano cerrado de la parcela FSC y SSC en la segunda trama de punto con PE en el eje x y el APC para el eje y.
- Ajustar la tensión de la PMT para el canal de fluorescencia de APC para que la señal APC para todas las poblaciones del grano tiene una intensidad de fluorescencia media (IMF) que se encuentra entre 1 x 10 1 y 5 x 10 3.
- Vortex el frasco de configuración PE cuentas por 30 s para volver a suspender las cuentas.
- Cuentas de transferencia 400 μL del PE a un tubo nuevo de FACS.
- Tubo de perlas de reemplazar los granos crudos del tubo desde el citómetro de flujo con el PE.
- Ajustar la tensión del tubo (PMT) fotomultiplicador para el PE fluorescencia canal ajuste para que la IMF de las cuentas PE cae entre el rango específico del lote encontrado aparece en el frasco de cuentas PE.
Nota: Las cuentas de instalación PE contienen granos del tamaño de una población (" cuentas de un " solamente).
7. Adquisición de datos
Nota: los procedimientos exactos asociados a la adquisición de datos en un instrumento dado pueden variar y dependen en el citómetro ' Especificaciones de configuración de s y el software de interfaz utilizado. Las siguientes instrucciones por lo tanto, se pretenden destacar los pasos requeridos en el análisis sin importar el citómetro empleado en el análisis.
- Verificar el citómetro de flujo se encuentra aún a la baja.
- Establece el número de eventos de grano adquirido a cerca de 300 por analito. Para un 13-plex panel esto equivale a adquirir 3.900 eventos combinada de ambas poblaciones de tamaño de grano (granos de A + B).
- Vortex cada muestra 5 s antes del análisis.
- Leer las muestras. Al leer las muestras, fijar el citómetro de flujo para el modo de configuración primero y espere hasta que se estabiliza la población de grano antes de cambiar al modo de adquisición.
- Usar nombres simples con numeración consecutiva para archivos de datos para facilitar el análisis de datos.
- Exportar sólo los eventos cerrados (A + B grano regiones) en lugar de total de eventos.
- Almacenar todos los archivos de FCS en la misma carpeta para cada ensayo. Si ejecuta ensayos múltiples, crear una carpeta independiente para cada ensayo.
Nota: los archivos de la FCS generados en el citómetro de flujo deben ser analizados usando el software de análisis de datos, que puede descargarse gratis 14.
- Instalar el software de análisis de datos en un PC.
- Transferir todos los archivos de ensayo FCS a la computadora que contiene el software de análisis.
- Conecte el dongle de clave de licencia (incluido en el kit) en un puerto USB del ordenador.
- Iniciar el software de análisis de datos.
- Haga clic en el azul " agregar archivos " botón situado en la parte superior de la pantalla.
- Vaya a la carpeta que contiene los archivos de la FCS de la prueba en el pop-up ventana que aparece.
- Haga clic en y arrastre todos los archivos de ensayo FCS del emergente ventana a la pantalla de software. Todos los archivos ahora deben aparecer en una lista de.
- Haga clic en el verde " siguiente " botón en la parte inferior derecha de la pantalla. Haga clic y mantenga pulsado para arrastrar el pequeño curva estándar azul botones (C7 a C0) a sus correspondientes archivos de FCS de la lista para definir la curva estándar.
- Haga clic en el verde " siguiente " botón en la parte inferior derecha de la pantalla para abrir el bloquea pop up ventana.
- En el lado izquierdo de la ventana bloquea escriba los nombres de ambos el A y regiones de grano B análisis objetivos de analito y su ID de asociado del grano (que se encuentra en el manual suministrado con el ensayo) en orden ascendente.
- Seleccionar la herramienta bloquea desde la parte superior de la pop-up ventana y utilizarlo para dibujar 2 puertas en la parcela FSC y SSC, alrededor de los granos de A y un segundo alrededor de los granos de B.
- Revisar la APC vs PE scatter diagramas que aparecen a continuación de la trama FSC y SSC que aparecen. Habrá 6 bandas de analito en las cuentas de A (diagrama izquierdo) y 7 bandas de analito en las cuentas de B (diagrama derecho).
Nota: Gates puede redibujarse manualmente seleccionando la herramienta de borrador en la parte superior y haga clic en para eliminar una determinada puerta. La herramienta bloquea puede luego ser seleccionada y aplicar manualmente una puerta a la región de banda deseada. - Haga clic en el verde " OK " botón para cerrar ventana emergente bloquea y volver a la lista de archivos FCS.
- Definir cualquier diluciones que fueron hechas a las muestras biológicas por derecho haga clic en un archivo determinado, seleccionando " dilución doble " en el menú y entrada el valor correcto.
- Haga clic en el verde " ejecutar " en la parte inferior derecha de la pantalla para generar las curvas estándar para el análisis y calcular las concentraciones de las muestras biológicas desconocidas.
Resultados
Este protocolo muestra cómo se puede utilizar una plataforma basada en grano de inmunoensayo para cuantificar simultáneamente perfiles del cytokine de muestras biológicas mediante un citómetro de flujo. Las muestras biológicas utilizadas en este ejemplo fueron sobrenadantes de cultivo celular de esplenocitos de ratón que habían sido previamente incubados bajo varias condiciones de activación, aunque el formato de ensayo también ha sido validado para su uso con muestras de suero o plasma.
Datos generados por el ensayo se utilizan para construir curvas estándar para todos los analitos en el panel multiplexado. El software de análisis de datos clasifica cada analito particular basado en un tamaño único del grano y la intensidad de la APC y cuantifica a través de un reportero de PE. Los rangos dinámicos, así como la sensibilidad del ensayo se demostró cuando se grafica en una escala log-log en la figura 2A. El software utiliza el análisis de datos y un algoritmo de ajuste curva de 5 parámetros para calcular con precisión no sólo las concentraciones de analitos en usuario proporcionado muestras biológicas pero también los límites de detección en el extremo alto y bajo de todas las curvas estándar (tabla 1 ). El formato descrito en el presente Protocolo también proporciona precisión de ensayo altamente robusto. En la figura 2B y 2C se presentan datos que representan la variabilidad intraensayo de cada analito. Dos concentraciones de proteína estándar independiente (alta y baja) fueron analizadas en un ensayo con 16 repeticiones para cada muestra. Figura 2 y 2D representan la variabilidad interensayo de analitos de tres ensayos independientes. Como con los experimentos de precisión inter-ensayo, altas y bajas concentraciones estándar se analizaron 3 réplicas en cada uno de los experimentos independientes. Mínima variabilidad en la concentración calculada de proteínas objetivo es claramente visible.
Para demostrar la utilidad de análisis cytometric basados en grano en interrogar a perfiles de expresión de citoquinas, esplenocitos de ratón fueron incubados bajo varias condiciones de activación. Además de un control sin estimular, las células también se incubaron con anticuerpos anti-CD3/anti-CD28, o forbol 12-miristato 13-acetato, LPS y ionomycin. Sobrenadantes de cultivo celular se recolectaron 48 h más tarde y ensayado mediante el panel de citoquinas helper T de ratón. Los resultados de la cuantificación de este experimento están representados en la figura 3 y el efecto de las condiciones de estimulación sobre las concentraciones de citoquinas está claramente delineado.
Los resultados descritos anteriormente son típicos, como el experimentador tiene técnica de laboratorio buena y sigue el protocolo de ensayo proporcionado. Fuentes de error en este formato de análisis pueden surgir de desviaciones en los tiempos de incubación, volúmenes de reactivo, se lava, o de la incorrecta configuración de PMT en el citómetro de flujo utilizado para analizar las muestras. En la Figura 4A una dispersión de un ensayo exitoso muestra 2 poblaciones distintas de grano que están claramente definidas. Esto puede compararse a Figura 4B, donde han llevado mala adherencia lavado y Protocolo a agregados de grano que difuminan las dos poblaciones. Además, según lo evidenciado por los escombros en el cuadrante izquierdo inferior, citómetro total eventos fueron exportados para análisis más bien que el formato preferido de sucesos de barrio cerrados. Citómetro apropiado configurar es fundamental para generar datos fiables en este ensayo. De PMT correcta figura 4 configuración permite la resolución de cada uno de los 6 analitos en el canal de clasificación de APC. Incorrecta configuración de PMT dará como resultado datos similares a la presentada en la figura 4 , donde el canal APC tiene poblaciones de grano con intensidades superpuestas de clasificación. Esto invalidará el ensayo ya que será imposible calcular las concentraciones de analito sin poblaciones claramente definidas de granos de ensayo.
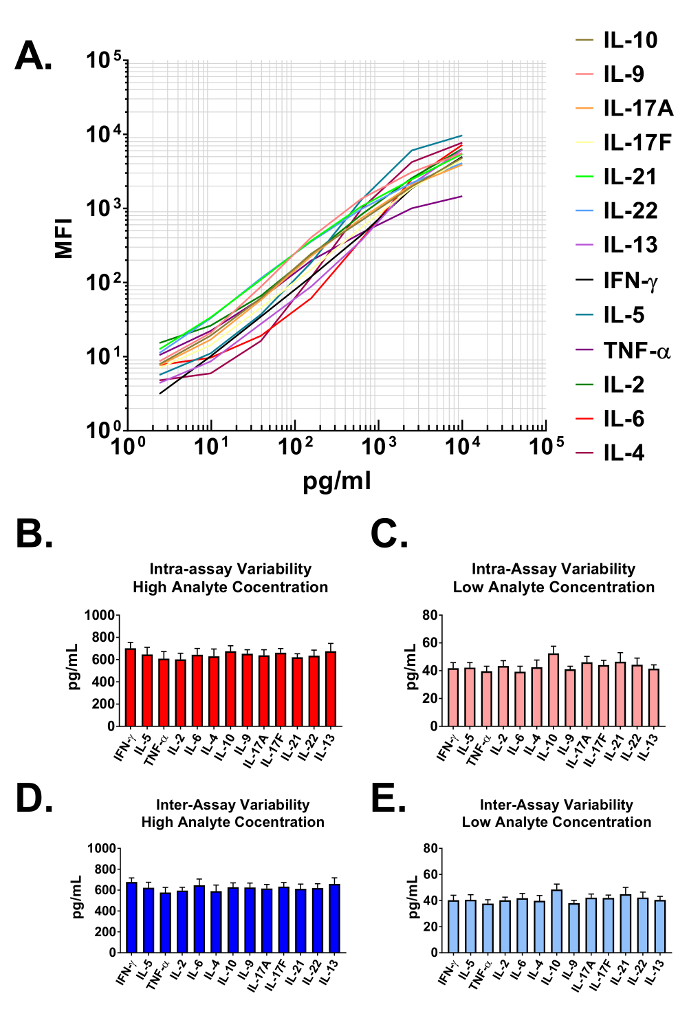
Figura 2: rangos de la curva estándar y precisión intraensayo. (A) una curva estándar representativa mediante el panel de Mouse T helper citoquinas. Una curva estándar se debe ejecutar con cada ensayo. (B-C) Precisión inter-ensayo. Se analizaron dos muestras con diferentes concentraciones de proteínas de la blanco (alta y baja) en un ensayo con 16 repeticiones para cada muestra. Los datos se representan como la media + desviación estándar. (D-E) Precisión inter-ensayo. Se analizaron dos muestras con diferentes concentraciones de proteínas de la blanco (alta y baja) en tres ensayos independientes con 3 repeticiones para cada muestra. Los datos se representan como la media + desviación estándar. Haga clic aquí para ver una versión más grande de esta figura.
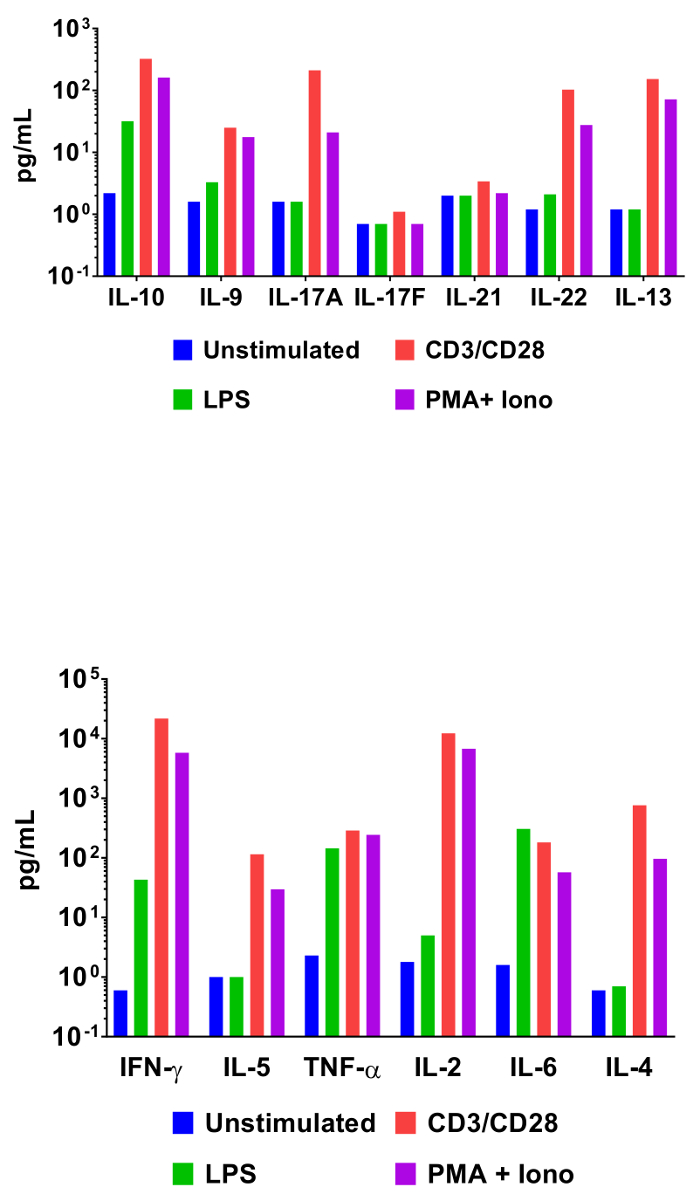
Figura 3: cuantificación de resultados representante. Esplenocitos de ratón (1 x 106 células) fueron cultivadas a 37 ° C + 5% CO2 en diversas condiciones: sin estimular, LPS (100 ng/mL), αCD3 (1 μg/mL revestido de placa) + αCD28 (1 μg/mL solubilidad), PMA (20 ng/mL) + Ionomycin (500 ng/mL). Sobrenadantes de cultivo fueron recogidos después de 48 h luego cuantificaron mediante el panel de citoquinas helper T de ratón. Perfiles de expresión diferencial de citocinas en respuesta a las condiciones de estimulación son claramente visibles. Haga clic aquí para ver una versión más grande de esta figura.

Figura 4: resultados de éxito vs ensayos fracasados. (A) pruebas acertados tendrán poblaciones de distinto grano para las poblaciones de microesfera. (B) ensayos fracasados tendrán separación de la población pobre, agregados de grano y demasiados eventos recogidos. (C) pruebas acertados han definido claramente intensidades en el canal de fluorescencia de clasificación que son fáciles de puerta y cuantificar. (D) ensayos infructuosos habrán mal separado y clasificación de intensidades que se superponen varias poblaciones de grano, haciendo difícil la cuantificación. Muchos de estos errores pueden evitarse a través del cuidado de adhesión al Protocolo de ensayo. Haga clic aquí para ver una versión más grande de esta figura.
| Curva estándar | Sensibilidad (pg/mL) | ||||
| Analito | Fórmula de ajuste | CV | R2 | Min. | Max. |
| IFN-Γ | 5-P.log(4.43, 14.40, 0.54, 9.11, 0.01) | 1.77% | 0.99 | 2.1 | 18105.0 |
| IL-5 | 5-P.log(4.36, 12.68, 0.64, 5.73, 0.36) | 1.32% | 1 | 1.9 | 14514.0 |
| TNF-Α | 5-P.log(4.36, 11.63, 0.79, 5.45, 0.37) | 1.48% | 0.99 | 1.9 | 20109.0 |
| IL-2 | 5-P.log(4.46, 12.62, 0.78, 5.07, 0.34) | 1,34% | 1 | 1.9 | 25109.0 |
| IL-6 | 5-P.log(4.58, 11.66, 0.64, 5.77, 0.37) | ||||
Tabla 1: ajuste de curvas estándar y sensibilidad del ensayo. El software de análisis de datos se utilizó para generar curvas estándar para todos los analitos en el ensayo multiplex. Este análisis completamente automatizado aplica una robusta 5 parámetro ajuste de la curva algoritmo a las normas de la serie de dilución de ensayo y también fue usado para definir la sensibilidad del ensayo con los límites teóricos de detección.
Discusión
Puede proporcionar el formato de ensayo descrito en el presente Protocolo robusta cuantificación de perfiles mediador soluble en muestras biológicas que al experimentador tiene técnica de buen laboratorio y cumple con el protocolo recomendado.
Hay varios pasos fundamentales que deben seguirse para garantizar resultados confiables. En primer lugar, las normas recombinantes se debe reconstituir en tampon de ensayo el tiempo completo recomendado y después sólo diluida en tubos de polipropileno. Tubos de poliestireno pueden promover la adherencia de las proteínas en la pared de los tubos, que pueden conducir a las señales de baja cuando se genera la curva estándar. En segundo lugar, agitando durante los pasos de incubación adecuada es fundamental. Sin agitación adecuada, las características de unión de los granos de ensayo pueden ser seriamente obstaculizadas. Además, el lavado apropiado entre los pasos de incubación también se requiere. El experimentador debe asegurarse de que exceso reactivos se extraen de los pozos antes de comenzar el siguiente paso en el protocolo. Configuración de instrumentos para el citómetro de flujo utilizado para cuestionar las cuentas de ensayo debe optimizarse así. En particular, valores de PMT se deben ajustar para asegurar la separación adecuada del grano y amplio rango dinámico de las curvas estándar. Finalmente, justo antes de que se analizan en el citómetro, granos debe agitarse para asegurar la suspensión adecuada y para evitar la formación de agregados que pueden sesgar los resultados.
Potencialmente, este protocolo puede ser modificado para analizar homogenados de tejido, así como los tipos de muestra en este manuscrito, siempre y cuando el investigador está dispuesto a optimizar su Protocolo de lisis antes de realizar ensayos de placa grande, completo. La técnica real por supuesto variará dependiendo del tipo de tejido; sin embargo, en general el protocolo debe usar un tampón de pH neutro que contiene concentraciones fisiológicas de sales iónicas, ningunos productos químicos desnaturalización y preferiblemente sin detergentes. Si se debe usar detergente, concentraciones de detergente no iónico deben mantenerse a un mínimo (por ejemplo, no más del 1%). El búfer debe contener también inhibidores de la proteasa suficiente para evitar la degradación proteolítica de proteínas de la blanco. Sin importar el protocolo de lisis utilizado, los preparativos finales deben centrifugarse para retirar las partículas antes del análisis.
En cuanto a limitaciones del análisis, las concentraciones de analito objetivos tipos de muestra biológica pueden variar mucho. Para cuantificar adecuadamente las concentraciones de analito, debe caer dentro de los valores superior e inferior de la curva estándar. Extrapolación de los valores de la curva no es necesariamente confiable y no se recomienda. Los investigadores por lo tanto deben optimizar las diluciones de las muestras de concretas en experiencias piloto antes de ejecutar un análisis completo de la placa. Además, la cuidadosa preparación de las muestras biológicas a ensayarse es fundamental para el éxito de este formato de ensayo. Las muestras lipémicas, hemolizadas o contienen residuos de proteínas afectan las interacciones de anticuerpos de antígenos que definen el principio fundamental de este inmunoensayo y hacer que los resultados no interpretables.
Este análisis es importante con respecto a los métodos existentes de cuantificación por varias razones. Cuando se ejecuta correctamente, el formato de ensayo puede cuantificar hasta 13 analitos de una muestra usando volúmenes mucho más pequeños que sería necesario analizar la muestra utilizando tradicionales ensayos de ELISA. Este formato de ensayo también no requiere instrumentación dedicada para llevar a cabo. Esto es una ventaja en comparación con otros inmunoensayos basados en grano disponibles en el mercado como el ensayo se puede ejecutar utilizando cualquier número de citómetros de flujo utilizado.
Investigación científica sobre el papel había jugado por citoquinas y factores secretados redes en salud y la enfermedad se está expandiendo. El formato de ensayo descrito en este manuscrito puede ser una herramienta valiosa para los investigadores tratando de entender estos fenómenos complejos y multifacéticos. Programas activos de investigación biomédica están comenzando a explorar el papel de las redes de citocinas en no sólo áreas generalizadas tales como inflamación6, sino también en el contexto de Estados de enfermedad específica como la aterosclerosis, el cáncer y la neuroinflamación 1516,de,17. Sin duda, investigación seguirá creciendo en estas áreas como la búsqueda de nuevas terapias tratar estas condiciones crónicas se expande. La necesidad de cuantificación precisa y fiable de los mediadores solubles asociada a estas enfermedades seguirá siendo una prioridad de investigación crítica.
Divulgaciones
Los autores son empleados de BioLegend, que fabrica los reactivos descritos en este manuscrito. Vigene Tech, Inc. desarrolló el software de análisis de datos LEGENDplex que se utiliza para analizar los datos generados por el ensayo descrito en este manuscrito.
Agradecimientos
Los autores quisieran agradecer las contribuciones de los inmuno-ensayos y equipo de desarrollo de producto para el desarrollo del ensayo. Además, nos gustaría agradecer a Vigene Tech, Inc. por su colaboración en el diseño de los paquetes de software de análisis de datos.
Materiales
| Name | Company | Catalog Number | Comments |
| Allegra 6R Centrifuge with MICROPLUS Carrier Adaptor | Beckman Coulter | 366816 | For LEGENDplex assays is run in microtubes, V-or U-bottom 96-well plate (optional) |
| LEGENDplex Mouse T helper Cytokine Panel | BioLegend | 740005 | Multiplex Immunoassay (13-plex) |
| Anti-mouse CD3ε antibody | BioLegend | 100314 | Clone 145-2C11, low endotoxin, azide free format |
| Anti-mouse CD28 antibody | BioLegend | 102112 | Clone 37.51, low endotoxin, azide free format |
| Vacuum Pump | EMD Millipore | WP6111560 | For LEGENDplex assays using filter plates (recommended) |
| Vacuum Manifold | EMD Millipore | MSVMHTS00 | For LEGENDplex assays using filter plates (recommended) |
| Sterile Disposable Reagent Reservoirs | Fisher Scientific | 07-200-130 | Or equivalent |
| Polypropylene MicroFACS Tubes | Fisher Scientific | 11-842-90 | For LEGENDplex assays is run in microtubes, V-or U-bottom 96-well plate (optional) |
| Pipette Kit | Fisher Scientific | 14-388-100 | Four pipette sizes (0.2-2µL, 2-20µL, 20-200µL, 100-1000µL) |
| Multi-Channel Pipette | Fisher Scientific | FA10011G | capable of dispensing 5 μL to 200 μL |
| Microplate Shaker | Fisher Scientific | 88880023 | Or equivalent |
| Microcentrifuge | Fisher Scientific | 75002410 | Or equivalent |
| FACS tubes | Fisher Scientific | NC9885747 | 12 x 75 mm round bottom |
| PMA (Phorbol 12-myristate 13-acetate) | Sigma-Aldrich | P8139 | |
| Lipopolysaccharide (LPS) | Sigma-Aldrich | L-8274 | Escherichia coli O26:B6 |
| Ionomycin | Sigma-Aldrich | I0634 | |
| Branson B200 Ultrasonic Cleaner | Sigma-Aldrich | Z305359 | Or equivalent |
| Microcentrifuge tubes | Sigma-Aldrich | Z666505 | 1.5 mL polypropylene tubes |
| Flow Cytometer | Various | Various | Cytometer equipped with single laser (488nm blue) or two lasers (488nm blue or 532nm green + 633nm Red) capable of reading emission wavelengths at 575nm and 660nm |
| 96-Well Polypropylene Plate | VWR | 82050-662 | For LEGENDplex assays is run in microtubes, V-or U-bottom 96-well plate (optional) |
Referencias
- Stanley, A. C., Lacy, P. Pathways for cytokine secretion. Physiology (Bethesda). 25, 218-229 (2010).
- Nicola, N. A. Cytokine pleiotropy and redundancy: a view from the receptor. Stem Cells. 12, (1994).
- Miyajima, A., Hara, T., Kitamura, T. Common subunits of cytokine receptors and the functional redundancy of cytokines. Trends Biochem Sci. 17, 378-382 (1992).
- Costantini, S., Castello, G., Colonna, G. Human Cytokinome: a new challenge for systems biology. Bioinformation. 5, 166-167 (2010).
- Capone, F., et al. Serum Cytokinome Profile Evaluation: A Tool to Define New Diagnostic and Prognostic Markers of Cancer Using Multiplexed Bead-Based Immunoassays. Mediators Inflamm. , 3064643 (2016).
- Schett, G., Elewaut, D., McInnes, I. B., Dayer, J. M., Neurath, M. F. How cytokine networks fuel inflammation: Toward a cytokine-based disease taxonomy. Nat Med. 19, 822-824 (2013).
- Tse, E., Kwong, Y. L. T-cell lymphoma: Microenvironment-related biomarkers. Semin Cancer Biol. 34, 46-51 (2015).
- Reale, M., Greig, N. H., Kamal, M. A. Peripheral chemo-cytokine profiles in Alzheimer's and Parkinson's diseases. Mini Rev Med Chem. 9, 1229-1241 (2009).
- Vistnes, M., Christensen, G., Omland, T. Multiple cytokine biomarkers in heart failure. Expert Rev Mol Diagn. 10, 147-157 (2010).
- Gobel, K., et al. Blood coagulation factor XII drives adaptive immunity during neuroinflammation via CD87-mediated modulation of dendritic cells. Nat Commun. 7, 11626 (2016).
- Palm, A. K., Friedrich, H. C., Kleinau, S. Nodal marginal zone B cells in mice: a novel subset with dormant self-reactivity. Sci Rep. 6, 27687 (2016).
- Yu, Y., et al. The transcription factor Bcl11b is specifically expressed in group 2 innate lymphoid cells and is essential for their development. J Exp Med. 212, 865-874 (2015).
- Pelly, V. S., et al. IL-4-producing ILC2s are required for the differentiation of TH2 cells following Heligmosomoides polygyrus infection. Mucosal Immunol. 9, 1407-1417 (2016).
- . LEGENDplex Multiplex Assays Available from: https://www.biolegend.com/legendplex (2017)
- Autieri, M. Pro- and anti-inflammatory networks in atherosclerosis. ISRN Vascular Medicine. 2012, (2012).
- West, N. R., et al. Emerging cytokine networks in colorectal cancer. Nat Rev Immunol. 15, 615-629 (2015).
- Becher, B., Spath, S., Goverman, J. Cytokine networks in neuroinflammation. Nat Rev Immunol. 17, 49-59 (2017).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados