Method Article
Multiplex-Zytokin Profilierung der stimulierten Maus Splenocyten mit Hilfe einer durchflusszytometrischen Bead-basierte Immunoassay-Plattform
In diesem Artikel
Zusammenfassung
Dieses Protokoll beschreibt die Quantifizierung der mehrere Zytokin Ziele gleichzeitig in Gewebekultur Überstände von stimulierten Maus Splenocyten Multiplex-Perle-basierte Immunoassay-Plattform mit einem Durchflusszytometer gesammelt.
Zusammenfassung
Perle-basierten Immunoassays beschäftigen das gleiche Grundprinzip als Sandwich-Immunoassays. Perlen, die unterscheiden sich durch Größe und interne Allophycocyanin (APC) Fluoreszenz-Intensität, sind Antikörper, die spezifisch für einen bestimmten Analyten konjugiert zu erfassen. Als nächstes wird eine ausgewählte Jury aus definierte Abscheidung Bead Sets mit einer biologischen Probe mit Ziel Analyten spezifisch für die Capture-Antikörper inkubiert. Eine biotinylierte Detektionsantikörper cocktail wird hinzugefügt, was führt zur Bildung von Capture Wulst-Analyten-Detektionsantikörper Sandwiches.
Zu guter Letzt wird Streptavidin-Phycoerythrin (SA-PE) hinzugefügt, die an biotinylierte Erkennung Antikörper, Bereitstellung von fluoreszierenden Signalintensitäten im Verhältnis zur Menge des gebundenen Analyten bindet. Die PE fluoreszierendes Signal der Analyt-spezifische Perlen Regionen wird mit Durchflusszytometrie quantifiziert und die Konzentrationen von bestimmten Analyten werden mit Daten-Analyse-Software und der Standardkurve generiert in der Probe bestimmt.
In diesem Experiment verwenden wir eine Maus T Helfer Zytokin Panel, um gleichzeitig die Konzentration von 13 separate Zytokin-Ziele in Gewebekultur Überstände von Maus Splenocyten kultivierten verschiedene stimulierende Bedingungen gesammelt zu quantifizieren.
Einleitung
In ihrer Rolle als lösliche Signalmoleküle vermitteln Zytokine die hochgradig koordinierten und multifaktorielle Prozesse, die Host Immunreaktionen zu regieren. Sie werden in allen Phasen des entzündlichen Prozesses, von der Initiierung, Auflösung, ausgedrückt und ein komplexes Zusammenspiel-Netzwerk, das eigene Synthese und dazu ihre zellulären Rezeptoren1gehört zu regulieren. Dieses Kommunikationsnetz ist geschichtet mit einer Komplexität, die die synergistischen oder antagonistischen Beziehungen, die eventuell existieren zwischen den einzelnen Komponenten hinausgeht. In der Tat sind viele Zytokine bekannt, überflüssig oder zumindest teilweise überlappenden Funktionen2,3teilen.
Integrierte Systeme Biologie-Ansätze, die gleichzeitig mehrere Zytokin Analyten zu quantifizieren sind derzeit zunehmend umfassenden Einblick in die einzigartige Cytokinomes zur Verfügung, die die Immunantwort mehrere Grunderkrankung zu orchestrieren 4,5erklärt. Diese Krankheit Staaten reichen von generalisierten Entzündung, Krebs, Neuro-degenerativen Erkrankungen und Herz-Kreislauf-Krankheit6,7,8,9.
Diese Zytokin-Netze können effektiv mit LEGENDplex Wulst-basierten Immunoassays abgefragt werden. Diese Tests basieren auf dem gleichen Prinzip wie das Sandwich Enzyme-Linked Immunosorbentprobe Assay (ELISA) und verwenden Fluoreszenz codiert Mikrosphären mit Capture Antikörper kovalent an ihrer Oberfläche befestigt. Diese Antikörper sind auf Flächen, die viel kleiner als die von traditionellen ELISA Formate immobilisiert. Dadurch können solche Tests mit weit weniger Probenvolumen durchgeführt werden, während bei gleichzeitiger Verringerung der unspezifischen Bindung und bietet Analyse von mehreren Analyten gemultiplext.
Der Test in diesem Protokoll beschriebenen nutzt diese Technologie, um bis zu 13 Ziele gleichzeitig quantitate. Daten aus dieser Assay können mit einer Vielzahl von gängigen fließen Cytometers bezogen werden und im Gegensatz zu anderen verfügbaren Tests erfordert nicht die Verwendung von speziellen Assay-spezifische Instrumentierung. Mit einem wachsenden Katalog von validierten Analyten Platten wurden diese Tests in mehreren biomedizinischen Forschung Projekte10,11,12,13verwendet.
Um zu demonstrieren, die Leichtigkeit und den Nutzen des Formates Assay in diesem Experiment, das wir eine Maus T Helfer Zytokin-Fenster verwenden, um quantitate trennen 13 Zytokin-Konzentrationen von Gewebekultur Überstände Maus Splenocyten kultiviert unter mehreren gewonnenen stimulierende Bedingungen. Neben Gewebekultur Überstände kann dieser Test auch mit Serum bzw. Plasma-Proben durchgeführt werden.
Protokoll
alle Tierversuche wurden nach NIH Empfehlungen innerhalb der Leitfaden für die Pflege und Verwendung von Labortieren durchgeführt und durch die IACUC bei BioLegend angenommen wurden.
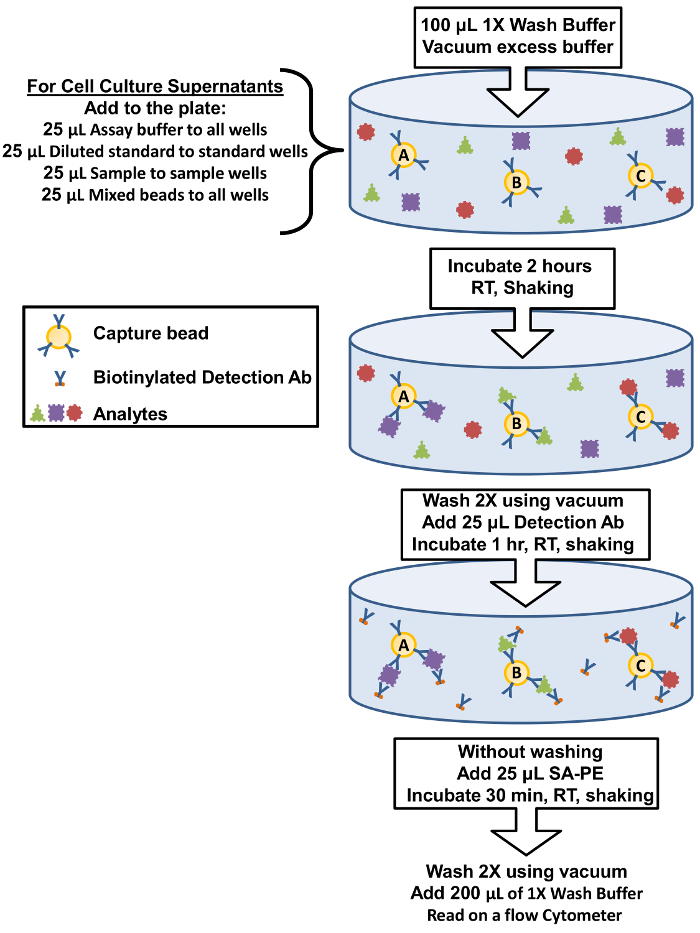
Abbildung 1: Platte-Assay-Verfahren-Zusammenfassung zu filtern. Das Protokoll für die Durchführung des Tests mit Filterplatten wird als Diagramm Darstellung wichtiger Test Komponenten und Inkubationsschritte dargestellt. Bitte klicken Sie hier für eine größere Version dieser Figur.
1. biologische Probenvorbereitung
Hinweis: die gewählte Lokalisation und das Volumen der Blutentnahme bleibt im Ermessen jedes einzelnen Forschers und hat keine Auswirkungen auf das Ergebnis des Tests. Jedoch sobald sie gewonnen werden, Proben behandelt werden entsprechend der unten aufgeführten Schritte.
- Vorbereitung von Serumproben
- sammeln die gewünschte Lautstärke des Blutes in bevorzugte Auflistung Rohr und lassen Sie es für mindestens 30 min. gerinnen
- Zentrifugieren die Probe für 10 min bei 1.000 x g bei Raumtemperatur.
- Serum und Assay sofort oder Aliquote in Polypropylen Mikrozentrifugenröhrchen entfernen und speichern Proben bei ≤-20 ° c
- Plasma-Proben-Vorbereitung
- sammeln die gewünschte Lautstärke des Blutes mit Ethylenediaminetetraacetic Säure (EDTA) beschichtet Blutentnahmeröhrchen.
- Zentrifuge für 10 min bei 1.000 x g bei Raumtemperatur innerhalb von 30 min Sammlung.
- Plasma und Assay sofort oder Aliquote in Polypropylen Mikrozentrifugenröhrchen entfernen und speichern Proben bei ≤-20 ° c
- Vorbereitung der Gewebekultur überstand Proben
- Zentrifugieren die Probe für 10 min bei 1.500 x g bei 4 ° C, Zellen und Schmutz zu entfernen.
- Überstand und Assay sofort oder Aliquote in Polypropylen Mikrozentrifugenröhrchen sammeln und Lagern bei ≤-20 ° c
- Auftauen von gefrorenen biologischen Proben
- komplett alle zuvor fixierten biologischen Proben auf Eis und Wirbel kurz vor dem Gebrauch auftauen. Mehr als 2 Frost/Tau-Zyklen zu vermeiden.
Hinweis: In diesem Protokoll verwendeten Proben stammen von BALB/c Maus Splenocyten Kultivierung (1 x 10 6 Zellen bei 37 ° C + 5 % CO 2 unter verschiedenen Bedingungen: unstimulierte, LPS (100 ng/mL), αCD3 (1 µg/mL Platte beschichtet) + αCD28 (1 µg/mL löslich), PMA (20 ng/mL) + Ionomycin (500 ng/mL). Kultur-Überstände wurden nach 48 h gesammelt und verarbeitet, wie beschrieben in Abschnitt 1.3.
- komplett alle zuvor fixierten biologischen Proben auf Eis und Wirbel kurz vor dem Gebrauch auftauen. Mehr als 2 Frost/Tau-Zyklen zu vermeiden.
2. Reagenz Vorbereitung
- Preparation of Pre-mixed Antibody-Immobilized Perlen
- Sonicate vorgemischte Perlen Flasche für 1 min in einem Sonikator Bad bei Raumtemperatur und dann Wirbel für 30 s vor dem Gebrauch. Wenn kein Sonikator Bad zur Verfügung steht, erhöhen die Wirbel 1 min.
- Vorbereitung der Waschpuffer
- bringen die 20 X Waschpuffer (20 X PBS mit 1 % Tween-20) mitgelieferten auf Raumtemperatur und Wirbel um alle Salze in Lösung zu bringen.
- 25 mL 20 x Waschpuffer mit 475 mL entionisiertem Wasser zu verdünnen. Store unbenutzten Teil 2 ° C bis 8 ° C bis zu einem Monat.
- Vorbereitung der Matrix B für die Verwendung mit Serum und Plasma Proben nur
- 5,0 mL Testpuffer (PBS mit 1 % BSA) enthalten im Kit, um die Flasche mit lyophilisierten Matrix B. erlauben mindestens 15 min für komplette Reconst hinzufügen Itution, dann Wirbel gut mischen. Übrig gebliebene rekonstituierte Matrix B können gespeichert werden, bei ≤-70 ° C bis zu einem Monat.
3. Standard-Vorbereitung
Hinweis: jede Analyten in diesem Panel hat eine Top standard Konzentration von 10.000 Pg/mL.
- Fügen Sie 250 µL Testpuffer die lyophilisierten Maus Th Zytokin Standard Cocktail wiederherzustellen. Mischen von kurz aufschütteln und lassen Sie das Fläschchen für 10 min. bei Raumtemperatur sitzen
- Übertragung den standard-Cocktail zu einem Polypropylen Microcentrifuge Schlauch mit der Bezeichnung " C7 ". Dies wird als standard oben verwendet werden.
- Label 6 Polypropylen Mikrozentrifugenröhrchen als C6, C5, C4, C3, C2 und C1.
- Fügen Sie 75 µL Testpuffer zu jedem dieser Röhren.
- Transfer 25 µL der oberen standard C7 C6 Rohr und Mischung von gut aufschütteln. Der C6-Standard werden.
- Weiter durchführen serielle Tipp 1:4 Verdünnungen mithilfe einer neuen Pipettieren für jedes Rohr 75 µL der Assay-Puffer in die nächste niedrigsten Standardrohr gefolgt von vortexen 25 µL der Vorgängernorm hinzu zu Normen C5, C4, C3 , C2 und C1. Verwenden Sie Testpuffer serienmäßig 0 Pg/mL (C0).
4. Probieren Sie Verdünnung
Hinweis: pilot Vorversuchen mit dieser Assay mit mehrere Verdünnungen erforderlich sein, um den am besten geeigneten Verdünnungsfaktor für einen bestimmten Satz von biologischen Proben zu bestimmen. Eine ordnungsgemäße Verdünnungsfaktor wird Konzentrationsberechnungen für die Probe produzieren, die im Rahmen der Standardkurve liegen. Die folgenden Schritte sind als Richtlinien gedacht und müssen je nach Probentyp empirisch ermittelt werden.
- Verdünnt Serum oder Plasma Proben 2-Fach mit Testpuffer (zB. 50 µL Probe mit 50 µL Testpuffer verdünnen) in Polypropylen Mikrozentrifugenröhrchen.
Hinweis: Wenn weitere Probenverdünnung erforderlich ist, sollten Verdünnungen mit Matrix B statt Testpuffer um genaue Messungen zu gewährleisten erfolgen. Hinzufügen von Serum oder Plasma-Proben ohne Verdünnung führt zu niedrigen Assay Genauigkeit und die Filterplatte verstopfen können. Matrix B besteht aus gepoolten Maus Serum erschöpft von endogenen Assay Zielen. Es dient als Verdünnungsmittel Serum oder Plasma Probe zur Matrix-Effekte zu vermeiden, die den Affekt analytischen Sensitivität von Immunoassays bekannt sind. - Test Handy-Kultur überstand Proben ohne Verdünnung.
Hinweis: Die Ebenen des Analyten variiert von Exemplar zu Exemplar. Überstand Proben mit einer frische Zubereitung ihrer entsprechenden Zellkulturmedium oder Testpuffer verdünnen.
5. Assay-Verfahren
Hinweis: der Test kann in Polypropylen Filterplatten, Mikro FACS Tuben oder V-Boden Mikrotiterplatten durchgeführt werden. Die Filter-Platte-Testverfahren empfiehlt sich wegen guten Probe zu Probe Konsistenz, Assay Robustheit und einfache Handhabung. Dieses Verfahren erfordert eine Vakuumfiltration Einheit zum Waschen (bereitgestellt durch den Endbenutzer).
- Alle Reagenzien auf Raumtemperatur (20-25 ° C) erwärmen vor dem Gebrauch zu ermöglichen.
- Satz der Filterplatte auf einem umgekehrten Teller Cover zu allen Zeiten während der Test-Setup und Inkubation Schritte, so dass die Unterseite der Platte keine Flächen nicht berührt, da es undicht verursachen.
- Die Platte aufrecht zu halten, während das gesamte Testverfahren, einschließlich Waschschritte, um nicht zu verlieren Perlen.
- Halten die Platte in den dunklen oder verpackt mit Alu-Folie für alle Inkubationsschritte.
- Laufen alle Standards und Proben als Duplikate, angeordnet auf der Platte in einer sequentiellen Reihenfolge.
- Durch Zugabe von 100 µL 1 x Waschpuffer in jede Vertiefung der Filterplatte Vornässen und lassen Sie es für 1 min bei Raumtemperatur (bei Verwendung von Filterboden Mikrotestplatte).
- Entfernen Puffervolumen mit dem Vakuum-Verteiler (5-10 s). Nicht mehr als 10 " Hg Vakuum. Tupfen Sie überschüssige Wash Puffern von der Unterseite der Platte durch Drücken der Plattenrandes auf einen Stapel von sauberen Papiertüchern. Platzieren Sie die Platte auf der umgekehrten Platte Deckeloberseite.
Hinweis: Schritte 5.1-5.2 können weggelassen werden, wenn den Test mit einem V-Boden Mikrotestplatte oder Micro FACS Rohre durchführen. - Für Kultur überstand Zellproben, alle Brunnen 25 µL Testpuffer hinzufügen. Die standard Brunnen 25 µL jeder Norm hinzufügen. Die Probe-Brunnen 25 µL jeder Probe hinzufügen.
- Zur Messung von Serum oder Plasma-Proben, die standard Brunnen 25 µL der Matrix B hinzufügen. Die Probe-Brunnen 25 µL Testpuffer hinzufügen. Die standard Brunnen 25 µL jeder Norm hinzufügen. Die Probe-Brunnen 25 µL jeder verdünnte Serum oder Plasma Probe hinzufügen.
- Vortex gemischte Perlen für 30 S. hinzufügen 25 µL gemischte Perlen in jede Vertiefung, schütteln Wulst Flasche zeitweise um Perle, die Abrechnung zu vermeiden. Das Endvolumen sollte 75 µL in jede Vertiefung nach der Zugabe von Perlen werden.
- Dichtplatte mit einer Platte-Versiegelung. Wickeln Sie die gesamte Platte, einschließlich der umgekehrten Platte Abdeckung mit Alu-Folie. Legen Sie die Platte auf Platte Shaker, sichern, und schütteln bei ca. 500 u/min für 2 h bei Raumtemperatur. Um Durchsickern zu verhindern, gelten Sie nicht positiven Druck auf die Platte Sealer.
- Ohne Invertierung, platzieren Sie die Platte auf dem Vakuum Verteiler und Vakuum wie zuvor anwenden und fügen Sie 200 µL 1 x Waschpuffer in jede Vertiefung.
- Entfernen Sie den Inhalt der Assay Platte Brunnen durch Vakuumfiltration. Tupfen Sie überschüssigen Waschpuffer von der Unterseite der Platte mit einer saugfähigen Unterlage oder Küchenpapier. Wiederholen Sie diesen Schritt noch einmal.
Hinweis: Wenn der Test durchgeführt wird-Röhren mit einem V-Boden Mikrotestplatte oder Micro FACS überspringen Schritte 5.12 und 5.13. Stattdessen Zentrifugieren die Platte bei 1.000 x g für 5 min bei Raumtemperatur, dann entfernen Sie den Überstand mit einer Mehrkanal-Pipette. - Fügen Sie 25 µL Erkennung Antikörper (siehe die Tabelle Materialien) in jede Vertiefung.
- Dichtplatte mit einem frischen Teller-Versiegelung. Die gesamte Platte, einschließlich der umgekehrten Platte Abdeckung mit Alu-Folie wickeln.
- Die Platte auf einem Teller-Shaker und schütteln bei ca. 500 u/min für 1 h bei Raumtemperatur.
- Ohne Staubsaugen, fügen Sie 25 µL Reagenz SA-PE direkt in jede Vertiefung. Keine Verdünnung des Reagenz ist notwendig.
- Dichtplatte mit einem frischen Teller-Versiegelung. Die gesamte Platte, einschließlich der umgekehrten Platte Abdeckung mit Alu-Folie wickeln.
- Die Platte auf einem Teller-Shaker und schütteln bei ca. 500 u/min für 30 min bei Raumtemperatur.
- Wiederholen Sie Schritt 5.13 oben.
- Fügen Sie 200 µL 1 x Waschpuffer zu jedem gut. Wieder auszusetzen, die Perlen auf einen Teller Shaker für 1 min.
- Mit einem Mehrkanal-pipettieren, Proben von der Filterplatte auf FACS Röhren Proben auf einem Durchflusszytometer lesen übertragen.
Hinweis: Probenvolumen kann von 200 µL auf 300 µL erhöht werden indem eine zusätzliche 100 µL 1 x Waschpuffer zu jedem Röhrchen, die Probe Trockenlauf zu vermeiden. Wenn nötige Proben bei gelagert werden können 4 ° C lichtgeschützt und analysiert am folgenden Tag. Jedoch verlängerte Probe Lagerung kann zu reduzierten Signal führen.
6. Flow Cytometer Set-up
Hinweis: um verlässliche Daten zu generieren, das Durchflusszytometer muss eingerichtet werden ordnungsgemäß vor der Datenerfassung. Dabei variieren für einzelne Benutzer in mancher Hinsicht abhängig von der Konfiguration des Instruments, der Test durchgeführt wird. Eine generalisierte Setup-Prozess für eine Cytometer PE und APC zu messen und ausgestattet mit den beiden 488 und 633 nm Laser wird besprochen unter.
- Starten Sie die Flow Cytometer und Erwerb Software nach Angaben des Herstellers ' s-Anweisungen mit dem Instrument.
- Erstellen Sie einen Dot-Plot mit FSC (forward Scatter) auf der X-Achse und SSC (Side Scatter) für die Y-Achse. Stellen Sie FSC und SSC auf linearen Modus.
- Erstellen Sie einen zweite Dot-Plot mit PE auf die X-Achse und APC für die Y-Achse. Dieses Grundstück sollte ein Protokoll-basierte Display-Modus eingestellt werden.
- Vortex das Fläschchen des rohen Beads enthalten im Kit für 30 s, die Perlen wieder auszusetzen. Diese Perlen enthalten eine interne APC-Farbstoff und bestehen aus zwei Größe Populationen.
- Transfer 400 µL der rohen Perlen zu einem neuen Schlauch FACS.
- Der Flow Cytometer Durchfluss zu niedrig eingestellt.
- Laufen die rohe Perlen, sorgfältig die Gain- und Spannung für FSC und SSC so angepasst, dass beide Größe Populationen dieser Perlen sichtbar getrennt und leicht zu Tor sind.
- Passen Sie den FSC-Schwellenwert um unerwünschte Ereignisse (z.B. Schmutz oder Luftblasen) auszuschließen.
- In FSC vs. SSC Plot zeichnen ein Tor, das umfasst alle Perlen Populationen.
Hinweis: Die rohe Perlen enthalten zwei Größe Populationen von Perlen, die kleinere " eine Perlenschnur " und die größeren " B Perle " Regionen. - Zeigen die geschlossene Wulst Populationen aus dem FSC vs. SSC-Plot auf dem zweiten Punkt Grundstück mit PE auf die X-Achse und APC für die Y-Achse.
- Die PMT-Spannung für den APC-Fluoreszenz-Kanal so einstellen, dass das APC-Signal für alle Bevölkerungsgruppen Perle eine mediane Fluoreszenzintensität (MFI) hat, das liegt zwischen 1 x 10 1 und 5 x 10 3.
- Vortex das Fläschchen von PE Setup Perlen für 30 s, die Perlen wieder auszusetzen.
- Transfer 400 µL des PE Perlen zu einem neuen Schlauch FACS.
- Ersetzen die rohe Perlen aus dem Durchflusszytometer mit der PE Rohr Perlen u.
- Anpassung die Photomultiplier (PMT) Röhrenspannung für die PE Fluoreszenz Kanal festlegen, so dass die MFI der PE-Perlen zwischen chargenspezifische gefunden fällt auf dem PE-Perlen-Fläschchen aufgeführt.
Hinweis: Die PE-Setup-Perlen enthalten Perlen aus einer Populationsgröße (" eine Perlenschnur " nur).
7. Die Datenerfassung
Hinweis: die genaue Verfahren in Zusammenhang mit der Erfassung von Daten auf einem bestimmten Instrument können variieren und sind abhängig von der Cytometer ' s Konfigurationsspezifikationen und die Schnittstellen-Software verwendet. Die folgenden Anweisungen sollen daher markieren Sie die erforderlichen Schritte im Test unabhängig von der Cytometer beschäftigt in der Probe genommen werden.
- Überprüfen die Durchflussmenge Cytometer befindet sich immer noch zu niedrig.
- Anzahl der Wulst Veranstaltungen bis ca. 300 pro Analyt erworben werden. Für einen 13-Plex Panel, dies entspricht einem Erwerb von 3.900 Veranstaltungen, kombiniert aus beiden Perlen Größe Populationen (A + B Perlen).
- Vortex jede Probe für 5 s vor der Analyse.
- Lesen Sie Proben. Beim Lesen von Proben der Durchflusszytometer zuerst auf Setup-Modus gesetzt und warten, bis Perlenpopulation stabilisiert wird, vor der Umstellung auf Akquisitionsmodus.
- Einfache Namen mit fortlaufende Nummerierung für Datendateien verwenden, um Datenanalyse zu erleichtern.
- Export nur geschlossenen Veranstaltungen (A + B bead Regionen) anstatt Gesamtereignisse.
- Speichern alle FCS-Dateien im selben Ordner für jedes Assay. Wenn mehrere Tests ausführen, erstellen Sie einen separaten Ordner für jedes Assay.
Hinweis: der FCS-Dateien auf das Durchflusszytometer generiert sollte analysiert werden, mit der Datenanalyse-Software, die kostenlos 14 heruntergeladen werden kann.
- Die Datenanalyse-Software auf einem PC zu installieren.
- Alle Assay FCS Dateien auf dem Computer, die Analyse-Software enthält, übertragen.
- Schließen die Lizenz Key Dongle (im Kit enthalten) an einen USB-Anschluss des Computers.
- Starten die Datenanalyse-Software.
- Klicken Sie auf das blaue " Dateien hinzufügen " Taste befindet sich am oberen Rand des Bildschirms.
- Navigieren Sie zu den Ordner mit der FCS-Dateien aus der Probe in die Pop-up-Fenster, das angezeigt wird.
- Klick und ziehen Sie alle Assay FCS-Dateien aus dem Pop-up-Fenster auf die Software-Anzeige. Alle Dateien sollten nun in einer Liste angezeigt.
- Klicken Sie auf die grünen " nächsten " Taste unten rechts auf dem Display. Mit der linken Maustaste und halten Sie, ziehen das kleine blaue Standardkurve Tasten (C7, C0) in ihre entsprechenden FCS-Dateien aus der Liste die Standardkurve definieren.
- Klicken Sie auf die grünen " nächsten " Taste unten rechts auf das Display, die gating Pop-up-Fenster zu öffnen.
- Auf der linken Seite des Fensters gating geben Sie die Namen der beiden A und B Wulst Regionen assay Analyten Ziele und ihre damit verbundenen Wulst-ID (gefunden in der Bedienungsanleitung im Lieferumfang der Assay) in aufsteigender Reihenfolge.
- Wählen Sie die gating-Werkzeug von der Spitze der Pop-up Fenster und verwenden, um 2 Tore in der FSC vs. SSC-Handlung, eine rund um die A-Perlen und eine zweite um die B-Perlen ziehen.
- Überprüfen die APC vs. PE Scatter plots, die unterhalb der FSC vs. SSC-Plot, die angezeigt werden. Werden 6 Analyten Bänder in den A-Perlen (linke Plot) und 7 Analyten Bänder in den B-Perlen (richtige Handlung).
Hinweis: Tore können manuell neu gezeichnet werden, indem Sie das Radiergummi-Werkzeug oben auswählen und klicken, um ein bestimmtes Tor zu löschen. Das gating Tool kann dann ausgewählt und verwendet, um ein Tor manuell auf die gewünschte Band Region anwenden. - Klicken Sie auf den grünen " "OK" "-Taste, um die gating Pop-up-Fenster schließen und zurück zur Liste der FCS Dateien.
- Definieren alle Verdünnungen, die biologischen Proben von rechts anklicken einer bestimmten Datei, Auswahl vorgenommen wurden " Verdünnung Falten " aus dem Menü und Eingabe der richtige Wert.
- Klicken Sie auf die grünen " laufen " unten auf die Schaltfläche rechts des Displays zu generieren die Standardkurven zum Test und berechnen die Konzentrationen von unbekannten biologischen Proben.
Ergebnisse
Dieses Protokoll wird veranschaulicht, wie eine Perle-basierte Immunoassay-Plattform verwendet werden kann, um gleichzeitig Cytokine Profile aus biologischen Proben mit einem Durchflusszytometer quantitate. Biologischen Proben, die in diesem Beispiel verwendeten waren Zelle Kultur Überstände von Maus Splenocyten, die zuvor verschiedene aktivierende Bedingungen inkubiert worden waren, obwohl der Assay-Format auch für die Verwendung mit Serum oder Plasma Proben validiert wurde.
Von der Assay generierte Daten werden verwendet, um Standardkurven für alle Analyte im Bedienfeld "Multiplex" zu konstruieren. Die Datenanalyse-Software jedes bestimmten Analyten basierend auf eine einzigartige Perle Größe und Intensität der APC klassifiziert und quantifiziert mit einem PE-Reporter. Die dynamische Bereiche sowie die Empfindlichkeit des Tests werden beim Plotten auf einer Log-Log-Skala in Abbildung 2Ademonstriert. Die Software nutzt Assay-Daten und eine 5-Parameterkurve passenden Algorithmus, um genau zu berechnen, nicht nur die Konzentration des Analyten in der Benutzer zur Verfügung gestellt biologische Proben, sondern auch der Nachweisgrenze auf die hohen und niedrigen Ende alle Standardkurven (Tabelle 1 ). In diesem Protokoll beschriebene Format bietet auch hochstabile Assay-Präzision. In Abbildung 2 b und 2 C werden Daten präsentiert die Intra-Assay-Variabilität der einzelnen Analyten Darstellung. Zwei separate standard Proteinkonzentrationen (hoch und niedrig) wurden in einem Test mit 16 Wiederholungen für jede Probe analysiert. Abbildung 2 und 2D stellen die Inter-Assay-Variabilität der Ziel Analyten von drei unabhängigen Tests. Wie mit den Intra-Assay-Präzision-Experimenten, hohe und niedrige standard Konzentrationen mit 3 analysiert wurden repliziert in den einzelnen unabhängigen Experimenten. Minimale Schwankungen in der berechneten Konzentration von Zielproteinen ist deutlich sichtbar.
Um das Dienstprogramm der Wulst-basierte durchflusszytometrischen Assays in Verhören Zytokin Expressionsprofile zu demonstrieren, wurden Maus Splenocyten unter verschiedenen aktivierende Bedingungen inkubiert. Zusätzlich eine unstimulierte Kontrolle wurden die Zellen auch mit LPS, Anti-CD3/Anti-CD28 Antikörper oder Phorbol Myristate-12 13-Acetat und Ionomycin inkubiert. Zelle Kultur Überstände wurden 48 h später und vermutlich mit der Maus T Helfer Zytokin Panel gesammelt. Die Quantifizierung Ergebnisse aus diesem Experiment sind in Abbildung 3 dargestellt und die Wirkung der Stimulation Bedingungen auf Zytokin-Konzentrationen ist klar abgegrenzt.
Die oben beschriebenen Ergebnisse sind typisch, solange der Experimentator gute Labortechnik hat und der zur Verfügung gestellten Test-Protokoll folgt. Fehlerquellen in diesem Assay-Format entstehen aus Abweichungen der Inkubationszeiten, Reagenz Bände, waschen, oder unsachgemäße PMT-Einstellungen auf das Durchflusszytometer verwendet, um die Proben zu analysieren. In Abbildung 4A eine Streuung zeigt Grundstück von einem erfolgreichen Test 2 unterschiedliche Wulst-Populationen, die klar definiert sind. Dies kann zu Abbildung 4 b, wo schlechte Einhaltung der Wasch- und Protokoll führten zu einer Perle Aggregate, die die beiden Populationen zu verwischen gegenübergestellt werden. Darüber hinaus wurden wie die Trümmer im unteren linken Quadranten belegt, total Cytometer Veranstaltungen für Analyse, anstatt das bevorzugte Format für geschlossene Veranstaltungen nur exportiert. Richtige Cytometer einrichten ist entscheidend für die zuverlässige Daten in diesem Assay zu erzeugen. In Abbildung 4 richtige PMT können Einstellungen für die Auflösung der einzelnen 6 Analyten in der APC-Klassifizierung-Kanal. Falsche Einstellungen der PMT führt Daten ähnlich dem in Abbildung 4 wo der APC-Kanal Wulst Populationen mit überlappenden Klassifizierung Intensitäten hat vorgestellt. Dies wird den Test ungültig, da es unmöglich Analytkonzentrationen ohne klar definierte Populationen von Assay Perlen berechnet werden.
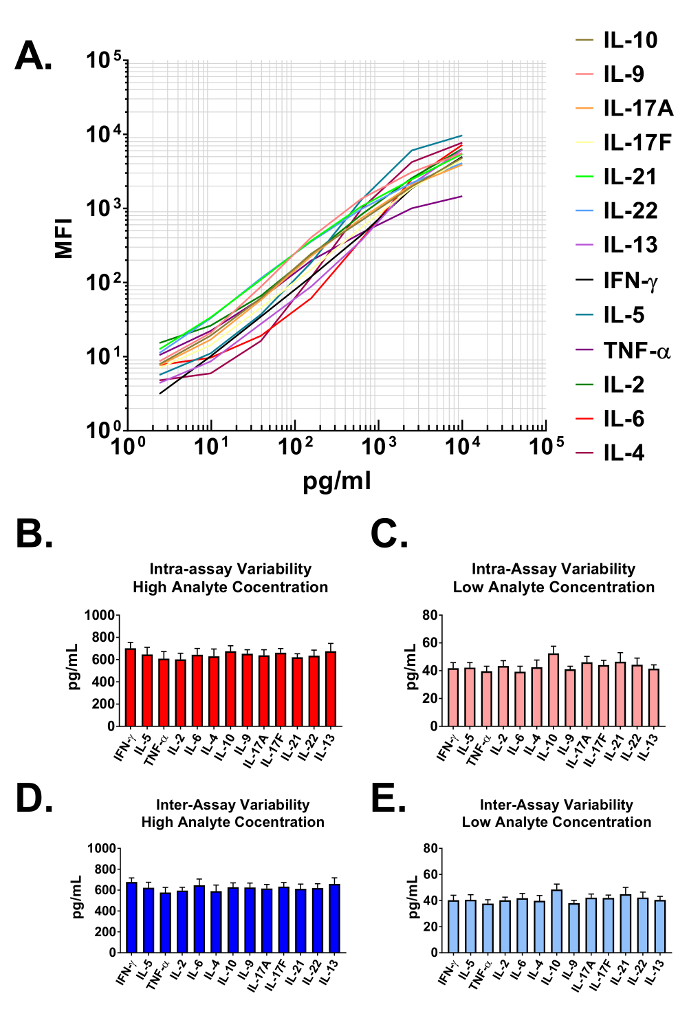
Abbildung 2: Standard Kurve reicht und Assay Präzision. (A) eine repräsentative standard Kurve mit der Maus T Helfer Zytokin-Panel erzeugt. Eine Standardkurve muss mit jedem Test ausgeführt werden. (B-C) Intra-Assay-Präzision. Zwei Proben mit verschiedenen Konzentrationen der Zielproteine (hoch und niedrig) wurden in einem Test mit 16 Wiederholungen für jede Probe analysiert. Daten sind als Mittelwert + Standardabweichung dargestellt. (D-E) Inter-Assay-Präzision. Zwei Proben mit verschiedenen Konzentrationen der Zielproteine (hoch und niedrig) wurden in drei unabhängigen Tests mit 3 Wiederholungen für jede Probe analysiert. Daten sind als Mittelwert + Standardabweichung dargestellt. Bitte klicken Sie hier für eine größere Version dieser Figur.
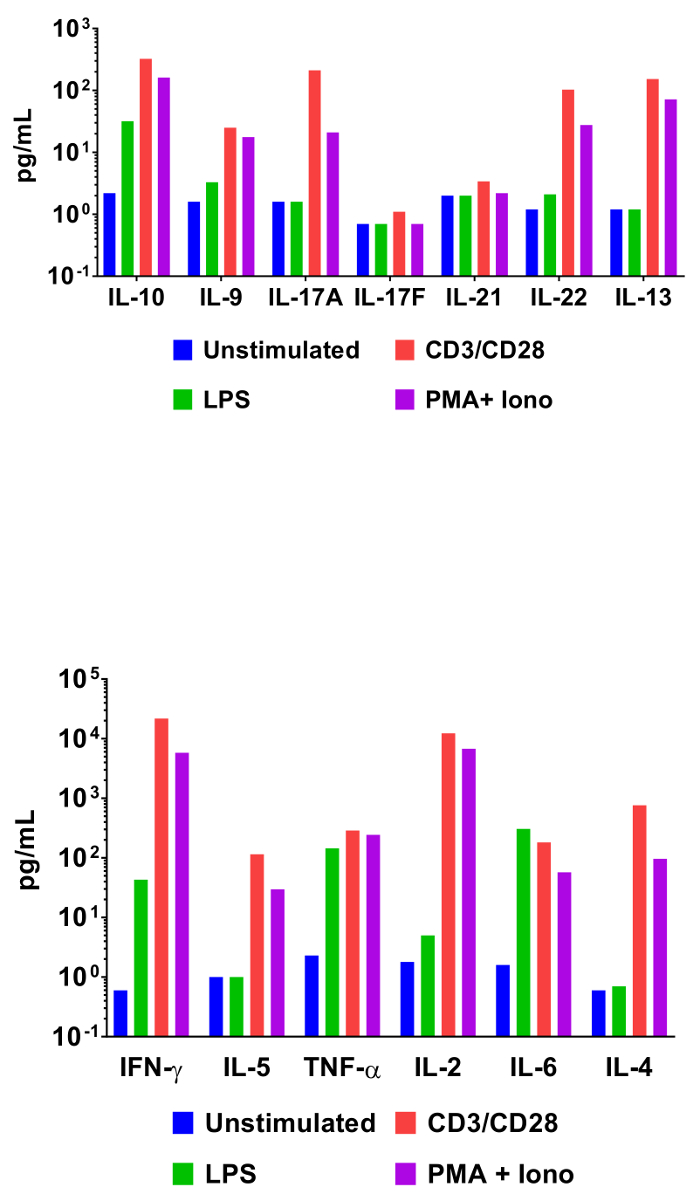
Abbildung 3: Quantifizierung der Vertreter Ergebnisse. Maus Splenocyten (1 x 106 Zellen) wurden bei 37 ° C + 5 % CO2 unter verschiedenen Bedingungen kultiviert: unstimulierte, LPS (100 ng/mL), αCD3 (1 µg/mL Platte beschichtet) + αCD28 (1 µg/mL löslich), PMA (20 ng/mL) + Ionomycin (500 ng/mL). Kultur-Überstände nach 48 h gesammelt wurden dann quantifiziert die Maus T Helfer Zytokin-Bedienfeld. Differenzielle Zytokin Expressionsprofile in Reaktion auf Anregung Bedingungen sind deutlich sichtbar. Bitte klicken Sie hier für eine größere Version dieser Figur.

Abbildung 4: ergibt sich aus erfolgreichen vs. erfolglosen Assays. (A) erfolgreiche Tests haben unterschiedliche Wulst Bevölkerung für die Bevölkerung in Mikrosphären. (B) erfolglos Assays haben arme Bevölkerung Trennung, Wulst Aggregate und zuviele gesammelten Ereignisse. (C) erfolgreiche Tests haben klar definierte Intensitäten in der Klassifizierung Fluoreszenz-Kanal, die sind einfach zu Tor und zu quantifizieren. (D) erfolglos Assays werden schlecht getrennt haben und Klassifizierung Intensitäten, die mehrere Perle dokumentierten und Quantifizierung schwierig zu überlappen. Viele dieser Fehler können durch sorgfältige Einhaltung der Assay-Protokolls vermieden werden. Bitte klicken Sie hier für eine größere Version dieser Figur.
| Standardkurve | Empfindlichkeit (Pg/mL) | ||||
| Analyten | Fit-Formel | CV | R2 | Min. | Max. |
| IFN-Γ | 5-P.log(4.43, 14.40, 0.54, 9.11, 0.01) | 1,77 % | 0.99 | 2.1 | 18105.0 |
| IL-5 | 5-P.log(4.36, 12.68, 0.64, 5.73, 0.36) | 1,32 % | 1 | 1.9 | 14514.0 |
| TNF-Α | 5-P.log(4.36, 11.63, 0.79, 5.45, 0.37) | 1,48 % | 0.99 | 1.9 | 20109.0 |
| IL-2 | 5-P.log(4.46, 12.62, 0.78, 5.07, 0.34) | 1,34 % | 1 | 1.9 | 25109.0 |
| IL-6 | 5-P.log(4.58, 11.66, 0.64, 5.77, 0.37) | ||||
Tabelle 1: Standard Kurvenanpassung und Assay Sensibilität. Die Datenanalyse-Software wurde verwendet, um generieren Standardkurven für alle Analyte in Multiplex-Assays enthalten. Dieser vollautomatische Auswertung gilt eine robuste 5-Parameter Kurvenanpassung Algorithmus an die Verdünnung Serie Standards von Assays und wurde auch verwendet, um Assay Empfindlichkeit mit der theoretischen Grenzen der Erkennung zu definieren.
Diskussion
In diesem Protokoll beschriebene Assay-Format bieten robuste Quantifizierung der löslichen Mediator Profile in biologischen Proben zur Verfügung gestellt des Experimentators hat gute Labortechnik und hält sich an die empfohlene Protokoll.
Es gibt mehrere wichtige Schritte, die befolgt werden müssen, um zuverlässige Ergebnisse zu gewährleisten. Erstens darf die rekombinante Standards in Testpuffer die volle Zeit empfohlen, und nach nur verdünnt in Polypropylenröhrchen wiederherzustellen. Polystyrol-Röhren können die Einhaltung der Proteine an der Wand der Rohre, fördern, die zu geringe Signale führen kann, Erstellung die Standardkurve. Zweite, richtige Schütteln während Inkubationsschritte ist von entscheidender Bedeutung. Ohne richtige Erregung können die Bindungseigenschaften der Assay-Perlen stark behindert werden. Darüber hinaus ist auch richtige waschen zwischen Inkubationsschritte erforderlich. Der Experimentator muss dafür sorgen, dass überschüssige Reagenzien aus dem Brunnen vor dem Beginn des nächsten Schritt im Protokoll entfernt werden. Instrumenten-Einstellungen für das Durchflusszytometer verwendet, um die Assay-Perlen zu verhören müssen auch optimiert werden. Insbesondere müssen RMZ Einstellungen angepasst werden, um richtige Wulst Trennung und breite dynamische Bereiche für die standard Kurven zu gewährleisten. Schließlich müssen nur vor auf die Cytometer analysiert, Perlen liegen verwirbelt, richtige Aufhängung zu versichern und die Bildung von Aggregaten die Ergebnisse verzerren können, zu vermeiden.
Dieses Protokoll kann möglicherweise geändert werden, um Gewebe Homogenates sowie die Probentypen diskutiert in dieser Handschrift zu analysieren, vorausgesetzt, die Forscher bereit, ihrer Lyse-Protokoll vor der Durchführung großer, volle Platte Assays zu optimieren. Die eigentliche Technik variiert natürlich je nach Gewebetyp; im Allgemeinen sollten das Protokoll jedoch einen neutralen pH-Puffer mit physiologischen Konzentrationen der ionische Salze, keine denaturierenden Chemikalien und vorzugsweise keine Reinigungsmittel verwenden. Wenn Waschmittel verwendet werden muss, sollte nicht-ionische Waschmittel Konzentrationen auf ein Minimum (z.B. nicht mehr als 1 %) gehalten werden. Der Puffer sollte auch ausreichend Protease-Inhibitoren zur Vermeidung proteolytischen Abbau von Zielproteinen enthalten. Unabhängig von der Lyse-Protokoll verwendet sollten die letzten Vorbereitungen zentrifugiert werden, um Partikel vor der Analyse zu entfernen.
Über Assay Einschränkungen können die Konzentrationen von Analyten Ziele in biologischen Probentypen stark variieren. Um richtig Analytkonzentrationen quantifizieren, müssen sie in der oberen und unteren Werten für die Standardkurve fallen. Extrapolation der Werte der Kurve ist nicht unbedingt zuverlässig und nicht empfehlen. Die Ermittler müssen daher Verdünnungen von ihrer bestimmten Proben in Pilotversuchen vor dem Ausführen eines voll-Platte-Assays optimieren. Darüber hinaus ist die sorgfältige Vorbereitung der biologischen Proben zu untersucht werden ausschlaggebend für den Erfolg des Datenformats Assay. Proben, die Lipämische, hämolysiert, oder enthalten Protein-Ablagerungen beeinträchtigen die Antigen Antikörper-Interaktionen, die das Grundprinzip dieses Immunoassay definieren sowie Rendern nicht interpretierbar sind.
Dieser Test ist in Bezug auf bestehende Quantifizierungsmethoden aus mehreren Gründen bedeutsam. Wenn richtig ausgeführt, kann der Assay-Format quantitate bis zu 13 Analyten aus einer Stichprobe mit weit geringeren Umfang als wäre erforderlich, um die Probe mit traditionellen ELISA-Assays zu analysieren. Dieser Assay-Format erfordert auch keine dedizierten Instrumentierung um durchgeführt werden. Dies ist ein Vorteil im Vergleich zu anderen handelsüblichen Bead-basierten Immunoassays, wie der Test mit einer beliebigen Anzahl von häufig verwendeten fließen Cytometers ausgeführt werden kann.
Wissenschaftliche Untersuchung über die Rolle gespielt von sezernierten Faktoren und Zytokin-Netzwerke in Gesundheit und Krankheit erweitert. In diesem Manuskript beschriebene Assay-Format kann ein wertvolles Werkzeug für die Forscher versuchen, diese vielschichtige und komplexe Phänomene zu verstehen. Aktive biomedizinische Forschungsprogramme fangen an, die Rolle der Zytokin-Netzwerke nicht nur Bereiche wie generalisierte Entzündung6, aber auch in Zusammenhang mit bestimmten Krankheitszuständen wie Arteriosklerose, Krebs und Neuroinflammation erkunden 15,16,17. Zweifellos wird die Untersuchung weiterhin in diesen Bereichen zu wachsen, wie die Suche nach neuer Therapeutika zur Behandlung dieser chronischen Erkrankungen erweitert. Die genaue und zuverlässige Quantifizierung der löslichen Mediatoren verbunden mit diesen Krankheiten wird eine kritische Forschungspriorität bleiben.
Offenlegungen
Die Autoren sind BioLegend, beschäftigt die Reagenzien beschrieben in diesem Manuskript herstellt. Vigene Tech, Inc. entwickelt die LEGENDplex Datenanalyse-Software, die verwendet wird, um Daten aus der Assay beschrieben in dieser Handschrift zu analysieren.
Danksagungen
Die Autoren möchten die Beiträge von Biomarkern und Immuno-Assays erkennen Product Development Team zur Entwicklung des Assays. Darüber hinaus möchten wir danken Vigene Tech, Inc. für deren gemeinsamen Anstrengungen bei der Gestaltung der Daten-Analyse-Software-Pakete.
Materialien
| Name | Company | Catalog Number | Comments |
| Allegra 6R Centrifuge with MICROPLUS Carrier Adaptor | Beckman Coulter | 366816 | For LEGENDplex assays is run in microtubes, V-or U-bottom 96-well plate (optional) |
| LEGENDplex Mouse T helper Cytokine Panel | BioLegend | 740005 | Multiplex Immunoassay (13-plex) |
| Anti-mouse CD3ε antibody | BioLegend | 100314 | Clone 145-2C11, low endotoxin, azide free format |
| Anti-mouse CD28 antibody | BioLegend | 102112 | Clone 37.51, low endotoxin, azide free format |
| Vacuum Pump | EMD Millipore | WP6111560 | For LEGENDplex assays using filter plates (recommended) |
| Vacuum Manifold | EMD Millipore | MSVMHTS00 | For LEGENDplex assays using filter plates (recommended) |
| Sterile Disposable Reagent Reservoirs | Fisher Scientific | 07-200-130 | Or equivalent |
| Polypropylene MicroFACS Tubes | Fisher Scientific | 11-842-90 | For LEGENDplex assays is run in microtubes, V-or U-bottom 96-well plate (optional) |
| Pipette Kit | Fisher Scientific | 14-388-100 | Four pipette sizes (0.2-2µL, 2-20µL, 20-200µL, 100-1000µL) |
| Multi-Channel Pipette | Fisher Scientific | FA10011G | capable of dispensing 5 μL to 200 μL |
| Microplate Shaker | Fisher Scientific | 88880023 | Or equivalent |
| Microcentrifuge | Fisher Scientific | 75002410 | Or equivalent |
| FACS tubes | Fisher Scientific | NC9885747 | 12 x 75 mm round bottom |
| PMA (Phorbol 12-myristate 13-acetate) | Sigma-Aldrich | P8139 | |
| Lipopolysaccharide (LPS) | Sigma-Aldrich | L-8274 | Escherichia coli O26:B6 |
| Ionomycin | Sigma-Aldrich | I0634 | |
| Branson B200 Ultrasonic Cleaner | Sigma-Aldrich | Z305359 | Or equivalent |
| Microcentrifuge tubes | Sigma-Aldrich | Z666505 | 1.5 mL polypropylene tubes |
| Flow Cytometer | Various | Various | Cytometer equipped with single laser (488nm blue) or two lasers (488nm blue or 532nm green + 633nm Red) capable of reading emission wavelengths at 575nm and 660nm |
| 96-Well Polypropylene Plate | VWR | 82050-662 | For LEGENDplex assays is run in microtubes, V-or U-bottom 96-well plate (optional) |
Referenzen
- Stanley, A. C., Lacy, P. Pathways for cytokine secretion. Physiology (Bethesda). 25, 218-229 (2010).
- Nicola, N. A. Cytokine pleiotropy and redundancy: a view from the receptor. Stem Cells. 12, (1994).
- Miyajima, A., Hara, T., Kitamura, T. Common subunits of cytokine receptors and the functional redundancy of cytokines. Trends Biochem Sci. 17, 378-382 (1992).
- Costantini, S., Castello, G., Colonna, G. Human Cytokinome: a new challenge for systems biology. Bioinformation. 5, 166-167 (2010).
- Capone, F., et al. Serum Cytokinome Profile Evaluation: A Tool to Define New Diagnostic and Prognostic Markers of Cancer Using Multiplexed Bead-Based Immunoassays. Mediators Inflamm. , 3064643 (2016).
- Schett, G., Elewaut, D., McInnes, I. B., Dayer, J. M., Neurath, M. F. How cytokine networks fuel inflammation: Toward a cytokine-based disease taxonomy. Nat Med. 19, 822-824 (2013).
- Tse, E., Kwong, Y. L. T-cell lymphoma: Microenvironment-related biomarkers. Semin Cancer Biol. 34, 46-51 (2015).
- Reale, M., Greig, N. H., Kamal, M. A. Peripheral chemo-cytokine profiles in Alzheimer's and Parkinson's diseases. Mini Rev Med Chem. 9, 1229-1241 (2009).
- Vistnes, M., Christensen, G., Omland, T. Multiple cytokine biomarkers in heart failure. Expert Rev Mol Diagn. 10, 147-157 (2010).
- Gobel, K., et al. Blood coagulation factor XII drives adaptive immunity during neuroinflammation via CD87-mediated modulation of dendritic cells. Nat Commun. 7, 11626 (2016).
- Palm, A. K., Friedrich, H. C., Kleinau, S. Nodal marginal zone B cells in mice: a novel subset with dormant self-reactivity. Sci Rep. 6, 27687 (2016).
- Yu, Y., et al. The transcription factor Bcl11b is specifically expressed in group 2 innate lymphoid cells and is essential for their development. J Exp Med. 212, 865-874 (2015).
- Pelly, V. S., et al. IL-4-producing ILC2s are required for the differentiation of TH2 cells following Heligmosomoides polygyrus infection. Mucosal Immunol. 9, 1407-1417 (2016).
- . LEGENDplex Multiplex Assays Available from: https://www.biolegend.com/legendplex (2017)
- Autieri, M. Pro- and anti-inflammatory networks in atherosclerosis. ISRN Vascular Medicine. 2012, (2012).
- West, N. R., et al. Emerging cytokine networks in colorectal cancer. Nat Rev Immunol. 15, 615-629 (2015).
- Becher, B., Spath, S., Goverman, J. Cytokine networks in neuroinflammation. Nat Rev Immunol. 17, 49-59 (2017).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten