Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Evaluación del impacto de la agregación de proteína sobre el estrés oxidativo celular en levaduras
En este artículo
Resumen
Agregación de la proteína provoca estrés oxidativo celular. Este protocolo describe un método de control de los Estados intracelulares de proteínas amyloidogenic y el estrés oxidativo asociado con ellos, mediante citometría de flujo. El enfoque se utiliza para estudiar el comportamiento de las variantes soluble y propensa a la agregación del péptido amiloide-β.
Resumen
Mal plegamiento de proteínas y acumulación en conformaciones amiloides han sido relacionadas con la aparición y progresión de varias enfermedades neurodegenerativas. Sin embargo, todavía hay poca información sobre cómo insoluble proteína agregados ejercen sus efectos tóxicos in vivo. Organismos modelo simples de procariotas y eucariotas, tales como bacterias y levadura, han contribuido significativamente a nuestra actual comprensión de los mecanismos detrás de la formación amiloide intracelular, la propagación de agregados y la toxicidad. En el presente Protocolo, el uso de levadura se describe como un modelo para analizar la relación entre la formación de agregados proteicos y su impacto sobre el estrés oxidativo celular. El método combina la detección del estado intracelular soluble/agregados de una proteína amyloidogenic con la cuantificación del daño oxidativo celular resultantes de su expresión mediante citometría de flujo (FC). Este enfoque es simple, rápida y cuantitativa. El estudio ilustra la técnica al correlacionar el estrés oxidativo celular causado por un amplio conjunto de variantes de péptido amiloide-β con sus propensiones respectiva agregación intrínseca.
Introducción
Proteostasis es un determinante fundamental de los procesos de acondicionamiento físico y el envejecimiento celular. En las células, se mantiene la homeostasis de proteínas por control de calidad de la proteína sofisticadas redes destinadas a asegurar la correcta refolding de Conformadores de proteínas mal plegadas por acompañantes o su proteólisis específica con varios mecanismos bien conservados1 ,2,3,4,5. Un gran número de estudios proporciona apoyo a la relación entre el inicio y la progresión de una amplia gama de enfermedades humanas y la falta de proteostasis, llevando a mal plegamiento de proteínas y agregación. Por ejemplo, la presencia de depósitos de proteína se considera una característica patológica de muchas enfermedades neurodegenerativas, como Alzheimer, Parkinson y Huntington enfermedades6,7,8, enfermedades prionogenic y amiloidosis no degenerativas9. Se ha sugerido que temprana asambleas oligomeric y protofibrillar en la reacción de agregación son los elicitores principal de citotoxicidad, establecer aberrantes interacciones con otras proteínas en el medio celular lleno de gente10. Además, inclusiones de proteína (PI) pueden transmitirse entre las células, propagando su efecto tóxico11,12. Por lo tanto, podría ser que la formación de PI de hecho podría constituir un mecanismo de desintoxicación que restringe la presencia de especies peligrosas agregadas a lugares específicos en la célula, donde pueden ser procesados o acumulados sin efectos secundarios importantes 13 , 14.
Estándar en vitro bioquímicos enfoques han aportado importante información sobre las diferentes especies que pueblan las reacciones de agregación y sus propiedades15,16. Sin embargo, las condiciones utilizadas en estos ensayos son claramente diferentes de los que ocurren dentro de la célula y, por lo tanto, cuestionan su importancia fisiológica. Debido a la notable conservación de vías celulares como control de calidad de la proteína, la autofagia o la regulación de redox celular estado17,18 entre eucariotas19,20,21 ,22,23, la levadura de florecimiento Saccharomyces cerevisiae (S. cerevisiae) ha surgido como un simple modelo celular privilegiado para el estudio de los determinantes moleculares de la agregación de proteínas y sus efectos citotóxicos asociados en entornos biológicamente relevantes24,25,26.
Propensión de agregación de la proteína es una característica intrínsecamente codificada en la secuencia primaria. Así, la formación de amiloide-como las estructuras se puede predecir basado en la identificación y evaluación de la potencia de promover la agregación de polipéptidos27regiones. Sin embargo, a pesar del éxito de bioinformáticas algoritmos para predecir las propiedades de agregación en vitro de secuencias de proteínas, son aún muy lejos de pronosticar cómo estas propensiones traducen en vivo impacto citotóxico. Estudios que aborden la relación entre el estado agregado de una proteína dada y su asociado daño celular de forma sistemática pueden ayudar a eludir esta limitación computacional. Esta conexión se aborda en el presente estudio, tomando ventaja de un amplio conjunto de variantes del péptido amiloide-β Aβ42 difieren sólo en un único residuo, pero mostrando un rango continuo de agregación propensiones en vivo28de. En particular, se describe un enfoque basado en FC para identificar las especies conformacionales responsables del daño oxidativo provocado por propensas a la agregación de proteínas en las células de levadura. La metodología ofrece muchas ventajas tales como simplicidad, capacidad de alto rendimiento y precisión para la medición cuantitativa. Este enfoque permitió confirmar ese juego un papel protector contra el estrés oxidativo de la PI.
Protocolo
1. cultivos de S. cerevisiae y expresión de la proteína
Nota: Variantes Aβ exhibición inclinaciones diferentes agregación relativa debido a una mutación en un solo residuo en la posición 19 (Phe19) del péptido de Aβ42 (figura 1A). Estas variantes de péptidos se han etiquetado con la proteína fluorescente verde (GFP), que actúa como una agregación de reportero (figura 1)29.
- Transforman plásmidos codifican para 20 variantes de Aβ42-GFP en células de levadura con un BY4741 fondo parental (MATa su3Δ1 leu2Δ0 met15Δ0 ura3Δ0)30. Cada plásmido codifica para un mutante de Aβ42 fusionado a GFP por un enlazador GSSGSSG. La plásmidos son pESC(-URA) e incluyen el marcador seleccionable URA329.
- Preparar el medio completo sintético sin uracilo (SC-URA) con los siguientes componentes: 1,9 g/L nitrógeno levadura base uracilo, sulfato de amonio 5 g/L y 900 mL de ddH2O combinado con 100 mL de glucosa 20% (p/v).
- Poner una colonia de las células de levadura transformadas en 20 mL de medio SC-URA que contiene 2% de glucosa y crecer los cultivos durante la noche a 30 ° C bajo agitación en rpm 210.
- Al día siguiente, crecen nuevas culturas de células de levadura por inoculación 100 μl de la cultura durante la noche en 5 mL de medio fresco de SC-URA.
- Un OD590 de 0.5, centrifugar los cultivos a 3.000 x g durante 4 min, descartar el sobrenadante y resuspender las células en el mismo volumen de medio fresco de SC-URA que contienen rafinosa 2%.
- Incubar a 30 ° C bajo agitación a 210 rpm por 30 min. Luego, centrifugar las células a 3.000 x g durante 4 min y descarte el sobrenadante. Resuspender las células en medio fresco de SC-URA que contiene 2% galactosa para inducir la expresión de proteína recombinante.
- Después de 16 h de inducir la expresión de proteínas en medio SC-URA que contiene galactosa 2%, 1 mL de las células inducidas en tubos estériles de microcentrífuga la cosecha por centrifugación a 3.000 x g durante 4 minutos.
Nota: Las diferencias más relevantes se producen después de un período de inducción de 16 h. inducida levadura las células que expresan variantes de Aβ42-GFP puede visualizarse por microscopía de fluorescencia para determinar la distribución de la proteína recombinante dentro de las células (figura 2B).
2. células de tinción
Nota: Células no-inducida son necesarias como control negativo para establecer un umbral fluorescente durante un análisis FC.
- Tomar las células de levadura inducida por h 16 y determinar su densidad óptica a 590 nm (OD590). Les diluido en un tampón de fosfato estéril salino (PBS) a un OD590 de 0,1.
- Preparar como sigue 1 x de tampón fosfato salino: Añadir 8 g de NaCl, 0,2 g de KCl, 1,44 g de Na2HPO4, y 0.24 g de KH2PO4 a 800 mL de ddH2O. llenar la solución hasta 1 L de ddH2O después de ajustar el pH a 7.4 con HCl. filtro el búfer a través de un filtro de 0.2 μm.
- Las suspensiones de células expresando la transferencia a tubos debidamente etiquetados (12 × 75 mm fondo redondo poliestireno expandido) y proteger de la luz. Asegúrese de que están listos para ser cargados en el citómetro de flujo para el análisis, junto con las células no inducidas y no manchado.
- Añadir la punta de prueba de estrés oxidativo (por ejemplo, rojo oscuro CellROX) a cada muestra a una concentración final de 5 μm e incubar las células en 30 ° C por 30 min en la oscuridad.
- Después de la incubación, lavar las células 3 veces con 1 x PBS y resuspender en el mismo volumen de tampón antes de su análisis por FC.
3. Análisis de citometría de flujo de
Nota: Usar una configuración FC con láseres apropiados y filtros para detectar GFP y señal de fluorescencia de punta de prueba de estrés oxidativo. Puede utilizarse un citómetro de flujo equipado con un láser de nm azul 488 para la detección de GFP y un 635 nm rojo para la detección de la fluorescencia de punta de prueba de estrés oxidativo. Adquisición de la fluorescencia de emisión de GFP y probe el estrés oxidativo se realiza con un 530/30 nm BP filtro y un 660/20 BP, respectivamente (tabla 1).
- Haga clic en Abra el Panel de hoja de cálculo nueva y crear los siguientes diagramas de punto de adquisición.
- Seleccione la Herramienta de diagrama Scatter de la barra de herramientas y crear un diagrama con las variables área de dispersión lateral (SSC-A) en el eje y frente a zona de dispersión hacia adelante (FSC-A) en una escala lineal en el eje x.
- Haga clic en la Herramienta de diagrama Scatter de la barra de herramientas y crear un diagrama con las variables área de FL1 (FITC) en el eje x vs. FL3-área (APC) en una escala logarítmica en el eje y.
- Haga clic en el icono Configuración de instrumentos . Haga clic en la ficha de compensación y ajuste todos los niveles de compensación. Haga clic en la ficha de adquisición y seleccione un número total de 20.000 eventos a registrar.
- Haga clic en el icono de Flujo para cambiar la velocidad de flujo a baja.
- Haga clic en la ficha de adquisición para comenzar a correr las células no inducidas, sin manchas y ajustar la tensión en los Parámetros fijados del instrumento del adelante y dispersión lateral hasta que la población se distribuye en el cuadrante de centro-izquierda.
- Haga clic en el icono de polígono para definir una región R1 alrededor de la población de la célula excepto restos de células y esta población cerrada P1 = R1 para todas las parcelas de punto fluorescente y representaciones de histograma (figura 3).
- Para ajustar la tensión de la PMT de la señal de fluorescencia, ejecute las células sin manchas en la trama de punto FL1-FL3, ajuste la ganancia en la ficha Configuración del instrumento , hasta que las células se distribuyen en el cuadrante izquierdo más bajo.
- Cambiar la muestra a las células inducidas para medir la fluorescencia de GFP. En la ficha Configuración del instrumento , establecer la ganancia de la FSC-A vs FL1 (FITC) cuando la población se distribuye en el cuadrante de abajo a la derecha. Definir la población de células positivas con una puerta (puerta P2).
- Cambio la muestra a la inducida mancharon la fluorescencia en una trama de puntos FSC-A vs FL3 (APC) para mostrar el estrés oxidativo de las células. Afine la ganancia hasta que la población celular se distribuye en el cuadrante superior izquierdo. Puerta de la población de células positivas en P3.
- Hacer dos parcelas histograma con el icono de histograma para representar a la fluorescencia de la célula. Para representar la intensidad de fluorescencia, asegúrese que es FL1 (FITC) que representa a la fluorescencia de GFP en el eje x, y FL3 (APC) que representa la fluorescencia en el eje y, aplicando las poblaciones P2 y P3 en una escala logarítmica, respectivamente.
Nota: Superposiciones de histograma son una buena manera de ilustrar las diferencias entre los perfiles de fluorescencia de las células que expresan diversas variantes Aβ.
4. inmunodetección de proteínas recombinantes
- Para determinar los niveles de expresión de la proteína, las culturas de levadura inducidas durante 16 horas por centrifugación a 4.000 x g durante 6 minutos la cosecha y almacenar los pellets de células a-80 ° C.
- Resuspender las células colectadas expresan la proteína recombinante por 16 h en PBS. Preparación de suspensiones celulares de 200 μL de cada mutante de Aβ42-GFP a una OD590 de 20.
- Para el análisis de la fracción de proteína total, cosechar 100 μl de cada mutante por centrifugación a 14.000 x g por 20 min y resuspender el pellet celular en el mismo volumen de tampón de lisis de la levadura Y-por (50 mM Tris-HCl pH 8.0, 1% DMSO, 200 mM NaCl, 1 mM EDTA que contiene 10 mg/mL SB3-14 (myr istyl sulfobetaína) complementado con fluoruro (PMSF) del phenylmethylsulfonyl de 1 mM).
- Incubar las muestras durante 20 min a temperatura ambiente en agitación suave.
- Determinar la concentración de extracto de proteína mediante el ensayo de Bradford. Carga hasta 5 μg de extracto de proteína de cada muestra en un gel de electroforesis de SDS-PAGE de acrilamida 15% y el blot en una membrana de difluoruro (PVDF) de polivinilideno a 100 V por 60 min.
- Preparar a un Bradford reactivo con 100 mg de azul brillante de Coomassie G-250 disuelto en 50 mL de etanol al 95% y agregar 100 mL de ácido fosfórico al 85% (w/v). Entonces, cuando el colorante se haya disuelto completamente, diluir la mezcla con ddH2O a 1 L y filtrar con papel de filtro de celulosa antes de uso.
Nota: El reactivo de Bradford debe ser marrón claro. - Preparar un estándar de albúmina de suero bovino (BSA) que van desde 5 a 100 μg de proteína en 1000 μl del reactivo de Bradford.
- Añadir 1-10 μl de proteína extraer 1 ml del reactivo de Bradford e incubar la mezcla a temperatura ambiente durante 5 minutos.
- Medir la absorbancia de los estándares y las muestras de extracto de proteína a OD595.
- Para el análisis, hacer un diagrama con los valores de absorbancia obtenidos para los estándares vs. μg de proteína. Basado en la curva estándar, determinar las concentraciones de las muestras originales de la cantidad de proteína, teniendo en cuenta el volumen y la dilución, si cualquier.
- Preparar a un Bradford reactivo con 100 mg de azul brillante de Coomassie G-250 disuelto en 50 mL de etanol al 95% y agregar 100 mL de ácido fosfórico al 85% (w/v). Entonces, cuando el colorante se haya disuelto completamente, diluir la mezcla con ddH2O a 1 L y filtrar con papel de filtro de celulosa antes de uso.
- Lavar la membrana con 1 x solución salina tamponada con Tris (TBS), 0,1% Tween 20 (TTBS) y bloquear usando leche descremada en polvo 5% (w/v) con 1 x TTBS.
- Para preparar 1 L de 10 x TBS, disolver 24 g de Tris base y 88 g de NaCl en 900 mL de H2Odd y ajustar el pH a 7,6. Añadir ddH2O a un volumen final de 1 L. Para una solución de 1 x, mezcle 1 parte de stock de x 10 con 9 partes de ddH2O.
- Incubar la membrana durante 1 h con un anticuerpo primario de β-amiloide 6E10 diluido 1:1,000 y por 1 h a temperatura ambiente con una cabra de anticuerpo secundario conjugado de anti-mouse IgG-HRP diluido 1:10, 000.
- Desarrollar membranas usando 1 mL de reactivo fluorescente Luminata y cuantificar bandas por análisis densitométrico como se describe en el paso 5.2.
5. Análisis de datos
- Para analizar los datos obtenidos por FC, crear una tabla mostrando la intensidad fluorescente media (IFM) y mediana de la fluorescencia con su correspondiente error estándar o el coeficiente de variación (CV) para la fluorescencia de GFP y niveles de estrés oxidativo.
- Haga clic en la ficha Inspector y el icono de estadísticas para personalizar los datos estadísticos que se requieren para cada canal.
Nota: Cuando se comparan las variantes de la proteína, convertir datos de valores de fluorescencia a una doblar-sobre fondo o resolución métrico (RD):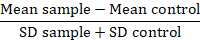
En esta métrica, la muestra media es la media fluorescencia de las muestras problema, el control media es la media fluorescencia de células utilizadas como control, y la SD es la desviación estándar de la media de la fluorescencia. Factor de RD es un índice más preciso que el valor de la IMF como no solo calcula la diferencia entre las muestras, sino también representa la difusión de los datos. Este cálculo es específicamente relevante al comparar datos de diferentes experimentos realizados en el tiempo. Una vez que se han calculado los RDs, el uso de una prueba estadística es recomendable confirmar la significación de las diferencias observadas entre las variantes de la proteína.
- Haga clic en la ficha Inspector y el icono de estadísticas para personalizar los datos estadísticos que se requieren para cada canal.
- De inmunodetección de proteínas, cuantificar los niveles de proteína recombinante por Western blot utilizando el software ImageJ.
- Antes del análisis de densitometría, convertir las imágenes raws originales de la membrana desarrollada en modo del archivo JPEG formato y escala de grises.
- Ajustar la sección Establecer mediciones del menú analizar valor de gris medio.
- Haga clic en la herramienta rectángulo de ImageJ y definir una región de interés (ROI) con un tamaño consistente para el análisis de densitometría de la banda. Centro de cada banda en la imagen de la membrana dentro del marco rectangular creado y registre la medida uno por uno usando Ctrl + M (el comando de medición).
- Medir el fondo local adyacente a cada banda, aplicación de la misma área de retorno de la inversión. Después de la medida, restar el fondo local correspondiente de cada banda individual.
Resultados
Este protocolo describe cómo utilizar una colección de 20 variantes del péptido de Aβ42 donde Phe19 ha sido mutado a todos natural proteinogenic aminoácidos28. Las propensiones de teórico de la agregación de estas proteínas pueden ser analizadas utilizando dos algoritmos bioinformáticos diferentes (AGGRESCAN y TANGO31,32). En ambos casos, este análisis representa una progresiva gradación de tende...
Discusión
Una amplia gama de enfermedades está ligada a la acumulación de proteínas mal plegadas en depósitos celulares6,7,8,33. Se han hecho muchos esfuerzos para desentrañar los mecanismos moleculares que desencadenan la aparición de estas enfermedades usando el enfoque computacional, que no tienen en las concentraciones de proteína de cuenta, o en la vitro se acerca, en la que la conce...
Agradecimientos
Materiales
| Name | Company | Catalog Number | Comments |
| Yeast cells BY4741 | ATCC | 201388 | Genotype: MATa his3Δ1 leu2Δ0 met15Δ0 ura3Δ0 |
| pESC(-Ura) plasmid | Agilent Genomics | 217454 | Yeast expression plasmid with a Gal promotor. Selectable marker URA3 |
| Yeast Synthetic Drop-out Medium Supplements | Sigma | Y1501 | Powder |
| Yeast Nitrogen Base Without Amino Acids | Sigma | Y0626 | Powder |
| Raffinose | Sigma | R7630 | Powder |
| Glucose | Sigma | G7021 | Powder |
| Galactose | Sigma | G0750 | Powder |
| Phosphate Buffered Saline (PBS) | Fisher Scientific | BP3991 | Solution 10X |
| CellROX Deep Red Reagent | Life Technologies | C10422 | Free radical cell-permeant fluorescent sensor, non-fluorescent while in a reduced state, and exhibits bright fluorescence upon oxidation by reactive oxygen species (ROS), with absorption/emission maxima at 644/665 nm. |
| Y-PER protein extraction reagent | Thermo Scientific | 78990 | Liquid cell lysis buffer |
| Acrylamide/Bis-acrylamide | Sigma | A6050 | Solution |
| Bradford dye reagent | Bio-Rad | 5000205 | Dye reagent for one-step determination of protein concentration |
| β-amyloid antibody 6E10 | BioLegend | 803001 | Mouse IgG1. The epitope lies within amino acids 3-8 of beta amyloid (EFRHDS). |
| Goat anti-mouse IgG-HRP conjugate | Bio-Rad | 1721011 | |
| Membrane Immobilon-P, PVDF | Millipore | IPVH00010 | |
| Luminata forte | Merk | WBLUF0100 | Premixed, ready to use chemiluminescent HRP detection reagent |
| Phenylmethanesulfonyl fluoride solution (PMSF) | Sigma | 93482 | Protease inhibitor. Dissolved at 0.1 M in ethanol |
| FACSCanto flow cytometer | BD Biosciences | 657338 | Equipped with a 488 nm blue laser for the detection of GFP, and 635 nm red laser / 530/30 nm BP filter and 660/20 BP filter |
| Mini Trans-Blot Electrophoresis Transfer cell | Bio-Rad | 1703930 | Protein transference system |
| Mini-PROTEAN Tetra Handcast Systems | Bio-Rad | 1658000FC | Electrophoresis system |
Referencias
- Frydman, J. Folding of newly translated proteins in vivo: the role of molecular chaperones. Annual Review of Biochemistry. 70, 603-647 (2001).
- Hartl, F. U., Bracher, A., Hayer-Hartl, M. Molecular chaperones in protein folding and proteostasis. Nature. 475, 324-332 (2011).
- Wong, E., et al. Molecular determinants of selective clearance of protein inclusions by autophagy. Nature Communications. 3, (2012).
- Winkler, J., Tyedmers, J., Bukau, B., Mogk, A. Chaperone networks in protein disaggregation and prion propagation. Journal of Structural Biology. 179, 152-160 (2012).
- Glickman, M. H., Ciechanover, A. The ubiquitin-proteasome proteolytic pathway: destruction for the sake of construction. Physiological Reviews. 82, 373-428 (2002).
- Dobson, C. M. Getting out of shape. Nature. 418, 729-730 (2002).
- Chiti, F., Dobson, C. M. Protein misfolding, functional amyloid, and human disease. Annual Review of Biochemistry. 75, 333-366 (2006).
- Aguzzi, A., O'Connor, T. Protein aggregation diseases: pathogenicity and therapeutic perspectives. Nature Reviews Drug Discovery. 9, 237-248 (2010).
- Rapezzi, C., et al. Transthyretin-related amyloidoses and the heart: a clinical overview. Nature Reviews Cardiology. 7, 398-408 (2010).
- Deas, E., et al. Alpha-synuclein oligomers interact with metal ions to induce oxidative stress and neuronal death in Parkinson's disease. Antioxidants & Redox Signaling. 24, 376-391 (2016).
- Brundin, P., Melki, R., Kopito, R. Prion-like transmission of protein aggregates in neurodegenerative diseases. Nature Reviews Molecular Cell Biology. 11, 301-307 (2010).
- Frost, B., Diamond, M. I. Prion-like mechanisms in neurodegenerative diseases. Nature Reviews Neuroscience. 11, 155-159 (2009).
- Arrasate, M., Mitra, S., Schweitzer, E. S., Segal, M. R., Finkbeiner, S. Inclusion body formation reduces levels of mutant huntingtin and the risk of neuronal death. Nature. 431, 805-810 (2004).
- Ross, C. A., Poirier, M. A. Opinion: what is the role of protein aggregation in neurodegeneration?. Nature Reviews Molecular Cell Biology. 6, 891-898 (2005).
- Mishra, R., Sjölander, D., Hammarström, P. Spectroscopic characterization of diverse amyloid fibrils in vitro by the fluorescent dye Nile red. Molecular BioSystems. 7, 1232-1240 (2011).
- Klunk, W. E., Jacob, R. F., Mason, R. P. Quantifying amyloid by congo red spectral shift assay. Methods in Enzymology. 309, 285-305 (1999).
- Khurana, V., Lindquist, S. Modelling neurodegeneration in Saccharomyces cerevisiae: why cook with baker's yeast?. Nature Reviews Neuroscience. 11, 436-449 (2010).
- Tenreiro, S., Outeiro, T. F. Simple is good: yeast models of neurodegeneration. FEMS Yeast Research. 10, 970-979 (2010).
- Moosavi, B., Mousavi, B., Macreadie, I. G. Yeast model of amyloid-β and Tau aggregation in Alzheimer's disease. Journal of Alzheimer's Disease. 47, 9-16 (2015).
- Tenreiro, S., Munder, M. C., Alberti, S., Outeiro, T. F. Harnessing the power of yeast to unravel the molecular basis of neurodegeneration. Journal of Neurochemistry. 127, 438-452 (2013).
- Yang, J., Hao, X., Cao, X., Liu, B., Nyström, T. Spatial sequestration and detoxification of huntingtin by the ribosome quality control complex. eLife. 5, (2016).
- Braun, R. J., Büttner, S., Ring, J., Kroemer, G., Madeo, F. Nervous yeast: modeling neurotoxic cell death. Trends in Biochemical Sciences. 35, 135-144 (2010).
- Figley, M. D., Gitler, A. D. Yeast genetic screen reveals novel therapeutic strategy for ALS. Rare Diseases. 1, e24420 (2013).
- Cooper, A. A., et al. Alpha-synuclein blocks ER-Golgi traffic and Rab1 rescues neuron loss in Parkinson's models. Science. 313, 324-328 (2006).
- Johnson, B. S., McCaffery, J. M., Lindquist, S., Gitler, A. D. A yeast TDP-43 proteinopathy model: exploring the molecular determinants of TDP-43 aggregation and cellular toxicity. Proceedings of the National Academy of Sciences of the United States of America. 105, 6439-6444 (2008).
- Bharathi, V., et al. Use of ade1 and ade2 mutations for development of a versatile red/white color assay of amyloid-induced oxidative stress in saccharomyces cerevisiae. Yeast. 33, 607-620 (2016).
- Pallarès, I., Ventura, S. Advances in the prediction of protein aggregation propensity. Current Medicinal Chemistry. , (2017).
- Villar-Piqué, A., Ventura, S. Protein aggregation propensity is a crucial determinant of intracellular inclusion formation and quality control degradation. Biochimica et Biophysica Acta. 1833, 2714-2724 (2013).
- Navarro, S., Villar-Piqué, A., Ventura, S. Selection against toxic aggregation-prone protein sequences in bacteria. Biochimica et Biophysica Acta. 1843, 866-874 (2014).
- Morell, M., de Groot, N. S., Vendrell, J., Avilés, F. X., Ventura, S. Linking amyloid protein aggregation and yeast survival. Molecular BioSystems. 7, 1121-1128 (2011).
- Conchillo-Solé, O., et al. AGGRESCAN: a server for the prediction and evaluation of "hot spots" of aggregation in polypeptides. BMC Bioinformatics. 8, 65 (2007).
- Fernandez-Escamilla, A. M., Rousseau, F., Schymkowitz, J., Serrano, L. Prediction of sequence-dependent and mutational effects on the aggregation of peptides and proteins. Nature Biotechnology. 22, 1302-1306 (2004).
- Renner, M., Melki, R. Protein aggregation and prionopathies. Pathologie Biologie.(Paris). 62, 162-168 (2014).
- Carija, A., Navarro, S., de Groot, N. S., Ventura, S. Protein aggregation into insoluble deposits protects from oxidative stress. Redox Biology. 12, 699-711 (2017).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados