JoVE 비디오를 활용하시려면 도서관을 통한 기관 구독이 필요합니다. 전체 비디오를 보시려면 로그인하거나 무료 트라이얼을 시작하세요.
Method Article
효 모 세포 산화 스트레스에 단백질 집단의 영향 평가
요약
단백질 집계 세포 산화 스트레스 elicits. 이 프로토콜 amyloidogenic 단백질의 세포내 주와 관련 된, cytometry 사용 하 여 산화 스트레스 모니터링 하는 방법을 설명 합니다. 접근은 아 밀 로이드 β 펩 티 드의 가용성과 집계 경향이 이체의 동작을 연구 하는 데 사용 됩니다.
초록
단백질 misfolding 및 녹말 체 conformations로 집계 발병 및 여러 신경 퇴행 성 질환의 진행에 관련 되어있다. 그러나, 여전히 약간의 정보는 어떻게 불용 성 단백질에 대 한 집계는 그들의 독성 효과 비보를 발휘. 단순 간결한 및 진 핵 모형 유기 체, 박테리아와 효 모, 등 세포내 녹말 체 형성, 집계 전파 및 독성 뒤에 있는 메커니즘의 우리의 현재 이해에 크게 기여 했다. 이 프로토콜에서 효를 사용 하 여 단백질의 형성 및 세포질 산화 스트레스에 미치는 영향 관계를 해 부 모델로 설명 합니다. 메서드는 amyloidogenic 단백질의 세포내 수용 성/집계 상태 감지 세포 산화 손상 cytometry (FC)을 사용 하 여 식에서 결과의 정량화 결합 합니다. 이 방법은 간단 하 고 빠르며, 양적입니다. 연구는 그들의 각각 고유한 집계 경향으로 아 밀 로이드 β 펩 티 드 이체의 큰 세트에 기인한 세포질 산화 스트레스를 연관 하 여 기법을 보여 줍니다.
서문
Proteostasis 셀 체력과 노화 프로세스의 근본적인 결정 이다. 셀, 네트워크는 올바른 refolding misfolded 단백질 conformers의 보호자 또는 그들의 타겟된 베이스로 몇 가지 잘 보존 메커니즘1 보장 하기 위한 정교한 단백질 품질 관리에 의해 단백질 항상성 유지 ,2,3,,45. 연구의 많은 발병과 인간의 질병의 광범위 한 범위와 proteostasis, 단백질 misfolding 및 집계의 실패의 진행 사이의 링크에 지원을 제공 합니다. 예를 들어, 단백질 예금의 존재는 많은 신경 퇴행 성 질환, 알츠하이머병, 파 킨 슨 병, 등, 헌팅턴의 질병6,7,8의 병 적인 특징을 간주 됩니다. prionogenic 질병, 고 퇴행 성 비 amyloidoses9. 그것은 집계 반응에서 초기 oligomeric 및 protofibrillar 어셈블리 붐비는 셀룰러 환경10다른 단백질 들과 탈 선 상호 작용을 설정 하는 세포 독성의 주요 elicitors는 건의 되었다. 또한, 전파 하는 그들의 독성 효과11,12셀 사이의 단백질 포함 (PI)를 전송할 수 있습니다. 따라서, PI의 대형 셀, 어디 처리 될 수 있다 또는 주요 부작용 없이 축적에서 특정 위치에 위험한 집계 된 종의 존재를 제한 하는 해독 메커니즘 구성 실제로 수 있습니다. 수 있습니다. 13 , 14.
표준 체 외에서 생 화 확 적인 접근 집계 반응와 그들의 속성15,16종에 대 한 중요 한 통찰력을 제공 했다. 그러나, 이러한 분석 실험에서 사용 하는 조건 셀 내에 발생 하는 명확 하 게 다른 있으며, 따라서 그들의 생리 적인 관련성 질문. 세포질 통로 단백질 품질 관리, autophagy, 진핵생물19,20,21 중 세포질 산화 환 원 상태17,18 의 규제 등의 주목할 만한 절약 때문에 ,,2223, 신진 효 모 Saccharomyces cerevisiae (S. cerevisiae) 단백질 집단의 분자 결정 요인 연구 특권 간단한 세포 모델 나왔다 그리고 생물학으로 관련 환경24,,2526에 관련 된 세포 독성 영향.
단백질 집계 성향 본질적으로 기본 순서로 인코딩 기능입니다. 따라서, 아 밀 로이드 같은 구조의 형성 예측할 수 있습니다 식별 및 집계 추진의 힘의 평가에 따라 지역 polypeptides27에. 그러나, 예측 단백질 시퀀스의 생체 외에서 집계 속성 bioinformatic 알고리즘의 성공에도 불구 하 고 그들은 여전히 이러한 경향 vivo에서 세포 독성 영향으로 번역 하는 어떻게 예측까지 있습니다. 체계적인 방식으로 주어진된 단백질의 집계 상태 및 그것의 관련 된 세포 손상을 사이 연결 하는 연구는 전산이 한계를 우회 하는 데 도움이 있습니다. 이 연결은 현재 연구, 아 밀 로이드 β 펩 티 드 Aβ42 단일 잔류물에만 서로 다른 하지만 집계 경향 vivo에서28의 연속 범위 표시의 이체의 큰 세트의 활용에서 해결 됩니다. 특히, 구조적 종 효 모 세포에서 집계 하기 쉬운 단백질에 의해 elicited 산화 손상에 대 한 회계를 식별 하는 FC 기반 접근 방식을 설명 합니다. 방법론에는 단순, 높은 처리 능력, 그리고 정확한 양적 측정 등 많은 이점을 제공합니다. 이 방법은 PI 놀이 산화 스트레스에 대 한 보호 역할을 확인 가능 합니다.
프로토콜
1. S. cerevisiae 문화와 단백질 표정
참고: Aβ 변종 전시 Aβ42 펩 티 드 (그림 1A)의 위치 19 (Phe19)에서 단일 잔류물에 돌연변이 인해 다른 상대 집계 경향. 이 펩 티 드 이체는 녹색 형광 단백질 (GFP), 집계 기자 (그림 1)29역할으로 태그 됩니다.
- 플라스 미드 BY4741 부모의 배경 (타 그의3Δ1 레이2Δ0 만난15Δ0 ura3Δ0)30효 모 세포로 20 Aβ42-GFP 변종에 대 한 인코딩 변환. 각 플라스 미드 GSSGSSG 링커에 의해 GFP를 융합 하는 Aβ42 돌연변이 인코딩합니다. 플라스 미드는 pESC(-URA) 이며 선택 가능한 마커 URA329를 포함.
- 다음 구성 요소와 uracil (사우스 캐롤라이나-우 라) 없이 합성 완전 한 매체 준비: 1.9 g/L 효 질소 uracil, 5 g/L 황산 암모늄과 900 mL ddH2O의 기본 20% (w/v) 포도 당 100ml와 결합.
- 2% 포도 당을 포함 하는 SC-우 라 중간의 20 mL로 변형 된 효 모 세포에의 한 식민지를 넣고 하룻밤 동요 210 rpm에서 30 ° C에서 문화를 성장.
- 다음 날, 신선한 SC-우 라 중간의 5 mL에 접종 100 µ L의 숙박 문화에 의해 새로운 효 모 세포 배양 성장.
- 에는 OD590 0.5, 원심 4 분 3000 x g에서 문화, 상쾌한, 삭제 고 resuspend 동일한 양의 신선한 SC-우 라 중 2% 유를 포함 하는 셀.
- 30 분 동안 210 rpm 동요에서 30 ° C에서 품 어. 그리고 4 분 3000 x g에서 세포를 원심는 상쾌한을 삭제 합니다. 재조합 형 단백질 표정 유도 2% 갈 락 토스를 포함 하는 신선한 SC-우 라 매체에 셀 resuspend
- 단백질 표정 2% 갈 락 토스를 포함 하는 사우스 캐롤라이나-우 라 중간에 유도의 16 h 후 4 분 3000 x g에서 원심 분리 하 여 살 균 microcentrifuge 관에 유도 세포의 1 mL를 수확.
참고: 가장 관련성이 높은 차이 발생 후 Aβ42 GFP 변종 표현 16 h. 유도 효 모 세포의 유도 기간 형광 현미경 검사 법 (그림 2B) 셀 안에 재조합 단백질 분포를 결정 하 여 구상 될 수 있다.
2. 세포 얼룩
참고: 신선한 비 유발 세포는 FC 분석 동안 형광 임계값을 설정 하는 부정적인 컨트롤으로 구성 해야 합니다.
- 16 h 유발 효 모 세포를가지고 고 590에서 그들의 광학 밀도 결정 nm (OD590). 0.1의 OD590 를 버퍼링 하는 멸 균 인산 염 분 (PBS)에 그들을 희석.
- 버퍼링 인산 염 분 x 1을 다음과 같이 준비: NaCl, KCl, 나2HPO4, 1.44 g의 0.2 g의 8 g을 추가 하 고 KH2포4 ddH2오의 800 mL의 0.24 g pH 7.4에 조정 후 1 L ddH2O의 솔루션 작성 HCl. 버퍼 0.2 µ m 필터를 통해 필터링.
- 레이블이 표시 된 튜브 (12 × 75 m m 라운드 하단 폴리스 티 렌)을 표현 하는 세포 정지를 전송 하 고 빛에서 그들을 보호. 그들은 비 유도 및 비-얼룩진 셀 함께 분석에 대 한 교류 cytometer에 로드할 준비가 다는 것을 확인 하십시오.
- 5 µ M의 최종 농도에 각 샘플을 (예를 들어, CellROX 딥 레드) 산화 스트레스 프로브를 추가 하 고 어둠 속에서 30 분 동안 30 ° C에 세포를 품 어.
- 부 화, 후 셀 1 x PBS로 3 회 세척 하 고 FC 그들의 분석 전에 버퍼의 동일한 볼륨에 그들을 resuspend.
3. 교류 Cytometry 분석
참고: 적절 한 레이저와 필터는 FC 설치를 사용 하 여 GFP와 산화 스트레스 프로브 형광 신호를 감지. 교류 cytometer GFP의 발견을 위한 488 nm 청색 레이저와 산화 스트레스 프로브 형광의 탐지를 위한 635 nm 빨간 레이저를 사용할 수 있습니다. GFP와 산화 스트레스 프로브 방출 형광의 인수는 각각 530/30 nm BP 필터와 660/20 BP 필터 수행 (표 1).
- 새 워크시트 패널 열기 에 클릭 하 고 다음 인수 점 플롯을 만들.
- 도구 모음에서 분산형 플롯 도구 를 선택 하 고 A를 만들기 음모 쪽 분산형-지역 변수 (SSC-)는 y 축 대 앞으로 피해 지역에 (FSC-A)는 x 축에 선형 눈금에.
- 도구 모음에서 분산형 플롯 도구 를 클릭 하 고 x 축 대에 변수 FL1 지역 (FITC)와 음모를 만들. FL3 지역 (APC)는 y 축에 로그 눈금.
- 악기 설정 아이콘을 클릭 합니다. 보상 탭을 클릭 하 고 모든 보상 수준을 설정 합니다. 인수 탭에서 클릭 하 고 기록 20000 이벤트의 총 수를 선택 합니다.
- 유량 아이콘 낮은흐름 속도 전환 하려면 클릭 합니다.
- 취득 탭 비 유도, 흠 없는 세포 실행 시작을 클릭 하 고 인구 중간 왼쪽 사분면에 배포 될 때까지 측면 분산형 및 앞으로의 악기 설정 에서 전압을 조정 합니다.
- 다각형 아이콘 세포 인구 세포 파편을 제외 하 고 주변 지역 R1 설정을 클릭 하 고이 문이 인구 p 1을 사용 하 여 모든 형광 점 플롯과 히스토그램 표시 (그림 3)에 대 한 R1 =.
- 형광 신호의 PMT 전압을 조정 하려면 셀 낮은 왼쪽된 사분면에 배포 될 때까지 튜닝 악기 설정 탭에서 이득 FL1 FL3 점 플롯에 흠 없는 셀을 실행 합니다.
- GFP 형광을 측정 하는 유도 셀에 샘플을 변경 합니다. 악기 설정 탭에서 인구 오른쪽 아래 사분면에 배포 될 때에는 FSC-A 대 FL1 (FITC) 이득을 설정 합니다. 게이트 (게이트 P2) 긍정적인 세포 인구를 정의 합니다.
- 변경 샘플 비 유발 산화 스트레스는 FSC-A 대 FL3 (APC) 도트 음모에 형광으로 표시 하려면 셀 스테인드. 튜닝 이득 셀 인구는 왼쪽 위 사분면에서 배 부 될 때까지. P3에 긍정적인 세포 인구 게이트입니다.
- 확인을 셀 형광을 나타내는 히스토그램 아이콘 두 히스토그램 플롯 합니다. 형광 강도 나타내는 FL1 (FITC) GFP 형광을 나타내는 x 축, 이며 FL3 (APC) 형광을 나타내는 y에 로그 눈금에 P2 및 P3 인구를 각각 적용 있는지 확인 합니다.
참고: 막대 그래프 오버레이 다른 Aβ 이체를 표현 하는 셀의 형광 프로필 간의 차이 설명 하기 위해 좋은 방법입니다.
4. 재조합 단백질 Immunodetection
- 단백질 표정 수준 결정, 16 h에 대 일 분, 4000 x g에서 원심 분리에 의해 유도 된 효 모 배양 수확을-80 ° c.에 셀 펠 릿 저장
- PBS에 16 h에 대 한 재조합 단백질을 표현 하는 수집 된 셀 resuspend 20의 OD590 를 각 Aβ42 GFP 돌연변이의 200 µ L 셀 정지를 준비 합니다.
- 총 단백질 분수 분석에 대 일 분 14000 x g에서 원심 분리 하 여 각 돌연변이의 100 µ L를 수확 하 고 효 모 세포의 용 해 버퍼 Y의 동일한 볼륨에 셀 펠 릿 resuspend-(50 mM Tris HCl pH 8.0, 1 %DMSO, 200 mM NaCl, 1 mM EDTA 10 mg/mL SB3-14 (myr 포함 된 당 istyl sulfobetaine) 보충 1 m m phenylmethylsulfonyl 불 (PMSF)).
- 온화한 동요에서 실 온에서 20 분에 대 한 샘플을 품 어.
- Bradford 분석 실험을 사용 하 여 단백질 추출 농도 결정 합니다. 15% 아크릴 SDS 페이지 젤 전기 이동 법에 60 분 100 V에서 polyvinylidene difluoride (PVDF) 막에 오 각 샘플의 단백질 추출의 최대 5 µ g를 로드 합니다.
- 브래드 퍼 드 Coomassie 화려한 블루 G-250의 100 밀리 그램으로 시 약 50ml 95% 에탄올에에서 용 해 하 고 85% (w/v) 인산의 100ml를 추가 준비. 다음, 염료 해산 완전히 ddH2O 1 l 혼합물을 희석 하 고 그냥 사용 하기 전에 셀 루 로스 필터 종이 통해 필터링.
참고: 브래드 퍼 드 시 해야 될 빛 갈색. - 소 혈 청 알 부 민 (BSA)의 브래드포드 시 약 1000 µ L에서 단백질의 5-100 µ g에서 배열의 표준 준비.
- 1-10 µ L 단백질의 브래드포드 시의 1 mL에 추출 하 고 품 어 5 분 동안 실내 온도에 혼합물을 추가 합니다.
- 표준과 마약595에서 단백질 추출 물 샘플의 흡 광도 측정 한다.
- 분석을 위해 표준 대에 대 한 얻은 흡 광도 값으로 음모를 확인 합니다. 단백질의 µ g입니다. 표준 곡선에 따라 달라 집니다, 있는 경우 볼륨 및 희석, 고려는 양의 단백질에서 원래 샘플의 농도 결정 합니다.
- 브래드 퍼 드 Coomassie 화려한 블루 G-250의 100 밀리 그램으로 시 약 50ml 95% 에탄올에에서 용 해 하 고 85% (w/v) 인산의 100ml를 추가 준비. 다음, 염료 해산 완전히 ddH2O 1 l 혼합물을 희석 하 고 그냥 사용 하기 전에 셀 루 로스 필터 종이 통해 필터링.
- 린스 Tris 버퍼 염 분 (TBS), x 1 막 0.1% Tween 20 (TTBS), 1 x TTBS의 5% (w/v) 무 지방 건조 우유를 사용 하 여 차단 하 고.
- 10 x TBS의 1 리터를 준비 하려면 기본 트리 스의 24 g와 H2Odd 900 mL에 NaCl의 88 g을 녹이 고 pH 7.6 조정. DdH2O l. 1의 최종 볼륨을 추가 10 x 재고 1 일부 ddH2o.의 9 부분 혼합 1 x 솔루션에 대 한
- 기본 β 아 밀 로이드 항 체 희석 6E10 1:1,000와 함께 1 시간에 대 한 막 품 어 그리고 이차 항 체 염소와 실 온에서 1 h에 대 한 반대로 마우스 IgG HRP 공액 희석 1:10, 000.
- 막 Luminata 형광 시 약의 1 mL를 사용 하 여 개발 및 단계 5.2에서에서 설명한 densitometric 분석 하 여 밴드를 계량 합니다.
5. 데이터 분석
- FC에서 얻은 데이터를 분석 하려면 표시 의미 형광 강도 (MFI) 테이블을 만듭니다와 그 해당 표준 오류 또는 GFP 형광 및 산화 스트레스 수준에 대 한 차이 (CV)의 계수 중간 형광.
- 관리자 탭 및 통계 아이콘 각 채널에 필요한 통계 데이터를 사용자 지정을 클릭 합니다.
참고: 비교할 때 단백질 이체, 접이식 배경 또는 해상도 통계 (RD)에 형광 값 데이터 변환: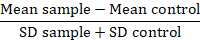
이 통계에서 평균 샘플 문제 샘플의 평균 형광, 평균 컨트롤은 컨트롤을 사용 하는 셀의 평균 형광 이며 SD는 평균 형광의 표준 편차. RD 요소 MFI 값 보다 더 정확한 인덱스는 그것 뿐만 아니라 샘플 뿐만 아니라 데이터의 확산을 위한 계정 사이의 차이 계산 합니다. 이 계산은 시간이 지남에 수행 다른 실험 데이터를 비교 하는 때 특히 적합 합니다. 일단는 RDs를 계산 통계 시험의 사용은 단백질 이체의 관찰 된 차이점의 중요성을 확인 하는 것이 좋습니다.
- 관리자 탭 및 통계 아이콘 각 채널에 필요한 통계 데이터를 사용자 지정을 클릭 합니다.
- 단백질 immunodetection에 대 한 재조합 단백질 수준 서쪽 오 점 ImageJ 소프트웨어를 사용 하 여 계량.
- Densitometry 분석 하기 전에 개발 된 막의 원래 raw 이미지를 JPEG 파일 형식와 회색조 모드 변환.
- 회색값 의미 분석 메뉴의 설정 측정 섹션을 조정 합니다.
- ImageJ에서 사각형 도구를 클릭 하 고 밴드 densitometry 분석에 대 한 일관 된 크기와 관심 (ROI)의 영역을 정의. 만든된 사각형 프레임 안에 막 이미지에 각 밴드 센터 고 측정 Ctrl + M (측정 명령)를 사용 하 여 하나 하나를 기록 합니다.
- 동일한 투자 수익 영역을 적용 하는 각 밴드에 인접 한 로컬 백그라운드를 측정 합니다. 측정 후 각각의 개별 밴드에서 해당 로컬 백그라운드를 뺍니다.
결과
이 프로토콜 어디 Phe19은 모든 자연 proteinogenic 아미노산28변경 되었습니다 Aβ42 펩 티 드의 20 이체의 컬렉션을 사용 하는 방법을 설명 합니다. 이 단백질의 이론적인 집계 경향 두 다른 bioinformatic 알고리즘 (AGGRESCAN와 탱고31,32)를 사용 하 여 분석할 수 있습니다. 두 경우 모두에서이 분석 집계 추세, ascribing, 일반?...
토론
다양 한 질병 세포 예금6,,78,33으로 misfolded 단백질의 축적에 연결 된다. 많은 노력 계정 단백질 농도를 고려 하지 않습니다, 계산 방법을 사용 하 여 이러한 질병의 발병을 트리거하는 분자 메커니즘을 해명 되었습니다 또는 생체 외에서 접근, 어떤 단백질 농도 반응 동안 상수 남아 있습니다. 그...
감사의 말
자료
| Name | Company | Catalog Number | Comments |
| Yeast cells BY4741 | ATCC | 201388 | Genotype: MATa his3Δ1 leu2Δ0 met15Δ0 ura3Δ0 |
| pESC(-Ura) plasmid | Agilent Genomics | 217454 | Yeast expression plasmid with a Gal promotor. Selectable marker URA3 |
| Yeast Synthetic Drop-out Medium Supplements | Sigma | Y1501 | Powder |
| Yeast Nitrogen Base Without Amino Acids | Sigma | Y0626 | Powder |
| Raffinose | Sigma | R7630 | Powder |
| Glucose | Sigma | G7021 | Powder |
| Galactose | Sigma | G0750 | Powder |
| Phosphate Buffered Saline (PBS) | Fisher Scientific | BP3991 | Solution 10X |
| CellROX Deep Red Reagent | Life Technologies | C10422 | Free radical cell-permeant fluorescent sensor, non-fluorescent while in a reduced state, and exhibits bright fluorescence upon oxidation by reactive oxygen species (ROS), with absorption/emission maxima at 644/665 nm. |
| Y-PER protein extraction reagent | Thermo Scientific | 78990 | Liquid cell lysis buffer |
| Acrylamide/Bis-acrylamide | Sigma | A6050 | Solution |
| Bradford dye reagent | Bio-Rad | 5000205 | Dye reagent for one-step determination of protein concentration |
| β-amyloid antibody 6E10 | BioLegend | 803001 | Mouse IgG1. The epitope lies within amino acids 3-8 of beta amyloid (EFRHDS). |
| Goat anti-mouse IgG-HRP conjugate | Bio-Rad | 1721011 | |
| Membrane Immobilon-P, PVDF | Millipore | IPVH00010 | |
| Luminata forte | Merk | WBLUF0100 | Premixed, ready to use chemiluminescent HRP detection reagent |
| Phenylmethanesulfonyl fluoride solution (PMSF) | Sigma | 93482 | Protease inhibitor. Dissolved at 0.1 M in ethanol |
| FACSCanto flow cytometer | BD Biosciences | 657338 | Equipped with a 488 nm blue laser for the detection of GFP, and 635 nm red laser / 530/30 nm BP filter and 660/20 BP filter |
| Mini Trans-Blot Electrophoresis Transfer cell | Bio-Rad | 1703930 | Protein transference system |
| Mini-PROTEAN Tetra Handcast Systems | Bio-Rad | 1658000FC | Electrophoresis system |
참고문헌
- Frydman, J. Folding of newly translated proteins in vivo: the role of molecular chaperones. Annual Review of Biochemistry. 70, 603-647 (2001).
- Hartl, F. U., Bracher, A., Hayer-Hartl, M. Molecular chaperones in protein folding and proteostasis. Nature. 475, 324-332 (2011).
- Wong, E., et al. Molecular determinants of selective clearance of protein inclusions by autophagy. Nature Communications. 3, (2012).
- Winkler, J., Tyedmers, J., Bukau, B., Mogk, A. Chaperone networks in protein disaggregation and prion propagation. Journal of Structural Biology. 179, 152-160 (2012).
- Glickman, M. H., Ciechanover, A. The ubiquitin-proteasome proteolytic pathway: destruction for the sake of construction. Physiological Reviews. 82, 373-428 (2002).
- Dobson, C. M. Getting out of shape. Nature. 418, 729-730 (2002).
- Chiti, F., Dobson, C. M. Protein misfolding, functional amyloid, and human disease. Annual Review of Biochemistry. 75, 333-366 (2006).
- Aguzzi, A., O'Connor, T. Protein aggregation diseases: pathogenicity and therapeutic perspectives. Nature Reviews Drug Discovery. 9, 237-248 (2010).
- Rapezzi, C., et al. Transthyretin-related amyloidoses and the heart: a clinical overview. Nature Reviews Cardiology. 7, 398-408 (2010).
- Deas, E., et al. Alpha-synuclein oligomers interact with metal ions to induce oxidative stress and neuronal death in Parkinson's disease. Antioxidants & Redox Signaling. 24, 376-391 (2016).
- Brundin, P., Melki, R., Kopito, R. Prion-like transmission of protein aggregates in neurodegenerative diseases. Nature Reviews Molecular Cell Biology. 11, 301-307 (2010).
- Frost, B., Diamond, M. I. Prion-like mechanisms in neurodegenerative diseases. Nature Reviews Neuroscience. 11, 155-159 (2009).
- Arrasate, M., Mitra, S., Schweitzer, E. S., Segal, M. R., Finkbeiner, S. Inclusion body formation reduces levels of mutant huntingtin and the risk of neuronal death. Nature. 431, 805-810 (2004).
- Ross, C. A., Poirier, M. A. Opinion: what is the role of protein aggregation in neurodegeneration?. Nature Reviews Molecular Cell Biology. 6, 891-898 (2005).
- Mishra, R., Sjölander, D., Hammarström, P. Spectroscopic characterization of diverse amyloid fibrils in vitro by the fluorescent dye Nile red. Molecular BioSystems. 7, 1232-1240 (2011).
- Klunk, W. E., Jacob, R. F., Mason, R. P. Quantifying amyloid by congo red spectral shift assay. Methods in Enzymology. 309, 285-305 (1999).
- Khurana, V., Lindquist, S. Modelling neurodegeneration in Saccharomyces cerevisiae: why cook with baker's yeast?. Nature Reviews Neuroscience. 11, 436-449 (2010).
- Tenreiro, S., Outeiro, T. F. Simple is good: yeast models of neurodegeneration. FEMS Yeast Research. 10, 970-979 (2010).
- Moosavi, B., Mousavi, B., Macreadie, I. G. Yeast model of amyloid-β and Tau aggregation in Alzheimer's disease. Journal of Alzheimer's Disease. 47, 9-16 (2015).
- Tenreiro, S., Munder, M. C., Alberti, S., Outeiro, T. F. Harnessing the power of yeast to unravel the molecular basis of neurodegeneration. Journal of Neurochemistry. 127, 438-452 (2013).
- Yang, J., Hao, X., Cao, X., Liu, B., Nyström, T. Spatial sequestration and detoxification of huntingtin by the ribosome quality control complex. eLife. 5, (2016).
- Braun, R. J., Büttner, S., Ring, J., Kroemer, G., Madeo, F. Nervous yeast: modeling neurotoxic cell death. Trends in Biochemical Sciences. 35, 135-144 (2010).
- Figley, M. D., Gitler, A. D. Yeast genetic screen reveals novel therapeutic strategy for ALS. Rare Diseases. 1, e24420 (2013).
- Cooper, A. A., et al. Alpha-synuclein blocks ER-Golgi traffic and Rab1 rescues neuron loss in Parkinson's models. Science. 313, 324-328 (2006).
- Johnson, B. S., McCaffery, J. M., Lindquist, S., Gitler, A. D. A yeast TDP-43 proteinopathy model: exploring the molecular determinants of TDP-43 aggregation and cellular toxicity. Proceedings of the National Academy of Sciences of the United States of America. 105, 6439-6444 (2008).
- Bharathi, V., et al. Use of ade1 and ade2 mutations for development of a versatile red/white color assay of amyloid-induced oxidative stress in saccharomyces cerevisiae. Yeast. 33, 607-620 (2016).
- Pallarès, I., Ventura, S. Advances in the prediction of protein aggregation propensity. Current Medicinal Chemistry. , (2017).
- Villar-Piqué, A., Ventura, S. Protein aggregation propensity is a crucial determinant of intracellular inclusion formation and quality control degradation. Biochimica et Biophysica Acta. 1833, 2714-2724 (2013).
- Navarro, S., Villar-Piqué, A., Ventura, S. Selection against toxic aggregation-prone protein sequences in bacteria. Biochimica et Biophysica Acta. 1843, 866-874 (2014).
- Morell, M., de Groot, N. S., Vendrell, J., Avilés, F. X., Ventura, S. Linking amyloid protein aggregation and yeast survival. Molecular BioSystems. 7, 1121-1128 (2011).
- Conchillo-Solé, O., et al. AGGRESCAN: a server for the prediction and evaluation of "hot spots" of aggregation in polypeptides. BMC Bioinformatics. 8, 65 (2007).
- Fernandez-Escamilla, A. M., Rousseau, F., Schymkowitz, J., Serrano, L. Prediction of sequence-dependent and mutational effects on the aggregation of peptides and proteins. Nature Biotechnology. 22, 1302-1306 (2004).
- Renner, M., Melki, R. Protein aggregation and prionopathies. Pathologie Biologie.(Paris). 62, 162-168 (2014).
- Carija, A., Navarro, S., de Groot, N. S., Ventura, S. Protein aggregation into insoluble deposits protects from oxidative stress. Redox Biology. 12, 699-711 (2017).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유