Для просмотра этого контента требуется подписка на Jove Войдите в систему или начните бесплатную пробную версию.
Method Article
Оценка влияния агрегации белков на сотовой окислительный стресс в дрожжи
В этой статье
Резюме
Агрегации белков вызывает сотовой оксидативного стресса. Этот протокол описывает метод для контроля внутриклеточных государства amyloidogenic белков и окислительного стресса, связанного с ними, используя проточной цитометрии. Этот подход используется для изучения поведения растворимых и подверженных агрегации вариантов пептида амилоид β.
Аннотация
Сворачиванию белков и агрегация в амилоида конформации связаны с появления и прогрессирования ряд нейродегенеративных заболеваний. Однако, есть еще немного информации о как нерастворимых белков агрегатов приложить их токсические эффекты в естественных условиях. Простая модель прокариот и эукариот организмов, таких как бактерии и дрожжи, значительно способствовали нашего нынешнего понимания механизмов за внутриклеточные амилоида формирования, распространения агрегатов и токсичности. В этом протоколе использование дрожжей описан как модель анализировать взаимосвязь между формирования белков агрегатов и их влияние на сотовой оксидативного стресса. Метод объединяет обнаружения внутриклеточных растворимый/агрегированы состояния amyloidogenic белка с количественная оценка сотовой оксидативного повреждения, вызванные его выражения, с помощью проточной цитометрии (FC). Этот подход является простым, быстрым и количественные. Исследование демонстрирует технику, корреляция сотовой окислительный стресс, вызванный большой набор вариантов амилоид β пептид с их соответствующих внутренних агрегации наклонности.
Введение
Proteostasis является основополагающим фактором, определяющим процессы фитнес и старение клеток. В клетках белок гомеостаз поддерживается контроль качества сложных белков, которые сетей, направленных на обеспечение правильного складывая смятых протеинов конформеров сопровождающих и/или их целенаправленных протеолиза с несколько хорошо сохранившихся механизмов1 ,-2,-3,-4,-5. Большое количество исследований поддерживать связь между наступлением и прогрессирование широкий спектр заболеваний человека и неудачи proteostasis, приводит к сворачиванию белков и агрегации. К примеру наличие белка месторождений считается патологическим отличительной чертой многих нейродегенеративных расстройств, таких как болезнь Альцгеймера, Паркинсона и Хантингтона заболеваний6,7,8, prionogenic заболевания и не дегенеративные amyloidoses9. Было высказано предположение о том, что раннего олигомерных и protofibrillar сборки в агрегации реакции являются основными elicitors цитотоксичность, установление аберрантных взаимодействиями с другими белками в переполненном клеточного окружения10. Кроме того белковых включений (PI) может передаваться между ячейками, пропагандирующих их токсический эффект11,12. Таким образом может быть, что формирование PI может действительно представляют собой механизм детоксикации, который ограничивает наличие опасных видов агрегированные в определенных местах в камере, где они могут быть обработаны или накопленные без серьезных побочных эффектов 13 , 14.
Стандарт в vitro биохимических подходов оказали важное понимание различных видов, которые заполняют агрегации реакций и их свойства,1516. Однако условия, используемые в этих анализов явно отличаются от тех, которые происходят в пределах ячейки и, таким образом, вопрос их физиологической значимости. Из-за заметным сохранение клеточных пути контроля качества белка, autophagy или регулирование сотовой окислительно-восстановительного состояния17,18 среди эукариот19,20,21 ,22,23, многообещающий дрожжей Saccharomyces cerevisiae (S. cerevisiae) стала привилегированных простой сотовая модель для изучения молекулярных детерминант агрегации белков и его связанные цитотоксическое влияние в биологически соответствующих средах24,25,26.
Склонность агрегации белков — это функция, по своей сути закодированы в первичной последовательности. Таким образом, формирование амилоид подобных структур можно предсказать на основе идентификации и оценки потенции агрегации содействия регионам в полипептиды27. Однако несмотря на успех bioinformatic алгоритмов предсказать свойства агрегации в vitro последовательностей белков, они все еще далеки от прогнозирования, как эти склонности перевести в vivo цитотоксическое влияние. Чтобы обойти это ограничение вычислительной может помочь исследования, которые касаются связи между агрегированных состояние данного белка и его связанные клеточных повреждений на систематической основе. Эта связь рассматривается в настоящем исследовании, воспользовавшись большой набор вариантов амилоид β пептида Aβ42 отличаются только в одном остатки, но отображение непрерывный спектр агрегации склонности в vivo28. В частности описывается FC-основанный подход к идентификации конформационные видов учета оксидативное повреждение, вызвал, подверженных агрегации белков в клетках дрожжей. Методология дает множество преимуществ, таких, как простота, высокой пропускная способность и точное количественное измерение. Этот подход позволил подтвердить, что PI играют защитную роль против окислительного стресса.
протокол
1. S. cerevisiae культур и выражения протеина
Примечание: Значения вариантов демонстрируют разные относительные агрегации наклонности из-за мутации в один остаток в позиции 19 (Phe19) Aβ42 пептид (рис. 1A). Эти пептиды варианты помечены Зеленый флуоресцентный белок (КГВ), который действует как агрегирование репортер (рис. 1)29.
- Трансформировать плазмид кодирования для 20 вариантов Aβ42-ГФП в дрожжевых клеток с BY4741 родительский фон (Мата его3Δ1 леи2Δ0 встретился с15Δ0 ura3Δ0)30. Каждый плазмида кодирует Aβ42 мутант, сливается с GFP компоновщиком GSSGSSG. Плазмиды pESC(-URA) и включают в себя маркер выбора URA329.
- Подготовить синтетических полного среднего без урацилом (SC-Ура) со следующими компонентами: 1,9 г/Л дрожжи азота базы без урацил, 5 г/Л сульфата аммония и 900 мл ddH2O в сочетании с 100 мл 20% (w/v) глюкозы.
- Положите один колонии преобразованные дрожжевых клеток в 20 мл среды SC-Ура, содержащий 2% глюкозы и расти культур на ночь на 30 ° C при агитации на 210 об/мин.
- Следующий день, растут новые культуры клеток дрожжей пересевать 100 мкл ночь культуры в 5 мл свежего SC-Ура среды.
- В ОД590 0.5 центрифуга культур на 3000 x g 4 мин, удалить супернатант и Ресуспензируйте клетки в том же объеме свежих средних SC-Ура, содержащий 2% Рафиноза.
- Инкубируйте на 30 ° C при агитации на 210 об/мин за 30 мин. Центрифуга клетки на 3000 x g 4 мин затем удалить супернатант. Ресуспензируйте клетки в свежих SC-Ура носитель, содержащий 2% галактозы побудить рекомбинантных белков.
- После 16 h побуждать выражение протеина в среде SC-Ура, содержащий 2% галактозы урожай 1 мл индуцированных клеток в стерильных microcentrifuge трубы центрифугированием в 3000 x g на 4 мин.
Примечание: Наиболее важные различия возникают после индукции период 16 h. индуцированные дрожжевых клеток выразить Aβ42-GFP варианты могут быть визуализированы путем микроскопии флуоресцирования для определения распределения рекомбинантных белков внутри клетки (рис. 2B).
2. ячейки пятнать
Примечание: Свежие-индуцированной клетки необходимы как отрицательный контроль учредить флуоресцентные порог во время анализа ФК.
- Возьмите 16 h индуцированной дрожжевых клеток и определить их оптической плотности на 590 Нм (590OD). Развести их в стерильных фосфатный буфер (PBS) OD590 0.1.
- Приготовляют следующим образом 1 x фосфатный буфер: добавить 8 г NaCl, 0,2 г KCl, 1,44 г Na2HPO4, и 0,24 г х2PO4 до 800 мл ddH2O. заполнить решение до 1 Л ddH2O после корректировки рН 7,4 с HCl. фильтр буфер через фильтр 0.2 мкм.
- Передать соответствующим образом помечены трубы (12 × 75 мм раунд дно полистирол) выражая клеточных суспензий и защищать их от света. Убедитесь, что они готовы быть загружены на проточный цитометр для анализа, вместе с-индуцированной и -окрашенных клеток.
- Добавьте зонд окислительного стресса (например, CellROX глубокий красный цвет) для каждого образца в конечной концентрации 5 мкм и инкубировать клетки на 30 ° C в течение 30 мин в темноте.
- После инкубации вымыть клетки 3 раза с ПБС и Ресуспензируйте их в такой же объем буфера перед их анализ по ФК.
3. поток Cytometry анализ
Примечание: Используйте ФК установки с соответствующим лазеры и фильтры для обнаружения GFP и сигнал флуоресценции зонда оксидативного стресса. Проточный цитометр оснащены 488 нм синий лазер для обнаружения GFP и 635 нм красный лазер для обнаружения флуоресценции зонда Оксидативный стресс может быть использован. Приобретение выбросов флуоресценции GFP и окислительного стресса зонд выполняется с 530/30 Нм ВР фильтр и фильтр 660/20 BP, соответственно (Таблица 1).
- Нажмите на Открыть новый лист панель и создать следующие участки точка приобретение.
- Выберите Точечная участок инструмент из панели инструментов и создать участок с переменными Scatter-Сиде (SSC-A) на оси y против вперед Scatter-области (FSC-A) в линейной шкалы на оси x.
- Нажмите кнопку Точечная участок инструмент из панели инструментов и создать участок с переменными значения параметров FL1-области (FITC) на оси против. FL3-области (APC) в логарифмическом масштабе шкалы по оси y.
- Щелкните значок Параметры инструментов . Перейдите на вкладку компенсации и задать все уровни компенсации. Нажмите на вкладку приобретения и выберите общее количество 20000 событий для записи.
- Щелкните значок Скорость потока , чтобы переключить скорость потока до минимума.
- Щелкните вкладку приобретать для запуска-индуцированной, безупречная клетки и отрегулировать напряжение в Инструмент настройки вперед и разброс стороне до тех пор, пока население распределяется в середине левый квадрант.
- Нажмите на значок многоугольник установить регион R1 вокруг популяции клеток, исключая ячейки мусор и использовать этот закрытый населения P1 = R1 для всех участков флуоресцентные точка и гистограмма представления (рис. 3).
- Чтобы настроить ПЛТ напряжение сигнала флуоресценции, запустите неокрашенных клетки в значения параметров FL1-FL3 точка сюжет, тюнинг выигрыш в вкладке Параметры инструмента до тех пор, пока клетки распространяются в нижнем левом квадранте.
- Измените образец индуцированных клетки для измерения флуоресценции GFP. На вкладке Настройка инструмента установите выигрыш в FSC-A против значения параметров FL1 (FITC) когда населения распространяется в правом нижнем квадранте. Определите положительный клеточный населения с ворот (ворота P2).
- Изменение образца для не индуцированной витражи ячейки для отображения оксидативного стресса, флуоресценции на участке FSC-A против FL3 (APC) точка. Настройка усиления до тех пор, пока популяции клеток распространяется в левом верхнем квадранте. Ворота положительный клеточный населения в P3.
- Сделайте две гистограммы участки с значок гистограммы представляют флуоресценции клеток. Представлять интенсивности флуоресценции, убедитесь, что значения параметров FL1 (FITC) представляющие GFP флуоресценции находится в оси x, и FL3 (APC) представляющие флюоресценция по оси y, применяя P2 и P3 населения в логарифмическом масштабе, соответственно.
Примечание: Гистограмма накладки являются хорошим способом для иллюстрации различий между профилями флуоресценции клеток, выражая различные варианты значения.
4. Рекомбинантный белок Immunodetection
- Чтобы определить уровнях выражения протеина, урожай культур дрожжей, индуцированной для 16 h центрифугированием в 4000 x g 6 мин и хранить клетки окатышей на-80 ° C.
- Ресуспензируйте собранных клетки, выражая рекомбинантных белков для 16 h в PBS. Подготовка 200 мкл клеточных суспензий каждого Aβ42-GFP мутант ОД590 20.
- Для анализа доли общего белка, урожай 100 мкл каждого мутант центрифугированием в 14.000 x g 20 мин и Ресуспензируйте гранулы клетки в том же объеме буфера lysis дрожжей Y-за (50 мм трис-HCl рН 8,0, 1% ДМСО, 200 мм NaCl, ЭДТА 1 мм, содержащие 10 мг/мл SB3-14 (myr istyl sulfobetaine) дополнены 1 мм phenylmethylsulfonyl фторид (PMSF)).
- Проинкубируйте образцы для 20 мин при комнатной температуре под мягким агитации.
- Определите концентрацию белка экстракт, используя assay Брадфорд. Загрузка до 5 мкг белка экстракта каждого образца на электрофорез геля акриламида 15% SDS-PAGE и пятно на мембрану винилидена фторид (PVDF) на 100 V 60 мин.
- Подготовьте Брэдфорд реагента с 100 мг Кумасси синим G-250 растворяют в 50 мл 95% этиловом спирте и добавить 100 мл 85% (w/v) фосфорной кислоты. Затем когда краситель полностью не растворится, разбавьте смесь с ddH2O до 1 Л и процеживают через фильтр целлюлозная непосредственно перед использованием.
Примечание: Брэдфорд Реагент должен быть светло-коричневый. - Подготовка стандарта бычьим сывороточным альбумином (БСА), начиная от 5-100 мкг белка в 1000 мкл Реагента Брэдфорд.
- Добавление 1-10 мкл белка экстракт 1 мл реагента Брэдфорд и инкубировать смесь при комнатной температуре в течение 5 мин.
- Измерьте absorbance стандартов и экстракт образцы протеина в ОД595.
- Для анализа сделайте сюжет с оптической плотности значения, полученные для стандартов против. мкг белка. На основе стандартной кривой, определите концентрации оригинальные образцы от количество белка, учитывая объем и разрежения, если таковые имеются.
- Подготовьте Брэдфорд реагента с 100 мг Кумасси синим G-250 растворяют в 50 мл 95% этиловом спирте и добавить 100 мл 85% (w/v) фосфорной кислоты. Затем когда краситель полностью не растворится, разбавьте смесь с ddH2O до 1 Л и процеживают через фильтр целлюлозная непосредственно перед использованием.
- Промойте мембрану с 1 x трис амортизированное saline (TBS), 0,1% 20 анимации (TTBS) и заблокировать его с помощью обезжиренное сухое молоко 5% (w/v) в 1 x TTBS.
- Для приготовления 1 Л 10 x TBS, распустить 24 g Tris база и 88 г NaCl в 900 мл H2Odd и отрегулировать pH 7.6. Добавить ddH2O окончательный объемом 1 л. 1 x решения смешайте 1 часть акций 10 x с 9 частей ddH2O.
- Проинкубируйте мембрану за 1 час с 1:1,000 6E10 разбавленный основное антитело β-амилоида и за 1 ч при комнатной температуре с козлом вторичное антитело конъюгат анти мыши IgG-ПХ разводят 1:10 000.
- Разработка мембраны с использованием 1 мл Luminata флуоресцентных реагентов и количественно полос денситометрических анализом, как описано в пункте 5.2.
5. анализ данных
- Чтобы проанализировать данные, полученные ФК, создайте таблицу отображение средней интенсивности флуоресценции (MFI) и средний флуоресценции с его соответствующей стандартной ошибки и/или коэффициент дисперсии (CV) для флуоресценции GFP и окислительного стресса.
- Нажмите на вкладку инспектора и статистики значок, чтобы настроить статистические данные, которые необходимы для каждого канала.
Примечание: При сравнении вариантов белка, преобразуйте флуоресценции значения данных в диван за фона или резолюции метрику (РР):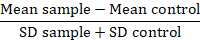
В этой метрике средний образец является среднее флуоресценции проблема образцов, средняя управления является среднее флуоресценции клеток, используемого как элемент управления, и SD — стандартное отклонение средней флуоресценции. RD фактором является более точный индекс, чем значение МФО, как он не только вычисляет разницу между образцами, но и счета для распространения данных. Этот расчет специально уместно при сравнении данных, поступающих из различных экспериментов со временем. После того, как были рассчитаны RDs, использование статистического теста рекомендуется подтвердить значимость наблюдаемых различий между вариантами белка.
- Нажмите на вкладку инспектора и статистики значок, чтобы настроить статистические данные, которые необходимы для каждого канала.
- Для белка immunodetection количественную оценку уровней рекомбинантных белков, Западная помарка, используя ImageJ программного обеспечения.
- Перед анализ денситометрии конвертировать в JPEG файла формата и серого режим оригинальных изображений raw развитых мембраны.
- Настройте Набор измерений раздел меню Analyze означаетзначение серый.
- Нажмите на инструмент прямоугольник с ImageJ и определить область интереса (ROI) с размером последовательного анализа денситометрия группы. Центр каждой полосы на изображении мембрана внутри созданного прямоугольную рамку и записывать измерения по одному с помощью Ctrl + M (команда измерения).
- Измеряют местные фон рядом с каждой группы, применение одной и той же области ROI. После измерения вычитайте соответствующие местные фон от каждой индивидуальной группы.
Результаты
Этот протокол описывает, как использовать коллекцию 20 вариантов Aβ42 пептид, где был мутировал Phe19 чтобы все аминокислоты природного proteinogenic28. Теоретические агрегации склонности этих белков могут быть проанализированы с использованием двух различных bioinfor...
Обсуждение
Широкий спектр заболеваний связано с накоплением смятых протеинов в клеточных вкладов6,,78,33. Многие усилия были сделаны разгадать молекулярные механизмы, которые вызывают наступления этих болезней с использованием ...
Благодарности
Материалы
| Name | Company | Catalog Number | Comments |
| Yeast cells BY4741 | ATCC | 201388 | Genotype: MATa his3Δ1 leu2Δ0 met15Δ0 ura3Δ0 |
| pESC(-Ura) plasmid | Agilent Genomics | 217454 | Yeast expression plasmid with a Gal promotor. Selectable marker URA3 |
| Yeast Synthetic Drop-out Medium Supplements | Sigma | Y1501 | Powder |
| Yeast Nitrogen Base Without Amino Acids | Sigma | Y0626 | Powder |
| Raffinose | Sigma | R7630 | Powder |
| Glucose | Sigma | G7021 | Powder |
| Galactose | Sigma | G0750 | Powder |
| Phosphate Buffered Saline (PBS) | Fisher Scientific | BP3991 | Solution 10X |
| CellROX Deep Red Reagent | Life Technologies | C10422 | Free radical cell-permeant fluorescent sensor, non-fluorescent while in a reduced state, and exhibits bright fluorescence upon oxidation by reactive oxygen species (ROS), with absorption/emission maxima at 644/665 nm. |
| Y-PER protein extraction reagent | Thermo Scientific | 78990 | Liquid cell lysis buffer |
| Acrylamide/Bis-acrylamide | Sigma | A6050 | Solution |
| Bradford dye reagent | Bio-Rad | 5000205 | Dye reagent for one-step determination of protein concentration |
| β-amyloid antibody 6E10 | BioLegend | 803001 | Mouse IgG1. The epitope lies within amino acids 3-8 of beta amyloid (EFRHDS). |
| Goat anti-mouse IgG-HRP conjugate | Bio-Rad | 1721011 | |
| Membrane Immobilon-P, PVDF | Millipore | IPVH00010 | |
| Luminata forte | Merk | WBLUF0100 | Premixed, ready to use chemiluminescent HRP detection reagent |
| Phenylmethanesulfonyl fluoride solution (PMSF) | Sigma | 93482 | Protease inhibitor. Dissolved at 0.1 M in ethanol |
| FACSCanto flow cytometer | BD Biosciences | 657338 | Equipped with a 488 nm blue laser for the detection of GFP, and 635 nm red laser / 530/30 nm BP filter and 660/20 BP filter |
| Mini Trans-Blot Electrophoresis Transfer cell | Bio-Rad | 1703930 | Protein transference system |
| Mini-PROTEAN Tetra Handcast Systems | Bio-Rad | 1658000FC | Electrophoresis system |
Ссылки
- Frydman, J. Folding of newly translated proteins in vivo: the role of molecular chaperones. Annual Review of Biochemistry. 70, 603-647 (2001).
- Hartl, F. U., Bracher, A., Hayer-Hartl, M. Molecular chaperones in protein folding and proteostasis. Nature. 475, 324-332 (2011).
- Wong, E., et al. Molecular determinants of selective clearance of protein inclusions by autophagy. Nature Communications. 3, (2012).
- Winkler, J., Tyedmers, J., Bukau, B., Mogk, A. Chaperone networks in protein disaggregation and prion propagation. Journal of Structural Biology. 179, 152-160 (2012).
- Glickman, M. H., Ciechanover, A. The ubiquitin-proteasome proteolytic pathway: destruction for the sake of construction. Physiological Reviews. 82, 373-428 (2002).
- Dobson, C. M. Getting out of shape. Nature. 418, 729-730 (2002).
- Chiti, F., Dobson, C. M. Protein misfolding, functional amyloid, and human disease. Annual Review of Biochemistry. 75, 333-366 (2006).
- Aguzzi, A., O'Connor, T. Protein aggregation diseases: pathogenicity and therapeutic perspectives. Nature Reviews Drug Discovery. 9, 237-248 (2010).
- Rapezzi, C., et al. Transthyretin-related amyloidoses and the heart: a clinical overview. Nature Reviews Cardiology. 7, 398-408 (2010).
- Deas, E., et al. Alpha-synuclein oligomers interact with metal ions to induce oxidative stress and neuronal death in Parkinson's disease. Antioxidants & Redox Signaling. 24, 376-391 (2016).
- Brundin, P., Melki, R., Kopito, R. Prion-like transmission of protein aggregates in neurodegenerative diseases. Nature Reviews Molecular Cell Biology. 11, 301-307 (2010).
- Frost, B., Diamond, M. I. Prion-like mechanisms in neurodegenerative diseases. Nature Reviews Neuroscience. 11, 155-159 (2009).
- Arrasate, M., Mitra, S., Schweitzer, E. S., Segal, M. R., Finkbeiner, S. Inclusion body formation reduces levels of mutant huntingtin and the risk of neuronal death. Nature. 431, 805-810 (2004).
- Ross, C. A., Poirier, M. A. Opinion: what is the role of protein aggregation in neurodegeneration?. Nature Reviews Molecular Cell Biology. 6, 891-898 (2005).
- Mishra, R., Sjölander, D., Hammarström, P. Spectroscopic characterization of diverse amyloid fibrils in vitro by the fluorescent dye Nile red. Molecular BioSystems. 7, 1232-1240 (2011).
- Klunk, W. E., Jacob, R. F., Mason, R. P. Quantifying amyloid by congo red spectral shift assay. Methods in Enzymology. 309, 285-305 (1999).
- Khurana, V., Lindquist, S. Modelling neurodegeneration in Saccharomyces cerevisiae: why cook with baker's yeast?. Nature Reviews Neuroscience. 11, 436-449 (2010).
- Tenreiro, S., Outeiro, T. F. Simple is good: yeast models of neurodegeneration. FEMS Yeast Research. 10, 970-979 (2010).
- Moosavi, B., Mousavi, B., Macreadie, I. G. Yeast model of amyloid-β and Tau aggregation in Alzheimer's disease. Journal of Alzheimer's Disease. 47, 9-16 (2015).
- Tenreiro, S., Munder, M. C., Alberti, S., Outeiro, T. F. Harnessing the power of yeast to unravel the molecular basis of neurodegeneration. Journal of Neurochemistry. 127, 438-452 (2013).
- Yang, J., Hao, X., Cao, X., Liu, B., Nyström, T. Spatial sequestration and detoxification of huntingtin by the ribosome quality control complex. eLife. 5, (2016).
- Braun, R. J., Büttner, S., Ring, J., Kroemer, G., Madeo, F. Nervous yeast: modeling neurotoxic cell death. Trends in Biochemical Sciences. 35, 135-144 (2010).
- Figley, M. D., Gitler, A. D. Yeast genetic screen reveals novel therapeutic strategy for ALS. Rare Diseases. 1, e24420 (2013).
- Cooper, A. A., et al. Alpha-synuclein blocks ER-Golgi traffic and Rab1 rescues neuron loss in Parkinson's models. Science. 313, 324-328 (2006).
- Johnson, B. S., McCaffery, J. M., Lindquist, S., Gitler, A. D. A yeast TDP-43 proteinopathy model: exploring the molecular determinants of TDP-43 aggregation and cellular toxicity. Proceedings of the National Academy of Sciences of the United States of America. 105, 6439-6444 (2008).
- Bharathi, V., et al. Use of ade1 and ade2 mutations for development of a versatile red/white color assay of amyloid-induced oxidative stress in saccharomyces cerevisiae. Yeast. 33, 607-620 (2016).
- Pallarès, I., Ventura, S. Advances in the prediction of protein aggregation propensity. Current Medicinal Chemistry. , (2017).
- Villar-Piqué, A., Ventura, S. Protein aggregation propensity is a crucial determinant of intracellular inclusion formation and quality control degradation. Biochimica et Biophysica Acta. 1833, 2714-2724 (2013).
- Navarro, S., Villar-Piqué, A., Ventura, S. Selection against toxic aggregation-prone protein sequences in bacteria. Biochimica et Biophysica Acta. 1843, 866-874 (2014).
- Morell, M., de Groot, N. S., Vendrell, J., Avilés, F. X., Ventura, S. Linking amyloid protein aggregation and yeast survival. Molecular BioSystems. 7, 1121-1128 (2011).
- Conchillo-Solé, O., et al. AGGRESCAN: a server for the prediction and evaluation of "hot spots" of aggregation in polypeptides. BMC Bioinformatics. 8, 65 (2007).
- Fernandez-Escamilla, A. M., Rousseau, F., Schymkowitz, J., Serrano, L. Prediction of sequence-dependent and mutational effects on the aggregation of peptides and proteins. Nature Biotechnology. 22, 1302-1306 (2004).
- Renner, M., Melki, R. Protein aggregation and prionopathies. Pathologie Biologie.(Paris). 62, 162-168 (2014).
- Carija, A., Navarro, S., de Groot, N. S., Ventura, S. Protein aggregation into insoluble deposits protects from oxidative stress. Redox Biology. 12, 699-711 (2017).
Перепечатки и разрешения
Запросить разрешение на использование текста или рисунков этого JoVE статьи
Запросить разрешениеСмотреть дополнительные статьи
This article has been published
Video Coming Soon
Авторские права © 2025 MyJoVE Corporation. Все права защищены