É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Avaliação do impacto da agregação da proteína no estresse oxidativo celular na levedura
Neste Artigo
Resumo
Agregação da proteína provoca estresse oxidativo celular. Este protocolo descreve um método para monitorar os Estados intracelulares de proteínas amiloidogénicos e o stress oxidativo associado a eles, usando citometria de fluxo. A abordagem é usada para estudar o comportamento de variantes do peptídeo β-amiloide solúveis e propensas a agregação.
Resumo
Enrolamento de proteínas e de agregação em conformações amiloides têm sido relacionados ao aparecimento e progressão de várias doenças neurodegenerativas. No entanto, ainda há pouca informação sobre proteína insolúvel como agregados exercem seus efeitos tóxicos em vivo. Organismos-modelo simples de procariotas e eucariotas, tais como bactérias e leveduras, têm contribuído significativamente para nossa compreensão atual dos mecanismos por trás da formação de amiloide intracelular, propagação de agregados e toxicidade. Neste protocolo, o uso de levedura é descrito como um modelo para dissecar a relação entre a formação de agregados de proteínas e seu impacto sobre o estresse oxidativo celular. O método combina a detecção de estado solúvel/agregados intracelular de uma proteína amiloidogénicos com a quantificação dos danos oxidativos celulares resultantes de sua expressão usando citometria de fluxo (FC). Essa abordagem é simples, rápido e quantitativo. O estudo ilustra a técnica correlacionando o estresse oxidativo celular, causado por um conjunto grande de variantes de peptídeo β-amiloide com suas propensões respectiva agregação intrínseca.
Introdução
Proteostasis é um determinante fundamental dos processos de adequação e envelhecimento celular. Nas células, homeostase de proteína é mantida pelo controle de qualidade de proteína sofisticadas redes visa garantir a correta dobrar de conformistas de misfolded proteínas por acompanhantes e/ou sua proteólise alvejado com vários mecanismos bem conservados1 ,2,3,4,5. Um grande número de estudos oferece suporte à ligação entre o início e a progressão de uma ampla gama de doenças humanas e o fracasso de proteostasis, levando ao enrolamento de proteínas e agregação. Por exemplo, a presença de depósitos de proteína é considerada uma característica patológica de muitas doenças neurodegenerativas, como Alzheimer, Parkinson e Huntington doenças6,7,8, doenças prionogenic e amyloidoses não-degenerativas9. Tem sido sugerido que cedo assemblies oligoméricos e protofibrillar na reação de agregação são os principais elicitores de citotoxicidade, estabelecendo interações aberrantes com outras proteínas no meio lotado de celular10. Além disso, inclusões de proteína (PI) podem ser transmitidos entre as células, seus efeitos tóxicos11,12de propagação. Portanto, pode ser que a formação de PI de fato pode constituir um mecanismo desintoxicante que restringe a presença de espécies perigosas agregados para locais específicos na célula, onde eles podem ser processados ou acumulados sem grandes efeitos colaterais 13 , 14.
Padrão em vitro abordagens bioquímicas forneceram importantes insights sobre as diferentes espécies que povoam as reações de agregação e suas propriedades de15,16. No entanto, as condições usadas nestes ensaios são claramente diferentes das que ocorrem dentro da célula e, portanto, questionar sua relevância fisiológica. Por causa de conservação notável dos caminhos celulares tais como controle de qualidade de proteína, autofagia ou o Regulamento do estado redox celular17,18 entre eucariotas19,20,21 ,22,23, a brotamento de levedura Saccharomyces cerevisiae (S. cerevisiae) surgiu como um modelo de celular simples privilegiado para estudar os determinantes moleculares de agregação da proteína e seu impacto associado citotóxico em ambientes biologicamente relevantes24,25,26.
Propensão de agregação de proteínas é uma característica inerentemente codificada na sequência principal. Assim, a formação de estruturas semelhantes a amiloide pode ser prevista com base na identificação e avaliação da potência de agregação-promover regiões em polipeptídeos27. No entanto, apesar do sucesso de bioinformatic algoritmos para prever as propriedades de agregação em vitro de sequências de proteínas, eles estão ainda longe de previsão como essas propensões traduzem na vivo citotóxicos impacto. Estudos que abordam a relação entre o estado agregado de uma determinada proteína e seu dano celular associado de forma sistemática podem ajudar a contornar essa limitação computacional. Esta conexão é abordada no presente estudo, aproveitando-se de um conjunto grande de variantes do peptídeo β-amiloide Aβ42 diferindo apenas em um único resíduo, mas indicando uma escala contínua de agregação propensões na vivo28. Em particular, uma abordagem baseada em FC para identificar a espécie conformacional representando o dano oxidativo eliciado propensas a agregação de proteínas nas células de levedura é descrita. A metodologia oferece muitas vantagens como simplicidade, capacidade de alta produtividade e precisão das medições quantitativo. Esta abordagem, foi possível confirmar essa jogada de PI um papel protetor contra o estresse oxidativo.
Protocolo
1. culturas de S. cerevisiae e expressão da proteína
Nota: Variantes Aβ apresentam propensões de agregação relativa diferente devido a uma mutação em um único resíduo na posição 19 (Phe19) do peptídeo Aβ42 (figura 1A). Essas variantes do peptide são marcados com a proteína fluorescente verde (GFP), que actua como uma agregação de repórter (Figura 1)29.
- Transforme os plasmídeos codificação para 20 variantes de Aβ42-GFP em células de levedura com um BY4741 fundo parental (MATa seu3Δ1 leu2Δ0 conheceu15Δ0 ura3Δ0)30. Cada plasmídeo codifica para um mutante de Aβ42 fundido a GFP por um vinculador GSSGSSG. Os plasmídeos são pESC(-URA) e incluem o marcador selecionável URA329.
- Prepare o suporte completo sintético sem uracil (SC-URA) com os seguintes componentes: 1,9 g/L nitrogênio levedura base sem uracil, 5g/L sulfato de amónio e 900 mL de DDQ2O combinado com 100 mL de glicose de 20% (p/v).
- Colocar uma colônia das células de fermento transformado em 20 mL do meio de SC-URA contendo 2% de glicose e crescem as culturas durante a noite a 30 ° C, sob agitação a 210 rpm.
- No dia seguinte, cresce novas culturas de células de levedura por inoculando 100 µ l de cultura durante a noite em 5 mL de meio fresco SC-URA.
- Em uma OD590 de 0.5, Centrifugar as culturas a 3.000 x g durante 4 min, desprezar o sobrenadante e ressuspender as células no mesmo volume de meio de SC-URA fresco contendo 2% rafinose.
- Incube a 30 ° C, sob agitação a 210 rpm por 30 min. Em seguida, Centrifugar as células a 3.000 x g, durante 4 min e descartar o sobrenadante. Ressuspender as células em meio de SC-URA fresco contendo 2% de galactose para induzir a expressão da proteína recombinante.
- Após 16 h de induzir a expressão de proteínas em meio de SC-URA contendo 2% de galactose, colha 1 mL de células induzidas em tubos estéreis microcentrifuga por centrifugação a 3.000 x g, durante 4 min.
Nota: As diferenças mais relevantes ocorrem após um período de indução de células de levedura induzida de 16 h. expressando variantes de Aβ42-GFP pode ser visualizado por microscopia de fluorescência para determinar a distribuição de proteína recombinante no interior das células (Figura 2B).
2. coloracao celular
Nota: As pilhas novas não induzida são necessárias como um controle negativo para estabelecer um limiar fluorescente durante uma análise FC.
- Levar as células de levedura induzida por h 16 e determinar sua densidade óptica, a 590 nm (OD590). Diluí-los em uma solução salina estéril tamponada de fosfato (PBS) para um OD590 de 0.1.
- Preparar 1 x solução salina tamponada fosfato como segue: adicionar 8 g de NaCl, 0,2 g de KCl, 1,44 g de Na2HPO4, e 0,24 g de KH2PO4 a 800 mL de DDQ2O. Preencha a solução até 1 L de DDQ2O depois de ajustar o pH para 7,4 com HCl. Filtre o buffer com um filtro de 0,2 µm.
- Transferir as suspensões celulares expressando para tubos apropriadamente rotulados (12 × 75 de poliestireno de hum fundo redondo) e protegê-los de luz. Certifique-se de que estão prontos para ser carregado no citômetro de fluxo para análise, juntamente com células não-induzido e não manchada.
- Adicionar a sonda de estresse oxidativo (por exemplo, CellROX vermelha) para cada amostra em uma concentração final de 5 µM e incube as celulas a 30 ° C por 30 min no escuro.
- Após a incubação, lavar as células 3 vezes com 1X PBS e ressuspendê-los no mesmo volume da reserva antes da sua análise pela FC.
3. fluxo Cytometry Analysis
Nota: Use uma configuração FC com lasers apropriados e filtros para detectar GFP e sinal de fluorescência de sonda de estresse oxidativo. Um citômetro de fluxo, equipado com um 488 nm azul do laser para a detecção de GFP e laser 635 nm vermelho para a detecção de fluorescência de sonda de stress oxidativo pode ser usado. Aquisição da fluorescência de emissão de GFP e sonda de estresse oxidativo é executada com um filtro de nm BP 530/30 e um filtro de BP 660/20, respectivamente (tabela 1).
- Clique em Abrir novo painel de planilha e crie os seguintes lotes do ponto de aquisição.
- Selecione a Ferramenta de plotagem Scatter na barra de ferramentas e criar um enredo com as variáveis Scatter-área lateral (SSC-A) sobre o eixo y vs Forward Scatter-área (FSC-A) em uma escala linear no eixo x.
- Clique na Ferramenta de plotagem Scatter na barra de ferramentas e criar um enredo com as variáveis FL1-área (FITC) sobre o eixo x vs. FL3-área (APC) em uma escala logarítmica no eixo y.
- Clique no ícone Configurações de instrumentos . Clique na guia de compensação e definir todos os níveis de compensação. Clique na guia de aquisição e selecione um número total de 20.000 eventos para ser gravado.
- Clique no ícone de Caudal para mudar a taxa de fluxo para baixo.
- Clique na guia de adquirir para começar a correr as células não-induzida, imaculadas e ajustar a tensão nas Configurações do instrumento do RCS e dispersão de lado até que a população é distribuída no quadrante médio esquerdo.
- Clique no ícone do polígono para definir uma região R1, em torno de população celular excluindo os restos da célula e usar esta população gated P1 = R1 para todos os lotes do ponto fluorescente e representações de histograma (Figura 3).
- Para ajustar a tensão de PGTO do sinal de fluorescência, execute as células imaculadas na trama ponto FL1-FL3, ajuste o ganho na guia Configuração do instrumento , até que as células são distribuídas no quadrante inferior esquerdo.
- Altere a amostra para as células induzidas para medir a fluorescência de GFP. Na guia Configuração do instrumento , defina o ganho no FSC-A vs FL1 (FITC) quando a população é distribuída no quadrante inferior direito. Defina a população de células positivas com um portão (portão P2).
- Mudança da amostra para o não-induzida manchado células para exibir o estresse oxidativo por fluorescência em um terreno de ponto FSC-A vs FL3 (APC). Ajuste o ganho até a população celular é distribuída no quadrante esquerdo superior. A população de células positivas em P3 de portão.
- Fazer duas parcelas de histograma com o ícone de histograma para representar a fluorescência da célula. Para representar a intensidade da fluorescência, certifique-se de FL1 (FITC) representando a fluorescência de GFP é no eixo x e FL3 (APC) representando a fluorescência é no eixo y, aplicando as populações P2 e P3 em uma escala logarítmica, respectivamente.
Nota: As sobreposições de histograma são uma boa maneira de ilustrar as diferenças entre os perfis de fluorescência das células expressando diferentes variantes Aβ.
4. recombinação da proteína Immunodetection
- Para determinar os níveis de expressão da proteína, colher as culturas de leveduras induzidas por 16 h por centrifugação a 4.000 x g durante 6 min e armazenar as pelotas de célula a-80 ° C.
- Ressuspender as células coletadas, expressando a proteína recombinante para 16 h em PBS. Prepare suspensões da pilha de 200 µ l de cada mutante de Aβ42-GFP para uma OD590 de 20.
- Para análise de fração de proteínas totais, colheita de 100 µ l de cada mutante por centrifugação a 14.000 x g por 20 min e ressuspender as pelotas de célula no mesmo volume de tampão de lise de leveduras Y-PER (50 mM Tris-HCl pH 8,0, 1% DMSO, 200 mM de NaCl, 1 mM EDTA, contendo 10 mg/mL (myr SB3-14 istyl sulfobetaine) suplementado com fluoreto de phenylmethylsulfonyl de 1 mM (PMSF)).
- Incube as amostras por 20 min em temperatura ambiente sob leve agitação.
- Determine a concentração de extrato de proteínas usando o ensaio de Bradford. Carrega até 5 µ g de extrato de proteína de cada amostra em um gel de electroforese de SDS-PAGE 15% acrilamida e borrão em uma membrana de difluoreto (PVDF) de polivinilideno a 100 V por 60 min.
- Prepare um Bradford reagente com 100 mg de azul de Coomassie brilhante G-250 dissolvido em 50 mL de etanol a 95% e adicionar 100 mL de ácido fosfórico 85% (p/v). Então, quando o corante é completamente dissolvido, diluir a mistura com o ddH2O de 1L e filtrar por papel de filtro de celulose imediatamente antes da utilização.
Nota: O reagente de Bradford deve ser marrom claro. - Prepare um padrão de albumina de soro bovino (BSA) variando de 5-100 µ g de proteína em 1.000 µ l de reagente de Bradford.
- Adicione 1-10 µ l de proteína extrair a 1 mL de reagente de Bradford e incubar a mistura à temperatura ambiente por 5 min.
- Medir a absorvância das normas e a proteína extrair amostras em OD595.
- Para a análise, fazer um gráfico com os valores de absorvância obtidos para os padrões vs. µ g de proteína. Com base na curva padrão, determine as concentrações das amostras originais da quantidade de proteína, considerando o volume e a diluição, se for o caso.
- Prepare um Bradford reagente com 100 mg de azul de Coomassie brilhante G-250 dissolvido em 50 mL de etanol a 95% e adicionar 100 mL de ácido fosfórico 85% (p/v). Então, quando o corante é completamente dissolvido, diluir a mistura com o ddH2O de 1L e filtrar por papel de filtro de celulose imediatamente antes da utilização.
- Lavar a membrana com 1 x salino Tris (TBS), 0.1% Tween 20 (TTBS) e bloqueá-lo usando leite seco desnatado 5% (p/v) em 1 x TTBS.
- Para preparar 1 L de 10 x TBS, dissolver 24 g de Tris base e 88 g de NaCl em 900 mL de H2Odd e ajustar o pH 7,6. Adicionar O ddH2até um volume final de 1 L. Para uma solução de 1 x, misturar 1 parte de estoque de x 10 com 9 peças de DDQ2O.
- Incubar a membrana para 1 h com um anticorpo primário de β-amiloide 6E10 diluído 1:1,000 e por 1h à temperatura ambiente com uma cabra de anticorpo secundário conjugado de anti-mouse IgG HRP diluído 01:10, 000.
- Desenvolver membranas usando 1 mL do reagente fluorescente Luminata e quantificar bandas por análise densitométricos, conforme descrito na etapa 5.2.
5. análise de dados
- Para analisar os dados obtidos pelo FC, criar uma tabela que exibe a média intensidade fluorescente (IFM) e fluorescência mediana com seu correspondente desvio-padrão e/ou o coeficiente de variação (CV) para os níveis de estresse oxidativo e fluorescência de GFP.
- Clique na guia de Inspetor e o ícone de estatísticas para personalizar os dados estatísticos que são necessários para cada canal.
Nota: Quando se comparam as variantes da proteína, converta dados valores de fluorescência para um fundo dobrável ou resolução métrica (RD):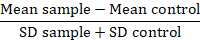
Nesta métrica, a amostra média é a média fluorescência das amostras do problema, o controle médio é a média fluorescência das células usado como um controle e o SD é o desvio-padrão da média fluorescência. Fator de RD é um índice mais exato do que o valor de IFM como ele não só calcula a diferença entre as amostras, mas também as contas para a divulgação dos dados. Este cálculo é especificamente relevante quando comparar dados provenientes de diferentes experiências realizadas ao longo do tempo. Uma vez que o RDs foram calculado, o uso de um teste estatístico é aconselhável confirmar a significância das diferenças observadas entre as variantes da proteína.
- Clique na guia de Inspetor e o ícone de estatísticas para personalizar os dados estatísticos que são necessários para cada canal.
- Para a proteína immunodetection, quantificar os níveis de proteína recombinante por Western blot utilizando software ImageJ.
- Antes da análise de densitometria, converta as imagens raw originais da membrana desenvolvida em modo JPEG arquivo formato e escala de cinza.
- Ajuste a seção Definir medições do menu Analyze para Dizer o valor de cinza.
- Clique na ferramenta retângulo do ImageJ e definir uma região de interesse (ROI) com um tamanho consistente para a análise de densitometria de banda. Centro de cada banda na imagem dentro da moldura retangular criada de membrana e registrar a medição de um por um, usando Ctrl + M (o comando de medição).
- Meça o fundo local adjacente à cada banda, aplicando a mesma área ROI. Após a medição, subtrai o fundo local correspondente de cada banda individual.
Resultados
Este protocolo descreve como empregar uma coleção de 20 variantes do peptide Aβ42 onde Phe19 alterou a todos os naturais proteinogenic aminoácidos28. A propensão de agregação teórica destas proteínas pode ser analisado usando dois algoritmos diferentes bioinformatic (AGGRESCAN e TANGO31,32). Em ambos os casos, esta análise processa uma gradação progressiva das tendências de agregação, atribui...
Discussão
Uma ampla gama de doenças está ligada ao acúmulo de misfolded proteínas em depósitos celulares6,7,8,33. Muitos esforços foram feitos para desvendar os mecanismos moleculares que desencadeiam o aparecimento destas doenças usando abordagens computacionais, que não levam em concentrações de proteína de conta, ou em vitro se aproxima, em que a concentração de proteína mantém...
Agradecimentos
Materiais
| Name | Company | Catalog Number | Comments |
| Yeast cells BY4741 | ATCC | 201388 | Genotype: MATa his3Δ1 leu2Δ0 met15Δ0 ura3Δ0 |
| pESC(-Ura) plasmid | Agilent Genomics | 217454 | Yeast expression plasmid with a Gal promotor. Selectable marker URA3 |
| Yeast Synthetic Drop-out Medium Supplements | Sigma | Y1501 | Powder |
| Yeast Nitrogen Base Without Amino Acids | Sigma | Y0626 | Powder |
| Raffinose | Sigma | R7630 | Powder |
| Glucose | Sigma | G7021 | Powder |
| Galactose | Sigma | G0750 | Powder |
| Phosphate Buffered Saline (PBS) | Fisher Scientific | BP3991 | Solution 10X |
| CellROX Deep Red Reagent | Life Technologies | C10422 | Free radical cell-permeant fluorescent sensor, non-fluorescent while in a reduced state, and exhibits bright fluorescence upon oxidation by reactive oxygen species (ROS), with absorption/emission maxima at 644/665 nm. |
| Y-PER protein extraction reagent | Thermo Scientific | 78990 | Liquid cell lysis buffer |
| Acrylamide/Bis-acrylamide | Sigma | A6050 | Solution |
| Bradford dye reagent | Bio-Rad | 5000205 | Dye reagent for one-step determination of protein concentration |
| β-amyloid antibody 6E10 | BioLegend | 803001 | Mouse IgG1. The epitope lies within amino acids 3-8 of beta amyloid (EFRHDS). |
| Goat anti-mouse IgG-HRP conjugate | Bio-Rad | 1721011 | |
| Membrane Immobilon-P, PVDF | Millipore | IPVH00010 | |
| Luminata forte | Merk | WBLUF0100 | Premixed, ready to use chemiluminescent HRP detection reagent |
| Phenylmethanesulfonyl fluoride solution (PMSF) | Sigma | 93482 | Protease inhibitor. Dissolved at 0.1 M in ethanol |
| FACSCanto flow cytometer | BD Biosciences | 657338 | Equipped with a 488 nm blue laser for the detection of GFP, and 635 nm red laser / 530/30 nm BP filter and 660/20 BP filter |
| Mini Trans-Blot Electrophoresis Transfer cell | Bio-Rad | 1703930 | Protein transference system |
| Mini-PROTEAN Tetra Handcast Systems | Bio-Rad | 1658000FC | Electrophoresis system |
Referências
- Frydman, J. Folding of newly translated proteins in vivo: the role of molecular chaperones. Annual Review of Biochemistry. 70, 603-647 (2001).
- Hartl, F. U., Bracher, A., Hayer-Hartl, M. Molecular chaperones in protein folding and proteostasis. Nature. 475, 324-332 (2011).
- Wong, E., et al. Molecular determinants of selective clearance of protein inclusions by autophagy. Nature Communications. 3, (2012).
- Winkler, J., Tyedmers, J., Bukau, B., Mogk, A. Chaperone networks in protein disaggregation and prion propagation. Journal of Structural Biology. 179, 152-160 (2012).
- Glickman, M. H., Ciechanover, A. The ubiquitin-proteasome proteolytic pathway: destruction for the sake of construction. Physiological Reviews. 82, 373-428 (2002).
- Dobson, C. M. Getting out of shape. Nature. 418, 729-730 (2002).
- Chiti, F., Dobson, C. M. Protein misfolding, functional amyloid, and human disease. Annual Review of Biochemistry. 75, 333-366 (2006).
- Aguzzi, A., O'Connor, T. Protein aggregation diseases: pathogenicity and therapeutic perspectives. Nature Reviews Drug Discovery. 9, 237-248 (2010).
- Rapezzi, C., et al. Transthyretin-related amyloidoses and the heart: a clinical overview. Nature Reviews Cardiology. 7, 398-408 (2010).
- Deas, E., et al. Alpha-synuclein oligomers interact with metal ions to induce oxidative stress and neuronal death in Parkinson's disease. Antioxidants & Redox Signaling. 24, 376-391 (2016).
- Brundin, P., Melki, R., Kopito, R. Prion-like transmission of protein aggregates in neurodegenerative diseases. Nature Reviews Molecular Cell Biology. 11, 301-307 (2010).
- Frost, B., Diamond, M. I. Prion-like mechanisms in neurodegenerative diseases. Nature Reviews Neuroscience. 11, 155-159 (2009).
- Arrasate, M., Mitra, S., Schweitzer, E. S., Segal, M. R., Finkbeiner, S. Inclusion body formation reduces levels of mutant huntingtin and the risk of neuronal death. Nature. 431, 805-810 (2004).
- Ross, C. A., Poirier, M. A. Opinion: what is the role of protein aggregation in neurodegeneration?. Nature Reviews Molecular Cell Biology. 6, 891-898 (2005).
- Mishra, R., Sjölander, D., Hammarström, P. Spectroscopic characterization of diverse amyloid fibrils in vitro by the fluorescent dye Nile red. Molecular BioSystems. 7, 1232-1240 (2011).
- Klunk, W. E., Jacob, R. F., Mason, R. P. Quantifying amyloid by congo red spectral shift assay. Methods in Enzymology. 309, 285-305 (1999).
- Khurana, V., Lindquist, S. Modelling neurodegeneration in Saccharomyces cerevisiae: why cook with baker's yeast?. Nature Reviews Neuroscience. 11, 436-449 (2010).
- Tenreiro, S., Outeiro, T. F. Simple is good: yeast models of neurodegeneration. FEMS Yeast Research. 10, 970-979 (2010).
- Moosavi, B., Mousavi, B., Macreadie, I. G. Yeast model of amyloid-β and Tau aggregation in Alzheimer's disease. Journal of Alzheimer's Disease. 47, 9-16 (2015).
- Tenreiro, S., Munder, M. C., Alberti, S., Outeiro, T. F. Harnessing the power of yeast to unravel the molecular basis of neurodegeneration. Journal of Neurochemistry. 127, 438-452 (2013).
- Yang, J., Hao, X., Cao, X., Liu, B., Nyström, T. Spatial sequestration and detoxification of huntingtin by the ribosome quality control complex. eLife. 5, (2016).
- Braun, R. J., Büttner, S., Ring, J., Kroemer, G., Madeo, F. Nervous yeast: modeling neurotoxic cell death. Trends in Biochemical Sciences. 35, 135-144 (2010).
- Figley, M. D., Gitler, A. D. Yeast genetic screen reveals novel therapeutic strategy for ALS. Rare Diseases. 1, e24420 (2013).
- Cooper, A. A., et al. Alpha-synuclein blocks ER-Golgi traffic and Rab1 rescues neuron loss in Parkinson's models. Science. 313, 324-328 (2006).
- Johnson, B. S., McCaffery, J. M., Lindquist, S., Gitler, A. D. A yeast TDP-43 proteinopathy model: exploring the molecular determinants of TDP-43 aggregation and cellular toxicity. Proceedings of the National Academy of Sciences of the United States of America. 105, 6439-6444 (2008).
- Bharathi, V., et al. Use of ade1 and ade2 mutations for development of a versatile red/white color assay of amyloid-induced oxidative stress in saccharomyces cerevisiae. Yeast. 33, 607-620 (2016).
- Pallarès, I., Ventura, S. Advances in the prediction of protein aggregation propensity. Current Medicinal Chemistry. , (2017).
- Villar-Piqué, A., Ventura, S. Protein aggregation propensity is a crucial determinant of intracellular inclusion formation and quality control degradation. Biochimica et Biophysica Acta. 1833, 2714-2724 (2013).
- Navarro, S., Villar-Piqué, A., Ventura, S. Selection against toxic aggregation-prone protein sequences in bacteria. Biochimica et Biophysica Acta. 1843, 866-874 (2014).
- Morell, M., de Groot, N. S., Vendrell, J., Avilés, F. X., Ventura, S. Linking amyloid protein aggregation and yeast survival. Molecular BioSystems. 7, 1121-1128 (2011).
- Conchillo-Solé, O., et al. AGGRESCAN: a server for the prediction and evaluation of "hot spots" of aggregation in polypeptides. BMC Bioinformatics. 8, 65 (2007).
- Fernandez-Escamilla, A. M., Rousseau, F., Schymkowitz, J., Serrano, L. Prediction of sequence-dependent and mutational effects on the aggregation of peptides and proteins. Nature Biotechnology. 22, 1302-1306 (2004).
- Renner, M., Melki, R. Protein aggregation and prionopathies. Pathologie Biologie.(Paris). 62, 162-168 (2014).
- Carija, A., Navarro, S., de Groot, N. S., Ventura, S. Protein aggregation into insoluble deposits protects from oxidative stress. Redox Biology. 12, 699-711 (2017).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados