Method Article
Edición del genoma mediada por CRISPR el hongo patógeno Candida albicans
* Estos autores han contribuido por igual
En este artículo
Resumen
Ingeniería eficiente del genoma de Candida albicans es fundamental para entender la patogénesis y el desarrollo de la terapéutica. Aquí, hemos descrito un protocolo de forma rápida y precisa modificar el genoma de C. albicans con CRISPR. El protocolo permite a los investigadores a introducir una gran variedad de modificaciones genéticas como mutaciones puntuales, inserciones y eliminaciones.
Resumen
Este método describe la eficiente CRISPR mediada por la edición del genoma del patógeno fúngico humano diploide Candida albicans. La edición del genoma mediada por CRISPR en C. albicans requiere Cas9, RNA de la guía y plantilla de reparación. Un plásmido que expresa un codón de levadura optimizado Cas9 (CaCas9) se ha generado. Guía secuencias directamente aguas arriba de un PAM sitio (NGG) son clonados en el vector de expresión Cas9. Una plantilla de reparación se hace por extensión de la cartilla en vitro. Cotransformation de la plantilla de la reparación y el vector en C. albicans conduce a la edición del genoma. Dependiendo de la plantilla de reparación utilizada, el investigador puede introducir cambios de nucleótidos, inserciones o eliminaciones. Como C. albicans es un diploide, se hacen las mutaciones en ambos alelos de un gen, siempre que los alelos A y B no abrigue SNPs que reparacion incorporación plantilla o interferir con la guía de orientación. Familias génicas plurinominales pueden editarse en paralelo existan adecuadas secuencias conservadas en todos los miembros de la familia. El sistema de C. albicans CRISPR descrito está flanqueado por sitios FRT y codifica flippase. Sobre inducción de flippase, el antibiótico marcador (CaCas9) y guía de RNA se extraen del genoma. Esto permite al investigador a realizar posterior al genoma. C. albicans CRISPR es una herramienta potente genética hongos y alteraciones menores a los protocolos descritos permiten la modificación de otras especies de hongos como C. glabrata, N. castellii, S. cerevisiae.
Introducción
Candida albicans es el hongo patógeno humano más frecuente1,2,3. Diferencias de comprensión entre C. albicans y mamífero biología molecular es fundamental para el desarrollo de la próxima generación de la terapéutica antifúngica. Esto requiere que los investigadores para poder rápidamente y con precisión genético manipular C. albicans.
Manipulación genética de C. albicans ha sido históricamente difícil. C. albicans no mantiene plásmidos, así todas las construcciones se deben incorporarse en el genoma. Además, C. albicans es diploide; por lo tanto, cuando la anulación de un gen o la introducción de una mutación, es importante asegurarse de ambas copias han sido cambiado4. Además, algunos loci de C. albicans son heterozigóticos, complicando aún más interrogatorio genético5. Para manipular genéticamente C. albicans, es típico realizar múltiples rondas de recombinación homóloga6. Sin embargo, la naturaleza diploide de genoma y laboriosa construcción desarrollo han hecho esto un proceso potencialmente tedioso, especialmente si se necesitan varios cambios. Estas limitaciones y la importancia médica de C. albicans exigen el desarrollo de nuevas tecnologías que permiten a los investigadores manipular más fácilmente el genoma de C. albicans .
Agrupadas regularmente otro corto repite palindrómico (CRISPR)-edición del genoma mediada es una poderosa herramienta que permite a los investigadores a cambio de la secuencia de un genoma. CRISPR requiere tres componentes: 1) la nucleasa Cas9 que hiende el ADN diana, 2) un 20 base RNA guía que se dirige a Cas9 a la secuencia de interés y 3) ADN plantilla incorpora el cambio previsto7, el sitio de la hendidura y la reparación 8. Una vez que la guía trae Cas9 para la secuencia del genoma blanco, Cas9 requiere una secuencia de motivo adyacente (PAM) de protospacer (NGG) directamente aguas arriba de la secuencia guía hender la DNA9. La exigencia de la guía de la base 20 y la secuencias de PAM proporciona un alto grado de especificidad de objetivos y límites objetivo escote.
CRISPR sistemas han sido diseñados para editar los genomas de un diverso conjunto de organismos y abordar una amplia variedad de problemas10. Se describe aquí es un protocolo CRISPR flexible y eficiente para la edición de un gen de C. albicans de interés. El experimento introduce un codón de parada en un gen, que causa la terminación de la traducción. Otras ediciones se pueden hacer dependiendo de la plantilla de reparación que se introduce. Un fragmento marcado con nourseothricin (Natr) que contiene levadura codón optimizado Cas9 (CaCas9) y una guía de RNA se incorpora en el genoma de C. albicans en un sitio neutral. Cotransformation con la plantilla de reparación codificación la mutación deseada lleva a la reparación de la hendidura por recombinación homóloga y la edición del genoma eficiente. Se describe a continuación está la edición de TPK2pero todo C. albicans open reading frames pueden ser objetivo varias veces de CRISPR. El sistema CRISPR está flanqueado por sitios FRT y puede extraerse el genoma de C. albicans por inducción de flippase codificada en el plásmido de expresión de CaCas9. El sistema de C. albicans CRISPR permite a investigadores con exactitud y rapidez de editar la C. albicans genoma11,12.
Protocolo
1. identificación y clonación de guía ARN secuencia
-
Identificación de la guía de la secuencia de ARN
- Identificar una 5'-NGG-3' secuencia de PAM cerca de donde se insertará el codón de parada. (Figura 1A) Etiquetado como todas las secuencias de PAM se encuentran en los primeros 100 pares de bases de TPK2 (figura 1A).
Nota: Guía secuencias dirigidas a cada marco de lectura abierto de C. albicans pueden encontrarse en http://osf.io/ARDTX 11,12. - Identificar la secuencia adelante guía Primer_3, que será el 20 bases directamente aguas arriba de un PAM NGG del sitio y no contendrá más de 5 Ts en una fila. Haga clic izquierdo en la base directamente aguas arriba de la NGG y arrastre el cursor de 20 bases, luego haga clic en la ficha de cartilla para agregar la cartilla.
- Identificar la secuencia Primer_3 inversa de la guía, que será el complemento a la secuencia de la guía hacia adelante.
Nota: Muestra son las guías que utilizan PAM_3 (figura 1B). - La cartilla con el botón derecho y seleccione "copiar datos de la cartilla". Pegar las secuencias en un programa de edición de texto.
- Identificar una 5'-NGG-3' secuencia de PAM cerca de donde se insertará el codón de parada. (Figura 1A) Etiquetado como todas las secuencias de PAM se encuentran en los primeros 100 pares de bases de TPK2 (figura 1A).
-
Añadir secuencias de proyección a oligos guía atrás y adelante para facilitar la clonación (cuadro 1, figura 1B).
- Agregue la secuencia de nucleótido ATTTG en el extremo 5' de la Primer_3 adelante guía antes de comprar.
- Añadir a G para el extremo 3' de la Primer_3 adelante guía antes de comprar.
- Agregar la secuencia de nucleótidos AAAAC en el extremo 5' de la Primer_3 inversa de guía antes de comprar.
- Añadir C al extremo 3' de la Primer_3 inversa de guía antes de comprar.
-
Digerir CaCas9 expresión vector pV1524 con BsmBI.
Nota: pV1524 contiene una ampicilina (Ampr) y nourseothricin (Natr) marcadores. Cas9 ha sido optimizado para codón para C. albicans.- Digerir el plásmido añadiendo: 2 μg de pv1524, 5 μL de 10 x Buffer, 1 μL de BsmBI y H2O a 50 μL en un tubo de 1,5 mL. Incubar a 55 ° C durante 20 min (alternativamente, digerir pv1524 durante 15 min con Esp3I, un isoschizomer de BsmBI, a 37 ° C.)
- Enfriar a temperatura ambiente (RT) y vuelta para 30 s a 2.348 x g para condensación en la parte inferior del tubo. Proceda al paso 1.4 o almacenar el plásmido digerido a-20 ° C.
-
Fosfatasa-tratar la columna vertebral digerida.
- Añadir 1 μL de fosfatasa intestinal becerro (CIP) a la mezcla de digestión e incubar a 37 ° C durante 1 h.
- Purificar el plásmido digerido utilizando un kit de potabilización de la reacción en cadena (PCR) polimerasa disponibles comercialmente (instrucciones incluidas con el kit) y eluir en 30 μL de tampón de elución (EB).
-
Fosforilan y recuece adelante guía Primer_3 y Primer_3 inversa de la guía.
- Añadir 0,5 μL de 100 μM adelante guía Primer_3, 0.5 μL de 100 μM Primer_3 inversa de la guía, 5 μL de tampón de ligasa de 10 x T4, 1 μL de la quinasa de T4 Polinucleótido y 43 μL de H2O a un tubo PCR.
- Añadir 5 μL de tampón de ligasa de 10 x T4, 1 μL de la quinasa de T4 Polinucleótido y 44 μL de grado biología molecular H2O en un segundo tubo PCR.
Nota: Esto servirá como control negativo. - Incubar las mezclas de reacción en un termociclador a 37 ° C durante 30 min, luego a 95 ° C durante 5 minutos.
- Enfriar la mezcla en la tasa más lenta de la rampa a 16 ° C a recueza, oligos. Luego coloque la mezcla de recocido oligo a 4 ° C.
-
Ligar los oligos recocido en pv1524 digerido de paso 1.4.3.
- Agregue lo siguiente a un tubo PCR: 1 μL de tampón de ligasa x T4 10, 0.5 μL de T4 ADN ligasa, 0.5 μL de mezcla de recocido oligo, digerido tratados con CIP purificada plásmido (20 – 40 ng) y H2O hasta un volumen total de 10 μL.
- Agregue lo siguiente a un tubo PCR: 1 μL de tampón de ligasa x T4 10, 0.5 μL de T4 ADN ligasa, digerido tratados con CIP vector purificado (20 – 40 ng), 1 μL de mezcla de control negativo y H2O hasta un volumen total de 10 μL.
- Incubar ambos tubos en un termociclador a 16 ° C por 30 min, luego a 65 ° C durante 10 minutos, luego enfriar a 25 ° C.
- Transforman químicamente competentes Escherichia coli DH5α utilizando un protocolo de transformación de choque de calor estándar a 5 μL de las mezclas de la ligadura. Seleccionar en medio LB Amp/Nat (200 μg/mL Amp, 50 μg/mL Nat).
Nota: Para seleccionar pV1524 y sus derivados en medios de comunicación de doble selección contrario, pérdida del módulo de Nat/CaCas9/guía por la supresión de FLP/FRT en bacterias. - Purificar plásmidos de cuatro transformantes mediante miniprep y la secuencia de inserción con la cartilla de la secuencia (tabla 1).
Nota: La mayoría del tiempo, secuencias 4 transformantes es suficiente para identificar al menos 1 copia correcta. - Guardar plásmidos que tienen la secuencia de RNA guía clonada de una sola vez en el BsmBI corte sitio a-20 ° C.
2. diseño y generación de reparación plantilla
- Inserte un codón de parada, con el botón izquierdo en la secuencia del gene y la adición de nucleótidos que codifican un codón y la restricción digestión sitio integrado (figura 1tabla 1).
Nota: La inserción será interrumpir la secuencia de PAM.
Nota: Un sitio de digestión de la restricción se incluirán en la secuencia de la plantilla de reparación para facilitar la eficiente detección de clones (figura 1). - Haga clic en 10 bases de aguas abajo de donde se hará la mutación y arrastrar que el cursor 60 bases río arriba. Haga clic en la ficha de cartilla para agregar la cartilla. Esto añadirá reparación plantilla Forward_3. Botón izquierdo del ratón 10 bases río arriba de donde se hará la mutación y arrastrar que el cursor 60 bases aguas abajo. Haga clic en la ficha de cartilla para agregar la cartilla. Esto añadirá reparación plantilla invertir 3. (Figura 1)
-
Realizar extensión de la cartilla para generar la plantilla de reparación.
- Añadir 1,2 μL de 100 μm reparación plantilla adelante la cartilla, 1,2 μL de 100 μm reparación plantilla inversa la cartilla, 6 μL de Deoxinucleótidos trifosfatos (dNTPs) (total concentración 40 mM), 6 μL de tampón, 0,6 μL de Taq polimerasa (3 unidades) y 45 μL de H2O para cada uno de los 4 PCR tubos.
- Realizar extensión de la cartilla entre 20 y 30 rondas de PCR. Las condiciones de extensión ejemplo: 2 min a 95 ° C, (30 s a 95 ° C, 1 min a 50 ° C, 1 min a 68 º C) x 34, 10 min a 68 º C.
- Combinar el contenido de los 4 tubos PCR en un tubo de 1,5 mL y utilizar un kit de potabilización de la polimerización en cadena para purificar productos en 50 μL de H2O.
- Cuantificar los productos de extensión de primer para asegurar suficiente ADN en la determinación de la absorbancia a 260 nm.
Nota: Concentración final típico del producto de la extensión de la cartilla es ~ 200-300 ng/μl.
3. transformación de C. albicans con reparación plantilla y plásmido
-
Hacer acetato de TE/litio.
- Mezcla 10 mM Tris-Cl, 1 mM EDTA, acetato de litio de 100 mM y H2O (todos solución stock pH 7.5) para alcanzar el volumen total de 50 mL.
-
Hacer placa
- Mezclar 40% PEG 3350, acetato de litio de 100 mM, 10 mM Tris-Cl, 1 mM EDTA y H2O (todos solución stock pH 7.5) para lograr el volumen total de 50 mL.
-
Digerir correctamente clonado plásmidos de paso 1.9.
- Añadir 10 μg de plásmido, 4 μL de tampón 10 x, 0.4 μL de 10 mg/mL de albúmina sérica bovina (BSA), 0,5 μl de KpnI, 0,5 μl de SacI y H20 y 40 μl de volumen en un tubo de 1,5 mL total. Incubar a 37 ° C durante la noche (figura 1).
-
Cultivar una cultura de la noche a la mañana de C. albicans SC5314, tipo prototroph, a 25 ° C en dextrosa peptona de levadura complementado con 0,27 mM uridina (YPD + Uri), idealmente a OD600 menos de 6.
- 5 unidades de OD600 células por transformación de pellets por giro durante 5 min a 2.348 x g y suspender el 5 OD600 células sedimentados en acetato de TE/litio de 100 μl.
- Agregue lo siguiente a un tubo de 1,5 mL en el orden indicado: 1) 100 μl de células de paso 3.4.1, 2) 40 μl de esperma de salmón cocida y enfriado rápido ADN (10 mg/mL), 3) 10 μg de digestión de plásmidos de paso 3.3.1, 4) 6 μg de reparación purificada plantilla y 5) 1 mL de la placa.
- Agregue lo siguiente a un tubo de 1,5 mL en el orden indicado: 1) 100 μl de células, 2) 40 μl de esperma de salmón cocida y enfriado rápido ADN (10 mg/mL), volumen de2O 3) H igual a la de transformación de ADN en el paso 3.5 y 4) 1 mL de la placa.
Nota: Esto servirá como control negativo. - Mezclar suavemente las transformaciones mediante pipeteo y dejar incubar a 25 ° C durante la noche.
- Calor del choque las células colocándolos en un baño de agua de 44 ° C durante 25 minutos.
- Gira por 5 min a 2.348 x g en una centrífuga de sobremesa y retire la mezcla de placa flotante. Lavar una vez añadiendo 1 mL de YPD + Uri y centrifugar nuevamente por 5min a 2.348 x g.
- Suspender las células en 0,1 mL de YPD + Uri e incubar en un tambor de rodillo o coctelera a 25 ° C durante la noche.
- Placa en YPD + Uri con 200 μg/mL nourseothricin. Las colonias aparecen en 2 a 5 días.
4. las rayas de colonias individuales
-
Divida un 100 x 15 mm placa de Petri en cuartos y cada cuadrante de la etiqueta.
- Toque una de las colonias de la placa de transformación con un palillo de dientes estéril o aplicador y raya en el lado más largo del cuadrante.
- Raya de colonias individuales utilizando una técnica aséptica y permitir que las colonias al crecer a 30 ° C durante 2 días.
5. Colonia PCR
-
Diseño de los cheque adelante cartilla (FCP) ~ 200 pares de bases corriente arriba del sitio de restricción que se introdujo y los verificación inversa primer (RCP) ~ 300 pares de bases corriente abajo.
- Añadir 0,3 μL de FCP, 0,3 μL de RCP, 0,3 μL de polimerasa termoestable (ExTaq 1.5 unidades), 3 μL de dNTPs (total concentración 40 mM), 3 μL de tampón ExTaq y 23 μL de H2O a un tubo de 1,5 mL.
Nota: Adición de 0,5 μL reacción dimetil sulfóxido (DMSO) puede mejorar la eficiencia de la PCR. - Añadir 0,25 μL de una colonia de levadura solo de paso 4.1.2 a la mezcla del paso 5.1.1, utilizando una pipeta P10 y teniendo cuidado de no para disturbar el agar.
- Amplificación de ADN por PCR y ejecutar 5 μL de la PCR en un gel para amplificación sea exitosa, teniendo cuidado de no para disturbar el precipitado de células en la parte inferior del tubo.
- Añadir 0,3 μL de FCP, 0,3 μL de RCP, 0,3 μL de polimerasa termoestable (ExTaq 1.5 unidades), 3 μL de dNTPs (total concentración 40 mM), 3 μL de tampón ExTaq y 23 μL de H2O a un tubo de 1,5 mL.
6. restricción digestión de Colony PCR
- Añada 10 μL de PCR producto (teniendo cuidado de no para no molestar el precipitado de células en la parte inferior del tubo), 3 μL de tampón, 1 μL de enzima de la restricción y 16 μL de H2O, luego incubar según las instrucciones del fabricante y resolver en un gel de agarosa para identificar corre ediciones de genoma de CT.
Nota: La enzima de restricción utilizada aquí es el sitio codificado en la plantilla de reparación específicos TPK2 .
7. ahorro de cepas
- Cultivar una cultura de la noche de las colonias que han sido confirmados por la digestión de la restricción en YPD + Uri a 30 ° C.
- Añadir 1 mL de la cultura y 1 mL de glicerol al 50% (lo que la concentración final de glicerol al 25%) a un tubo adecuado para el almacenamiento a-80 ° C.
- Almacenar los clones correcta a-80 ° C.
Nota: Tensiones correctas pueden almacenarse a-80 ° C durante muchos años.
8. eliminación de Natr marcador
- Planta correcta raya en maltosa de levadura peptona (YPMaltose) (2% maltosa).
- Elegir una colonia de la placa del veteado y la levadura durante 48 h a 30 ° C en maltosa de 20 g/L de líquido YPMaltose de la cultura.
- 200-400 células en maltosa de 20 g/L de YPMaltose de la placa e incubar a 30 ° C por 24 h.
- Replicar la placa en YPMaltose y YPMaltose 200 μg/mL NAT.
- Incubar a 30 ° C por 24 h.
Nota: Las colonias que ya no crecen en YPMaltose 200 μg/mL nourseothricin pero crecen en YPMaltose han perdido el Natr marcador (CaCAS9) y RNA de la guía. - Excepto las cepas que han perdido el Natr marcador (CaCAS9) y guía de RNA como hecho en pasos 7.1 – 7.3.
Nota: Un plásmido similar, pV1393, utiliza el SAP2 frente a un promotor MAL2 . Crecimiento en YCB-BSA inducen flippase y eliminación de Natr si pV1393 se utiliza para la edición de genes.
Resultados
Secuencias de guía RNAs y plantillas de reparación que C. albicans TPK2, una subunidad catalítica de la quinasa de c-AMP, fueron diseñados según las directrices sugeridas arriba. Secuencias se muestran en la (tabla 1, figura 1). Guía RNAs fueron clonados en vectores de expresión de CaCas9 y cotransformed con reparación plantilla tipo C. albicans. Un sitio de digestión de restricción EcoRI y codones de parada en la plantilla de reparación alteran el sitio de PAM y facilitan la detección de mutantes correcta (figura 1). Transformantes fueron con las colonias solo reflejos y defendidos por digestión de restricción y PCR de Colonia para la incorporación de la plantilla de reparación (figura 2). Digestión de restricción distingue rápidamente tipo salvaje de secuencias mutantes.
| Nombre de oligonucleótidos | Secuencia del oligonucleótido |
| Guía hacia adelante Primer_3 | ATTTGgggtgaactatttgttcgccG |
| Guía inversa Primer_3 | AAAACggcgaacaaatagttcacccC |
| Reparación plantilla Forward_3 | tcagcaatatcagcaacaatttcaacaaccgcagcaacaactttatTAAGAATTCggcga |
| Reparación plantilla Reverse_3 | attttgtccagtttgggctgcagcagggtgaactatttgttcgccGAATTCTTAataaag |
| Cartilla de verificación hacia adelante | ttaaagaaacttcacatcaccaag |
| Cartilla de verificación inversa | actttgatagcataatatctaccat |
| Cartilla de la secuencia | ggcatagctgaaacttcggccc |
Tabla 1: lista de oligo nucleótidos utilizada para este estudio. Sitios para fines de clonación se capitalizan y en negrita en las secuencias de la cartilla guía. Secuencias en la plantilla de reparación que mutan el ADN genómico se capitalizan y en negrita.
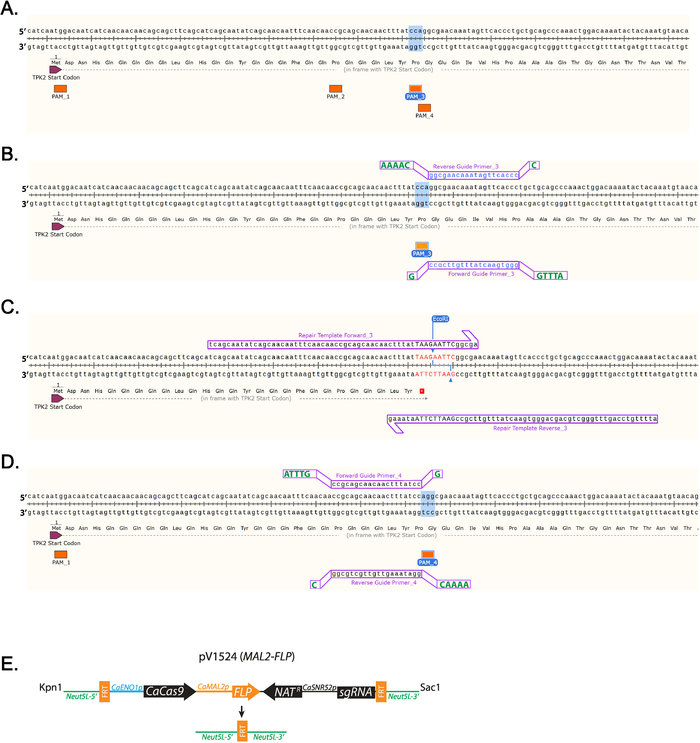
Figura 1 : Diagrama de la guía de diseño de plantilla de ARN y reparación. Etiquetado (A) de todas las secuencias de PAM en los primeros 100 nucleótidos de TPK2. Secuencia de PAM 3 (PAM_3) se destaca, ya es la secuencia que hemos utilizado en este estudio. (B) Guía de ARN diseño usando PAM_3. Imprimaciones base 20 diseñados usando SnapGene son minúsculas y azul. Bases adicionales necesarios para la clonación son mayúsculas y verde se muestra la compensación. (C) reparación cartillas de plantilla que inserte un codón TAA y EcoRI sitio se insertan en el TPK2 marco de lectura. El ADN que difiere de la secuencia de tipo salvaje es rojo y mayúsculas. (D) ejemplo de cómo una guía está diseñada en la cadena positiva de ADN mediante PAM_4. (E) diagrama esquemático del pV1524 después de la clonación de la guía de RNA y la digestión con KpnI y SacI. Neut5L-5' y Neut5L-3' objetivo el vector en el sitio Neut5L en el genoma de C. albicans . CaENO1p es el promotor que impulsa la expresión de la levadura-optimizado CaCas9. NATr es la cinta de resistencia nourseothricin. CaSNR52p es el promotor conducir expresión de RNA guía (sgRNA). FRT sitios son troceados y recombinados por flippase (FLP) quitar el cassette CRISPR a flippase expresión. Un esquema similar a (E) fue publicado por Vyas et al. 11. por favor haga clic aquí para ver una versión más grande de esta figura.
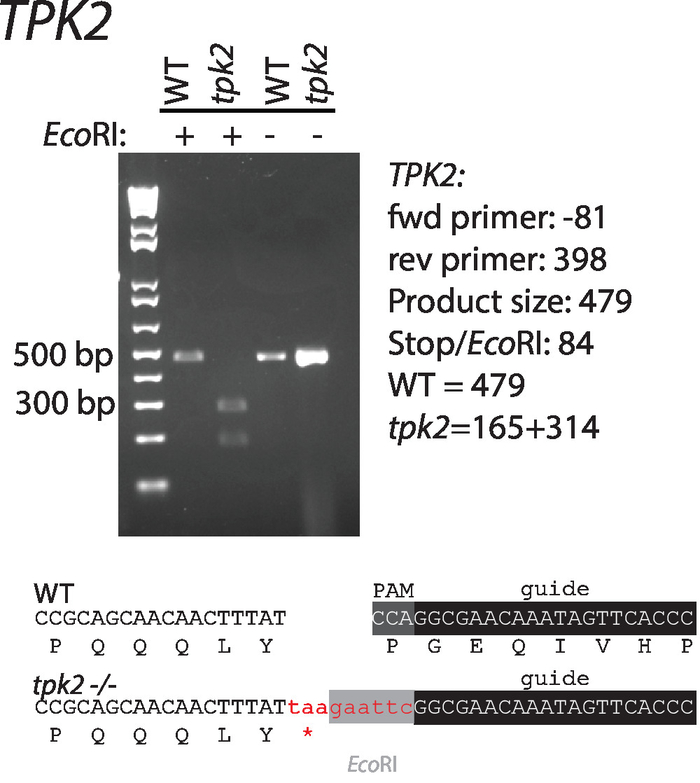
Figura 2 : Introducción y confirmación de un codón de parada y el sitio de restricción EcoRI a TPK2. Cebadores utilizados para la amplificación se enumeran en la tabla 1. Abajo el gel se muestran secuencias de tipo salvaje y mutantes. Esta figura ha sido modificada desde Vyas et al11. Haga clic aquí para ver una versión más grande de esta figura.
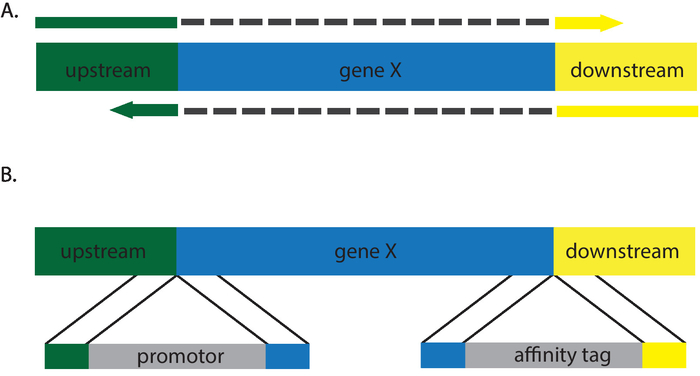
Figura 3 : Dibujos animados de descripción de plantillas de reparación que generará supresiones (A) y (B) inserciones. Gris guiones en (A) representan secuencias intervinientes no presentes en los iniciadores de la plantilla de reparación. Haga clic aquí para ver una versión más grande de esta figura.
Discusión
C. albicans CRISPR eficientemente modifica el genoma de C. albicans . pV1524 codifica una levadura Cas9 optimizado de codón y está diseñado de modo que los investigadores pueden clonar fácilmente secuencias guía RNA aguas abajo de la CaSNR52 promotor (figura 1)11. Se debe asegurar que una sola copia de la secuencia de la guía ha sido clonada en vectores de expresión CaCas9 por secuenciación, como copias adicionales impedirá la edición del genoma. Si constantemente se introducen múltiples copias de la guía, se debe bajar la concentración de la guía de recocido en la ligadura. El vector y el protocolo descrito permiten atacar cualquier gene de C. albicans . Si bien C. albicans es diploide, sólo una simple transformación es necesaria para ambos alelos de un gen de destino. Además, el carácter procesual de la edición del genoma CRISPR-CaCas9 permite a los investigadores a varios miembros de familias génicas. Muchas familias de genes secretadas aspartil proteasas (PAE) como proteínas aglutinina-como secuencia (ALS) son importantes para la virulencia de C. albicans . CRISPR edición del genoma facilitará la investigación de estas familias de genes.
Los protocolos descritos introducen a un codón de parada en un marco de lectura abierto, dando por resultado el equivalente fenotípico de un valor nulo (figura 2). Una amplia variedad de alteraciones genéticas puede hacerse mediante la variación de la plantilla de reparación. Absurdo, sin sentido y mutaciones silenciosas pueden insertarse mediante recombinación con una plantilla de reparaciones correspondiente. Incorporación de un sitio de restricción optimiza la proyección de la planta, como los sin deben ser examinados mediante la secuenciación de12,13. Además, C. albicans CRISPR permite a los investigadores generar inserciones y deleciones, lo que es un sistema ideal para insertar etiquetas de afinidad, realizar swaps promotor y generar agujeros ciegos (figura 3). Selección de transformantes correcta para estas mutaciones es más laborioso, ya que es necesario secuenciar las ediciones para confirmar la correcta incorporación de las plantillas de reparación. Además, Southern blot puede ser necesario para copias adicionales de un gen no se han insertado en ubicaciones adicionales en el genoma. El requisito del sitio NGG PAM lugares leves limitaciones en las regiones del genoma que pueden ser objeto. El desarrollo de sistemas alternativos de CRISPR que utiliza nucleasas alternativas como Cpf1 o variaciones en el Cas9 tener/va aliviar muchas de estas limitaciones14. A conocimiento de los investigadores en este momento, estos sistemas no se han aplicado todavía a C. albicans.
El sistema CRISPR descrito en el protocolo anterior se ha desarrollado tal que puede ser aplicado en una gran variedad de especies como Saccharomyces cerevisiae, Naumouozyma castellii, patógeno humano glabrata de la Candida11 . Transformación y la edición eficaz de estas levaduras requiere pequeños cambios en el protocolo descrito, pero el marco para la edición de estos genomas alternativos es notablemente similar a la descrita para C. albicans12. Además, la levadura proporciona un excelente mecanismo para el desarrollo de procedimientos de edición genómica. En la levadura, cuando ADE2 es transformado, un precursor de la ruta de biosíntesis de adenina se acumula, rojo que da vuelta las células. Este fenotipo fácilmente observable permite a los investigadores identificar células editadas y solucionar rápidamente problemas genoma edición de protocolos. Combinado con la caja de herramientas de biología molecular amplia disponible para hongos, protocolos para la edición de numerosas especies de levaduras han sido desarrollados15,16. Un amplio uso de la tecnología de edición de genoma de hongos tiene el potencial para un impacto significativo en una amplia variedad de disciplinas científicas.
CRISPR ha mejorado enormemente la eficiencia de la ingeniería del genoma de C. albicans, pero hasta la fecha CRISPR no se ha utilizado para realizar pantallas genoma de C. albicans. Los protocolos actuales requieren una reparación plantilla para introducir mutaciones, como la nonhomologous terminan uniendo vía en C. albicans es ineficiente12. La generación de reparación plantilla oligos para cada gen es una barrera significativa a la ejecución de pantallas de todo el genoma. La confluencia de los costos de disminución de la síntesis de ADN y los avances tecnologías CRISPR hará más factible desarrollo de bibliotecas de la canceladura. Por ejemplo, la expresión de una plantilla de reparación desde el vector de CaCas9 allana el camino para el desarrollo de las bibliotecas de plásmido sostenible que cada gen11. Además, protocolos CRISPR Candida transitorios que no requieren de vectores de expresión de CaCas9 incorporar en el genoma de C. albicans han sido desarrollados17. Además, aumentó guía expresión aumenta genoma edición eficacia18. Estos y otros avances tecnologías CRISPR, son cruciales para el desarrollo de pantallas de todo el genoma de C. albicans19,20,21,22.
El genoma de C. albicans es diploide, pero una y alelos B no son siempre idénticos5. Tal heterocigoto ofrece tanto retos como oportunidades. Si uno pretende atacar ambos alelos, deben usarse un sitio de PAM, secuencia guía y plantilla de reparación que actúa en ambas copias del gen. Sin embargo, dependiendo de polimorfismos de nucleótido único presentes en un gen, el sistema CRISPR C.albicans permite a los investigadores a un solo alelo. Tal precisión tiene el potencial para permitir a los investigadores a examinar las diferencias funcionales entre alelos. Dirigidas a alelos específicos deben hacerse con cuidado, como se ha observado la pérdida del heterocigoto (LOH) en un alelo o de un cromosoma entero. Cuando se edita solo C. albicans alelos, uno debe examinar adyacentes secuencias de ADN para determinar si un clon ha mantenido un perfil SNP diploide. Además, efectos off-target están bastante bajos para C. albicans CRISPR y secuenciación del genoma entero puede ser considerado para las cepas claves.
Divulgaciones
Los autores no tienen nada que revelar.
Agradecimientos
Los autores agradecen a Dr. Gennifer Mager para lectura y comentarios sobre el manuscrito. Este trabajo fue apoyado por fondos de inicio de laboratorio de Ball State University y NIH-1R15AI130950-01 a D.A.B.
Materiales
| Name | Company | Catalog Number | Comments |
| Chemicals | |||
| Agar | BD Bacto | 214010 | |
| agarose | amresco | 0710-500G | |
| Ampicillan | Sigma-Aldrich | A9518 | |
| Bacto Peptone | BD Bacto | 211677 | |
| Bsmb1 | New England Biolabs | R0580L | |
| Calf intestinal phosphatase (CIP) | New England Biolabs | M0290L | |
| Cut Smart Buffer | New England Biolabs | B7204S | |
| Dimethyl Sulfoxide (DMSO) | Sigma-Aldrich | D8418 | |
| dNTPs | New England Biolabs | N0447L | |
| Ethylenediaminetetraacetic acid (EDTA) | Sigma-Aldrich | 3609 | |
| Glacial Acetic Acid | Sigma-Aldrich | 2810000ACS | |
| Glucose | BDH VWR analytical | BDH9230 | |
| Glycerol | Sigma-Aldrich | 49767 | |
| Kpn1 | New England Biolabs | R3142L | |
| LB-Medium | MP | 3002-032 | |
| Lithium Acetate | Sigma-Aldrich | 517992 | |
| L-Tryptophan | Sigma-Aldrich | T0254 | |
| Maltose | Sigma-Aldrich | M5885 | |
| Molecular Biology Water | Sigma-Aldrich | W4502 | |
| NEB3.1 Buffer | New England Biolabs | B7203S | |
| Nourseothricin | Werner Bioagents | 74667 | |
| Poly(ethylene glycol) PEG 3350 | Sigma-Aldrich | P4338 | |
| Sac1 | New England Biolabs | R3156L | |
| Salmon Sperm DNA | Invitrogen | AM9680 | |
| T4 Polynucleotide kinase | New England Biolabs | M0201S | |
| T4 DNA ligase | New England Biolabs | M0202L | |
| Taq polymerase | New England Biolabs | M0267X | |
| Tris HCl | Sigma-Aldrich | T3253 | |
| uridine | Sigma-Aldrich | U3750 | |
| Yeast Extract | BD Bacto | 212750 | |
| Equipment | |||
| Electrophoresis Appartus | |||
| Incubator | |||
| Microcentifuge | |||
| PCR machine | |||
| Replica Plating Apparatus | |||
| Rollerdrum or shaker | |||
| Spectrophotometer | |||
| Waterbath |
Referencias
- Pfaller, M. A., Diekema, D. J. Epidemiology of invasive candidiasis: a persistent public health problem. Clinincal Microbiology Review. 20 (1), 133-163 (2007).
- Magill, S. S., et al. Multistate point-prevalence survey of health care-associated infections. New England Journal of Medicine. 370 (13), 1198-1208 (2014).
- Wisplinghoff, H., et al. Nosocomial bloodstream infections in US hospitals: analysis of 24,179 cases from a prospective nationwide surveillance study. Clinical Infectious Disease. 39 (3), 309-317 (2004).
- Jones, T., et al. The diploid genome sequence of Candida albicans. Proceedings of the National Academy of Science USA. 101 (19), 7329-7334 (2004).
- Muzzey, D., Schwartz, K., Weissman, J. S., Sherlock, G. Assembly of a phased diploid Candida albicans genome facilitates allele-specific measurements and provides a simple model for repeat and indel structure. Genome Biology. 14 (9), (2013).
- Morschhauser, J., Michel, S., Staib, P. Sequential gene disruption in Candida albicans by FLP-mediated site-specific recombination. Molecular Microbiololgy. 32 (3), 547-556 (1999).
- Lau, V., Davie, J. R. The discovery and development of the CRISPR system in applications in genome manipulation. Biochemistry and Cell Biology. 95 (2), 203-210 (2017).
- Doudna, J. A., Charpentier, E. Genome editing. The new frontier of genome engineering with CRISPR-Cas9. Science. 346 (6213), 1258096 (2014).
- Anders, C., Niewoehner, O., Duerst, A., Jinek, M. Structural basis of PAM-dependent target DNA recognition by the Cas9 endonuclease. Nature. 513 (7519), 569-573 (2014).
- Reardon, S. Welcome to the CRISPR zoo. Nature. 531 (7593), 160-163 (2016).
- Vyas, V. K., et al. New CRISPR Mutagenesis Strategies Reveal Variation in Repair Mechanisms among Fungi. mSphere. 3 (2), (2018).
- Vyas, V. K., Barrasa, M. I., Fink, G. R. A Candida albicans CRISPR system permits genetic engineering of essential genes and gene families. Science Advances. 1 (3), e1500248 (2015).
- Evans, B. A., et al. Restriction digest screening facilitates efficient detection of site-directed mutations introduced by CRISPR in C. albicans UME6. PeerJ. 6, e4920 (2018).
- Kim, D., et al. Genome-wide analysis reveals specificities of Cpf1 endonucleases in human cells. Nature Biotechnology. 34 (8), 863-868 (2016).
- Tarasava, K., Oh, E. J., Eckert, C. A., Gill, R. T. CRISPR-enabled tools for engineering microbial genomes and phenotypes. Biotechnology Journal. , e1700586 (2018).
- Raschmanova, H., Weninger, A., Glieder, A., Kovar, K., Vogl, T. Implementing CRISPR-Cas technologies in conventional and non-conventional yeasts: Current state and future prospects. Biotechnology Advances. 36 (3), 641-665 (2018).
- Min, K., Ichikawa, Y., Woolford, C. A., Mitchell, A. P. Candida albicans Gene Deletion with a Transient CRISPR-Cas9 System. mSphere. 1 (3), (2016).
- Ng, H., Dean, N. Dramatic Improvement of CRISPR/Cas9 Editing in Candida albicans by Increased Single Guide RNA Expression. mSphere. 2 (2), (2017).
- Huang, M. Y., Woolford, C. A., Mitchell, A. P. Rapid Gene Concatenation for Genetic Rescue of Multigene Mutants in Candida albicans. mSphere. 3 (2), (2018).
- Shapiro, R. S., et al. A CRISPR-Cas9-based gene drive platform for genetic interaction analysis in Candida albicans. Nature Microbiology. 3 (1), 73-82 (2018).
- Grahl, N., Demers, E. G., Crocker, A. W., Hogan, D. A. Use of RNA-Protein Complexes for Genome Editing in Non-albicans Candida Species. mSphere. 2 (3), (2017).
- Nguyen, N., Quail, M. M. F., Hernday, A. D. An Efficient, Rapid, and Recyclable System for CRISPR-Mediated Genome Editing in Candida albicans. mSphere. 2 (2), (2017).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados