Method Article
Candida albicans insan mantar patojen genom düzenleme CRISPR-aracılı
* Bu yazarlar eşit katkıda bulunmuştur
Bu Makalede
Özet
Candida albicans verimli genom mühendislik patogenez ve tedavi gelişimini anlamak için önemlidir. Burada, hızlı ve doğru CRISPR kullanarak C. albicans genom düzenlemek için bir iletişim kuralı açıklanan. Protokol müfettişler çok çeşitli noktasını mutasyonlar, eklemeler ve silmeler dahil olmak üzere genetik değişiklik tanıtmak sağlar.
Özet
Bu yöntem verimli aracılı CRISPR genom diploit insan mantar patojen düzenleme açıklar Candida albicans. CRISPR-aracılı genom C. albicans içinde düzenleme gerektirir Cas9, RNA rehberlik ve onarım şablon. Bir Maya kodon ifade bir plazmid Cas9 optimize (CaCas9) oluşturulur. Kılavuzu dizileri doğrudan ters yönde bir PAM üzerinden site (NGG) Cas9 ifade vektör klonlanmış. Onarım şablon daha sonra astar uzantısı vitrotarafından yapılır. C. albicans içine onarım şablon ve vektör cotransformation genom düzenleme için yol açar. Kullanılan tamir şablonuna bağlı olarak, araştırmacı nükleotit değişiklikleri, eklemeleri veya silmeleri tanıtabilirsiniz. C. albicans bir diploid olduğu için A ve B gen Hedefleme Rehberi ile müdahale veya şablon birleşme onarmak SNPs liman değil koşuluyla mutasyonlar bir genin her iki gen içinde yapılır. Uygun korunmuş dizileri tüm aile üyelerinde varsa multimember gen aileler paralel olarak düzenlenebilir. Tanımlanan C. albicans CRISPR sistemini FRT siteleri tarafından çevrili olduğunu ve flippase kodlar. Flippase indüksiyon antibiyotik marker (CaCas9) ve Kılavuzu RNA genomu kaldırılır. Bu araştırmacı genom sonraki düzenlemeleri gerçekleştirmek izin verir. C. albicans ve CRISPR bir güçlü mantar genetik mühendisliği araçtır küçük değişiklikler açıklanan protokollere C. glabrata, N. castellii ve S. cerevisiaedahil olmak üzere diğer mantar türler değiştirmesine.
Giriş
Candida albicans en yaygın insan mantar patojen1,2,3' tür. C. albicans ve memeli moleküler biyoloji arasındaki farklar anlama antifungal tedavi yeni nesil gelişimi için önemlidir. Bu hızlı ve doğru bir şekilde genetik olarak C. albicansmanipüle edebilmek için Müfettişler gerektirir.
C. albicans genetik manipülasyon tarihsel zorlu olmuştur. C. albicans plazmid sahip değilse, böylece tüm yapıları genom dahil gerekir. Ayrıca, C. albicans diploit; Bu nedenle, her iki kopya değiştirilmiş4bir gen vurma veya bir mutasyon tanıtımı, emin olmak önemli olmuştur. Buna ek olarak, bazı C. albicans loci Heterozigoz, daha da zorlaştıran genetik sorgulama5bulunmaktadır. C. albicansgenetik olarak işlemek için birden fazla mermi Homolog rekombinasyon6gerçekleştirmek için normaldir. Ancak, genom ve zahmetli yapı geliştirme diploit doğası yaptık bu potansiyel olarak sıkıcı bir süreç, özellikle birden çok değişiklik gerekiyorsa. Bu sınırlamalar ve C. albicans tıbbi önemi daha kolay C. albicans genom işlemek müfettişler etkinleştirmek yeni teknolojilerin geliştirilmesini talep ediyorum.
Düzenli olarak interspaced kısa palindromik yineler (CRISPR) kümelenmiş-aracılı genom düzenleme olduğunu araştırmacılar genom sırasını değiştirmek güçlü bir araç. CRISPR üç bileşenleri gerektirir: hedef DNA cleaves 1) Cas9 nükleaz, 2) 20 Cas9 faiz sıra için hedef Kılavuzu RNA ve temel 3) bölünme sitesi onarır ve hedeflenen değişim7, birleştirmek şablon DNA onarım 8. Cas9 istemek protospacer bitişik motifi (PAM) sıra (NGG) bir kez rehber Cas9 hedef genom serisine getiriyor, doğrudan DNA9ayırmak için Kılavuzu sırasının ters yönde. 20 temel Kılavuzu ve PAM dizileri gereksinimini özgüllük hedefleme yüksek derecede sağlar ve hedef kapalı bölünme sınırlar.
CRISPR sistemleri farklı birtakım organizmaların genleri düzenlemek ve çok çeşitli sorunlar10mücadele için tasarlanmıştır. Burada açıklanan ilgi C. albicans gen düzenleme için bir esnek, verimli CRISPR iletişim kuralıdır. Deneme bir kodonu çeviri fesih neden bir gen için tanıtır. Diğer düzenlemeler tanıtıldı onarım şablonuna bağlı olarak yapılabilir. Bir parça nourseothricin (Natr) ile işaretlenmiş Maya içeren kodon-optimize Cas9 (CaCas9) ve bir rehber RNA C. albicans genom tarafsız bir siteye eklenmiştir. Cotransformation istenen mutasyon kodlama onarım şablonuyla Homolog rekombinasyon ve verimli genom düzenleyerek bölünme onarmak için yol açar. Aşağıda açıklanan TPK2ama tüm C. albicans çerçeveler birden çok kez CRISPR tarafından hedeflenebilir okuma açık düzenleme var. CRISPR sistem FRT siteleri tarafından çevrili olduğunu ve C. albicans genom CaCas9 ifade plazmid kodlanmış flippase indüksiyon tarafından kaldırılabilir. C. albicans CRISPR sisteminin hızlı ve doğru C. albicans genom11,12düzenlemek müfettişler sağlar.
Protokol
1. kimlik ve Kılavuzu RNA dizisi klonlama
-
Kılavuzu RNA dizisinin tanımlaması
- Bir 5'-NGG-3 tanımlamak ' PAM sıra yakın nerede kodonu eklenir. (Şekil 1A) Etiketli tüm PAM dizileri ilk 100 baz çiftine TPK2 (Şekil 1A) bulunur.
Not: Her C. albicans açık okuma çerçevesi Hedefleme Rehberi dizileri http://osf.io/ARDTX 11,12, bulunabilir. - 20 olacaktır ileri Kılavuzu Primer_3 sıra tanımlamak üsleri doğrudan akıntıya karşı bir NGG PAM site ve 5'ten fazla içermez Ts üst üste. Sol tıklayın doğrudan üsteki akıntıya karşı NGG ve sürükle imleci 20 üsleri, astar eklemek için astar sekmesinde sol tıklatın.
- İleri Kılavuzu serisi tamamlayacak-ecek var olmak kılavuz Primer_3 ters sıra tanımlayın.
Not: PAM_3 kullanma kılavuzları gösterilmiştir (Şekil 1B). - Astar sağ tıklatıp "astar veri Kopyala". Dizileri bir metin düzenleme programı yapıştırın.
- Bir 5'-NGG-3 tanımlamak ' PAM sıra yakın nerede kodonu eklenir. (Şekil 1A) Etiketli tüm PAM dizileri ilk 100 baz çiftine TPK2 (Şekil 1A) bulunur.
-
Çıkıntı dizileri (Tablo 1, Şekil 1B) klonlama kolaylaştırmak için ileri ve ters Kılavuzu oligos ekleyin.
- Nükleotid dizisi ATTTG 5' İleri Kılavuzu Primer_3 için satın almadan önce ekleyin.
- G 3' İleri Kılavuzu Primer_3 için satın almadan önce ekleyin.
- Nükleotid dizisi AAAAC 5' ters Kılavuzu Primer_3 için satın almadan önce ekleyin.
- C 3' ters Kılavuzu Primer_3 için satın almadan önce ekleyin.
-
Özet CaCas9 ifade vektör pV1524 ile BsmBI.
Not: pV1524 bir ampisilin (Ampr) ve nourseothricin (Natr) işaretleri içerir. Cas9 kodon- C. albicansiçin en iyi duruma getirilmiş oldu.- Plazmid ekleyerek Özet: pv1524, 10 arabellek x 5 μL, BsmBI ve H 1 μL 2 μg2O 1,5 mL tüp içinde 50 μL için. 20 dk. 55 ° C'de kuluçkaya (alternatif olarak, pv1524 ile Esp3I, BsmBI, bir isoschizomer 37 ° C'de 15 dakika sindirmek)
- Oda sıcaklığında (RT) ve 30 tur serin tüp altına yoğunlaşma getirmek için 2,348 x g s. 1.4 adım geçin veya sindirilir plazmid-20 ° C'de depolayın
-
Fosfataz-sindirilir omurga tedavi.
- Buzağı bağırsak fosfataz (CIP) 1 μL sindirim karışıma ekleyin ve 1 h için 37 ° C'de kuluçkaya.
- Piyasada bulunan polimeraz zincir reaksiyonu (PCR) arıtma kiti (kit ile sağlanan yönergeleri) kullanarak sindirilir plazmid arındırmak ve elüsyon arabellek (EB) 30 μL içinde elute.
-
Fazdan ve İleri Kılavuzu Primer_3 ve ters Kılavuzu Primer_3 tavlama.
- 100 μM ileri 0.5 μL Kılavuzu Primer_3, 100 mikron 0.5 μL ekleyin ters Kılavuzu Primer_3, 10 x T4 ligaz arabelleği 5 μL, T4 polinükleotit kinaz 1 μL ve H2O PCR Tube 43 μL.
- 10 x T4 ligaz arabelleği 5 μL, T4 polinükleotit kinaz 1 μL ve moleküler biyoloji-grade H2O 44 μL ikinci PCR tüpte ekleyin.
Not: Bu negatif kontrol görev yapacak. - Bir thermocycler sonra 95 ° C'de 5 min için 30 dk 37 ° C'de reaksiyon karışımları kuluçkaya.
- En yavaş rampa oranı 16 ° C tavlamak için karisimin serin, oligos. 4 ° C'de tavlanmış oligo karışımı yerleştirin
-
Tavlanmış oligos adım 1.4.3 sindirilmiş pv1524 içine ligate.
- PCR tüp aşağıdakileri ekleyin: 10 x T4 ligaz arabellek, T4 DNA ligaz 0.5 μL, tavlanmış oligo Mix, 0.5 μL 1 μL sindirilmiş CIP tedavi saf plazmid (20-40 ng) ve H2O 10 μL toplam hacim için.
- PCR tüp aşağıdakileri ekleyin: 10 x T4 ligaz arabelleği, T4 DNA ligaz, 0.5 μL 1 μL arıtılmış vektör CIP tedavi (20-40 ng), negatif kontrol karışımı ve H2O kadar 10 μL toplam hacim 1 μL sindirilir.
- Bir thermocycler için 30 dk 16 ° C'de, sonra 10 dk 65 ° C'de hem tüplerde kuluçkaya, daha sonra 25 ° C'ye serin
- Tüp ligasyonu karışımları 5 μL standart Isı şok dönüştürme protokolü kullanılarak kimyasal olarak yetkili Escherichia coli DH5α dönüşümü. LB Amp/Nat medyada (200 μg/mL ampul, 50 μg/mL Nat) seçin.
Not: PV1524 ve türevleri Çift Kişilik seçim medya seçmek için başarısızlık FLP/FRT eksizyon bakterilerde tarafından Nat/CaCas9/rehber modülü kaybolmasına neden olur. - Plazmid dört transformants üzerinden miniprep tarafından arındırmak ve sıralama astar (Tablo 1) ile ekleme sıra sıra.
Not: Çoğu zaman 4 transformants sıralama, en az 1 doğru klon tanımlamak için yeterli değil. - BsmBI sitesi-20 ° C'de kesmek içine bir kez klonlanmış Kılavuzu RNA dizisi var plazmid kaydetmek
2. tasarımı ve onarım şablon nesil
- Bir kodonu gen sırayla seçeneğini ve bir stop kodonu ve kısıtlama sindirim sitesi (Şekil 1 cTablo 1) kodlamak nükleotit ekleyerek yerleştirin.
Not: Ekleme PAM sıra bozabilir olacaktır.
Not: Bir kısıtlama sindirim site klonların (Şekil 1 c) verimli tarama kolaylaştırmak için onarım şablon sırada yer alır. - 10 mutasyon nerede yapılacak, aşağı üsleri ve imleci 60 akıntıya karşı üsleri sürükle sol tıklatın. Sol astar eklemek için astar sekmesini tıklatın. Bu onarım şablon Forward_3 ekler. 10 mutasyon nerede yapılacak, akıntıya karşı üsleri ve imleci 60 aşağı üsleri sürükle sol tıklatın. Sol astar eklemek için astar sekmesini tıklatın. Bu onarım şablon ters 3 eklemek olacaktır. (Şekil 1 c)
-
Onarım şablon oluşturmak için astar uzantısı gerçekleştirin.
- 100 μm kalınlığında onarım şablon ileri astar, 1.2 μL 100 mikron onarım şablon ters astar, deoxynucleotide trifosfatlardan (dNTPs) 6 μL 1.2 μL ekleyin (Toplam konsantrasyonu 40 mM), 6 μL arabellek, 0.6 μL Taq polimeraz (3 adet) ve H2O her 4 PCR 45 μL tüpler.
- Astar uzantısı PCR 20 ve 30 mermi arasında çalışan yerine getirilir. Örnek uzantısı koşulları: 95 ° C'de 2 dk (30 s 95 ° c, 50 ° c, 68 ° C'de 1 dk 1 dk) x 34, 68 ° C'de 10 dakika
- Tüm 4 PCR tüpleri bir 1,5 mL tüp içeriğini birleştirmek ve H2o 50 μL ürünlerinde arındırmak için PCR arıtma setini kullanın
- 260 absorbans belirlenerek yeterli DNA sağlamak için astar uzantılı ürünler quantitate nm.
Not: Astar uzantısı ürün tipik son konsantrasyonu olduğunu ~ 200 – 300 ng/µL.
3. C. albicans onarım şablon ve plazmid ile dönüştürme
-
TE/Lityum asetat olun.
- Mix 10 mM Tris-Cl, 1 mM EDTA, 100 mM Lityum asetat ve H2O (tüm hisse senedi çözüm pH 7.5) 50 mL toplam hacmi elde etmek için.
-
TABAK hazırla
- %40 3350 PEG, 100 mM Lityum asetat, 10 mM Tris-Cl, 1 mM EDTA ve H2O (tüm hisse senedi çözüm pH 7.5) 50 mL toplam hacmi elde etmek için karıştırın.
-
Doğru klonlanmış plazmid adım 1.9 sindirmek.
- 0.5 µL KpnI, sacı 0.5 µL ve H20-40 µL hacmi 1.5 mL tüp içinde toplam, plazmid, 10 x arabelleği 4 µL, 10 mg/mL Sığır serum albumin (BSA) 0.4 µL 10 μg ekleyin. 37 ° C gece (Şekil 1 d) kuluçkaya.
-
C. albicans SC5314, 25 ° c 0,27 mM Uridine (YPD + URI), OD600 6'dan daha az ideal desteklenmiş Maya pepton dekstroz içinde vahşi-türü prototroph bir gecede kültür büyümek.
- 2,348 x g de 5 dk iplik tarafından 5 OD600 adet hücre dönüşüm başına cips ve 100 µL TE/Lityum asetat pelleted hücrelerinin 5 OD600 askıya alma.
- 1.5 mL tüp listelendikleri sırada aşağıdaki eklemek: 1) 100 µL hücre adım 3.4.1, haşlanmış ve hızlı soğutmalı somon sperm DNA (10 mg/mL), plazmid sindirim adım 3.3.1, saflaştırılmış onarım şablonunun 4) 6 µg 3) 10 µg 2) 40 µL ve 5) plaka 1 mL.
- 1.5 mL tüp listelendikleri sırada aşağıdaki eklemek: 1) 100 µL hücre, haşlanmış ve hızlı soğutmalı somon sperm DNA (10 mg/mL), 3) H2O birim DNA adım 3.5 ve plaka 4) 1 mL dönüştürme eşit 2) 40 µL.
Not: Bu bir negatif kontrol görev yapacak. - Dönüştürmeleri karışımı yavaşça pipetting tarafından ve 25 ° C de gecede kuluçkaya izin.
- Isı şok hücreleri için 25 dk 44 ° C su banyosu yerleştirerek.
- 2,348 x g benchtop santrifüj, 5 min için spin ve süpernatant plaka karışımı çıkarın. Bir kez de 2,348 x g1 mL YPD + URI ve 5 min için tekrar santrifüj ekleyerek yıkayın.
- YPD + URI 0.1 mL hücrelerde askıya alma ve bir rulo davul veya shaker 25 ° C'de gecede kuluçkaya.
- YPD + URI plaka ile 200 μg/mL nourseothricin. Koloniler 2-5 gün içinde görünür.
4. tek kolonileri için çizgiler
-
Bir 100 mm x 15 mm Petri kabına dört eşit parçaya bölün ve her çeyrekte etiket.
- Bir dönüşüm plaka bir steril kürdan veya aplikatör ve çizgi ile kolonilerin çeyreği yan en uzun dokunun.
- Aseptik bir teknikle tek kolonileri için çizgi ve 30 ° C de 2 gün boyunca büyümeye kolonileri sağlar.
5. koloni PCR
-
Tasarım ileriye doğru onay astar (FCP) ~ 200 baz çifti akıntıya karşı kısıtlama sitesinin kullanılmaya başlandı ve ters onay astar (RCP) ~ 300 baz çifti aşağı.
- FCP, RCP, 0.3 μL opsonins polimeraz (ExTaq 1,5 adet), dNTPs 3 μL 0.3 μL 0.3 μL ekleyin (Toplam konsantrasyonu 40 mM), 3 ExTaq arabellek ve H2O 1,5 mL tüp için 23 μL μL.
Not: 0.5 μL/tepki dimetil sülfoksit (DMSO) eklenmesi PCR verimliliği artırabilirsiniz. - Bir tek Maya koloninin 0,25 μL 4.1.2 adımından adım P10 pipet ucu kullanarak ve agar bozmamaya dikkat çekici 5.1.1 karışıma ekleyin.
- DNA PCR tarafından yükseltmek ve 5 μL PCR güçlendirme tüp altındaki hücre Pelet bozmamaya dikkat çekici başarılı olduğundan emin olmak için bir jel çalıştırın.
- FCP, RCP, 0.3 μL opsonins polimeraz (ExTaq 1,5 adet), dNTPs 3 μL 0.3 μL 0.3 μL ekleyin (Toplam konsantrasyonu 40 mM), 3 ExTaq arabellek ve H2O 1,5 mL tüp için 23 μL μL.
6. kısıtlama sindirim koloninin PCR
- PCR (hücre Pelet tüpün dibinde rahatsız değil için Bakımı) ürün, 3 μL arabelleği, Restriksiyon enzimi 1 μL ve H2O, 16 μL 10 μL ekleyin sonra üreticinin talimatlarına göre kuluçkaya ve corre tanımlamak için bir özel jel gidermek CT genom düzenlemeleri.
Not: Burada kullanılan Restriksiyon enzimi TPK2 belirli onarım şablonunda kodlanmış sitedir.
7. suşları tasarruf
- Bir gecede kültür YPD + URI kısıtlaması sindirim 30 ° C'de tarafından teyit edilmiştir kolonilerden büyümek
- Bir tüp-80 ° C'de muhafazası için uygun 1 mL kültür ve 1 mL % 50 gliserol (gliserol son konsantrasyonu % 25'e getirerek) eklemek
- -80 ° C'de doğru klonlar depolamak
Not: Doğru suşları-80 ° C'de uzun yıllar saklanabilir.
8. Natr Marker kaldırılması
- Maya pepton maltoz (YPMaltose) üzerine çizgi doğru transformant (% 2 maltoz).
- Bir koloni çizgili plaka almak ve sıvı YPMaltose 20 g/L maltoz içinde 30 ° C'de 48 h için Maya kültür.
- YPMaltose 20 g/L maltoz hücrelerdeyse 200-400 plaka ve 24 saat için 30 ° C'de kuluçkaya.
- YPMaltose ve YPMaltose 200 μg/mL Nat. tabağa çoğaltmak
- 24 h 30 ° C'de kuluçkaya.
Not: Artık YPMaltose 200 μg/mL nourseothricin üzerinde büyümek ama YPMaltose üzerinde büyümek kolonileri Natr işaretleyici (CaCAS9) kaybetmiş ve RNA Kılavuzu. - 7,1-7,3 adımdan gibi Natr işaretleyici (CaCAS9) ve Kılavuzu RNA kaybetmiş suşları kaydedin.
Not: Benzer bir plazmid, pV1393, SAP2 MAL2 promotor karşı kullanır. PV1393 gen düzenleme için kullanılırsa büyümesini YCB-BSA üzerinde flippase ve Natr kaldırılması teşvik.
Sonuçlar
Dizileri RNA'ların ve C. albicans TPK2, hedef onarım şablonları bir c-AMP kinaz katalitik alt birim Kılavuzu, yukarıda önerilen yönergelere göre tasarlanmıştır. Dizileri (Tablo 1, Şekil 1') gösterilir. Kılavuzu RNA'ların CaCas9 ifade vektörel çizimler klonlanmış ve vahşi-türü C. albicansşablonunda onarım ile cotransformed. Bir EkoRI kısıtlama sindirim sitesi ve stop kodon onarım şablonundaki PAM sitesi bozabilir ve tarama doğru mutantlar (Şekil 1) için kolaylaştırmak. Transformants için tek kolonileri çizgili ve koloni PCR ve kısıtlama sindirim onarım şablon (Şekil 2)birleşme için tarafından tarandı. Kısıtlama sindirim hızlı bir şekilde mutasyona uğramış dizileri vahşi tipi ayırt eder.
| Oligonükleotid adı | Oligonükleotid sıra |
| İleri Kılavuzu Primer_3 | ATTTGgggtgaactatttgttcgccG |
| Geriye doğru rehber Primer_3 | AAAACggcgaacaaatagttcacccC |
| Onarım şablon Forward_3 | tcagcaatatcagcaacaatttcaacaaccgcagcaacaactttatTAAGAATTCggcga |
| Onarım şablon Reverse_3 | attttgtccagtttgggctgcagcagggtgaactatttgttcgccGAATTCTTAataaag |
| Ön onay astar | ttaaagaaacttcacatcaccaag |
| Geriye doğru onay astar | actttgatagcataatatctaccat |
| Sıralama astar | ggcatagctgaaacttcggccc |
Tablo 1: Bu çalışma için kullanılan oligo nükleotit listesi. Amaçlı klonlama için eklenen siteler büyük harfle ve kalın Kılavuzu astar olarak serileri. Genomik DNA'sı mutasyona onarım şablon serilerinde büyük harfle yazılmış ve kalın.
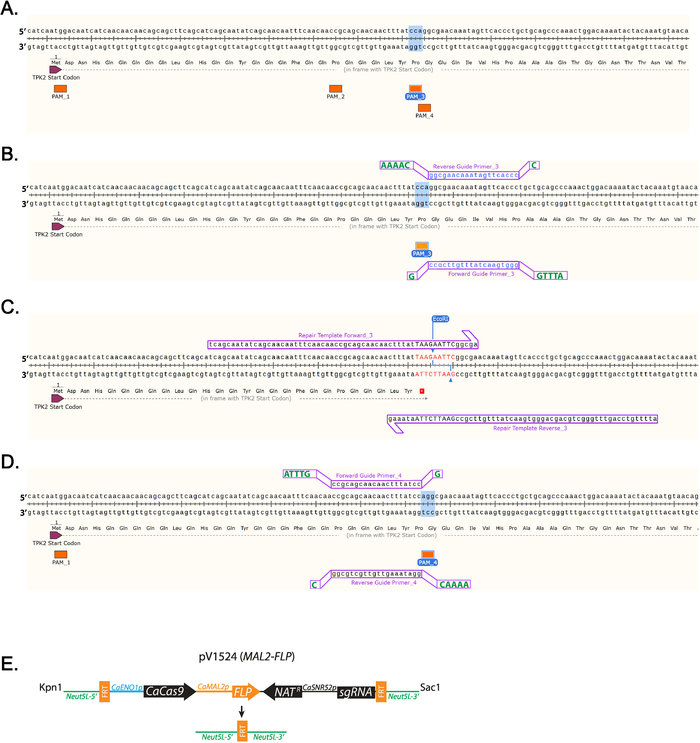
Resim 1 : Diyagramı RNA ve onarım şablon tasarım kılavuzunun. TPK2ilk 100 nükleotit tüm PAM serilerinde(a)etiketleme. Bu çalışmada kullanılan sıraya olduğu gibi PAM sıra 3 (PAM_3) vurgulanır. (B) Kılavuzu RNA tasarım PAM_3 kullanarak. SnapGene kullanılarak tasarlanmış 20 temel astar hem küçük hem mavi. İlave üsler klonlama için gerekli mahsup büyük ve yeşil gösterilmiştir. (C) onarım bir tad kodonu ve EkoRI site eklemek şablon astar çerçeve okuma TPK2 eklenir. Kırmızı hem de büyük vahşi tipi serisinden farklı DNA'sı. (D) örnek nasıl bir rehber PAM_4 kullanarak DNA pozitif iplikçiğe tasarlanmıştır. (E) pV1524 Kılavuzu RNA ve sindirim KpnI ve sacı klonlama sonra Şematik diyagramı. Neut5L-5' ve Neut5L-3' taki C. albicans genom Neut5L bölgesine vektör hedef. CaENO1p Maya için optimize edilmiş CaCas9ifade sürücüler promotor var. NATr nourseothricin direnç kaset var. CaSNR52p Kılavuzu RNA ifade (sgRNA) sürüş promotor var. FRT siteleri i ciddi ve flippase ifade üzerine CRISPR kaset kaldırma flippase (FLP) tarafından recombined. (E) benzer bir şematik Vyas ve arktarafından yayımlandı. 11. Bu rakam daha büyük bir versiyonunu görüntülemek için buraya tıklayınız.
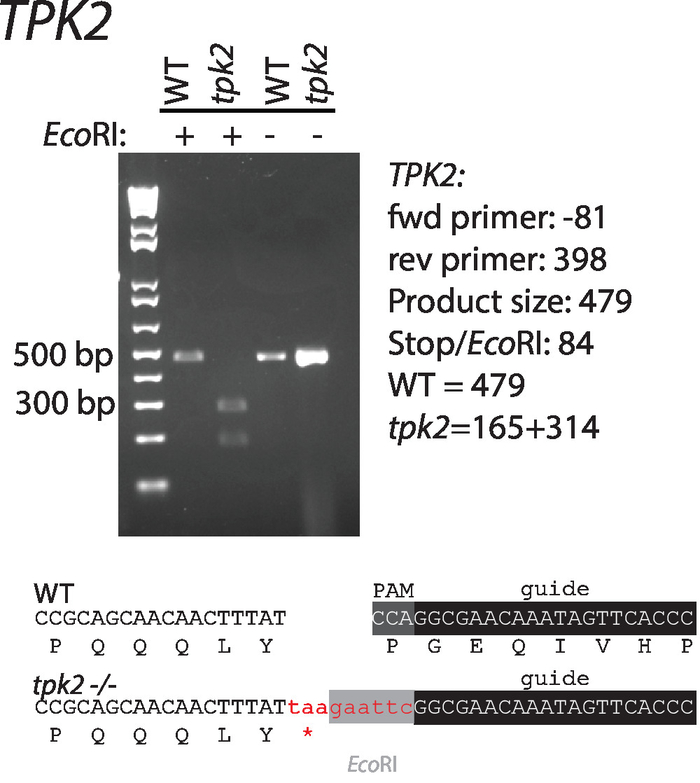
Resim 2 : Giriş ve kodonu ve EkoRI kısıtlama siteye TPK2. Amplifikasyon için kullanılan astar Tablo 1' de listelenmiştir. Vahşi-türü ve mutant diziler jel gösterilmiştir. Bu rakam Vyas vd.11' den değiştirildi. Bu rakam daha büyük bir versiyonunu görüntülemek için buraya tıklayınız.
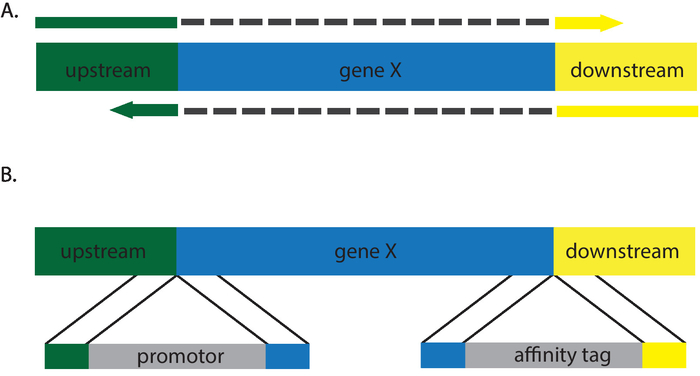
Şekil 3 : Çizgi film (A) silmelere ve eklemelere (B) üretecektir onarım şablonları açıklaması. Gri tire (a) tasvir aradan geçen diziler onarım şablon astar içinde mevcut değil. Bu rakam daha büyük bir versiyonunu görüntülemek için buraya tıklayınız.
Tartışmalar
C. albicans CRISPR C. albicans genom verimli bir şekilde düzenler. pV1524 kodlar bir Maya kodon-optimize Cas9 ve müfettişler-ebilmek kolayca clone Kılavuzu RNA dizisi aşağı CaSNR52 organizatörü (Şekil 1)11öyle ki tasarlanmıştır. Ek kopyalarını genom düzenleme engel olarak Kılavuzu dizi yalnızca tek bir kopyasını CaCas9 ifade vektörel çizimler sıralama tarafından kopyalamış ki sağlanması gerekir. Çok bol-in belgili tanımlık kılavuz sürekli olarak tanıttı, bir tüp ligasyonu içinde kullanılan tavlanmış Kılavuzu konsantrasyonu düşük olmalıdır. Vektör ve açıklanan protokol bırakmak herhangi bir C. albicans gen hedef. C. albicans diploit olmasına rağmen sadece tek bir dönüşüm bir genin her iki gen hedeflemek için gereklidir. Ayrıca, CRISPR-CaCas9 genom düzenleme ilerleyicidir doğası araştırmacılar birden çok gen ailelerinin üyeleri hedeflemesini sağlar. Salgılanan aspartyl proteaz (SAP) ve agglutinin gibi sıra proteinler (ALS) gibi pek çok gen aile C. albicans virülans için önemlidir. CRISPR genom düzenleme bu gen ailelerin incelenmesi kolaylaştıracaktır.
Yukarıda açıklanan protokoller fenotipik eşdeğer bir null (Şekil 2) kaynaklanan bir açık okuma çerçevesi bir stop kodon tanıtın. Çok çeşitli genetik değişiklikler onarım şablonu değiştirerek yapılabilir. Saçma, missense ve Sessiz mutasyonlar rekombinasyon uygun onarım şablonuyla yoluyla eklenebilir. O olmadan12,13sıralama tarafından taranması gerekir gibi bir kısıtlama site birleşme transformant tarama, akıcılık. Buna ek olarak, C. albicans CRISPR araştırmacılar eklemeler ve silmeler, oluşturmak için yapım o benzeşme etiketleri eklemek, promotor takas yapmak ve altını gizleme (Şekil 3) oluşturmak için ideal bir sistem sağlar. Onarım şablonları doğru birleşme onaylamak için düzenlemeler sıralamak gerekli olduğu gibi Bu mutasyonlar için doğru transformants için tarama daha zahmetli. Ayrıca, Southern blot başka bir gen kopyalarını, genom ek yerlerdeki eklenmiş değil emin olmak gerekli olabilir. NGG PAM sitesi şartı hedeflenebilir genom bölgelerinde hafif sınırlamalar yerleştirir. Cpf1 veya Cas9 sistemi çeşitleri var / gibi kullanım alternatif enzimler birçok bu sınırlamalar14hafifletmek alternatif CRISPR sistemlerinin geliştirilmesi. Müfettişler kadarıyla şu anda, bu sistemler henüz C. albicansiçin uygulanmadı.
Öyle ki bu tür Saccharomyces cerevisiae, Naumouozyma castelliive insan patojen Candida glabrata11 dahil zengin çeşitte uygulanabilir Yukarıdaki iletişim kuralında tanımlanan CRISPR sistemini geliştirdi . Dönüşüm ve verimli bu Maya düzenleme açıklanan protokolünde hafif değişiklikler gerektiriyor, ancak bu alternatif genleri düzenleme çerçevesinde son derece C. albicans12için açıklanan benzer. Ayrıca, Maya yordamlar düzenleme genom geliştirmek için mükemmel bir mekanizma sağlar. Ne zaman ADE2 mutasyona uğramış, Maya, adenin biyosentezi yolu habercisi, hücreleri kırmızıya döndüğü birikir. Bu kolayca gözlemlenebilir fenotip düzenlenen hücrelerini tanımlamak ve hızlı bir şekilde iletişim kuralları düzenleme genom gidermek müfettişler sağlar. Mantarlar için kullanılabilir geniş moleküler biyoloji araç kutusu ile birlikte, çok sayıda Maya türü düzenleme için protokol Gelişmiş15,16olmuştur. Mantarlar genom düzenleme teknolojisi geniş bir uygulama çeşitli bilimsel disiplinlerin önemli ölçüde etkisi potansiyeline sahiptir.
CRISPR C. albicansDairesi'nde genom verimliliğini büyük ölçüde iyileşmiştir, ama CRISPR bugüne kadar Genom geniş ekranlar C. albicansiçinde gerçekleştirmek için kullanılmamış. Verimsiz12yolu C. albicans içinde katılma nonhomologous sonu olduğu gibi geçerli iletişim kuralları mutasyonlar, tanıtmak için bir onarım şablon gerektirir. Her gen için onarım şablon oligos nesil Genom geniş ekranlar yürütülmesi için önemli bir engeldir. CRISPR teknolojik gelişmeler ve DNA sentezi azalmıştır maliyeti izdiham silme kütüphaneleri geliştirilmesi daha uygun hale getirecek. Örneğin, ifade CaCas9 vektör onarım şablonundan her gen11hedef sürdürülebilir plazmid kütüphaneleri geliştirilmesi için yol açıyor. Ayrıca, CaCas9 ifade vektör C. albicans genom dahil gerekli olmayan geçici Candida CRISPR iletişim kuralları Gelişmiş17olmuştur. Buna ek olarak, verimliliği18düzenleme kılavuzu ifade artar genom arttı. Bunlar ve diğer gelişmeler CRISPR teknolojileri, Genom geniş ekranlar C. albicans19,20,21,22gelişimi için çok önemli.
C. albicans genom diploid ama A ve B gen değil her zaman aynı5. Böyle heterozygosity zorluklar ve fırsatlar sağlar. Bir iki gen hedef hedefliyor verirseniz bir PAM sitesi, kılavuz dizisi ve gen her iki kopyası üzerinde hareket edecek onarım şablon kullanılmalıdır. Ancak, tek nükleotid polimorfizmleri bir gen mevcut bağlı olarak, tek bir gen hedeflemek müfettişler C.albicans CRISPR sistemi sağlar. Böyle hassas müfettişler alelleri arasında işlevsel farklılıklar incelemek izin potansiyeline sahiptir. Kaybı heterozygosity (LOH) adlı bir gen ya da tüm bir kromozom gözlemlediği gibi belirli allelleri hedefleme dikkatli bir şekilde yapılmalıdır. Tek C. albicans düzenlerken gen, bir klon diploit SNP profil ettirmiştir belirlemek için bitişik DNA dizileri incelemek gerekir. Buna ek olarak, hedef kapalı etkileri C. albicans CRISPR için oldukça düşüktür, ancak tüm genom sıralama anahtar suşları için kabul edilebilir.
Açıklamalar
Yazarlar ifşa gerek yok.
Teşekkürler
Yazarlar Dr Gennifer Mager okuma için teşekkür ederiz ve el yazması üzerinde yararlı yorumlar. Bu eser Ball State University laboratuvar başlangıç fonlar ve NIH-1R15AI130950-01 D.A.B. tarafından desteklenmiştir
Malzemeler
| Name | Company | Catalog Number | Comments |
| Chemicals | |||
| Agar | BD Bacto | 214010 | |
| agarose | amresco | 0710-500G | |
| Ampicillan | Sigma-Aldrich | A9518 | |
| Bacto Peptone | BD Bacto | 211677 | |
| Bsmb1 | New England Biolabs | R0580L | |
| Calf intestinal phosphatase (CIP) | New England Biolabs | M0290L | |
| Cut Smart Buffer | New England Biolabs | B7204S | |
| Dimethyl Sulfoxide (DMSO) | Sigma-Aldrich | D8418 | |
| dNTPs | New England Biolabs | N0447L | |
| Ethylenediaminetetraacetic acid (EDTA) | Sigma-Aldrich | 3609 | |
| Glacial Acetic Acid | Sigma-Aldrich | 2810000ACS | |
| Glucose | BDH VWR analytical | BDH9230 | |
| Glycerol | Sigma-Aldrich | 49767 | |
| Kpn1 | New England Biolabs | R3142L | |
| LB-Medium | MP | 3002-032 | |
| Lithium Acetate | Sigma-Aldrich | 517992 | |
| L-Tryptophan | Sigma-Aldrich | T0254 | |
| Maltose | Sigma-Aldrich | M5885 | |
| Molecular Biology Water | Sigma-Aldrich | W4502 | |
| NEB3.1 Buffer | New England Biolabs | B7203S | |
| Nourseothricin | Werner Bioagents | 74667 | |
| Poly(ethylene glycol) PEG 3350 | Sigma-Aldrich | P4338 | |
| Sac1 | New England Biolabs | R3156L | |
| Salmon Sperm DNA | Invitrogen | AM9680 | |
| T4 Polynucleotide kinase | New England Biolabs | M0201S | |
| T4 DNA ligase | New England Biolabs | M0202L | |
| Taq polymerase | New England Biolabs | M0267X | |
| Tris HCl | Sigma-Aldrich | T3253 | |
| uridine | Sigma-Aldrich | U3750 | |
| Yeast Extract | BD Bacto | 212750 | |
| Equipment | |||
| Electrophoresis Appartus | |||
| Incubator | |||
| Microcentifuge | |||
| PCR machine | |||
| Replica Plating Apparatus | |||
| Rollerdrum or shaker | |||
| Spectrophotometer | |||
| Waterbath |
Referanslar
- Pfaller, M. A., Diekema, D. J. Epidemiology of invasive candidiasis: a persistent public health problem. Clinincal Microbiology Review. 20 (1), 133-163 (2007).
- Magill, S. S., et al. Multistate point-prevalence survey of health care-associated infections. New England Journal of Medicine. 370 (13), 1198-1208 (2014).
- Wisplinghoff, H., et al. Nosocomial bloodstream infections in US hospitals: analysis of 24,179 cases from a prospective nationwide surveillance study. Clinical Infectious Disease. 39 (3), 309-317 (2004).
- Jones, T., et al. The diploid genome sequence of Candida albicans. Proceedings of the National Academy of Science USA. 101 (19), 7329-7334 (2004).
- Muzzey, D., Schwartz, K., Weissman, J. S., Sherlock, G. Assembly of a phased diploid Candida albicans genome facilitates allele-specific measurements and provides a simple model for repeat and indel structure. Genome Biology. 14 (9), (2013).
- Morschhauser, J., Michel, S., Staib, P. Sequential gene disruption in Candida albicans by FLP-mediated site-specific recombination. Molecular Microbiololgy. 32 (3), 547-556 (1999).
- Lau, V., Davie, J. R. The discovery and development of the CRISPR system in applications in genome manipulation. Biochemistry and Cell Biology. 95 (2), 203-210 (2017).
- Doudna, J. A., Charpentier, E. Genome editing. The new frontier of genome engineering with CRISPR-Cas9. Science. 346 (6213), 1258096 (2014).
- Anders, C., Niewoehner, O., Duerst, A., Jinek, M. Structural basis of PAM-dependent target DNA recognition by the Cas9 endonuclease. Nature. 513 (7519), 569-573 (2014).
- Reardon, S. Welcome to the CRISPR zoo. Nature. 531 (7593), 160-163 (2016).
- Vyas, V. K., et al. New CRISPR Mutagenesis Strategies Reveal Variation in Repair Mechanisms among Fungi. mSphere. 3 (2), (2018).
- Vyas, V. K., Barrasa, M. I., Fink, G. R. A Candida albicans CRISPR system permits genetic engineering of essential genes and gene families. Science Advances. 1 (3), e1500248 (2015).
- Evans, B. A., et al. Restriction digest screening facilitates efficient detection of site-directed mutations introduced by CRISPR in C. albicans UME6. PeerJ. 6, e4920 (2018).
- Kim, D., et al. Genome-wide analysis reveals specificities of Cpf1 endonucleases in human cells. Nature Biotechnology. 34 (8), 863-868 (2016).
- Tarasava, K., Oh, E. J., Eckert, C. A., Gill, R. T. CRISPR-enabled tools for engineering microbial genomes and phenotypes. Biotechnology Journal. , e1700586 (2018).
- Raschmanova, H., Weninger, A., Glieder, A., Kovar, K., Vogl, T. Implementing CRISPR-Cas technologies in conventional and non-conventional yeasts: Current state and future prospects. Biotechnology Advances. 36 (3), 641-665 (2018).
- Min, K., Ichikawa, Y., Woolford, C. A., Mitchell, A. P. Candida albicans Gene Deletion with a Transient CRISPR-Cas9 System. mSphere. 1 (3), (2016).
- Ng, H., Dean, N. Dramatic Improvement of CRISPR/Cas9 Editing in Candida albicans by Increased Single Guide RNA Expression. mSphere. 2 (2), (2017).
- Huang, M. Y., Woolford, C. A., Mitchell, A. P. Rapid Gene Concatenation for Genetic Rescue of Multigene Mutants in Candida albicans. mSphere. 3 (2), (2018).
- Shapiro, R. S., et al. A CRISPR-Cas9-based gene drive platform for genetic interaction analysis in Candida albicans. Nature Microbiology. 3 (1), 73-82 (2018).
- Grahl, N., Demers, E. G., Crocker, A. W., Hogan, D. A. Use of RNA-Protein Complexes for Genome Editing in Non-albicans Candida Species. mSphere. 2 (3), (2017).
- Nguyen, N., Quail, M. M. F., Hernday, A. D. An Efficient, Rapid, and Recyclable System for CRISPR-Mediated Genome Editing in Candida albicans. mSphere. 2 (2), (2017).
Yeniden Basımlar ve İzinler
Bu JoVE makalesinin metnini veya resimlerini yeniden kullanma izni talebi
Izin talebiDaha Fazla Makale Keşfet
This article has been published
Video Coming Soon
JoVE Hakkında
Telif Hakkı © 2020 MyJove Corporation. Tüm hakları saklıdır