Method Article
CRISPR-vermittelten Genom-Bearbeitung von menschlichen pilzliche Erreger Candida albicans
* Diese Autoren haben gleichermaßen beigetragen
In diesem Artikel
Zusammenfassung
Effiziente Genom Engineering von Candida Albicans ist entscheidend für das Verständnis der Pathogenese und die Entwicklung von Therapeutika. Hier haben wir ein Protokoll, um schnell und präzise bearbeiten das C. Albicans -Genom mit CRISPR beschrieben. Das Protokoll ermöglicht Ermittler, eine Vielzahl von genetischen Veränderungen einschließlich Punktmutationen, Einfügungen und Löschungen einzuführen.
Zusammenfassung
Diese Methode beschreibt die effiziente CRISPR vermittelte Genom-Bearbeitung der diploiden menschlichen pilzliche Erreger Candida Albicans. CRISPR-vermittelten Genom-Bearbeitung in C. Albicans erfordert Cas9, RNA zu führen, und Vorlage zu reparieren. Ein Plasmid mit dem Ausdruck ein Hefe-Codon optimiert Cas9 (CaCas9) generiert wurde. Führen Sie Sequenzen direkt stromaufwärts von einem PAM Website (NGG) sind in der Cas9 Ausdruck Vektor kloniert. Eine Reparatur Vorlage erfolgt dann durch Primer Extension in Vitro. Cotransformation der Reparatur Vorlage und Vektor in C. Albicans führt zu Genom-Bearbeitung. Abhängig von der Reparatur Vorlage verwendet kann der Prüfer Nukleotid Änderungen, Einfügungen oder Löschungen einzuführen. Als C. Albicans einen diploiden ist, sind Mutationen in beide Allele eines Gens, erfolgen, sofern die Allele A und B nicht SNPs Hafen zu tun, die mit Guide targeting stören oder Vorlage Einarbeitung zu reparieren. Bestehend Genfamilien können parallel bearbeitet werden, wenn geeignete konservierte Sequenzen in alle Familienmitglieder vorhanden sind. Das beschriebene C. Albicans CRISPR-System ist flankiert von FRT Websites und Flippase kodiert. Bei der Induktion von Flippase werden die Antibiotika Marker (CaCas9) und Guide RNA aus dem Genom entfernt. Dadurch kann den Prüfer nachträgliche Bearbeitungen, das Genom durchführen. C. Albicans CRISPR ist ein leistungsfähiges Pilz Gentechnik-Werkzeug und geringfügige Änderungen zu den beschriebenen Protokollen erlauben die Änderung der anderen Pilzarten, darunter C. Glabrata, N. Castellii, und S. Cerevisiae.
Einleitung
Candida Albicans ist die am weitesten verbreitete menschliche pilzliche Erreger1,2,3. Verständnis für Unterschiede zwischen C. Albicans und Säugetieren Molekularbiologie ist entscheidend für die Entwicklung der nächsten Generation der antimykotischen Therapie. Dies erfordert Ermittler, schnell und präzise genetisch C. Albicansmanipulieren zu können.
Genmanipulation von C. Albicans seit jeher schwierig. C. Albicans Plasmide nicht beibehalten, so müssen alle Konstrukte in das Genom integriert werden. Darüber hinaus ist C. Albicans diploid; deshalb beim ausschlagen eines Gens oder eine Mutation einzuführen, es wichtig ist, sicherzustellen, dass haben beide Kopien veränderte4gegeben. Darüber hinaus sind einige C. Albicans Loci heterozygot, genetische Verhör5weiter zu erschweren. C. Albicansgenetisch zu manipulieren, ist es üblich, mehrere Runden der homologen Rekombination6durchführen. Jedoch die diploide Natur des Genoms und aufwändige Konstruktion Entwicklung habe diese möglicherweise langwierigen Prozess, vor allem, wenn mehrere Änderungen erforderlich sind. Diese Einschränkungen und die medizinische Bedeutung von C. Albicans fordern die Entwicklung neuer Technologien, die Ermittler, leichter zu manipulieren, das Genom von C. Albicans ermöglichen.
Gruppierten regelmäßig dazwischen kurze palindromische Wiederholungen (CRISPR)-vermittelten Genom-Bearbeitung ist ein leistungsfähiges Werkzeug, mit dem Forscher die Sequenz eines Genoms ändern kann. CRISPR erfordert drei Komponenten: (1) die Cas9-Nuklease das Ziel DNA spaltet, (2) eine 20 base Guide RNA, die Cas9 richtet sich an die Reihenfolge des Interesses, und (3) reparieren Schablone DNA, die Reparaturen der Spaltstelle und verfügt über die beabsichtigte Änderung7, 8. Sobald der Guide die Zielsequenz Genom Cas9 bringt, Cas9 erfordert eine Protospacer angrenzenden Motiv (PAM) Sequenz (NGG) unmittelbar vor der Führer-Sequenz, die die DNA9Spalten. Die Anforderung für die 20 base Guide und PAM-Sequenzen bietet ein hohes Maß an targeting Spezifität und Ziel Spaltung begrenzt.
CRISPR-Systeme wurden so bearbeiten Sie das Erbgut von einer vielfältigen Reihe von Organismen und zur Bekämpfung einer Vielzahl von Problemen10. Hier beschrieben ist eine flexible, effiziente CRISPR-Protokoll für die Bearbeitung eines C. Albicans -Gens von Interesse. Das Experiment führt ein Stopcodon zu einem Gen verursacht Übersetzung Kündigung. Abhängig von der Reparatur-Vorlage, die eingeführt wird, können andere Änderungen vorgenommen werden. Ein Fragment mit Nourseothricin (NatR) gekennzeichnet, die Hefe Codon-optimierte Cas9 (CaCas9) und eine Anleitung RNA ist integriert in das Genom von C. Albicans an einem neutralen Ort. Cotransformation mit der Reparatur Vorlage Codierung die gewünschte Mutation führt um zu der Spaltung durch homologe Rekombination und effiziente Genom-Bearbeitung zu reparieren. Nachstehend ist die Bearbeitung von TPK2, aber alle C. Albicans offen lesen, dass Frames mehrere Male durch CRISPR ausgerichtet werden können. Das CRISPR-System ist flankiert von FRT Websites und kann aus dem Genom von C. Albicans durch Induktion von Flippase auf dem CaCas9 Ausdruck Plasmid kodiert entfernt werden. Das C. Albicans CRISPR-System ermöglicht Ermittler, C. Albicans Genom11,12exakt und schnell zu bearbeiten.
Protokoll
1. Identifikation und Klonen von Guide RNA-Sequenz
-
Identifikation des Guide RNA-Sequenz
- Identifizieren Sie eine 5'-NGG-3 "PAM-Sequenz in der Nähe, wo das Stopp-Codon eingefügt werden soll. (Abbildung 1A) Mit der Bezeichnung finden alles, was PAM-Sequenzen in den ersten 100 Basenpaaren des TPK2 (Abbildung 1A).
Hinweis: Reiseführer-Sequenzen, die Ausrichtung auf jeder offenen Leserahmen von C. Albicans finden Sie bei http://osf.io/ARDTX 11,12. - Identifizieren der vorwärts Guide Primer_3-Sequenz, die die 20 werden Basen direkt stromaufwärts von einem NGG PAM Website und enthält nicht mehr als 5 Ts in Folge. Mit der linken Maustaste auf der Basis direkt stromaufwärts von der NGG und ziehen Sie der Cursor 20 Basen, dann klicken Sie auf die Registerkarte "Grundierung" hinzufügen die Grundierung.
- Identifizieren Sie die Reverse Guide Primer_3-Sequenz, die die Ergänzung zu der Sequenz nach vorn Guide sein wird.
Hinweis: Gezeigt werden Führungen, die PAM_3 verwenden (Abbildung 1 b). - Mit der rechten Maustaste der Grundierung und wählen Sie "Primer Daten kopieren". Fügen Sie die Sequenzen in ein Textbearbeitungsprogramm ein.
- Identifizieren Sie eine 5'-NGG-3 "PAM-Sequenz in der Nähe, wo das Stopp-Codon eingefügt werden soll. (Abbildung 1A) Mit der Bezeichnung finden alles, was PAM-Sequenzen in den ersten 100 Basenpaaren des TPK2 (Abbildung 1A).
-
Forward und Reverse Guide Oligos zu klonen (Tabelle 1, Abbildung 1 b) erleichtern fügen Sie Überhang Sequenzen hinzu.
- 5'-Ende des vorwärts Guide Primer_3 die Nukleotidsequenz ATTTG vor dem Kauf hinzufügen.
- 3' Ende der vorwärts Guide Primer_3 G vor dem Kauf hinzufügen.
- 5'-Ende des Reverse Guide Primer_3 die Nukleotidsequenz AAAAC vor dem Kauf hinzufügen.
- Die 3' Ende des Reverse Guide Primer_3 C vor dem Kauf hinzu.
-
Digest CaCas9 Ausdruck Vektor pV1524 mit BsmBI.
Hinweis: pV1524 enthält eine Ampicillin (AmpR) und Nourseothricin (NatR) Markierungen. Cas9 wurde für C. AlbicansCodon optimiert.- Das Plasmid zu verdauen, indem Sie hinzufügen: 2 μg pv1524, 5 μl 10-fach Puffer, 1 μL BsmBI und H2O bis 50 μL in einem 1,5 mL-Tube. Bei 55 ° C für 20 min. inkubieren (Alternativ verdauen pv1524 für 15 min mit Esp3I, eine Isoschizomer des BsmBI, bei 37 ° c)
- Cool auf Raumtemperatur (RT) und Spin für 30 s bei 2.348 X g , Kondensation an der Unterseite des Rohres zu bringen. Fahren Sie mit Schritt 1.4 oder speichern das verdaute Plasmid bei-20 ° C.
-
Vergnügen Sie Phosphatase-vorverdaute Rückgrat.
- Die Verdauung Mischung 1 μL der Kalb intestinale Phosphatase (CIP) hinzu und Inkubation bei 37 ° C für 1 h.
- Reinigen Sie das verdaute Plasmid mit einem handelsüblichen Polymerase Kettenreaktion (PCR) Reinigung Kit (mit Kit gelieferten Anweisungen) und in 30 μl der Elution Buffer (EB) eluieren.
-
Phosphorylieren und Tempern nach vorn Guide Primer_3 und Reverse Guide Primer_3.
- Fügen Sie 0,5 μL von 100 μM nach vorn Guide Primer_3, 0,5 μL von 100 μM Reverse Guide Primer_3, 5 μL der 10 x T4 Ligase Puffer, 1 μl T4-Polynukleotid-Kinase und 43 μL von H2O zu einem PCR-Schlauch.
- Fügen Sie 5 μL der 10 x T4 Ligase Puffer, 1 μl T4-Polynukleotid-Kinase und 44 μL der Molekularbiologie-Klasse H2O in einer zweiten PCR-Röhre.
Hinweis: Dies wird als negative Kontrolle dienen. - Inkubieren Sie die Reaktionsgemischen in einem Thermocycler bei 37 ° C für 30 min, dann bei 95 ° C für 5 min.
- Abkühlen der Mischung mit der langsamsten Ramp-Rate auf 16 ° C zu Tempern, Oligos. Legen Sie dann die geglühten Oligo-Mischung bei 4 ° c
-
Verbinden Sie die geglühten Oligos in verdaute pv1524 aus Schritt 1.4.3.
- Fügen Sie Folgendes zu einem PCR-Rohr: 1 μl 10 x T4 Ligase Puffer, 0,5 μl T4 DNA-Ligase, 0,5 μL des geglühten Oligo-Mix, verdaut CIP behandelt gereinigtes Plasmid (20 – 40 ng) und H2O auf ein Gesamtvolumen von 10 μL.
- Fügen Sie Folgendes zu einem PCR-Rohr: 1 μl 10 x T4 Ligase Puffer, 0,5 μl T4 DNA-Ligase, verdaut CIP behandelt gereinigten Vektor (20 – 40 ng), 1 μL der Negativkontrolle Mischung und H2O bis zu einem Gesamtvolumen von 10 μL.
- Beide Röhren in einem Thermocycler bei 16 ° C für 30 min, dann bei 65 ° C für 10 min inkubieren, dann auf 25 ° c abkühlen lassen
- Verwandeln Sie 5 μL der Ligatur Mischungen in chemisch kompetente Escherichia coli DH5α mit einem standard Hitze Schock Umwandlung Protokoll. Wählen Sie auf LB Amp/Nat Medien (200 μg/mL Amp, 50 μg/mL Nat).
Hinweis: PV1524 und seine Derivate auf doppelte Auswahl Medien wählen wird Verlust von Nat/CaCas9/Guide-Modul von FLP/FRT Exzision in Bakterien Nichtbeachtung. - Plasmide aus vier Transformanten durch Miniprep zu reinigen, und Sequenz der Einfügereihenfolge Sequenzierung Primer (Tabelle 1).
Hinweis: Die meiste Zeit, Sequenzierung 4 Transformanten ist ausreichend, um mindestens 1 richtige Klon zu identifizieren. - Plasmide, die die Anleitung RNA ein einziges Mal in die BsmBI schneiden Website bei-20 ° c kloniert Sequenz zu retten
2. Gestaltung und Erstellung von Reparatur-Vorlage
- Legen Sie ein Stopcodon indem mit der linken Maustaste in die Gensequenz und Nukleotide, die eine Stop-Codon und Einschränkung Verdauung-Website (Abbildung 1Tabelle 1) kodieren.
Hinweis: Die Einfügung unterbricht die PAM-Sequenz.
Hinweis: Eine Einschränkung Verdauung Website werden nacheinander die Reparatur Vorlage ermöglichen effiziente Sortierung der Klone (Abbildung 1) berücksichtigt. - Mit der linken Maustaste 10 Basen flussabwärts von wo die Mutation erfolgt und ziehen Sie der Cursor 60 stromaufwärts Basen. Klicken Sie auf die Registerkarte "Grundierung" hinzufügen die Grundierung. Dadurch wird die Reparatur Vorlage Forward_3 hinzugefügt. Mit der linken Maustaste 10 Basen stromaufwärts von wo die Mutation erfolgt und ziehen Sie der Cursor 60 flussabwärts Basen. Klicken Sie auf die Registerkarte "Grundierung" hinzufügen die Grundierung. Dadurch wird die Reparatur Vorlage Reverse 3 hinzugefügt. (Abbildung 1)
-
Führen Sie Grundierung Erweiterung um die Reparatur-Vorlage generieren.
- Hinzufügen von 100 μm Reparatur Vorlage forward Primer, 1,2 μl von 100 μm Reparatur Vorlage rückwärts-Primer, 6 μL Deoxynucleotide Triphosphate (dNTPs) 1,2 μl (volle Konzentration 40 mM), 6 μl Puffer, 0,6 μL der Taq Polymerase (3 Einheiten) und 45 μl von H2O zu jedem der 4 PCR Röhren.
- Zwischen 20 und 30 Runden von PCR durchführen Sie Primer Erweiterung. Beispiel-Erweiterung Bedingungen: 2 min bei 95 ° C (30 s bei 95 ° C, 1 min bei 50 ° C, 1 min bei 68 ° C) X 34, 10 min bei 68 ° C.
- Kombinieren Sie alle 4 PCR-Rohre in einer 1,5 mL Tube Inhalt und verwenden Sie eine PCR-Reinigung-Kit, um Produkte in 50 μL der H2O. reinigen
- Quantitate Grundierung Erweiterungsprodukte um sicherzustellen, dass genügend DNA Bestimmung der Absorption bei 260 nm.
Hinweis: Typische Endkonzentration von der Grundierung Erweiterungsprodukt ist ~ 200-300 ng/µL.
3. Umwandlung von C. Albicans mit Reparatur Vorlage und Plasmid
-
TE/Lithium Acetat zu machen.
- Mischung 10 mM Tris-Cl, 1 mM EDTA 100 mM Lithium Acetat und H2O (alle Stammlösung pH 7,5), 50 mL Gesamtvolumen zu erreichen.
-
Platte zu machen
- Mischen Sie 40 % PEG 3350, 100 mM Lithium Acetat, 10 mM Tris-Cl, 1 mM EDTA und H2O (alle Stammlösung pH 7,5) um 50 mL Gesamtvolumen zu erreichen.
-
Richtig geklont Plasmide aus Schritt 1,9 zu verdauen.
- Fügen Sie 10 µg Plasmid, 4 µL 10-fach Puffer, 0.4 µL 10 mg/mL Rinderserumalbumin (BSA), 0,5 µL KpnI, 0,5 µL SacI und H20 bis 40 µL Gesamtvolumen in einem 1,5 mL-Tube. Inkubation bei 37 ° C über Nacht (Abbildung 1).
-
Wachsen Sie eine Übernachtung Kultur von C. Albicans SC5314, Wildtyp Prototroph bei 25 ° C in Hefe Pepton Traubenzucker ergänzt mit 0,27 mM Uridin (YPD + Uri), im Idealfall auf weniger als 6 OD600 .
- Pellet-5 OD600 Einheiten von Zellen pro Umwandlung von Spinnerei für 5 min bei 2.348 X g und 5 OD600 gebeizte Zellen in 100 µL TE/Lithium Acetat auszusetzen.
- Fügen Sie Folgendes zu einer 1,5 mL-Tube in der angegebenen Reihenfolge: (1) 100 µL Zellen aus Schritt 3.4.1, 2) 40 µL gekocht und schnell abgekühlt Lachs Samenzellen DNA (10 mg/mL), 3) 10 µg Plasmid Verdauung aus Schritt 3.3.1, (4) 6 µg gereinigten Reparatur Vorlage und 5) 1 mL der Platte.
- Fügen Sie Folgendes zu einer 1,5 mL-Tube in der angegebenen Reihenfolge: (1) 100 µL Zellen, (2) 40 µL gekocht und schnell abgekühlt Lachs Samenzellen DNA (10 mg/mL), (3) H2O Volumen gleich, dass DNA in Schritt 3,5 und 4) 1 mL der Platte zu verwandeln.
Hinweis: Dies wird als eine negative Kontrolle dienen. - Mischen Sie die Transformationen durch Pipettieren vorsichtig und lassen Sie über Nacht bei 25 ° C inkubieren.
- Hitze Schock die Zellen, indem man sie in ein Wasserbad 44 ° C für 25 min.
- Für 5 min bei 2.348 X g in einer Zentrifuge Benchtop drehen Sie und entfernen Sie die Platte Mischung überstand. Durch Zugabe von 1 mL der YPD + Uri und Zentrifuge wieder für 5 min bei 2.348 X geinmal waschen.
- Unterbrechen Sie die Zellen in 0,1 mL YPD + Uri und über Nacht auf einer Bandage oder Shaker bei 25 ° C inkubieren.
- Platte auf YPD + Uri mit 200 μg/mL Nourseothricin. Kolonien erscheinen in 2 – 5 Tage.
(4) Streifen für die einzelnen Kolonien
-
Teilen Sie einen 100 mm x 15 mm Petrischale Vierteln, und beschriften Sie jeden Quadranten.
- Tippen Sie auf eine der Kolonien von der Transformation-Platte mit einer sterilen Zahnstocher oder Applikator und Streifen auf der längsten Seite des Quadranten.
- Streifen für die einzelnen Kolonien mit einem aseptischen Technik und die Kolonien für 2 Tage bei 30 ° C wachsen lassen.
(5) Kolonie PCR
-
Die nach vorne Check Grundierung (FCP) ~ 200 Basenpaare stromaufwärts des Standortes Einschränkung eingeführt wurde und die reverse Check Grundierung (RCP) ~ 300 Basenpaare stromabwärts zu entwerfen.
- Hinzufügen von 0,3 μL des FCP, 0,3 μL der RCP, 0,3 μL der thermostabile Polymerase (ExTaq 1,5 Einheiten), 3 μL von dNTPs (volle Konzentration 40 mM), 3 μl ExTaq-Puffer und 23 μL von H2O, eine 1,5 mL-Tube.
Hinweis: Zugabe von 0,5 μL/Reaktion Dimethyl Sulfoxid (DMSO) kann PCR-Effizienz verbessern. - Die Mischung aus Schritt 5.1.1, mit einer Pipettenspitze P10 und kümmert sich nicht um den Agar stören fügen Sie 0,25 μl einer einzigen Hefe-Kolonie aus Schritt 4.1.2 hinzu.
- Verstärken Sie DNA durch PCR und 5 μl des PCR auf ein Gel um sicherzustellen, dass die Verstärkung ist erfolgreich, kümmert sich nicht um die Zelle Pellet am unteren Ende der Röhre zu stören.
- Hinzufügen von 0,3 μL des FCP, 0,3 μL der RCP, 0,3 μL der thermostabile Polymerase (ExTaq 1,5 Einheiten), 3 μL von dNTPs (volle Konzentration 40 mM), 3 μl ExTaq-Puffer und 23 μL von H2O, eine 1,5 mL-Tube.
6. Beschränkungsverdauung der Kolonie PCR
- Fügen Sie 10 μl des PCR-Produkt (kümmert sich nicht um die Zelle Pellet am Boden des Röhrchens nicht stören), 3 μL des Puffers 1 μL der Restriktionsenzym und 16 μL von H2O, dann brüten nach Herstellerangaben und Beschlussfassung über ein Agarosegel Corre identifizieren CT-Genom-Bearbeitungen.
Hinweis: Das Restriktionsenzym verwendet hier ist der Ort in der TPK2 bestimmten Reparatur Vorlage codiert.
7. speichern Stämme
- Wachsen einer Übernachtung Kultur aus den Kolonien, die durch Beschränkungsverdauung YPD + Uri bei 30 ° c bestätigt wurden
- Fügen Sie 1 mL der Kultur und 1 mL 50 % Glycerin (womit die letztendliche Konzentration an Glycerin auf 25 %) zu einem Schlauch lagerfähig bei-80 ° C.
- Speichern Sie die richtigen Klone bei-80 ° C.
Hinweis: Richtige Stämme können bei-80 ° C für viele Jahre gespeichert werden.
8. Entfernung von NatR Marker
- Korrekte Transformant Streifen auf Hefe Pepton Maltose (YPMaltose) (2 % Maltose).
- Wählen Sie eine Kolonie von der gestreiften Platte und Kultur der Hefe für 48 h bei 30 ° C im flüssigen YPMaltose 20 g/L Maltose.
- Platte 200 – 400 Zellen auf YPMaltose 20 g/L Maltose und Inkubation bei 30 ° C für 24 h.
- Replizieren Sie die Platte auf YPMaltose und YPMaltose 200 μg/mL Nat.
- Inkubation bei 30 ° C für 24 h.
Hinweis: Kolonien, die nicht mehr wachsen auf YPMaltose 200 μg/mL Nourseothricin, sondern auf YPMaltose wachsen die Nat-R -Markierung (CaCAS9) verloren haben und RNA zu führen. - Speichern Sie die Stämme, die die NatR Marker (CaCAS9) und Guide RNA wie in Schritten 7.1-7.3 verloren haben.
Hinweis: Eine ähnliche Plasmid, pV1393, nutzt die SAP2 im Gegensatz zu einem MAL2 -Promotor. Wachstum auf YCB – BSA wird Flippase und Entfernung von NatR induzieren, wenn pV1393, für die Bearbeitung von gen verwendet wird.
Ergebnisse
Sequenzen von guide RNAs und Reparatur-Vorlagen, die auf C. Albicans TPK2, eine c-AMP-Kinase katalytische Untereinheit, wurden nach den oben beschriebenen Richtlinien entwickelt. Sequenzen werden (Tabelle 1, Abbildung 1) angezeigt. Guide-RNAs waren in CaCas9 Ausdruck Vektoren kloniert und cotransformiert mit Reparatur Vorlage in Wildtyp C. Albicans. Ein EcoRI Einschränkung Verdauung Website und Stop-Codons in der Reparatur-Vorlage die PAM-Website zu stören und zu erleichtern-Screening für korrekte Mutanten (Abbildung 1). Transformanten wurden für die einzelnen Kolonien durchzogen und abgeschirmt durch Kolonie-PCR und Einschränkung Verdauung für den Einbau der Reparatur Vorlage (Abbildung 2). Beschränkungsverdauung unterscheidet schnell Wildtyp mutierte Sequenzen.
| Oligonukleotid-Name | Oligonukleotid Sequenz |
| Nach vorne Guide Primer_3 | ATTTGGggtgaactatttgttcgccG |
| Umgekehrte Guide Primer_3 | AAAACGgcgaacaaatagttcacccC |
| Reparatur-Vorlage Forward_3 | TcagcaatatcagcaacaatttcaacaaccgcagcaacaactttatTAAGAATTCggcga |
| Reparatur-Vorlage Reverse_3 | AttttgtccagtttgggctgcagcagggtgaactatttgttcgccGAATTCTTAataaag |
| Nach vorne Check Primer | ttaaagaaacttcacatcaccaag |
| Reverse Check Primer | actttgatagcataatatctaccat |
| Sequenzierung Primer | ggcatagctgaaacttcggccc |
Tabelle 1: Liste der Oligo-Nukleotide, die für diese Studie verwendet. Websites, die für das Klonen Zwecke hinzugefügt werden groß geschrieben und in der Führer-Primer Fett-Sequenzen. Sequenzen in der Reparatur-Vorlage, die die genomische DNA mutieren werden aktiviert und Fett.
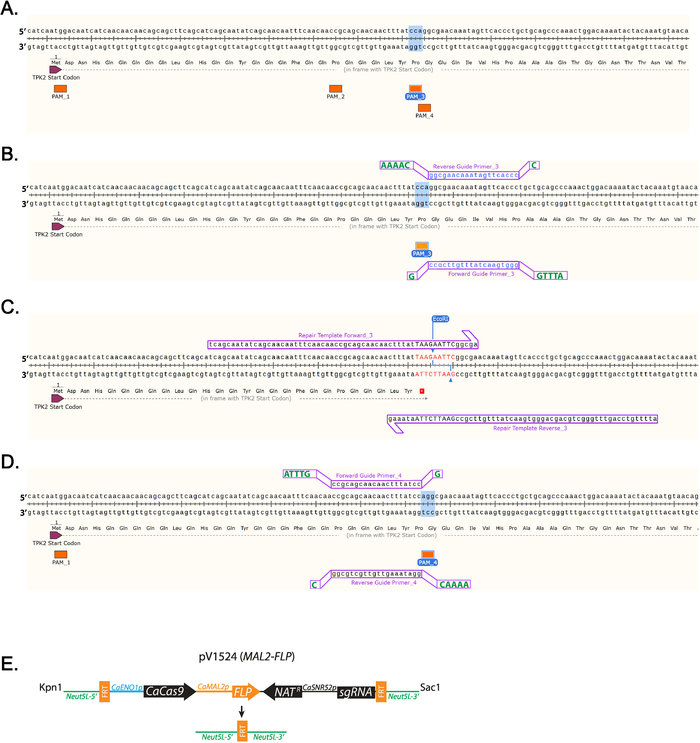
Abbildung 1 : Diagramm der Führer RNS und Reparatur Vorlagenentwurf. (A) Kennzeichnung der PAM-Sequenzen in den ersten 100 Nukleotide der TPK2. PAM Sequenz 3 (PAM_3) wird hervorgehoben, denn das ist die Reihenfolge in dieser Studie verwendet. (B) Guide RNA design mit PAM_3. 20 Basis Primer mit SnapGene entworfen sind Kleinbuchstaben und blau. Weitere Basen erforderlich für das Klonen sind Großbuchstaben und grün Offset angezeigt. (C) Reparatur Vorlage Zündkapseln, die ein Stopcodon TAA und EcoRI-Site einfügen werden in die TPK2 lesen Frame eingefügt. DNA, die aus der Wildtyp Sequenz unterscheidet sich ist rot und Großbuchstaben. (D) Beispiel dafür, wie eine Anleitung auf dem positiven Strang der DNA mit Hilfe PAM_4 konzipiert ist. (E) schematische Darstellung der pV1524 nach dem Klonen des Leitfadens RNA und Verdauung mit KpnI und SacI. Neut5L-5' und Neut5L-3 "Ziel den Vektor auf der Neut5L Website in das Genom von C. Albicans . CaENO1p ist der Promotor, der Ausdruck von der Hefe-optimierte CaCas9antreibt. NAT-R ist die Nourseothricin-Widerstand-Kassette. CaSNR52p ist der Promotor Guide RNA Ausdruck (SgRNA) fahren. FRT Aufstellungsorte sind gespalten und von Flippase (FLP) Entfernen der CRISPR-Kassette auf Flippase Ausdruck rekombiniert. Eine schematische Darstellung ähnlich (E) wurde von Vyas Et al.veröffentlicht. 11. Bitte klicken Sie hier für eine größere Version dieser Figur.
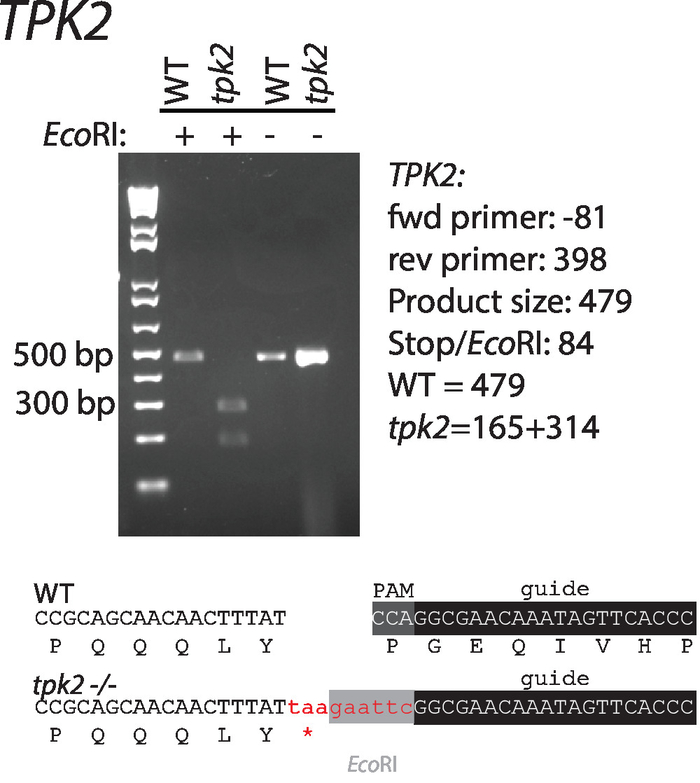
Abbildung 2 : Einführung und Bestätigung eines Stopp-Codon und EcoRI Einschränkung Website, TPK2. Primer zur Verstärkung sind in Tabelle 1aufgeführt. Wildtyp und Mutante-Sequenzen finden Sie unten das Gel. Diese Zahl wurde von Vyas Et Al.11geändert. Bitte klicken Sie hier für eine größere Version dieser Figur.
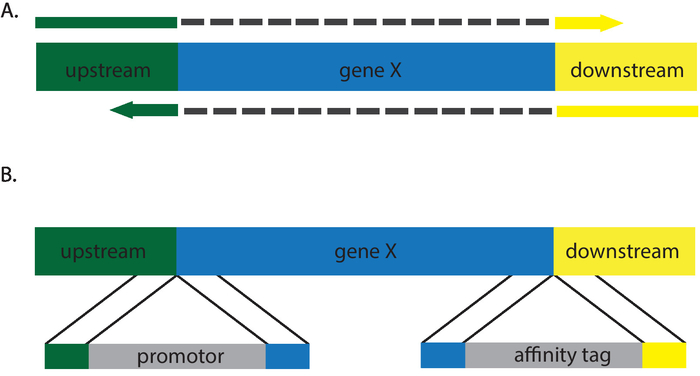
Abbildung 3 : Cartoon-Beschreibung der Reparatur-Vorlagen, die Löschungen (A) und (B) Einfügungen generieren wird. Grauen Striche in (A) zeigen dazwischenliegende Sequenzen in die Reparatur Vorlage Primer nicht vorhanden. Bitte klicken Sie hier für eine größere Version dieser Figur.
Diskussion
C. Albicans CRISPR effizient bearbeitet das Genom von C. Albicans . pV1524 kodiert eine Hefe-Codon-optimierte Cas9 und ist so konzipiert, dass Ermittler leicht Guide RNA-Sequenzen flussabwärts des CaSNR52 Promotor (Abbildung 1)11Klonen können. Es muss sichergestellt werden, dass nur eine einzige Kopie der Anleitung Sequenz in CaCas9 Expressionsvektoren geklont wurde durch Sequenzierung, da Extrakopien Genom-Bearbeitung behindern werden. Wenn mehrere Kopien des Leitfadens konsequent eingeführt werden, sollte man die Konzentration des geglühten Guide in Ligatur verwendet senken. Der Vektor und beschriebenen Protokoll erlauben targeting von C. Albicans -gen. Obwohl C. Albicans diploid ist, muss nur eine einzelne Transformation beide Allele eines Gens zu richten. Darüber hinaus ermöglicht die prozessualen Natur der CRISPR-CaCas9 Genom-Bearbeitung Forscher mehrere Mitglieder von Genfamilien ausrichten. Viele Genfamilien wie die sekretierten Aspartyl Proteasen (SAPS) und Agglutinin-Sequenz Proteine (ALS) sind wichtig für C. Albicans Virulenz. CRISPR Genom-Bearbeitung erleichtert die Untersuchung dieser gen-Familien.
Die oben beschriebenen Protokolle stellen ein Stopcodon eine offene Leseraster, wodurch das phänotypische Äquivalent von einem Null-Wert (Abbildung 2). Eine Vielzahl von genetischen Veränderungen kann durch Variation der Reparatur Vorlage erfolgen. Unsinn und Missense Stille Mutationen können durch Rekombination mit einem entsprechenden Reparatur-Template eingefügt werden. Gründung einer Einschränkung Website optimiert Transformant Screening, als diejenigen ohne untersucht werden müssen, durch Sequenzierung12,13. Darüber hinaus ermöglicht C. Albicans CRISPR Forscher erzeugen Einfügungen und Löschungen, so dass es ein ideales System Affinität-Tags einfügen, Promoter Swaps, und generieren Knockouts (Abbildung 3). Screening für korrekte Transformanten für diese Mutationen ist aufwendiger, da es notwendig, um die Änderungen bestätigen korrekte Einbindung der Reparatur Vorlagen zu sequenzieren. Südlicher Fleck möglicherweise darüber hinaus muss sichergestellt werden, dass zusätzliche Kopien eines Gens nicht an weiteren Standorten im Genom eingefügt wurden. Die Anforderung des Standortes NGG PAM stellt leichte Einschränkungen auf die Regionen des Genoms, die ausgerichtet werden können. Die Entwicklung von alternativen CRISPR-Systemen, Einsatz alternativer Nukleasen wie Cpf1 oder Variationen über das Cas9-System haben/werden viele von diesen Einschränkungen14lindern. Die Ermittler wissen zu diesem Zeitpunkt wurden diese Systeme nicht noch zu C. Albicansübernommen.
Im obigen Protokoll beschriebene CRISPR-System wurde entwickelt, so dass es in einer Vielzahl von Arten, einschließlich Saccharomyces Cerevisiae, Naumouozyma Castelliiund der menschlichen Erreger Candida Glabrata11 angewendet werden können . Transformation und effiziente Bearbeitung von diese Hefe erfordert leichte Änderungen an den beschriebenen Protokoll, aber der Rahmen für die Bearbeitung dieser alternativen Genome ist bemerkenswert ähnlich, die für C. Albicans12beschrieben. Darüber hinaus Hefe bieten einen ausgezeichnete Mechanismus Genom Bearbeitung von Verfahren zu entwickeln. Hefe wenn ADE2 mutiert ist, sammelt sich in eine Vorstufe zu der Adenin-Biosynthese-Weg, die Zellen rot zu drehen. Diese leicht beobachtbaren Phänotyp ermöglicht Ermittler bearbeitete Zellen erkennen und schnell beheben Genom Protokolle bearbeiten. In Kombination mit der umfangreichen Molekularbiologie-Toolbox für Pilze, wurden die Protokolle für die Bearbeitung zahlreicher Hefearten entwickelten15,16. Eine breite Anwendung des Genoms Technik in Pilzen hat das Potenzial, eine Vielzahl von wissenschaftlichen Disziplinen erheblich beeinträchtigen.
CRISPR hat sich stark verbessert die Effizienz des Genoms Engineering in C. Albicans, aber bisher CRISPR nicht verwendet wurde, um Genom breite Bildschirme in C. Albicansdurchführen. Aktuelle Protokolle erfordern Reparatur Vorlage einzuführen Mutationen, wie nonhomologous Ende C. Albicans Weg mitmachen ineffizient12ist. Die Generation der Reparatur Vorlage Oligos für jedes Gen ist ein erhebliches Hindernis für die Ausführung der genomweiten Bildschirme. Zusammenfluss von verringerten Kosten der DNA-Synthese und Forderungen an CRISPR-Technologien wird Entwicklung von Löschung Bibliotheken mehr möglich machen. Zum Beispiel ebnet Ausdruck einer Reparatur Vorlage aus dem CaCas9-Vektor den Weg für die Entwicklung von nachhaltigen Plasmid-Bibliotheken, die auf jedes Gen11. Darüber hinaus wurden vorübergehende Candida CRISPR-Protokolle, die keine CaCas9 Expressionsvektor integriert in das Genom von C. Albicans entwickelten17. Darüber hinaus erhöht Reiseführer Ausdruck erhöht Genom Bearbeitung Effizienz18. Diese und andere Forderungen an CRISPR-Technologien, sind entscheidend für die Entwicklung der genomweiten Bildschirme in C. Albicans19,20,21,22.
Das Genom von C. Albicans ist diploiden, aber A und B-Allele sind nicht immer identisch5. Solchen Heterozygotie bietet Herausforderungen und Chancen. Wenn man beide Allele ausrichten soll, müssen eine PAM-Website, Leitfaden Sequenz und Reparatur-Vorlage, die auf beide Kopien des Gens wird verwendet werden. Jedoch ermöglicht abhängig von Einzel-Nukleotid-Polymorphismen, die in einem gen, C.albicans CRISPR System Ermittler auf ein einzelnes Allel abzielen. Diese Präzision hat das Potenzial, Ermittler, funktionalen Unterschiede zwischen Allele untersuchen zu lassen. Ausrichtung auf bestimmte Allele muss sorgfältig erfolgen wie Verlust der Heterozygotie (LOH) auf ein Allel oder ein ganzes Chromosom festgestellt wurde. Bei der Bearbeitung von einzelnen C. Albicans Allele, muss man prüfen, neben DNA-Sequenzen, um festzustellen, ob ein Klon ein diploider SNP-Profil beibehalten hat. Darüber hinaus Ziel Effekte sind ziemlich niedrig für C. Albicans CRISPR, aber kompletten Genoms kann für wichtige Stämme in Betracht gezogen werden.
Offenlegungen
Die Autoren haben nichts preisgeben.
Danksagungen
Die Autoren danken Dr. Gennifer Mager für Lesung und hilfreichen Kommentare auf das Manuskript. Unterstützt wurde diese Arbeit von Ball State University Labor Start Fonds und NIH-1R15AI130950-01, D.A.B.
Materialien
| Name | Company | Catalog Number | Comments |
| Chemicals | |||
| Agar | BD Bacto | 214010 | |
| agarose | amresco | 0710-500G | |
| Ampicillan | Sigma-Aldrich | A9518 | |
| Bacto Peptone | BD Bacto | 211677 | |
| Bsmb1 | New England Biolabs | R0580L | |
| Calf intestinal phosphatase (CIP) | New England Biolabs | M0290L | |
| Cut Smart Buffer | New England Biolabs | B7204S | |
| Dimethyl Sulfoxide (DMSO) | Sigma-Aldrich | D8418 | |
| dNTPs | New England Biolabs | N0447L | |
| Ethylenediaminetetraacetic acid (EDTA) | Sigma-Aldrich | 3609 | |
| Glacial Acetic Acid | Sigma-Aldrich | 2810000ACS | |
| Glucose | BDH VWR analytical | BDH9230 | |
| Glycerol | Sigma-Aldrich | 49767 | |
| Kpn1 | New England Biolabs | R3142L | |
| LB-Medium | MP | 3002-032 | |
| Lithium Acetate | Sigma-Aldrich | 517992 | |
| L-Tryptophan | Sigma-Aldrich | T0254 | |
| Maltose | Sigma-Aldrich | M5885 | |
| Molecular Biology Water | Sigma-Aldrich | W4502 | |
| NEB3.1 Buffer | New England Biolabs | B7203S | |
| Nourseothricin | Werner Bioagents | 74667 | |
| Poly(ethylene glycol) PEG 3350 | Sigma-Aldrich | P4338 | |
| Sac1 | New England Biolabs | R3156L | |
| Salmon Sperm DNA | Invitrogen | AM9680 | |
| T4 Polynucleotide kinase | New England Biolabs | M0201S | |
| T4 DNA ligase | New England Biolabs | M0202L | |
| Taq polymerase | New England Biolabs | M0267X | |
| Tris HCl | Sigma-Aldrich | T3253 | |
| uridine | Sigma-Aldrich | U3750 | |
| Yeast Extract | BD Bacto | 212750 | |
| Equipment | |||
| Electrophoresis Appartus | |||
| Incubator | |||
| Microcentifuge | |||
| PCR machine | |||
| Replica Plating Apparatus | |||
| Rollerdrum or shaker | |||
| Spectrophotometer | |||
| Waterbath |
Referenzen
- Pfaller, M. A., Diekema, D. J. Epidemiology of invasive candidiasis: a persistent public health problem. Clinincal Microbiology Review. 20 (1), 133-163 (2007).
- Magill, S. S., et al. Multistate point-prevalence survey of health care-associated infections. New England Journal of Medicine. 370 (13), 1198-1208 (2014).
- Wisplinghoff, H., et al. Nosocomial bloodstream infections in US hospitals: analysis of 24,179 cases from a prospective nationwide surveillance study. Clinical Infectious Disease. 39 (3), 309-317 (2004).
- Jones, T., et al. The diploid genome sequence of Candida albicans. Proceedings of the National Academy of Science USA. 101 (19), 7329-7334 (2004).
- Muzzey, D., Schwartz, K., Weissman, J. S., Sherlock, G. Assembly of a phased diploid Candida albicans genome facilitates allele-specific measurements and provides a simple model for repeat and indel structure. Genome Biology. 14 (9), (2013).
- Morschhauser, J., Michel, S., Staib, P. Sequential gene disruption in Candida albicans by FLP-mediated site-specific recombination. Molecular Microbiololgy. 32 (3), 547-556 (1999).
- Lau, V., Davie, J. R. The discovery and development of the CRISPR system in applications in genome manipulation. Biochemistry and Cell Biology. 95 (2), 203-210 (2017).
- Doudna, J. A., Charpentier, E. Genome editing. The new frontier of genome engineering with CRISPR-Cas9. Science. 346 (6213), 1258096 (2014).
- Anders, C., Niewoehner, O., Duerst, A., Jinek, M. Structural basis of PAM-dependent target DNA recognition by the Cas9 endonuclease. Nature. 513 (7519), 569-573 (2014).
- Reardon, S. Welcome to the CRISPR zoo. Nature. 531 (7593), 160-163 (2016).
- Vyas, V. K., et al. New CRISPR Mutagenesis Strategies Reveal Variation in Repair Mechanisms among Fungi. mSphere. 3 (2), (2018).
- Vyas, V. K., Barrasa, M. I., Fink, G. R. A Candida albicans CRISPR system permits genetic engineering of essential genes and gene families. Science Advances. 1 (3), e1500248 (2015).
- Evans, B. A., et al. Restriction digest screening facilitates efficient detection of site-directed mutations introduced by CRISPR in C. albicans UME6. PeerJ. 6, e4920 (2018).
- Kim, D., et al. Genome-wide analysis reveals specificities of Cpf1 endonucleases in human cells. Nature Biotechnology. 34 (8), 863-868 (2016).
- Tarasava, K., Oh, E. J., Eckert, C. A., Gill, R. T. CRISPR-enabled tools for engineering microbial genomes and phenotypes. Biotechnology Journal. , e1700586 (2018).
- Raschmanova, H., Weninger, A., Glieder, A., Kovar, K., Vogl, T. Implementing CRISPR-Cas technologies in conventional and non-conventional yeasts: Current state and future prospects. Biotechnology Advances. 36 (3), 641-665 (2018).
- Min, K., Ichikawa, Y., Woolford, C. A., Mitchell, A. P. Candida albicans Gene Deletion with a Transient CRISPR-Cas9 System. mSphere. 1 (3), (2016).
- Ng, H., Dean, N. Dramatic Improvement of CRISPR/Cas9 Editing in Candida albicans by Increased Single Guide RNA Expression. mSphere. 2 (2), (2017).
- Huang, M. Y., Woolford, C. A., Mitchell, A. P. Rapid Gene Concatenation for Genetic Rescue of Multigene Mutants in Candida albicans. mSphere. 3 (2), (2018).
- Shapiro, R. S., et al. A CRISPR-Cas9-based gene drive platform for genetic interaction analysis in Candida albicans. Nature Microbiology. 3 (1), 73-82 (2018).
- Grahl, N., Demers, E. G., Crocker, A. W., Hogan, D. A. Use of RNA-Protein Complexes for Genome Editing in Non-albicans Candida Species. mSphere. 2 (3), (2017).
- Nguyen, N., Quail, M. M. F., Hernday, A. D. An Efficient, Rapid, and Recyclable System for CRISPR-Mediated Genome Editing in Candida albicans. mSphere. 2 (2), (2017).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten