Method Article
CRISPR-mediata Editing genomico agente patogeno fungoso umano Candida albicans
* Questi autori hanno contribuito in egual misura
In questo articolo
Riepilogo
Ingegneria di genoma efficiente dei albicans del Candida è fondamentale per comprendere la patogenesi e lo sviluppo di terapeutica. Qui, abbiamo descritto un protocollo per rapidità e precisione modificare il genoma di c. albicans utilizzando CRISPR. Il protocollo consente ai ricercatori di introdurre un'ampia varietà di modificazioni genetiche, tra cui le mutazioni puntiformi, inserzioni e delezioni.
Abstract
Questo metodo viene descritto l'editing genomico efficiente CRISPR mediata dell'agente patogeno fungoso umana diploide Candida albicans. L'editing genomico CRISPR-mediata in c. albicans richiede Cas9, guida RNA e la riparazione di modello. Un plasmide esprimente un codone di lievito ottimizzato Cas9 (CaCas9) è stato generato. Sequenze di guida direttamente a Monte da un PAM sito (NGG) vengono clonati nel vettore di espressione di Cas9. Un modello di riparazione quindi fatta dal primer extension in vitro. Cotransformation del modello di riparazione e vettoriale in c. albicans conduce a editing genomico. A seconda del modello di riparazione usato, lo sperimentatore può introdurre cambiamenti del nucleotide, inserimenti o eliminazioni. Come albicans del c. è un diploide, mutazioni sono realizzate in entrambi gli alleli di un gene, purché gli alleli A e B non harbor SNPs che interferire con la Guida di targeting o incorporazione di modello di riparazione. Famiglie geniche multimembro possono essere modificate in parallelo, in presenza di sequenze conservate adatti a tutti i membri della famiglia. Il sistema di c. albicans CRISPR descritto è affiancato da siti FRT e codifica flippase. All'induzione di flippase, il marcatore antibiotico (CaCas9) e guida RNA vengono rimossi dal genoma. Questo consente al ricercatore di eseguire successive modifiche al genoma. C. albicans CRISPR è uno strumento potente fungine ingegneria genetica, e piccole modifiche ai protocolli descritti consentono la modifica di altre specie fungine, tra cui glabrata del c., N. castellii e S. cerevisiae.
Introduzione
Candida albicans è il più prevalente agente patogeno fungoso umano1,2,3. Comprensione delle differenze tra albicans del c. e biologia molecolare dei mammiferi è fondamentale per lo sviluppo della prossima generazione di terapie antifungine. Questo richiede investigatori per poter rapidamente e con precisione geneticamente manipolare c. albicans.
Manipolazione genetica di albicans del c. è stata storicamente impegnativo. C. albicans non mantiene plasmidi, così tutti i costrutti devono essere incorporati nel genoma. Inoltre, c. albicans è diploide; Pertanto, quando buttando giù un gene o l'introduzione di una mutazione, è importante garantire che entrambe le copie sono state cambiate4. Inoltre, alcuni loci di albicans del c. sono eterozigoti, complicando ulteriormente genetico interrogatorio5. Per manipolare geneticamente albicans del c., è tipico per eseguire più cicli di ricombinazione omologa6. Tuttavia, la natura diploide del genoma e laborioso costrutto sviluppo hanno reso questo un processo potenzialmente noioso, specialmente se sono necessarie modifiche multiple. Queste limitazioni e l'importanza medica di albicans del c. richiedono lo sviluppo di nuove tecnologie che permettono ai ricercatori di manipolare più facilmente il genoma di c. albicans .
Cluster regolarmente interspaziati breve palindromi ripetizioni (CRISPR)-editing genomico mediata è un potente strumento che permette ai ricercatori di modificare la sequenza di un genoma. CRISPR richiede tre componenti: 1) la nucleasi Cas9 che fende il DNA bersaglio, 2) un 20 RNA guida destinata a Cas9 alla sequenza di interesse di base e 3) riparazione del DNA di modello che ripara il sito di clivaggio e incorpora la modifica prevista7, 8. Una volta che la Guida porta Cas9 alla sequenza del genoma di destinazione, Cas9 richiede una sequenza di motivi adiacenti (PAM) protospacer (NGG) direttamente a Monte della sequenza guida fendere il DNA9. Il requisito per i 20 Guida base e le stesse sequenze di PAM fornisce un elevato grado di specificità di targeting e limita fenditura fuori bersaglio.
CRISPR sistemi sono stati progettati per modificare il genoma di un insieme diversificato di organismi e affrontare un'ampia varietà di problemi10. Descritto qui è un protocollo CRISPR, flessibile ed efficiente per l'editing di un gene di albicans del c. di interesse. L'esperimento introduce un codone di arresto di un gene, che causa la terminazione della traduzione. Altre modifiche possono essere fatte a seconda del modello di riparazione che è stato introdotto. Un frammento contrassegnato con nourseothricin (Natr) contenenti lievito ottimizzato codone Cas9 (CaCas9) ed una guida RNA è incorporata il genoma di c. albicans presso un campo neutro. Cotransformation con il modello di riparazione codifica la mutazione desiderata conduce alla riparazione del clivaggio di ricombinazione omologa ed editing genomico efficiente. Descritto di seguito è la redazione di TPK2, ma tutti i albicans del c. open frame possono essere mirati più volte da CRISPR di lettura. Il sistema CRISPR è affiancato da siti FRT e può essere rimosso dal genoma albicans del c. di induzione di flippase codificato sul plasmide di espressione di CaCas9. Il sistema di c. albicans CRISPR consente ai ricercatori di esattamente e rapidamente modificare il genoma di albicans del c. 11,12.
Protocollo
1. identificazione e clonazione di guida RNA sequenza
-
Identificazione della sequenza di RNA Guida
- Identificare un 5'-NGG-3' sequenza di PAM vicino dove verrà inserito il codone di stop. (Figura 1A) Con l'etichetta PAM sequenze si trovano tutti nelle prime 100 coppie di basi di TPK2 (Figura 1A).
Nota: Sequenze di guida ogni frame di lettura aperta di albicans del c. di targeting possono essere trovati alla http://osf.io/ARDTX 11,12. - Identificare la sequenza avanti guida Primer_3, che sarà il 20 basi direttamente a Monte di una NGG PAM del sito e non conterrà più di 5 Ts in una riga. Tasto sinistro del mouse sulla base direttamente a Monte della NGG e trascinare il cursore 20 basi, quindi fare clic sulla scheda per aggiungere il primer primer.
- Identificare la sequenza inversa guida Primer_3, che sarà il complemento alla sequenza avanti guida.
Nota: Vengono riportate le guide che utilizzano PAM_3 (Figura 1B). - Il primer di destro e selezionare "copia dati primer". Incollare le sequenze in un editor di testo.
- Identificare un 5'-NGG-3' sequenza di PAM vicino dove verrà inserito il codone di stop. (Figura 1A) Con l'etichetta PAM sequenze si trovano tutti nelle prime 100 coppie di basi di TPK2 (Figura 1A).
-
Aggiungere sequenze di sporgenza oligos Forward e Reverse guida per facilitare la clonazione (tabella 1, Figura 1B).
- Aggiungere la sequenza nucleotidica ATTTG all'estremità 5' della Primer_3 di guida in avanti prima di acquistare.
- Aggiungere G all'estremità 3' della Primer_3 di guida in avanti prima di acquistare.
- Aggiungere la sequenza nucleotidica AAAAC all'estremità 5' della Primer_3 di guida invertire prima dell'acquisto.
- Aggiungere C all'estremità 3' della Primer_3 di guida invertire prima dell'acquisto.
-
Digest CaCas9 espressione vettoriale pV1524 con BsmBI.
Nota: pV1524 contiene un ampicillina (Ampr) e nourseothricin (Natr) marcatori. Cas9 è stato ottimizzato per codone per c. albicans.- Digerire il plasmide aggiungendo: 2 μg di pv1524, 5 μL di tampone 10x, 1 μL di BsmBI e H2O a 50 μL in una provetta da 1,5 mL. Incubare a 55 ° C per 20 minuti (in alternativa, digerire pv1524 per 15 min con Esp3I, un isoschizomer di BsmBI, a 37 ° C.)
- Raffreddare a temperatura ambiente (TA) e spin per 30 s a 2.348 x g per portare la condensazione alla parte inferiore del tubo. Procedere con il passo 1.4 o memorizzare il plasmide digerito a-20 ° C.
-
Fosfatasi-ossequio la spina dorsale digerita.
- Aggiungere 1 μL di fosfatasi intestinale di vitello (CIP) per la miscela di digestione e incubare a 37 ° C per 1 h.
- Purificare il plasmide digerito utilizzando un kit di purificazione di reazione a catena (PCR) polimerasi commercialmente disponibile (le istruzioni fornite con il kit) ed eluire esso a 30 μL di tampone di eluizione (EB).
-
Fosforilare e tempri avanti guida Primer_3 e Reverse Primer_3 guida.
- Aggiungere 0,5 μL di 100 μM avanti guida Primer_3, 0,5 μL di 100 μM invertire guida Primer_3, 5 μL di tampone di ligasi x T4 10, 1 μL della chinasi di polinucleotide T4 e 43 μL di H2O a un tubo PCR.
- Aggiungere 5 µ l di tampone di ligasi 10 x T4, 1 μL della chinasi di polinucleotide T4 e 44 μL di biologia molecolare-grade H2O in un secondo tubo PCR.
Nota: Questo servirà come controllo negativo. - Incubare le miscele di reazione in un termociclatore a 37 ° C per 30 min, poi a 95 ° C per 5 min.
- Raffreddare la miscela al più lento tasso di rampa a 16 ° C per temprare i, i oligos. Quindi inserire la miscela di oligo ricotto a 4 ° C.
-
Legare i oligos ricotto in pv1524 digerito dal punto 1.4.3.
- Aggiungere quanto segue in una provetta PCR: 1 μL di 10 x T4 ligasi buffer, 0,5 μL di T4 DNA ligasi, 0,5 μL di miscela di oligo ricotto, digerito CIP-trattati purificato il plasmide (20 – 40 ng) e H2O a un volume totale di 10 μL.
- Aggiungere quanto segue in una provetta PCR: 1 μL di tampone di 10 x T4 ligasi, 0,5 μL di T4 DNA ligasi, digerito vettoriale purificata CIP-trattati (20 – 40 ng), 1 μL di miscela di controllo negativo e H2O fino a un volume totale di 10 μL.
- Incubare entrambi i tubi in un termociclatore a 16 ° C per 30 min, poi al 65 ° C per 10 minuti, poi raffreddare a 25 ° C.
- 5 μL delle miscele di legatura si trasformano in chimicamente competenti Escherichia coli DH5α utilizza un protocollo di trasformazione scossa di calore standard. Selezionare su supporti LB Amp/Nat (Amp, 50 μg/mL Nat 200 μg/mL).
Nota: Fallimento per selezionare pV1524 e suoi derivati sui media doppia selezione comporterà perdita di Nat/CaCas9/guida modulo dall'asportazione di FLP/FRT nei batteri. - Purificare i plasmidi da quattro transformants di miniprep e sequenza la sequenza di inserimento con primer di sequenziamento (tabella 1).
Nota: la maggior parte del tempo, sequenziamento 4 trasformanti è sufficiente per identificare almeno 1 clone corretto. - Salvare i plasmidi che hanno la sequenza di RNA guida clonata una sola volta nel BsmBI tagliare sito a-20 ° C.
2. progettazione e generazione di modello di riparazione
- Inserire un codone di arresto sinistro nella sequenza genica e aggiungendo nucleotidi che codificano un codone e restrizione digestione sito sportello (Figura 1tabella 1).
Nota: L'inserimento interromperà la sequenza di PAM.
Nota: Un sito di restrizione digestione sarà tra la sequenza di modello di riparazione per facilitare efficiente selezione dei cloni (Figura 1). - Sinistro del mouse 10 basi a valle di dove avverrà la mutazione e trascinare che il cursore 60 basi a Monte. Fare clic sulla scheda per aggiungere il primer primer. Questo aggiungerà riparazione modello Forward_3. Sinistro del mouse 10 basi a Monte di dove avverrà la mutazione e trascinare che il cursore 60 basi a valle. Fare clic sulla scheda per aggiungere il primer primer. Questo aggiungerà riparazione modello Reverse 3. (Figura 1)
-
Eseguire estensione dell'iniettore per generare il modello di riparazione.
- Aggiungere 1,2 μL di 100 novità di forward primer μm riparazione modello, 1,2 μL di 100 novità di reverse primer μm riparazione modello, 6 μL di deossinucleotidi trifosfati (dNTPs) (concentrazione totale 40 mM), 6 μL di tampone, 0,6 μL di Taq polimerasi (3 unità) e 45 μL di H2O a ciascuno dei 4 PCR tubi.
- Eseguire estensione dell'iniettore di corsa tra 20 e 30 giri di PCR. Condizioni di estensione di esempio: 2 min a 95 ° C (30 s a 95 ° C, 1 min a 50 ° C, 1 min a 68 ° C) x 34, 10 min a 68 ° C.
- Combinare il contenuto di tutte le 4 provette PCR in una provetta da 1,5 mL e utilizzare un kit di purificazione di PCR per purificare i prodotti in 50 μL di H2O.
- Quantificare i prodotti di estensione del primer per assicurare sufficiente DNA determinando l'assorbanza a 260 nm.
Nota: Concentrazione finale tipico del prodotto estensione primer è ~ 200-300 ng / µ l.
3. trasformazione dei albicans del c. con modello di riparazione e plasmide
-
Fare acetato di TE/litio.
- Miscelare 10 mM Tris-Cl, 1 mM EDTA, acetato di litio 100 mM e H2O (tutti gli stock soluzione pH 7,5) per ottenere il volume totale di 50 mL.
-
Fare piastra
- Mescolare 40% PEG 3350, acetato di litio 100 mM, 10 mM Tris-Cl, 1 mM EDTA e H2O (tutti gli stock soluzione pH 7,5) per ottenere il volume totale di 50 mL.
-
Digerire correttamente clonato plasmidi dal passaggio 1,9.
- Aggiungere 10 μg di plasmide, 4 µ l di tampone 10x, 0,4 µ l di 10 mg/mL albumina di siero bovino (BSA), 0,5 µ l di KpnI, 0,5 µ l di SacI e H20 a 40 µ l di volume in una provetta da 1,5 mL totale. Incubare a 37 ° C durante la notte (Figura 1).
-
Far crescere una cultura durante la notte di c. albicans SC5314, selvaggio-tipo prototroph, a 25 ° C in destrosio di lievito peptone completato con 0,27 mM uridina (YPD + Uri), idealmente a OD600 meno di 6.
- A pellet 5 unità di600 OD di cellule per trasformazione di filatura per 5 min a 2.348 x g e sospendere il 5 OD600 delle cellule pellettate in acetato di TE/litio 100 µ l.
- Aggiungere quanto segue in una provetta da 1,5 mL nell'ordine indicato: 1) 100 µ l di cellule dal punto 3.4.1, 2) 40 µ l di sperma di salmone bollito e raffreddato a rapido DNA (10 mg/mL), 3) 10 µ g di digestione del plasmide dal punto 3.3.1, 4) 6 µ g di modello di riparazione purificato e 5) 1 mL di piastra.
- Aggiungere quanto segue in una provetta da 1,5 mL nell'ordine indicato: 1) 100 µ l di cellule, 2) 40 µ l di sperma di salmone bollito e raffreddato a rapido del DNA (10 mg/mL), volume di2O 3) H uguale a quello della trasformazione del DNA nel passaggio 3.5 e 4) 1 mL di piastra.
Nota: Questo servirà come controllo negativo. - Mescolare delicatamente le trasformazioni pipettando e lasciare Incubare a 25 ° C durante la notte.
- Calore di scossa le cellule ponendoli in bagnomaria a 44 ° C per 25 min.
- Girare per 5 min a 2.348 x g in una centrifuga da banco e rimuovere la miscela di piastra surnatante. Lavare una volta aggiungendo 1 mL di YPD + Uri e centrifugare nuovamente per 5 min a 2.348 x g.
- Sospendere le cellule in 0,1 mL di YPD + Uri e incubare su un tamburo rullo o in uno shaker a 25 ° C durante la notte.
- Piastra su YPD + Uri con 200 μg/mL nourseothricin. Colonie apparirà in 2 – 5 giorni.
4. striature per singole colonie
-
Dividere un 100 mm x 15 mm di Petri in quarti ed etichettare ogni quadrante.
- Toccare una delle colonie dalla piastra di trasformazione con stuzzicadenti sterile o applicatore e striscia attraverso il lato più lungo del quadrante.
- Striscia per singole colonie usando una tecnica asettica e consentire le colonie a crescere a 30 ° C per 2 giorni.
5. PCR della Colonia
-
Progettare il controllo avanti primer (FCP) ~ 200 paia di basi a Monte del sito di restrizione è stata introdotta e il controllo reverse primer (RCP) ~ 300 paia di basi a valle.
- Aggiungere 0,3 μL di FCP, 0.3 μL di RCP, 0.3 μL di polimerasi termostabile (ExTaq 1.5 unità), 3 μL di dNTPs (concentrazione totale 40 mM), 3 μL di tampone di ExTaq e 23 μL di H2O a una provetta da 1,5 mL.
Nota: Aggiunta di 0,5 μL/reazione dimetil solfossido (DMSO) può migliorare l'efficienza PCR. - Aggiungere 0.25 μL di una colonia di lievito singolo passaggio 4.1.2 alla miscela dal punto 5.1.1, utilizzando una punta di pipetta P10, facendo attenzione a non per disturbare l'agar.
- Amplificare il DNA mediante PCR ed eseguire 5 μL della PCR su un gel per garantire l'amplificazione è successo, avendo cura di non per disturbare il pellet cellulare nella parte inferiore del tubo.
- Aggiungere 0,3 μL di FCP, 0.3 μL di RCP, 0.3 μL di polimerasi termostabile (ExTaq 1.5 unità), 3 μL di dNTPs (concentrazione totale 40 mM), 3 μL di tampone di ExTaq e 23 μL di H2O a una provetta da 1,5 mL.
6. restrizione digestione di PCR della Colonia
- Aggiungere 10 μL di PCR prodotto (prendersi cura non per non disturbare il pellet cellulare nella parte inferiore del tubo), 3 μL di tampone, 1 μL di enzima di restrizione e 16 μL di H2O, quindi incubare secondo le istruzioni del produttore e risolvere su un gel di agarosio per identificare corre modifiche di genoma di CT.
Nota: L'enzima di restrizione usato qui è il sito codificato nel TPK2 riparazione specifico modello.
7. risparmio ceppi
- Far crescere una cultura durante la notte dalle colonie che sono state confermate da digestione di limitazione in YPD + Uri a 30 ° C.
- Aggiungere 1 mL di coltura e 1 mL di glicerolo al 50% (portando la concentrazione finale di glicerolo al 25%) a un tubo adatto per la conservazione a-80 ° C.
- Memorizzare i cloni corretti a-80 ° C.
Nota: Corretti ceppi possono essere memorizzati a-80 ° C per molti anni.
8. rimozione di Natr Marker
- Striatura transformant corretta sul maltosio di lievito peptone (YPMaltose) (2% maltosio).
- Scegli una colonia dalla piastra striata e il lievito per 48 h a 30 ° C in maltosio di 20 g/L YPMaltose liquido della coltura.
- Piastra di 200 – 400 celle il maltosio 20 g/L YPMaltose e incubare a 30 ° C per 24 h.
- Replicare la piastra al YPMaltose e YPMaltose 200 μg/mL NAT.
- Incubare a 30 ° C per 24 h.
Nota: Colonie che non crescono su YPMaltose 200 μg/mL nourseothricin ma crescono sulla YPMaltose hanno perso il Natr marker (CaCAS9) e guidano di RNA. - Salvare i ceppi che hanno perso il Natr marker (CaCAS9) e guida RNA come fatto nei passaggi 7.1-7.3.
Nota: Un plasmide simile, pV1393, utilizza il SAP2 al contrario di un promotore di MAL2 . Crescita sul YCB – BSA indurrà flippase e rimozione di Natr se pV1393 viene utilizzato per l'editing di gene.
Risultati
Sequenze di guida RNAs e riparazione modelli destinati a albicans del c. TPK2, una subunità catalitica della chinasi c-AMP, sono stati progettati secondo le linee guida suggerite sopra. Sequenze sono illustrate nella (tabella 1, Figura 1). Guida RNAs sono stati clonati in vettori di espressione di CaCas9 e cotransformed con modello di riparazione nel selvaggio-tipo c. albicans. Un sito di digestione di restrizione EcoRI e codoni di stop nel modello riparazione danneggiare il sito di PAM e facilitano la selezione per corrette mutanti (Figura 1). Trasformanti sono stati striati per singole colonie e proiettati dalla digestione di PCR e restrizione di Colonia per l'incorporazione del modello riparazione (Figura 2). Digestione di limitazione distingue rapidamente selvaggio-tipo da sequenze mutanti.
| Nome del oligonucleotide | Sequenza di oligonucleotidi |
| Guida avanti Primer_3 | ATTTGgggtgaactatttgttcgccG |
| Invertire la guida Primer_3 | AAAACggcgaacaaatagttcacccC |
| Riparazione modello Forward_3 | tcagcaatatcagcaacaatttcaacaaccgcagcaacaactttatTAAGAATTCggcga |
| Riparazione modello Reverse_3 | attttgtccagtttgggctgcagcagggtgaactatttgttcgccGAATTCTTAataaag |
| Check Forward Primer | ttaaagaaacttcacatcaccaag |
| Check Reverse Primer | actttgatagcataatatctaccat |
| Primer di sequenziamento | ggcatagctgaaacttcggccc |
Tabella 1: elenco dei nucleotidi oligo utilizzati per questo studio. Siti aggiunti per la clonazione scopi sono in maiuscolo e in grassetto nell'iniettore guida sequenze. Sequenze nel modello di riparazione che mutano il DNA genomico sono in maiuscolo e in grassetto.
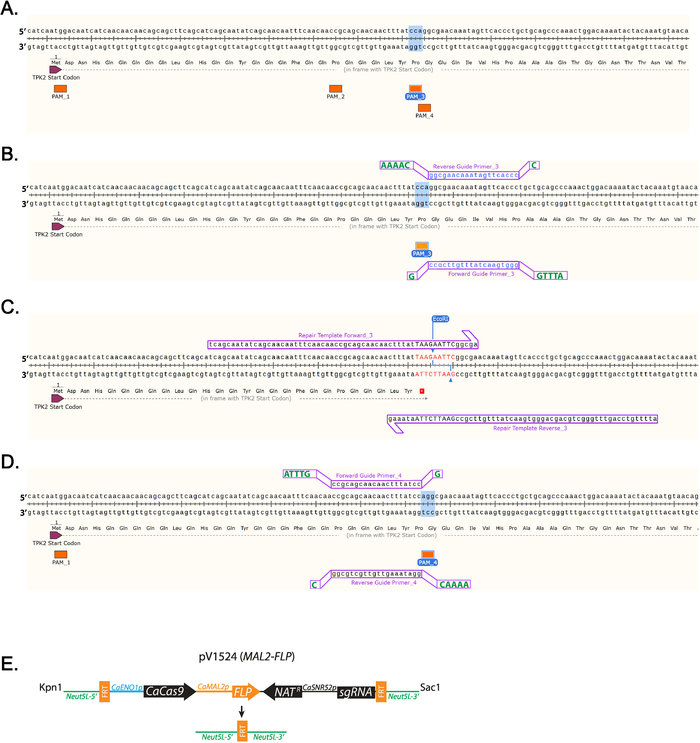
Figura 1 : Diagramma di guida di disegno del modello di RNA e riparazione. (A) etichettatura di tutte le sequenze di PAM nei primi 100 nucleotidi di TPK2. PAM sequenza 3 (PAM_3) è evidenziata, come che è la sequenza utilizzata in questo studio. (B) guida RNA progettare utilizzando PAM_3. 20 Primer base disegnati utilizzando SnapGene sono minuscole e blu. Ulteriori basi necessarie per la clonazione sono maiuscole e verde indicato offset. (C) riparazione iniettori modello che inserire un codone di stop TAA ed EcoRI sito vengono inseriti nella TPK2 cornice di lettura. DNA che differisce dalla sequenza wild-type è rosso e maiuscolo. (D) esempio di come una guida è stata progettata sul filo positivo del DNA usando PAM_4. (E) diagramma schematico di pV1524 dopo la clonazione della Guida RNA e digestione con KpnI e SacI. Neut5L-5' e Neut5L-3' il vettore al sito Neut5L del genoma di c. albicans di destinazione. CaENO1p è il promotore che spinge l'espressione del lievito-ottimizzato CaCas9. NATr è la cassetta di resistenza nourseothricin. CaSNR52p è il promotore Guida Guida espressione del RNA (sgRNA). FRT siti sono spaccati e ricombinati di flippase (FLP) rimozione della cartuccia del CRISPR su flippase espressione. Uno schema simile alla (E) è stato pubblicato da Vyas et al. 11. Clicca qui per visualizzare una versione più grande di questa figura.
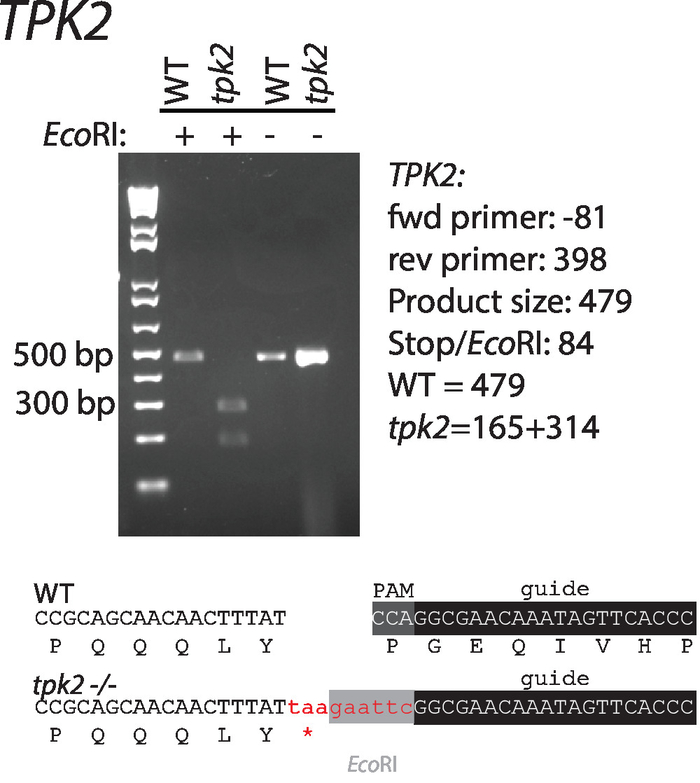
Figura 2 : Introduzione e conferma di un codone di arresto e il sito di restrizione EcoRI a TPK2. Primer usati per l'amplificazione sono elencati nella tabella 1. Sequenze wild-type e mutanti sono indicate sotto il gel. Questa figura è stata modificata da Vyas et al.11. Clicca qui per visualizzare una versione più grande di questa figura.
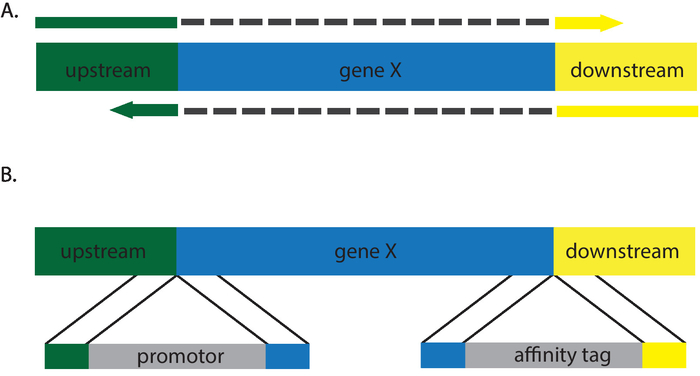
Figura 3 : Cartoon Descrizione dei modelli di riparazione che genererà eliminazioni (A) e (B) inserimenti. Grigio trattini in (A) rappresentano sequenze d'intervento non è presente nei primer di modello di riparazione. Clicca qui per visualizzare una versione più grande di questa figura.
Discussione
C. albicans CRISPR efficiente consente di modificare il genoma di c. albicans . pV1524 consente di codificare un lievito codone-ottimizzato Cas9 ed è progettato tali che gli investigatori possono facilmente clonare sequenze di guida RNA a valle del promotore (Figura 1) CaSNR52 11. È necessario accertarsi che una sola copia della sequenza guida è stata clonata in vettori di espressione CaCas9 mediante sequenziamento, come copie extra impedirà l'editing genomico. Se più copie della guida vengono introdotti in modo coerente, uno dovrebbe abbassare la concentrazione della Guida ricotta utilizzata nella legatura. Il vettore e il protocollo descritto consentono di targeting di qualsiasi gene c. albicans . Anche se i albicans del c. è diploide, solo una singola trasformazione è necessaria per indirizzare entrambi gli alleli di un gene. Inoltre, la natura processive del CRISPR-CaCas9 editing genomico consente ai ricercatori di più membri di famiglie geniche di destinazione. Molte famiglie geniche quali la secrezione aspartil proteasi (SAPS) e le proteine agglutinina-come sequenza (ALS) sono importanti per la virulenza di c. albicans . CRISPR editing genomico faciliterà l'indagine di queste famiglie geniche.
I protocolli descritti sopra introducono un codone di stop per un open reading frame, conseguente l'equivalente fenotipica di un valore null (Figura 2). Un'ampia varietà di alternanze genetiche può avvenire mediante la variazione del modello di riparazione. Sciocchezze, missenso e mutazioni silenti possono essere inserite tramite ricombinazione con un modello di correzione appropriata. Incorporazione di un sito di restrizione snellisce transformant screening, come quelli senza deve essere schermati mediante sequenziamento12,13. Inoltre, c. albicans CRISPR consente ai ricercatori di generare inserimenti ed eliminazioni, che lo rende un sistema ideale per inserire tag affinità, eseguire consente di scambiare promotor e generare Ko (Figura 3). Screening per trasformanti corretto per queste mutazioni è più laborioso, come è necessario sequenziare le modifiche per confermare la corretta incorporazione dei modelli di riparazione. Inoltre, macchia del sud può essere necessaria garantire ulteriori copie di un gene non sono state inserite in altre posizioni nel genoma. Il requisito del sito NGG PAM posti lievi limitazioni sulle regioni del genoma che possono essere mirati. Lo sviluppo di sistemi alternativi di CRISPR che uso alternativo nucleasi come Cpf1 o variazioni sul sistema Cas9 sono/saranno alleviare molti di questi limiti14. A conoscenza degli investigatori in questo momento, questi sistemi non sono ancora stati applicati al c. albicans.
Il sistema CRISPR descritto nel protocollo di cui sopra è stato sviluppato tale che può essere applicato in un'ampia varietà di specie tra cui Saccharomyces cerevisiae, Naumouozyma castelliie l'agente patogeno umano glabrata del Candida11 . Trasformazione e un editing efficiente di questi lieviti richiede lievi modifiche al protocollo descritto, ma il quadro per la modifica di questi genomi alternativi è notevolmente simile a quello descritto per i albicans del c.12. Inoltre, lievito forniscono un eccellente meccanismo di sviluppare procedure di editing genomico. In lievito, quando ADE2 è mutato, un precursore per la via di biosintesi di adenina si accumula, trasformando le cellule rosse. Questo fenotipo facilmente osservabile permette ai ricercatori di identificare celle modificate e rapidamente risolvere i protocolli di modifica del genoma. Combinato con il toolbox di biologia molecolare estesa disponibile per i funghi, protocolli per la modifica di numerose specie di lievito sono stati sviluppati15,16. Una vasta applicazione di tecnologia di editing del genoma nei funghi ha il potenziale per un impatto significativo sulle più svariate discipline scientifiche.
CRISPR ha notevolmente migliorato l'efficienza di ingegneria del genoma di c. albicans, ma ad oggi CRISPR non è stato utilizzato per eseguire schermi di grandi dimensioni del genoma di c. albicans. Attuali protocolli richiedono un modello di riparazione per introdurre le mutazioni, come la nonhomologous fine partecipare via c. albicans è inefficiente12. La generazione di riparazione modello oligos per ogni gene è un ostacolo significativo alla realizzazione di schermi di tutto il genoma. Confluenza dei costi in diminuzione della sintesi del DNA e gli avanzamenti alle tecnologie CRISPR renderà la sviluppo di librerie di eliminazione più fattibile. Per esempio, espressione di un modello di riparazione dal vettore CaCas9 spiana la strada per lo sviluppo di librerie di plasmide sostenibile che ogni gene11di destinazione. Inoltre, protocolli CRISPR Candida transitorie che non necessitano di vettore di espressione CaCas9 incorporano nel genoma di c. albicans sono stati sviluppati17. Inoltre, aumentato guida espressione aumenta genoma efficienza18di editing. Questi ed altri avanzamenti alle tecnologie CRISPR, sono cruciali per lo sviluppo di schermi di genoma in albicans del c.19,20,21,22.
Il genoma di c. albicans è diploide, ma A e B alleli non sono sempre identici5. Tali eterozigosi fornisce entrambe le sfide e le opportunità. Se uno si propone di entrambi gli alleli di destinazione, deve essere utilizzati un sito PAM, sequenza di guida e modello di riparazione che agirà su entrambe le copie del gene. Tuttavia, dipendendo dalla polimorfismi a singolo nucleotide presente in un gene, il sistema CRISPR c consente ai ricercatori di un singolo allele di destinazione. Tale precisione ha il potenziale per consentire ai ricercatori di esaminare le differenze funzionali tra alleli. Targeting per alleli specifici deve essere fatta con attenzione, come perdita di eterozigosi (LOH) a un allele o di un intero cromosoma è stato osservato. Quando si modifica singola c. albicans alleli, uno deve esaminare sequenze di DNA adiacenti per determinare se un clone ha mantenuto un profilo SNP diploide. Inoltre, fuori bersaglio effetti sono abbastanza bassi per CRISPR albicans del c. , ma sequenziamento dell'intero genoma può essere considerato per ceppi chiave.
Divulgazioni
Gli autori non hanno nulla a rivelare.
Riconoscimenti
Gli autori ringraziano il Dr. Gennifer Mager per lettura e commenti utili sul manoscritto. Questo lavoro è stato sostenuto da Ball State University Laboratorio avvio fondi e NIH-1R15AI130950-01 al d.a.b.
Materiali
| Name | Company | Catalog Number | Comments |
| Chemicals | |||
| Agar | BD Bacto | 214010 | |
| agarose | amresco | 0710-500G | |
| Ampicillan | Sigma-Aldrich | A9518 | |
| Bacto Peptone | BD Bacto | 211677 | |
| Bsmb1 | New England Biolabs | R0580L | |
| Calf intestinal phosphatase (CIP) | New England Biolabs | M0290L | |
| Cut Smart Buffer | New England Biolabs | B7204S | |
| Dimethyl Sulfoxide (DMSO) | Sigma-Aldrich | D8418 | |
| dNTPs | New England Biolabs | N0447L | |
| Ethylenediaminetetraacetic acid (EDTA) | Sigma-Aldrich | 3609 | |
| Glacial Acetic Acid | Sigma-Aldrich | 2810000ACS | |
| Glucose | BDH VWR analytical | BDH9230 | |
| Glycerol | Sigma-Aldrich | 49767 | |
| Kpn1 | New England Biolabs | R3142L | |
| LB-Medium | MP | 3002-032 | |
| Lithium Acetate | Sigma-Aldrich | 517992 | |
| L-Tryptophan | Sigma-Aldrich | T0254 | |
| Maltose | Sigma-Aldrich | M5885 | |
| Molecular Biology Water | Sigma-Aldrich | W4502 | |
| NEB3.1 Buffer | New England Biolabs | B7203S | |
| Nourseothricin | Werner Bioagents | 74667 | |
| Poly(ethylene glycol) PEG 3350 | Sigma-Aldrich | P4338 | |
| Sac1 | New England Biolabs | R3156L | |
| Salmon Sperm DNA | Invitrogen | AM9680 | |
| T4 Polynucleotide kinase | New England Biolabs | M0201S | |
| T4 DNA ligase | New England Biolabs | M0202L | |
| Taq polymerase | New England Biolabs | M0267X | |
| Tris HCl | Sigma-Aldrich | T3253 | |
| uridine | Sigma-Aldrich | U3750 | |
| Yeast Extract | BD Bacto | 212750 | |
| Equipment | |||
| Electrophoresis Appartus | |||
| Incubator | |||
| Microcentifuge | |||
| PCR machine | |||
| Replica Plating Apparatus | |||
| Rollerdrum or shaker | |||
| Spectrophotometer | |||
| Waterbath |
Riferimenti
- Pfaller, M. A., Diekema, D. J. Epidemiology of invasive candidiasis: a persistent public health problem. Clinincal Microbiology Review. 20 (1), 133-163 (2007).
- Magill, S. S., et al. Multistate point-prevalence survey of health care-associated infections. New England Journal of Medicine. 370 (13), 1198-1208 (2014).
- Wisplinghoff, H., et al. Nosocomial bloodstream infections in US hospitals: analysis of 24,179 cases from a prospective nationwide surveillance study. Clinical Infectious Disease. 39 (3), 309-317 (2004).
- Jones, T., et al. The diploid genome sequence of Candida albicans. Proceedings of the National Academy of Science USA. 101 (19), 7329-7334 (2004).
- Muzzey, D., Schwartz, K., Weissman, J. S., Sherlock, G. Assembly of a phased diploid Candida albicans genome facilitates allele-specific measurements and provides a simple model for repeat and indel structure. Genome Biology. 14 (9), (2013).
- Morschhauser, J., Michel, S., Staib, P. Sequential gene disruption in Candida albicans by FLP-mediated site-specific recombination. Molecular Microbiololgy. 32 (3), 547-556 (1999).
- Lau, V., Davie, J. R. The discovery and development of the CRISPR system in applications in genome manipulation. Biochemistry and Cell Biology. 95 (2), 203-210 (2017).
- Doudna, J. A., Charpentier, E. Genome editing. The new frontier of genome engineering with CRISPR-Cas9. Science. 346 (6213), 1258096 (2014).
- Anders, C., Niewoehner, O., Duerst, A., Jinek, M. Structural basis of PAM-dependent target DNA recognition by the Cas9 endonuclease. Nature. 513 (7519), 569-573 (2014).
- Reardon, S. Welcome to the CRISPR zoo. Nature. 531 (7593), 160-163 (2016).
- Vyas, V. K., et al. New CRISPR Mutagenesis Strategies Reveal Variation in Repair Mechanisms among Fungi. mSphere. 3 (2), (2018).
- Vyas, V. K., Barrasa, M. I., Fink, G. R. A Candida albicans CRISPR system permits genetic engineering of essential genes and gene families. Science Advances. 1 (3), e1500248 (2015).
- Evans, B. A., et al. Restriction digest screening facilitates efficient detection of site-directed mutations introduced by CRISPR in C. albicans UME6. PeerJ. 6, e4920 (2018).
- Kim, D., et al. Genome-wide analysis reveals specificities of Cpf1 endonucleases in human cells. Nature Biotechnology. 34 (8), 863-868 (2016).
- Tarasava, K., Oh, E. J., Eckert, C. A., Gill, R. T. CRISPR-enabled tools for engineering microbial genomes and phenotypes. Biotechnology Journal. , e1700586 (2018).
- Raschmanova, H., Weninger, A., Glieder, A., Kovar, K., Vogl, T. Implementing CRISPR-Cas technologies in conventional and non-conventional yeasts: Current state and future prospects. Biotechnology Advances. 36 (3), 641-665 (2018).
- Min, K., Ichikawa, Y., Woolford, C. A., Mitchell, A. P. Candida albicans Gene Deletion with a Transient CRISPR-Cas9 System. mSphere. 1 (3), (2016).
- Ng, H., Dean, N. Dramatic Improvement of CRISPR/Cas9 Editing in Candida albicans by Increased Single Guide RNA Expression. mSphere. 2 (2), (2017).
- Huang, M. Y., Woolford, C. A., Mitchell, A. P. Rapid Gene Concatenation for Genetic Rescue of Multigene Mutants in Candida albicans. mSphere. 3 (2), (2018).
- Shapiro, R. S., et al. A CRISPR-Cas9-based gene drive platform for genetic interaction analysis in Candida albicans. Nature Microbiology. 3 (1), 73-82 (2018).
- Grahl, N., Demers, E. G., Crocker, A. W., Hogan, D. A. Use of RNA-Protein Complexes for Genome Editing in Non-albicans Candida Species. mSphere. 2 (3), (2017).
- Nguyen, N., Quail, M. M. F., Hernday, A. D. An Efficient, Rapid, and Recyclable System for CRISPR-Mediated Genome Editing in Candida albicans. mSphere. 2 (2), (2017).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon