Method Article
En Vivo Reprogramación Directa de Células Gliales Residentes en Interneuronas por Inyección Intracerebral de Vectores Virales
En este artículo
Resumen
Este protocolo tiene como objetivo generar interneuronas directamente reprogramadas in vivo, utilizando un sistema viral basado en AAV en el cerebro y un reportero de FPSG impulsado por la sinapsina FLEX, que permite la identificación celular y el análisis posterior in vivo.
Resumen
Convertir la glia residente en el cerebro en neuronas funcionales y específicas del subtipo in vivo proporciona un paso hacia el desarrollo de terapias alternativas de reemplazo celular mientras que también crea herramientas para estudiar el destino celular in situ. Hasta la fecha, ha sido posible obtener neuronas a través de la reprogramación in vivo, pero el fenotipo preciso de estas neuronas o cómo maduran no se ha analizado en detalle. En este protocolo, describimos una conversión más eficiente y una identificación específica de la célula de las neuronas reprogramadas in vivo, utilizando un sistema vectorial viral basado en AAV. También proporcionamos un protocolo para la evaluación funcional de la maduración neuronal de las células reprogramadas. Mediante la inyección de vectores de flip-escision (FLEX), que contienen los genes de reportero impulsados por la reprogramación y la sinapsina a tipos de células específicas en el cerebro que sirven como el objetivo para la reprogramación celular. Esta técnica permite la fácil identificación de las neuronas recién reprogramadas. Los resultados muestran que las neuronas reprogramadas obtenidas maduran funcionalmente con el tiempo, reciben contactos sinápticos y muestran propiedades electrofisiológicas de diferentes tipos de interneuronas. Usando los factores de transcripción Ascl1, Lmx1a y Nurr1, la mayoría de las células reprogramadas tienen propiedades de las interneuronas que contienen parrebúta y parvalbumina.
Introducción
El objetivo general de este método es convertir eficientemente la glia residente del cerebro in vivo en neuronas funcionales y específicas del subtipo, como las interneuronas que expresan parvalbumina. Esto proporciona un paso adelante hacia el desarrollo de una terapia de reemplazo celular alternativo para enfermedades cerebrales sin la necesidad de una fuente celular exógena. También crea una herramienta para estudiar los cambios de destino celular in situ.
El cerebro tiene una capacidad limitada para generar nuevas neuronas. Por lo tanto, en las enfermedades neurológicas, hay una necesidad de fuentes celulares exógenas para la reparación del cerebro. Para ello, diferentes fuentes de células han sido sometidas a intensas investigaciones a lo largo de los años, incluyendo células de tejido primario, células derivadas de células madre y células reprogramadas1,2,3. La reprogramación directa de las células cerebrales residentes en las neuronas es un enfoque reciente que podría proporcionar un método atractivo para la reparación del cerebro, ya que utiliza las propias células del paciente para generar nuevas neuronas dentro del cerebro. Hasta la fecha, varios informes han mostrado in vivo reprogramación a través de la entrega de vectores virales en el cerebro4,5,6,78,9 en diferentes regiones cerebrales como la corteza, la médula espinal, elestriado y el cerebro medio 5,10,11, así como en el cerebro intacto y lesionado5,8,11,12. Tanto las neuronas inhibitorias comolas excitatorias se han obtenido 4,8, pero el fenotipo preciso o funcionalidad de estas células aún no se ha analizado en detalle.
En este protocolo, describimos una reprogramación más eficiente y una identificación específica de la célula de las neuronas reprogramadas in vivo. Proporcionamos un protocolo para la evaluación funcional de la maduración neuronal y la caracterización del fenotipo basada en la funcionalidad y los rasgos inmunohistoquímicos.
Usamos un vector AAV inducible cre y un reportero de GFP para identificar las neuronas reprogramadas in vivo. Esta elección de vectores virales tiene la ventaja de infectar células divisorias y no divisorias del cerebro,aumentando el número de células objetivo, como alternativa al uso del retrovirus 7,8. Un reportero FLEX (GFP, por sus tantos) basado en neuronas, nos permitió detectar específicamente las neuronas recién generadas. Estudios anteriores han utilizado promotores específicos de subtipos para la reprogramación in vivo7,9, que también permiten la expresión de reprogramación de genes y reporteros en tipos de células específicas. Sin embargo, ese método requiere una mayor identificación de las neuronas reprogramadas mediante el análisis postmortem de la coexpresión del reportero y los marcadores neuronales. El uso de un reportero específico de neuronas, como el descrito en este documento, permite una identificación directa. Esto proporciona una prueba directa de una conversión exitosa y permite una identificación celular en vivo que se requiere para la electrofisiología de abrazadera de parche.
Protocolo
Todos los procedimientos experimentales se llevaron a cabo en virtud de la Directiva de la Unión Europea (2010/63/UE) y fueron aprobados por el comité ético para el uso de animales de laboratorio en la Universidad de Lund y el Departamento de Agricultura de Suecia (Jordbruksverket). Los ratones se alojan en un ciclo de luz/oscuridad de 12 h con acceso ad libitum a alimentos y agua.
1. Vectores virales
- Clonación de vectores AAV
- Para crear vectores AAV5 inducibles cre-inducibles, inserte cDNA para GFP, Ascl1, Lmx1a y NR4A2 (Nurr1) en una orientación inversa flanqueada por dos pares de secuencias heterotipadas y antiparalelas de volteo-escisión LoxP (FLEX) (Tablade materiales). Para la inserción de ADNc, utilice una columna vertebral como pAAV-Cba-FLEX o una con una estructura similar, que contenga secuencias FLEX y un promotor de beta actina de pollo (CBA).
- Inserte cada ADNc individual (por ejemplo, Ascl1) en la columna vertebral mediante la reacción en cadena de la polimerasa (PCR) y las enzimas de restricción. Express GFP bajo el control de un promotor de sinapsina y Ascl1, Lmx1a, Nurr1 bajo el control de un promotor DE CBA ubicuamente expresado.
NOTA: Asegúrese de tener ADN libre de endotoxinas utilizando kits específicos de aislamiento de ADN sin endotoxinas. - Realice la secuenciación y el análisis de restricciones en las construcciones antes de su uso, con el fin de comprobar el éxito del paso de clonación.
- Producción de vectores virales AAV5
PRECAUCION: Consulte las pautas locales de bioseguridad al tratar el virus asociado a adeno (AAV). En Suecia, el AAV utilizado en este protocolo requiere el nivel de bioseguridad 2 (BSL-2).- Células HEK293T de semilla con medios de cultivo estándar (DMEM+Glutamax + 10% FBS + penicilina (100 U/ml) estreptomicina (100 g/ml), ver Tabla de materiales) en matraces T175 a una densidad de 3 x 106 células por matraz. Cuente 5 matraces por lote de AAV y planifique 6 lotes a la vez.
- Cuando las células alcancen una confluencia del 50-70%, prepare la siguiente mezcla para la transfección (por matraz de 175 cm 2).
- En un tubo de centrífuga de 50 ml, agregue cantidades equimolares de plásmido vectorial y plásmido auxiliar de la serie pDG (pDP5, pDP6), con una cantidad total de 72 g por matraz de 175 cm 2.
- Agregue el buffer Tris-EDTA (buffer TE, 10 mM Tris-HCl, 1 mM EDTA) a un volumen final de 144 mL.
- Añadir agua ultrapura para que el volumen total se convierta en 1296 ml y mezclar.
- Añadir 144 sL de 2,5 M CaCl2 y mezclar. Añadir 1,92 ml de HEPES Tampona tampona (HBS) (1,5 mM Na2HPO 4,140 mM NaCl, 50 mM HEPES) a la solución de ADN y mezclar inmediatamente mediante vórtice.
- Incubar a temperatura ambiente (RT) durante exactamente 60 s. Transfiera la solución a medios de cultivo celular fresco de 28 ml y mezcle.
- Sustituya el medio de los matraces por un medio de cultivo celular que contenga una mezcla de transfección.
- Tres días después de la transfección, cosechar las células vertiendo el medio de los matorrales en un recipiente desechable para residuos y añadir 5 ml de tampón de cosecha (EDTA añadido a la salina tamponada de fosfato, ver Tabla de materiales,DPBS a una concentración final de 5 mM) a una concentración de 5 mM) en cada matraz para permitir el desprendimiento celular.
- Vierta la solución celular en un tubo centrífugo de 50 ml. Agregue otros 4 ml de DPBS a cada matraz para enjuagar las celdas restantes y la piscina con la primera solución celular. Centrifugar las células cosechadas a 1.000 x g durante 5 min a 4oC.
- Después de la centrifugación, retire el sobrenadante y disuelva los gránulos en 15 ml de tampón de lisis (50 mM Tris-HCl pH 8.5, 150 mM NaCl, 1 mM MgCl2)por vórtice.
- Congeleen el baño co2-hielo/etanol durante 15 minutos y guárdelos en un congelador de -20 oC. Descongelar el silbido celular cosechado en un baño de agua a 37 oC antes de su uso.
- Purificación de vectores virales AAV5
- Realizar purificación AAV por Iodixanol Gradient Ultracentrifugation13 y utilizar tubos de sellado ultracentrífugos con centrifugación a 350.000 x g durante 1 h y 45 min en RT.
- Utilice una jeringa de 10 ml con una aguja de 18G e inserte aproximadamente 2 mm por debajo del borde de fase del 40/60% con el bisel hacia arriba para extraer la fase que contiene AAV. Asegúrese de detenerse antes de llegar a la banda de proteína después de que se haya extraído 5-6 ml.
- Almacene los extractos de gradiente en botellas de vidrio autoclavedos a 4 oC. Evite almacenar tiempos más tiempos que durante la noche.
- Diluir el extracto de gradiente de Iodixanol 3 veces mediante el pipeteo lentamente en 12 ml de almacenamiento de iodixanol Elution (IE) (20 mM Tris-HCl pH 8.0, 15 mM NaCl) mientras se arremolina.
- Purificar y concentrar el gradiente de Iodixanol diluido a través de un filtro de intercambio de aniones. Empuje lentamente a través de una velocidad no más rápida que 1 gota / s. Empuje 3 ml de tampón IE lentamente a través del filtro para lavarlo.
- Elute en una unidad de filtro centrífugo con 1-2 ml de tampón de elución (20 mM Tris-HCl pH 8.0;250 mM NaCl). Agregue DPBS al dispositivo a un volumen final de 4 ml. Centrífuga a 2.000 x g a RT hasta que se deja menos de 0,5 ml en el filtro. Retire el líquido de la parte inferior del tubo, rellene con 4 ml de DPBS y vuelva a centrifugar. Repita este paso dos veces más. Asegúrese de que el volumen del vector concentrado en el filtro es de unos 200 s después del último paso de centrifugación.
- Retire el vector concentrado de 200 l utilizando una pipeta y empuje el concentrado a través de un filtro de 0,22 mm para esterilizarlo. Aliquot 200-Linto un vial de vidrio de 9 mm con inserto entrelazado (Tablade Materiales).
NOTA: Las existencias de vectores AAV5 se pueden almacenar en congeladores de -80 oC para un almacenamiento prolongado, o a 4 oC si se utilizan en un plazo de 2 semanas.
- Determinación del vector viral AAV5
- Determinar el titer AAV5 utilizando la cadena de polimerasa cuantitativa estándar Reaction (qPCR) con imprimaciones para la secuencia inverted Terminal Repeat (ITR)y una sonda de 5 'FAM / 3'BHQ1 para la secuencia ITR (Tablade materiales). Utilice una curva estándar lograda con cantidades conocidas de ITR que contengan plásmido. Cada vector AAV debe tener un rango de 1E+14 – 1E+15 copias del genoma por mililitro si se utiliza la secuencia ITR para la determinación del títer.
NOTA: Una producción exitosa de virus AAV5 da existencias con valoradores en el rango mínimo de 3E+13 – 7E+13 Unidades/mL.
- Determinar el titer AAV5 utilizando la cadena de polimerasa cuantitativa estándar Reaction (qPCR) con imprimaciones para la secuencia inverted Terminal Repeat (ITR)y una sonda de 5 'FAM / 3'BHQ1 para la secuencia ITR (Tablade materiales). Utilice una curva estándar lograda con cantidades conocidas de ITR que contengan plásmido. Cada vector AAV debe tener un rango de 1E+14 – 1E+15 copias del genoma por mililitro si se utiliza la secuencia ITR para la determinación del títer.
2. Inyección de factores de reprogramación en el cerebro
- Configuración de animales, colocación estereotaxia y perforación
Nota:Este protocolo se centra en el uso de Ascl1, Lmx1a y Nurr1 (ALN) para la reprogramación de la glia NG2 en interneuronas. En nuestra experiencia, las interneuronas de un fenotipo similar se pueden obtener utilizando otras combinaciones de factores11.- Antes de la cirugía, prepare la mezcla viral que contiene los vectores Cba-FLEX-Ascl1, Cba-FLEX-Lmx1a, Cba-FLEX-Nurr1 y el vector reportero Syn-FLEX-GFP. Añadir cada una de las existencias preparadas en la sección 1 a la mezcla final, de modo que la solución viral final tenga el 5% de cada uno de los factores de reprogramación (Ascl1, Lmx1a y Nurr1) y el 10% de la construcción del reportero (5% A, 5% L, 5% N y 10% GFP).
NOTA: Las mezclas vectoriales AAV5 se pueden almacenar a 4 oC y conservarse para su uso futuro in vivo. - Anestesiar el ratón usando 2% de isoflurano en una mezcla de aire y óxido nitroso (N2O) en una proporción de 4:1. Controle la respiración del animal observando los movimientos del diafragma. Durante la cirugía, mantenga la anestesia con 1-1,5 % de isoflurano.
NOTA: El modelo de ratón descrito por la presente consiste en una cepa de ratón que expresa específicamente Cre en NG2 glia. La reprogramación in vivo se puede lograr utilizando diferentes cepas de ratón que expresan Cre en otras poblaciones de células gliales (por ejemplo, astrocitos14). - Una vez que el animal está completamente anestesiado (por ejemplo, relajación muscular completa y sin respuesta a un pellizco en la almohadilla del pie), afeitar el área alrededor del sitio de la incisión y llevar al animal al marco estereotaxico.
- Para mantener la temperatura corporal del animal durante la cirugía, coloque una almohadilla de calentamiento en la base del marco estereotaxico. Coloque el ratón sobre una toalla de papel limpia y seca.
- Coloque cuidadosamente la cabeza del ratón en las barras de los oídos. Si se coloca correctamente, no se debe observar ningún movimiento lateral de la cabeza. Ajuste la barra de la oreja izquierda a 4 mm, antes de comenzar a colocar el ratón en el marco estereotaxico.
- Fije la barra dental en su lugar, y luego apriete la barra nasal. Asegúrese de que la cabeza no se mueva en ninguna dimensión y apunte hacia adelante (la línea media es perpendicular al plano de las barras de orejas). Aplique pomada oftálmica para la protección ocular.
- Antes del inicio de la cirugía, administrar la analgesia adecuada (porejemplo, 0,05 mg/kg de buprenorfina, subcutánea).
- Limpie el área de la incisión con una gasa de algodón o una toallita empapada con 70% EtOH, sin acercarse a la zona ocular.
NOTA: Para la inyección viral intracerebral, se utiliza una jeringa de 5 o 10 ml adaptada con un capilar de vidrio tirado. Los capilares de vidrio se extraen con un tirador de micropipetas, lo que resulta en un capilar con una punta muy fina, lo que minimizará la invasividad del procedimiento. Para adaptar el capilar en la jeringa, utilice un trozo de tubo de goma sobre la conexión entre la aguja de la jeringa y el capilar de vidrio y derrita con una fuente de calor (por ejemplo, un encendedor). Una conexión estrecha entre las dos piezas se asegurará de que no haya fugas de líquido durante la inyección. Antes de comenzar la cirugía, pruebe esto llenando la jeringa con salina y expulsando el líquido de la jeringa. - Haga una incisión de aproximadamente 0,5-0,8 cm a lo largo de la línea media de la cabeza. Corte a través de capas cutáneas y subcutáneas, con un bisturí.
- Ensanchar las aletas de la piel a cada lado de la incisión. Limpie la incisión de cualquier sangre y raspa las capas subcutáneas con un brote de algodón.
- Mueva el brazo M/L-D/V del marco estereotaxico en su lugar (por encima del animal) y fíjelo.
NOTA: El marco estereotaxico permite ajustar la jeringa a lo largo del eje Anterior/Posterior (Eje A/P, Y), Medial/Lateral (M/L, eje X) y Dorsal/Ventral (Eje D/V, Z). - Mueva la jeringa a lo largo de los diferentes ejes del marco estereotaxico, para llevar la punta del capilar de vidrio justo por encima de bregma (el punto de unión donde se encuentran las diferentes placas de cráneo).
- Asegúrese de que la punta capilar esté perfectamente recta tanto en los planos A/P como en los planos M/L. En caso de una bregma ambigua, tome el promedio de las suturas laterales y de línea media.
- Cuando la punta del capilar se coloca correctamente por encima de bregma, restablezca los valores M/L y A/P a 0,0 en el contador de coordenadas digitales.
- Para asegurarse de que la cabeza del animal está en una posición perfectamente plana, utilice el contador de coordenadas digitales para medir el valor de la coordenada D/V, cuando el brazo A/P esté en +2,0 y -2,0 (M/L a 0,0), así como cuando el brazo M/L esté en +2,0 y -2,0 (A/P a 0,0). Ajuste la altura de la barra dental y las barras de los oídos en consecuencia.
- Mueva la jeringa a las coordenadas deseadas para la inyección de vectores virales en el estriado (A/P a +1.0; M/L -2.0, en relación con bregma).
- Levante ligeramente la jeringa y, mirando el lugar de la inyección a través del microscopio, taladre un agujero utilizando un taladro dental en las coordenadas de la inyección. Comience a perforar en el sitio, trabajando de manera circular y suave.
NOTA: No ponga demasiada presión hacia abajo, ya que la broca debe ser lo suficientemente afilada como para atravesar el hueso sin fuerza adicional. Evite la perforación larga y sostenida, ya que esto crea calor. - Al final de la perforación, compruebe que la dura mater permanezca intacta y expuesta para inyección.
- Antes de la cirugía, prepare la mezcla viral que contiene los vectores Cba-FLEX-Ascl1, Cba-FLEX-Lmx1a, Cba-FLEX-Nurr1 y el vector reportero Syn-FLEX-GFP. Añadir cada una de las existencias preparadas en la sección 1 a la mezcla final, de modo que la solución viral final tenga el 5% de cada uno de los factores de reprogramación (Ascl1, Lmx1a y Nurr1) y el 10% de la construcción del reportero (5% A, 5% L, 5% N y 10% GFP).
- Preparación de la configuración de la jeringa
- Coloque un trozo de gasa de algodón sobre la incisión abierta y enjuague la jeringa con solución salina.
- Después del lavado, tome una burbuja de aire de 1-2 l, seguida de 1 l de solución que contenga los vectores virales, evitando cualquier burbuja involuntaria. Asegúrese de que la solución viral se puede visualizar fácilmente debajo de la burbuja de aire, mientras se inyecta.
- Inyección viral
- Retire la gasa de algodón del sitio de la incisión y baje la jeringa con el brazo D/V del marco estereotaxico. A medida que se acerca la superficie del cráneo, mire cuidadosamente a través del microscopio y mida el nivel de D/V de dura mater — debe abultarligeraligeramente bajo una presión suave.
- Mientras toca el dura mater con la punta del capilar, ajuste la coordenada D/V a 0.0.
- Baje la jeringa, progresando lentamente a la profundidad deseada (D/V -2.7, en relación con la dura mater). Asegúrese de que la trayectoria esté libre de fragmentos óseos, de modo que no se observe ninguna flexión de la aguja/capilar.
NOTA: Las coordenadas presentadas se refieren a una inyección de los factores de reprogramación en el estriado de ratones NG2-Cre. Se pueden utilizar coordenadas correspondientes a otras regiones cerebrales. Pruebe siempre las coordenadas para inyección primero utilizando un tinte (por ejemplo, Trypan Blue), inyección de perlas de color o un vector viral de reportero antes de la inyección de factores de reprogramación en el cerebro de una nueva cepa de ratón. Si se inyecta Trypan Blue, los animales necesitan ser sacrificados inmediatamente después de la cirugía y el cerebro diseccionado. Los cerebros frescos se pueden cortar usando un microtome, mientras que congelado, y el sitio de la inyección determinado por la visualización de la posición del tinte en el cerebro. Si las coordenadas de prueba utilizando cuentas de colores, es posible determinar el sitio de la inyección en cerebros perfundidos y cortados una semana después de la inyección. Alternativamente, se puede utilizar una inyección con un vector viral que lleva un gen reportero, y el sitio de inyección determinado en cerebros cortados perfundidos. - Inyectar 1 l de la solución viral a una velocidad de 0,4 l/min. Cuando se inyecte todo el volumen, espere un período de difusión de 2 minutos antes de la retirada de la jeringa.
- Después de la difusión, retraiga lentamente la jeringa hasta que la punta del capilar esté completamente fuera del cerebro.
- Coloque un trozo de gasa de algodón sobre la herida y enjuague la jeringa con solución salina.
- Saque el brazo M/L-D/V del marco estereotaxico fuera del área de trabajo.
- Procedimientos de cierre y postoperatorio de heridas
- Suturar cuidadosamente la incisión usando hilo de sutura.
NOTA: Todos los hilos de sutura utilizados en animales inyectados deben desecharse en una taza/vial que contenga solución de detergente antiviral. La misma solución se utiliza para limpiar todas las superficies que rodean el área de cirugía que han estado en contacto con materiales quirúrgicos. Todas las herramientas quirúrgicas se lavan a fondo y se autoclaven al final de cada día de cirugía. - Retire el animal del marco estereotaxico y colóquelo en una estación postoperatoria, que incluye una jaula limpia y calentada, acceso a alimentos y agua, y donde el animal permanece hasta que esté completamente despierto. Durante este período, monitoree al animal de cerca hasta que se recupere la conciencia.
- No abarque animales operados con animales no operados hasta que los primeros se hayan recuperado completamente de la cirugía.
- Supervisar los animales operados todos los días. Dependiendo de las suturas utilizadas, asegúrese de que se retiran si es necesario. A todos los animales se les administró Bupenorphinum (Temgesic a 1ml/kg) subcutáneo como cuidado postoperatorio.
NOTA: Si opera con la misma jeringa en dos días consecutivos, lave con agua, seguida de etanol 70% y agua de nuevo. Deje la jeringa llena de agua durante la noche, para permitir la disolución de cualquier posible residuo.
- Suturar cuidadosamente la incisión usando hilo de sutura.
3. Grabaciones electrofisiológicas
- Preparación de rebanadas de tejido para electrofisiología
PRECAUCION: Una preparación de tejido bien ejecutada es necesaria para lograr buenas grabaciones electrofisiológicas. Prepare la habitación cuidadosamente y coloque las herramientas para la perfusión y la disección sobre hielo.- Preparar la solución de Krebs para perfusión, disección y corte en frío y oxigenado (95% O2 y 5% CO2) para perfusión, disección y corte (preparar esto el mismo día a partir del stock 10x diluyendo la solución en stock en agua ultrapura y añadiendo NaHCO3 y glucosa). Los componentes de la solución Krebs en mM (después de la dilución a 1x) son: 126 NaCl, 2.5 KCl, 1.2 NaH2PO4 H2O, 1,3 MgCl2x 6H2O, y 2,4 CaCl26H2O, 22 NaHCO3, 10 glucosa. Ajuste el pH de la solución a 7.4.
- Realice una calibración (Vibracheck) para el vibratome con una nueva cuchilla de afeitar.
- Encienda el bloque de enfriamiento del vibratoma (o llene la cámara circundante con hielo), llene la cámara de corte con solución Krebs para oxigenación con 95 % O2 y 5 % CO2 al menos 30 minutos antes de su uso.
- Ponga un plato de Petri en hielo y llénelo con solución de Krebs oxigenada. Coloque la hoja, las tijeras y la placa de montaje sobre hielo.
PRECAUCION: Lleve la jaula que contiene el ratón a la habitación al menos 1 h antes de iniciar el procedimiento de aclimatación. El estrés tiene un efecto negativo sobre la condición de las secciones del tejido cerebral. - Anestesia terminalmente el ratón inyectando una sobredosis de Pentobarbital y deja que el animal se duerma. Cuando el reflejo de parpadeo está fuera y el animal no responde a los estímulos del dolor, perfumar transcardialmente el animal con solución Krebs helada durante 2-3 min (a una velocidad de 10-20 mL/min).
- Rápidamente, pero con cuidado, diseccionar el cerebro y ponerlo boca abajo en el Petri-plato que se coloca en el hielo (que contiene la solución Krebs).
NOTA: Con el fin de comparar la maduración funcional de las neuronas reprogramadas, sacrificar los animales para las grabaciones en diferentes puntos de tiempo de inyección post-viral. Evaluar las propiedades de disparo de distintos subtipos de neuronas en función de la literatura existente15 . - Haga un corte coronal a lo largo del cerebelo medio y pegue este lado en la placa de montaje (también helada) para el corte de vibratome.
- Sumerja el cerebro pegado cuidadosamente en la cámara tampón en el vibratomo.
PRECAUCION: Tenga cuidado de no tocar la cuchilla de afeitar para su propia seguridad, y para que la hoja permanezca calibrada. - Corte desde la parte más rostral del cerebro hasta el nivel estriado a alta velocidad. A continuación, corte el estriado coronalmente a 275 mm a velocidad lenta (0,10 mm/s).
- Después de cada corte, retire cuidadosamente el lado estriado no inyectado (por ejemplo, con una aguja doblada) y transfiera el lado inyectado a otro vial en un baño de agua, que contenga una red inferior (Krebs oxigenados en RT) colocados en un baño de agua. Mantenga esto en RT hasta que se corten todas las secciones.
- Aumentar lentamente la temperatura del baño de agua a 37 oC y dejarlo durante 30 minutos. A continuación, apague el calentador y deje enfriar hasta RT.
NOTA: En este punto, puede pausar hasta que comience a grabar. Las secciones duran 4-6 h.
- Grabaciones de abrazaderas de parche de celda completa
- Transfiera la primera sección de tejido a la cámara de registro sumergida en un flujo continuo de solución krebs. Monte la sección con pesos ligeros y sumerja el objetivo.
- Identificar la región estrialista en el microscopio y buscar neuronas gFP positivas (reprogramadas). Seleccione una neurona extensa en morfología y no cubierta por haces de fibra o vasos sanguíneos.
- Preparar pipetas de vidrio borosilicato (3-7 M) para parchear y llenar con la siguiente solución intracelular (en mM): 122.5 gluconato de potasio, 12.5 KCl, 0.2 EGTA, 10 HEPES, 2 MgATP, 0.3 Na3GTP y 8 NaCl, ajustado a pH 7.3 con KOH.
- Para el llenado de biocittina, añadir 1 mg de sal de biocictina a 1 ml de solución interna y vórtice.
PRECAUCION: Asegúrese de filtrar la solución interna con biocitina, ya que esto puede obstruir el electrodo. - Fije la pipeta de vidrio al electrodo de grabación y sumerja en la solución. Compruebe la resistencia del electrodo. Luego acércate lentamente a la célula con la pipeta, manteniendo una ligera presión positiva en el electrodo para evitar taponar la punta.
PRECAUCION: Tenga cuidado de realizar un seguimiento de su célula mientras desciende el electrodo y no blanquear la fluorescencia en la célula (es decir, apague la lámpara de fluorescencia cuando no la necesite). - Enjuague el tejido circundante cuidadosamente con la presión positiva del electrodo y acérquese a la célula con el electrodo. Localice el electrodo justo encima de la célula y descienda hasta que el electrodo toque la membrana. Hacer un sello Giga-o entre el electrodo y la membrana celular, y con pulsos de presión negativa, romper la membrana para crear un parche de células enteras.
PRECAUCION: Las neuronas reprogramadas son sensibles. Tenga cuidado al aplicar parches y no poner demasiada presión negativa al alcanzar un sello Giga-o al abrir la membrana celular. Además, parchear a los animales que son más viejos requiere práctica y paciencia ya que su tejido conectivo es más grueso y es más difícil visualizar las neuronas. - Compruebe los potenciales de membrana de reposo inmediatamente después de romper en el modo de abrazadera de corriente y anote para su análisis.
- En la abrazadera de corriente, mantenga la celda entre -60 mV a -80 mV e inyecte corrientes de 500 ms de -20 pA a +90 pA, con incrementos de 10 pA para inducir potenciales de acción.
- Transfiera a la abrazadera de voltaje y mida las corrientes de potasio rectificadoras de sodio hacia adentro y retardadas a pasos de despolarizantes de 10 mV.
NOTA: Las grabaciones de abrazaderas de voltaje se evalúan mejor utilizando una solución interna diferente, compuesta de productos químicos que sujetan la membrana de manera más eficiente. Sin embargo, para las neuronas reprogramadas, el número de células que se pueden registrar de es pocos y el número de secciones de tejido con estas neuronas es limitado. Por lo tanto, es una ventaja registrar tanto en la abrazadera de corriente como en la abrazadera de voltaje desde la misma celda. - En el modo de abrazadera de voltaje, registre la actividad postsináptica espontánea a -70 mV. Distinguir los eventos postsinápticos inhibitorios en un potencial de membrana de 0 mV de eventos excitatorios a -70 mV.
- Después de alcanzar una línea de base estable, añadir Picrotoxina, el antagonista del receptor GABA A, a la solución Krebs que fluye en la cámara de registro, para extraer eventos excitatorios (añadir una solución de stock de picrotoxina a una concentración final de 50 mM en separado jeringa tampón que está conectada a la perfusión de la cámara. Conecte la salida del búfer de la cámara a una bolsa de vacío para su eliminación segura).
- Después de 20 minutos, añadir CNQX (20 mM), un antagonista AMPA, a la solución Krebs que fluye en la cámara de registro, para bloquear los eventos inhibitorios (utilizando el mismo procedimiento que para Picrotoxin). Dejar actuar otros 20 minutos y luego lavar con la solución Krebs. Retire la sección de tejido de la cámara.
- Después de terminar las grabaciones, realice un análisis fuera de línea de las corrientes postsinápticas excitatorias espontáneas (EPSC) y las corrientes postsinápticas inhibidoras (IPSC) utilizando una detección de eventos de umbral (>5 pA) en el programa de análisis.
- Durante toda la grabación, la célula se llenará lentamente con la solución interna que contiene biocitina. Mantenga el parche durante al menos 20 minutos antes de retirar lentamente el electrodo para lograr el llenado completo de la célula.
PRECAUCION: Tenga cuidado de no tener ninguna presión positiva en el electrodo que podría destruir el análisis morfológico consecutivo de las células después. - Para la visualización de biocitos, coloque la sección tisular en un 4 % de paraformaldehído (PFA) durante la noche a 4oC. Enjuague la sección en un tampón de fosfato potásico de 0,02 M (KPBS) con un 0,1 % de Titón. A continuación, mancha para la estreptavidina- Cy3 (1: 600 en KPBS-T, 2 h), para la identificación de la célula llena de biocitos y la visualización morfológica de la neurona reprogramada.
NOTA: El relleno de biocitetina se puede utilizar para el análisis morfológico y las ilustraciones de las neuronas parcheadas.
4. Inmunohistoquímica, Estereología y Cuantificación
NOTA: Dedicar un grupo específico de ratones para la inmunohistoquímica, ya que las secciones de tejido utilizadas para la electrofisiología no son óptimas para la inmunohistoquímica.
- Anestetiza ratones con una inyección i.p. de una sobredosis de pentobarbital y monta el animal para perfusión.
- Perfumar transcardialmente los ratones, primero con una solución salina para eliminar la sangre, y luego con hielo-frío 4% PFA.
- Diseccionar los cerebros y post-fijar durante al menos 12 h en 4% PFA.
- Poner los cerebros en 25% solución de sacarosa (para la crioprotección) durante aproximadamente 12 h.
NOTA: Los cerebros están listos para ser cortados cuando se hunden en la solución de sacarosa del 25%, lo que indica que la sacarosa ha penetrado en todo el cerebro del ratón. - Hacer un corte coronal a través del cerebelo, utilizar la parte plana, más caudal del cerebro para colocar el cerebro en un microtome y fijarlo en su lugar utilizando el compuesto de temperatura de corte óptima (OCT).
- Cortar el cerebro en rodajas coronales con un espesor de 35 mm y dividir el cerebro en series consecutivas colocadas en viales o pozos que contienen 0.02 M KPBS.
- Procesar las secciones de inmunohistoquímica utilizando anticuerpos contra GFP y marcadores interneuronas como GAD65/67, PV, Chat (colina acetiltransferasa) y NPY (Neuropéptido Y), según los protocolos estándar16, 17.
NOTA: Las rodajas cerebrales se pueden mantener a 4 oC o -20 oC en solución anticongelante durante largos períodos. - Una vez finalizada la tinción, monte las secciones en una corredera de vidrio y cúbralas con un cubreobjetos de vidrio, utilizando una solución de montaje para fijarlo en su lugar. Deje que los portaobjetos se sequen toda la noche.
NOTA: Para nuestros estudios, los cerebros enteros se cortan en 1:8 serie (es decir, cada vial que contiene secciones tendrá un octavo del cerebro del ratón11). - Analice las secciones utilizando una fluorescencia invertida y/o un microscopio confocal.
PRECAUCION: La microscopía fluorescente es útil para obtener una visión general de los resultados y capturar imágenes para calcular la eficiencia de la reprogramación. Para un análisis más detallado de la identidad fenotípica mediante la observación de células de doble positivo, se deben utilizar imágenes confocales. - Para la cuantificación de diferentes marcadores expresados en GFP+ neuronas en el estriado, contar el número de células doble-positivas en relación con el número total de células GFP+ (es decir, las neuronas reprogramadas), en al menos dos campos de cada sección estriado.
- Para determinar el número total extrapolado aproximado de neuronas reprogramadas por cerebro, cuente las neuronas GFP+ presentes en el estriado de una de las series cerebrales cortadas anteriormente, y luego multiplique por el número total de series.
NOTA: Diferentes métodos de cuantificación se pueden utilizar para expresar la eficiencia de reprogramación, el número de neuronas reprogramadas por animal y el porcentaje de neuronas que expresan ciertos marcadores fenotípicos. Para obtener más información, consulte la publicación anterior11. - Analice las diferencias entre las condiciones que utilizan Graph Pad Prism o similar.
NOTA: Para el cálculo de la eficiencia, inyectamos ratones NG2-Cre con los vectores de conversión ALN dependientes de CRE y el reportero de GFP. Para estimar la eficiencia de conversión, también inyectamos animales con un GFP dependiente de Cre bajo el omnipresente promotor de cba, haciendo que todas las células objetivo GFP+. Usando esta comparación, estimamos que 66.81% - 38.38% de las células dirigidas convertidas en neuronas. Para la validación de la expresión de cada uno de los genes, se puede realizar una doble tinción inmunofluorescente complementaria para GFP/Ascl1, GFP/Lmx1a y GFP/Nurr1. Además, la secuenciación del genoma, como la secuenciación de ARN de una sola célula, puede revelar la presencia de cada uno de los genes en las células reprogramadas.
Resultados
La inyección de vectores AAV se utiliza para reprogramar con éxito las células glia ng2 residentes en neuronas en el estriado del ratón NG2-Cre (Figura1A). Para apuntar específicamente a NG2 glia, los vectores FLEX con genes de reprogramación/reportero, se insertan en una dirección antisentido y están flanqueados por dos pares de sitios loxP antiparalelos y heterotípicos (Figura1B). Cada uno de los tres genes de reprogramación (Ascl1, Lmx1a y Nurr1) se coloca bajo el control del omnipresente promotor de cba en vectores individuales. Con el fin de asegurarse de que la expresión GFP se limita a las neuronas reprogramadas que se originan en una célula de expresión Cre, GFP se coloca bajo el control del promotor de la sinapsina específica de la neurona, (también en un vector FLEX).
El uso de la combinación de reprogramación y construcciones de reportero permite la generación de neuronas gFP positivas en el estriado del ratón (Figura1C,C'). El uso de la construcción del reportero sin la presencia de genes de reprogramación no produce neuronas positivas en gFP (Figura1D).
Las neuronas reprogramadas que están llenas de biocitetina son visibles después de la inmuno-tinción post mortem (Figura2A). Si la conversión es exitosa, debe haber una morfología neuronal extensa. Los registros electrofisiológicos de las neuronas reprogramadas muestran la presencia de conexiones funcionales postsinápticas con medidas de actividad espontáneas (Figura 2B,C). Esto se puede bloquear con bloqueador GABAérgico o glutamatérgico ionotrópico (Picrotoxina o CNQX), lo que sugiere entrada sináptica excitatoria e inhibitoria a las neuronas reprogramadas. La aparición de actividad espontánea aumenta con el tiempo posterior a la inyección viral (Figura2D), lo que indica una maduración gradual.
Los potenciales de acción inducidos por la corriente están presentes en las neuronas funcionales. Las posibilidades de acción aumentan en número con el tiempo después de la conversión (Figura2E). Esto indica además maduración en la función neuronal. En una neurona inmadura, la corriente inducirá ninguno o muy pocos potenciales de acción (Figura2F).
Los patrones de disparo de una neurona son específicos del tipo celular, ya que depende de factores como la morfología de las células y la expresión de canal15. Los patrones registrados en las neuronas reprogramadas in vivo pueden distinguirse en grupos y compararse con los de los subtipos neuronales endógenos, por ejemplo, interneuronas de pico rápido (Tipo de célula B, Figura 3B)u otros tipos de células (Figura3A,C,D ). Las diferencias electrofisiológicas observadas pueden confirmarse por la presencia de marcadores de subtipo específicos y co-expresión con GFP (Figura3E-H). En conjunto, estos datos indican que las neuronas reprogramadas presentes en el estriado tienen propiedades de diferentes tipos de interneuronas, como parvalbumina-, ChAt- y NPY- expresando interneuronas, así como la identidad de neurona spinospino media estriada (DARPP32+) ( Figura 3E -H).
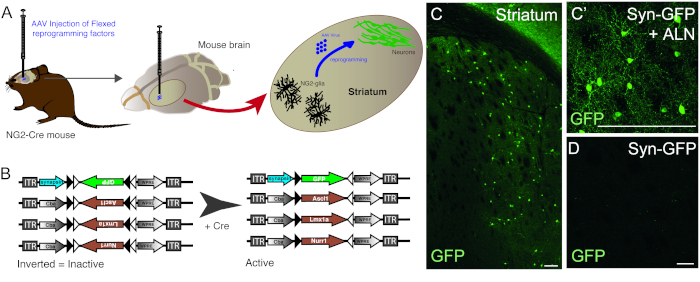
Figura 1: Reprogramación in vivo de la glia NG2 residente en neuronas. (A) Representación esquemática de la reprogramación in vivo mediada por el virus AAV de la glia estriada NG2. (B) Representación esquemática de construcciones AAV5 FLEX utilizadas para la reprogramación in vivo, en la que la expresión génica está regulada por la expresión Cre en las células objetivo. (C y C') Neuronas reprogramadas in vivo, resultantes de la inyección de Syn-GFP + ALN en el Estriado. (D) Ausencia de neuronas reprogramadas cuando no se añaden factores de reprogramación al cóctel viral, y sólo se inyecta la construcción del reportero in vivo. Barras de escala a 100 mm (C),25 mm (C'), 25 mm (D). Haga clic aquí para ver una versión más grande de esta figura.
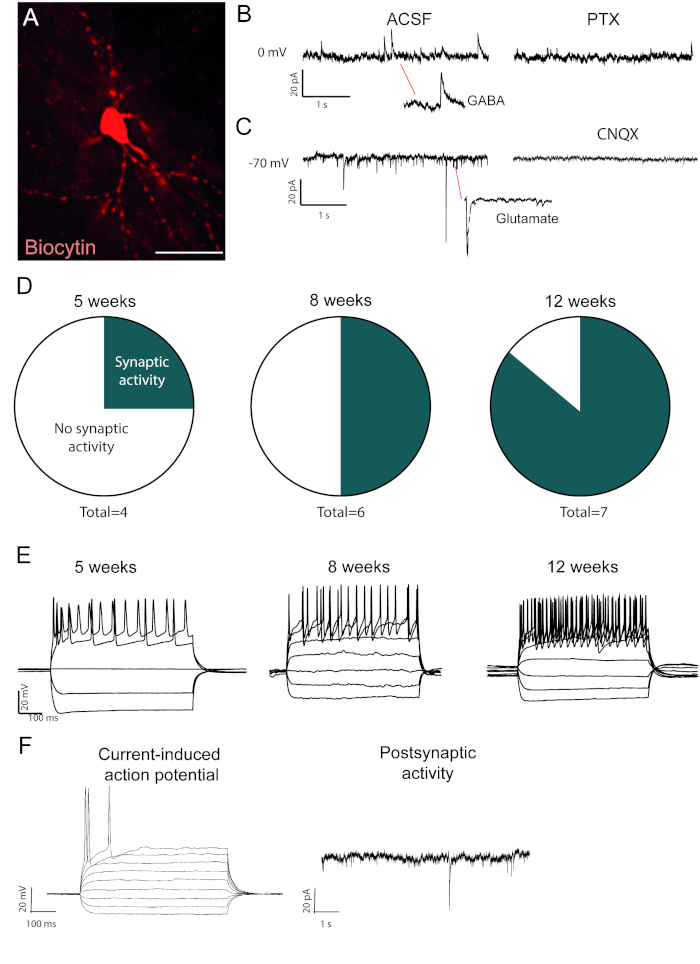
Figura 2: Las neuronas reprogramadas in vivo son funcionales y muestran maduración con el tiempo. (A) La neurona reprogramada llena de biocitotina, muestra morfología neuronal madura, incluyendo espinas dendríticas. Rastros muestra (B) actividad inhibitoria (GABAérgica) que está bloqueada con picrotoxina, un antagonista del receptor GABAA y (C) actividad excitatoria que está bloqueada con CNQX, un antagonista del receptor AMPA. (D) El número de neuronas con actividad postsináptica aumenta con el tiempo. (E) Las neuronas parcheadas muestran disparo repetitivo ya a las 5 semanas después de la inyección (con.p.i.) y continúan mostrando que a las 8 y 12 p.p.i. (F) El potencial de acción inducido por corriente y la actividad postsináptica de una neurona inmadura, mostrando pocos sinápticos eventos y pocos potenciales de acción en comparación con B y D. Barra de escala 25 mm Haga clic aquí para ver una versión más grande de esta figura.
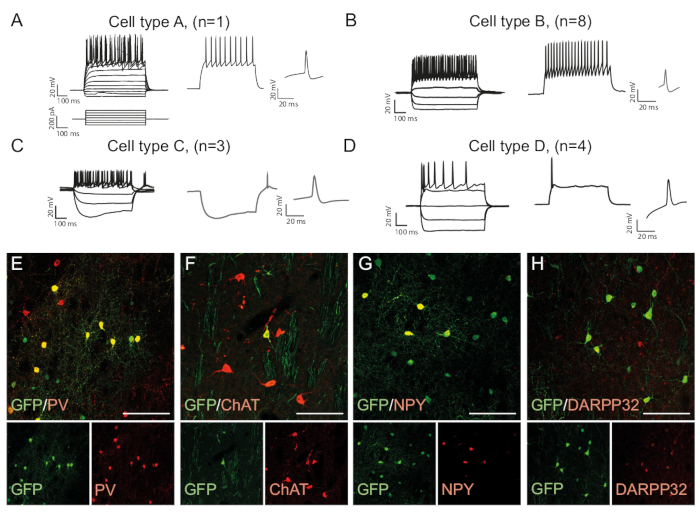
Figura 3: Las neuronas reprogramadas in vivo muestran propiedades inmunohistoquímicas y electrofisiológicas de las interneuronas estriales. (A-D) Los patrones de disparo de las neuronas reprogramadas in vivo pueden ser de tipos distintos: (A) Tipo A es similar a la neurona espinosa media endógena (DARPP32+); (B) similar a las interneuronas de pico rápido (PV+); (C) similar a las neuronas de pico de umbral bajo con hundimiento prominente (NPY+); (D) neuronas disparando con gran post-hiperpolarización (Chat+). (E-H) Imágenes confocales que muestran la colocalización de la PFP y los marcadores interneuronas PV (E), ChAT (F), NPY (G) y el marcador de neurona de proyección DARPP32 (H). Todas las barras de escala de 50 mm. Haga clic aquí para ver una versión más grande de esta figura.
Discusión
La reprogramación directa in vivo se puede lograr utilizando vectores AAV FLEX en cepas de ratón C-expressing. Es importante tener en cuenta que se han observado diferencias entre las cepas del ratón con respecto a la eficacia de reprogramación. Para la reprogramación in vivo en el estriado, la línea de ratón NG2-Cre ha demostrado ser la más eficiente en comparación con otras cepas. Antes de comenzar a utilizar una nueva cepa animal, es importante comprobar las pautas del proveedor de ratón con respecto a la expresión de Cre a lo largo del tiempo, ya que la edad de los animales a menudo afecta a la especificidad de esta expresión proteica. En nuestros estudios, los animales mayores de 12 semanas no se utilizaron para la reprogramación in vivo, ya que existía un riesgo para la expresión de Cre en células distintas de NG2 glia. Se recomienda la presencia y el seguimiento constantes de los animales de control inyectados únicamente con la construcción Synapsin-FLEX-GFP. Esto permite el seguimiento de los animales en busca de células positivas en la PTF que no deben estar presentes si no se utilizan genes de reprogramación (ALN).
Para identificar las neuronas recién reprogramadas, un método de identificación específico de la neurona como el descrito en este protocolo es de suma importancia. Esto permite una adecuada identificación y distinción de las neuronas reprogramadas de las células circundantes endógenas que es de particular relevancia cuando la reprogramación en una región homotópica.
También es importante orientar la estructura correcta para la inyección viral. Por lo tanto, la cirugía estereotaxia para la inyección viral es importante y necesita ser abordada con precisión, especialmente cuando se dirige a estructuras más pequeñas del cerebro.
Hemos demostrado anteriormente11 que no es fiable predecir el resultado de la reprogramación in vivo basada en experimentos de reprogramación in vitro utilizando los mismos factores de reprogramación. Por lo tanto, todos los factores de interés deben probarse in vivo. En nuestras manos, muchas combinaciones de factores diferentes dan el mismo subtipo de neuronas (es decir, interneuronas11 in vivo) a pesar del hecho de que estos genes han estado implicados en el desarrollo de otras neuronas.
La abrazadera de parche de células enteras para neuronas reprogramadas es una técnica delicada y el procesamiento de tejidos es importante para un buen resultado. La perfusión con solución Krebs helada mejora la calidad del tejido. Además, las neuronas parcheadas necesitan ser tratadas cuidadosamente. Incluso si la maduración y la identidad fenotípica de las neuronas reprogramadas se pueden evaluar un poco utilizando una abrazadera de parche de células enteras, estas células no son totalmente comparables a sus contrapartes endógenas. Se deben utilizar tipos adicionales de análisis, como la secuenciación del genoma (por ejemplo, la secuenciación de ARN) para confirmar aún más la identidad celular reprogramada.
La técnica descrita en este documento se podría considerar para el desarrollo de futuras terapias donde se necesita reemplazo neuronal en el cerebro. Aunque la reprogramación in vivo todavía está en sus primeras etapas y la traducción a los seres humanos aún no está prevista, esta técnica podría proporcionar un método para evaluar la función génica exógena en el cerebro y estudiar la maduración celular in vivo.
Divulgaciones
Los autores no tienen nada que revelar.
Agradecimientos
Marcella Birtele ha sido financiada por el Programa Horizonte 2020 de la Unión Europea (H2020-MSCA-ITN-2015) en virtud de las redes de formación innovadora Marie Sk-odowska-Curie y el Acuerdo de Subvención No 676408. Daniella Rylander Ottosson ha sido financiada por el Consejo Sueco de Investigación (2017-01234).
Materiales
| Name | Company | Catalog Number | Comments |
| REAGENTS FOR AAV5 CLONING AND VIRAL VECTOR PREPARATION | |||
| pAAV-CA-FLEX | AddGene | 38042 | |
| Ascl1 | AddGene | 67291 | NM_008553.4 |
| Lmx1a | AddGene | 33013 | NM_0033652.5 |
| Nurr1 | AddGene | 35000 | NM_013613.2 |
| GFP-syn | AddGene | 30456 | |
| LoxP (FLEX) sequence 1 | GATCTccataacttcgtataaagtatcctatac gaagttatatcaaaataggaagaccaatgcttc accatcgacccgaattgccaagcatcaccatcg acccataacttcgtataatgtatgctatacgaa gttatactagtcccgggaaggcgaagacgcgga agaggctctaga | ||
| LoxP (FLEX) sequences 2 | tactagtataacttcgtataggatactttatac gaagttatcattgggattcttcctattttgatc caagcatcaccatcgaccctctagtccacatct caccatcgacccataacttcgtatagcatacat tatacgaagttatgtccctcgaagaggttcgaa ttcgtttaaacGGTACCCTCGAC | ||
| pDP5 | Plasmid Factory | PF435 | |
| pDP6 | Plasmid Factory | PF436 | |
| Phosphate-Buffered Saline (PBS) | Thermo Fisher Scientific | 10010023 | |
| FBS (Fetal bovine serum) | Thermo Fisher Scientific | 10500064 | |
| Penicillin streptomycin | Thermo Fisher Scientific | 15140122 | |
| DMEM (Dulbecco's Modified Eagle Medium)+ Glutamax | Thermo Fisher Scientific | 61965026 | |
| DPBS (Dulbecco's Phosphate Buffer Saline) | Thermo Fisher Scientific | 14190094 | |
| HEK293 cells | Thermo Fisher Scientific | 85120602-1VL | |
| Flasks | BD Falcon | 10078780 | 175 cm2 |
| Tris H-CL | Sigma Aldrich | 10812846001 | For TE buffer use 10 mM, pH 8.0; for lysis buffer use 50mM, pH 8.5; for IE buffer use 20 mM, pH 8.0; for elution buffer use 20 mM, pH 8.0 |
| EDTA | EDTA: Invitrogen | EDTA: AM9260G | For TE buffer use 1 mM EDTA |
| Ultrapure water | see Ultrapure water system | ||
| CaCl2 | SigmaAldrich | C5080 | 2.5 M |
| Dulbecco´s phosphate-buffered saline (DPBS) | Thermo Fisher Scientific | 14190136 | |
| NaCl | Sigma-Aldrich | S3014 | FOR HBS use 140 mM; for Lysis Buffer use 150 mM; for IE buffer use 15 mM; for elution buffer use 250 mM |
| MgCl2 | Sigma-Aldrich | M8266-100G | For lysis Buffer: 1 mM |
| Ultracentrifuge sealing tubes | Beckman Coulter | Quick-Seal® Polypropylene Tube | |
| OptiPrep™ Density Gradient Medium | Sigma Aldrich | D1556-250ML | |
| 10-mL syringe-18G needle | BD | 305064 | |
| Laboratory glass bottles | VWR | ? | |
| Anion exchange filter | PALL laboratory | MSTG25Q6 | Acrodisc unit with Mustang Q membrane |
| Centrifugal filter unit | Merck | Z740210-24EA | Amicon Ultra-4 device |
| Endotoxin-Free Plasmid DNA Isolation Kits | Thermo Fisher Scientific | A33073 | |
| Na2PO4 | Sigma-Aldrich | S7907 | |
| HEPES | Sigma-Aldrich | H7523 | 15 mL |
| Falcon tube | Thermo Fisher Scientific | Corning 352196 | |
| Falcon tube | Thermo Fisher Scientific | Corning 352070 | |
| Glass Vials | Novatech | 30209-1232 | CGGCCTCAGFGAGCGA |
| Forward Primer for Inverted Terminal Repeat (ITR) sequence | GGAACCCCTAGTGATGGAGTT | ||
| Reverse Primer for Inverted Terminal Repeat (ITR) sequence | CACTCCCTCTCTGCGCGCTCG | ||
| 5´FAM / 3´BHQ1 probe | Jena Bioscience | ||
| 0.22 mm filter | Merck | SLGV004SL | Millex-GV filter |
| EQUIPMENT AAV5 VIRAL VECTOR PREPARATION | |||
| Freezer -20 °C | |||
| Freezer -80 °C | |||
| Fridge +4 °C | |||
| AAV viral room | |||
| Ultrapure Water system | Merck | Milli-Q® IQ 7000 | |
| Vortex mixer | VWR | 444-0004 | |
| Ultracentrifuge | Beckman Coulter | Beckman Optima LE-80K Ultracentrifuge | |
| Autoclave | Tuttnauer | 2540 EL | |
| Polymerase Chain Reaction (PCR) | BioRad | C1000 Touch Thermal Cycler | |
| Quantitative PCR (qPCR) | Roche | LightCycler® 480 System | |
| Centrifuge | Thermo Fisher Scientific | Sorvall ST16 | |
| Water Bath | Thermo Fisher Scientific | TSGP02 | |
| ANIMAL MODEL | |||
| NG2-Cre mice | Jackson | NG2-CrexB6129, Stock #008533 | |
| REAGENTS FOR INJECTION OF REPROGRAMMING FACTORS INTO THE BRAIN | |||
| Water | |||
| Saline | Apoteket AB | 70% | |
| Ethanol | Solveco | ||
| Isolfurane | Apoteket AB | Dilute to 1% solution with warm water. | |
| Virkon | Viroderm | 7511 | |
| Pentobarbital | Apoteket AB | P0500000 | i.p. for terminal procedure at the dose of 60 mg/ml. |
| Sucrose | Merck | S0389-500G | |
| Trypan Blu | Thermo Fisher Scientific | 15250061 | |
| Retrobeads | Lumafluor | R170 | |
| Buprenorphine | Apoteket AB | ||
| EQUIPMENT FOR INJECTION OF REPROGRAMMING FACTORS INTO THE BRAIN | From RSG Solingen. | ||
| Scissors | VWR | 233-1552 | From Biochem. |
| Tweezers | VWR | 232-0007 | |
| Forcep | VWR | 232-0120 | |
| Scalpel holder | VWR | RSGA106.621 | Number 20. |
| Scalpel | VWR | RSGA106.200 | |
| Stereotaxic frame | Stoelting Europe | 51500D | |
| Mouse ear bars | Stoelting Europe | 51648 | 5 ul |
| Syringe with Removable needle | Hamilton Company | 65 | 0,75 inner diameter and 1.5 outer diameter. |
| Glass capilaries | Stoelting | 50811 | |
| Glass capillary puller | Sutter company | P-1000 | |
| Dental drill | Agnthos AB | 1464 | |
| Shaver | Agnthos AB | GT420 | |
| Isoflurane Chamber and pump | Agnthos AB | 8323101 | 2 mL |
| Syringes | Merck | Z118400-30EA | 25G |
| Needles | Merck | Z192414 Aldrich | |
| Mouse and neonatal rat adaptor for stereotaxic fram | Stoelting | 51625 | |
| Heating pad | Braintree scientific, inc | 53800M | From Covidien 2187. |
| Cotton gauze | Fisher Scientific | 22-037-902 | |
| Rubber tube | Elfa Distrelec | HFT-A-9.5/4.8 PO-X BK 150 | |
| Cotton swabs | Fisher Scientific | 18-366-473 | |
| REAGENTS FOR WHOLE-CELL PATCH CLAMP RECORDINGS | |||
| NaCl | Sigma-Aldrich | S3014 | |
| KCl | Sigma-Aldrich | P9333 | |
| NaH2PO4-H2O | Sigma-Aldrich | S9638 | |
| MgCl2-6 H2O | Sigma-Aldrich | M2670 | |
| CaCl2-6 H2O | Sigma-Aldrich | C8106 | |
| MgSO4-7 H2O | Sigma-Aldrich | 230391 | |
| NaHCO3 | Sigma-Aldrich | S5761 | |
| Glucose | Sigma-Aldrich | G7021 | see Ultrapure Water system |
| Ultrapure Water | Prepare 1 or 2M. | ||
| KOH | Sigma-Aldrich | P5958-500G | |
| K-D-gluconate | Sigma-Aldrich | G4500 Sigma | |
| KCl | Sigma-Aldrich | P9541 | |
| KOH-EGTA (Etilene glycol-bis-N-tetracetic acid) | Sigma-Aldrich | E3889 Sigma | |
| KOH- Hepes acid (N-2-hydroxyethylpiperazine-N’-2-ethanesulfonic acid) | Sigma-Aldrich | H7523 | |
| NaCl | Sigma-Aldrich | S3014 | |
| Mg2ATP | Sigma-Aldrich | A9187 | |
| Na3GTP | Sigma-Aldrich | G8877 | |
| Biocytin | Sigma-Aldrich | B4261 | |
| Picrotoxin | Merck | P1675 Sigma | |
| CNQX | Merck | C239 Sigma | |
| Ice | |||
| EQUIPMENT FOR WHOLE-CELL PATCH CLAMP RECORDINGS | |||
| Borosilicate glass pipette | Sutter Company | B150-86-10 | |
| Glass capillary puller | Sutter company | P-1000 | |
| Vibratome | Leica | Leica VT1000 S | |
| WaterBath | Thermo Fisher Scientific | TSGP02 | |
| Clampfit software | Molecular Devices | ||
| Multiclamp software | Molecular Devices | ||
| REAGENTS FOR IMMUNOHISTOCHEMISTRY | Use at a concentration of 4%. CAUTION: PFA is a potent fixative. Avoid ingestion and contact with skin. | ||
| Paraformaldehyde (PFA) | Merck Millipore | 1040051000 | Use at a concentration of 0.1%. |
| Triton X-100 | Fisher Scientific | 10254640 | Donkey.Use at a concentration of 1 : 400. |
| Serum | Merck Millipore | S30-100ML | Reconstitute the powder in Milli-Q water to 1 mg/mL. Aliquot and store at -20°C, light sensitive. Use at a concentration of 1 : 500. |
| 4′,6-Diamidino-2′-phenylindole dihydrochloride (DAPI) | Sigma Aldrich | D9542 | 1: 600 in KPBS-T. |
| streptavidin- Cy3 | Thermo Fisher Scientific | 434315 | 1:5000, rabbit. |
| Anti-GAD65/67 | Abcam | 1:2000, mouse. | |
| Anti-PV | Sigma | P3088 | 1:200, goat. |
| Anti-Chat | Merk | AB143 | 1:5000, rabbit. |
| Anti-NPY | Immunostar | 22940 | |
| K2HPO4 | Sigma-Aldrich | P3786 | |
| NaCl | Sigma-Aldrich | S3014 | |
| KH2PO4 | Sigma-Aldrich | NIST200B | 1:1000, chicken. |
| Anti-GFP | Abcam | ab13970 | |
| Mounting solution | Merck | 10981 | Polyvinyl alcohol mounting medium with DABCO®, antifading |
| OCT | Agar Scientific | ||
| Glycerol | Sigma-Aldrich | G9012-100ML | |
| Distilled water | |||
| Anti-freeze solution | Tissue Pro Technology | AFS05-1000N, 1000 mL/ea. | |
| EQUIPMENT FOR IMMUNOHISTOCHEMISTRY | |||
| Inverted fluorescence microscope | Leica | DMI6000 B | |
| Confocal microscope | Leica | TCS SP8 laser scanning confocal microscope. | |
| Prism | GraphPad | ||
| Microtome | Leica | SM2010R |
Referencias
- Barker, R. A., Parmar, M., Studer, L., Takahashi, J. Human Trials of Stem Cell-Derived Dopamine Neurons for Parkinson's Disease: Dawn of a New Era. Cell Stem Cell. 21 (5), 569-573 (2017).
- Southwell, D. G., et al. Interneurons from embryonic development to cell-based therapy. Science. 344 (6180), 1240622 (2014).
- Parmar, M., Torper, O., Drouin-Ouellet, J. Cell-based therapy for Parkinson's disease: A journey through decades toward the light side of the Force. European Journal of Neuroscience. , (2018).
- Torper, O., et al. In Vivo Reprogramming of Striatal NG2 Glia into Functional Neurons that Integrate into Local Host Circuitry. Cell Reports. 12 (3), 474-481 (2015).
- Su, Z., Niu, W., Liu, M. L., Zou, Y., Zhang, C. L. In vivo conversion of astrocytes to neurons in the injured adult spinal cord. Nature Communication. 5, 3338 (2014).
- Liu, Y., et al. Ascl1 Converts Dorsal Midbrain Astrocytes into Functional Neurons In Vivo. Journal of Neuroscience. 35 (25), 9336-9355 (2015).
- Guo, Z., et al. In vivo direct reprogramming of reactive glial cells into functional neurons after brain injury and in an Alzheimer's disease model. Cell Stem Cell. 14 (2), 188-202 (2014).
- Grande, A., et al. Environmental impact on direct neuronal reprogramming in vivo in the adult brain. Nature Communication. 4, 2373 (2013).
- Niu, W., et al. In vivo reprogramming of astrocytes to neuroblasts in the adult brain. Nature Cell Biology. 15 (10), 1164-1175 (2013).
- Weinberg, M. S., Criswell, H. E., Powell, S. K., Bhatt, A. P., McCown, T. J. Viral Vector Reprogramming of Adult Resident Striatal Oligodendrocytes into Functional Neurons. Molecular Therapy. 25 (4), 928-934 (2017).
- Pereira, M., et al. Direct Reprogramming of Resident NG2 Glia into Neurons with Properties of Fast-Spiking Parvalbumin-Containing Interneurons. Stem Cell Reports. 9 (3), 742-751 (2017).
- Rivetti di Val Cervo, P., et al. Induction of functional dopamine neurons from human astrocytes in vitro and mouse astrocytes in a Parkinson's disease model. Nature Biotechnology. 35 (5), 444-452 (2017).
- Crosson, S. M., Dib, P., Smith, J. K., Zolotukhin, S. Helper-free Production of Laboratory Grade AAV and Purification by Iodixanol Density Gradient Centrifugation. Molecular Therapy Methods & Clinial Development. 10, 1-7 (2018).
- Torper, O., et al. Generation of induced neurons via direct conversion in vivo. Proceedings of the National Academy of Sciences of the United States of America. 110 (17), 7038-7043 (2013).
- Kawaguchi, Y., Kubota, Y. Correlation of physiological subgroupings of nonpyramidal cells with parvalbumin- and calbindinD28k-immunoreactive neurons in layer V of rat frontal cortex. Journal of Neurophysiology. 70 (1), 387-396 (1993).
- Grealish, S., et al. The A9 dopamine neuron component in grafts of ventral mesencephalon is an important determinant for recovery of motor function in a rat model of Parkinson's disease. Brain. 133 (Pt 2), 482-495 (2010).
- Thompson, L., Barraud, P., Andersson, E., Kirik, D., Bjorklund, A. Identification of dopaminergic neurons of nigral and ventral tegmental area subtypes in grafts of fetal ventral mesencephalon based on cell morphology, protein expression, and efferent projections. Journal of Neuroscience. 25 (27), 6467-6477 (2005).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados