Method Article
Producción de E. coli-expresada Nanopartículas de Proteína Auto-Ensamblaje para Vacunas Que Requieren Presentación de Epitope Trimérico
En este artículo
Resumen
Aquí se proporciona un método detallado que describe la purificación, el enjuimiento y la caracterización de nanopartículas de proteínas autoensamblables (SAPN) para su uso en el desarrollo de vacunas.
Resumen
Las nanopartículas de proteínas autoensambladas (SAPN) funcionan como pantallas de antígeno repetitivo y se pueden utilizar para desarrollar una amplia gama de vacunas para diferentes enfermedades infecciosas. En este artículo demostramos un método para producir un núcleo SAPN que contiene un conjunto de paquete de seis hélices (SHB) que es capaz de presentar antígenos en una conformación trimé. Describimos la expresión del SHB-SAPN en un sistema de E. coli, así como los pasos de purificación de proteínas necesarios. Incluimos un paso de lavado de isopropanol para reducir el lipopolisacárido bacteriano residual. Como una indicación de la identidad y pureza de la proteína, la proteína reaccionó con anticuerpos monoclonales conocidos en los análisis de manchas occidentales. Después de volver a plegar, el tamaño de las partículas cayó en el rango esperado (20 a 100 nm), que fue confirmado por dispersión dinámica de la luz, análisis de seguimiento de nanopartículas y microscopía electrónica de transmisión. La metodología descrita aquí está optimizada para el SHB-SAPN, sin embargo, con sólo ligeras modificaciones se puede aplicar a otras construcciones de SAPN. Este método también es fácilmente transferible a la producción a gran escala para la fabricación de GMP para vacunas humanas.
Introducción
Si bien el desarrollo de vacunas tradicionales se ha centrado en los patógenos inactivados o atenuados, el enfoque de las vacunas modernas se ha desplazado hacia las vacunas subunidades1. Este enfoque puede conducir a una respuesta más específica y potencialmente más eficaces a los candidatos a vacunas. Sin embargo, uno de los principales inconvenientes es que las vacunas subunidades no sonpartículas como organismos enteros, lo que puede resultar en una inmunogenicidad reducida 2. Una nanopartícula como sistema de visualización de antígenos repetitivo puede tener los beneficios tanto del enfoque de la vacuna subunidad dirigida como de la naturaleza particulada de todo el organismo1,3.
Entre los tipos existentes de nanovacunas, los conjuntos de proteínas racionalmente diseñados permiten el diseño y desarrollo decandidatos a vacunas que pueden presentar múltiples copias del antígeno potencialmente en una conformación nativa 1,4 ,5,6. Un ejemplo de estos conjuntos de proteínas son las nanopartículas de proteínaauto (SAPN)7. Los SAPN se basan en dominios de bobina sin bobina y se expresan tradicionalmente en Escherichia coli8. Se han desarrollado candidatos a vacunas SAPN para una variedad de enfermedades como elpaludismo, el SRAS, la gripe, la toxoplasmosis y el VIH-1 9,10,11,12,13 , 14 , 15 , 16 , 17 , 18 , 19. El diseño de cada candidato a SAPN es específico del patógeno de interés, sin embargo, las técnicas de producción, purificación y redistribución son generalmente ampliamente aplicables.
Uno de nuestros intereses actuales es una vacuna eficaz contra el VIH-1. En RV144, el único ensayo clínico de fase III de una vacuna contra el VIH-1 que demostró una eficacia modesta, el menor riesgo de infección se correlacionó con los anticuerpos IgG con el bucle V1V2 de la proteína envolvente20,21. Se cree que la presentación trimérica nativa de esta región es importante para la inmunogenicidad protectora22. Para presentar el bucle V1V2 lo más cerca posible de la conformación nativa, desarrollamos una prueba de la principal vacuna SAPN candidata que contenía el paquete de seis hélices posterior a la fusión del sobre VIH-1 (SHB) para presentar el bucle V1V2 en la conformación correcta9 . Este candidato fue reconocido por anticuerpos monoclonales conocidos a la proteína de envoltura VIH-1. Los ratones inmunizados con V1V2-SHB-SAPN elevaron anticuerpos específicos V1V2, que, lo más importante, están vinculados a gp70 V1V2, los epítopos conformacionales correctos9. El núcleo SHB-SAPN podría tener otras funciones más allá del papel como portador para el bucle HIV-1 V1V2. Aquí describimos una metodología detallada para la expresión, purificación, redistribución y validación del núcleo SHB-SAPN. La selección de la secuencia, el diseño de nanopartículas, la clonación molecular y la transformación de E. coli se han descrito previamente9.
Protocolo
1. Expresión de la proteína SHB-SAPN en E. coli BL21(DE3)
- Mezclar 95 ml del componente A y 5 ml del componente B del medio en un matraz Erlenmeyer de vidrio estéril de 2 L según las instrucciones del fabricante (véase la Tabla de Materiales). Añadir ampicilina a una concentración final de 100 g/ml.
- Inocular los medios de comunicación con E. coli de un cultivo de acciones de glicerol previamente establecido. Cultivo de incubación a 30oC con agitación a 200 rotaciones por minuto (rpm) durante 48 h.
NOTA: La culata usada de E. coli BL21 (DE3) contenía el vector de expresión resistente a la ampicilina23 con el gen SHB-SAPN. Aunque el protocolo general de los medios recomienda 24 h de incubación a 37oC, 48 h de incubación a 30oC dio un mayor rendimiento para SHB-SAPN. - Transfiera el cultivo a dos tubos cónicos de 50 ml. Centrifugar los tubos a 4.000 x g durante 10 min con un rotor de ángulo fijo a 4oC. Retire el sobrenadante y guarde el pellet para cosechar células.
NOTA: El pellet celular puede procesarse inmediatamente o congelarse a -80 oC hasta su uso.
2. Lisis de E. coli BL21(DE3) por sonicación
NOTA: Utilice material plástico y cristalería no pirogénicos horneados a 250 oC durante al menos 30 minutos. El TCEP es necesario en los buffers durante este protocolo si el antígeno visualizado contiene los bonos S-S. Sólo para el núcleo SHB-SAPN, la presencia de TCEP en los búferes no es esencial.
- Preparar tampón libre de imidazol (8 M Urea, 50 mM de fosfato sódico monobásico, base Tris de 20 mM, 5 mM TCEP) pH 8.0 (ajustado con 5 N NaOH) y filtrarlo utilizando una unidad de filtración de vacío de 0,22 m.
- Resuspender las células peletadas (del paso 1.3) con 40 ml de tampón libre de imidazol en un tubo cónico de 50 ml. Sonicar las células resuspendidas con una sonda sobre hielo durante 5 min (4 s de sonicación, 6 s de descanso) con una salida de sonicación de 150 W.
- Centrifugar el lisado celular (40 ml) a 29.000 x g a 4 oC durante 25 minutos en un rotor de ángulo fijo para generar sobrenadante clarificado. Transfiera el sobrenadante a un matraz estéril de 150 ml y deseche el pellet. Diluir el sobrenadante a 100 ml utilizando el búfer libre de imidazol (más adelante en el protocolo denominado "muestra").
NOTA: Este paso de dilución es necesario para evitar que la presión del sistema FPLC se vuelva demasiado alta durante la carga de lisado en la columna.
3. Purificación de proteínas usando una hiscolumna
NOTA: Este protocolo se realizó utilizando un instrumento FPLC, pero se puede adaptar al flujo de gravedad.
- Preparar los siguientes tampones y filtrarlos utilizando una unidad de filtración de botella de vacío de 0,22 m: (i) tampón libre de imidazol "Buffer A" (8 M Urea, 50 mM de fosfato sódico monobásico, base Tris de 20 mM, 5 mM TCEP) pH 8.0; (ii) tampón de imidazol de 500 mM "Buffer B" (8 M Urea, 50 mM de fosfato sódico monobásico, base Tris de 20 mM, 5 mM TCEP, 500 mM de Imidazol) pH 8.0; y (iii) lavado de isopropanol (20 mM Tris, 60% isopropanol) pH 8.0.
NOTA: el pH para cada buffer se ajustó con 5 NaOH. - Equilibrar la columna.
NOTA: Para la producción a escala de laboratorio, este protocolo utiliza una columna His preempaquetada de 5 ml, pero se puede utilizar cualquier columna de mayor tamaño.- Abra el software FPLC y haga clic en la opción Nuevo método. Se abrirá inmediatamente en el menú Configuración del método. En el menú desplegable para la posición de columna, elija el puerto C1 3.
- En el menú desplegable Mostrar por técnica, elija afinidad. En el menú desplegable Tipo de columna elija otros, Histrap HP, 5 mL. El volumen de columna y las cajas de presión se ajustarán automáticamente a los valores adecuados.
- Haga clic en el botón Esquema del método. Arrastre los siguientes botones desde el menú emergente Biblioteca de fases: equilibrio, aplicación de ejemplo,lavado de columnasy elución junto a la flecha en ese orden exacto. Cierre el menú Biblioteca de fases.
- Haga clic en el botón de equilibrio. Los valores enumerados en la tabla deben ser "buffer inicial B" (4%), "buffer final B" (4%) y "volume (CV)" 5.
- Haga clic en el cuadro Aplicación de ejemplo. En el cuadro de carga de muestra, haga clic en el botón de opción Inyectar muestra en columna con bombade muestra . Asegúrese de que la casilla situada junto a la configuración Usar caudal del método esté marcada en la caja de inyección de muestra con bomba del sistema. Junto al cuadro de volumen en el lado derecho de la pantalla, cambie el valor a 20 ml.
- Haga clic en el botón De lavado de columnas. Los valores enumerados en la tabla deben ser "buffer inicial B" (4%), "buffer final B" (4%) y "volume (CV)" 5. Junto al esquema de recopilación de fracciones, desactive el cuadro Habilitar.
- Haga clic en el botón de elución. Los valores enumerados en la tabla deben ser "buffer inicial B" 0%, "buffer final B" 100% y "volume (CV)" 5. Junto al esquema de recogida de fracciones, haga clic en el cuadro Habilitar. Deshaga clic en Usar tamaño de fracción de la configuración del método y ajuste el tamaño de fracción a 4 ml en el cuadro de relleno a continuación.
- Haga clic en el botón Guardar como en la parte superior del software. Asigne al archivo el nombre "equilibration".
- Conecte una columna His preempaquetada de 5 ml al puerto de columna 3 correspondiente en el FPLC. Tanto el tubo de la bomba A como la bomba B, así como el tubo de la bomba de muestra deben colocarse en agua desionizada filtrada de 0,22 m. Ejecute el programa de equilibrio.
- Coloque tanto la bomba A como la bomba B, así como el tubo de la bomba de muestra en el búfer libre de imidazol (Buffer A) y vuelva a ejecutar el protocolo de equilibrio.
- Enlazar la muestra a la columna y purificar la proteína.
- Abra el software FPLC y haga clic en la opción Nuevo método. Se abrirá inmediatamente en el menú Configuración del método. En el menú desplegable de Posición de columna, elija El puerto C1 3. En el menú desplegable Mostrar por técnica, elija afinidad. En el menú desplegable tipo de columna elija others, Histrap HP, 5 mL. El volumen de columna y los cuadros de presión se establecerán automáticamente en los valores adecuados.
- Haga clic en el botón Esquema del método. Arrastre los botones del menú emergente Biblioteca de fases: equilibrio, aplicaciónde muestra, lavado de columnas (Lavado 1), lavado de columnas (Lavado 2), lavado de columnas (Lavado 3) y Elución junto a la flecha en ese orden exacto. Cierre el menú Biblioteca de fases.
- Haga clic en el botón Equilibración. Los valores enumerados en la tabla deben ser "buffer inicial B" 4%, "buffer final B" 4% y "volumen (CV)" 5.
- Haga clic en el cuadro Aplicación de ejemplo. En el cuadro de carga de muestra, haga clic en el botón de opción para Inject sample en la columna con la bombade muestra . Asegúrese de que la casilla situada junto a la configuración Usar caudal del método esté marcada en la caja de inyección de muestra con bomba del sistema.
- Junto al cuadro de volumen en el lado derecho de la pantalla, cambie el valor a 100 ml. Junto al esquema de recopilación de fracciones, haga clic en el botón Habilitar. Deshaga clic en el cuadro Usar tamaño de fracción de la configuración del método y, a continuación, cambie el tamaño de fracción a 4 ml.
- Haga clic en el primer botón de lavado de columna (Lavado 1). Los valores enumerados en la tabla deben ser "buffer inicial B" 4%, "buffer final B" 4% y "volumen (CV)" 10. Junto al esquema de recopilación de fracciones, haga clic en el cuadro Habilitar. Deshaga clic en Usar tamaño de fracción de la configuración del método y, a continuación, cambie el tamaño de fracción a 4 ml.
- Haga clic en el botón de lavado de la segunda columna (Lavado 2). Los valores enumerados en la tabla deben ser "buffer inicial B" 0%, "buffer final B" 0% y "volumen (CV)" 5. Junto al esquema de recopilación de fracciones, haga clic en el cuadro Habilitar. Deshaga clic en Usar tamaño de fracción de la configuración del método y, a continuación, cambie el tamaño de fracción a 4 ml.
- Haga clic en el botón de lavado de la tercera columna (Lavado 3). Los valores enumerados en la tabla deben ser "buffer inicial B" 0%, "buffer final B" 0% y "volumen (CV)" 5. Junto al esquema de recopilación de fracciones, haga clic en Habilitar. Deshaga clic en el cuadro Usar tamaño de fracción de la configuración del método y, a continuación, cambie el tamaño de fracción a 4 ml.
- Haga clic en el botón Elution. En la tabla, haga clic con el botón derecho en la información que aparece y, en el menú que aparece, haga clic en Eliminar paso. Arrastre el botón de degradado isocrático a la tabla dos veces, de modo que haya dos entradas.
- El valor de la primera entrada debe ser "buffer inicial B" 30%, "buffer final B" 30% y "Volume (CV)" 10. El valor de la segunda entrada debe ser "buffer inicial B" 100%, "buffer final B" 100% y "volume (CV)" 10. Junto al esquema de recopilación de fracciones, haga clic en el botón Habilitar. Haga clic en el cuadro situado junto a Usar tamaño de fracción a partir dela configuración del método .
- Haga clic en el botón Guardar como en la parte superior del software. Asigne al archivo el nombre "purificación". Bomba Un tubo del FPLC debe colocarse en el tampón de lavado libre de imidazol, mientras que el tubo de la bomba B debe colocarse en el tampón de 500 mM de imidazol. El tubo de la bomba de muestra debe colocarse en la muestra de 100 ml.
- Ejecute el programa de "purificación" y espere el momento en que se necesita el 60% de isopropanol (Lavado 2). Pausa el programa, mueva la bomba Un tubo del lavado sin imidazol al lavado de isopropanol al 60%. Reinicie el programa.
- Una vez completado el paso de isopropanol, detenga el programa de nuevo y mueva la bomba un tubo de nuevo en el tampón de lavado libre de imidazol. Reinicie el programa de purificación (el resto de la ejecución está automatizada).
4. Evaluación de pureza e identificación de proteínas por SDS-PAGE
- Combine todas las fracciones correspondientes a (i) flujo a través (el lisado celular que no se une a la columna Su columna), (ii) Lavado 1, (iii) Lavado 3 (60% lavado isopropanol), y (iv) Lavado 3 en tubos cónicos separados de 50 ml. No combine las fracciones de 2 ml de los pasos de elución.
- Mezclar 15 l de cada una de las fracciones agrupadas y todas las fracciones de los pasos de elución con 2 tampón de muestra de Laemmli en un tubo de microcentrífuga de 0,5 ml y desnaturalizarlos a 95 oC durante 10 minutos.
- Mientras que la proteína desnaturaliza, configurar el aparato de gel de funcionamiento con 3 geles de poliacrilamida prefabricados 4-20% sin manchas en 1 tampón de carrera Tris-glicina SDS-PAGE.
- Cargar 8 l de marcador de peso molecular en el primer pozo y 30 l de muestra desnaturalizada a los otros pozos del gel. Ejecuta los geles a 200 V hasta que el tinte llegue al fondo del gel (alrededor de 30 min). Retire los geles del aparato y enjuague brevemente con agua desionizada. Imagen del gel inmediatamente utilizando el sistema de imágenes sin manchas.
- Identificar las fracciones que contienen bandas proteicas con el tamaño correcto (18,07 kDa). Agrupa todas estas fracciones.
5. Identificación de proteínas por mancha occidental
- Ejecuta una mancha occidental usando un anticuerpo His-specific (anti-6x HisTag) y un anticuerpo específico de SHB (167-D-IV) para identificar la proteína purificada de longitud completa. El anticuerpo anti-6x HisTag reconoce la terminal N de la proteína y el anticuerpo 167-D-IV reconoce la terminal C que demuestra la presencia de la proteína de longitud completa.
- Determinar la concentración proteica de la (i) flujo a través, (ii) Lavado 1, (iii) Lavado 2 (60% lavado de isopropanol), (iv) Lavado 3, y todas las fracciones de los pasos de elución con el instrumento espectrómetro a una absorbancia de 280 nm. Generar diluciones que contengan 100 ng de proteína en 15 ml de tampón libre de imidazol para cada uno de estos grupos.
- Añadir 15 l de 2 x tampón de muestra de Laemmli a cada 15 l de las muestras y desnaturalizarlas como en el paso 4.1. Una vez finalizada la desnaturalización, gire por los tubos para asegurarse de que toda la proteína se puede transferir.
- Cargue las muestras y el marcador premanchado en un gel de poliacrilamida prefabricado de 4-20% libre de manchas. Ejecute la electroforesis a 200 V hasta que el frente del tinte alcance la parte inferior del gel.
- Mientras el gel funciona, hacer 1 L de TBS-T (20 mM Tris, 150 mM NaCl, y 0.1% Tween 20) y 200 mL de 5% de leche sin grasa en TBS-T.
- Utilice un sistema de transferencia de manchas occidentales para transferir proteínas a una membrana de nitrocelulosa. Utilice una pila de transferencia premontada y coloque el gel sobre ella. Configurar el sistema para funcionar a 25 V durante 7 min. Compruebe la presencia del marcador premanchado en la membrana de nitrocelulosa indicando una transferencia completa.
NOTA: Todos los pasos subsiguientes se realizan en un agitador orbital a 100 rpm a temperatura ambiente (RT). - Una vez completada la transferencia, lave las manchas dos veces con TBS-T durante 10 minutos cada una.
- Bloquear las membranas de nitrocelulosa (blot) con 5% de leche sin grasa en TBS-T durante al menos 1 h. Lavar las manchas dos veces con TBS-T durante 10 minutos cada una.
- Diluir los anticuerpos primarios 167-D-IV y anti-6x HisTag a 1 mg/ml en TBS-T (stock Ab). Diluir el stock Abs de la 167-D-IV 10,000-fold y el anti-6x HisTag 5,000-fold añadiendo 2 l de la acción 167-D-IV y 4 l de la acción anti-6x anticuerpos HisTag a dos tubos diferentes que contienen 20 ml de TBS-T. Añadir el volumen total de 20 ml de anticuerpos primarios, uno a cada mancha, e incubar manchas durante 1 h. Lavar manchas dos veces con TBS-T durante 10 min.
- Diluir 4 ml de 1 mg/ml del anticuerpo secundario antihumano ratón conjugado con fosfatasa alcalina en 20 ml de TBS-T (1:5.000 dilución). Diluir 4 ml de 1 mg/ml del anticuerpo secundario antiratón de cabra conjugado con fosfatasa alcalina en 20 ml de TBS-T (1:5.000 dilución). Añadir anticuerpos secundarios a las manchas correspondientes.
NOTA: El anticuerpo secundario antihumano se une a 167-D y el antiratón se une al Anti-6x HisTag. Lave las manchas dos veces con TBS-T durante 10 min. - Agregue suficiente sustrato de fosfatasa alcalina BCIP/NBT para cubrir las manchas. Desarrolle las manchas durante unos 10 minutos hasta que aparezcan las bandas. Enjuague las manchas con agua fría del grifo y déjelas secar antes de escanearlas con un escáner de cama plana.
6. Redoblar el SHB-SAPN
- Añadir la proteína agrupada (10-u201220 mL en total) a un casete de diálisis cortado con peso molecular de 10 kDa y marcarla en urea de 8 M, Tris de 20 mM, glicerol al 5%, 5 mM de TCEP pH 8.5 a RT (18-u201226 oC) durante la noche.
- Dializar lentamente la urea de la muestra disminuyendo la concentración de urea en el tampón de diálisis paso a paso en 2 M cada 2 h. A una concentración de urea de 2 M, mueva el aparato de diálisis a 4 oC (no utilice TCEP en el búfer de diálisis de este paso). Terminar el redoblamiento marcando la muestra en urea de 120 mM, Tris de 20 mM, glicerol al 5%, pH 8,5 a 4 oC durante la noche.
- Retire la proteína replegada (SHB-SAPN) del casete de diálisis. Filtre el SHB-SAPN utilizando un filer de jeringa de fluoruro de polivinilideno (PVDF) de 0,22 m. Aliquot SHB-SAPN en tubos estériles y congelarlos a -80 oC, dejando al menos 100 ml en RT para análisis posteriores.
7. Validación de partículas por tamaño y apariencia
- Dispersión dinámica de la luz (DLS)
- Medir el tamaño medio de partícula del SHB-SAPN por los siguientes parámetros: seleccionar proteína como material, crear un tampón complejo para urea de 120 mM, Tris de 20 mM, 5% de glicerol 25 oC para la temperatura, seleccionar cubetas desechables para el análisis, seleccionar medición, establecida para 5 corridas.
- Agregue 45 l de SHB-SAPN a una cubeta desechable y ejecute el software haciendo clic en la flecha verde. Seleccione el volumen de porcentaje para la lectura.
- Análisis de seguimiento de nanopartículas (NTA)
- Diluir la muestra en 1:20 en el búfer de redoblamiento. Hacer 10 ml de muestra diluida.
- Con jeringas de 3 ml, enjuague el instrumento NTA con el tampón de redoblamiento y cargue aproximadamente 1,5 ml de muestra para equilibrar el instrumento. Utilice el resto de la muestra para el análisis.
- Cree un nuevo SOP en el software NTA haciendo clic en la pestaña SOP. En la pestaña cambie el número de capturas a 3 y cambie el tiempo de captura a 30 s. Pulse el botón de enfoque automático en el lado izquierdo de la pantalla para enfocar la muestra. Utilice la perilla de enfoque manual en el lado de la máquina para ajustar el enfoque.
- Ejecute el SOP creado cuando el sistema solicite cargar un pequeño volumen de muestra con la jeringa. Después de que el sistema haya tomado todas las capturas, aparecerá automáticamente la pantalla de análisis. Deslice la barra límite de detección para que todas las partículas reales estén marcadas con cruces rojas. Pulse el botón de análisis de ejecución y el análisis comenzará automáticamente.
- Microscopía electrónica de transmisión (TEM)
- Películas de soporte TEM de cobre de malla de forma de descarga de resplandor/carbono 400.
- Añadir 3 l de muestra a una concentración de 0,075 mg/ml a la rejilla durante 30 s. Salte del líquido con un papel de filtro.
- Lave la rejilla con 3 l de agua desionizada tres veces, cada vez que se mete el agua con papel de filtro.
- Añadir 3 l de acetato de ursiol al 0,5% de acetato de orina a la película de soporte y dejar que se quede durante 30 s. Salte la mayor parte del acetato de uranyl pero deje una película delgada en la superficie. Deje que las muestras se sequen antes de tomar imágenes en el microscopio electrónico de transmisión.
- Muestras de imagen a 80 kV en un TEM.
8. Determinación de los niveles de endotoxina en las muestras utilizando un ensayo cinético de lisato de limulus amoebocyte (LAL)
- Retire el kit y las muestras del refrigerador y deje que se equilibren a RT.
- Para realizar este ensayo, se requiere un lector de placas con un bloque de calor y la capacidad de leer las muestras durante 40 lecturas a una longitud de onda de 405 nm a 37 oC. Escriba una plantilla de programa para que los pozos se lean cada 150 s para identificar el punto de tiempo de inicio (el OD aumentó en 0,2 en comparación con la primera lectura).
- Diluir la muestra a la concentración de dosis de inmunización en PBS.
NOTA: El pH del búfer de redoblamiento está fuera del rango del ensayo LAL. La dilución de la muestra en PBS establecerá el pH en el rango aceptable. - Resuspender la endotoxina de control en el volumen adecuado de agua LAL libre de endotoxinas según lo determinado por el certificado de análisis para generar 50 EU/ml. Vórtice vigorosamente el vial durante 15 minutos para asegurar la resuspensión completa de la endotoxina.
- Genere la curva estándar de endotoxina saque una dilución serial de 10 veces en viales de vidrio en el rango de 50 UE a 0,005 UE/ml. Para cada dilución, añadir 0,1 ml de la dilución anterior a 0,9 ml de agua LAL. Vórtice vigorosamente después de la combinación durante 1 min.
- Añada diluciones de curva estándar y muestras SHB-SAPN por duplicado a una placa de 96 pocillos. Utilice el agua LAL como un control negativo en duplicado también. Preincubar la placa a 37oC durante 15 min.
- Hacia el final de la incubación, resuspenda el vial del reactivo de ensayo con 2,6 ml de agua LAL. Mezcle suavemente el contenido con una pipeta serológica.
- Añadir 100 l del reactivo de ensayo a cada pocto de la placa de 96 pocillos. Mueva rápidamente la placa al lector de placas y ejecute la plantilla de programa escrita en el paso 8.2.
- Una vez completado el programa, genere una curva estándar utilizando el valor de registro de los controles frente al valor de registro de la hora de inicio. Utilice la fórmula generada a partir de esta curva para calcular la concentración de endotoxinas en las muestras.
Resultados
El SHB-SAPN completamente ensamblado que se muestra aquí se basa en secuencias proteicas (Figura1A)que se prevé que se plieguen en una partícula que contenga 60 copias del monómero (Figura1B). La Figura 2 proporciona un esquema del método para la producción, purificación e identificación del núcleo SHB-SAPN. E. coli de una población de glicerol que contenía un vector de expresión pPep-T con secuencia genética del núcleo SHB-SAPN fueron inducidos en BL21 (DE3) E. coli. Las células bacterianas se cultivaron con éxito y se lisó bajo condiciones de desnaturalización y reducción.
El lisato de célula total se utilizó para purificar los monómeros SHB-SAPN mediante FPLC utilizando una columna Ni2+ (Figura3A). El cromatógrafo FPLC demuestra que la proteína eluyó tanto a 150 mM como a 500 mM de imidazol (Figura3A). El cromatograma también muestra otros dos picos a 185 ml y 210 ml de volumen total correspondiente al lavado de isopropanol y al lavado sin imidazol, respectivamente. Las fracciones y la pureza de la proteína recombinante fueron identificadas por geles SDS-PAGE gradiente (Figura3B). La proteína de interés se localizó principalmente en las fracciones 68-u201279 (278-u2012300 mL de volumen total). Estas fracciones se combinaron para análisis posteriores. Western blot con anticuerpo anti-His (N-terminal) y 167-D-IV anticuerpo (C-terminus) indicó que las fracciones agrupadas eran de hecho la proteína de interés (Figura4A,B). Estas manchas también demostraron la presencia de los multimers SHB-SAPN. Los lavados anteriores y las fracciones de elución tendían a contener una mayor concentración de proteína multimerizada y, por lo tanto, estaban excluidos.
Las muestras que contenían los monómeros proteicos de interés se plegaron en el SHB-SAPN completamente ensamblado mediante diálisis. La distribución del tamaño de partícula se determinó mediante el análisis de seguimiento de DLS y nanopartículas (Figura5A,B). El DLS identificó partículas con un diámetro hidrodinámico medio Z de 67 nm, mientras que el sistema NTA midió un tamaño medio de 81 nm. Las ligeras diferencias de tamaño se debieron a las técnicas de dimensionamiento de partículas, sinembargo, el tamaño de ambos análisis estaba en el rango esperado de 20-u2012100 nm 8,24,25. Los SHB-SAPN fueron visualizados por TEM y las imágenes mostraban partículas individuales bien formadas con la distribución del tamaño obtenida de las dos técnicas de dimensionamiento de partículas (Figura5C).
Durante la purificación de la proteína, la columna fue lavada con isopropanol para disminuir la contaminación por LPS en el producto final SHB-SAPN. Para verificar si el nivel de endotoxina era aceptable para la inmunización, la concentración de LPS en muestras SHB-SAPN purificadas con o sin el paso de lavado de isopropanol se determinó mediante un ensayo cinético de LAL. Los resultados indicaron que el lavado de isopropanol disminuyó los niveles de endotoxina de >0,25 UE/g a 0,010 EU/g de proteína SHB-SAPN (Tabla 1).
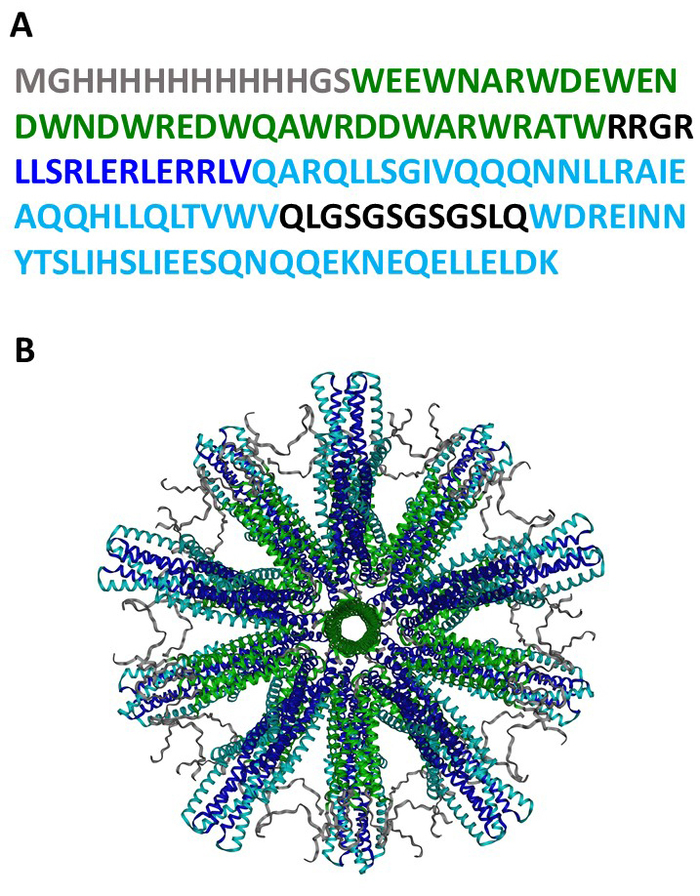
Figura 1: Secuencia y estructura proteica SHB-SAPN. (A) La secuencia de aminoácidos del monómero SHB-SAPN. (B) Modelo informático de la estructura del núcleo SHB-SAPN completamente ensamblado que consta de 60 monómeros proteicos. Esquema de color para secuencias de aminoácidos: Gris - HisTag; Verde - pentamer; Borde de diseño azul oscuro de novo; Paquete azul claro de seis hélices. Haga clic aquí para ver una versión más grande de esta figura.
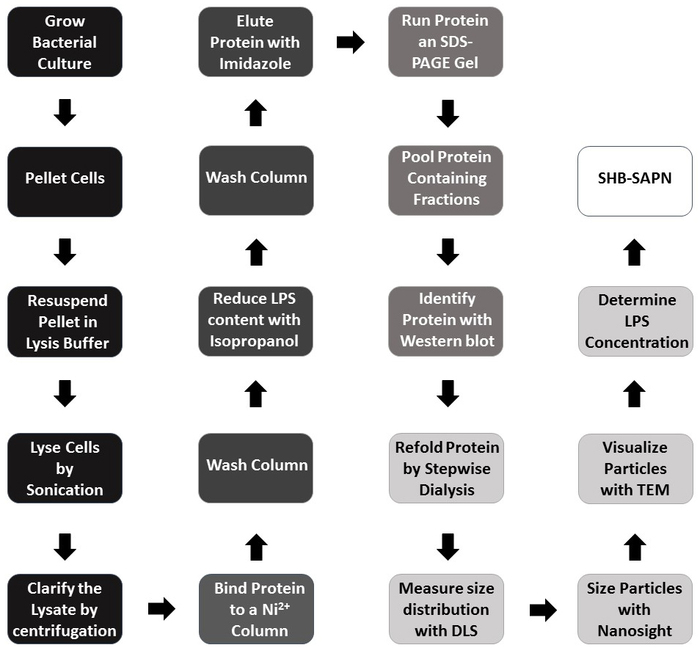
Figura 2: Diagrama de flujo de la protocolo para la producción SHB-SAPN. Esquema de color: Negro - expresión del monómero proteico en E. coli; Gris oscuro - purificación de monómeros; Gris medio - identificación del monómero; Gris claro : redoblamiento y caracterización; Blanco: producto SHB-SAPN completamente ensamblado. En pasos etiquetados con gris oscuro y medio, la proteína está bajo condiciones desnaturalizantes y reductoras. Haga clic aquí para ver una versión más grande de esta figura.
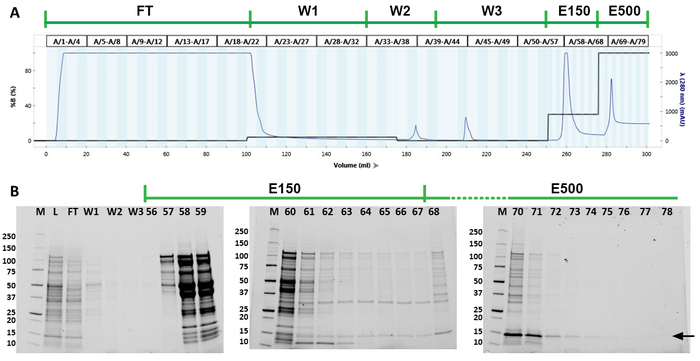
Figura 3: Purificación de proteínas de los monómeros SHB-SAPN. (A) Cromatógrafo de la purificación FPLC. La línea verde sobre el cromatograma indica los pasos de purificación. La línea azul en el cromatograma representa la densidad óptica de las fracciones a 280 nm de longitud de onda. La línea negra muestra qué porcentaje del tampón B (urea de 8 M, Tris de 20 mM, fosfato sódico de 50 mM monobásico, 5 mM TCEP, 500 mM de Imidazol, pH 8.5) que se utilizó en cada etapa de la purificación. (B) Geles SDS-PAGE de las fracciones agrupadas del lisado (L), el flujo a través (FT), el primer lavado (W1), el lavado de isopropanol (W2), el tercer lavado (W3) y las fracciones individuales de los pasos de elucion de 150 mM de imidazol (E150) y 500 mM de imidazol (E500) Purificación. Los marcadores moleculares en el primer carril (M) identifican bandas entre 10 y 250 kDa. La proteína diana está indicada por una flecha negra. Haga clic aquí para ver una versión más grande de esta figura.
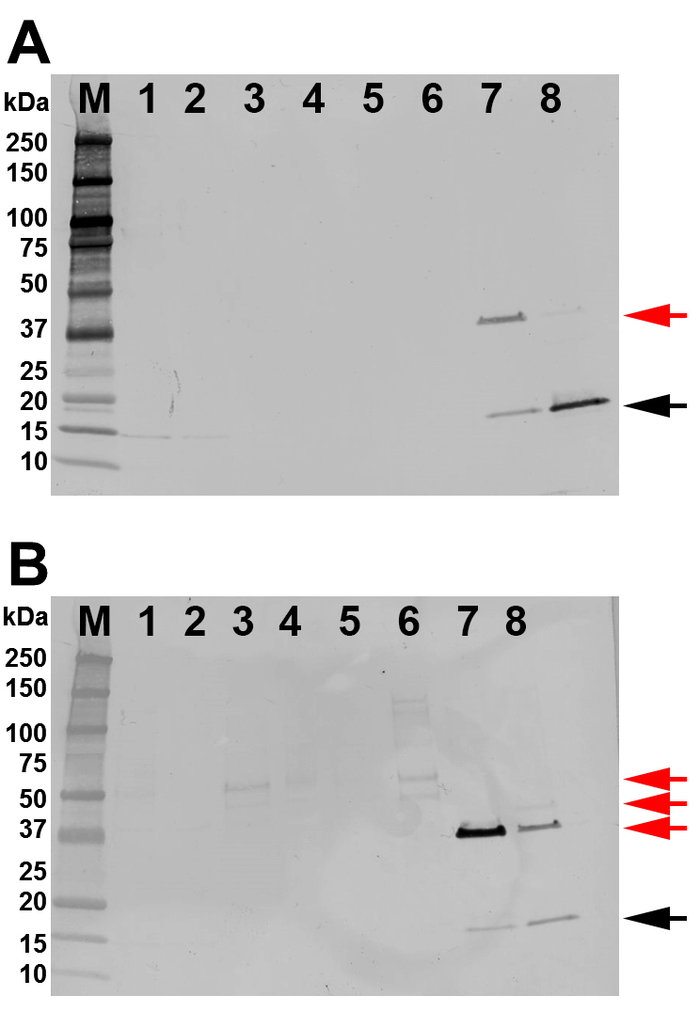
Figura 4: Identificación de la proteína por mancha occidental. Todos los carriles están cargados con 100 ng de proteína. (A) Resultados de una mancha occidental con anti-6x HisTag. (B) Resultados de una mancha occidental con un anticuerpo monoclonal 167-D-IV HIV-1. Los carriles están etiquetados como: M - marcador de peso molecular; 1 o lisado; 2 - Flujo a través; 3o primer lavado; 4o lavado de isopropanol (segundo lavado); 5o tercer lavado; 6 fracciones de volumen agrupado 56-u201261 (primer pico de elución), 7 facciones de volumen agrupado 62-u201267 (entre los dos picos), 8 fracciones de volumen agrupado 68-u201278 (segundo pico de elución). La proteína objetivo con el tamaño de banda esperado de 18,07 kDa como la banda monomérica SHB-SAPN está indicada por una flecha negra. Las bandas adicionales en los carriles 7 y 8 son dimers, trimers y multimers del SHB-SAPN (flecha roja). Haga clic aquí para ver una versión más grande de esta figura.
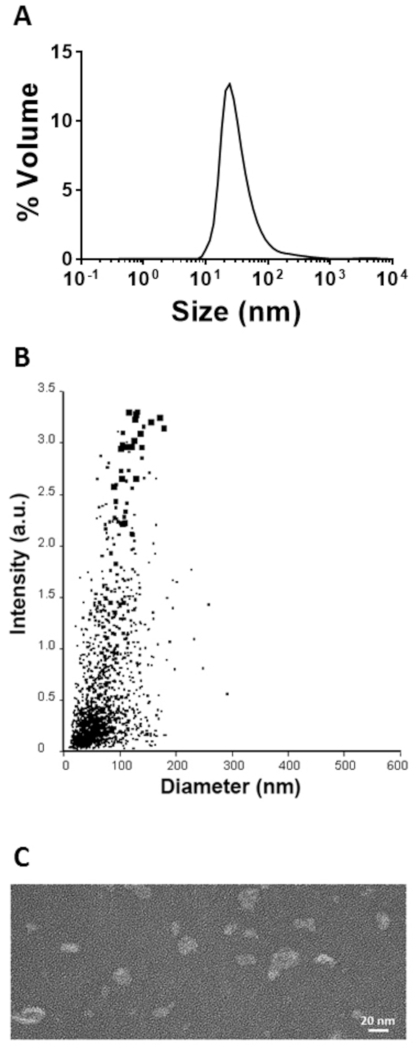
Figura 5: Caracterización del SHB-SAPN redoblado. (A). Distribución del tamaño de partícula determinada por DLS. ( B ) Tamañodepartícula determinado por el seguimiento de nanopartículas (sistema). (C) Visualización de las partículas SHB-SAPN por TEM. Haga clic aquí para ver una versión más grande de esta figura.
| Muestra | Endotoxina (EU/mL) | Endotoxina (UE/g de proteína) |
| SAPN con lavado de isopropanol | 2.02 | 0.01 |
| SAPN sin lavado de isopropanol | >50 | >0.25 |
| Control negativo | Por debajo del nivel de detección | N/A |
Tabla 1: Niveles de endotoxina de SHB-SAPN sióndos. Los niveles de endotoxina en muestras SHB-SAPN purificadas con o sin un lavado de isopropanol se presentan tanto como unidades de endotoxina/ml como endotoxinas/g de proteína SHB-SAPN.
Discusión
La nanotecnología proporciona muchas ventajas y soluciones para el desarrollo de vacunas subunidades. Las nanovacunas pueden presentar antígenos repetidamente como partículas al sistema inmunitario huésped aumentando la inmunogenicidad26. Si bien hay muchos tipos diferentes de nanovacunas, creemos que las compuestas por proteína según novo parecen ser el enfoque más fuerte para el desarrollo de vacunas1. Pueden ser diseñados sin ninguna homología de secuencia a las proteínas anfitrionas y presentar el antígeno de interés en una conformación similar a la nativa, al tiempo que proporcionan un bajo costo de producción y altos rendimientos del producto. Un ejemplo de este enfoque es la tecnología SAPN, que hemos aplicado a las vacunas contra múltiples enfermedades infecciosas7. Abordando las dificultades en el desarrollo de la vacuna VIH-1, hemos diseñado un núcleo único SHB-SAPN para presentar eficazmente el antígeno V1V2 en una conformación trimé nativasimilar9. Muchas metas de vacunas, en particular para enfermedades virales, están presentes como recortadores27. Este fenómeno indica que nuestro diseño de SAPN tiene amplias implicaciones para el desarrollo de vacunas subunidades.
En este método, demostramos cómo producir SHB-SAPNs en un sistema de expresión de E. coli. Expresamos altos rendimientos de proteína (alrededor de 6 mg/100 ml de cultivo). La proteína contenía 10 histidinas y fue fácilmente purificada usando una cromatografía de afinidad de metal inmovilizada con columna Ni2+. Esta longitud de la His-Tag se encontró que es el óptimo para el mayor rendimiento proteico. La proteína purificada contenía la longitud completa de la proteína diseñada como lo indica la presencia tanto de la repetición HisTag de N-terminal como de la heptad terminal C. Utilizamos técnicas ampliamente aceptadas y las optimizamos para la expresión, producción y caracterización del núcleo SHB-SAPN. La falta de producción de la proteína de longitud completa durante el desarrollo de un SAPN que contenga un nuevo epítopo proteico podría indicar un problema de expresión del gen en la célula huésped. Si sucede, el gen y el sistema de expresión deben ser rediseñados y adaptados al protocolo descrito. La modificación del tiempo de sonicación o la intensidad también puede aumentar la concentración de la proteína de longitud completa pronosticada.
Las partículas redobladas estaban en el rango de tamaño esperado (20 a 100 nm)8,24,25 según lo determinado por el ANÁLISis de seguimiento de DLS y nanopartículas. Estos resultados se confirmaron aún más mediante el uso de TEM. Si hay problemas en este paso, normalmente se debe a un problema con el pH o la fuerza iónica del búfer de redoblamiento. Cuando se detectan partículas de gran tamaño en las técnicas de tamaño de partículas, indica la agregación, que se puede evitar aumentando el pH del búfer de redoblamiento. Si las partículas no son detectadas por DLS, verifique la concentración de la proteína y verifique el pH del tampón. La concentración final de proteína para DLS debe ser de al menos 100 g/ml. Si la concentración no es el problema, indica la abundancia de partículas pequeñas e incompletamente formadas, cuya concentración puede reducirse disminuyendo el pH. Alternativamente, la concentración de cloruro de sodio se puede ajustar al rango óptimo para minimizar la presencia de partículas con tamaño no deseado.
Por último, mediante el uso de un paso de lavado de isopropanol durante la purificación pudimos reducir la contaminación del LPS desde el huésped E. coli a 0,01 UE / g de SAPN que está por debajo del límite de la Administración de Alimentos y Medicamentos (FDA) de 5 UE/kg de peso corporal para productos inyectables 28. Este nivel se puede reducir aún más mediante el uso de una columna de intercambio de aniones también conocida como columna Q. Si todavía hay altos niveles de endotoxina, compruebe todos los materiales que se utilizaron para la preparación de tampón. Recuerde utilizar sólo cristalería despirogenada y cristalería libre de endotoxinas en este método.
Estos resultados indican que hemos desarrollado con éxito un método para producir el núcleo SHB-SAPN que se puede utilizar para estudios de inmunización preclínica. Este método con sólo ligeras modificaciones, si las hay, se puede aplicar a la purificación de SHB-SAPNs cuando se añade un antígeno de interés. El uso de este método como punto de partida uno de los cambios principales se encuentra en el paso de elución. Diferentes proteínas eluyen a diferentes concentraciones de imidazol que deben determinarse experimentalmente. La otra diferencia importante podría ser la composición del búfer de redoblamiento. La optimización requeriría probar diferentes condiciones de pH, así como fortalezas iónicas.
Teniendo en cuenta el trabajo futuro, sólo se necesitan dos ligeras modificaciones para permitir la aplicación humana del SHB-SAPN. La primera es que el vector de expresión necesita ser cambiado a un marcador seleccionable de resistencia a la kanamicina debido a la alergia a la ampicilina en los seres humanos29. El otro requisito importante de la fabricación de proteínas para uso humano es producir el SHB-SAPN en medios libres de productos animales. Un estudio a pequeña escala ya indicó un rendimiento razonable de proteína en un medio de base vegetal. El trabajo presentado aquí es fácilmente escalable para la producción definitiva de GMP como se ha demostrado con un candidato a la vacuna contra el paludismo, FMP01416. Esta producción FMP014-SAPN a gran escala incluía tanto el intercambio de aniones como los pasos de intercambio catiónico para reducir aún más el contenido de LPS y Ni2+ del producto final. Este SAPN con expreses bacterianos ya se ha ampliado para un próximo ensayo clínico de Fase 1/2a.
Divulgaciones
Las opiniones expresadas son las de los autores y no deben interpretarse como las posiciones del Ejército de los Estados Unidos o del Departamento de Defensa. Peter Burkhard tiene un interés en la empresa llamada Alpha-O Peptides AG y tiene patentes sobre la tecnología. Los demás autores no tienen afiliación o participación financiera con ninguna empresa con un interés financiero en el tema presentado en este documento.
Agradecimientos
Este trabajo fue apoyado por un acuerdo de cooperación (W81XWH-11-2-0174) entre la Fundación Henry M Jackson para el Avance de la Medicina Militar, Inc., y el Departamento de Defensa de los Estados Unidos. El anticuerpo anti-VIH-1 gp41 mAb 167-D IV fue recibido de la Dra. Susan Zolla-Pazner a través del Programa de Reactivos de SIDA de NIH.
Materiales
| Name | Company | Catalog Number | Comments |
| 10x Tris/Glycine/SDS | BioRad | 1610732 | 1 L |
| 2-Mercaptoethanol | BioRad | 1610710 | 25 mL |
| 2-propanol | Fisher | BP26181 | 4 L |
| 2x Laemmli Sample Buffer | BioRad | 1610737 | 30 mL |
| 40ul Cuvette Pack of 100 with Stoppers | Malvern Panalytical | ZEN0040 | 100 pack |
| 4–20% Mini-PROTEAN TGX Precast Protein Gels, 10-well, 30 µl | BioRad | 4561093 | 10 pack |
| Ampicillin | Fisher | BP1760-25 | 25 g |
| Anti-6X His tag antibody [HIS.H8] | AbCam | ab18184 | 100 mg |
| Anti-HIV-1 gp41 Monoclonal (167-D IV) | AIDS Reagent Repository | 11681 | 100 mg |
| BCIP/NBT Substrate, Solution | Southern Biotech | 0302-01 | 100 mL |
| Corning Disposable Vacuum Filter/Storage Systems | Fisher | 09-761-108 | A variety of sizes |
| Formvar/Carbon 400 mesh, Copper approx. grid hole size: 42µm | Ted Pella, Inc | 01754-F | 25 pack |
| GE Healthcare 5 mL HisTrap HP Prepacked Columns | GE HealthCare | 45-000-325 | 5 pack |
| Glycerol | Fisher | BP229-4 | 4 L |
| Goat Anti-Mouse IgG H&L (Alkaline Phosphatase) | ABCam | ab97020 | 1 mg |
| Imidazole | Fisher | O3196-500 | 500 g |
| Instant NonFat Dry Milk | Quality Biological | A614-1003 | 10 pack |
| Kinetic-QCL Kinetic Chormogenic LAL Assay | Lonza Walkersville | 50650U | 192 Test Kit |
| LAL Reagent Grade Multi-well Plates | Lonza Walkersville | 25-340 | 1 plate |
| Magic Media E. coli Expression Medium | ThermoFisher | K6803 | 1 L |
| MilliporeSigma Millex Sterile Syringe 0.22 mm Filters | Millipore | SLGV033RB | 250 pack |
| Mouse Anti-Human IgG Fc-AP | Southern Biotech | 9040-04 | 1.0 mL |
| One Shot BL21 Star (DE3) Chemically Competent E. coli | ThermoFisher | C601003 | 20 vials |
| Precision Plus Protein Unstained Protein Standards, Strep-tagged recombinant, | BioRad | 1610363 | 1 mL |
| Slide-A-Lyzer Dialysis Cassettes, 10K MWCO, 12 mL | ThermoFisher | 66810 | 8 pack |
| Sodium Chloride | Fisher | BP358-212 | 2.5 kg |
| Sodium Phosphate Monobasic | Fisher | BP329-500 | 500 g |
| Tris Base | Fisher | BP152-1 | 1 kg |
| Tris-(2-carboxyethyl)phosphine hydrochloride | Biosynth International | C-1818 | 100 g |
| Uranyl Acetate, Reagent, A.C.S | Electron Micoscopy Services | 541-09-3 | 25 g |
| Urea | Fisher | BP169-500 | 2.5 kg |
| Whatman qualitative filter paper | Sigma Aldrich | WHA10010155 | pack of 500 |
| Name | Company | Catalog Number | Comments |
| Equipment | |||
| ChromLab Software ver 4 | BioRad | 12009390 | Software |
| Epoch 2 Microplate Spectrophotometer | BioTek | EPOCH2 | Plate Reader |
| Fiberlite F14-14 x 50cy Fixed-Angle Rotor | ThermoFisher | 096-145075 | Rotor |
| Gel Doc EZ Gel Documentation System | BioRad | 1708270 | Gel Imager for Stain free Gels |
| JEOL TEM | JEOL | 1400 | Transmission Electron Microscope |
| Mini-PROTEAN Tetra Vertical Electrophoresis Cell for Mini Precast Gels | BioRad | 1658004 | To run gels |
| NanoDrop One Microvolume UV-Vis Spectrophotometer | ThermoFisher | ND-ONE-W | For Protein Concentration |
| NanoSight NS300 | Malvern Panalytical | Particle Sizing | |
| NanoSight NTA software NTA | Malvern Panalytical | Particle Sizing | |
| New Brunswick Innova 44/44R | Eppendorf | M1282-0000 | Incubator/Shaker |
| NGC Quest 10 Chromatography System | BioRad | 7880001 | FPLC to aid in protein purification |
| PELCO easiGlow Glow Discharge Cleaning System | Ted Pella, INC | 91000S | To clean grids |
| PowerPac Universal Power Supply | BioRad | 1645070 | To run gels |
| Rocker Shaker | Daigger | EF5536A | For Western |
| Sonifer 450 | Branson | also known as 096-145075 | Sonicator |
| Thermo Scientific Sorvall LYNX 4000 Superspeed Centrifuge | ThermoFisher | 75-006-580 | Centrifuge |
| Trans-Blot Turbo Mini Nitrocellulose Transfer Packs | BioRad | 1704158 | For Western |
| Trans-Blot Turbo Transfer System | BioRad | 1704150 | For Western |
| Vortex-Genie 2 | Daigger | EF3030A | Vortex |
| Zetasizer Nano ZS | Malvern Panalytical | Particle Sizing | |
| Zetasizer Software | Malvern Panalytical | Particle Sizing |
Referencias
- Karch, C. P., Burkhard, P. Vaccine technologies: From whole organisms to rationally designed protein assemblies. Biochemical Pharmacology. 120, 1-14 (2016).
- Snapper, C. M. Distinct Immunologic Properties of Soluble Versus Particulate Antigens. Frontiers in Immunology. 9, 598(2018).
- Kelly, H. G., Kent, S. J., Wheatley, A. K. Immunological basis for enhanced immunity of nanoparticle vaccines. Expert Review of Vaccines. , 1-12 (2019).
- Yeates, T. O. Geometric Principles for Designing Highly Symmetric Self-Assembling Protein Nanomaterials. Annual Review of Biophysics. 46, 23-42 (2017).
- Marcandalli, J., et al. Induction of Potent Neutralizing Antibody Responses by a Designed Protein Nanoparticle Vaccine for Respiratory Syncytial Virus. Cell. 176 (6), 1420-1431 (2019).
- Ross, J. F., et al. Decorating Self-Assembled Peptide Cages with Proteins. ACS Nano. 11 (8), 7901-7914 (2017).
- Karch, C. P., Matyas, G. R., Burkhard, P., Beck, Z. Self-Assembling Protein Nanoparticles: implications for HIV-1 vaccine development. Nanomedicine (Lond). 13 (17), 2121-2125 (2018).
- Raman, S., Machaidze, G., Lustig, A., Aebi, U., Burkhard, P. Structure-based design of peptides that self-assemble into regular polyhedral nanoparticles). Nanomedicine. 2 (2), 95-102 (2006).
- Karch, C. P., et al. Design and characterization of a self-assembling protein nanoparticle displaying HIV-1 Env V1V2 loop in a native-like trimeric conformation as vaccine antigen. Nanomedicine. , (2018).
- Karch, C. P., et al. The use of a P. falciparum specific coiled-coil domain to construct a self-assembling protein nanoparticle vaccine to prevent malaria. Journal of Nanobiotechnology. 15 (1), 62(2017).
- Li, J., et al. A self-adjuvanted nanoparticle based vaccine against infectious bronchitis virus. PLoS One. 13 (9), e0203771(2018).
- Wahome, N., et al. Conformation-specific display of 4E10 and 2F5 epitopes on self-assembling protein nanoparticles as a potential HIV vaccine. Chemical Biology & Drug Design. 80 (3), 349-357 (2012).
- El Bissati, K., et al. Effectiveness of a novel immunogenic nanoparticle platform for Toxoplasma peptide vaccine in HLA transgenic mice. Vaccine. 32 (26), 3243-3248 (2014).
- Kaba, S. A., et al. A nonadjuvanted polypeptide nanoparticle vaccine confers long-lasting protection against rodent malaria. Journal of Immunology. 183 (11), 7268-7277 (2009).
- Kaba, S. A., et al. Protective antibody and CD8+ T-cell responses to the Plasmodium falciparum circumsporozoite protein induced by a nanoparticle vaccine. PLoS One. 7 (10), e48304(2012).
- Seth, L., et al. Development of a self-assembling protein nanoparticle vaccine targeting Plasmodium falciparum Circumsporozoite Protein delivered in three Army Liposome Formulation adjuvants. Vaccine. 35 (41), 5448-5454 (2017).
- Kaba, S. A., et al. Self-assembling protein nanoparticles with built-in flagellin domains increases protective efficacy of a Plasmodium falciparum based vaccine. Vaccine. 36 (6), 906-914 (2018).
- El Bissati, K., et al. Protein nanovaccine confers robust immunity against Toxoplasma. NPJ Vaccines. 2, 24(2017).
- Karch, C. P., et al. Vaccination with self-adjuvanted protein nanoparticles provides protection against lethal influenza challenge. Nanomedicine. 13 (1), 241-251 (2017).
- Haynes, B. F., et al. Immune-correlates analysis of an HIV-1 vaccine efficacy trial. New England Journal of Medicine. 366 (14), 1275-1286 (2012).
- Rerks-Ngarm, S., et al. Vaccination with ALVAC and AIDSVAX to prevent HIV-1 infection in Thailand. New England Journal of Medicine. 361 (23), 2209-2220 (2009).
- O' Connell, R. J., Kim, J. H., Excler, J. L. The HIV-1 gp120 V1V2 loop: structure, function and importance for vaccine development. Expert Review of Vaccines. 13 (12), 1489-1500 (2014).
- Babapoor, S., et al. A Novel Vaccine Using Nanoparticle Platform to Present Immunogenic M2e against Avian Influenza Infection. Influenza Research and Treatment. 2011, 126794(2011).
- Indelicato, G., Burkhard, P., Twarock, R. Classification of self-assembling protein nanoparticle architectures for applications in vaccine design. Royal Society Open Science. 4 (4), 161092(2017).
- Indelicato, G., et al. Principles Governing the Self-Assembly of Coiled-Coil Protein Nanoparticles. Biophysical Journal. 110 (3), 646-660 (2016).
- Doll, T. A., Raman, S., Dey, R., Burkhard, P. Nanoscale assemblies and their biomedical applications. Journal of the Royal Society Interface. 10 (80), 20120740(2013).
- Rey, F. A., Lok, S. M. Common Features of Enveloped Viruses and Implications for Immunogen Design for Next-Generation Vaccines. Cell. 172 (6), 1319-1334 (2018).
- Bacterial Endotoxins. United States Pharmacopeia (USP). , Ch. 85 (2011).
- Points to Consider (PTC) in the Characterization of Cell Lines Used to Produce Biologicals. , Food and Drug Adminstration. Maryland. Available from: https://www.fda.gov/media/76255/download (1993).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados