Method Article
Herstellung von E. coli-exprimierenden selbstmontierenden Protein-Nanopartikeln für Impfstoffe, die eine trimerische Epitop-Präsentation erfordern
In diesem Artikel
Zusammenfassung
Hier wird eine detaillierte Methode zur Beschreibung der Reinigung, Umfaltung und Charakterisierung selbstassemblierender Protein-Nanopartikel (SAPNs) für die Impfstoffentwicklung beschrieben.
Zusammenfassung
Selbstzusammenstellung sammle Protein-Nanopartikel (SAPNs) fungieren als repetitive Antigen-Displays und können zur Entwicklung einer breiten Palette von Impfstoffen gegen verschiedene Infektionskrankheiten verwendet werden. In diesem Artikel zeigen wir eine Methode zur Herstellung eines SAPN-Kerns, der eine Sechs-Helix-Bundle(SHB)-Baugruppe enthält, die Inderantigene in einer trimerischen Konformation darstellen kann. Wir beschreiben die Expression des SHB-SAPN in einem E. coli-System sowie die notwendigen Proteinreinigungsschritte. Wir haben einen Isopropanol-Waschschritt aufgenommen, um das verbleibende bakterielle Lipopolysaccharid zu reduzieren. Als Hinweis auf die Proteinidentität und Reinheit reagierte das Protein mit bekannten monoklonalen Antikörpern in Western-Blot-Analysen. Nach dem Umfalten fiel die Größe der Teilchen in den erwarteten Bereich (20 bis 100 nm), was durch dynamische Lichtstreuung, Nanopartikel-Tracking-Analyse und Transmissionselektronenmikroskopie bestätigt wurde. Die hier beschriebene Methodik ist für den SHB-SAPN optimiert, kann jedoch nur geringfügig auf andere SAPN-Konstrukte angewendet werden. Diese Methode ist auch leicht auf die Großproduktion für die GMP-Herstellung für Menschliche Impfstoffe übertragbar.
Einleitung
Während sich die traditionelle Impfstoffentwicklung auf die inaktivierten oder abgeschwächten Krankheitserreger konzentrierte, hat sich der Fokus moderner Impfstoffe auf Untereinheitsimpfstoffe verlagert1. Dieser Ansatz kann zu einer gezielteren Reaktion und potenziell wirksameren Impfstoffkandidaten führen. Einer der Hauptnachteile ist jedoch, dass Impfstoffe aus Untereinheiten keine Partikel wie ganze Organismen sind, was zu einer reduzierten Immunogenität führen kann2. Ein Nanopartikel als repetitives Antigen-Anzeigesystem kann sowohl die Vorteile des zielgerichteten Impfstoffansatzes der Untereinheit als auch die Partikelart des gesamten Organismus1,3haben.
Unter den bestehenden Arten von Nanoimpfstoffen ermöglichen rationell gestaltete Proteinassemblys das Design und die Entwicklung von Impfstoffkandidaten, die mehrere Kopien des Antigens möglicherweise in einer nativen Konformium darstellen können1,4 ,5,6. Ein Beispiel für diese Proteinassemblys sind die selbstzusammenstellungsreichenden Protein-Nanopartikel (SAPNs)7. SAPNs basieren auf Coiled-Coil-Domains und werden traditionell in Escherichia coli8ausgedrückt. SAPN-Impfstoffkandidaten wurden für eine Vielzahl von Krankheiten wie Malaria, SARS, Grippe, Toxoplasmose und HIV-19,10,11,12,13 entwickelt. , 14 , 15 , 16 , 17 , 18 , 19. Die Konzeption jedes SAPN-Kandidaten ist spezifisch für den Pathogen von Interesse, jedoch sind die Herstellungs-, Reinigungs- und Umfaltungstechniken im Allgemeinen anwendbar.
Eines unserer aktuellen Interessen ist ein wirksamer HIV-1-Impfstoff. In RV144 – der einzigen klinischen Phase-III-Studie eines HIV-1-Impfstoffs, die eine bescheidene Wirksamkeit zeigte – korrelierte das reduzierte Infektionsrisiko mit IgG-Antikörpern mit der V1V2-Schleife des Hüllproteins20,21. Die native trimerische Darstellung dieser Region gilt als wichtig für die schützende Immunogenität22. Um die V1V2-Schleife möglichst nah an der nativen konformen Konformation zu präsentieren, haben wir einen Proof of Principle SAPN-Impfstoffkandidaten entwickelt, der den HIV-1-Hüllumschlag nach der Fusion (SHB) enthielt, um die V1V2-Schleife in die richtige Konformation 9 zu präsentieren. . Dieser Kandidat wurde von bekannten monoklonalen Antikörpern gegen HIV-1-Hüllkurvenprotein erkannt. Mäuse, die mit V1V2-SHB-SAPN immunisiert wurden, hoben V1V2-spezifische Antikörper auf, die vor allem an gp70 V1V2, die richtigen Konformalepitope9gebunden sind. Der SHB-SAPN-Kern könnte über die Rolle als Träger der HIV-1 V1V2-Schleife hinaus andere Funktionen haben. Hier beschreiben wir eine detaillierte Methodik für den Ausdruck, die Reinigung, das Umfalten und die Validierung des SHB-SAPN-Kerns. Die Sequenzauswahl, das Nanopartikeldesign, das molekulare Klonen und die Transformation von E. coli wurden zuvor beschrieben9.
Protokoll
1. Expression des SHB-SAPN-Proteins in E. coli BL21(DE3)
- 95 ml des Bauteils A und 5 ml des Bestandteils B des Mediums in einem 2 L sterilen Glas Erlenmeyer Kolben nach Herstelleranleitung mischen (siehe Materialtabelle). Fügen Sie Ampicillin zu einer Endkonzentration von 100 g/ml hinzu.
- Impfen Sie die Medien mit E. coli aus einer zuvor etablierten Glycerin-Aktienkultur. Inkubationskultur bei 30 °C mit Schütteln bei 200 Umdrehungen pro Minute (Rpm) für 48 h.
HINWEIS: Der verwendete E. coli BL21 (DE3) Bestand enthielt den Ampicillinresistenten Expressionsvektor23 mit dem SHB-SAPN-Gen. Obwohl das allgemeine Protokoll der Medien 24 h Inkubation bei 37 °C empfiehlt, ergaben 48 h Inkubation bei 30 °C eine höhere Ausbeute für SHB-SAPN. - Übertragen Sie die Kultur auf zwei 50 ml konische Röhren. Zentrifugieren Sie die Rohre bei 4.000 x g für 10 min mit einem festen Winkelrotor bei 4 °C. Entfernen Sie den Überstand und speichern Sie das Pellet, um Zellen zu ernten.
HINWEIS: Das Zellpellet kann entweder sofort verarbeitet oder bei -80 °C bis zur Verwendung eingefroren werden.
2. Lyse von E. coli BL21(DE3) durch Beschallung
HINWEIS: Verwenden Sie nichtpyrogene Kunststoffe und Glaswaren, die bei 250 °C für mindestens 30 min gebacken werden. Tris(2-Carboxyethyl)phosphin (TCEP) als Reduktionsmittel bricht die Disulfidbindungen innerhalb und zwischen Proteinen. TCEP ist in den Puffern während dieses Protokolls notwendig, wenn das angezeigte Antigen S-S-Bindungen enthält. Nur für den SHB-SAPN-Kern ist das Vorhandensein von TCEP in den Puffern nicht unbedingt erforderlich.
- Imidazol-freier Puffer (8 M Harnstoff, 50 mM Natriumphosphat monobasic, 20 mM Tris-Basis, 5 mM TCEP) pH 8.0 (angepasst mit 5 N NaOH) vorbereiten und mit einer 0,22 m Vakuumflaschenfiltrationseinheit filtern.
- Die Pelletzellen (ab Schritt 1.3) mit 40 ml Imidazol-freier Puffer in einem 50 ml konischen Rohr wieder aufsetzen. Beschallen Sie die resuspendierten Zellen mit einer Sonde auf Eis für 5 min (4 s Beschallung, 6 s Ruhe) mit einer Beschallungsleistung von 150 W.
- Zentrifugieren Sie das Zelllysat (40 ml) bei 29.000 x g bei 4 °C für 25 min in einem festen Winkelrotor, um geklärten Überstand zu erzeugen. Den Überstand in einen 150 ml sterilen Kolben geben und das Pellet entsorgen. Verdünnen Sie den Überstand auf 100 ml mit dem Imidazol-freien Puffer (später im Protokoll, das als "Probe" bezeichnet wird).
HINWEIS: Dieser Verdünnungsschritt ist erforderlich, um zu verhindern, dass der Druck des FPLC-Systems während der Lysatbelastung der Säule zu hoch wird.
3. Proteinreinigung mit einer His-Säule
HINWEIS: Dieses Protokoll wurde mit einem FPLC-Instrument durchgeführt, kann aber an den Gravitationsfluss angepasst werden.
- Bereiten Sie die folgenden Puffer vor und filtern Sie sie mit einer 0,22 m-Vakuumflaschenfiltrationseinheit: (i) Imidazol-freier Puffer "Puffer A" (8 M Harnstoff, 50 mM Natriumphosphat monobasic, 20 mM Tris-Basis, 5 mM TCEP) pH 8.0; ii) 500 mM Imidazolpuffer "Puffer B" (8 M Harnstoff, 50 mM Natriumphosphat monobasic, 20 mM Tris-Basis, 5 mM TCEP, 500 mM Imidazol) pH 8,0; und (iii) Isopropanolwäsche (20 mM Tris, 60% Isopropanol) pH 8.0.
HINWEIS: der pH-Wert für jeden Puffer wurde mit 5 N NaOH eingestellt. - Gleichgewichte die His-Kolumne.
HINWEIS: Für die Produktion im Labormaßstab verwendet dieses Protokoll eine vorverpackte His-Spalte mit einer Größe von 5 ml, aber jede größere Spalte kann verwendet werden.- Öffnen Sie die FPLC-Software und klicken Sie auf die Option Neue Methode. Es wird sofort zum Menü "Methodeneinstellungen" geöffnet. Wählen Sie im Dropdown-Menü für Spaltenposition C1-Port 3.
- Wählen Sie im Dropdown-Menü Gezeigt nach Technik Affinitätaus. Wählen Sie im Dropdown-Menü "Spalte" andere aus, Histrap HP, 5 ml. Das Spaltenvolumen und die Druckkästen werden automatisch auf die entsprechenden Werte gesetzt.
- Klicken Sie auf die Schaltfläche "Methode". Ziehen Sie die folgenden Schaltflächen aus dem Popupmenü der Phasenbibliothek: Ausgleich, Beispielanwendung, Spaltenwäscheund Elution neben dem Pfeil in genau dieser Reihenfolge. Schließen Sie das Menü Phasenbibliothek.
- Klicken Sie auf die Schaltfläche "Ausgleich". Die in der Tabelle aufgeführten Werte sollten "Anfangspuffer B" (4 %), "Endpuffer B" (4 %) und "Volume (CV)" 5 sein.
- Klicken Sie auf das Anwendungsfeld Beispiel. Klicken Sie in der Musterladebox auf das Optionsfeld für Inject Sample on column with sample pump. Stellen Sie sicher, dass das Kästchen neben dem Durchfluss von Methodeneinstellungen verwenden in der Probeninjektion mit Systempumpenbox aktiviert ist. Ändern Sie neben dem Lautstärkefeld auf der rechten Seite des Bildschirms den Wert auf 20 ml.
- Klicken Sie auf die Schaltfläche Spaltenwäsche. Die in der Tabelle aufgeführten Werte sollten "Anfangspuffer B" (4 %), "Endpuffer B" (4 %) und "Volume (CV)" 5 sein. Klicken Sie neben dem Bruchsammlungsschema auf das Feld Aktivieren.
- Klicken Sie auf die Elution-Schaltfläche. Die in der Tabelle aufgeführten Werte sollten "Anfangspuffer B" 0%, "Endpuffer B" 100% und "Volume (CV)" 5 sein. Klicken Sie neben dem Brucherfassungsschema auf das Feld Aktivieren. Klicken Sie im Einfüllfeld unten auf die Wertfraktionsgröße aus Methodeneinstellungen verwenden, und passen Sie die Bruchgröße auf 4 ml an.
- Klicken Sie auf die Schaltfläche Speichern als oben in der Software. Benennen Sie die Datei "Ausgleich".
- Schließen Sie eine 5 ml vorverpackte His-Säule an den entsprechenden Säulenanschluss 3 auf dem FPLC an. Sowohl Pumpe A- und Pumpen-B-Schläuche als auch die Probenpumpenschläuche sollten in 0,22 m gefiltertes deionisiertes Wasser gelegt werden. Führen Sie das Ausgleichsprogramm aus.
- Legen Sie sowohl Pumpe A als auch Pumpe B sowie den Probenpumpenschlauch in den Imidazol-freien Puffer (Puffer A) und führen Sie das Ausgleichsprotokoll erneut aus.
- Binden Sie die Probe an die Säule und reinigen Sie das Protein.
- Öffnen Sie die FPLC-Software und klicken Sie auf die Option Neue Methode. Es wird sofort zum Menü "Methodeneinstellungen" geöffnet. Wählen Sie im Dropdown-Menü für Spaltenposition C1-Port 3. Wählen Sie im Dropdown-Menü Gezeigt nach Technik Affinitätaus. Wählen Sie im Dropdown-Menü des Spaltentyps andere aus, Histrap HP, 5 ml. Das Spaltenvolumen und die Druckkästen werden automatisch auf die entsprechenden Werte gesetzt.
- Klicken Sie auf die Schaltfläche "Methode". Ziehen Sie die Schaltflächen aus dem Popup-Menü phase-Bibliothek: Ausgleich, Beispielanwendung, Spaltenwäsche (Wash 1), Spaltenwäsche (Wash 2), Spaltenwäsche (Wash 3) und Elution neben dem Pfeil in diesem genaue Reihenfolge. Schließen Sie das Menü Phasenbibliothek.
- Klicken Sie auf die Schaltfläche "Ausgleich". Die in der Tabelle aufgeführten Werte sollten "Anfangspuffer B" 4%, "Endpuffer B" 4% und "Volume (CV)" 5 sein.
- Klicken Sie auf das Anwendungsfeld Beispiel. Klicken Sie in der Musterladebox auf das Optionsfeld für Inject Probe auf Spalte mit Probenpumpe. Stellen Sie sicher, dass das Kästchen neben dem Durchfluss von Methodeneinstellungen verwenden in der Probeninjektion mit Systempumpenbox aktiviert ist.
- Ändern Sie neben dem Lautstärkefeld auf der rechten Seite des Bildschirms den Wert auf 100 ml. Klicken Sie neben dem Brucherfassungsschema auf die Schaltfläche Aktivieren. Deaktivieren Sie das Feld Bruchgröße aus Methodeneinstellungen verwenden, und ändern Sie dann die Bruchgröße auf 4 ml.
- Klicken Sie auf die erste Spaltenwaschtaste (Wash 1). Die in der Tabelle aufgeführten Werte sollten "Anfangspuffer B" 4%, "Endpuffer B" 4% und "Volume (CV)" 10 sein. Klicken Sie neben dem Bruchsammlungsschema auf das Feld Aktivieren. Klicken Sie auf die Größe der Fraktion verwenden aus den Methodeneinstellungen, und ändern Sie dann die Bruchgröße auf 4 ml.
- Klicken Sie auf die zweite Spalte Waschtaste (Wash 2). Die in der Tabelle aufgeführten Werte sollten "Anfangspuffer B" 0%, "Endpuffer B" 0% und "Volume (CV)" 5 sein. Klicken Sie neben dem Bruchsammlungsschema auf das Feld Aktivieren. Klicken Sie auf die Größe der Fraktion verwenden aus den Methodeneinstellungen, und ändern Sie dann die Bruchgröße auf 4 ml.
- Klicken Sie auf die dritte Spalte Waschtaste (Wash 3). Die in der Tabelle aufgeführten Werte sollten "Anfangspuffer B" 0%, "Endpuffer B" 0% und "Volume (CV)" 5 sein. Klicken Sie neben dem Bruchsammlungsschema auf Aktivieren. Deaktivieren Sie das Feld Bruchgröße aus Methodeneinstellungen verwenden, und ändern Sie dann die Bruchgröße auf 4 ml.
- Klicken Sie auf die Elution-Schaltfläche. Klicken Sie in der Tabelle mit der rechten Maustaste auf die aufgeführten Informationen, und klicken Sie im angezeigten Menü auf Schritt löschen. Ziehen Sie die isokratische Farbverlaufsschaltfläche zweimal auf die Tabelle, sodass es zwei Einträge gibt.
- Der Wert für den ersten Eintrag sollte "Anfangspuffer B" 30%, "Endpuffer B" 30% und "Volume (CV)" 10 lauten. Der Wert für den zweiten Eintrag sollte "Anfangspuffer B" 100%, "Endpuffer B" 100% und "Volume (CV)" 10 lauten. Klicken Sie neben dem Brucherfassungsschema auf die Schaltfläche Aktivieren. Klicken Sie auf das Feld neben Bruchbruchgröße aus Methodeneinstellungenverwenden .
- Klicken Sie oben auf der Software auf die Schaltfläche Speichern unter. Benennen Sie die Datei "Reinigung". Pumpe Ein Schlauch des FPLC sollte in den imidazolfreien Waschpuffer gelegt werden, während Pumpe B-Schläuche in den 500 mM Imidazolpuffer gelegt werden sollten. Der Probenpumpenschlauch sollte in die 100 ml-Probe gelegt werden.
- Führen Sie das Programm "Reinigung" aus und warten Sie auf die Zeit, zu der das 60% Isopropanol benötigt wird (Wash 2). Halten Sie das Programm an, bewegen Sie Pumpe A Schläuche aus der Imidazol-freien Wäsche in die 60% Isopropanol-Waschanlage. Starten Sie das Programm neu.
- Sobald der Isopropanol-Schritt abgeschlossen ist, halten Sie das Programm erneut an und bewegen Sie Pumpe A-Schläuche zurück in den Imidazol-freien Waschpuffer. Starten Sie das Reinigungsprogramm neu (der Rest des Laufs ist automatisiert).
4. Reinheitsbewertung und Proteinidentifikation durch SDS-PAGE
- Kombinieren Sie alle Fraktionen, die (i) durchfließen (das Zelllysat, das nicht an die Seine Säule gebunden ist), (ii) Waschen 1, (iii) Waschen 3 (60% Isopropanolwäsche) und (iv) Waschen 3 in separaten 50 ml konischen Röhren. Kombinieren Sie die 2 ml-Fraktionen aus den Elutionsschritten nicht.
- Mischen Sie 15 l aus jeder der gepoolten Fraktionen und alle Fraktionen aus Elutionsschritten mit 2x Laemmli Probenpuffer in einem 0,5 ml Mikrozentrifugenrohr und denaturieren Sie sie bei 95 °C für 10 min.
- Während das Protein denaturiert, richten Sie das Gellaufgerät mit 3 fleckenfreien 4–20% vorgefertigten Polyacrylamid-Gelen in 1x Tris-Glycin SDS-PAGE Laufpuffer ein.
- Laden Sie 8 l Molekulargewichtsmarker auf den ersten Brunnen und 30 l denaturierte Probe auf die anderen Brunnen des Gels. Führen Sie die Gele bei 200 V, bis die Farbstofffront auf den Boden des Gels trifft (ca. 30 min). Entfernen Sie die Gele aus dem Gerät und spülen Sie kurz mit entionisiertem Wasser ab. Stellen Sie das Gel sofort mit dem fleckenfreien Bildgebungssystem ab.
- Identifizieren Sie die Fraktionen, die Proteinbänder mit der richtigen Größe (18,07 kDa) enthalten. Bündeln Sie alle diese Brüche.
5. Proteinidentifikation durch Western Blot
- Führen Sie einen westlichen Blot mit einem His-spezifischen Antikörper (Anti-6x HisTag) und einem SHB-spezifischen Antikörper (167-D-IV) aus, um das gereinigte Voll-Längenprotein zu identifizieren. Der Anti-6x HisTag Antikörper erkennt die N-Endstation des Proteins und der 167-D-IV-Antikörper erkennt die C-Endstation, die das Vorhandensein des vollfänglich enthatonischen Proteins demonstriert.
- Bestimmen Sie die Proteinkonzentration des (i) Durchflusses, (ii) Waschen 1, (iii) Waschen 2 (60% Isopropanolwäsche), (iv) Wash 3, und alle Fraktionen aus den Elutionsschritten mit dem Spektrometer-Instrument bei einer Absorption von 280 nm. Generieren Sie Verdünnungen, die 100 ng Protein in 15 L Imidazol-freien Puffer für jede dieser Gruppen enthalten.
- Fügen Sie zu jeder 15-L-Probe 15 L des 2x Laemmli-Probenpuffers hinzu und denaturieren Sie sie wie in Schritt 4.1. Sobald die Denaturierung abgeschlossen ist, drehen Sie die Röhrchen nach unten, um sicherzustellen, dass das gesamte Protein übertragen werden kann.
- Proben und den vorgefärbten Marker in ein fleckenfreies 4–20% vorgefertigtes Polyacrylamidgel einteilen. Führen Sie die Elektrophorese bei 200 V aus, bis die Farbfront den Boden des Gels erreicht.
- Während das Gel läuft, machen Sie 1 L TBS-T (20 mM Tris, 150 mM NaCl und 0,1% Tween 20) und 200 ml 5% fettfreie Milch in TBS-T.
- Verwenden Sie ein Western Blot Transfersystem, um Protein auf eine Nitrocellulosemembran zu übertragen. Verwenden Sie einen vormontierten Transferstapel und legen Sie das Gel darauf. Richten Sie das System auf 25 V für 7 min. Überprüfen Sie, ob der vorgefärbte Marker auf der Nitrozellulosemembran angezeigt wird, was auf eine vollständige Übertragung hinweist.
HINWEIS: Alle nachfolgenden Schritte werden auf einem Orbital-Shaker ausgeführt, der bei Raumtemperatur (RT) bei 100 Umdrehungen pro Minute eingestellt ist. - Sobald der Transfer abgeschlossen ist, spülen Sie Blots zweimal mit TBS-T für jeweils 10 min.
- Die Nitrocellulosemembranen (Blot) mit 5% fettfreier Milch in TBS-T für mindestens 1 h blockieren. Blots zweimal mit TBS-T für jeweils 10 min waschen.
- Verdünnen Sie die primären 167-D-IV- und Anti-6x HisTag-Antikörper auf 1 mg/ml in TBS-T (Lager Ab). Verdünnen Sie die Lager Abs des 167-D-IV 10.000-fachen und des Anti-6x HisTag 5.000-fach, indem Sie 2 l des Bestands 167-D-IV und 4 l des Lagers anti-6x HisTag Antikörper zu zwei verschiedenen Röhren hinzufügen, die 20 ml TBS-T enthalten. Fügen Sie das gesamte 20 ml Volumen der primären Antikörper, einen zu jedem Fleck, und brüten Flecken für 1 h. Blots zweimal mit TBS-T für 10 min waschen.
- Verdünnung von 4 l von 1 mg/ml des mausantihumanen Sekundärantikörpers konjugiert mit alkalischer Phosphatase in 20 ml TBS-T (1:5.000 Verdünnung). Verdünnung von 4 l von 1 mg/ml des Ziegenanti-Maus-Sekundärantikörpers konjugiert mit alkalischer Phosphatase in 20 ml TBS-T (1:5.000 Verdünnung). Fügen Sie sekundäre Antikörper zu den entsprechenden Blots hinzu.
HINWEIS: Der antihumane Sekundärantikörper bindet an 167-D und die Anti-Maus an den Anti-6x HisTag. Blots zweimal mit TBS-T für 10 min waschen. - Fügen Sie genügend BCIP/NBT alkalische Phosphatase Substrat, um die Flecken zu decken. Entwickeln Sie die Flecken für ca. 10 min, bis Bänder auftreten. Spülen Sie Flecken mit kaltem Leitungswasser und lassen Sie sie trocknen, bevor Sie mit einem Flachbettscanner scannen.
6. Umfalten der SHB-SAPN
- Fügen Sie das gepoolte Protein (10-u201220 ml insgesamt) zu einer 10 kDa-Molekulargewichts-Abschneide-Dialysekassette hinzu und dialysieren Sie es über Nacht in 8 M Harnstoff, 20 mM Tris, 5% Glycerin, 5 mM TCEP pH 8,5 bei RT (18-u201226 °C).
- Dialysieren Sie den Harnstoff langsam aus der Probe, indem Sie die Harnstoffkonzentration im Dialysepuffer schrittweise um 2 M alle 2 h verringern. Bei einer Harnstoffkonzentration von 2 M das Dialysegerät auf 4 °C bewegen (verwenden Sie TCEP im Dialysepuffer aus diesem Schritt nicht). Beenden Sie die Umfaltung, indem Sie die Probe über Nacht in 120 mM Harnstoff, 20 mM Tris, 5% Glycerin, pH 8,5 bei 4 °C diagnosigen.
- Entfernen Sie das umgeklappte Protein (SHB-SAPN) aus der Dialysekassette. Filtern Sie den SHB-SAPN mit einem 0,22 m Polyvinylidenfluorid (PVDF) Spritzenfiler. Aliquot SHB-SAPN in sterile Schläuche ein- und gefrieren sie bei -80 °C, so dass für nachfolgende Analysen mindestens 100 l bei RT übrig bleiben.
7. Validierung von Partikeln nach Größe und Aussehen
- Dynamische Lichtstreuung (DLS)
- Messen Sie die mittlere Partikelgröße des SHB-SAPN anhand der folgenden Parameter: Protein als Material auswählen, einen komplexen Puffer für 120 mM Harnstoff, 20 mM Tris, 5% Glycerin 25 °C für die Temperatur erstellen, Einwegküvetten für die Analyse auswählen, automatische Messung, eingestellt auf 5 Durchläufe.
- Fügen Sie 45 L SHB-SAPN zu einer Einweg-Küvette hinzu und führen Sie die Software durch Klicken auf den grünen Pfeil aus. Wählen Sie das prozentuale Volumen für die Auslesung aus.
- Nanopartikel-Tracking-Analyse (NTA)
- Verdünnen Sie die Probe im Umfaltungspuffer um 1:20. Machen Sie 10 ml verdünnte Probe.
- Mit 3 ml Spritzen das NTA-Instrument mit dem Umfaltpuffer spülen und ca. 1,5 ml Probe belasten, um das Instrument auszueinemt. Verwenden Sie den Rest der Stichprobe für die Analyse.
- Erstellen Sie ein neues SOP in der NTA-Software, indem Sie auf die Registerkarte SOP klicken. Ändern Sie unter der Registerkarte die Anzahl der Aufnahmen auf 3 und ändern Sie die Aufnahmezeit auf 30 s. Drücken Sie die Autofokus-Taste auf der linken Seite des Bildschirms, um das Sample in den Fokus zu rücken. Verwenden Sie den manuellen Fokusknopf an der Seite der Maschine, um den Fokus zu optimieren.
- Führen Sie das erstellte SOP aus, wenn das System auffordert, ein kleines Volumen der Probe mit der Spritze zu beladen. Nachdem das System alle Aufnahmen gemacht hat, wird automatisch der Analysebildschirm angezeigt. Schieben Sie den Erfassungsbegrenzungsbalken so, dass alle realen Partikel mit roten Kreuzen markiert sind. Drücken Sie die Schaltfläche "Ausführungsanalyse", und die Analyse beginnt automatisch.
- Transmissionselektronenmikroskopie (TEM)
- Glühöffnung Formvar/Carbon 400 Mesh Kupfer TEM Stützfolien.
- Fügen Sie 3 l der Probe mit einer Konzentration von 0,075 mg/ml für 30 s ins Gitter. Wick aus der Flüssigkeit mit einem Filterpapier.
- Waschen Sie das Gitter dreimal mit 3 l entionisiertem Wasser, wobei das Wasser jedes Mal mit Filterpapier abgeleitet wird.
- Fügen Sie dem Stützfilm 3 l von 0,5% Uranylacetat hinzu und lassen Sie ihn 30 s lang sitzen. Wick den größten Teil des Uranylacetats abstoßen, aber einen dünnen Film auf der Oberfläche lassen. Lassen Sie die Proben trocknen, bevor Sie sie auf dem Transmissionselektronenmikroskop abbilden.
- Bildproben bei 80 kV auf einem TEM.
8. Bestimmung der Endotoxinspiegel in den Proben mit einem kinetischen Limulus-Amoebocytlysat (LAL)-Assay
- Entfernen Sie das Kit und die Proben aus dem Kühlschrank und lassen Sie rt ausdemalibrate.
- Um diesen Test durchzuführen, ist ein Plattenleser mit einem Wärmeblock und der Möglichkeit, die Proben für 40 Lesevorgänge bei einer Wellenlänge von 405 nm bei 37 °C zu lesen, erforderlich. Schreiben Sie eine Programmvorlage, damit alle 150 s Brunnen gelesen werden, um den Anfangszeitpunkt zu identifizieren (die OD erhöhte sich im Vergleich zum ersten Lesevorgang um 0,2).
- Verdünnen Sie die Probe auf die Impfdosiskonzentration in PBS.
HINWEIS: Der pH-Wert des Umfaltungspuffers liegt außerhalb des Bereichs des LAL-Assays. Die Verdünnung der Probe in PBS setzt den pH-Wert auf den akzeptablen Bereich. - Setzen Sie das Kontrollendotoxin in dem entsprechenden Volumen des endotoxinfreien LAL-Wassers, wie durch das Analysezertifikat zur Erzeugung von 50 EU/ml bestimmt, wieder aus. Die Durchstechflasche 15 min lang kräftig wirbeln, um eine vollständige Resuspension des Endotoxins zu gewährleisten.
- Generieren Sie die Endotoxin-Standardkurve, indem Sie eine 10-fache serielle Verdünnung in Glasfläschchen im Bereich von 50 EU bis 0,005 EU/ml vorformen. Fügen Sie für jede Verdünnung 0,1 ml der vorherigen Verdünnung auf 0,9 ml LAL-Wasser hinzu. Wirbel kräftig nach Derkombination für 1 min.
- Fügen Sie Standard-Kurvenverdünnungen und SHB-SAPN-Proben in Duplikat zu einer 96-Well-Platte hinzu. Verwenden Sie LAL-Wasser auch als Negativkontrolle in Duplikat. Die Platte 15 min bei 37 °C vorbrüten.
- Gegen Ende der Inkubation die Testreagenzdurchstechflasche mit 2,6 ml LAL-Wasser wieder aufsetzen. Mischen Sie den Inhalt vorsichtig mit einer serologischen Pipette.
- Fügen Sie jedem Brunnen der 96-Well-Platte 100 l des Assay-Reagenzes hinzu. Bewegen Sie die Platte schnell auf den Plattenleser und führen Sie die in Schritt 8.2 geschriebene Programmvorlage aus.
- Nachdem das Programm abgeschlossen ist, generieren Sie eine Standardkurve mit dem Protokollwert der Steuerelemente im Vergleich zum Protokollwert der Einseinstellungszeit. Verwenden Sie die aus dieser Kurve generierte Formel, um die Endotoxinkonzentration in den Proben zu berechnen.
Ergebnisse
Der hier gezeigte vollständig montierte SHB-SAPN basiert auf Proteinsequenzen (Abbildung 1A), die sich voraussichtlich in ein Teilchen falten, das 60 Kopien des Monomers enthält (Abbildung 1B). Abbildung 2 enthält einen Überblick über die Methode für die Produktion, Reinigung und Identifizierung des SHB-SAPN-Kerns. E. coli aus einem Glycerinbestand, der einen pPep-T-Expressionsvektor mit Gensequenz des SHB-SAPN-Kerns enthielt, wurde in BL21 (DE3) E. coli induziert. Bakterielle Zellen wurden erfolgreich angebaut und unter denaturierenden und reduzierenden Bedingungen lysiert.
Das gesamte Zelllysat wurde verwendet, um SHB-SAPN-Monomere durch FPLC mit einer Ni 2+-Spalte zu reinigen (Abbildung 3A). Der FPLC-Chromatograph zeigt, dass Proteine, die sowohl bei 150 mM als auch bei 500 mM Imidazol eluiert wurden (Abbildung 3A). Das Chromatogramm zeigt auch zwei weitere Spitzen bei 185 ml bzw. 210 ml Gesamtvolumen, die der Isopropanolwäsche bzw. der Imidazol-freien Wäsche entsprechen. Die Fraktionen und die Reinheit des rekombinanten Proteins wurden durch Gradienten-SDS-PAGE-Gele identifiziert (Abbildung 3B). Das protein von interest wurde in erster Linie in den Fraktionen 68'u201279 (278'u2012300 ml Gesamtvolumen) lokalisiert. Diese Fraktionen wurden für weitere Analysen kombiniert. Western Blot mit Anti-His-Antikörper (N-Terminal) und 167-D-IV-Antikörper (C-Terminus) deutete darauf hin, dass die gepoolten Fraktionen tatsächlich das Protein von Interesse waren (Abbildung 4A,B). Diese Flecken zeigten auch die Präsenz der SHB-SAPN Multimer. Frühere Washes und Elutionsfraktionen enthielten tendenziell eine höhere Konzentration an multimerisiertem Protein und wurden daher ausgeschlossen.
Die Proben, die die von Interesse sindden Proteinmonomere enthielten, wurden durch Dialyse in den vollständig montierten SHB-SAPN gefaltet. Die Partikelgrößenverteilung wurde durch DLS- und Nanopartikel-Tracking-Analyse bestimmt (Abbildung 5A,B). Die DLS identifizierte Partikel mit einem Z-durchschnittlichen hydrodynamischen Durchmesser von 67 nm, während das NTA-System eine mittlere Größe von 81 nm maß. Die leichten Größenunterschiede waren auf die Partikelgrößentechniken zurückzuführen, die Größe beider Analysen lag jedoch im erwarteten Bereich von 20-u2012100 nm8,24,25. SHB-SAPNs wurden von TEM visualisiert und die Bilder zeigten wohlgeformte Einzelteilchen mit der Größenverteilung, die aus den beiden Partikelgrößentechniken gewonnen wurde (Abbildung 5C).
Während der Reinigung des Proteins wurde die Säule mit Isopropanol gewaschen, um die LPS-Kontamination im endgültigen SHB-SAPN-Produkt zu verringern. Um zu überprüfen, ob der Endotoxinspiegel für die Immunisierung akzeptabel war, wurde die Konzentration von LPS in SHB-SAPN-Proben, die mit oder ohne Isopropanol-Waschschritt gereinigt wurden, durch einen kinetischen LAL-Assay bestimmt. Die Ergebnisse zeigten, dass die Isopropanolwäsche die Endotoxinwerte von >0,25 EU/g auf 0,010 EU/g SHB-SAPN-Protein verringerte (Tabelle 1).
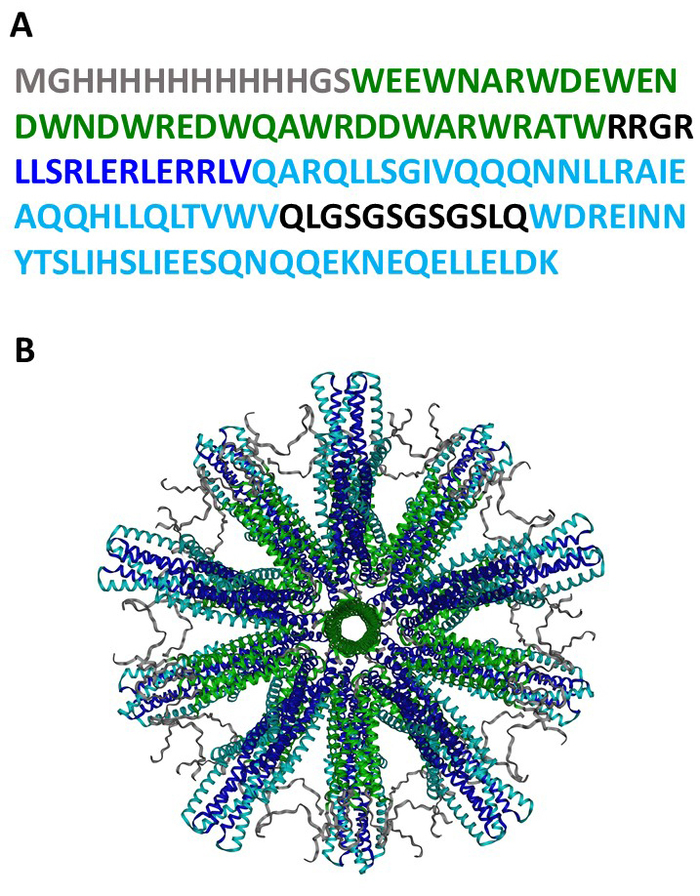
Abbildung 1: SHB-SAPN-Proteinsequenz und -struktur. (A) Die Aminosäuresequenz des SHB-SAPN-Monomers. (B) Computermodell der Struktur des vollständig montierten SHB-SAPN-Kerns bestehend aus 60 Proteinmonomeren. Farbschema für Aminosäuresequenzen: Grau = HisTag; Grün = Pentamer; Dunkelblau = de novo entworfen Trimmer; Hellblau = Sechs-Helix-Bundle. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
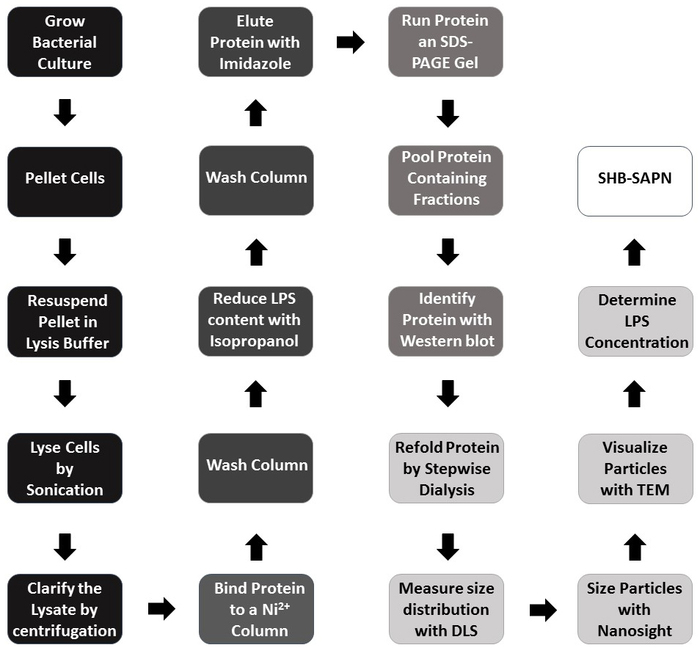
Abbildung 2: Flussdiagramm der Protokoll für die SHB-SAPN-Produktion. Farbschema: Schwarz = Expression des Proteinmonomers in E. coli; Dunkelgrau = Monomerreinigung; Mittelgrau = Monomer-Identifikation; Hellgrau = Umfalten und Charakterisieren; Weiß = vollständig montiertes SHB-SAPN-Produkt. In Schritten, die mit dunkel und mittelgrau gekennzeichnet sind, ist das Protein unter denaturierenden und reduzierenden Bedingungen. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
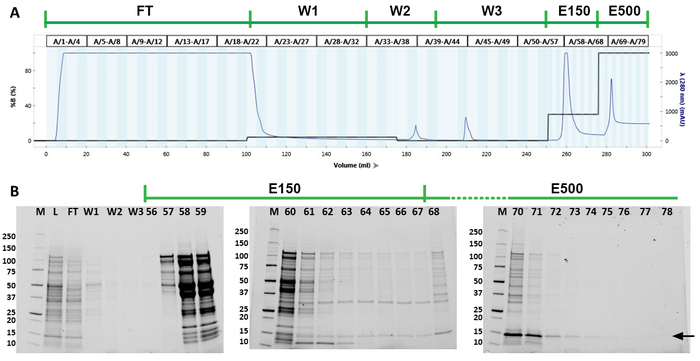
Abbildung 3: Proteinreinigung der SHB-SAPN-Monomere. (A) Chromatograph aus der FPLC-Reinigung. Die grüne Linie über dem Chromatogramm zeigt die Reinigungsschritte an. Blaue Linie im Chromatogramm stellt die optische Dichte der Fraktionen bei 280 nm Wellenlänge dar. Die schwarze Linie zeigt, welcher Prozentsatz des Puffers B (8 M Harnstoff, 20 mM Tris, 50 mM Natriumphosphat monobasic, 5 mM TCEP, 500 mM Imidazol, pH 8,5) in jeder Phase der Reinigung verwendet wurde. (B) SDS-PAGE Gele der gepoolten Fraktionen aus dem Lysat (L), durchfließen (FT), Erste Wäsche (W1), Isopropanolwäsche (W2), dritte Waschung (W3) und einzelne Fraktionen aus dem 150 mM Imidazol (E150) und 500 mM Imidazol (E500) reinigung. Molekulare Marker in der ersten Spur (M) identifizieren Bänder zwischen 10 und 250 kDa. Das Zielprotein wird durch einen schwarzen Pfeil angezeigt. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
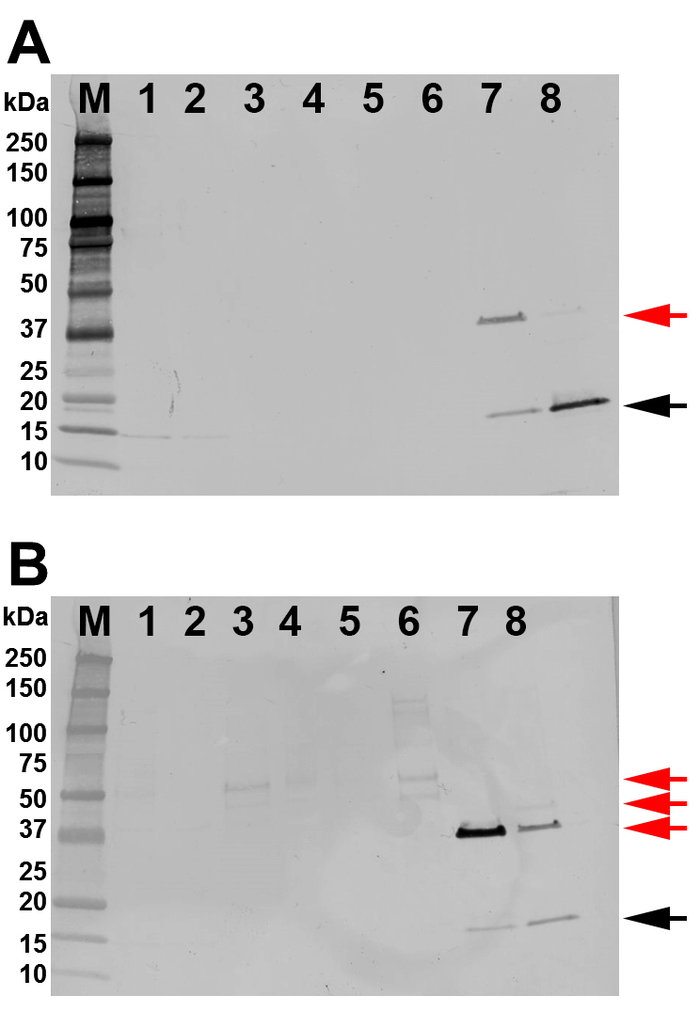
Abbildung 4: Identifizierung des Proteins durch Western Blot. Alle Bahnen sind mit 100 ng Protein beladen. (A) Ergebnisse eines westlichen Blots mit Anti-6x HisTag. (B) Ergebnisse eines westlichen Flecks mit einem 167-D-IV HIV-1 monoklonalen Antikörper. Bahnen sind wie beschriftet als: M = Molekulargewichtsmarker; 1 = Lysat; 2 = Durchfließen; 3 = erste Wäsche; 4 = Isopropanolwäsche (zweite Wäsche); 5 = dritte Wäsche; 6 = gepoolte Volumenfraktionen 56-u201261 (erste Elutionsspitze), 7 = gepoolte Volumenfraktionen 62-u2201267 (zwischen den beiden Spitzen), 8 = gepoolte Volumenfraktionen 68-u201278 (zweite Elutionsspitze). Zielprotein mit der erwarteten Bandgröße von 18,07 kDa als monomeres SHB-SAPN-Band wird durch einen schwarzen Pfeil angezeigt. Zusätzliche Bänder in den Bahnen 7 und 8 sind Dimer, Trimmer und Multimer des SHB-SAPN (roter Pfeil). Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
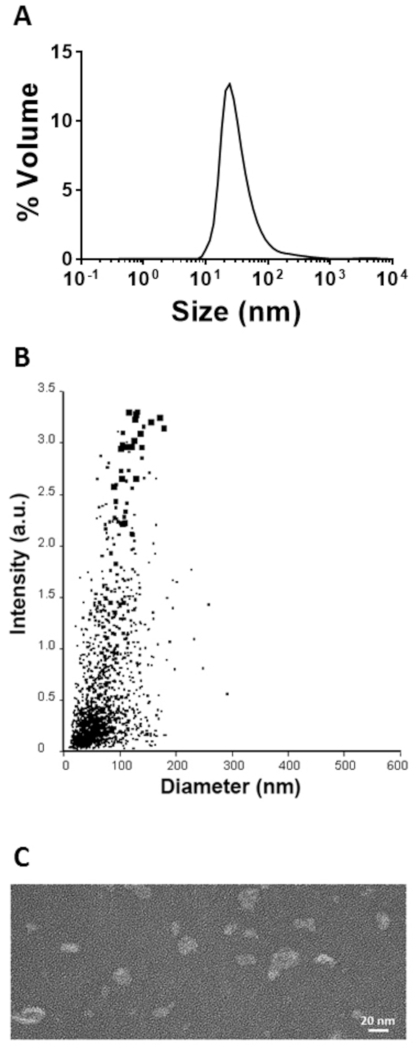
Abbildung 5: Charakterisierung des neu gefalteten SHB-SAPN. (A). Partikelgrößenverteilung, die von DLS bestimmt wird. (B) Partikelgröße, die durch Nanopartikelverfolgung (System) bestimmt wird. (C) Visualisierung der SHB-SAPN-Partikel durch TEM. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
| probe | Endotoxin (EU/ml) | Endotoxin (EU/g Protein) |
| SAPN mit Isopropanol Wash | 2.02 | 0.01 |
| SAPN ohne Isopropanol Wash | >50 | >0,25 |
| Negative Kontrolle | Unterhalb der Erkennungsstufe | N/A |
Tabelle 1: Endotoxingehalte von umgeklappten SHB-SAPNs. Endotoxingehalte in SHB-SAPN-Proben, die mit oder ohne Isopropanolwäsche gereinigt werden, sowohl als Endotoxineinheiten/ml als auch als Endotoxineinheiten/g SHB-SAPN-Protein dargestellt.
Diskussion
Die Nanotechnologie bietet viele Vorteile und Lösungen für die Entwicklung von Impfstoffen aus Untereinheiten. Nanoimpfstoffe können wiederholt Antigene als Partikel für das Wirtsimmunsystem darstellen und die Immunogenität erhöhen26. Obwohl es viele verschiedene Arten von Nanoimpfstoffen gibt, glauben wir, dass solche, die aus de novo designten Proteinen bestehen, der stärkste Ansatz für die Impfstoffentwicklung zu sein scheinen1. Sie können ohne Sequenzhomologie zu den Wirtsproteinen entwickelt werden und präsentieren das Antigen von Interesse in der Nähe von nativen-ähnlichen Konformationen bei gleichzeitig niedrigen Produktionskosten und hohen Produkterträgen. Ein Paradebeispiel für diesen Ansatz ist die SAPN-Technologie, die wir auf Impfstoffe gegen mehrere Infektionskrankheiten angewendet haben7. Um die Schwierigkeiten bei der Entwicklung von HIV-1-Impfstoffen zu beheben, haben wir einen einzigartigen SHB-SAPN-Kern entwickelt, um das V1V2-Antigen effektiv in einer nativen trimeren Konformation9zu präsentieren. Viele Impfziele, insbesondere bei Viruserkrankungen, sindals Trimmer 27 vorhanden. Dieses Phänomen deutet darauf hin, dass unser SAPN-Design weitreichende Auswirkungen auf die Entwicklung von Subunit-Impfstoffen hat.
Mit dieser Methode zeigen wir, wie SHB-SAPNs in einem E. coli-Expressionssystem produziert werden. Wir drückten hohe Proteinerträge aus (ca. 6 mg/100 ml Kultur). Das Protein enthielt 10 Histidine und wurde leicht mit einer immobilisierten Metallaffinitätschromatographie mit Ni2+ Säule gereinigt. Diese Länge des His-Tags erwies sich als das Optimum für den höchsten Proteinertrag. Das gereinigte Protein enthielt die volle Länge des entworfenen Proteins, wie durch das Vorhandensein des N-terminalen HisTag und der C-Terminal-Heptad-Wiederholung angezeigt wird. Wir haben weithin akzeptierte Techniken eingesetzt und für den Ausdruck, die Produktion und die Charakterisierung des SHB-SAPN-Kerns optimiert. Ein Mangel an Produktion des Voll-Längen-Proteins während der Entwicklung eines SAPN, das ein neues Protein-Epitop enthält, könnte auf ein Expressionsproblem des Gens in der Wirtszelle hindeuten. Wenn dies geschieht, müssen das Gen und das Expressionssystem neu gestaltet und an das beschriebene Protokoll angepasst werden. Eine Änderung der Beschallungszeit oder -intensität kann auch die Konzentration des vorhergesagten Voll-Längen-Proteins erhöhen.
Umgeklappte Partikel lagen im erwarteten Größenbereich (20 bis 100 nm)8,24,25, wie durch DLS- und Nanopartikel-Tracking-Analyse bestimmt. Diese Ergebnisse wurden durch die Verwendung von TEM weiter bestätigt. Wenn es in diesem Schritt Probleme gibt, ist dies normalerweise auf ein Problem mit dem pH- oder Ionenstärke des Umfaltpuffers zurückzuführen. Wenn großformatige Partikel auf den Partikelgrößentechniken erkannt werden, zeigt dies eine Aggregation an, die durch Erhöhung des pH-Werts des Umfaltpuffers vermieden werden kann. Wenn die Partikel nicht von DLS erkannt werden, überprüfen Sie die Konzentration des Proteins und überprüfen Sie den pH-Wert des Puffers. Die endgültige Proteinkonzentration für DLS sollte mindestens 100 g/ml betragen. Wenn die Konzentration nicht das Problem ist, zeigt sie die Fülle kleiner, unvollständig gebildeter Teilchen an, deren Konzentration durch Senkung des pH-Werts reduziert werden kann. Alternativ kann die Natriumchloridkonzentration auf den optimalen Bereich eingestellt werden, um das Vorhandensein von Partikeln mit unerwünschter Größe zu minimieren.
Schließlich konnten wir durch die Verwendung eines Isopropanol-Waschschritts während der Reinigung die kontaminierende LPS vom Wirt E. coli auf 0,01 EU/g SAPN reduzieren, was unter dem Grenzwert von 5 EU/kg Körpergewicht für injizierbare Produkte liegt. 28. Dieser Pegel kann durch die Verwendung einer Anionenaustauschsäule, auch bekannt als Q-Spalte, weiter reduziert werden. Wenn noch hohe Konzentrationen an Endotoxin vorhanden sind, überprüfen Sie alle Materialien, die für die Puffervorbereitung verwendet wurden. Denken Sie daran, nur depyrogenierte Glaswaren und Endotoxin freie Kunststoffe in dieser Methode zu verwenden.
Diese Ergebnisse deuten darauf hin, dass wir erfolgreich eine Methode zur Herstellung des SHB-SAPN-Kerns entwickelt haben, die für präklinische Immunisierungsstudien verwendet werden kann. Diese Methode mit nur geringfügigen Änderungen, wenn überhaupt, kann auf die Reinigung von SHB-SAPNs angewendet werden, wenn ein Antigen von Interesse hinzugefügt wird. Mit dieser Methode als Ausgangspunkt ist eine der wichtigsten Änderungen im Elutionsschritt. Verschiedene Proteine werden in unterschiedlichen Imidazolkonzentrationen elute, die experimentell bestimmt werden müssen. Der andere große Unterschied könnte die Zusammensetzung des Umfaltungspuffers sein. Die Optimierung würde die Prüfung verschiedener pH-Bedingungen sowie ionischer Stärken erfordern.
Im Hinblick auf die zukünftige Arbeit sind nur zwei geringfügige Änderungen erforderlich, um eine menschliche Anwendung des SHB-SAPN zu ermöglichen. Die erste ist, dass der Expressionsvektor aufgrund der Ampicillinallergie beim Menschen in einen kanamycin-resistenzwählbaren Marker geändert werden muss29. Die andere wichtige Anforderung der Proteinherstellung für den menschlichen Gebrauch ist die Herstellung des SHB-SAPN in tierproduktfreien Medien. Eine kleine Studie zeigte bereits einen angemessenen Proteinertrag in einem pflanzlichen Medium. Die hier vorgestellte Arbeit ist für die ultimative GMP-Produktion leicht skalierbar, wie mit einem Malaria-Impfstoff-Kandidaten, FMP01416,demonstriert wurde. Diese groß angelegte FMP014-SAPN-Produktion umfasste sowohl den Anionenaustausch als auch die Kationenaustauschschritte, um den LPS- und Ni2+-Gehalt aus dem Endprodukt weiter zu reduzieren. Dieses bakterielle SAPN wurde bereits für eine bevorstehende klinische Phase 1/2a-Studie skaliert.
Offenlegungen
Die geäußerten Ansichten sind die der Autoren und sollten nicht so ausgelegt werden, dass sie die Positionen der US-Armee oder des Verteidigungsministeriums repräsentieren. Peter Burkhard hat Interesse an der Alpha-O Peptides AG und hat Patente auf die Technologie. Die anderen Autoren haben keine Zugehörigkeit oder finanzielle Beteiligung an einem Unternehmen mit einem finanziellen Interesse an dem in diesem Papier vorgestellten Thema.
Danksagungen
Diese Arbeit wurde durch eine Kooperationsvereinbarung (W81XWH-11-2-0174) zwischen der Henry M Jackson Foundation for the Advancement of Military Medicine, Inc. und dem US-Verteidigungsministerium unterstützt. Der Anti-HIV-1 gp41 mAb 167-D IV Antikörper wurde von Dr. Susan Zolla-Pazner über das NIH AIDS Reagenzprogramm erhalten.
Materialien
| Name | Company | Catalog Number | Comments |
| 10x Tris/Glycine/SDS | BioRad | 1610732 | 1 L |
| 2-Mercaptoethanol | BioRad | 1610710 | 25 mL |
| 2-propanol | Fisher | BP26181 | 4 L |
| 2x Laemmli Sample Buffer | BioRad | 1610737 | 30 mL |
| 40ul Cuvette Pack of 100 with Stoppers | Malvern Panalytical | ZEN0040 | 100 pack |
| 4–20% Mini-PROTEAN TGX Precast Protein Gels, 10-well, 30 µl | BioRad | 4561093 | 10 pack |
| Ampicillin | Fisher | BP1760-25 | 25 g |
| Anti-6X His tag antibody [HIS.H8] | AbCam | ab18184 | 100 mg |
| Anti-HIV-1 gp41 Monoclonal (167-D IV) | AIDS Reagent Repository | 11681 | 100 mg |
| BCIP/NBT Substrate, Solution | Southern Biotech | 0302-01 | 100 mL |
| Corning Disposable Vacuum Filter/Storage Systems | Fisher | 09-761-108 | A variety of sizes |
| Formvar/Carbon 400 mesh, Copper approx. grid hole size: 42µm | Ted Pella, Inc | 01754-F | 25 pack |
| GE Healthcare 5 mL HisTrap HP Prepacked Columns | GE HealthCare | 45-000-325 | 5 pack |
| Glycerol | Fisher | BP229-4 | 4 L |
| Goat Anti-Mouse IgG H&L (Alkaline Phosphatase) | ABCam | ab97020 | 1 mg |
| Imidazole | Fisher | O3196-500 | 500 g |
| Instant NonFat Dry Milk | Quality Biological | A614-1003 | 10 pack |
| Kinetic-QCL Kinetic Chormogenic LAL Assay | Lonza Walkersville | 50650U | 192 Test Kit |
| LAL Reagent Grade Multi-well Plates | Lonza Walkersville | 25-340 | 1 plate |
| Magic Media E. coli Expression Medium | ThermoFisher | K6803 | 1 L |
| MilliporeSigma Millex Sterile Syringe 0.22 mm Filters | Millipore | SLGV033RB | 250 pack |
| Mouse Anti-Human IgG Fc-AP | Southern Biotech | 9040-04 | 1.0 mL |
| One Shot BL21 Star (DE3) Chemically Competent E. coli | ThermoFisher | C601003 | 20 vials |
| Precision Plus Protein Unstained Protein Standards, Strep-tagged recombinant, | BioRad | 1610363 | 1 mL |
| Slide-A-Lyzer Dialysis Cassettes, 10K MWCO, 12 mL | ThermoFisher | 66810 | 8 pack |
| Sodium Chloride | Fisher | BP358-212 | 2.5 kg |
| Sodium Phosphate Monobasic | Fisher | BP329-500 | 500 g |
| Tris Base | Fisher | BP152-1 | 1 kg |
| Tris-(2-carboxyethyl)phosphine hydrochloride | Biosynth International | C-1818 | 100 g |
| Uranyl Acetate, Reagent, A.C.S | Electron Micoscopy Services | 541-09-3 | 25 g |
| Urea | Fisher | BP169-500 | 2.5 kg |
| Whatman qualitative filter paper | Sigma Aldrich | WHA10010155 | pack of 500 |
| Name | Company | Catalog Number | Comments |
| Equipment | |||
| ChromLab Software ver 4 | BioRad | 12009390 | Software |
| Epoch 2 Microplate Spectrophotometer | BioTek | EPOCH2 | Plate Reader |
| Fiberlite F14-14 x 50cy Fixed-Angle Rotor | ThermoFisher | 096-145075 | Rotor |
| Gel Doc EZ Gel Documentation System | BioRad | 1708270 | Gel Imager for Stain free Gels |
| JEOL TEM | JEOL | 1400 | Transmission Electron Microscope |
| Mini-PROTEAN Tetra Vertical Electrophoresis Cell for Mini Precast Gels | BioRad | 1658004 | To run gels |
| NanoDrop One Microvolume UV-Vis Spectrophotometer | ThermoFisher | ND-ONE-W | For Protein Concentration |
| NanoSight NS300 | Malvern Panalytical | Particle Sizing | |
| NanoSight NTA software NTA | Malvern Panalytical | Particle Sizing | |
| New Brunswick Innova 44/44R | Eppendorf | M1282-0000 | Incubator/Shaker |
| NGC Quest 10 Chromatography System | BioRad | 7880001 | FPLC to aid in protein purification |
| PELCO easiGlow Glow Discharge Cleaning System | Ted Pella, INC | 91000S | To clean grids |
| PowerPac Universal Power Supply | BioRad | 1645070 | To run gels |
| Rocker Shaker | Daigger | EF5536A | For Western |
| Sonifer 450 | Branson | also known as 096-145075 | Sonicator |
| Thermo Scientific Sorvall LYNX 4000 Superspeed Centrifuge | ThermoFisher | 75-006-580 | Centrifuge |
| Trans-Blot Turbo Mini Nitrocellulose Transfer Packs | BioRad | 1704158 | For Western |
| Trans-Blot Turbo Transfer System | BioRad | 1704150 | For Western |
| Vortex-Genie 2 | Daigger | EF3030A | Vortex |
| Zetasizer Nano ZS | Malvern Panalytical | Particle Sizing | |
| Zetasizer Software | Malvern Panalytical | Particle Sizing |
Referenzen
- Karch, C. P., Burkhard, P. Vaccine technologies: From whole organisms to rationally designed protein assemblies. Biochemical Pharmacology. 120, 1-14 (2016).
- Snapper, C. M. Distinct Immunologic Properties of Soluble Versus Particulate Antigens. Frontiers in Immunology. 9, 598(2018).
- Kelly, H. G., Kent, S. J., Wheatley, A. K. Immunological basis for enhanced immunity of nanoparticle vaccines. Expert Review of Vaccines. , 1-12 (2019).
- Yeates, T. O. Geometric Principles for Designing Highly Symmetric Self-Assembling Protein Nanomaterials. Annual Review of Biophysics. 46, 23-42 (2017).
- Marcandalli, J., et al. Induction of Potent Neutralizing Antibody Responses by a Designed Protein Nanoparticle Vaccine for Respiratory Syncytial Virus. Cell. 176 (6), 1420-1431 (2019).
- Ross, J. F., et al. Decorating Self-Assembled Peptide Cages with Proteins. ACS Nano. 11 (8), 7901-7914 (2017).
- Karch, C. P., Matyas, G. R., Burkhard, P., Beck, Z. Self-Assembling Protein Nanoparticles: implications for HIV-1 vaccine development. Nanomedicine (Lond). 13 (17), 2121-2125 (2018).
- Raman, S., Machaidze, G., Lustig, A., Aebi, U., Burkhard, P. Structure-based design of peptides that self-assemble into regular polyhedral nanoparticles). Nanomedicine. 2 (2), 95-102 (2006).
- Karch, C. P., et al. Design and characterization of a self-assembling protein nanoparticle displaying HIV-1 Env V1V2 loop in a native-like trimeric conformation as vaccine antigen. Nanomedicine. , (2018).
- Karch, C. P., et al. The use of a P. falciparum specific coiled-coil domain to construct a self-assembling protein nanoparticle vaccine to prevent malaria. Journal of Nanobiotechnology. 15 (1), 62(2017).
- Li, J., et al. A self-adjuvanted nanoparticle based vaccine against infectious bronchitis virus. PLoS One. 13 (9), e0203771(2018).
- Wahome, N., et al. Conformation-specific display of 4E10 and 2F5 epitopes on self-assembling protein nanoparticles as a potential HIV vaccine. Chemical Biology & Drug Design. 80 (3), 349-357 (2012).
- El Bissati, K., et al. Effectiveness of a novel immunogenic nanoparticle platform for Toxoplasma peptide vaccine in HLA transgenic mice. Vaccine. 32 (26), 3243-3248 (2014).
- Kaba, S. A., et al. A nonadjuvanted polypeptide nanoparticle vaccine confers long-lasting protection against rodent malaria. Journal of Immunology. 183 (11), 7268-7277 (2009).
- Kaba, S. A., et al. Protective antibody and CD8+ T-cell responses to the Plasmodium falciparum circumsporozoite protein induced by a nanoparticle vaccine. PLoS One. 7 (10), e48304(2012).
- Seth, L., et al. Development of a self-assembling protein nanoparticle vaccine targeting Plasmodium falciparum Circumsporozoite Protein delivered in three Army Liposome Formulation adjuvants. Vaccine. 35 (41), 5448-5454 (2017).
- Kaba, S. A., et al. Self-assembling protein nanoparticles with built-in flagellin domains increases protective efficacy of a Plasmodium falciparum based vaccine. Vaccine. 36 (6), 906-914 (2018).
- El Bissati, K., et al. Protein nanovaccine confers robust immunity against Toxoplasma. NPJ Vaccines. 2, 24(2017).
- Karch, C. P., et al. Vaccination with self-adjuvanted protein nanoparticles provides protection against lethal influenza challenge. Nanomedicine. 13 (1), 241-251 (2017).
- Haynes, B. F., et al. Immune-correlates analysis of an HIV-1 vaccine efficacy trial. New England Journal of Medicine. 366 (14), 1275-1286 (2012).
- Rerks-Ngarm, S., et al. Vaccination with ALVAC and AIDSVAX to prevent HIV-1 infection in Thailand. New England Journal of Medicine. 361 (23), 2209-2220 (2009).
- O' Connell, R. J., Kim, J. H., Excler, J. L. The HIV-1 gp120 V1V2 loop: structure, function and importance for vaccine development. Expert Review of Vaccines. 13 (12), 1489-1500 (2014).
- Babapoor, S., et al. A Novel Vaccine Using Nanoparticle Platform to Present Immunogenic M2e against Avian Influenza Infection. Influenza Research and Treatment. 2011, 126794(2011).
- Indelicato, G., Burkhard, P., Twarock, R. Classification of self-assembling protein nanoparticle architectures for applications in vaccine design. Royal Society Open Science. 4 (4), 161092(2017).
- Indelicato, G., et al. Principles Governing the Self-Assembly of Coiled-Coil Protein Nanoparticles. Biophysical Journal. 110 (3), 646-660 (2016).
- Doll, T. A., Raman, S., Dey, R., Burkhard, P. Nanoscale assemblies and their biomedical applications. Journal of the Royal Society Interface. 10 (80), 20120740(2013).
- Rey, F. A., Lok, S. M. Common Features of Enveloped Viruses and Implications for Immunogen Design for Next-Generation Vaccines. Cell. 172 (6), 1319-1334 (2018).
- Bacterial Endotoxins. United States Pharmacopeia (USP). , Ch. 85 (2011).
- Points to Consider (PTC) in the Characterization of Cell Lines Used to Produce Biologicals. , Food and Drug Adminstration. Maryland. Available from: https://www.fda.gov/media/76255/download (1993).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten