Method Article
Produção de E. coli-expressa auto-montagem de nanopartículas de proteína para vacinas que requerem Trimeric epitope apresentação
Neste Artigo
Resumo
Um método detalhado é fornecido aqui descrevendo a purificação, o redobramento, e a caracterização de nanopartículas de proteína Self-montagem (SAPNs) para o uso no desenvolvimento da vacina.
Resumo
A auto-montagem de nanopartículas de proteína (SAPNs) função como exposições de antígeno repetitivo e pode ser usado para desenvolver uma ampla gama de vacinas para diferentes doenças infecciosas. Neste artigo nós demonstramos um método para produzir um núcleo de SAPN que contem um conjunto do feixe da seis-hélice (SHB) que seja capaz de apresentar antígenos em uma conformação trimérico. Nós descrevemos a expressão do SHB-SAPN em um sistema de E. coli , assim como as etapas necessárias da purificação da proteína. Nós incluímos uma etapa da lavagem do isopropanol para reduzir o lipopolysaccharide bacteriano residual. Como uma indicação da identidade e da pureza da proteína, a proteína reagiu com os anticorpos monoclonais conhecidos em análises ocidentais do borrão. Após o redobramento, o tamanho das partículas caiu na escala esperada (20 a 100 nanômetro), que foi confirmado pelo espalhamento de luz dinâmico, pela análise de seguimento da nanopartícula, e pela microscopia de elétron da transmissão. A metodologia descrita aqui é otimizada para o SHB-SAPN, porém, com apenas ligeiras modificações pode ser aplicada a outros constructos SAPN. Este método também é facilmente transferível para a produção em grande escala para a fabricação de GMP para vacinas humanas.
Introdução
Enquanto o desenvolvimento tradicional da vacina se concentrou nos patógenos inativados ou atenuados, o foco das vacinas modernas mudou para as vacinas de subunidades1. Esta abordagem pode levar a uma resposta mais orientada, e potencialmente mais eficaz vacina candidatos. No entanto, um dos principais inconvenientes é que as vacinas subunitárias não são particulados como organismos inteiros que podem resultar em imunogenicidade reduzida2. Uma nanopartícula como um sistema de exibição de antígeno repetitivo pode ter os benefícios tanto da abordagem da vacina de subunidade direcionada quanto da natureza particulada de todo o organismo1,3.
Entre os tipos existentes de nanovaccines, os conjuntos de proteína projetados racionalmente permitem o projeto e o desenvolvimento de candidatos vacinais que podem apresentar cópias múltiplas do antígeno potencial em uma conformação nativo-como1,4 ,5,6. Um exemplo desses conjuntos proteicos são as nanopartículas proteicas Automontantes (SAPNs)7. Os SAPNs são baseados em domínios bobinados e são tradicionalmente expressos em Escherichia coli8. Os candidatos à vacina SAPN foram desenvolvidos para diversas doenças como malária, SARS, influenza, toxoplasmose e HIV-19,10,11,12,13 , 14 anos de , 15 anos de , 16 anos de , 17 anos de , 18 anos de , o projeto 19. The de cada candidato de SAPN é específico ao micróbio patogénico do interesse, entretanto, a produção, a purificação, e as técnicas de redobramento são geralmente amplamente aplicáveis.
Um dos nossos interesses actuais é uma vacina eficaz para o VIH-1. Em RV144 — o único ensaio clínico de fase III de umavacina contra oVIH-1 que demonstrou eficácia modesta — o risco reduzido de infeção foi correlacionado com anticorpos IgG ao ciclo V1V2 da proteína envelope20,21. O nativo-como a apresentação trimérico desta região é pensado para ser importante para a imunogenicidade protetora22. Para apresentar o laço de V1V2 em tão perto ao nativo-como a conformação como possível, nós desenvolvemos uma prova do candidato da vacina do princípio SAPN que continha o pacote da seis-hélice da borne-fusão do envelope de HIV-1 (SHB) para apresentar o laço V1V2 na conformação correta9 . Este candidato foi reconhecido por anticorpos monoclonais conhecidos à proteína do envelope de HIV-1. Camundongos imunizados com V1V2-SHB-SAPN levantaram V1V2 anticorpos específicos, que, mais importante, vinculados a gp70 V1V2, os epítopos conformacionais corretos9. O núcleo de SHB-SAPN poderia ter outras funções além do papel como um portador para o laço de HIV-1 V1V2. Aqui nós descrevemos uma metodologia detalhada para a expressão, a purificação, o redobramento, e a validação do núcleo de SHB-SAPN. A seleção da seqüência, o projeto da nanopartícula, o clonagem molecular, e a transformação de e. coli foram descritos previamente9.
Protocolo
1. expressão da proteína SHB-SAPN em E. coli BL21 (de3)
- Misturar 95 mL do componente A e 5 mL do componente B da mídia em um frasco Erlenmeyer de vidro estéril de 2 L de acordo com as instruções do fabricante (consulte a tabela de materiais). Adicionar ampicilina a uma concentração final de 100 μg/mL.
- Inoculate os meios com E. coli de uma cultura previamente estabelecida do estoque do glicerol. Incubar cultura a 30 ° c com agitação a 200 rotações por minuto (rpm) para 48 h.
Nota: o estoque usado de E. coli BL21 (de3) continha o vetor de expressão resistente à ampicilina23 com o gene SHB-SAPN. Embora o protocolo geral da mídia recomende 24 h de incubação a 37 ° c, 48 h de incubação a 30 ° c proporcionou maior rendimento para SHB-SAPN. - Transfira a cultura para 2 50 mL de tubos cônicos. Centrifugue os tubos a 4.000 x g durante 10 min com um rotor de ângulo fixo a 4 ° c. Retire o sobrenadante e guarde o pellet para colher células.
Nota: o pellet celular pode ser processado imediatamente ou congelado em-80 ° c até o uso.
2. lise de E. coli BL21 (de3) por sonication
Nota: Use plasticware pirogénica e os produtos vidreiros cozidos em 250 ° c para pelo menos 30 min. tris (2-carboxyethyl) phosphine (TCEP) como um agente de redução quebra as ligações do dissulfeto dentro e entre proteínas. O TCEP é necessário nos bufferes durante este protocolo se o antígeno indicado contem as ligações S-S. Apenas para o núcleo SHB-SAPN, a presença de TCEP nos buffers não é essencial.
- Preparar o tampão livre de imidazol (8 M de ureia, 50 mM de fosfato de sódio monobásico, 20 mM TRIS base, 5 mM TCEP) pH 8,0 (ajustado com 5 N NaOH) e filtrá-lo usando uma unidade de filtração de vácuo de 0,22 μm.
- Ressuscitou as células peletizadas (da etapa 1,3) com 40 mL de tampão livre de imidazol em tubo cônico de 1 50 mL. SONICATE as células ressuscipended com uma sonda no gelo por 5 min (4 s de sonication, 6 s de descanso) com uma saída sonication de 150 W.
- Centrifugue o lisado celular (40 ml) em 29.000 x g em 4 ° c por 25 minutos em um rotor de ângulo fixo para gerar o sobrenadante esclarecido. Transfira o sobrenadante para um balão estéril de 150 mL e descarte o pellet. Diluir o sobrenadante para 100 mL utilizando o tampão livre de imidazol (mais tarde no protocolo referido como "amostra").
Nota: esta etapa de diluição é necessária para evitar que a pressão do sistema FPLC se torne demasiado elevada durante o carregamento de lisado na coluna.
3. purificação de proteínas usando um seu-coluna
Nota: Este protocolo foi realizado utilizando um instrumento FPLC, mas pode ser adaptado ao fluxo de gravidade.
- Prepare os seguintes buffers e filtre-os usando uma unidade de filtração de garrafas de vácuo de 0,22 μm: (i) buffer livre de imidazol "buffer A" (8 M de ureia, fosfato de sódio monobásico de 50 mM, base Tris de 20 mM, TCEP de 5 mM) pH 8,0; (II) 500 mM imidazol tampão "tampão B" (8 M de ureia, 50 mM fosfato de sódio monobásico, 20 mM TRIS base, 5 mM TCEP, 500 mM imidazol) pH 8,0; e (III) lavagem com isopropanol (20 mM Tris, 60% isopropanol) pH 8,0.
Nota: o pH para cada tampão foi ajustado com 5 N NaOH. - Equilibram a coluna dele.
Observação: para a produção em escala de laboratório, este protocolo usa um 5 mL pré-embalados his-Column, mas qualquer coluna de tamanho maior pode ser usada.- Abra o software FPLC e clique na opção novo método . Ele abrirá imediatamente para o menu de configurações do método . o menu suspenso para a posição da coluna escolha C1 porta 3.
- No menu suspenso mostrado pela técnica , escolha Affinity. No menu suspenso tipo de coluna , escolha outros, histrap HP, 5 ml. O volume da coluna e as caixas de pressão serão automaticamente definidos para os valores apropriados.
- Clique no botão de contorno do método . Arraste os seguintes botões do menu pop-up da biblioteca de fases : equilibração, aplicação de amostra, lavagem de colunae eluição ao lado da seta nessa ordem exata. Feche o menu da biblioteca de fases .
- Clique no botão equilíbrio . Os valores listados na tabela devem ser "buffer inicial B" (4%), "buffer final B" (4%) e "volume (CV)" 5.
- Clique na caixa de aplicação de exemplo . Na caixa de carregamento de amostra, clique no botão de opção para injetar amostra na coluna com bomba de amostra. Certifique-se de que a caixa ao lado da taxa de fluxo de uso das configurações do método seja verificada na injeção de amostra com caixa de bomba do sistema. Ao lado da caixa de volume no lado direito da tela, altere o valor para 20 ml.
- Clique no botão de lavagem da coluna . Os valores listados na tabela devem ser "buffer inicial B" (4%), "buffer final B" (4%) e "volume (CV)" 5. Ao lado do esquema de coleta de frações, Desclique na caixa habilitar.
- Clique no botão de eluição . Os valores listados na tabela devem ser "buffer inicial B" 0%, "buffer final B" 100% e "volume (CV)" 5. Ao lado do esquema de coleta de frações, clique na caixa Ativar . Desclique o tamanho da fração de uso das configurações do método e ajuste o tamanho da fração para 4 ml na caixa de preenchimento abaixo.
- Clique no botão salvar como na parte superior do software. Nomeie o arquivo de "equilibração".
- Conecte um 5 mL pré-embalado sua coluna à porta de coluna correspondente 3 no FPLC. A tubulação da bomba a e da bomba B assim como a tubulação da bomba da amostra deve ser coloc na água deionizada filtrada do μm 0,22. Executar o programa de equilibração.
- Coloc a bomba a e a bomba B assim como a tubulação da bomba da amostra no amortecedor imidazole-livre (amortecedor a) e funcione o protocolo do equilíbrio outra vez.
- Vincular a amostra à coluna e purificar a proteína.
- Abra o software FPLC e clique na opção novo método . Ele abrirá imediatamente para o menu de configurações do método . o menu suspenso para a posição da coluna escolha C1 porta 3. No menu suspenso mostrado pela técnica , escolha Affinity. No menu suspenso tipo de coluna , escolha outros, Histrap HP, 5 ml. O volume da coluna e as caixas de pressão serão automaticamente definidos para os valores apropriados.
- Clique no botão de contorno do método . Arraste os botões do menu pop-up da biblioteca de fases : equilíbrio, aplicação de amostra, lavagem de coluna (lavagem 1), lavagem de coluna (lavagem 2), lavagem de coluna (lavagem 3) e eluição ao lado da seta em que ordem exata. Feche o menu da biblioteca de fases .
- Clique no botão equilibration . Os valores listados na tabela devem ser "buffer inicial B" 4%, "buffer final B" 4% e "volume (CV)" 5.
- Clique na caixa de aplicação de exemplo . Na caixa de carregamento da amostra, clique no botão de opção para Iinjetar amostra na coluna com bomba de amostra. Certifique-se de que a caixa ao lado da taxa de fluxo de uso das configurações do método seja verificada na injeção de amostra com caixa de bomba do sistema.
- Ao lado da caixa de volume no lado direito da tela, altere o valor para 100 mL. Ao lado do esquema de coleta de fração, clique no botão Ativar . Desclique na caixa usar tamanho de fração de configurações de método e altere o tamanho da fração para 4 ml.
- Clique no botão de lavagem da primeira coluna (Wash 1). Os valores listados na tabela devem ser "buffer inicial B" 4%, "buffer final B" 4% e "volume (CV)" 10. Ao lado do esquema de coleta de frações, clique na caixa habilitar . Desclique o tamanho da fração de uso das configurações do método e altere o tamanho da fração para 4 ml.
- Clique no botão de lavagem da segunda coluna (Wash 2). Os valores listados na tabela devem ser "buffer inicial B" 0%, "buffer final B" 0% e "volume (CV)" 5. Ao lado do esquema de coleta de frações, clique na caixa habilitar . Desclique o tamanho da fração de uso das configurações do método e altere o tamanho da fração para 4 ml.
- Clique no botão de lavagem da terceira coluna (Wash 3). Os valores listados na tabela devem ser "buffer inicial B" 0%, "buffer final B" 0% e "volume (CV)" 5. Ao lado do esquema de coleta de frações, clique em Enable. Desclique na caixa usar tamanho de fração de configurações de método e altere o tamanho da fração para 4 ml.
- Clique no botão de Elution . Na tabela clique com o botão direito do mouse nas informações listadas e, no menu que aparece, clique em excluir etapa. Arraste o botão de gradiente isocrático para a tabela duas vezes, para que haja duas entradas.
- O valor para a primeira entrada deve ler "buffer inicial B" 30%, "buffer final B" 30% e "volume (CV)" 10. O valor para a segunda entrada deve ler "buffer inicial B" 100%, "buffer final B" 100% e "volume (CV)" 10. Ao lado do esquema de coleta de fração, clique no botão Ativar . Clique na caixa ao lado de usar o tamanho da fração das configurações do método.
- Clique no botão salvar como na parte superior do software. Nomeie o arquivo de "purificação". A tubulação da bomba a do FPLC deve ser coloc no amortecedor imidazol-livre da lavagem quando a tubulação da bomba B deve ser coloc no amortecedor do imidazol de 500 milímetros. O tubo da bomba de amostra deve ser colocado na amostra de 100 mL.
- Execute o programa de "purificação" e aguarde o momento em que o isopropanol 60% é necessário (Wash 2). Pause o programa, mova a tubulação da bomba a da lavagem imidazole-livre na lavagem do isopropanol de 60%. Reinicie o programa.
- Uma vez que a etapa do isopropanol é terminada, pause o programa outra vez e mova a tubulação da bomba a de novo no amortecedor imidazole-livre da lavagem. Reinicie o programa de purificação (o resto da corrida é automatizado).
4. avaliação da pureza e identificação da proteína por SDS-PAGE
- Combine todas as frações correspondentes a (i) fluxo-through (o lisado da pilha que não se vinculou à coluna his), (II) lavagem 1, (III) lavagem 3 (lavagem do isopropanol de 60%), e (IV) lavagem 3 em tubos cônicos separados de 50 ml. Não combine as frações de 2 mL das etapas de eluição.
- Misture 15 μL de cada uma das frações agrupadas e todas as frações das etapas de eluição com 2x tampão de amostra de Laemmli em um tubo de microcentrífuga de 0,5 mL e desnaturá-los a 95 ° c por 10 min.
- Quando as denatures da proteína, ajustaram acima o instrumento running do gel com os 3 géis pré-moldados mancha-livres do poliacrilamida em 1x Tris-Glycine SDS-PAGE que executa o amortecedor.
- Carregar 8 μL de marcador de peso molecular para o primeiro poço e 30 μL de amostra desnaturada para os outros poços do gel. Executar os géis em 200 V até que a frente corante atinge o fundo do gel (cerca de 30 min). Retire os géis do aparelho e enxague brevemente com água desionizada. Imagem do gel imediatamente usando o sistema de imagem livre de manchas.
- Identifique as frações que contêm bandas de proteínas com o tamanho correto (18, 7 kDa). Piscina todas essas frações.
5. identificação da proteína pelo borrão ocidental
- Funcione um borrão ocidental usando um anticorpo his-specific (anti-6x HisTag) e um anticorpo SHB-específico (167-D-IV) para identidade a proteína completo purified. O anticorpo de anti-6x HisTag reconhece o término de N da proteína e o anticorpo 167-D-IV reconhece o término de C que demonstra a presença da proteína full-length.
- Determine a concentração proteica do (i) fluxo-através de, (II) lavagem 1, (III) lavagem 2 (60% lavagem do isopropanol), (IV) lavagem 3, e todas as frações das etapas da eluição com o instrumento do espectrómetro em uma absorvância de 280 nanômetro. Gerar diluições que contenham 100 ng de proteína em 15 μL de tampão livre de imidazol para cada um destes grupos.
- Adicionar 15 μL de tampão de amostra de 2x Laemmli a cada 15 μL das amostras e desnaturá-los como no passo 4,1. Uma vez que a desnaturação é terminada gire para baixo os tubos para assegurar-se de que toda a proteína possa ser transferida.
- Amostras de carga e o marcador pré-manchado em um gel de poliacrilamida pré-moldado sem mancha 4 – 20%. Funcione a electroforese em 200 V até que a parte dianteira da tintura alcangue a parte inferior do gel.
- Enquanto o gel corre, fazer 1 L de TBS-T (20 mM Tris, 150 mM NaCl, e 0,1% Tween 20) e 200 mL de 5% de leite não gordo em TBS-T.
- Use um sistema de transferência de Western blot para transferir proteínas para uma membrana de nitrocelulose. Use uma pilha de transferência pré-montada e coloque o gel sobre ele. Configure o sistema para rodar a 25 V durante 7 min. Verifique a presença do marcador pré-manchado na membrana de nitrocelulose indicando uma transferência completa.
Nota: todas as etapas subseqüentes são executadas em um agitador orbital ajustado em 100 RPM na temperatura ambiente (RT). - Uma vez que a transferência é terminada, lave bordos duas vezes com TBS-T por 10 minutos cada um.
- Bloqueie as membranas de nitrocelulose (blot) com 5% de leite não gordo em TBS-T durante pelo menos 1 h. Lave as borrões duas vezes com TBS-T durante 10 min cada.
- Diluir os anticorpos primários 167-D-IV e anti-6x HisTag para 1 mg/mL em TBS-T (estoque AB). Diluir o estoque ABS do 167-D-IV 10.000-fold e o anti-6x HisTag 5.000-fold adicionando 2 μL do estoque 167-D-IV e 4 μL do estoque anti-6x HisTag anticorpos a dois tubos diferentes contendo 20 mL de TBS-T. Adicione o volume total de 20 mL de anticorpos primários, um a cada borrão, e incubar manchas por 1 h. Lave borrões duas vezes com TBS-T por 10 min.
- Diluir 4 μL de 1 mg/mL do anticorpo secundário anti-humano do rato conjugado com fosfatase alcalina em 20 mL de TBS-T (diluição de 1:5000). Diluir 4 μL de 1 mg/mL do anticorpo secundário de cabra anti-rato conjugado com fosfatase alcalina em 20 mL de TBS-T (diluição de 1:5000). Adicione anticorpos secundários a manchas correspondentes.
Nota: o anticorpo secundário anti-humano liga-se a 167-D e o anti-rato liga-se ao anti-6x HisTag. Lave borchas duas vezes com TBS-T por 10 min. - Adicione o suficiente substrato de fosfatase alcalina BCIP/NBT para cobrir os blots. Desenvolva os borrões por aproximadamente 10 minutos até que as faixas apareçam. Enxágüe manchas com água fria da torneira e deixe-os secar antes de digitalizar com um scanner de mesa.
6. redobrar o SHB-SAPN
- Adicione a proteína agrupada (10 \ u201220 ml total) a um 10 kDa peso molecular cortado gaveta de diálise e Dialize-lo em 8 M de ureia, 20 mm Tris, 5% glicerol, 5 mm TCEP pH 8,5 em RT (18 \ u201226 ° c) durante a noite.
- Lentamente Dialize a ureia fora da amostra, diminuindo a concentração de ureia no tampão de diálise Stepwise por 2 M cada 2 h. Numa concentração de 2 M de ureia, mova o aparelho de diálise para 4 ° c (não utilize TCEP no tampão de diálise a partir deste passo). Termine o redobramento atividade a amostra em 120 de ureia do milímetro, 20 milímetros Tris, 5% glicerol, pH 8,5 em 4 ° c durante a noite.
- Retire a proteína redobrada (SHB-SAPN) da gaveta de diálise. Filtre o SHB-SAPN usando um Filer da seringa do fluoreto do polyvinylidene de 0,22 μm (PVDF). Aliquot SHB-SAPN em tubos estéreis e congele-os a-80 ° c, deixando pelo menos 100 μL em RT para análises subsequentes.
7. validação de partículas por tamanho e aparência
- Espalhamento de luz dinâmico (DLS)
- Meça o tamanho de partícula médio do SHB-SAPN pelos seguintes parâmetros: selecione a proteína como o material, crie um amortecedor complexo para a ureia de 120 milímetros, 20 milímetros Tris, 5% glicerol 25 ° c para a temperatura, selecione as abotoas descartáveis para a análise, seleto automático medição, definido para 5 execuções.
- Adicione 45 μL de SHB-SAPN a uma cubeta descartável e execute o software estalando na seta verde. Selecione o volume percentual para a leitura.
- Análise de rastreamento de nanopartículas (NTA)
- Diluir a amostra por 1:20 no buffer de redobramento. Fazer 10 mL de amostra diluída.
- Utilizando seringas de 3 mL, lave o instrumento NTA com o tampão de redobramento e carregue cerca de 1,5 mL de amostra para equilibrar o instrumento. Use o resto da amostra para a análise.
- Crie um novo SOP no software NTA clicando na guia SOP . a guia alterar o número de capturas para 3 e alterar o tempo de captura para 30 s. Pressione o botão autofocus no lado esquerdo da tela para colocar a amostra em foco. Use o botão de focagem manual no lado da máquina para afinar o foco.
- Execute o SOP criado, quando o sistema solicitar a carga de um pequeno volume de amostra com a seringa. Após o sistema ter tomado todas as capturas que irá automaticamente trazer a tela de análise. Deslize a barra de limite de detecção para que todas as partículas reais sejam marcadas com cruzes vermelhas. Pressione o botão de análise de execução e a análise começará automaticamente.
- Microscopia eletrônica de transmissão (TEM)
- Brilho de descarga formvar/carbono 400 malha cobre TEM filmes de apoio.
- Adicionar 3 μL de amostra a uma concentração de 0, 75 mg/mL na grelha durante 30 s. Wick fora do líquido usando um papel de filtro.
- Lave a grelha com 3 μL de água deionizada três vezes, cada vez que wicking fora da água com papel de filtro.
- Adicionar 3 μL de 0,5% de acetato de uranilo ao filme de apoio e permitir que ele se sente por 30 s. Wick fora a maior parte do acetato de uranilo, mas deixar um filme fino sobre a superfície. Permita que as amostras SECem antes da imagem latente eles no microscópio de elétron da transmissão.
- Amostras de imagem em 80 kV em um TEM.
8. determinação dos níveis de endotoxina nas amostras utilizando um ensaio cinético de Limulus amebócito lisado (LAL)
- Retire o kit e as amostras do frigorífico e deixe equilibrar a RT.
- Para realizar este ensaio, é necessário um leitor de placas com um bloco de calor e a capacidade de ler as amostras para 40 leituras em um comprimento de onda de 405 nm a 37 ° c. Escreva um modelo de programa para que os poços são lidos a cada 150 s para identificar o ponto de tempo de início (o OD aumentou 0,2 em comparação com a primeira leitura).
- Diluir a amostra para a concentração de dose de imunização em PBS.
Nota: o pH do tampão de redobramento está fora do intervalo do ensaio LAL. A diluição da amostra em PBS definirá o pH para o intervalo aceitável. - Suspender a endotoxina de controle no volume adequado de água LAL livre de endotoxina, conforme determinado pelo certificado de análise para gerar 50 EU/mL. Vórtice vigorosamente o frasco para injetáveis durante 15 min para garantir a ressuspensão completa da endotoxina.
- Gere a curva padrão da endotoxina preformando uma diluição de série de 10 vezes em frascos de vidro na escala de 50 UE a 0, 5 UE/mL. Para cada diluição, adicionar 0,1 mL da diluição anterior a 0,9 mL de água LAL. Vortex vigorosamente após a combinação por 1 min.
- Adicione diluições de curva padrão e amostras SHB-SAPN em duplicado para uma placa 96-well. Use a água LAL como um controle negativo em duplicado também. Pré-incubar a placa a 37 ° c durante 15 min.
- No final da incubação, suspender o frasco para injetáveis de reagente de ensaio com 2,6 mL de água LAL. Misture suavemente o conteúdo com uma pipeta serológica.
- Adicionar 100 μL do reagente do ensaio a cada poço da placa de 96 poços. Mova rapidamente a placa para o leitor de placas e execute o modelo de programa escrito na etapa 8,2.
- Depois que o programa for concluído, gere uma curva padrão usando o valor de log dos controles versus o valor de log do tempo de início. Use a fórmula gerada a partir desta curva para calcular a concentração de endotoxina nas amostras.
Resultados
A SHB-SAPN totalmente montada mostrada aqui é construída sobre sequências proteicas (Figura 1a) que se prevê dobrar em uma partícula que contenha 60 exemplares do monómero (Figura 1b). A Figura 2 fornece um contorno do método para a produção, purificação e identificação do núcleo SHB-SAPN. E. coli de um estoque do glicerol que contivesse um vetor da expressão de PPEP-T com seqüência do gene do núcleo de SHB-SAPN foram induzidos em BL21 (de3) E. coli. As células bacterianas foram cultivadas e lisadas com sucesso condições de desnaturação e redução.
O lisado total da pilha foi usado para purificar monômeros de SHB-SAPN por FPLC usando uma coluna de Ni2 + (Figura 3a). O cromatografo de FPLC demonstra que a proteína eluída em 150 milímetros e no imidazol de 500 milímetros (Figura 3a). O cromatograma também mostra dois outros picos a 185 mL e 210 mL de volume total correspondente à lavagem de isopropanol e à lavagem livre de imidazol, respectivamente. As frações e a pureza da proteína recombinante foram identificadas por géis de SDS-PAGE gradiente (Figura 3B). A proteína de interesse foi principalmente localizada em frações 68 \ u201279 (278 \ u2012300 mL de volume total). Estas frações foram combinadas para análises complementares. O borrão ocidental com anticorpo anti-his (N-terminal) e o anticorpo 167-D-IV (C-Terminus) indicaram que as frações agrupadas eram certamente a proteína do interesse (figura 4a, B). Estes borrões também demonstraram a presença dos multimers SHB-SAPN. As frações anteriores de lavagem e de eluição tenderam a conter uma maior concentração de proteínas multimerizadas e, portanto, foram excluídas.
As amostras que continham os monómeros proteicos de interesse foram dobradas para o SHB-SAPN totalmente montado por diálise. A distribuição do tamanho das partículas foi determinada pelo DLS e pela análise de rastreamento de nanopartículas (Figura 5a, B). O DLS identificou partículas com um diâmetro hidrodinâmico da Z-média de 67 nm, enquanto o sistema NTA mediu um tamanho médio de 81 nm. As pequenas diferenças de tamanho foram devidas às técnicas de dimensionamento de partículas, porém o tamanho de ambas as análises estava na faixa esperada de 20 \ u2012100 nm8,24,25. Os SHB-SAPNs foram visualizados pelo Met e as imagens mostraram partículas individuais bem formadas com a distribuição de tamanho obtida a partir das duas técnicas de dimensionamento de partículas (Figura 5C).
Durante a purificação da proteína, a coluna foi lavada com isopropanol para diminuir a contaminação do LPS no produto final SHB-SAPN. Para verificar se o nível de endotoxina era aceitável para imunização, a concentração de LPS em amostras de SHB-SAPN purificadas com ou sem o passo de lavagem de isopropanol foi determinada por um ensaio cinético de LAL. Os resultados indicaram que a lavagem do isopropanol diminuiu os níveis de endotoxina de > 0,25 EU/μg para 0, 10 EU/μg de proteína SHB-SAPN (tabela 1).
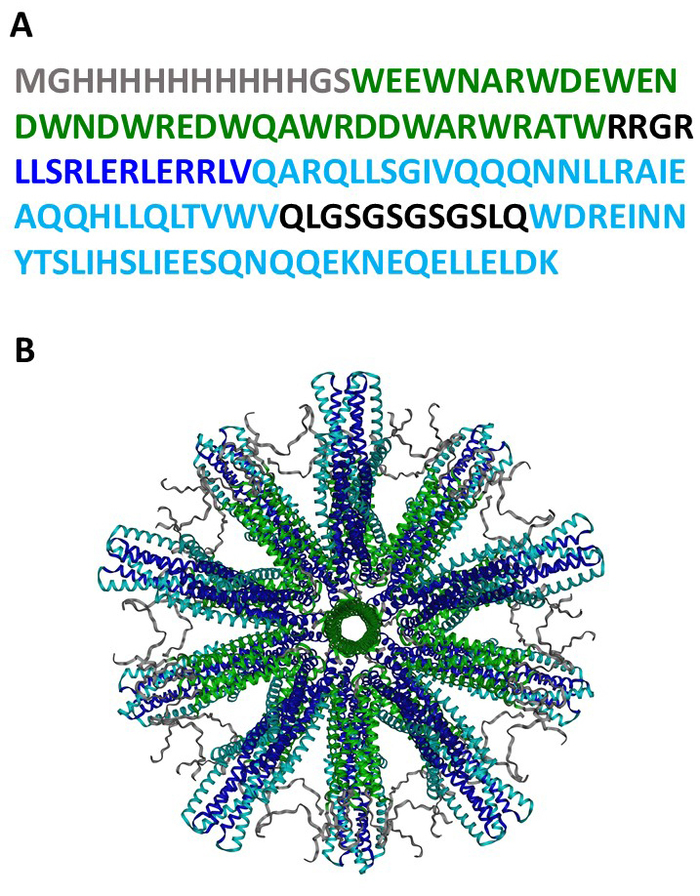
Figura 1: seqüência e estrutura da proteína SHB-SAPN. (A) a sequência de aminoácidos do monómero SHB-SAPN. (B) modelo informático da estrutura do núcleo inteiramente montado de SHB-SAPN que consiste em 60 monomers da proteína. Esquema de cores para sequências de aminoácidos: Grey = HisTag; Verde = pentamer; Azul escuro = de novo projetado trimer; Azul claro = feixe de seis-hélice. Por favor clique aqui para ver uma versão maior desta figura.
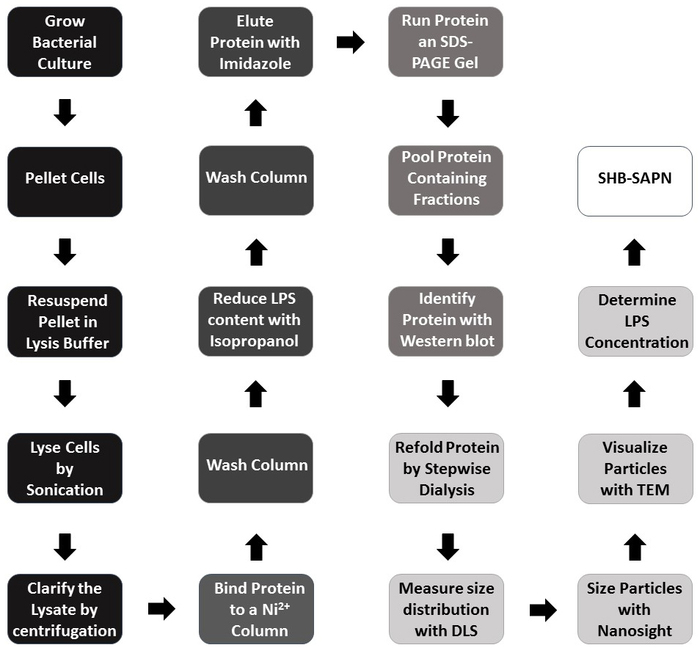
Figura 2: fluxograma da protocolo para a produção de SHB-SAPN. Esquema de cores: preto = expressão do monômero proteico em E. coli; Cinza escuro = purificação do monómero; Cinza médio = identificação do monómero; Cinza claro = redobramento e caracterização; Branco = totalmente montado SHB-SAPN produto. Em etapas rotuladas com cinza escuro e médio, a proteína está condições de desnaturação e redução. Por favor clique aqui para ver uma versão maior desta figura.
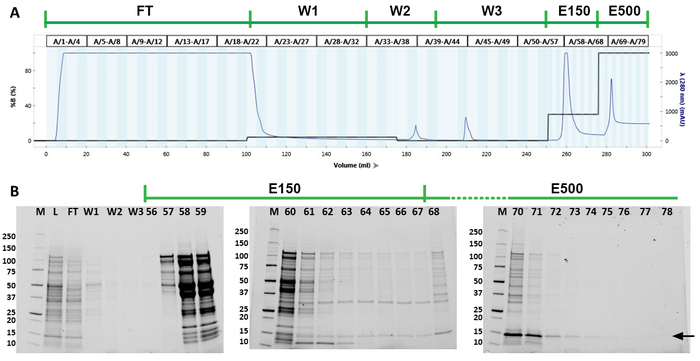
Figura 3: purificação proteica dos monômeros SHB-SAPN. (A) cromatógrafo da purificação do FPLC. A linha verde acima do cromatograma indica as etapas de purificação. A linha azul no cromatograma representa a densidade óptica das frações a 280 nm de comprimento de onda. A linha preta mostra o percentual de tampão B (8 M de ureia, 20 mM Tris, 50 mM de fosfato de sódio monobásico, 5 mM TCEP, 500 mM imidazol, pH 8,5) que foi utilizado em cada estágio da purificação. (B) géis de SDS-PAGE das frações agrupadas do lisado (L), fluxo através (ft), primeira lavagem (W1), lavagem do isopropanol (W2), terceira lavagem (w3), e frações individuais dos 150 milímetros imidazol (E150) e 500 milímetros imidazol (E500) etapas da eluição do Purificação. Marcadores moleculares na primeira pista (M) identificam bandas entre 10 e 250 kDa. A proteína alvo é indicada por uma seta preta. Por favor clique aqui para ver uma versão maior desta figura.
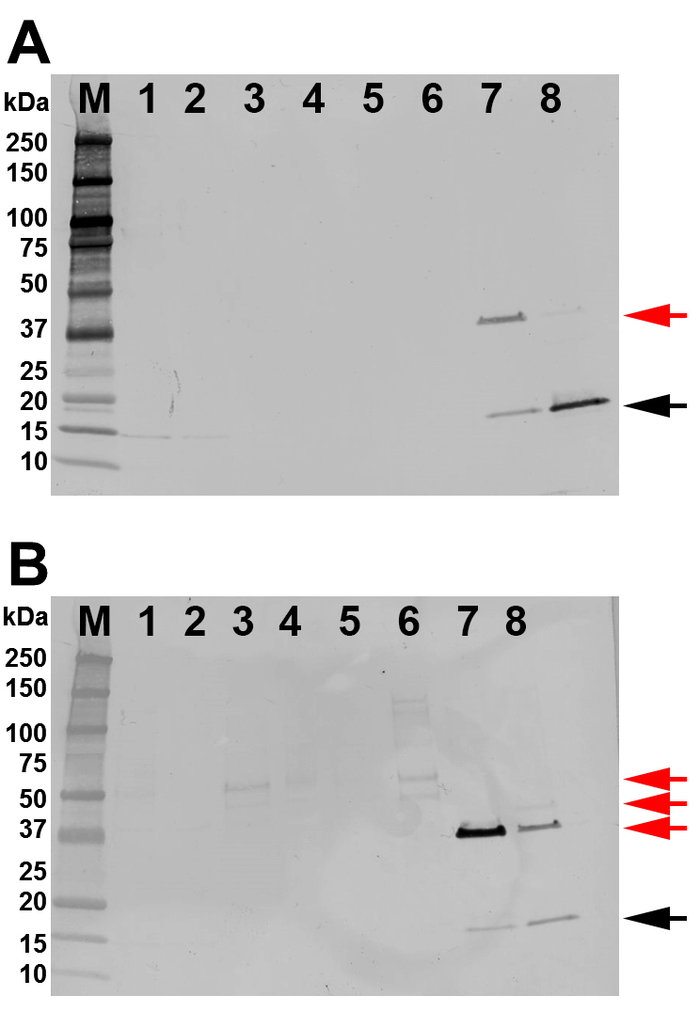
Figura 4: identificação da proteína por Western Blot. Todas as pistas são carregadas com 100 ng de proteína. (A) resultados de um borrão ocidental com anti-6x histag. (B) resultados de um borrão ocidental com um anticorpo monoclonal 167-D-IV HIV-1. As pistas são rotuladas como: M = marcador de peso molecular; 1 = lisado; 2 = fluxo através; 3 = primeira lavagem; 4 = lavagem de isopropanol (segunda lavagem); 5 = terceira lavagem; 6 = frações de volume agrupadas 56 \ u201261 (primeiro pico de eluição), 7 = facções de volume agrupadas 62 \ u201267 (entre os dois picos), 8 = frações de volume agrupadas 68 \ u201278 (segundo pico de eluição). A proteína alvo com o tamanho de banda esperado de 18, 7 kDa como a banda monomérica SHB-SAPN é indicada por uma seta preta. Bandas extras nas faixas 7 e 8 são dímeros, trimers e multários do SHB-SAPN (seta vermelha). Por favor clique aqui para ver uma versão maior desta figura.
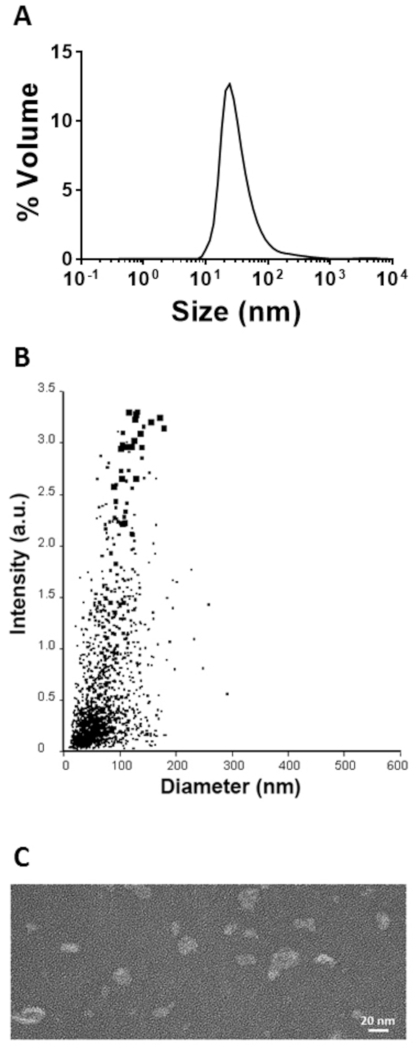
Figura 5: caracterização do SHB-SAPN redobrado. (A). distribuição granulométrica conforme determinado pela DLS. (B) granulometria determinada pelo rastreamento de nanopartículas (sistema). (C) visualização das partículas de SHB-SAPN por tem. Por favor clique aqui para ver uma versão maior desta figura.
| Amostra | Endotoxina (EU/mL) | Endotoxina (EU/μg de proteína) |
| SAPN com isopropanol Wash | 2, 2 | 0, 1 |
| SAPN sem isopropanol Wash | > 50 anos | > 0,25 |
| Controle negativo | Abaixo do nível de detecção | N/A |
Tabela 1: níveis de endotoxina de SHB-SAPNs redobrado. Os níveis de endotoxina em amostras de SHB-SAPN purificados com ou sem lavagem de isopropanol apresentaram-se como unidades de endotoxina/mL e unidades de endotoxina/μg de proteína SHB-SAPN.
Discussão
A nanotecnologia oferece muitas vantagens e soluções para o desenvolvimento de vacinas de subunidades. Nanovaccines pode repetidamente apresentar antígenos como particulados ao sistema imunológico hospedeiro aumentando a imunogenicidade26. Embora existam muitos tipos diferentes de nanovaccines, acreditamos que os compostos de proteína projetada de novo parecem ser a abordagem mais forte para o desenvolvimento da vacina1. Podem ser projetados sem nenhuma homologia da seqüência às proteínas do anfitrião e apresentar o antígeno do interesse perto do nativo-como a conformação ao fornecer o baixo custo de produção e rendimentos elevados do produto. Um excelente exemplo dessa abordagem é a tecnologia SAPN, que temos aplicado a vacinas contra múltiplas doenças infecciosas7. Abordando as dificuldades no desenvolvimento da vacina HIV-1, nós projetamos um núcleo SHB-SAPN original para apresentar eficazmente o antígeno de V1V2 em uma conformação de trimérico nativa-como9. Muitos alvos vacinais, particularmente para doenças virais, estão atuais como trímeros27. Este fenômeno indica que nosso projeto de SAPN tem implicações largas para o desenvolvimento de vacinas do subunidade.
Neste método, demonstramos como produzir SHB-SAPNs em um sistema de expressão de E. coli . Nós expressamos rendimentos elevados da proteína (aproximadamente 6 mg/100 mL da cultura). A proteína continha 10 histidinas e foi facilmente purificada usando uma cromatografia de afinidade metálica imobilizada com a coluna Ni2 + . Este comprimento do his-tag foi encontrado para ser o ideal para o rendimento o mais elevado da proteína. A proteína purificada continha o comprimento total da proteína projetada como indicado pela presença do N-terminal histag e da repetição de criselefantina terminal C. Foram utilizadas técnicas amplamente aceitas e otimizadas para a expressão, produção e caracterização do núcleo SHB-SAPN. A falta da produção da proteína full-length durante o desenvolvimento de um SAPN que contem um epítopo novo da proteína poderia indicar um problema da expressão do gene na pilha de anfitrião. Se isso acontecer, o gene e o sistema de expressão devem ser redesenhados e adaptados ao protocolo descrito. A modificação do tempo ou da intensidade da sonicação também pode aumentar a concentração da proteína de comprimento total prevista.
As partículas redobradas estavam na escala esperada do tamanho (20 a 100 nanômetro)8,24,25 como determinado por DLS e pela análise de seguimento da nanopartícula. Esses resultados foram confirmados com o uso de TEM. Se há uns problemas nesta etapa, é normalmente devido a um problema com o pH ou a força iónica do amortecedor de redobramento. Quando partículas de tamanho grande são detectadas nas técnicas de dimensionamento de partículas, ela indica agregação, que pode ser evitada aumentando o pH do buffer de redobramento. Se as partículas não forem detectadas pela DLS, verifique a concentração da proteína e verifique o pH do tampão. A concentração final de proteínas para DLS deve ser de pelo menos 100 μg/mL. Se a concentração não é o problema, indica a abundância de partículas pequenas, incompletamente formadas, cuja concentração pode ser reduzida diminuindo o pH. Alternativamente, a concentração do cloreto de sódio pode ser ajustada à escala a melhor para minimizar a presença de partículas com tamanho indesejado.
Finalmente, usando uma etapa da lavagem do isopropanol durante a purificação nós pudemos reduzir contaminando LPS do anfitrião e. coli a 0, 1 eu/ΜG de SAPN que está abaixo do limite da administração do alimento e da droga (FDA) de 5 UE/quilograma do peso corporal para produtos injetáveis o nível 28. this pode mais ser reduzido usando uma coluna da troca do aníon igualmente conhecida como a coluna de Q. Se os altos níveis de endotoxina ainda estão presentes, verifique todos os materiais que foram utilizados para a preparação do tampão. Recorde usar somente os produtos vidreiros depyrogenated e o plasticware livre da endotoxina neste método.
Estes resultados indicam que nós desenvolvemos com sucesso um método para produzir o núcleo de SHB-SAPN que pode ser usado para estudos pre-Clinical da imunização. Este método com apenas ligeiras modificações, se houver, pode ser aplicado à purificação de SHB-SAPNs quando um antígeno de interesse é adicionado. Usando esse método como um ponto de partida uma das principais alterações está na etapa de eluição. Proteínas diferentes eluir em concentrações diferentes do imidazol que devem ser determinadas experimentalmente. A outra diferença principal pode ser a composição do buffer de redobramento. A otimização exigiria testar diferentes condições de pH, bem como forças iônicas.
Em consideração do trabalho futuro, somente duas modificações ligeiras são necessárias para permitir a aplicação humana do SHB-SAPN. O primeiro é que o vetor da expressão precisa de ser mudado a um marcador selecionável da resistência do canamicina devido à alergia da ampicilina nos seres humanos29. A outra exigência principal da fabricação da proteína para o uso humano é produzir o SHB-SAPN em meios produto-livres animais. Um estudo de pequena escala já indicou um rendimento razoável de proteínas em uma mídia baseada em plantas. O trabalho apresentado aqui é facilmente escalável para a produção final GMP como demonstrado com um candidato a vacina contra a malária, FMP01416. Esta produção de grande escala FMP014-SAPN incluiu tanto a troca aniónica e as etapas de troca catiónica para reduzir ainda mais LPS e Ni2 + conteúdo do produto final. Este SAPN bacteriano-expressado foi aumentado já para um próximo ensaio clínico de fase 1/2a.
Divulgações
Os pontos de vista expressos são os dos autores e não devem ser interpretados para representar as posições do exército dos EUA ou do departamento de defesa. Peter Burkhard tem um interesse na empresa chamada Alpha-O peptídeos AG e tem patentes sobre a tecnologia. Os demais autores não têm afiliação ou envolvimento financeiro com qualquer empresa com interesse financeiro no assunto apresentado neste artigo.
Agradecimentos
Este trabalho foi apoiado por um acordo cooperativo (W81XWH-11-2-0174) entre a Fundação Henry M Jackson para o avanço da medicina militar, Inc., e o departamento de defesa dos EUA. O anticorpo anti-HIV-1 gp41 mAb 167-D IV foi recebido do Dr. Susan Zolla-Pazner através do programa de reagente de AIDS da NIH.
Materiais
| Name | Company | Catalog Number | Comments |
| 10x Tris/Glycine/SDS | BioRad | 1610732 | 1 L |
| 2-Mercaptoethanol | BioRad | 1610710 | 25 mL |
| 2-propanol | Fisher | BP26181 | 4 L |
| 2x Laemmli Sample Buffer | BioRad | 1610737 | 30 mL |
| 40ul Cuvette Pack of 100 with Stoppers | Malvern Panalytical | ZEN0040 | 100 pack |
| 4–20% Mini-PROTEAN TGX Precast Protein Gels, 10-well, 30 µl | BioRad | 4561093 | 10 pack |
| Ampicillin | Fisher | BP1760-25 | 25 g |
| Anti-6X His tag antibody [HIS.H8] | AbCam | ab18184 | 100 mg |
| Anti-HIV-1 gp41 Monoclonal (167-D IV) | AIDS Reagent Repository | 11681 | 100 mg |
| BCIP/NBT Substrate, Solution | Southern Biotech | 0302-01 | 100 mL |
| Corning Disposable Vacuum Filter/Storage Systems | Fisher | 09-761-108 | A variety of sizes |
| Formvar/Carbon 400 mesh, Copper approx. grid hole size: 42µm | Ted Pella, Inc | 01754-F | 25 pack |
| GE Healthcare 5 mL HisTrap HP Prepacked Columns | GE HealthCare | 45-000-325 | 5 pack |
| Glycerol | Fisher | BP229-4 | 4 L |
| Goat Anti-Mouse IgG H&L (Alkaline Phosphatase) | ABCam | ab97020 | 1 mg |
| Imidazole | Fisher | O3196-500 | 500 g |
| Instant NonFat Dry Milk | Quality Biological | A614-1003 | 10 pack |
| Kinetic-QCL Kinetic Chormogenic LAL Assay | Lonza Walkersville | 50650U | 192 Test Kit |
| LAL Reagent Grade Multi-well Plates | Lonza Walkersville | 25-340 | 1 plate |
| Magic Media E. coli Expression Medium | ThermoFisher | K6803 | 1 L |
| MilliporeSigma Millex Sterile Syringe 0.22 mm Filters | Millipore | SLGV033RB | 250 pack |
| Mouse Anti-Human IgG Fc-AP | Southern Biotech | 9040-04 | 1.0 mL |
| One Shot BL21 Star (DE3) Chemically Competent E. coli | ThermoFisher | C601003 | 20 vials |
| Precision Plus Protein Unstained Protein Standards, Strep-tagged recombinant, | BioRad | 1610363 | 1 mL |
| Slide-A-Lyzer Dialysis Cassettes, 10K MWCO, 12 mL | ThermoFisher | 66810 | 8 pack |
| Sodium Chloride | Fisher | BP358-212 | 2.5 kg |
| Sodium Phosphate Monobasic | Fisher | BP329-500 | 500 g |
| Tris Base | Fisher | BP152-1 | 1 kg |
| Tris-(2-carboxyethyl)phosphine hydrochloride | Biosynth International | C-1818 | 100 g |
| Uranyl Acetate, Reagent, A.C.S | Electron Micoscopy Services | 541-09-3 | 25 g |
| Urea | Fisher | BP169-500 | 2.5 kg |
| Whatman qualitative filter paper | Sigma Aldrich | WHA10010155 | pack of 500 |
| Name | Company | Catalog Number | Comments |
| Equipment | |||
| ChromLab Software ver 4 | BioRad | 12009390 | Software |
| Epoch 2 Microplate Spectrophotometer | BioTek | EPOCH2 | Plate Reader |
| Fiberlite F14-14 x 50cy Fixed-Angle Rotor | ThermoFisher | 096-145075 | Rotor |
| Gel Doc EZ Gel Documentation System | BioRad | 1708270 | Gel Imager for Stain free Gels |
| JEOL TEM | JEOL | 1400 | Transmission Electron Microscope |
| Mini-PROTEAN Tetra Vertical Electrophoresis Cell for Mini Precast Gels | BioRad | 1658004 | To run gels |
| NanoDrop One Microvolume UV-Vis Spectrophotometer | ThermoFisher | ND-ONE-W | For Protein Concentration |
| NanoSight NS300 | Malvern Panalytical | Particle Sizing | |
| NanoSight NTA software NTA | Malvern Panalytical | Particle Sizing | |
| New Brunswick Innova 44/44R | Eppendorf | M1282-0000 | Incubator/Shaker |
| NGC Quest 10 Chromatography System | BioRad | 7880001 | FPLC to aid in protein purification |
| PELCO easiGlow Glow Discharge Cleaning System | Ted Pella, INC | 91000S | To clean grids |
| PowerPac Universal Power Supply | BioRad | 1645070 | To run gels |
| Rocker Shaker | Daigger | EF5536A | For Western |
| Sonifer 450 | Branson | also known as 096-145075 | Sonicator |
| Thermo Scientific Sorvall LYNX 4000 Superspeed Centrifuge | ThermoFisher | 75-006-580 | Centrifuge |
| Trans-Blot Turbo Mini Nitrocellulose Transfer Packs | BioRad | 1704158 | For Western |
| Trans-Blot Turbo Transfer System | BioRad | 1704150 | For Western |
| Vortex-Genie 2 | Daigger | EF3030A | Vortex |
| Zetasizer Nano ZS | Malvern Panalytical | Particle Sizing | |
| Zetasizer Software | Malvern Panalytical | Particle Sizing |
Referências
- Karch, C. P., Burkhard, P. Vaccine technologies: From whole organisms to rationally designed protein assemblies. Biochemical Pharmacology. 120, 1-14 (2016).
- Snapper, C. M. Distinct Immunologic Properties of Soluble Versus Particulate Antigens. Frontiers in Immunology. 9, 598(2018).
- Kelly, H. G., Kent, S. J., Wheatley, A. K. Immunological basis for enhanced immunity of nanoparticle vaccines. Expert Review of Vaccines. , 1-12 (2019).
- Yeates, T. O. Geometric Principles for Designing Highly Symmetric Self-Assembling Protein Nanomaterials. Annual Review of Biophysics. 46, 23-42 (2017).
- Marcandalli, J., et al. Induction of Potent Neutralizing Antibody Responses by a Designed Protein Nanoparticle Vaccine for Respiratory Syncytial Virus. Cell. 176 (6), 1420-1431 (2019).
- Ross, J. F., et al. Decorating Self-Assembled Peptide Cages with Proteins. ACS Nano. 11 (8), 7901-7914 (2017).
- Karch, C. P., Matyas, G. R., Burkhard, P., Beck, Z. Self-Assembling Protein Nanoparticles: implications for HIV-1 vaccine development. Nanomedicine (Lond). 13 (17), 2121-2125 (2018).
- Raman, S., Machaidze, G., Lustig, A., Aebi, U., Burkhard, P. Structure-based design of peptides that self-assemble into regular polyhedral nanoparticles). Nanomedicine. 2 (2), 95-102 (2006).
- Karch, C. P., et al. Design and characterization of a self-assembling protein nanoparticle displaying HIV-1 Env V1V2 loop in a native-like trimeric conformation as vaccine antigen. Nanomedicine. , (2018).
- Karch, C. P., et al. The use of a P. falciparum specific coiled-coil domain to construct a self-assembling protein nanoparticle vaccine to prevent malaria. Journal of Nanobiotechnology. 15 (1), 62(2017).
- Li, J., et al. A self-adjuvanted nanoparticle based vaccine against infectious bronchitis virus. PLoS One. 13 (9), e0203771(2018).
- Wahome, N., et al. Conformation-specific display of 4E10 and 2F5 epitopes on self-assembling protein nanoparticles as a potential HIV vaccine. Chemical Biology & Drug Design. 80 (3), 349-357 (2012).
- El Bissati, K., et al. Effectiveness of a novel immunogenic nanoparticle platform for Toxoplasma peptide vaccine in HLA transgenic mice. Vaccine. 32 (26), 3243-3248 (2014).
- Kaba, S. A., et al. A nonadjuvanted polypeptide nanoparticle vaccine confers long-lasting protection against rodent malaria. Journal of Immunology. 183 (11), 7268-7277 (2009).
- Kaba, S. A., et al. Protective antibody and CD8+ T-cell responses to the Plasmodium falciparum circumsporozoite protein induced by a nanoparticle vaccine. PLoS One. 7 (10), e48304(2012).
- Seth, L., et al. Development of a self-assembling protein nanoparticle vaccine targeting Plasmodium falciparum Circumsporozoite Protein delivered in three Army Liposome Formulation adjuvants. Vaccine. 35 (41), 5448-5454 (2017).
- Kaba, S. A., et al. Self-assembling protein nanoparticles with built-in flagellin domains increases protective efficacy of a Plasmodium falciparum based vaccine. Vaccine. 36 (6), 906-914 (2018).
- El Bissati, K., et al. Protein nanovaccine confers robust immunity against Toxoplasma. NPJ Vaccines. 2, 24(2017).
- Karch, C. P., et al. Vaccination with self-adjuvanted protein nanoparticles provides protection against lethal influenza challenge. Nanomedicine. 13 (1), 241-251 (2017).
- Haynes, B. F., et al. Immune-correlates analysis of an HIV-1 vaccine efficacy trial. New England Journal of Medicine. 366 (14), 1275-1286 (2012).
- Rerks-Ngarm, S., et al. Vaccination with ALVAC and AIDSVAX to prevent HIV-1 infection in Thailand. New England Journal of Medicine. 361 (23), 2209-2220 (2009).
- O' Connell, R. J., Kim, J. H., Excler, J. L. The HIV-1 gp120 V1V2 loop: structure, function and importance for vaccine development. Expert Review of Vaccines. 13 (12), 1489-1500 (2014).
- Babapoor, S., et al. A Novel Vaccine Using Nanoparticle Platform to Present Immunogenic M2e against Avian Influenza Infection. Influenza Research and Treatment. 2011, 126794(2011).
- Indelicato, G., Burkhard, P., Twarock, R. Classification of self-assembling protein nanoparticle architectures for applications in vaccine design. Royal Society Open Science. 4 (4), 161092(2017).
- Indelicato, G., et al. Principles Governing the Self-Assembly of Coiled-Coil Protein Nanoparticles. Biophysical Journal. 110 (3), 646-660 (2016).
- Doll, T. A., Raman, S., Dey, R., Burkhard, P. Nanoscale assemblies and their biomedical applications. Journal of the Royal Society Interface. 10 (80), 20120740(2013).
- Rey, F. A., Lok, S. M. Common Features of Enveloped Viruses and Implications for Immunogen Design for Next-Generation Vaccines. Cell. 172 (6), 1319-1334 (2018).
- Bacterial Endotoxins. United States Pharmacopeia (USP). , Ch. 85 (2011).
- Points to Consider (PTC) in the Characterization of Cell Lines Used to Produce Biologicals. , Food and Drug Adminstration. Maryland. Available from: https://www.fda.gov/media/76255/download (1993).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados