Method Article
Production de E. coli-les nanoparticules de protéines auto-assemblantes exprimées pour les vaccins nécessitant une présentation de l'épitope trimmérique
Dans cet article
Résumé
Une méthode détaillée est fournie ici décrivant la purification, le repliement et la caractérisation des nanoparticules de protéines auto-assemblantes (SAPN) pour le développement de vaccins.
Résumé
Les nanoparticules de protéines auto-assemblantes (SAPN) fonctionnent comme des écrans d'antigènes répétitifs et peuvent être utilisées pour développer un large éventail de vaccins pour différentes maladies infectieuses. Dans cet article nous démontrons une méthode pour produire un noyau de SAPN contenant un ensemble de faisceau de six-helix (SHB) qui est capable de présenter des antigènes dans une conformation trimeric. Nous décrivons l'expression du SHB-SAPN dans un système d'E. coli, ainsi que les étapes nécessaires de purification de protéine. Nous avons inclus une étape de lavage d'isopropanol pour réduire le lipopolysaccharide bactérien résiduel. Comme indication de l'identité et de la pureté de protéine, la protéine a réagi avec les anticorps monoclonaux connus dans les analyses occidentales de tache. Après le repli, la taille des particules est tombée dans la plage prévue (20 à 100 nm), ce qui a été confirmé par la diffusion dynamique de la lumière, l'analyse du suivi des nanoparticules et la microscopie électronique de transmission. La méthodologie décrite ici est optimisée pour le SHB-SAPN, cependant, avec seulement de légères modifications, il peut être appliqué à d'autres constructions SAPN. Cette méthode est également facilement transférable à la production à grande échelle pour la fabrication de GMP pour les vaccins humains.
Introduction
Alors que le développement traditionnel des vaccins s'est concentré sur les agents pathogènes inactivés ou atténués, l'accent des vaccins modernes s'est déplacé vers les vaccins sous-unis1. Cette approche peut mener à une réponse plus ciblée et à des candidats vaccins potentiellement plus efficaces. Cependant, l'un des principaux inconvénients est que les vaccins sous-unis ne sont pas des particules comme des organismes entiers qui peuvent entraîner une immunogénicité réduite2. Une nanoparticule comme système répétitif d'affichage d'antigène peut avoir les avantages de l'approche ciblée de vaccin de sous-unité aussi bien que la nature particulaire de l'organisme entier1,3.
Parmi les types existants de nanovaccins, les assemblages de protéines conçus rationnellement permettent la conception et le développement de vaccins candidats qui peuvent présenter plusieurs copies de l'antigène potentiellement dans une conformation indigène1,4 ,5,6. Un exemple de ces assemblages de protéines sont les nanoparticules de protéines auto-assemblantes (SAPNs)7. Les SAPN sont basés sur des domaines enroulés et sont traditionnellement exprimés en Escherichia coli8. Les vaccins SAPN candidats ont été développés pour une variété de maladies telles que le paludisme, le SRAS, la grippe, la toxoplasmose, et le VIH-19,10,11,12,13 , 14 (en) , 15 Annonces , 16 Annonces , 17 Annonces , 18 ans, états-unis qui , 19. La conception de chaque candidat SAPN est spécifique à l'agent pathogène d'intérêt, cependant, les techniques de production, de purification et de repliement sont généralement largement applicables.
L'un de nos intérêts actuels est un vaccin efficace contre le VIH-1. Dans RV144, le seul essai clinique de phase III d'un vaccin contre le VIH-1 qui a démontré une efficacité modeste, le risque réduit d'infection a été corrélé avec les anticorps IgG à la boucle V1V2 de la protéine enveloppe20,21. La présentation trimeric indigène-comme de cette région est pensée pour être importante pour l'immunogénicité protectrice22. Pour présenter la boucle V1V2 dans la conformation native aussi proche que possible, nous avons développé une preuve de principe du vaccin SAPN candidat qui contenait l'enveloppe VIH-1 paquet de six hélices (SHB) pour présenter la boucle V1V2 dans la conformation correcte9 . Ce candidat a été identifié par les anticorps monoclonaux connus à la protéine d'enveloppe de HIV-1. Les souris immunisées avec V1V2-SHB-SAPN ont soulevé des anticorps spécifiques V1V2, qui, plus important encore, liés au gp70 V1V2, l'épitopes conformationnelle correcte9. Le noyau SHB-SAPN pourrait avoir d'autres fonctions au-delà du rôle de porteur de la boucle V1V2 VIH-1. Nous décrivons ici une méthodologie détaillée pour l'expression, la purification, le repliement et la validation du noyau SHB-SAPN. La sélection de la séquence, la conception des nanoparticules, le clonage moléculaire et la transformation d'E. coli ont déjà été décrits9.
Protocole
1. Expression de la protéine SHB-SAPN dans E. coli BL21(DE3)
- Mélanger 95 ml de composant A et 5 ml de composant B du support dans un flacon Erlenmeyer en verre stérile de 2 L selon les instructions du fabricant (voir le Tableau des matériaux). Ajouter l'ampicilline à une concentration finale de 100 g/mL.
- Inoculer les médias avec E. coli d'une culture de stock de glycérol précédemment établie. Incuber la culture à 30 oC avec des secousses à 200 rotations par minute (rpm) pendant 48 h.
REMARQUE : Le stock utilisé de E. coli BL21 (DE3) contenait le vecteur d'expression résistant à l'ampicilline23 avec le gène SHB-SAPN. Bien que le protocole général des médias recommande 24 h d'incubation à 37 oC, 48 h d'incubation à 30 oC ont donné un rendement plus élevé pour le SHB-SAPN. - Transférer la culture dans deux tubes coniques de 50 ml. Centrifuger les tubes à 4 000 x g pendant 10 min avec un rotor à angle fixe à 4 oC. Retirez le supernatant et enregistrez le granule pour récolter les cellules.
REMARQUE : Le granule cellulaire peut être traité immédiatement ou congelé à -80 oC jusqu'à ce qu'il soit utilisé.
2. Lysis de E. coli BL21(DE3) par sonication
REMARQUE : Utilisez des plastiques et des verreries non pyrogéniques cuits au four à 250 oC pendant au moins 30 min. Tris(2-carboxyethyl)phosphine (TCEP) comme agent réducteur brise les liaisons de disulfure à l'intérieur et entre les protéines. TCEP est nécessaire dans les tampons pendant ce protocole si l'antigène affiché contient des liaisons S-S. Pour le noyau SHB-SAPN seulement, la présence de TCEP dans les tampons n'est pas essentielle.
- Préparer un tampon sans imidazole (8 M Urea, 50 mM de phosphate de sodium monobaselique, base Tris de 20 mM, 5 mM TCEP) pH 8,0 (ajusté avec 5 N NaOH) et filtrer à l'aide d'une unité de filtration de bouteille sous vide de 0,2 m.
- Resuspendre les cellules granulées (à partir de l'étape 1.3) avec 40 ml de tampon sans imidazole dans un tube conique de 50 ml. Sonicate les cellules suspendues avec une sonde sur la glace pendant 5 min (4 s de sonication, 6 s de repos) avec une sortie de sonication de 150 W.
- Centrifuger le lysate cellulaire (40 ml) à 29 000 x g à 4 oC pendant 25 min dans un rotor à angle fixe pour générer un supernatant clarifié. Transférer le supernatant dans un flacon stérile de 150 ml et jeter la pastille. Diluer le supernatant à 100 ml à l'aide du tampon sans imidazole (plus tard dans le protocole appelé « échantillon »).
REMARQUE : Cette étape de dilution est nécessaire pour éviter que la pression du système FPLC ne devienne trop élevée pendant le chargement du lysate sur la colonne.
3. Purification des protéines à l'aide d'une colonne
REMARQUE : Ce protocole a été exécuté à l'aide d'un instrument FPLC, mais il peut être adapté au débit gravitationnel.
- Préparer les tampons suivants et les filtrer à l'aide d'une unité de filtration de bouteille sous vide de 0,22 m : (i) tampon sans imidazole « Tampon A » (8 M Urea, 50 mM de phosphate de sodium monobasic, base Tris de 20 mM, 5 mM TCEP) pH 8,0; (ii) tampon imidazole de 500 mM «Buffer B» (8 M Urea, 50 mM de phosphate de sodium monobasic, base Tris de 20 mM, 5 mM TCEP, 500 mM Imidazole) pH 8,0; et (iii) lavage d'isopropanol (20 mM Tris, 60% isopropanol) pH 8.0.
REMARQUE : le pH pour chaque tampon a été ajusté avec 5 N NaOH. - Équilibrez la colonne De Sa.
REMARQUE : Pour la production à l'échelle du laboratoire, ce protocole utilise une colonne préemballée de 5 ml, mais toute colonne de plus grande taille peut être utilisée.- Ouvrez le logiciel FPLC et cliquez sur l'option Nouvelle méthode. Il s'ouvrira immédiatement au menu Paramètres Méthode. Sous le menu déroulant pour la position de colonne choisissez C1 port 3.
- Sur le menu montré par technique déposer choisir affinité. Sur le menu de type Colonne déroulant choisir d'autres, Histrap HP, 5 mL. Le volume de la colonne et les cases de pression seront automatiquement réglés sur les valeurs appropriées.
- Cliquez sur le bouton Decontour Méthode. Faites glisser les boutons suivants du menu popup de la bibliothèque Phase : équilibre, applicationd'échantillon, lavage de colonne,et élution à côté de la flèche dans cet ordre exact. Fermez le menu de la bibliothèque Phase.
- Cliquez sur le bouton d'équilibre. Les valeurs indiquées dans le tableau doivent être « tampon initial B » (4 %), « tampon final B » (4 %) et « volume (CV) » 5.
- Cliquez sur la case d'application Sample. Dans la boîte de chargement de l'échantillon, cliquez sur le bouton radio pour Injecter l'échantillon sur la colonne avec pompe d'échantillon. Assurez-vous que la boîte à côté du débit d'utilisation des paramètres de la méthode est vérifiée dans l'injection d'échantillon avec la boîte de pompe du système. A côté de la boîte de volume sur le côté droit de l'écran changer la valeur à 20 ml.
- Cliquez sur le bouton de lavage Colonne. Les valeurs indiquées dans le tableau doivent être « tampon initial B » (4 %), « tampon final B » (4 %) et « volume (CV) » 5. A côté du schéma de collecte de fractions unclick la boîte Enable.
- Cliquez sur le bouton d'élution. Les valeurs indiquées dans le tableau doivent être « tampon initial B » 0 %, « tampon final B » 100 % et « volume (CV) » 5. À côté du schéma de collecte de fractions, cliquez sur la case Activer. Décochez la taille de la fraction d'utilisation à partir des paramètres de la méthode et ajustez la taille des fractions à 4 ml dans la boîte de remplissage ci-dessous.
- Cliquez sur l'enregistrement comme bouton sur le dessus du logiciel. Nommez le fichier "équilibrité".
- Connectez une colonne préemballée de 5 mL à la colonne correspondante du port 3 sur le FPLC. Les tubes de la pompe A et de la pompe B ainsi que le tube de la pompe de l'échantillon doivent être placés dans de l'eau déionisée filtrée de 0,22 m. Exécutez le programme d'équilibre.
- Placez à la fois la pompe A et la pompe B ainsi que le tube de pompe échantillon dans le tampon sans imidazole (Buffer A) et exécutez le protocole d'équilibre à nouveau.
- Lier l'échantillon à la colonne et purifier la protéine.
- Ouvrez le logiciel FPLC et cliquez sur l'option Nouvelle méthode. Il s'ouvrira immédiatement au menu Paramètres Méthode. Sous le menu déroulant pour la position colonne choisissez C1 port 3. Sur le menu montré par technique déposer choisir affinité. Sur le type de colonne menu déroulant choisir d'autres, Histrap HP, 5 mL. Le volume de la colonne et les cases de pression seront automatiquement réglés sur les valeurs appropriées.
- Cliquez sur le bouton Decontour Méthode. Faites glisser les boutons du menu popup de la bibliothèque Phase : équilibre,application d'échantillon, lavage de colonne (Wash 1), lavage de colonne (Wash 2), lavage de colonne (Wash 3), et Elution à côté de la flèche dans ce ordre exact. Fermez le menu de la bibliothèque Phase.
- Cliquez sur le bouton Équilibre. Les valeurs indiquées dans le tableau doivent être « tampon initial B » 4 %, « tampon final B » 4 % et « volume (CV) » 5.
- Cliquez sur la case d'application Sample. Dans la boîte de chargement de l'échantillon cliquez sur le bouton radio pour inject échantillon sur la colonne avec pompe échantillon. Assurez-vous que la boîte à côté du débit d'utilisation des paramètres de la méthode est vérifiée dans l'injection d'échantillon avec la boîte de pompe du système.
- À côté de la boîte de volume sur le côté droit de l'écran changer la valeur à 100 ml. À côté du schéma de collecte de fractions, cliquez sur le bouton Activer. Décochez la taille de la fraction d'utilisation à partir de la boîte de réglages de méthode, puis modifiez la taille de la fraction à 4 ml.
- Cliquez sur le premier bouton de lavage de colonne (Wash 1). Les valeurs indiquées dans le tableau doivent être « tampon initial B » 4 %, « tampon final B » 4 % et « volume (CV) » 10. À côté du schéma de collecte de fractions, cliquez sur la case Activer. Décochez la taille de la fraction d'utilisation à partir des paramètres de la méthode, puis modifiez la taille de la fraction à 4 ml.
- Cliquez sur le bouton de lavage de la deuxième colonne (Wash 2). Les valeurs indiquées dans le tableau doivent être « tampon initial B » 0 %, « tampon final B » 0 % et « volume (CV) » 5. À côté du schéma de collecte de fractions, cliquez sur la case Activer. Décochez la taille de la fraction d'utilisation à partir des paramètres de la méthode, puis modifiez la taille de la fraction à 4 ml.
- Cliquez sur le bouton de lavage de la troisième colonne (Wash 3). Les valeurs indiquées dans le tableau doivent être « tampon initial B » 0 %, « tampon final B » 0 % et « volume (CV) » 5. À côté du schéma de collecte de fractions cliquez sur l'Activer . Décochez la taille de la fraction d'utilisation à partir de la boîte de réglages de méthode, puis modifiez la taille de la fraction à 4 ml.
- Cliquez sur le bouton Elution. Dans le tableau à droite cliquez sur les informations énumérées et, sur le menu qui vient, cliquez sur Supprimer l'étape. Faites glisser le bouton de gradient isocratique sur la table deux fois, de sorte qu'il y ait deux entrées.
- La valeur de la première entrée doit être la suivante : « tampon initial B » 30 %, « tampon final B » 30 % et « Volume (CV) » 10. La valeur de la deuxième entrée doit être la suivante : « tampon initial B » 100 %, « tampon final B » 100 % et « volume (CV) » 10. À côté du schéma de collecte de fractions, cliquez sur le bouton Activer. Cliquez sur la boîte à côté de la taille de fraction d'utilisation des paramètres de la méthode.
- Cliquez sur le bouton Enregistrer comme sur le dessus du logiciel. Nommez le fichier "purification". Pompe Un tube du FPLC doit être placé dans le tampon de lavage sans imidazole tandis que le tube de la pompe B doit être placé dans le tampon imidazole de 500 mM. Le tube de la pompe de l'échantillon doit être placé dans l'échantillon de 100 ml.
- Exécutez le programme de « purification » et attendez le moment où l'isopropanol de 60 % est nécessaire (Wash 2). Pause du programme, déplacer la pompe Un tube de l'imidazole-libre de lavage dans le lavage de 60% isopropanol. Redémarrez le programme.
- Une fois l'étape isopropanol terminée, mettre en pause le programme à nouveau et déplacer la pompe A tube de retour dans le tampon de lavage sans imidazole. Redémarrez le programme de purification (le reste de la course est automatisé).
4. Évaluation de la pureté et identification des protéines par SDS-PAGE
- Combinez toutes les fractions correspondant à (i) flow-through (le lysate cellulaire qui ne se lie pas à sa colonne), (ii) Laver 1, (iii) Laver 3 (60% isopropanol lavage), et (iv) Laver 3 dans des tubes coniques séparés de 50 ml. Ne pas combiner les fractions de 2 ml des étapes d'élution.
- Mélanger 15 L de chacune des fractions mises en commun et toutes les fractions des étapes d'élution avec 2x laemmli échantillon tampon dans un tube microcentrifuge de 0,5 mL et les dénaturer à 95 oC pendant 10 min.
- Tandis que les dénatures de protéine, installez l'appareil de fonctionnement de gel avec 3 gels de polyacrylamide prémoulés de 4 à 20% dans le tampon de fonctionnement 1x Tris-glycine SDS-PAGE.
- Chargez 8 ll de marqueur de poids moléculaire au premier puits et 30 l d'échantillon dénaturé aux autres puits du gel. Exécuter les gels à 200 V jusqu'à ce que le front de teinture frappe le fond du gel (environ 30 min). Retirer les gels de l'appareil et rincer brièvement à l'eau déionisée. Imagez le gel immédiatement à l'aide du système d'imagerie sans taches.
- Identifiez les fractions qui contiennent des bandes protéiques de la bonne taille (18,07 kDa). Mettez en commun toutes ces fractions.
5. Identification des protéines par tache occidentale
- Exécuter une tache occidentale à l'aide d'un anticorps spécifique (anti-6x HisTag) et un anticorps spécifique SHB (167-D-IV) pour identifier la protéine purifiée pleine longueur. L'anticorps anti-6x HisTag reconnaît le terminus N de la protéine et l'anticorps 167-D-IV reconnaît le terminus C démontrant la présence de la protéine pleine longueur.
- Déterminer la concentration en protéines du (i) flow-through, (ii) Wash 1, (iii) Wash 2 (60% isopropanol wash), (iv) Wash 3, et toutes les fractions de l'élution étapes avec l'instrument spectromètre à une absorption de 280 nm. Générer des dilutions qui contiennent 100 ng de protéines dans 15 'L de tampon sans imidazole pour chacun de ces groupes.
- Ajouter 15 lde de tampon d'échantillon 2x Laemmli à chaque 15 l des échantillons et les dénaturer comme à l'étape 4.1. Une fois la dénaturation terminée, déspinez les tubes pour s'assurer que toutes les protéines peuvent être transférées.
- Chargez les échantillons et le marqueur pré-taché dans un gel polyacrylamide prémoulé sans taches de 4 à 20 %. Faites fonctionner l'électrophoresis à 200 V jusqu'à ce que le front de teinture atteigne le fond du gel.
- Pendant que le gel fonctionne, faire 1 L de TBS-T (20 mM Tris, 150 mM NaCl, et 0,1% Tween 20) et 200 ml de 5% de lait non gras dans TBS-T.
- Utilisez un système de transfert de taches occidentales pour transférer les protéines sur une membrane de nitrocellulose. Utilisez une pile de transfert pré-assemblée et placez le gel dessus. Configurez le système pour fonctionner à 25 V pendant 7 min. Vérifier la présence du marqueur pré-taché sur la membrane de nitrocellulose indiquant un transfert complet.
REMARQUE : Toutes les étapes suivantes sont effectuées sur un shaker orbital réglé à 100 tr/min à température ambiante (RT). - Une fois le transfert terminé, laver deux fois avec le SCT-T pendant 10 min chacun.
- Bloquer les membranes de nitrocellulose (blot) avec 5 % de lait non gras dans le SCT-T pendant au moins 1 h. Laver deux fois avec le SCT-T pendant 10 min chacun.
- Diluer les anticorps primaires 167-D-IV et anti-6x HisTag à 1 mg/mL dans tbS-T (stock Ab). Diluer le stock Abs du 167-D-IV 10,000-fold et l'anti-6x HisTag 5,000-fold en ajoutant 2 L du stock 167-D-IV et 4 -L du stock anti-6x HisTag anticorps à deux tubes différents contenant 20 mL de TBS-T. Ajouter le volume total de 20 ml d'anticorps primaires, un à chaque tache, et couver des taches pendant 1 h. Laver les taches deux fois avec tbsSE-T pendant 10 min.
- Diluer 4 'L de 1 mg/mL de l'anticorps secondaire anti-humain de souris conjugué avec la phosphatase alcaline dans 20 mL de TBS-T (1:5,000 dilution). Diluer 4 'L de 1 mg/mL de l'anticorps secondaire anti-souris de chèvre conjugué à la phosphatase alcaline dans 20 mL de TBS-T (1:5,000 dilution). Ajouter des anticorps secondaires aux taches correspondantes.
REMARQUE: L'anticorps secondaire anti-humain se lie à 167-D et l'anti-souris se lie à l'anti-6x HisTag. Laver les taches deux fois avec tbS-T pendant 10 min. - Ajouter suffisamment de substrat de phosphatase alcaline BCIP/NBT pour couvrir les taches. Développer les taches pendant environ 10 min jusqu'à ce que les bandes apparaissent. Rincer les taches avec l'eau froide du robinet et les laisser sécher avant de les scanner à l'eau à plat.
6. Repliez le SHB-SAPN
- Ajouter la protéine mise en commun (10'u201220 mL au total) à un poids moléculaire de 10 kDa coupé la cassette de dialyse et dialyser en 8 M d'urée, 20 mM Tris, 5% de glycérol, 5 mM TCEP pH 8,5 à RT (18'u201226 'C) du jour au lendemain.
- Dialysez lentement l'urée de l'échantillon en diminuant la concentration d'urée dans le tampon de dialyse dans le sens de l'étape de 2 M tous les 2 h. À une concentration d'urée de 2 M, déplacer l'appareil de dialyse à 4 oC (ne pas utiliser de TCEP dans le tampon de dialyse de cette étape). Terminer le repliement en dialysant l'échantillon en 120 mM d'urée, 20 mM de Tris, 5 % de glycérol, pH 8,5 à 4 oC pendant la nuit.
- Retirer la protéine repliée (SHB-SAPN) de la cassette de dialyse. Filtrer le SHB-SAPN à l'aide d'un fileur de seringues en polyvinylide polyvinylide (PVDF) de 0,22 m. Aliquot SHB-SAPN en tubes stériles et les congeler à -80 oC, laissant au moins 100 L à RT pour des analyses ultérieures.
7. Validation des particules par taille et apparence
- Diffusion dynamique de la lumière (DLS)
- Mesurer la taille moyenne des particules du SHB-SAPN par les paramètres suivants : sélectionner la protéine comme matériau, créer un tampon complexe pour 120 mm d'urée, 20 mM Tris, 5 % de glycérol 25 oC pour la température, sélectionner les cuvettes jetables pour l'analyse, sélectionner les mesure, fixée à 5 pistes.
- Ajouter 45 lde de SHB-SAPN à une cuvette jetable et exécuter le logiciel en cliquant sur la flèche verte. Sélectionnez le volume de pourcentage pour la lecture.
- Analyse du suivi des nanoparticules (NTA)
- Diluer l'échantillon par 1:20 dans le tampon de repli. Faire 10 ml d'échantillon dilué.
- À l'aide de seringues de 3 ml, rincer l'instrument NTA avec le tampon de repliage et charger environ 1,5 ml d'échantillon pour équilibrer l'instrument. Utilisez le reste de l'échantillon pour l'analyse.
- Créez un nouveau SOP dans le logiciel NTA en cliquant sur l'onglet SOP. Sous l'onglet, changer le nombre de captures à 3 et changer le temps de capture à 30 s. Appuyez sur le bouton autofocus sur le côté gauche de l'écran pour mettre l'échantillon au point. Utilisez le bouton de mise au point manuel sur le côté de la machine pour affiner la mise au point.
- Exécuter le SOP créé, lorsque le système invite charger un petit volume d'échantillon avec la seringue. Une fois que le système a pris toutes les captures, il fera automatiquement monter l'écran d'analyse. Faites glisser la barre de limite de détection de sorte que toutes les particules réelles soient marquées de croix rouges. Appuyez sur le bouton d'analyse d'exécution et l'analyse commencera automatiquement.
- Microscopie électronique de transmission (TEM)
- Glow décharge formvar/carbon 400 maille cuivre TEM films de soutien.
- Ajouter 3 ll d'échantillon à une concentration de 0,075 mg/mL sur la grille pendant 30 s. Wick hors du liquide à l'aide d'un papier filtre.
- Laver la grille avec 3 ll d'eau déionisée trois fois, chaque fois en évacuant l'eau avec du papier filtre.
- Ajouter 3 'L de 0.5% d'acétate uranyl au film de support et lui permettre de s'asseoir pendant 30 s. Wick hors de la plupart de l'acétate uranyl, mais laisser un film mince sur la surface. Laisser sécher les échantillons avant de les imagerier au microscope électronique de transmission.
- Échantillons d'images à 80 kV sur un TEM.
8. Détermination des niveaux d'endotoxine dans les échantillons à l'aide d'un lysate d'amoebocyte de limulus cinétique (LAL)
- Retirer le kit et les échantillons du réfrigérateur et laisser l'équilibre vers RT.
- Pour effectuer cet analyse, un lecteur de plaque avec un bloc de chaleur et la possibilité de lire les échantillons pendant 40 lectures à une longueur d'onde de 405 nm à 37 oC est nécessaire. Rédigez un modèle de programme de sorte que les puits soient lus tous les 150 s pour identifier le point de début (l'OD a augmenté de 0,2 par rapport à la première lecture).
- Diluer l'échantillon à la concentration de dose d'immunisation dans le SCP.
REMARQUE : Le pH du tampon de repli age est en dehors de la portée de l'assay LAL. La dilution de l'échantillon dans PBS fixera le pH à la plage acceptable. - Resuspendre l'endotoxine de contrôle dans le volume approprié d'eau LAL sans endotoxine, tel que déterminé par le certificat d'analyse pour générer 50 UE/mL. Vortex vigoureusement le flacon pendant 15 min pour assurer la résuspension complète de l'endotoxine.
- Générez la courbe standard de l'endotoxine en préformant une dilution sérielle 10 fois dans des flacons de verre de l'ordre de 50 UE à 0,005 EU/mL. Pour chaque dilution, ajouter 0,1 ml de la dilution précédente à 0,9 ml d'eau LAL. Vortex vigoureusement après combinaison pendant 1 min.
- Ajoutez des dilutions courbes standard et des échantillons de SHB-SAPN en double à une plaque de 96 puits. Utilisez également l'eau LAL comme un contrôle négatif en double. Préincuber la plaque à 37 oC pendant 15 min.
- Vers la fin de l'incubation, suspendre à nouveau le flacon de réactif d'assay avec 2,6 ml d'eau LAL. Mélanger délicatement le contenu avec une pipette sérologique.
- Ajouter 100 l de réactif d'assay à chaque puits de la plaque de 96 puits. Déplacez rapidement la plaque vers le lecteur de plaque et exécutez le modèle de programme écrit à l'étape 8.2.
- Une fois le programme terminé, générer une courbe standard en utilisant la valeur journal des contrôles par rapport à la valeur journal du temps d'début. Utilisez la formule générée à partir de cette courbe pour calculer la concentration d'endotoxines dans les échantillons.
Résultats
Le SHB-SAPN entièrement assemblé ici est construit sur des séquences protéiques (Figure 1A) qui sont prévues pour se plier en une particule qui contient 60 copies du monomère (Figure 1B). La figure 2 fournit un aperçu de la méthode de production, de purification et d'identification du noyau SHB-SAPN. E. coli d'un stock de glycérol qui contenait un vecteur d'expression pPep-T avec la séquence génétique du noyau SHB-SAPN ont été induits dans BL21 (DE3) E. coli. Des cellules bactériennes ont été avec succès cultivées et lysées sous des conditions dénaturantes et de réduction.
Le lysate total des cellules a été utilisé pour purifier les monomères SHB-SAPN par le FPLC à l'aide d'une colonne Ni2 ( Figure 3A). Le chromatographe FPLC démontre que les protéines ont élifié à 150 mm et 500 mM d'imidazole (figure 3A). Le chromatogramme montre également deux autres pics à 185 ml et 210 ml de volume total correspondant au lavage d'isopropanol et au lavage sans imidazole, respectivement. Les fractions et la pureté de la protéine recombinante ont été identifiées par les gels SDS-PAGE de gradient (figure3B). La protéine d'intérêt était principalement située en fractions 68-u201279 (278-u2012300 mL volume total). Ces fractions ont été combinées pour d'autres analyses. Western blot with anti-His antibody (N-terminal) and 167-D-IV antibody (C-terminus) indicated that the pooled fractions were indeed the protein of interest (Figure 4A,B). Ces taches ont également démontré la présence des multimères SHB-SAPN. Les lavages et les fractions d'élution antérieurs avaient tendance à contenir une concentration plus élevée de protéines multimerisées et étaient donc exclus.
Les échantillons qui contenaient les monomères protéiques d'intérêt ont été pliés dans le SHB-SAPN entièrement assemblé par dialyse. La distribution de la taille des particules a été déterminée par l'analyse du dLS et du suivi des nanoparticules (figure5A,B). Le DLS a identifié des particules d'un diamètre hydrodynamique moyen De 67 nm, tandis que le système NTA a mesuré une taille moyenne de 81 nm. Les légères différences de taille étaient dues aux techniques de dimensionnement des particules, mais la taille des deux analyses était de l'ordre de 20'u2012100 nm8,24,25. Les SHB-SAPN ont été visualisés par TEM et les images montraient des particules individuelles bien formées avec la distribution de la taille obtenue à partir des deux techniques de dimensionnement des particules (figure5C).
Pendant la purification de la protéine, la colonne a été lavée avec l'isopropanol pour diminuer la contamination de LPS dans le produit final de SHB-SAPN. Pour vérifier si le niveau d'endotoxine était acceptable pour l'immunisation, la concentration de LPS dans les échantillons de SHB-SAPN purifiéavec ou sans l'étape de lavage d'isopropanol a été déterminée par un analyse cinétique de LAL. Les résultats ont indiqué que le lavage à l'isopropanol a diminué les niveaux d'endotoxine s'est élevé à 0,05 eu/g à 0,010 UE/g de protéines SHB-SAPN (tableau1).
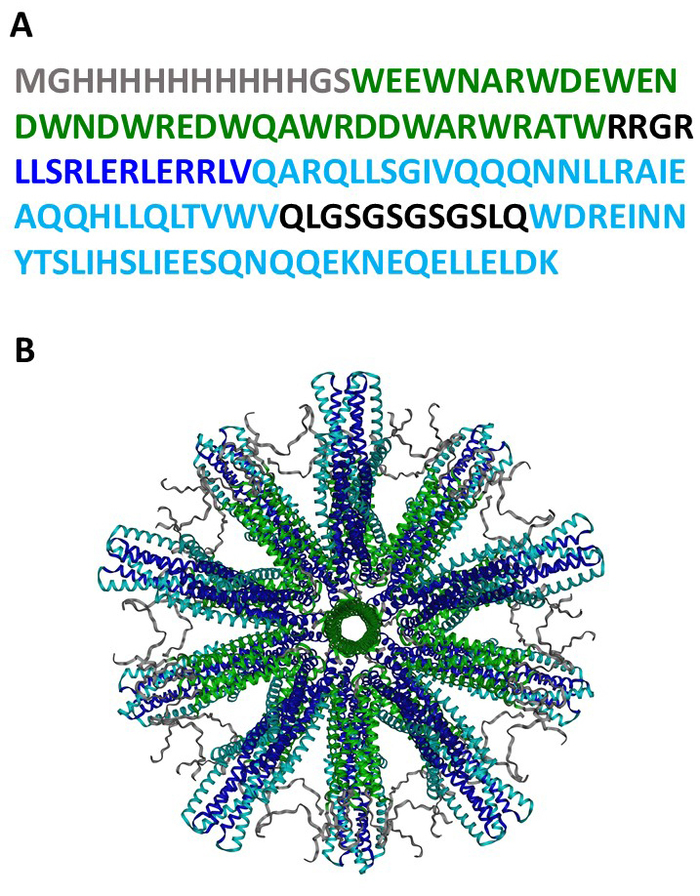
Figure 1 : séquence et structure des protéines SHB-SAPN. (A) La séquence d'acides aminés du monomère SHB-SAPN. (B) Modèle informatique de la structure du noyau SHB-SAPN entièrement assemblé composé de 60 monomères protéiques. Schéma de couleurs pour les séquences d'acides aminés: Gris - HisTag; Vert et pentamer; Trimer conçu en bleu foncé et de novo; Bleu clair et paquet de six hélices. Veuillez cliquer ici pour voir une version plus grande de ce chiffre.
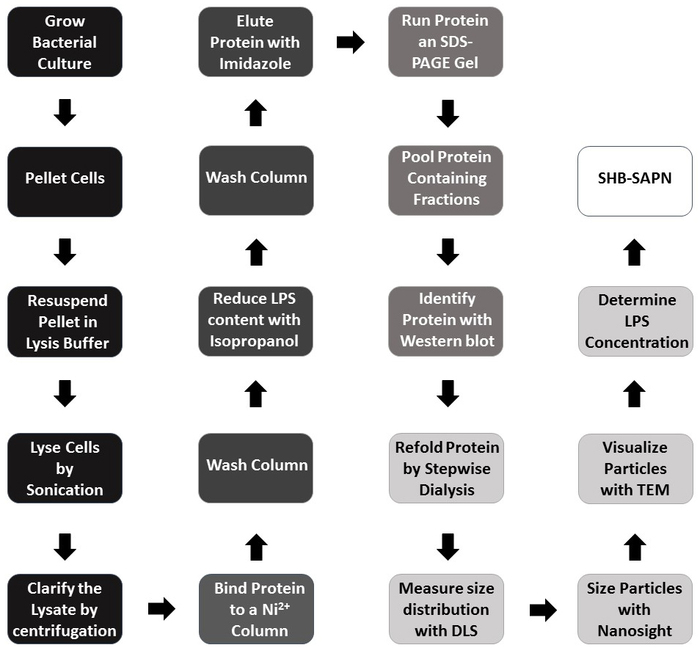
Figure 2 : Diagramme de flux de la protocole pour la production sHB-SAPN. Schéma de couleurs : Noir et expression du monomère protéique chez E. coli; Gris foncé et purification monomère; Identification gris moyen et monomère; Gris clair et repliement et caractérisation; Produit SHB-SAPN entièrement assemblé. Dans les étapes étiquetées avec gris foncé et moyen, la protéine est sous dénaturation et réduire les conditions. Veuillez cliquer ici pour voir une version plus grande de ce chiffre.
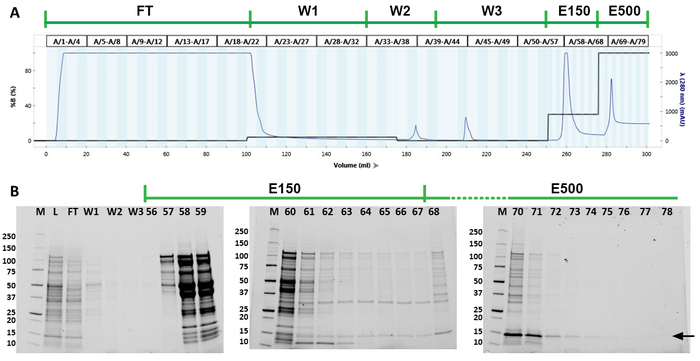
Figure 3 : Purification protéique des monomères SHB-SAPN. (A) Chromatographe de la purification FPLC. La ligne verte au-dessus du chromatogramme indique les étapes de purification. La ligne bleue dans le chromatogramme représente la densité optique des fractions à 280 nm longueur d'onde. La ligne noire montre quel pourcentage de tampon B (8 M urée, 20 mM Tris, 50 mM de phosphate de sodium monobasic, 5 mM TCEP, 500 mM Imidazole, pH 8,5) qui a été utilisé à chaque étape de la purification. (B) Gels SDS-PAGE des fractions mises en commun du lysate (L), écoulement à travers (FT), premier lavage (W1), lavage d'isopropanol (W2), troisième lavage (W3), et fractions individuelles de l'imidazole 150 mM (E150) et 500 mM imidazole (E500) étapes d'élution de l'ilution 150 mM (E150) et 500 mM imidazole (E500) étapes d'élution de l'ilution 150 mM (E150) et 500 mM imidazole (E500) étapes d'élution de l'ilution 150 mM (E150) et 500 mM imidazole (E500) étapes d'élution de la purification. Les marqueurs moléculaires de la première voie (M) identifient les bandes entre 10 et 250 kDa. La protéine cible est indiquée par une flèche noire. Veuillez cliquer ici pour voir une version plus grande de ce chiffre.
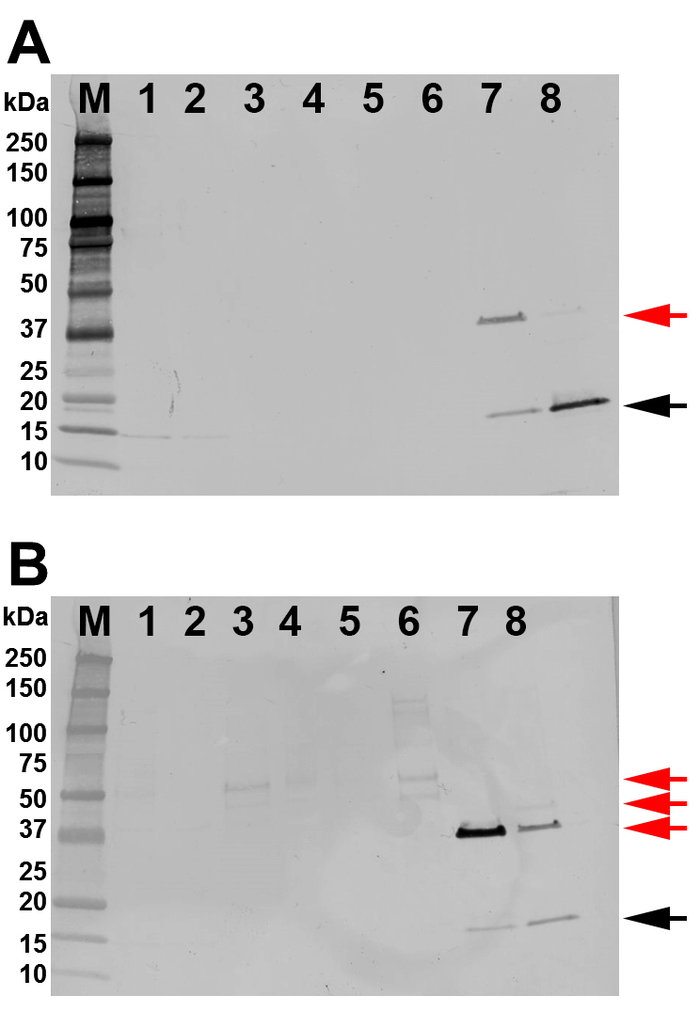
Figure 4 : Identification de la protéine par tache occidentale. Toutes les voies sont chargées de 100 ng de protéines. (A) Résultats d'une tache occidentale avec anti-6x HisTag. (B) Résultats d'une tache occidentale avec un anticorps monoclonal 167-D-IV HIV-1. Les voies sont étiquetées comme : M et marqueur de poids moléculaire; 1 lysate; 2 - Flux à travers; 3 - premier lavage; 4 - lavage à l'isopropanol (deuxième lavage); 5 - troisième lavage; 6 - fractions de volume en commun 56-u201261 (premier pic d'élution), 7 - factions de volume en commun 62-u201267 (entre les deux pics), 8 fractions de volume en commun 68-u201278 (deuxième pic d'élution). Protéine cible avec la taille prévue de la bande de 18,07 kDa comme la bande monomeric SHB-SAPN est indiquée par une flèche noire. Les bandes supplémentaires dans les voies 7 et 8 sont des douilles, des trimers et des multimères du SHB-SAPN (flèche rouge). Veuillez cliquer ici pour voir une version plus grande de ce chiffre.
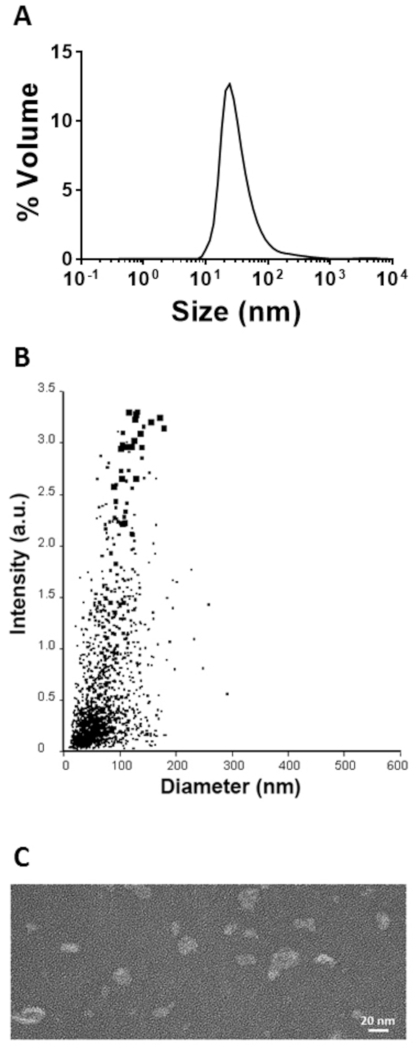
Figure 5 : Caractérisation du SHB-SAPN replié. (A. Distribution de la taille des particules telle que déterminée par DLS. (B) Taille des particules déterminée par le suivi des nanoparticules (système). (C) Visualisation des particules SHB-SAPN par TEM. Veuillez cliquer ici pour voir une version plus grande de ce chiffre.
| échantillon | Endotoxine (UE/mL) | Endotoxine (UE/g de protéines) |
| SAPN avec lavage d'isopropanol | 2.02 | 0.01 |
| SAPN sans lavage isopropanol | À la 50e | 0,25 euros |
| Contrôle négatif | En dessous du niveau de détection | ne s'applique pas |
Tableau 1 : Niveaux d'endotoxine des SHB-SAPN repliés. Les niveaux d'endotoxine dans les échantillons de SHB-SAPN purifiés avec ou sans lavage d'isopropanol présentés comme unités d'endotoxine/mL et unités d'endotoxine/g de protéine DeHB-SAPN.
Discussion
La nanotechnologie offre de nombreux avantages et solutions pour le développement de vaccins sous-unis. Les nanovaccins peuvent présenter à plusieurs reprises des antigènes comme particules au système immunitaire hôte augmentant l'immunogénicité26. Bien qu'il existe de nombreux types différents de nanovaccins, nous croyons que ceux composés de protéines de novo conçu semblent être l'approche la plus forte pour le développement de vaccins1. Ils peuvent être conçus sans aucune homologie de séquence aux protéines hôtes et présenter l'antigène d'intérêt dans près de conformation indigène-comme tout en fournissant le coût de production bas et les rendements élevés de produit. Un excellent exemple de cette approche est la technologie SAPN, que nous avons appliquée aux vaccins contre de multiples maladies infectieuses7. En abordant les difficultés dans le développement du vaccin CONTRE le VIH-1, nous avons conçu un noyau SHB-SAPN unique pour présenter efficacement l'antigène V1V2 dans une conformation trimérique indigène9. De nombreuses cibles vaccinales, en particulier pour les maladies virales, sont présentes en tant que trimestres27. Ce phénomène indique que notre conception SAPN a de vastes implications pour le développement de vaccins sous-unitaires.
Dans cette méthode, nous démontrons comment produire des SHB-SAPNs dans un système d'expression E. coli. Nous avons exprimé des rendements élevés de protéines (environ 6 mg/100 ml de culture). La protéine contenait 10 histidines et a été facilement purifiée à l'aide d'une chromatographie d'affinité métallique immobilisée avec la colonne Ni2. Cette longueur du His-Tag s'est avérée optimale pour le rendement protéique le plus élevé. La protéine purifiée contenait la pleine longueur de la protéine conçue comme indiqué par la présence du HisTag N-terminal et de la répétition de l'heptade terminal C. Nous avons utilisé des techniques largement acceptées et les avons optimisées pour l'expression, la production et la caractérisation du noyau SHB-SAPN. L'absence de la production de la protéine pleine longueur pendant le développement d'un SAPN contenant un nouvel épitope de protéine pourrait indiquer un problème d'expression du gène dans la cellule hôte. Si cela se produit, le gène et le système d'expression doivent être repensés et adaptés au protocole décrit. La modification du temps ou de l'intensité de la sonication peut également augmenter la concentration de la protéine pleine longueur prévue.
Les particules repliées se trouvaient dans la plage de taille prévue (20 à 100 nm)8,24,25 selon l'analyse du DLS et du suivi des nanoparticules. Ces résultats ont été confirmés par l'utilisation de TEM. S'il y a des problèmes dans cette étape, c'est normalement dû à un problème avec le pH ou la force ionique du tampon repliant. Lorsque des particules de grande taille sont détectées sur les techniques de dimensionnement des particules, il indique l'agrégation, qui peut être évitée en augmentant le pH du tampon de repliage. Si les particules ne sont pas détectées par DLS, vérifiez la concentration de la protéine et vérifiez le pH du tampon. La concentration finale de protéines pour le DLS devrait être d'au moins 100 g/mL. Si la concentration n'est pas le problème, elle indique l'abondance de petites particules incomplètement formées, dont la concentration peut être réduite en diminuant le pH. Alternativement, la concentration de chlorure de sodium peut être ajustée à la plage optimale pour minimiser la présence de particules de taille non désirée.
Enfin, en utilisant une étape de lavage de l'isopropanol pendant la purification, nous avons pu réduire la contamination du LPS de l'hôte E. coli à 0,01 UE / G de SAPN qui est en dessous de la limite de la Food and Drug Administration (FDA) de 5 UE / kg de poids corporel pour les produits injectables 28. Ce niveau peut être encore réduit en utilisant une colonne d'échange d'anion également connue sous le nom de colonne Q. Si des niveaux élevés d'endotoxine sont encore présents, vérifiez tous les matériaux qui ont été utilisés pour la préparation tampon. N'oubliez pas d'utiliser uniquement de la verrerie depyrogenated et de la plasticware sans endotoxine dans cette méthode.
Ces résultats indiquent que nous avons mis au point avec succès une méthode pour produire le noyau SHB-SAPN qui peut être utilisé pour des études de vaccination préclinique. Cette méthode avec seulement de légères modifications, le cas échéant, peut être appliquée à la purification des SHB-SAPNs lorsqu'un antigène d'intérêt est ajouté. L'utilisation de cette méthode comme point de départ l'un des changements majeurs est dans l'étape d'élution. Différentes protéines s'élipent à différentes concentrations d'imidazoles qui doivent être déterminées expérimentalement. L'autre différence majeure pourrait être la composition du tampon repliant. L'optimisation nécessiterait de tester différentes conditions de pH ainsi que des forces ioniques.
En compte tenu des travaux futurs, seules deux légères modifications sont nécessaires pour permettre l'application humaine du SHB-SAPN. La première est que le vecteur d'expression doit être changé en un marqueur sélectionnable de résistance à la kanamycine en raison de l'allergie à l'ampicilline chez l'homme29. L'autre exigence majeure de la fabrication de protéines à usage humain est de produire le SHB-SAPN dans les médias sans produits animaux. Une étude à petite échelle a déjà indiqué un rendement raisonnable de protéines dans un média à base de plantes. Les travaux présentés ici sont facilement évolutifs pour la production finale de GMP comme l'a démontré un candidat vaccin contre le paludisme, FMP01416. Cette production FMP014-SAPN à grande échelle comprenait à la fois l'échange d'anion et les étapes d'échange de cation pour réduire davantage le contenu LPS et Ni2 du produit final. Ce SAPN à exprime bactérienne a déjà été mis à l'échelle pour un prochain essai clinique de phase 1/2a.
Déclarations de divulgation
Les opinions exprimées sont celles des auteurs et ne doivent pas être interprétées comme représentant les positions de l'armée américaine ou du ministère de la Défense. Peter Burkhard a un intérêt dans la société appelée Alpha-O Peptides AG et a des brevets sur la technologie. Les autres auteurs n'ont aucune affiliation ou implication financière avec une société ayant un intérêt financier sur le sujet présenté dans le présent document.
Remerciements
Ce travail a été soutenu par un accord de coopération (W81XWH-11-2-0174) entre la Fondation Henry M Jackson pour l'avancement de la médecine militaire, Inc., et le ministère de la Défense des États-Unis. L'anticorps anti-VIH-1 gp41 mAb 167-D IV a été reçu de la Dre Susan Zolla-Pazner dans le cadre du Programme de réactifs du sida des NIH.
matériels
| Name | Company | Catalog Number | Comments |
| 10x Tris/Glycine/SDS | BioRad | 1610732 | 1 L |
| 2-Mercaptoethanol | BioRad | 1610710 | 25 mL |
| 2-propanol | Fisher | BP26181 | 4 L |
| 2x Laemmli Sample Buffer | BioRad | 1610737 | 30 mL |
| 40ul Cuvette Pack of 100 with Stoppers | Malvern Panalytical | ZEN0040 | 100 pack |
| 4–20% Mini-PROTEAN TGX Precast Protein Gels, 10-well, 30 µl | BioRad | 4561093 | 10 pack |
| Ampicillin | Fisher | BP1760-25 | 25 g |
| Anti-6X His tag antibody [HIS.H8] | AbCam | ab18184 | 100 mg |
| Anti-HIV-1 gp41 Monoclonal (167-D IV) | AIDS Reagent Repository | 11681 | 100 mg |
| BCIP/NBT Substrate, Solution | Southern Biotech | 0302-01 | 100 mL |
| Corning Disposable Vacuum Filter/Storage Systems | Fisher | 09-761-108 | A variety of sizes |
| Formvar/Carbon 400 mesh, Copper approx. grid hole size: 42µm | Ted Pella, Inc | 01754-F | 25 pack |
| GE Healthcare 5 mL HisTrap HP Prepacked Columns | GE HealthCare | 45-000-325 | 5 pack |
| Glycerol | Fisher | BP229-4 | 4 L |
| Goat Anti-Mouse IgG H&L (Alkaline Phosphatase) | ABCam | ab97020 | 1 mg |
| Imidazole | Fisher | O3196-500 | 500 g |
| Instant NonFat Dry Milk | Quality Biological | A614-1003 | 10 pack |
| Kinetic-QCL Kinetic Chormogenic LAL Assay | Lonza Walkersville | 50650U | 192 Test Kit |
| LAL Reagent Grade Multi-well Plates | Lonza Walkersville | 25-340 | 1 plate |
| Magic Media E. coli Expression Medium | ThermoFisher | K6803 | 1 L |
| MilliporeSigma Millex Sterile Syringe 0.22 mm Filters | Millipore | SLGV033RB | 250 pack |
| Mouse Anti-Human IgG Fc-AP | Southern Biotech | 9040-04 | 1.0 mL |
| One Shot BL21 Star (DE3) Chemically Competent E. coli | ThermoFisher | C601003 | 20 vials |
| Precision Plus Protein Unstained Protein Standards, Strep-tagged recombinant, | BioRad | 1610363 | 1 mL |
| Slide-A-Lyzer Dialysis Cassettes, 10K MWCO, 12 mL | ThermoFisher | 66810 | 8 pack |
| Sodium Chloride | Fisher | BP358-212 | 2.5 kg |
| Sodium Phosphate Monobasic | Fisher | BP329-500 | 500 g |
| Tris Base | Fisher | BP152-1 | 1 kg |
| Tris-(2-carboxyethyl)phosphine hydrochloride | Biosynth International | C-1818 | 100 g |
| Uranyl Acetate, Reagent, A.C.S | Electron Micoscopy Services | 541-09-3 | 25 g |
| Urea | Fisher | BP169-500 | 2.5 kg |
| Whatman qualitative filter paper | Sigma Aldrich | WHA10010155 | pack of 500 |
| Name | Company | Catalog Number | Comments |
| Equipment | |||
| ChromLab Software ver 4 | BioRad | 12009390 | Software |
| Epoch 2 Microplate Spectrophotometer | BioTek | EPOCH2 | Plate Reader |
| Fiberlite F14-14 x 50cy Fixed-Angle Rotor | ThermoFisher | 096-145075 | Rotor |
| Gel Doc EZ Gel Documentation System | BioRad | 1708270 | Gel Imager for Stain free Gels |
| JEOL TEM | JEOL | 1400 | Transmission Electron Microscope |
| Mini-PROTEAN Tetra Vertical Electrophoresis Cell for Mini Precast Gels | BioRad | 1658004 | To run gels |
| NanoDrop One Microvolume UV-Vis Spectrophotometer | ThermoFisher | ND-ONE-W | For Protein Concentration |
| NanoSight NS300 | Malvern Panalytical | Particle Sizing | |
| NanoSight NTA software NTA | Malvern Panalytical | Particle Sizing | |
| New Brunswick Innova 44/44R | Eppendorf | M1282-0000 | Incubator/Shaker |
| NGC Quest 10 Chromatography System | BioRad | 7880001 | FPLC to aid in protein purification |
| PELCO easiGlow Glow Discharge Cleaning System | Ted Pella, INC | 91000S | To clean grids |
| PowerPac Universal Power Supply | BioRad | 1645070 | To run gels |
| Rocker Shaker | Daigger | EF5536A | For Western |
| Sonifer 450 | Branson | also known as 096-145075 | Sonicator |
| Thermo Scientific Sorvall LYNX 4000 Superspeed Centrifuge | ThermoFisher | 75-006-580 | Centrifuge |
| Trans-Blot Turbo Mini Nitrocellulose Transfer Packs | BioRad | 1704158 | For Western |
| Trans-Blot Turbo Transfer System | BioRad | 1704150 | For Western |
| Vortex-Genie 2 | Daigger | EF3030A | Vortex |
| Zetasizer Nano ZS | Malvern Panalytical | Particle Sizing | |
| Zetasizer Software | Malvern Panalytical | Particle Sizing |
Références
- Karch, C. P., Burkhard, P. Vaccine technologies: From whole organisms to rationally designed protein assemblies. Biochemical Pharmacology. 120, 1-14 (2016).
- Snapper, C. M. Distinct Immunologic Properties of Soluble Versus Particulate Antigens. Frontiers in Immunology. 9, 598(2018).
- Kelly, H. G., Kent, S. J., Wheatley, A. K. Immunological basis for enhanced immunity of nanoparticle vaccines. Expert Review of Vaccines. , 1-12 (2019).
- Yeates, T. O. Geometric Principles for Designing Highly Symmetric Self-Assembling Protein Nanomaterials. Annual Review of Biophysics. 46, 23-42 (2017).
- Marcandalli, J., et al. Induction of Potent Neutralizing Antibody Responses by a Designed Protein Nanoparticle Vaccine for Respiratory Syncytial Virus. Cell. 176 (6), 1420-1431 (2019).
- Ross, J. F., et al. Decorating Self-Assembled Peptide Cages with Proteins. ACS Nano. 11 (8), 7901-7914 (2017).
- Karch, C. P., Matyas, G. R., Burkhard, P., Beck, Z. Self-Assembling Protein Nanoparticles: implications for HIV-1 vaccine development. Nanomedicine (Lond). 13 (17), 2121-2125 (2018).
- Raman, S., Machaidze, G., Lustig, A., Aebi, U., Burkhard, P. Structure-based design of peptides that self-assemble into regular polyhedral nanoparticles). Nanomedicine. 2 (2), 95-102 (2006).
- Karch, C. P., et al. Design and characterization of a self-assembling protein nanoparticle displaying HIV-1 Env V1V2 loop in a native-like trimeric conformation as vaccine antigen. Nanomedicine. , (2018).
- Karch, C. P., et al. The use of a P. falciparum specific coiled-coil domain to construct a self-assembling protein nanoparticle vaccine to prevent malaria. Journal of Nanobiotechnology. 15 (1), 62(2017).
- Li, J., et al. A self-adjuvanted nanoparticle based vaccine against infectious bronchitis virus. PLoS One. 13 (9), e0203771(2018).
- Wahome, N., et al. Conformation-specific display of 4E10 and 2F5 epitopes on self-assembling protein nanoparticles as a potential HIV vaccine. Chemical Biology & Drug Design. 80 (3), 349-357 (2012).
- El Bissati, K., et al. Effectiveness of a novel immunogenic nanoparticle platform for Toxoplasma peptide vaccine in HLA transgenic mice. Vaccine. 32 (26), 3243-3248 (2014).
- Kaba, S. A., et al. A nonadjuvanted polypeptide nanoparticle vaccine confers long-lasting protection against rodent malaria. Journal of Immunology. 183 (11), 7268-7277 (2009).
- Kaba, S. A., et al. Protective antibody and CD8+ T-cell responses to the Plasmodium falciparum circumsporozoite protein induced by a nanoparticle vaccine. PLoS One. 7 (10), e48304(2012).
- Seth, L., et al. Development of a self-assembling protein nanoparticle vaccine targeting Plasmodium falciparum Circumsporozoite Protein delivered in three Army Liposome Formulation adjuvants. Vaccine. 35 (41), 5448-5454 (2017).
- Kaba, S. A., et al. Self-assembling protein nanoparticles with built-in flagellin domains increases protective efficacy of a Plasmodium falciparum based vaccine. Vaccine. 36 (6), 906-914 (2018).
- El Bissati, K., et al. Protein nanovaccine confers robust immunity against Toxoplasma. NPJ Vaccines. 2, 24(2017).
- Karch, C. P., et al. Vaccination with self-adjuvanted protein nanoparticles provides protection against lethal influenza challenge. Nanomedicine. 13 (1), 241-251 (2017).
- Haynes, B. F., et al. Immune-correlates analysis of an HIV-1 vaccine efficacy trial. New England Journal of Medicine. 366 (14), 1275-1286 (2012).
- Rerks-Ngarm, S., et al. Vaccination with ALVAC and AIDSVAX to prevent HIV-1 infection in Thailand. New England Journal of Medicine. 361 (23), 2209-2220 (2009).
- O' Connell, R. J., Kim, J. H., Excler, J. L. The HIV-1 gp120 V1V2 loop: structure, function and importance for vaccine development. Expert Review of Vaccines. 13 (12), 1489-1500 (2014).
- Babapoor, S., et al. A Novel Vaccine Using Nanoparticle Platform to Present Immunogenic M2e against Avian Influenza Infection. Influenza Research and Treatment. 2011, 126794(2011).
- Indelicato, G., Burkhard, P., Twarock, R. Classification of self-assembling protein nanoparticle architectures for applications in vaccine design. Royal Society Open Science. 4 (4), 161092(2017).
- Indelicato, G., et al. Principles Governing the Self-Assembly of Coiled-Coil Protein Nanoparticles. Biophysical Journal. 110 (3), 646-660 (2016).
- Doll, T. A., Raman, S., Dey, R., Burkhard, P. Nanoscale assemblies and their biomedical applications. Journal of the Royal Society Interface. 10 (80), 20120740(2013).
- Rey, F. A., Lok, S. M. Common Features of Enveloped Viruses and Implications for Immunogen Design for Next-Generation Vaccines. Cell. 172 (6), 1319-1334 (2018).
- Bacterial Endotoxins. United States Pharmacopeia (USP). , Ch. 85 (2011).
- Points to Consider (PTC) in the Characterization of Cell Lines Used to Produce Biologicals. , Food and Drug Adminstration. Maryland. Available from: https://www.fda.gov/media/76255/download (1993).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon