Method Article
Imágenes 3D de muestras de tejido blando utilizando un método de tinción específica de rayos X y tomografía computarizada nanoscópica
* Estos autores han contribuido por igual
En este artículo
Resumen
Se presenta un protocolo para la visualización 3D de estructuras microscópicas de tejido mediante el uso de un método de tinción específico de rayos X diseñado para la tomografía computarizada de rayos X.
Resumen
Demostramos un método basado en laboratorio que combina microCT de rayos X y nanoCT con una mancha de rayos X específica, que se dirige al citoplasma celular. El protocolo descrito es fácil de aplicar, rápido y adecuado para muestras de tejidos blandos más grandes. La metodología presentada permite la caracterización de estructuras tisulares cruciales en tres dimensiones y se demuestra en un riñón de ratón entero. El enfoque multiescala permite tomar imágenes de todo el riñón del ratón y soporta la selección de nuevos volúmenes de interés, que se adquieren con resoluciones más altas que van en el rango de nanómetros. De este modo, se reproduce la morfología de tejido blando con un nivel de detalle similar al de las imágenes de microscopía de luz histológica correspondientes. Se obtienen conocimientos más profundos sobre la configuración 3D de las estructuras tisulares sin impedir más investigaciones a través de métodos histológicos.
Introducción
La caracterización completa de muestras de tejido blando requiere información sobre la microestructura del tejido 3D. El estándar de oro actual para los análisis de muestras de tejidos blandos es la histopatología. El tejido y la morfología celular de la muestra se exploran en 2D dentro de regiones seleccionadas de interés (ROI) utilizando microscopía óptica1. Este método, sin embargo, tiene varios inconvenientes. La preparación de la muestra requiere mucho tiempo, es complicada, destructiva y propensa a los artefactos. Las diapositivas microscópicas producidas proporcionan sólo información 2D paralela al plano de sección. A menudo el número de secciones histológicas, que se investigan, está restringido debido a las limitaciones de tiempo2,3.
En los últimos años, el campo de la histología 3D ha evolucionado. Aquí, las rebanadas de tejido virtual de cualquier plano espacial deseado son accesibles. Esto permite el seguimiento de las estructuras a lo largo de la muestra, lo que conduce a una comprensión más profunda de la arquitectura del tejido 3D y los cambios estructurales asociados con diferentes patologías. Se han desarrollado varios métodos para lograr la generación de datos de volumen 3D. Van desde enfoques basados en secciones seriales, que utilizan microscopía de luz o electrónica4,5,6,7,8, hasta métodos de imagen de cara de bloque, como imágenes 3D episcópicas o microscopía electrónica de barrido de cara de bloque7,8,9. Sin embargo, todos los métodos mencionados implican la sección o la destrucción completa de la muestra, lo que no permite más investigaciones. La resolución obtenida depende en gran medida de que el proceso de seccionamiento sea propenso a los artefactos como se describe en la histología convencional. Estos métodos también sufren de artefactos de alineación.
Las técnicas de imágenes de rayos X 3D, como la tomografía computarizada microscópica y nanoscópica (microCT y nanoCT) aspiran a generar datos 3D de alta resolución sin destruir la muestra de tejido. Hasta ahora, el débil contraste de atenuación de rayos X de los tejidos blandos y el acceso limitado a altas resoluciones en un entorno de laboratorio han deteriorado su uso para la visualización 3D de estructuras microscópicas de tejidos. Los recientes avances hacia una TC de rayos X de alta resolución basada en laboratorio permiten resoluciones muy por debajo de 1 m10,11,12,13.
La falta de contraste en el tejido blando en las imágenes de rayos X convencionales basadas en la atenuación se compensa con agentes de tinción, que mejoran el contraste de atenuación de rayos X. Los agentes de tinción conocidos a partir de otras técnicas de diagnóstico por imágenes como el tetróxido de osmio (OsO4),el yoduro de potasio (IKI) o el ácido fosfotúgótico (PTA) se utilizan a menudo14,15,16,17, 18,19,20,21,22,23,24,25. Agentes de tinción que permiten (i) focalización biológica específica, (ii) tinción homogénea y completa, (iii) fácil manejo, (iv) penetración rápida del tejido sin crear artefactos como anillos de difusión, (v) tinción de tejido grande y denso, y (vi) se requiere compatibilidad total con la histopatología para establecer la TC de rayos X como herramienta para la visualización 3D de estructuras microscópicas de tejido. En este trabajo, mostramos cómo se preparan muestras de tejido blando para la toma de imágenes por TC de rayos X con una mancha de rayos X específica del citoplasma basada en eosina que cumple con los requisitos indicados anteriormente26.
El enfoque de imágenes multiescala garantiza la evaluación de la calidad de las tinciones a través de una medición general de microCT y la selección de volúmenes de interés (VOI) para más investigaciones de alta resolución. La calidad de la tinción se analiza centrándose en parámetros de tinción como (i) integridad, (ii) apariencia de anillos de difusión, (iii) mejora del contraste, (iv) apariencia de artefactos de TC como rayas y (v) homogeneidad. La configuración de nanoCT basada en laboratorio, que utiliza aumento geométrico para alcanzar resoluciones de hasta 100 nm, visualiza la morfología de los tejidos blandos en (sub)-nivel celular10,27. Un análisis comparativo de las rodajas de nanoCT con las correspondientes imágenes histológicas de microscopía de luz confirma la reproducción de la arquitectura tisular con detalles similares a nivel microscópico en 2D, permitiendo la caracterización histopatológica del tejido Muestra. Este protocolo de vídeo detallado tiene como objetivo sensibilizar y destacar el potencial de esta metodología, ya que la herramienta de imagen de tejidos blandos 3D no destructiva es de interés para una amplia comunidad científica como zoólogos, biólogos y salud Profesionales.
Protocolo
Precaución: Consulte todas las fichas de datos de seguridad de materiales (MSDS) relevantes antes de su uso. Varios de los productos químicos utilizados en el protocolo son agudamente tóxicos y cancerígenos. Utilice todas las prácticas de seguridad apropiadas al realizar el protocolo de tinción, incluido el uso de controles de ingeniería (capucha de humo, guantera) y equipo de protección personal (gafas de seguridad, guantes, abrigo de laboratorio, pantalones de longitud completa, zapatos cerrados de punta).
Animales utilizados:
La vivienda de animales se llevó a cabo en el Klinikum rechts der Isar, Universidad Técnica de Múnich de conformidad con las directrices de la Unión Europea 2010/63. La remoción de órganos fue aprobada de un comité interno de protección animal de Klinikum rechts der Isar, Munich, Alemania (número de referencia interno 4-005-09). Todos los procedimientos se ajustaban a las directrices y reglamentos pertinentes. Todos los laboratorios son inspeccionados para comprobar si están de acuerdo con los principios de buenas prácticas de laboratorio de la OCDE.
1. Protocolo de tinción de eosina
- Para fijar muestras de tejido blando, llene un tubo centrífugo de 50 ml con una solución fijativa que contenga 9,5 ml de solución de formaldehído (FA) al 4% (v/v) y 0,5 ml de ácido acético glacial (AA).
NOTA: Prepare la solución FA recién a partir de una solución FA libre de ácido del 37% estabilizada con aproximadamente un 10% de metanol. Diluir aún más la solución de FA con la solución salina tamponada de fosfato de Dulbecco (DPBS). Elija DPBS sin calcio y magnesio. Conservar la solución de FA diluida no más de un mes. Durante la acidificación, el pH de la solución fijativa está cambiando de neutro a aproximadamente 3.
ADVERTENCIA: Debido a que la FA es aguda tóxica para órganos, corrosiva y cancerígena, el uso de una campana de humo es obligatorio y se debe utilizar el equipo personal de protección adecuado.- Agregue la muestra de tejido blando recién removida a un tubo centrífugo de 50 ml y refrigere el tubo centrífugo de 50 ml durante 24-72h.
NOTA: El protocolo se puede pausar aquí. - Lave la muestra de tejido blando con solución DPBS durante 1h.
- Agregue la muestra de tejido blando recién removida a un tubo centrífugo de 50 ml y refrigere el tubo centrífugo de 50 ml durante 24-72h.
- Para manchar la muestra de tejido blando fijada (por ejemplo, un riñón de ratón entero), coloque el tejido blando en 2 ml de solución de tinción Y de eosina e incubar la muestra durante 24 h. Mantenga la muestra en una placa de agitación horizontal para un balanceo suave (ca. 60 rpm) durante la incubación Proceso.
NOTA: La solución de tinción Y de eosina tiene una concentración del 30% (p/v) en agua destilada. Elija el volumen de la solución de tinción de manera que la muestra esté completamente cubierta por la solución de tinción y permita que la muestra se mueva libremente dentro del contenedor de la muestra. El tiempo de incubación puede diferir para otras muestras y debe ajustarse en consecuencia. - Después de la tinción, retire la muestra de tejido blando cuidadosamente del recipiente de la muestra.
- Retire cuidadosamente el exceso de agente de tinción con papel tisú de celulosa.
- Coloque la muestra de tejido blando en un recipiente de muestra cónica por encima de una fase de vapor de etanol para su almacenamiento y uso posterior.
NOTA: El recipiente de muestra cónica siempre debe contener unas gotas de etanol del 70% (v/v) en la parte inferior del tubo para mantener húmeda la muestra de tejido blando y prevenir artefactos.
2. Imágenes microCT de rayos X
NOTA: Las mediciones de microCT de rayos X se realizaron con un escáner microCT, que ofrece mediciones de TC de visión general (la capacidad de crear imágenes de toda la muestra dentro del campo de visión (FOV)) y el rendimiento de las mediciones de TC de alta resolución (la capacidad de enfocarse en en un volumen de interés deseado (VOI) de la misma muestra) hasta 1 m.
- Monte la muestra de tejido blando en un soporte de muestra adecuado. Asegurar un ajuste ajustado de la muestra en el soporte de la muestra para evitar que la muestra se mueva durante las mediciones de TC de rayos X.
- En el caso del riñón de ratón teñido: Preparar un portamuestras con dos tubos de centrífuga, mediante el que se corta la parte inferior de un tubo. Pegue los dos tubos centrífugos juntos utilizando adhesivo de dos componentes. Asegurar una alineación recta de los tubos centrífugos alrededor del eje de rotación. Espere a que el adhesivo se endurezque.
- Una vez que el soporte de muestra esté listo para su uso, transfiera el riñón del ratón al tubo de centrífuga intacto, que contiene unas gotas de etanol del 70% (v/v) en la parte inferior del tubo.
NOTA: La estabilidad de la muestra es crucial. Tómese el tiempo para preparar la muestra para las mediciones de TC de rayos X. La muestra de tejido blando se mantiene durante una fase de vapor de etanol para mantener la muestra húmeda durante las mediciones de TC de rayos X y evitar que la muestra de tejido blando se contratraiga y otros artefactos. La muestra de tejido blando no debe estar en contacto con el disolvente para evitar la acumulación del disolvente alrededor de la muestra durante la medición de TC de rayos X, lo que podría provocar el movimiento de la muestra durante la medición o podría causar problemas durante la reconstrucción. Si el soporte de la muestra no permite sujetar el disolvente en la parte inferior, se puede colocar un papel de celulosa humedecido con etanol del 70% (v/v) en el soporte de la muestra. Cabe señalar que no se observaron artefactos de contracción debido al etanol solvente.
NOTA: El protocolo se puede pausar aquí.
- Después de una alineación cuidadosa de la muestra, elija los parámetros de adquisición para obtener la mejor calidad de imagen. En el caso de los datos microCT presentados, adquiera el escaneo a una tensión máxima de 50 kV, una corriente de 3,5 W utilizando 1601 proyecciones distribuidas equitativamente en 360o.
NOTA: Los parámetros de adquisición para la exploración por TC de visión general se eligieron para obtener la mejor calidad de imagen. Como tal, el objetivo de la cámara 0.39x fue elegido para cubrir toda la muestra dentro del campo de visión (FOV). Esto dio lugar a un tamaño de píxel efectivo de 12 m. El tiempo de exposición de 2 s por proyección proporcionó una buena relación señal-ruido. El ROI para el análisis CT de alta resolución se identificó utilizando los datos de microCT del análisis de visión general. Los escáneres MicroCT a menudo incorporan una herramienta de software integrada, que permite la selección precisa del ROI determinado. Para los datos CT de alta resolución, se eligió el objetivo de la cámara 4x, lo que resultó en un tamaño de píxel efectivo de 3,3 m. Aquí, se necesitaba un tiempo de exposición de 15 s por proyección.
NOTA: El protocolo se puede pausar aquí. - Después de la adquisición de los datos de TC de rayos X, procesar las proyecciones en consecuencia para la reconstrucción del volumen 3D. En el caso de los datos microCT presentados: Reconstruya los datos de TC de rayos X con el software integrado.
NOTA: Las representaciones de volumen de los datos de microCT que se muestran en la Figura 1 y la Figura 2 se generaron mediante un software de visualización.
NOTA: El protocolo se puede pausar aquí.
3. Imágenes nanoCT de rayos X
NOTA: El escáner nanoCT de rayos X se ha desarrollado internamente. El instrumento libre de lentes está equipado con una fuente de rayos X de nanofoco y un detector de recuento de un solo fotón. Se pueden generar datos 3D con resoluciones de hasta 100 nm10. Generalmente, los sistemas nanoCT, incluidos los que tienen óptica de rayos X, están disponibles comercialmente y no se limitan al escáner nanoCT descrito.
- Preparación de muestras NanoCT
- Prepare los VMI de la muestra de tejido blando. Cortar el tejido blando en trozos muy pequeños de aproximadamente 0,5 mm de longitud de borde usando un bisturí y un estereomicroscopio. En el caso del riñón del ratón: Corte el riñón del ratón en dos mitades a lo largo del eje más largo. Tome la mitad del riñón del ratón y prepare diferentes regiones anatómicas como la corteza renal y la médula renal.
NOTA: La otra mitad del riñón del ratón se transfirió a histopatología, donde la muestra se incrustó en parafina y se procesó en consecuencia para producir las secciones histológicas típicas como se ve en la Figura 3c y la Figura 3d . - Transfiera las piezas pequeñas antes del primer paso de deshidratación a un nuevo plato de Petri, donde permanecen para todos los pasos posteriores.
- Deshidratar las muestras utilizando concentraciones (todas v/v) en %: 50, 60, 70, 80, 90, 96 y 100 etanol equilibrados con agua destilada. Realice cada paso de deshidratación durante 1 h cada uno.
NOTA: El protocolo se puede pausar aquí. Mantenga las piezas de tejido pequeño en 100% de etanol durante la noche. - Punto crítico seco (CPD) las pequeñas piezas de tejido.
NOTA: La aplicación de CPD permite la deshidratación completa de la muestra de tejido mediante el intercambio del disolvente (aquí etanol) con el agente de secado (aquí CO2). Esto ha sido necesario para garantizar que la muestra se puede montar en los portamuestras del nanoCT, no se mueve durante la medición y se puede colocar muy cerca de la fuente de rayos X para permitir un mejor aumento geométrico. La configuración de nanoCT se basa en un mero aumento geométrico, definiéndose el factor de ampliación como la distancia de origen a detector sobre la distancia de origen a muestra. La técnica de secado fue introducida por primera vez por Anderson para preservar la estructura 3D de especímenes biológicos para la microscopía electrónica28. Bray29proporciona una visión general de la técnica.- Rellene previamente la cámara de vacío con 100% de etanol. Transfiera las piezas de tejido pequeño en una cápsula microporosa y colóquelas en la cámara de vacío del CPD.
NOTA: Dado que la alta presión está implicada en el proceso de CPD, asegúrese de que todas las partes del CPD, en particular los accesorios, estén intactas y que el sistema esté correctamente cerrado. - Enfríe la cámara a 6-8oC y llénelo conco.líquido 2 .
- Mientras se agita, espere 3 minutos para permitir la mezcla adecuada de los dos componentes. Escurra cuidadosamente la cámara. Asegúrese de que el soporte de la muestra aún esté cubierto con disolvente. Repita este paso diez veces para permitir la sustitución completa del etanol porCO2 dentro de la muestra.
- Después del llenado final de la cámara con CO2,calentar la máquina hasta el punto crítico de CO2 (31 oC y 73,8 bar) seguido de una liberación muy lenta del CO2 gaseoso durante un tiempo de 30 min.
NOTA: La liberación del gas debe llevarse a cabo muy lentamente, ya que de lo contrario se puede condensar agua en la muestra. Asegúrese de que la temperatura no caiga por debajo del punto crítico de CO2. Sólo abra la máquina CPD cuando se haya liberado toda la presión del sistema. - Retire las piezas de tejido CPD rápidamente de la máquina y guárdelas en una nueva placa Petri almacenada en un desecador antes de su uso posterior.
NOTA: El protocolo se puede pausar aquí.
- Rellene previamente la cámara de vacío con 100% de etanol. Transfiera las piezas de tejido pequeño en una cápsula microporosa y colóquelas en la cámara de vacío del CPD.
- Prepare los VMI de la muestra de tejido blando. Cortar el tejido blando en trozos muy pequeños de aproximadamente 0,5 mm de longitud de borde usando un bisturí y un estereomicroscopio. En el caso del riñón del ratón: Corte el riñón del ratón en dos mitades a lo largo del eje más largo. Tome la mitad del riñón del ratón y prepare diferentes regiones anatómicas como la corteza renal y la médula renal.
- Monte las piezas de tejido CPD en un soporte de muestra adecuado. Asegúrese de un ajuste ajustado de la muestra en el soporte de la muestra para evitar que la muestra se mueva durante las mediciones de TC. En el caso de las piezas de tejido renal del ratón CPD: Pegue las piezas de tejido con superpegamento a un soporte de muestra.
NOTA: Cualquier movimiento no deseado de la muestra durante las mediciones de TC puede causar problemas durante la reconstrucción del volumen, especialmente cuando se adquiere un conjunto de datos con tamaño de voxel nanométrico.
NOTA: El protocolo se puede pausar aquí. - Después de una alineación cuidadosa de la muestra, elija los parámetros de adquisición para obtener la mejor calidad de imagen. En el caso de los datos nanoCT presentados: Adquiera proyecciones a una tensión máxima de 60 kV con 1599 proyecciones distribuidas equitativamente en 360o y un tamaño de voxel de aproximadamente 400 nm.
NOTA: Una sola medición de TC adquirida a un tamaño de vóxel de 400 nm tiene un FOV de 75 m en la dirección del eje de rotación (vertical) y aproximadamente 560 m en la dirección perpendicular al eje de rotación (horizontal). Para investigar volúmenes más grandes, se puede lograr una extensión del FOV a lo largo del eje de rotación combinando múltiples escaneos en diferentes posiciones verticales. Además, se pueden realizar exploraciones por tomografía local para medir muestras con un diámetro de muestra más grande perpendicular al eje de rotación que el FOV de una tomografía computarizada global. Los datos nanoCT se adquirieron con un tiempo de exposición de 4 s por proyección. Como tal, el tiempo total de adquisición por conjunto de datos fue de aproximadamente 3,5 h.
NOTA: El protocolo se puede pausar aquí. - Después de la adquisición de los datos de TC, procesar las proyecciones en consecuencia para la reconstrucción del volumen 3D.
- En el caso de los datos nanoCT presentados, normalice las proyecciones adquiridas con imágenes de campo plano. Mejore la nitidez de las proyecciones utilizando un algoritmo de desconvolución Richardson-Lucy30,31. Utilice una función gaussiana simétrica rotacional con una desviación estándar de un píxel como kernel de desconvolución.
- Aplique el algoritmo de recuperación de fase de Paganin a las imágenes afiladas para aumentar el contraste de tejido blando. Establezca los parámetros del algoritmo para optimizar la calidad de imagen32. Reconstruya las proyecciones preprocesadas con un algoritmo de proyección posterior filtrada de última generación.
NOTA: La Figura 3 evalúa los datos de nanoCT obtenidos con las secciones histológicas correspondientes, que tenían aproximadamente 7 m de espesor. Por lo tanto, se generaron rebanadas de proyección de intensidad mínima de 18 sectores de nanoCT adyacentes con un espesor virtual de aproximadamente 7 m mediante el cálculo del valor mínimo para cada píxel en los sectores correspondientes. Se utilizó un software de visualización para representar el volumen de los datos nanoCT, que se muestra en la Figura 4.
Resultados
La Figura 1 muestra los sectores de TC y la representación de volúmenes de datos de microCT de baja resolución que resaltan la mejora del contraste después de la tinción. La Figura 2 muestra los sectores de TC y la representación por volumen de datos de microCT de alta resolución derivados de una tomografía local de todo el riñón del ratón. La Figura 3 muestra los sectores ct de los datos nanoCT en comparación con las secciones histológicas correspondientes. La Figura 4 muestra la representación por segmentos de TC y volumen de datos nanoCT que resaltan los detalles estructurales a nivel celular. La medición de microCT de baja resolución permite una visión general de todo el órgano y ayuda a identificar volúmenes de interés (VOI) para la medición de microCT de alta resolución. A través de este enfoque multiescala, se determina el VOI para el nanoCT. El nanoCT permite una visión muy detallada de la muestra de tejido blando a nivel celular. El estudio comparativo con la correspondiente sección histológica destaca la plena compatibilidad con la histopatología. Aquí, el enfoque de imágenes multimodales está confirmando los resultados obtenidos con ambas modalidades.
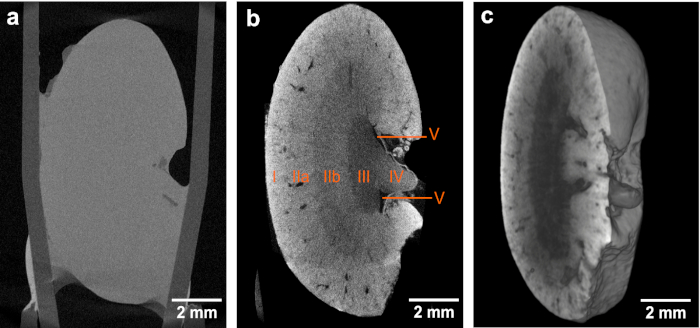
Figura 1. Sectores de TC y procesamiento de volumen de los datos de microCT de baja resolución. (a,b) Imágenes generales del mismo riñón del ratón antes y después de la tinción, respectivamente, destacando la mejora del contraste obtenida después de la aplicación del protocolo de tinción basado en eosina. Ambos conjuntos de datos microCT se adquirieron utilizando parámetros de adquisición idénticos. El tamaño de voxel en ambos conjuntos de datos es de 12 m. La mejora del contraste lograda en (b) permite la identificación de las siguientes regiones estructurales anatómicas: Cortex (I), médula exterior (II) con mayor distinción en las rayas externas de la médula exterior (IIa) y las rayas internas de la médula exterior (Ib), médula interna (III), papila (IV) y pelvis renal (V). (c) Representación por volumen de datos de microCT que muestran una sección sagital virtual a través de todo el riñón del ratón. Esta cifra ha sido modificada de Busse y M'ller et al.26Por favor, haga clic aquí para ver una versión más grande de esta cifra.

Figura 2. Procesamiento por tomografía computarizada y volumen de datos de microCT de alta resolución derivados del mismo riñón de ratón después de la aplicación del protocolo de tinción basado en eosina desarrollado. (a) La esquina izquierda muestra la imagen microCT de vista general resaltando el ROI (cuadro azul) de la imagen de alta resolución mostrada. Se pueden identificar las siguientes regiones estructurales anatómicas: Corteza (I), médula externa (II) con mayor distinción en las rayas externas de la médula externa (IIa) y las rayas internas de la médula externa (IIb), la médula interna (III), el cáliz menor (IV) y los vasos (V y VI). (b) Volumen de interés de los datos microCT de alta resolución adquiridos con un tamaño de voxel de 3,3 m. Se muestra la región de la médula y una sección virtual a través de un vaso derivado de una tomografía local de todo el riñón. Esta cifra ha sido modificada de Busse y M'ller et al.26. Haga clic aquí para ver una versión más grande de esta figura.

Figura 3. Rebanadas de TC de datos nanoCT (a,b) en comparación con las secciones histológicas (c,d) derivadas del mismo riñón de ratón después de la aplicación del protocolo de tinción basado en eosina desarrollado. (a) La imagen nanoCT de la misma muestra de riñón de ratón después de la tinción, disección y CPD muestra estructuras detalladas de la región (IIb) que se ven en la Figura 1 y la Figura 2. Estas se conocen como extremidades ascendentes gruesas del bucle de Henle. (b) Rebanada de proyección de intensidad mínima derivada del mismo conjunto de datos nanoCT que se muestra en(a ) con un espesor de corte virtual de aproximadamente 7 m, lo que permite una visualización clara de los núcleos celulares. (c) Sección histológica representativa que muestra las extremidades ascendentes gruesas del bucle de Henle con visualización clara de los núcleos celulares y el borde del pincel. La sección histológica tiene un espesor aproximado de 7 m y se obtuvo de la misma muestra renal de ratón después de la tinción e incrustación aplicada a la eosina en un bloque de parafina. (d) Sección histológica representativa con aplicación de hematoxilina de tinción de contador que resalte los núcleos celulares en púrpura. Preparación de la sección histológica cercana a la sección que se muestra en (c) con espesor aproximado de 7 m. Esta cifra ha sido modificada de Busse y M'ller et al.26Por favor, haga clic aquí para ver una versión más grande de esta cifra.

Figura 4. Procesamiento por tomografía computarizada y volumen de datos nanoCT. (a) La imagen nanoCT de la misma muestra de riñón de ratón que muestra las estructuras conocidas como extremidades ascendentes gruesas del bucle de Henle. Esta es una vista detallada de la región (IIb) que se ve en la Figura 1 y la Figura 2 adquirida a partir de una pequeña pieza del riñón con un tamaño de voxel de aproximadamente 400 nm. La preparación de la muestra implicó la tinción, disección y CPD. (b) La representación del volumen de los datos nanoCT que visualizan la estructura 3D de las extremidades ascendentes gruesas de los bucles de Henle. Esta cifra ha sido modificada de Busse y M'ller et al.26Por favor, haga clic aquí para ver una versión más grande de esta cifra.
Discusión
Actualmente, la eosina se utiliza como protocolo histológico estándar para etiquetar el citoplasma celular. El agente de tinción se aplica como una solución acuosa del 0,1% (p/v) a rodajas microscópicas de tejido blando (generalmente cortadas con un espesor de 2-10 m)33. La aplicación de este protocolo histológico estandarizado a muestras de tejido 3D como un riñón de ratón entero no da lugar a una imagen de TC mejorada de contraste de atenuación. Por un lado, esto se puede atribuir a las propiedades de baja atenuación intrínseca del tejido blando para las energías de rayos X típicamente utilizadas de los sistemas de microCT basados en laboratorio. Por lo general, el tejido blando se compone principalmente de carbono, hidrógeno, oxígeno y nitrógeno34,y por lo tanto, no resulta en la mejora del contraste. Por otro lado, la baja concentración de eosina utilizada para la tinción fue el factor limitante. A pesar de que una molécula de eosina contiene cuatro átomos de bromuro (alto número atómico de bromo con Z a 3534),no se cumplieron los niveles de sensibilidad requeridos para la toma de imágenes por TC de rayos X.
Para superar este desafío de bajo contraste de atenuación, se investigaron varias concentraciones de eosina. Una limitación es aquí la máxima solubilidad de la eosina en el agua, que es 30% (p/v) en una solución acuosa. Mejor mejora del contraste de atenuación dentro del tejido blando se observó con la mayor concentración de eosina, que se esperaba de acuerdo con la Ley Lambert-Beer. Por lo tanto, el protocolo de tinción final se llevó a cabo con la mayor concentración.
La pregunta de cómo preparar el tejido blando de manera óptima a nivel molecular para el procedimiento de tinción para mejorar aún más la mejora del contraste fue respondida por el ajuste del pH. Aquí, la acidificación de la muestra de tejido blando durante la fijación o antes de la tinción se encontró que es crucial. Esto también fue demostrado por Hong et al.35. La mayor acumulación de agente de tinción dentro del citoplasma celular por el ácido se logró a través de interacciones iónicas mejoradas, que fueron el resultado de la protonación de las cadenas laterales de aminoácidos de proteínas y péptidos presentes dentro del citoplasma celular. En la Figura 1a,bse muestra un resultado representativo que destaca la mejora del contraste en comparación con una muestra de tejido blando no manchado. Aquí, se logró una visión estructural de todo un riñón de ratón que visualiza regiones anatómicas cruciales como la corteza, la médula, la papila y la pelvis renal.
El protocolo de tinción presentado es fácil de aplicar y contiene sólo tres pasos. Los reactivos necesarios son fácilmente accesibles. El tiempo total de tinción de 24 horas es rápido para una tinción de órgano entero, lo que permite la visualización 3D de muestras de tejidos blandos(Figura 1c, Figura 2b y Figura 4b) en un entorno de laboratorio en un entorno de laboratorio en múltiples escalas hacia abajo a nivel celular. Cabe señalar que el tiempo general de tinción y el volumen de la solución de tinción necesaria podrían solicitar algunas adaptaciones dependiendo de la naturaleza de la muestra. Sin embargo, el protocolo de tinción basado en eosina es adecuado para la tinción de órganos enteros, que luego permite imágenes microCT de alta resolución de órganos enteros. No se observaron artefactos de contracción debido al etanol solvente, que se utilizó para mantener la muestra húmeda durante las mediciones de microCT. Se requieren pasos de preparación adicionales para la creación de imágenes nanoCT, lo que permite la investigación de piezas de tejido más pequeñas recuperadas de la muestra original. Con respecto a futuras aplicaciones histopatológicas, los escaneos de resumen proporcionarán información valiosa sobre las regiones y estructuras anatómicas alteradas, lo que permite la determinación de los ROC como se muestra en la Figura 2a. Estos pueden ser estudiados en 3D por microCT(Figura 1c y Figura 2b) o nanoCT (Figura 4b) y evaluados en 2D con histología(Figura 3).
Otra fuerza del protocolo se ve en la plena compatibilidad con la histopatología con respecto al procedimiento de tinción H&E. La aplicación del procedimiento de tinción basado en eosina a muestras a granel no impide nuevas investigaciones histológicas(Figura 3), aunque la concentración de eosina aplicada sea mucho mayor en comparación con la solución de tinción histológica. La rebanada nanoCT con un espesor virtual de aproximadamente 400 nm(Figura 3a)se compara ya muy bien con la sección histológica(Figura 3c), que se deriva de la muestra de tejido blando correspondiente. Teniendo en cuenta el espesor aproximado de una sección histológica con 7-10 m, la generación de rodajas de proyección de intensidad mínima de los datos nanoCT(Figura 3b), que corresponden a un espesor virtual de aproximadamente 7 m, permite una mejor comparación con la sección histológica (Figura 3c). Aquí, los núcleos celulares se revelan claramente como área de no atenuación como eosina específicamente mancha proteínas y péptidos en el citoplasma celular33.
La aplicación de una mayor tinción de contador con métodos histológicos estándar es posible, a pesar de que se ha invertido el orden de la tinción en comparación con el procedimiento de tinción histológica estándar. Comenzando primero con el protocolo de tinción basado en eosina desarrollado para TC, seguido de la tinción de contrarresta de esas secciones histológicas basadas en eosina con hematoxilina, permite una compatibilidad completa y da como resultado una tinción de alta calidad que muestra la forma esperada de apariencia. La tinción específica de los núcleos celulares con la hematoxilina agria de Mayer se aplicó a la sección histológica resaltando los núcleos celulares en púrpura(Figura 3d). La aplicación de la tinción de contador histológico se limita actualmente a la mancha H. Hay que evaluar otras manchas de contador histológico estándar, como la base periódica del ácido Schiff, Elastica van Gieson o La plata Gomori, así como la compatibilidad con técnicas inmunohistológicas.
El protocolo de tinción basado en la eosina permite (i) la orientación específica del citoplasma celular, (ii) la tinción homogénea y completa, (iii) la implementación fácil, (iv) la penetración rápida del tejido sin crear artefactos como anillos de difusión, (v) la tinción de muestras de tejidos blandos grandes y densas, y (vi) plena compatibilidad con la histopatología con respecto a la mancha de H&E. Estos requisitos son importantes para permitir la visualización de TC de rayos X de alta resolución de tejido blando hasta el nivel celular. En combinación con los dispositivos nanoCT desarrollados recientemente12,36,37, generación no destructiva de rodajas histológicas virtuales que son comparables en contraste y resolución a los datos histológicos convencionales se hace posible. Este enfoque combinado permitirá el establecimiento de la TC de rayos X como una herramienta valiosa para la visualización 3D de estructuras microscópicas de tejidos.
Divulgaciones
Los autores no tienen nada que revelar.
Agradecimientos
Agradecemos al Dr. Enken Drecoll las discusiones histológicas y al equipo extremadamente útil de Excillum AB, Suecia. Reconocemos el apoyo financiero a través del DFG Cluster of Excellence Munich Center for Advanced Photonics (MAP) y el DFG Gottfried Wilhelm Leibniz Program. Además, este proyecto de investigación ha recibido financiación del programa de investigación e innovación Union Horizon 2020 de Europa en el marco del Acuerdo de Subvención Marie Sk-odowska-Curie No. H2020-MSCA-IF-2015-703745-CONSALT.
Materiales
| Name | Company | Catalog Number | Comments |
| 50-ml centrifuge tube by Falcon | VWR | 734-0453 | |
| Formaldehyde solution, 37% | Carl Roth | CP10.2 | acid-free, stabilized with ~10% MeOH |
| Glacial acetic acid | Alfa Aesar | 36289.AP | |
| Eosin Y disodium salt | Sigma-Aldrich | E4382 | certified by Biological Stain Commission |
| Phosphate Buffered Saline (PBS) | Merck | L1825 | Dulbecco's formualtion, w/o calcium and magnesium |
| Sample Tubes by Nalgene | Carl Roth | ATK5.1 | |
| Rocking Shaker ST5 | CAT | 60281-0000 | |
| Cellulose tissue paper | VWR | 115-0600 | |
| Forceps, by USBECK Laborgeräte | VWR | 232-0096 | |
| Microcentrifuge tubes by Eppendorf | VWR | 211-2120 | safe-lock, 2.0 ml |
| Ethanol absolute by Baker Analyzed | VWR | 80252500 | |
| Disposable safety scalpel by Aesculap | VWR | AESCBA210 | |
| Petri dish by Sterilin | VWR | 391-2019 | |
| Plastic pasteur pipette | Carl Roth | EA68.1 | graduated, 1 ml |
| Desiccator by Duran | VWR | SCOT247826954 | |
| Silicone grease by Bayer | Sigma-Aldrich | 85404 | high-vacuum |
| Carbon dioxide cylinder with standpipe | Linde | 3700113 | 10 kg, short |
| micro-porous treatment capsule | PLANO GmbH | 4614 | pore size 78 µm (B) |
| Bal-Tec CPD 030 | Bal-Tec AG | CO2 as drying agent | |
| Stemi 2000-C stereomicroscope with KL 1500 LCD | Zeiss | this stereomicroscope has been updated(1) | |
| Zeiss Xradia Versa 500 | Zeiss | this microCT scanner has been updated(2) | |
| Avizo Fire 8.1 | Thermo Fisher Scientific | ||
| PILATUS detector as part of the nanoCT scanner | Dectris | single-photon counting detector(4,5); there are commercially availble nanoCT systems available (6,7) | |
| nanofocus X-ray source as part of the nanoCT scanner | Excillum | high-flux nanofocus X-ray transmission tube(3); there are commercially availble nanoCT systems available(6,7) | |
| (1) Germany, Z. ZEISS product information: ZEISS stereomicroscopes https://www.micro-shop.zeiss.com/de/de/system/Stereomikroskope/1006> (September 06, 2019). | |||
| (2) Germany, Z. ZEISS product information: ZEISS Xradia 510 Versa https://www.zeiss.com/microscopy/int/products/x-ray-microscopy/zeiss-xradia-510-versa.html> (April 10, 2019). | |||
| (3) Nachtrab, F. et al. Development of a Timepix based detector for the NanoXCT project. Journal of Instrumentation 10 (11), C11009, (2015). | |||
| (4) Kraft, P. et al. Performance of single-photon-counting PILATUS detector modules. Journal of Synchrotron Radiation 16 (3), 368-375, (2009). | |||
| (5) Kraft, P. et al. Characterization and calibration of PILATUS detectors. IEEE Transactions on Nuclear Science 56 (3), 758-764, (2009). | |||
| (6) Germany, Z. ZEISS product information: ZEISS Xradia 810 Ultra https://www.zeiss.com/microscopy/int/products/x-ray-microscopy/xradia-810-ultra.html> (April 9 2019). | |||
| (7) Company, G. E. GE product information: Phoenix nanotom m, https://www.gemeasurement.com/sites/gemc.dev/files/geit-31344en_nanotom_m_0517.pdf> (April 10, 2019). |
Referencias
- Suvarna, S. K., Layton, C., Bancroft, J. D. . Theory and Practice of Histological Techniques. 7th edn. , (2013).
- Chatterjee, S. Artefacts in histopathology. Journal of Oral and Maxillofacial Pathology. 18 (4), 111-116 (2014).
- McInnes, E. Artefacts in histopathology. Comparative Clinical Pathology. 13 (3), 100-108 (2005).
- Andreasen, A., Drewes, A., Assentoft, J., Larsen, N. Computer-assisted alignment of standard serial sections without use of artificial land-marks. A practical approach to the utilization of incomplete information in 3-d reconstruction of the hippocampal region. Journal of Neuroscience Methods. 45 (3), 199-207 (1992).
- Braverman, M. S., Braverman, I. M. Three-dimensional reconstructions of objects from serial sections using a microcomputer graphics system. Journal of Investigative Dermatology. 86 (3), 290-294 (1986).
- Denk, W., Hortsmann, H. Serial block-face scanning electron microscopy to reconstruct three-dimensional tissue nanostructure. PLoS Biology. 2 (11), 329 (2004).
- Mohun, T. J., Weninger, J. W. Imaging heart development using high-resolution episcopic microscopy. Current Opinion in Genetics and Development. 21, 573-578 (2011).
- Weninger, J. W., Meng, S., Streicher, J., Müller, G. B. A new episcopic method for rapid 3-d reconstruction: applications in anatomy and embryology. Anatomy and Embryology. 197 (5), 341-348 (1998).
- Odgaard, A., Andersen, K., Melsen, F., Gundersen, H. J. G. A Direct Method for Fast 3-Dimensional Serial Reconstruction. Journal of Microscopy. 159, 335-342 (1990).
- Müller, M., et al. Myoanatomy of the velevt worm leg revealed by labratory-based nanofocus X-ray source tomography. Proceedings of the National Academy of Sciences of the United States of America. 114 (47), 12378-12383 (2017).
- Salomon, M., Hanke, R., Krüger, P., Uhlmann, N., Voland, V. Realization of a computed tomography setup to achieve resolutions below 1 µm. Nuclear Instruments and Methods in Physics Research Section A. 591, 50-53 (2008).
- Tkachuk, A., et al. X-ray computed tomography in Zernike phase contrast mode at 8 keV with 50-nm resolution using Cu rotating anode X-ray source. Zeitschrift für Kristallographie. 222, 650-655 (2007).
- Withers, P. J. X-ray nanotomography. Materials Today. 10, 26-34 (2007).
- Jahn, H., et al. Evaluation of contrasting techniques for X-ray imaging of velvet worms (Onychophora). Journal of Microscopy. 270 (3), 343-358 (2018).
- Martins de S. e Silva, J., et al. Three-dimensional non-destructive soft-tissue visualization with X-ray staining micro-tomography. Scientific Reports. 5, 14088 (2015).
- Metscher, B. D. MicroCT for Developmental Biology: A Versatile Tool for High-Contrast 3D Imaging at Histological Resolutions. Developmental Dynamics. 238, 632-640 (2009).
- Metscher, B. D. MicroCT for comparative morphology: simple staining methods allow high-contrast 3D imaging of diverse non-mineralized animal tissues. BMC Physiology. 9, 11 (2009).
- Mitzutani, R., et al. X-Ray Microtomographic Imaging of Three-Dimensional Structure of Soft Tissues. Tissue Engineering Part C: Methods. 14 (4), 359-363 (2008).
- Pauwels, E., Loo, D. v., Cornillie, P., Brabant, L., Hoorebeke, L. v. An exploratory study of contrast agents for soft tissue visualization by means of high resolution X-ray computed tomography imaging. Journal of Microscopy. 250, 21-31 (2013).
- Degenhardt, K., Wright, A. C., Horng, D., Padmanabhan, A., Epstein, J. A. Rapid 3D phenotyping of cardiovascular development in mouse embryos by micro-CT with iodine staining. Circulation: Cardiovascular Imaging. 3 (3), 314-322 (2010).
- Dullin, C., et al. μCT of ex-vivo stained mouse hearts and embryos enables a precise match between 3D virtual histology, classical histology and immunochemistry. PLOS ONE. 12 (2), 0170597 (2017).
- Jeffrey, N. S., Stephenson, R. S., Gallagher, J. A., Cox, P. Micro-computed tomography with iodine staining resolves the arrangement of muscle fibres. Journal of Biomechanics. 44, 189-192 (2011).
- Johnson, J. T., et al. Virtual Histology of Transgenic Mouse Embryos for High-Throughput Phenotyping. PLOS Genetics. 2, 61 (2006).
- Leszczyński, B., et al. Visualization and Quantitative 3D Analysis of Intraocular Melanoma and Its Vascularization in a Hamster Eye. International Journal of Molecular Sciences. 19 (2), 332 (2018).
- Mizutani, R., Suzuki, Y. X-ray microtomography in biology. Mircon. 43, 104-115 (2012).
- Busse, M., et al. Three-dimensional virtual histology enabled through cytoplasm-specific X-ray stain for microscopic and nanoscopic computed tomography. Proceedings of the National Academy of Sciences of the United States of America. 115 (10), 2293-2298 (2018).
- Müller, M., et al. Non-destructive high-resolution 3D virtual histology enabled through a cell nucleus-specific stain for X-ray computed tomography. Scientific Reports. 8, 17855 (2018).
- . ZEISS product information: ZEISS Xradia 510 Versa Available from: https://www.zeiss.com/microscopy/int/products/x-ray-microscopy/zeiss-xradia-510-versa.html (2019)
- Nachtrab, F., et al. Development of a Timepix based detector for the NanoXCT project. Journal of Instrumentation. 10 (11), 11009 (2015).
- Kraft, P., et al. Performance of single-photon-counting PILATUS detector modules. Journal of Synchrotron Radiation. 16 (3), 368-375 (2009).
- Kraft, P., et al. Characterization and calibration of PILATUS detectors. IEEE Transactions on Nuclear Science. 56 (3), 758-764 (2009).
- . ZEISS product information: ZEISS Xradia 810 Ultra Available from: https://www.zeiss.com/microscopy/int/products/x-ray-microscopy/zeiss-xradia-510-versa.html (2019)
- Anderson, T. F. Techniques for the preservation of three-dimensional structure in preparing specimens for the electron microscope. Transactions of the New York Academy of Sciences. 13 (4), 130-134 (1951).
- Bray, D., Williams, J. R., Clifford, A. A. . Supercritical Fluid Methods and Protocols. Methods in Biotechnology. 13, 235-243 (2000).
- Lucy, L. B. An iterative technique for the rectification of observed distributions. The Astronomical Journal. 79, 745-765 (1974).
- Richardson, W. H. Bayesian-Based Iterative Method of Image Restoration. The Journal of the Optical Society of America. 62 (1), 55-59 (1972).
- Paganin, F., Mayo, S. C., Gureyev, T. E., Miller, P. R., Wilkins, S. W. Simultaneous phase and amplitude extraction from a single defocused image of a homogeneous object. Journal of Microscopy. 206, 33-40 (2002).
- Riedelsheimer, B., Büchl-Zimmermann, S., Mulisch, M., Welsch, U. . Mikroskopische Technik. , 193-194 (2015).
- Hubbell, J. H., Seltzer, S. M. Tables of Xray mass attenuation coefficients and mass energy-absorption coefficients from 1 keV to 92 keV and 48 additional substances of dosimetric interest, Table 3. National Institute of Standards and Technology. , (1995).
- Hong, H. Y., Yoo, G. S., Choi, J. K. An Eosin Y Method for Protein Determination in Solution. Analytical Letters. 32 (12), 2427-2442 (1999).
- . GE product information: Phoenix nanotom m Available from: https://www.gemeasurement.com/sites/gemc.dev/files/geit-31344en_nanotom_m_0517.pdf (2019)
- Dierick, M., et al. Recent Micro-CT Scanner Developments at UGCT. Nuclear Instruments and Methods in Physics Research Section B. 324, 35-40 (2014).
- Kastner, J., Plank, B., Heinzl, C. Advanced X computed tomography methods: High resolution CT, quantitative CT, 4DCT and phase contrast CT. Proceedings of Digital Industrial Radiology and Computed Tomography. , 120-132 (2015).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoExplorar más artículos
This article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados