Method Article
3D-Bildgebung von Weichgewebeproben mit einer Röntgen-spezifischen Färbemethode und nanoskopischer Computertomographie
* Diese Autoren haben gleichermaßen beigetragen
In diesem Artikel
Zusammenfassung
Ein Protokoll zur 3D-Visualisierung mikroskopischer Gewebestrukturen mit Hilfe einer Röntgen-spezifischen Färbemethode für die Röntgen-Computertomographie wird vorgestellt.
Zusammenfassung
Wir zeigen eine laborbasierte Methode, die RöntgenmikroCT und NanoCT mit einem spezifischen Röntgenfleck kombiniert, der auf das Zellzytoplasma abzielt. Das beschriebene Protokoll ist einfach anzuwenden, schnell und für größere Weichgewebeproben geeignet. Die vorgestellte Methodik ermöglicht die Charakterisierung wichtiger Gewebestrukturen in drei Dimensionen und wird auf einer ganzen Mausniere demonstriert. Der multiskalige Ansatz ermöglicht das Abbilden der gesamten Mausniere und unterstützt die Auswahl weiterer Interessensvolumina, die mit höheren Auflösungen bis in den Nanometerbereich erfasst werden. Dabei wird die Weichgewebemorphologie mit einer ähnlichen Detailebene wie die entsprechenden histologischen Lichtmikroskopiebilder reproduziert. Tiefere Einblicke in die 3D-Konfiguration von Gewebestrukturen werden ohne weitere Untersuchungen durch histologische Methoden gewonnen.
Einleitung
Die vollständige Charakterisierung von Weichgewebeproben erfordert Informationen über die 3D-Gewebemikrostruktur. Der aktuelle Goldstandard für Weichgewebeprobenanalysen ist die Histopathologie. Die Gewebe- und Zellmorphologie der Probe wird in 2D in ausgewählten Interessengebieten (ROIs) mittels optischer Mikroskopie1untersucht. Diese Methode hat jedoch mehrere Nachteile. Die Vorbereitung der Probe ist zeitaufwändig, kompliziert, zerstörerisch und anfällig für Artefakte. Die produzierten mikroskopischen Dias liefern nur 2D-Informationen parallel zur Schnittebene. Oft ist die Anzahl der untersuchten histologischen Abschnitte aus Zeitgründen2,3begrenzt.
In den letzten Jahren hat sich der Bereich der 3D-Histologie weiterentwickelt. Hier sind virtuelle Gewebescheiben aus jeder gewünschten räumlichen Ebene zugänglich. Dies ermöglicht die Verfolgung von Strukturen in der gesamten Stichprobe, was zu einem tieferen Verständnis der 3D-Gewebearchitektur und struktureller Veränderungen im Zusammenhang mit verschiedenen Pathologien führt. Zur Generierung von 3D-Volumendaten wurden verschiedene Methoden entwickelt. Sie reichen von seriellen, abschnittsbasierten Ansätzen, die entweder Licht- oder Elektronenmikroskopie4,5,6,7,8, bis hin zu Block-Face-Bildgebungsmethoden wie episkopischer 3D-Bildgebung verwenden. oder Block-Face-Rasterelektronenmikroskopie7,8,9. Alle genannten Methoden beinhalten jedoch entweder die Schnitt- oder Zerlegung der Probe vollständig, was keine weiteren Untersuchungen zulässt. Die erhaltene Auflösung hängt stark davon ab, dass der Schnittprozess anfällig für Artefakte ist, wie in der konventionellen Histologie beschrieben. Diese Methoden leiden auch unter Ausrichtungsartefakten.
3D-Röntgenbildgebungstechniken wie mikroskopische und nanoskopische Computertomographie (microCT und nanoCT) streben danach, hochauflösende 3D-Daten ohne Zerstörung der Gewebeprobe zu erzeugen. Bisher haben der schwache Röntgendämpfungskontrast von Weichgewebe und der eingeschränkte Zugang zu hohen Auflösungen in einer Laborumgebung ihre Verwendung für die 3D-Visualisierung mikroskopischer Gewebestrukturen beeinträchtigt. Jüngste Fortschritte in Richtung laborbasierter, hochauflösender Röntgen-CT ermöglichen Auflösungen weit unter 1 m10,11,12,13.
Der fehlende Kontrast im Weichgewebe in der konventionellen Dämpfungs-basierten Röntgenbildgebung wird durch Färbemittel kompensiert, die den Röntgendämpfungskontrast verbessern. Färbemittel, die aus anderen bildgebenden Verfahren wie Osmiumtetroxid (OsO4), Jodkaliumjodid (IKI) oder Phosphotungstiksäure (PTA) bekannt sind, werden häufig14,15,16,17, 18,19,20,21,22,23,24,25. Färbemittel, die (i) spezifische biologische Spezifizien, (ii) homogene und vollständige Färbung, (iii) einfache Handhabung, (iv) schnelles Eindringen des Gewebes ohne Entstehung von Artefakten wie Diffusionsringe, (v) große und dichte Gewebefärbung und (vi) ermöglichen Die volle Kompatibilität mit der Histopathologie ist erforderlich, um Röntgen-CT als Werkzeug zur 3D-Visualisierung mikroskopischer Gewebestrukturen zu etablieren. In dieser Arbeit zeigen wir, wie Weichgewebeproben für die Röntgen-CT-Bildgebung mit einem zytoplasma-spezifischen Röntgenfleck auf Basis von Eosin vorbereitet werden, der die oben genannten Anforderungenerfüllt.
Der multiskale Bildgebungsansatz gewährleistet die Bewertung der Färbequalität durch eine Übersichts-MikroCT-Messung und die Auswahl von VoIs (Volumes of Interest) für weitere hochauflösende Untersuchungen. Die Färbequalität wird unter dem Schwerpunkt auf Färbeparametern wie (i) Vollständigkeit, (ii) Aussehen von Diffusionsringen, (iii) Kontrastverbesserung, (iv) Aussehen von CT-Artefakten wie Streifen und (v) Homogenität analysiert. Das laborbasierte NanoCT-Setup, das die geometrische Vergrößerung verwendet, um Auflösungen bis zu 100 nm zu erreichen, visualisiert die Weichgewebemorphologie auf der (sub)zellulären Ebene10,27. Eine vergleichende Analyse der nanoCT-Scheiben mit entsprechenden histologischen Lichtmikroskopiebildern bestätigt die Reproduktion der Gewebearchitektur mit ähnlichen Details auf mikroskopischer Ebene in 2D und ermöglicht so die histopathologische Charakterisierung des Gewebes probe. Dieses detaillierte Videoprotokoll soll das Bewusstsein für diese Methode schärfen und das Potenzial dieser Methode als zerstörungsfreies 3D-Weichgewebe-Bildgebungswerkzeug hervorheben, das für eine breite wissenschaftliche Gemeinschaft wie Zoologen, Biologen und Gesundheit von Interesse ist. Profis.
Protokoll
Achtung: Bitte beachten Sie vor der Verwendung alle relevanten Sicherheitsdatenblätter (MSDS). Mehrere der im Protokoll verwendeten Chemikalien sind akut toxisch und krebserregend. Bitte verwenden Sie alle geeigneten Sicherheitspraktiken bei der Durchführung des Färbeprotokolls, einschließlich der Verwendung von technischen Steuerungen (Rauchhaube, Handschuhkasten) und persönlicher Schutzausrüstung (Schutzbrille, Handschuhe, Labormantel, Hose in voller Länge, geschlossene Schuhe).
Verwendete Tiere:
Die Tierhaltung erfolgte am Klinikum rechts der Isar, Technische Universität München nach den Richtlinien der Europäischen Union 2010/63. Die Organentnahme wurde von einem internen Tierschutzausschuss des Klinikumrechts der Isar, München, genehmigt (interne Referenznummer 4-005-09). Alle Verfahren entsprachen den einschlägigen Richtlinien und Vorschriften. Alle Laboratorien werden nach den OECD-Grundsätzen der guten Laborpraxis geprüft.
1. Eosin-Färbeprotokoll
- Um Weichgewebeproben zu fixieren, füllen Sie ein 50-ml-Zentrifugenrohr mit einer fixativen Lösung, die 9,5 ml 4% (v/v) Formaldehydlösung (FA) und 0,5 ml Eisessigsäure (AA) enthält.
HINWEIS: Bereiten Sie die FA-Lösung frisch aus einer 37% säurefreien FA-Lösung vor, die mit ca. 10% Methanol stabilisiert ist. Verdünnen Sie die FA-Lösung mit der Phosphatgepufferte Saline (DPBS) von Dulbecco weiter. Wählen Sie DPBS ohne Kalzium und Magnesium. Bewahren Sie die verdünnte FA-Lösung nicht länger als einen Monat auf. Während der Versauerung ändert sich der pH-Wert der fixativen Lösung von neutral auf ca. 3.
VORSICHT: Da FA akut organtoxisch, korrosiv und krebserregend ist, ist die Verwendung einer Dunstabzugshaube obligatorisch und es muss eine geeignete persönliche Schutzausrüstung verwendet werden.- Fügen Sie die frisch entfernte Weichgewebeprobe in ein 50-ml-Zentrifugenrohr und kühlen Sie das 50-ml-Zentrifugenrohr für 24-72h.
HINWEIS: Das Protokoll kann hier angehalten werden. - Waschen Sie die Weichgewebeprobe mit DPBS-Lösung für 1h.
- Fügen Sie die frisch entfernte Weichgewebeprobe in ein 50-ml-Zentrifugenrohr und kühlen Sie das 50-ml-Zentrifugenrohr für 24-72h.
- Um die fixierte Weichteilprobe (z.B. eine ganze Mausniere) zu färben, legen Sie das Weichgewebe in 2 ml Eosin-Y-Färbungslösung und inkubieren Sie die Probe 24 h. Halten Sie die Probe auf einer horizontalen Schüttelplatte für ein glattes Schaukeln (ca. 60 Umdrehungen pro Minute) während der Inkubation verfahren.
HINWEIS: Die Eosin-Y-Färbungslösung hat eine Konzentration von 30% (w/v) in destilliertem Wasser. Wählen Sie das Volumen der Färbelösung so aus, dass die Probe vollständig von der Färbelösung abgedeckt ist, und lassen Sie die Probe sich frei im Probenbehälter bewegen. Die Inkubationszeit kann bei anderen Proben abweichen und muss entsprechend angepasst werden. - Nach der Färbung die Weichteilprobe vorsichtig aus dem Probenbehälter entfernen.
- Entfernen Sie vorsichtig überschüssiges Färbemittel mit Zellulose-Gewebepapier.
- Legen Sie die Weichgewebeprobe in einen konischen Probenbehälter über einer Ethanoldampfphase zur Lagerung und weiteren Verwendung.
HINWEIS: Der konische Probenbehälter muss immer ein paar Tropfen 70% (v/v) Ethanol an der Unterseite des Rohres enthalten, um die Weichgewebeprobe feucht zu halten und Artefakte zu verhindern.
2. Röntgen-MikroCT-Bildgebung
ANMERKUNG: Die Röntgen-MikroCT-Messungen wurden mit einem MikroCT-Scanner durchgeführt, der Übersichts-CT-Messungen (die Möglichkeit, die gesamte Probe innerhalb des Sichtfeldes (FOV)) abzubilden) und die Leistung von hochauflösenden CT-Messungen (die Fähigkeit, auf einem gewünschten Zinsvolumen (VOI) derselben Probe) bis zu 1 m.
- Montieren Sie die Weichgewebeprobe an einem geeigneten Probenhalter. Stellen Sie sicher, dass die Probe eng am Probenhalter angebracht ist, um zu verhindern, dass sich die Probe während der Röntgen-CT-Messungen bewegt.
- Bei der gefärbten Mausniere: Bereiten Sie einen Probenhalter mit zwei Zentrifugenrohren vor, wobei der Boden eines Rohres abgeschnitten wird. Kleben Sie die beiden Zentrifugenrohre mit Zweikomponenten-Klebstoff zusammen. Stellen Sie eine gerade Ausrichtung der Zentrifugenrohre um die Rotationsachse sicher. Warten Sie, bis der Klebstoff aushärtet ist.
- Sobald der Probenhalter einsatzbereit ist, übertragen Sie die Mausniere in das intakte Zentrifugenrohr, das ein paar Tropfen 70% (v/v) Ethanol an der Unterseite des Rohres hält.
HINWEIS: Die Stabilität der Probe ist von entscheidender Bedeutung. Nehmen Sie sich Zeit, um die Probe für Röntgen-CT-Messungen vorzubereiten. Die Weichgewebeprobe wird über eine Ethanoldampfphase gehalten, um die Probe während der Röntgen-CT-Messungen feucht zu halten und eine Schrumpfung der Weichgewebeprobe und andere Artefakte zu verhindern. Die Weichgewebeprobe sollte nicht mit dem Lösungsmittel in Berührung kommen, um die Ansammlung des Lösungsmittels um die Probe während der Röntgen-CT-Messung zu vermeiden, was während der Messung zu einer Probenbewegung führen oder während der Rekonstruktion zu Problemen führen könnte. Lässt der Probenhalter kein Lösungsmittel an der Unterseite zu, kann ein mit 70 % (v/v) Ethanol befeuchtetes Zellulosepapier in den Probenhalter gelegt werden. Es sei darauf hingewiesen, dass Schrumpfartefakte aufgrund des Lösungsmittels Ethanol nicht beobachtet wurden.
HINWEIS: Das Protokoll kann hier angehalten werden.
- Wählen Sie nach sorgfältiger Ausrichtung der Probe Erfassungsparameter für die beste Bildqualität aus. Bei den vorgelegten microCT-Daten erfassen Sie den Scan bei einer Spitzenspannung von 50 kV, einem Strom von 3,5 W mit 1601 Projektionen, die gleichmäßig über 360° verteilt sind.
HINWEIS: Die Erfassungsparameter für den Übersichts-CT-Scan wurden für die beste Bildqualität ausgewählt. Als solches wurde das 0,39-fache Kameraziel gewählt, um die gesamte Probe im Sichtfeld (FOV) abzudecken. Dies führte zu einer effektiven Pixelgröße von 12 m. Die Belichtungszeit von 2 s pro Projektion lieferte ein gutes Signal-Rausch-Verhältnis. Der ROI für den hochauflösenden CT-Scan wurde anhand der microCT-Daten aus dem Übersichtsscan ermittelt. MicroCT-Scanner verfügen oft über ein integriertes Software-Tool, das eine präzise Auswahl des ermittelten ROI ermöglicht. Für die hochauflösenden CT-Daten wurde das 4x Kameraobjektiv gewählt, was zu einer effektiven Pixelgröße von 3,3 m führte. Hier war eine Belichtungszeit von 15 s pro Projektion erforderlich.
HINWEIS: Das Protokoll kann hier angehalten werden. - Nach Erfassung der Röntgen-CT-Daten werden die Projektionen entsprechend für die Rekonstruktion des 3D-Volumens verarbeitet. Bei den vorgestellten microCT-Daten: Rekonstruieren Sie die Röntgen-CT-Daten mit der integrierten Software.
HINWEIS: Die Volumen-Renderings der in Abbildung 1 und Abbildung 2 dargestellten microCT-Daten wurden mit einer Visualisierungssoftware generiert.
HINWEIS: Das Protokoll kann hier angehalten werden.
3. Röntgen-NanoCT-Bildgebung
HINWEIS: Der Röntgen-NanoCT-Scanner wurde eigens entwickelt. Das linsenfreie Instrument ist mit einer Nanofokus-Röntgenquelle und einem Single-Photon-Zähldetektor ausgestattet. 3D-Daten mit Auflösungen bis zu 100 nm können10generiert werden. Im Allgemeinen sind nanoCT-Systeme, einschließlich solche mit Röntgenoptik, kommerziell erhältlich und nicht auf den beschriebenen NanoCT-Scanner beschränkt.
- NanoCT Probenvorbereitung
- Bereiten Sie VOIs der Weichgewebeprobe vor. Schneiden Sie das Weichgewebe mit einem Skalpell und einem Stereomikroskop in sehr kleine Stücke von ca. 0,5 mm Kantenlänge. Im Falle der Maus niere: Schneiden Sie die Mausniere in zwei Hälften entlang der längsten Achse. Nehmen Sie die eine Hälfte der Mausniere und bereiten Sie verschiedene anatomische Regionen wie Nierenrinde und Nierenmedulla.
ANMERKUNG: Die andere Hälfte der Mausniere wurde in die Histopathologie übertragen, wo die Probe in Paraffin eingebettet und entsprechend verarbeitet wurde, um die typischen histologischen Abschnitte gemäß Abbildung 3c und Abbildung 3d zu ergeben. . - Übertragen Sie die kleinen Stücke vor dem ersten Austrocknungsschritt in eine neue Petrischale, wo sie für alle nachfolgenden Schritte verbleiben.
- Dehydrieren Sie die Proben mit Konzentrationen (alle v/v) in %: 50, 60, 70, 80, 90, 96 und 100 Ethanol, das mit destilliertem Wasser ausgeglichen ist. Führen Sie jeden Austrocknungsschritt für jeweils 1 h aus.
HINWEIS: Das Protokoll kann hier angehalten werden. Bewahren Sie die kleinen Gewebestücke über Nacht in 100% Ethanol auf. - Kritischer Punkt trocken (CPD) die kleinen Gewebestücke.
HINWEIS: Die Anwendung von CPD ermöglicht die vollständige Austrocknung der Gewebeprobe durch den Austausch des Lösungsmittels (hier Ethanol) mit dem Trocknungsmittel (hier CO2). Dies war notwendig, um sicherzustellen, dass die Probe an den Probenhaltern des nanoCT montiert werden kann, sich während der Messung nicht bewegt und sehr nahe an der Röntgenquelle positioniert werden kann, um eine optimale geometrische Vergrößerung zu ermöglichen. Das nanoCT-Setup basiert auf einer reinen geometrischen Vergrößerung, wobei der Vergrößerungsfaktor als Quell-Zu-Detektor-Abstand über die Quell-zu-Probe-Entfernung definiert wird. Die Trocknungstechnik wurde zuerst von Anderson eingeführt, um die 3D-Struktur biologischer Proben für die Elektronenmikroskopie zu erhalten28. Eine Übersicht über die Technik bietet Bray29.- Füllen Sie die Vakuumkammer mit 100% Ethanol vor. Übertragen Sie die kleinen Gewebeteile in eine mikroporöse Kapsel und legen Sie sie in die Vakuumkammer der CPD. Schließen Sie das System.
HINWEIS: Da Hochdruck im CPD-Prozess involviert ist, stellen Sie sicher, dass alle Teile der CPD, insbesondere die Armaturen, intakt sind und das System ordnungsgemäß geschlossen ist. - Kühlen Sie die Kammer auf 6- 8°C und füllen Sie sie mit flüssigem CO2auf.
- Warten Sie beim Rühren 3 min, um eine ordnungsgemäße Mischung der beiden Komponenten zu ermöglichen. Die Kammer vorsichtig abtropfen lassen. Stellen Sie sicher, dass der Probenhalter noch mit Lösungsmittel bedeckt ist. Wiederholen Sie diesen Schritt zehnmal, um einen vollständigen Austausch von Ethanol durch CO2 innerhalb der Probe zu ermöglichen.
- Nach der Endbefüllung der Kammer mitCO2erhitzen Sie die Maschine bis zum kritischen Punkt von CO2 (31 °C und 73,8bar), gefolgt von einer sehr langsamen Freisetzung des gasförmigenCO2 über eine Zeit von 30 min.
HINWEIS: Die Freisetzung des Gases sollte sehr langsam erfolgen, da sich sonst Kondenswasser auf der Probe bilden kann. Stellen Sie sicher, dass die Temperatur nicht unter den kritischen Punkt von CO2fällt. Öffnen Sie die CPD-Maschine nur, wenn der gesamte Druck aus dem System gelöst wurde. - Entfernen Sie die CPD-Gewebeteile schnell von der Maschine und bewahren Sie sie vor der weiteren Verwendung in einer neuen Petrischale in einem Trockenhaus auf.
HINWEIS: Das Protokoll kann hier angehalten werden.
- Füllen Sie die Vakuumkammer mit 100% Ethanol vor. Übertragen Sie die kleinen Gewebeteile in eine mikroporöse Kapsel und legen Sie sie in die Vakuumkammer der CPD. Schließen Sie das System.
- Bereiten Sie VOIs der Weichgewebeprobe vor. Schneiden Sie das Weichgewebe mit einem Skalpell und einem Stereomikroskop in sehr kleine Stücke von ca. 0,5 mm Kantenlänge. Im Falle der Maus niere: Schneiden Sie die Mausniere in zwei Hälften entlang der längsten Achse. Nehmen Sie die eine Hälfte der Mausniere und bereiten Sie verschiedene anatomische Regionen wie Nierenrinde und Nierenmedulla.
- Montieren Sie die CPD-Gewebeteile an einem geeigneten Probenhalter. Stellen Sie sicher, dass die Probe eng am Probenhalter angebracht ist, um zu verhindern, dass sich die Probe während der CT-Messungen bewegt. Bei der CPD-Maus Nierengewebestücke: Kleben Sie die Gewebestücke mit Superkleber an einen Probenhalter.
HINWEIS: Jede unerwünschte Bewegung der Probe während CT-Messungen kann Probleme bei der Volumenrekonstruktion verursachen - insbesondere beim Erfassen eines Datensatzes mit Nanometer-Voxelgröße.
HINWEIS: Das Protokoll kann hier angehalten werden. - Wählen Sie nach sorgfältiger Ausrichtung der Probe Erfassungsparameter für die beste Bildqualität aus. Bei den vorgestellten nanoCT-Daten: Erfassen Sie Projektionen bei einer Spitzenspannung von 60 kV mit 1599 Projektionen, die gleichmäßig über 360° und eine Voxelgröße von ca. 400 nm verteilt sind.
ANMERKUNG: Eine einzelne CT-Messung, die bei einer Voxelgröße von 400 nm erfasst wird, hat einen FOV von 75 m in Richtung der Rotationsachse (vertikal) und etwa 560 m in Richtung senkrecht zur Rotationsachse (horizontal). Um größere Volumina zu untersuchen, kann eine Erweiterung des FOV entlang der Rotationsachse erreicht werden, indem mehrere Scans an verschiedenen vertikalen Positionen kombiniert werden. Darüber hinaus können lokale Tomographie-Scans durchgeführt werden, um Proben mit einem größeren Probendurchmesser zu messen, der senkrecht zur Rotationsachse liegt als vom FOV eines globalen CT-Scans angegeben. Die NanoCT-Daten wurden mit einer Belichtungszeit von 4 s pro Projektion erfasst. Die Gesamterfassungszeit pro Datensatz betrug etwa 3,5 h.
HINWEIS: Das Protokoll kann hier angehalten werden. - Nach Erfassung der CT-Daten werden die Projektionen entsprechend für die Rekonstruktion des 3D-Volumens verarbeitet.
- Bei den vorgestellten nanoCT-Daten normalisieren Sie die erfassten Projektionen mit Flachfeldbildern. Verbessern Sie die Schärfe der Projektionen, indem Sie einen Richardson-Lucy-Dekonvolutionalgorithmus30,31verwenden. Verwenden Sie eine rotationssymmetrische Gaußsche Funktion mit einer Standardabweichung von einem Pixel als Dekonvolution-Kernel.
- Wenden Sie Paganins Phasenabrufalgorithmus auf die geschärften Bilder an, um den Weichgewebekontrast zu erhöhen. Legen Sie die Parameter des Algorithmus fest, um die Bildqualität32zu optimieren. Rekonstruieren Sie die vorverarbeiteten Projektionen mit einem hochmodernen gefilterten Rückprojektionsalgorithmus.
ANMERKUNG: Abbildung 3 wertet die erhaltenen NanoCT-Daten mit entsprechenden histologischen Abschnitten aus, die etwa 7 m dick waren. Daher wurden durch die Berechnung des Mindestwertes für jedes Pixel in den relevanten Slices minimale Intensitätsprojektionsscheiben von 18 benachbarten NanoCT-Slices mit einer virtuellen Dicke von ca. 7 m erzeugt. Eine Visualisierungssoftware wurde verwendet, um das Volumen der nanoCT-Daten zu rendern, die in Abbildung 4dargestellt ist.
Ergebnisse
Abbildung 1 zeigt CT-Slices und Volumenwiedergabe von MikroCT-Daten mit niedriger Auflösung, die die Kontrastverbesserung nach der Färbung hervorheben. Abbildung 2 zeigt CT-Slices und Volumenwiedergabe von hochauflösenden MicroCT-Daten, die aus einer lokalen Tomographie der gesamten Mausniere abgeleitet wurden. Abbildung 3 zeigt CT-Slices von NanoCT-Daten im Vergleich zu den entsprechenden histologischen Abschnitten. Abbildung 4 zeigt die CT-Slice- und Volumenwiedergabe von NanoCT-Daten, die strukturelle Details auf zellulärer Ebene hervorheben. Die low-resolution microCT-Messung ermöglicht einen Überblick über das gesamte Organ und hilft, Interessenvoe (VOIs) für die hochauflösende MikroCT-Messung zu identifizieren. Durch diesen multiskalen Ansatz wird das VOI für den nanoCT bestimmt. Das nanoCT ermöglicht eine sehr detaillierte Ansicht der Weichgewebeprobe auf zellulärer Ebene. Die vergleichende Studie mit der entsprechenden histologischen Sektion zeigt die volle Kompatibilität mit der Histopathologie. Hier bestätigt der multimodale Bildgebungsansatz die Ergebnisse, die mit beiden Modalitäten erzielt wurden.
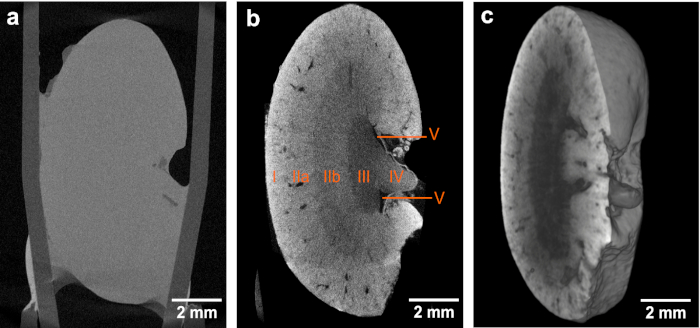
Abbildung 1. CT-Slices und Volumen-Rendering der microCT-Daten mit niedriger Auflösung. (a,b) Übersichtsbilder der gleichen Mausniere vor bzw. nach der Färbung, wobei die Kontrastverbesserung hervorgehoben wird, die nach Anwendung des eosinbasierten Färbeprotokolls erzielt wurde. Beide MicroCT-Datensätze wurden mit identischen Erfassungsparametern erfasst. Die Voxelgröße in beiden Datensätzen beträgt 12 m. Die in (b) erzielte Kontrastverbesserung ermöglicht die Identifizierung der folgenden anatomischen Strukturbereiche: Cortex (I), äußere Medulla (II) mit weiterer Unterscheidung in äußeren Streifen der äußeren Medulla (IIa) und inneren Streifen der äußeren Medulla (IIb), innere Medulla (III), Papille (IV) und Nierenbecken (V). (c) Volumenwiedergabe von microCT-Daten, die einen virtuellen sagittalen Abschnitt durch die gesamte Mausniere zeigen. Diese Zahl wurde von Busse und Müller et al.26geändert. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 2. CT-Slice- und Volumen-Rendering von hochauflösenden MicroCT-Daten, die nach Anwendung des entwickelten eosinbasierten Färbeprotokolls von derselben Mausniere abgeleitet wurden. (a) In der linken Ecke wird das Übersichts-MicroCT-Bild angezeigt, das den ROI (blaue Box) für das angezeigte hochauflösende Bild hervorhebt. Folgende anatomische Strukturbereiche sind identifizierbar: Cortex (I), äußere Medulla (II) mit weiterer Unterscheidung in äußeren Streifen der äußeren Medulla (IIa) und inneren Streifen der äußeren Medulla (IIb), innere Medulla (III), kleiner Kelch (IV) und Gefäße (V und VI). (b) Volumen der Interessenwiedergabe der hochauflösenden MikroCT-Daten, die mit einer Voxelgröße von 3,3 m erfasst wurden. Die Medulla-Region und ein virtueller Abschnitt durch ein Gefäß, das aus einer lokalen Tomographie der gesamten Niere abgeleitet ist, wird gezeigt. Diese Zahl wurde von Busse und Müller et al.26geändert. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 3. CT-Slices von NanoCT-Daten (a,b) im Vergleich zu den histologischen Abschnitten (c,d), die nach Anwendung des entwickelten eosinbasierten Färbeprotokolls aus derselben Mausniere abgeleitet wurden. (a) Das NanoCT-Bild derselben Mausnierenprobe nach Färbung, Sezieren und CPD zeigt detaillierte Strukturen der Region (IIb) in Abbildung 1 und Abbildung 2. Diese sind als dick aufsteigende Gliedmaßen der Schleife von Henle bekannt. (b) Projektionssegment mit minimaler Intensität, abgeleitet aus demselben NanoCT-Datensatz, der in (a) mit einer virtuellen Scheibendicke von ca. 7 m dargestellt ist, was eine klare Visualisierung der Zellkerne ermöglicht. (c) Repräsentativer histologischer Abschnitt mit dicken aufsteigenden Gliedmaßen der Henle-Schleife mit klarer Visualisierung von Zellkernen und Pinselrand. Der histologische Abschnitt hat eine ungefähre Dicke von 7 m und wurde aus der gleichen Mausnierenprobe nach der aufgetragenen Eosin-basierten Färbung und Einbettung in einen Paraffinblock gewonnen. (d) Repräsentativer histologischer Abschnitt mit Anwendung von Gegenfleck Hämatoxylin, der die Zellkerne in Violett hervorhebt. Vorbereitung des histologischen Abschnitts in der Nähe des in (c) dargestellten Abschnitts mit einer ungefähren Dicke von 7 m. Diese Zahl wurde von Busse und Müller et al.26geändert. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 4. CT-Slice- und Volumen-Rendering von NanoCT-Daten. (a) Das nanoCT-Bild der gleichen Maus-Nierenprobe, das die Strukturen zeigt, die als dick aufsteigende Gliedmaßen der Schleife von Henle bekannt sind. Dies ist eine detaillierte Ansicht der Region (IIb) in Abbildung 1 und Abbildung 2, die von einem kleinen Stück der Niere mit einer Voxelgröße von etwa 400 nm gewonnen wurde. Die Vorbereitung der Probe umfasste Färbung, Sezieren und CPD. (b) Volumenwiedergabe von NanoCT-Daten, die die 3D-Struktur dick aufsteigender Gliedmaßen der Henle-Schleifen visualisieren. Diese Zahl wurde von Busse und Müller et al.26geändert. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Diskussion
Derzeit wird Eosin als standard histologisches Protokoll verwendet, um das Zellzytoplasma zu kennzeichnen. Das Färbemittel wird als 0,1% (w/v) wässrige Lösung auf mikroskopische Weichgewebescheiben aufgetragen (in der Regel mit einer Dicke von 2-10 m geschnitten)33. Die Anwendung dieses standardisierten histologischen Protokolls auf 3D-Gewebeproben wie eine ganze Mausniere führt nicht zu einem Dämpfungskontrast verbessertes CT-Bild. Einerseits ist dies auf die geringen intrinsischen Dämpfungseigenschaften von Weichgewebe für typischerweise verwendete Röntgenenergien von laborbasierten MikroCT-Systemen zurückzuführen. In der Regel besteht Weichgewebe hauptsächlich aus Kohlenstoff, Wasserstoff, Sauerstoff und Stickstoff34und führt daher nicht zu einer Kontrastverbesserung. Auf der anderen Seite war die geringe Konzentration von Eosin für die Färbung der begrenzende Faktor. Obwohl ein Eosinmolekül vier Bromidatome (hohes Atomelement Brom mit Z = 3534) enthält, wurden die für die Röntgen-CT-Bildgebung erforderlichen Empfindlichkeitsniveaus nicht erreicht.
Um diese Herausforderung des niedrigen Dämpfungskontrasts zu bewältigen, wurden mehrere Konzentrationen von Eosin untersucht. Eine Einschränkung ist hier die maximale Löslichkeit von Eosin in Wasser, die 30% (w/v) in einer wässrigen Lösung beträgt. Die beste Dämpfungskontrastverbesserung innerhalb des Weichgewebes wurde mit der höchsten Eosinkonzentration beobachtet, die nach dem Lambert-Biergesetz erwartet wurde. Daher wurde das endgültige Färbeprotokoll mit der höchsten Konzentration durchgeführt.
Die Frage, wie das Weichgewebe auf molekularer Ebene optimal auf das Färbeverfahren vorbereitet werden kann, um die Kontrastverbesserung weiter zu verbessern, wurde durch pH-Anpassung beantwortet. Hierbei erwies sich die Versauerung der Weichgewebeprobe während der Fixierung oder vor der Färbung als entscheidend. Dies zeigte auch Hong et al.35. Die höhere Ansammlung von Färbemitteln innerhalb des Zellzytoplasmas durch die Säure wurde durch verbesserte ionische Wechselwirkungen erreicht, die ein Ergebnis der Protonierung von Aminosäureseitenketten von Proteinen und Peptiden im Zellzytoplasma waren. Abbildung 1a,bzeigt ein repräsentatives Ergebnis, das die Kontrastverbesserung im Vergleich zu einer ungefärbten Weichgewebeprobe hervorhebt. Hier wurde ein struktureller Überblick über eine ganze Mausniere erreicht, die entscheidende anatomische Regionen wie Kortex, Medulla, Papille und Nierenbecken visualisiert.
Das vorgestellte Färbeprotokoll ist einfach anzuwenden und enthält nur drei Schritte. Die erforderlichen Reagenzien sind leicht zugänglich. Die Gesamtfärbezeit von 24 Stunden ist schnell für eine Vollorganfärbung, die die 3D-Visualisierung von Weichgewebeproben (Abbildung 1c, Abbildung 2b und Abbildung 4b) in einer Laborumgebung unter mehrere skalieren auf zelluläre Ebene. Es sei darauf hingewiesen, dass die gesamte Färbezeit und das Volumen der benötigten Färbelösung je nach Art der Probe einige Anpassungen verlangen können. Dennoch eignet sich das eosinbasierte Färbeprotokoll für die Vollorganfärbung, die dann eine hochauflösende MikroCT-Bildgebung ganzer Organe ermöglicht. Schrumpfartefakte aufgrund des Lösungsmittels Ethanol, das verwendet wurde, um die Probe während der MikroCT-Messungen feucht zu halten, wurden nicht beobachtet. Für die NanoCT-Bildgebung sind zusätzliche Präparationsschritte erforderlich, die die Untersuchung kleinerer Gewebeteile aus der Originalprobe ermöglichen. Im Hinblick auf zukünftige histopathologische Anwendungen liefern die Übersichtsscans wertvolle Einblicke in veränderte anatomische Regionen und Strukturen, die die Bestimmung von ROIs ermöglichen, wie in Abbildung 2adargestellt. Diese können in 3D nach mikroCT (Abbildung 1c und Abbildung 2b) oder nanoCT (Abbildung 4b) untersucht und in 2D mit Histologie ausgewertet werden ( Abbildung3).
Eine weitere Stärke des Protokolls liegt in der vollständigen Kompatibilität mit der Histopathologie in Bezug auf das H&E-Färbeverfahren. Die Anwendung des eosinbasierten Färbeverfahrens auf Massenproben behindert weitere histologische Untersuchungen nicht (Abbildung 3), obwohl die angewandte Eosinkonzentration im Vergleich zur histologischen Färbelösung viel höher ist. Die nanoCT-Scheibe mit einer virtuellen Dicke von ca. 400 nm (Abbildung 3a) vergleicht sich bereits sehr gut mit dem histologischen Abschnitt (Abbildung 3c), der aus der entsprechenden Weichgewebeprobe abgeleitet wurde. Unter Berücksichtigung der ungefähren Dicke eines histologischen Abschnitts mit 7-10 m ermöglicht die Erzeugung von Projektionsscheiben mit minimaler Intensität der NanoCT-Daten (Abbildung 3b), die einer virtuellen Dicke von ca. 7 m entsprechen, eine besserer Vergleich mit dem histologischen Abschnitt (Abbildung 3c). Hierbei werden die Zellkerne eindeutig als Nichtdämpfungsbereich offenbart, da Eosin protein- und peptiden im Zellzytoplasma33spezifisch färbt.
Die Anwendung weiterer Gegenfärbung mit standard histologischen Methoden ist möglich, obwohl die Reihenfolge der Färbung im Vergleich zum standard histologischen Färbungsverfahren umgekehrt wurde. Beginnend mit dem entwickelten eosinbasierten Färbeprotokoll für CT, gefolgt von der Gegenfärbung dieser eosinbasierten histologischen Abschnitte mit Hämatoxylin, ermöglicht eine vollständige Kompatibilität und führt zu einer hochwertigen Färbung, die die erwartete Form anzeigt. des Aussehens. Die Zellkerne-spezifische Färbung mit Mayers saurem Hämatoxylin wurde auf den histologischen Abschnitt aufgetragen, der die Zellkerne in Violett hervorhebt (Abbildung 3d). Die Anwendung der histologischen Gegenfärbung ist derzeit auf den H-Fleck beschränkt. Andere histologische Gegenfärbungen wie periodische Säure SchiffsBasis, Elastica van Gieson oder Gomori Silber müssen bewertet und die Kompatibilität mit immunhistologischen Techniken getestet werden.
Das eosinbasierte Färbeprotokoll ermöglicht (i) zellzytoplasmaspezifisches Targeting, (ii) homogene und vollständige Färbung, (iii) einfache Umsetzung, (iv) schnelles Eindringen des Gewebes ohne Entstehung von Artefakten wie Diffusionsringen, (v) die Färbung große und dichte Weichgewebeproben und (vi) volle Kompatibilität mit der Histopathologie in Bezug auf den H&E-Fleck. Diese Anforderungen sind wichtig, um eine hochauflösende Röntgen-CT-Visualisierung von Weichgewebe bis auf zelluläre Ebene zu ermöglichen. In Kombination mit den kürzlich entwickelten nanoCT-Geräten12,36,37, zerstörungsfreie Erzeugung von virtuellen histologischen Slices, die im Kontrast und Auflösung mit herkömmlichen histologischen Daten vergleichbar sind möglich gemacht wird. Dieser kombinierte Ansatz wird die Etablierung von Röntgen-CT als wertvolles Werkzeug für die 3D-Visualisierung mikroskopischer Gewebestrukturen ermöglichen.
Offenlegungen
Die Autoren haben nichts zu verraten.
Danksagungen
Wir danken Dr. Enken Drecoll für die histologischen Diskussionen und dem äußerst hilfsbereiten Team von Excillum AB, Schweden. Wir würdigen die finanzielle Unterstützung durch das DFG-Exzellenzcluster München Center for Advanced Photonics (MAP) und das DFG Gottfried Wilhelm Leibniz Programm. Darüber hinaus wurde dieses Forschungsprojekt aus dem Forschungs- und Innovationsprogramm Union Horizont 2020 im Rahmen des Marie-Skodowska-Curie-Zuschussabkommens Nr. H2020-MSCA-IF-2015-703745-CONSALT.
Materialien
| Name | Company | Catalog Number | Comments |
| 50-ml centrifuge tube by Falcon | VWR | 734-0453 | |
| Formaldehyde solution, 37% | Carl Roth | CP10.2 | acid-free, stabilized with ~10% MeOH |
| Glacial acetic acid | Alfa Aesar | 36289.AP | |
| Eosin Y disodium salt | Sigma-Aldrich | E4382 | certified by Biological Stain Commission |
| Phosphate Buffered Saline (PBS) | Merck | L1825 | Dulbecco's formualtion, w/o calcium and magnesium |
| Sample Tubes by Nalgene | Carl Roth | ATK5.1 | |
| Rocking Shaker ST5 | CAT | 60281-0000 | |
| Cellulose tissue paper | VWR | 115-0600 | |
| Forceps, by USBECK Laborgeräte | VWR | 232-0096 | |
| Microcentrifuge tubes by Eppendorf | VWR | 211-2120 | safe-lock, 2.0 ml |
| Ethanol absolute by Baker Analyzed | VWR | 80252500 | |
| Disposable safety scalpel by Aesculap | VWR | AESCBA210 | |
| Petri dish by Sterilin | VWR | 391-2019 | |
| Plastic pasteur pipette | Carl Roth | EA68.1 | graduated, 1 ml |
| Desiccator by Duran | VWR | SCOT247826954 | |
| Silicone grease by Bayer | Sigma-Aldrich | 85404 | high-vacuum |
| Carbon dioxide cylinder with standpipe | Linde | 3700113 | 10 kg, short |
| micro-porous treatment capsule | PLANO GmbH | 4614 | pore size 78 µm (B) |
| Bal-Tec CPD 030 | Bal-Tec AG | CO2 as drying agent | |
| Stemi 2000-C stereomicroscope with KL 1500 LCD | Zeiss | this stereomicroscope has been updated(1) | |
| Zeiss Xradia Versa 500 | Zeiss | this microCT scanner has been updated(2) | |
| Avizo Fire 8.1 | Thermo Fisher Scientific | ||
| PILATUS detector as part of the nanoCT scanner | Dectris | single-photon counting detector(4,5); there are commercially availble nanoCT systems available (6,7) | |
| nanofocus X-ray source as part of the nanoCT scanner | Excillum | high-flux nanofocus X-ray transmission tube(3); there are commercially availble nanoCT systems available(6,7) | |
| (1) Germany, Z. ZEISS product information: ZEISS stereomicroscopes https://www.micro-shop.zeiss.com/de/de/system/Stereomikroskope/1006> (September 06, 2019). | |||
| (2) Germany, Z. ZEISS product information: ZEISS Xradia 510 Versa https://www.zeiss.com/microscopy/int/products/x-ray-microscopy/zeiss-xradia-510-versa.html> (April 10, 2019). | |||
| (3) Nachtrab, F. et al. Development of a Timepix based detector for the NanoXCT project. Journal of Instrumentation 10 (11), C11009, (2015). | |||
| (4) Kraft, P. et al. Performance of single-photon-counting PILATUS detector modules. Journal of Synchrotron Radiation 16 (3), 368-375, (2009). | |||
| (5) Kraft, P. et al. Characterization and calibration of PILATUS detectors. IEEE Transactions on Nuclear Science 56 (3), 758-764, (2009). | |||
| (6) Germany, Z. ZEISS product information: ZEISS Xradia 810 Ultra https://www.zeiss.com/microscopy/int/products/x-ray-microscopy/xradia-810-ultra.html> (April 9 2019). | |||
| (7) Company, G. E. GE product information: Phoenix nanotom m, https://www.gemeasurement.com/sites/gemc.dev/files/geit-31344en_nanotom_m_0517.pdf> (April 10, 2019). |
Referenzen
- Suvarna, S. K., Layton, C., Bancroft, J. D. . Theory and Practice of Histological Techniques. 7th edn. , (2013).
- Chatterjee, S. Artefacts in histopathology. Journal of Oral and Maxillofacial Pathology. 18 (4), 111-116 (2014).
- McInnes, E. Artefacts in histopathology. Comparative Clinical Pathology. 13 (3), 100-108 (2005).
- Andreasen, A., Drewes, A., Assentoft, J., Larsen, N. Computer-assisted alignment of standard serial sections without use of artificial land-marks. A practical approach to the utilization of incomplete information in 3-d reconstruction of the hippocampal region. Journal of Neuroscience Methods. 45 (3), 199-207 (1992).
- Braverman, M. S., Braverman, I. M. Three-dimensional reconstructions of objects from serial sections using a microcomputer graphics system. Journal of Investigative Dermatology. 86 (3), 290-294 (1986).
- Denk, W., Hortsmann, H. Serial block-face scanning electron microscopy to reconstruct three-dimensional tissue nanostructure. PLoS Biology. 2 (11), 329 (2004).
- Mohun, T. J., Weninger, J. W. Imaging heart development using high-resolution episcopic microscopy. Current Opinion in Genetics and Development. 21, 573-578 (2011).
- Weninger, J. W., Meng, S., Streicher, J., Müller, G. B. A new episcopic method for rapid 3-d reconstruction: applications in anatomy and embryology. Anatomy and Embryology. 197 (5), 341-348 (1998).
- Odgaard, A., Andersen, K., Melsen, F., Gundersen, H. J. G. A Direct Method for Fast 3-Dimensional Serial Reconstruction. Journal of Microscopy. 159, 335-342 (1990).
- Müller, M., et al. Myoanatomy of the velevt worm leg revealed by labratory-based nanofocus X-ray source tomography. Proceedings of the National Academy of Sciences of the United States of America. 114 (47), 12378-12383 (2017).
- Salomon, M., Hanke, R., Krüger, P., Uhlmann, N., Voland, V. Realization of a computed tomography setup to achieve resolutions below 1 µm. Nuclear Instruments and Methods in Physics Research Section A. 591, 50-53 (2008).
- Tkachuk, A., et al. X-ray computed tomography in Zernike phase contrast mode at 8 keV with 50-nm resolution using Cu rotating anode X-ray source. Zeitschrift für Kristallographie. 222, 650-655 (2007).
- Withers, P. J. X-ray nanotomography. Materials Today. 10, 26-34 (2007).
- Jahn, H., et al. Evaluation of contrasting techniques for X-ray imaging of velvet worms (Onychophora). Journal of Microscopy. 270 (3), 343-358 (2018).
- Martins de S. e Silva, J., et al. Three-dimensional non-destructive soft-tissue visualization with X-ray staining micro-tomography. Scientific Reports. 5, 14088 (2015).
- Metscher, B. D. MicroCT for Developmental Biology: A Versatile Tool for High-Contrast 3D Imaging at Histological Resolutions. Developmental Dynamics. 238, 632-640 (2009).
- Metscher, B. D. MicroCT for comparative morphology: simple staining methods allow high-contrast 3D imaging of diverse non-mineralized animal tissues. BMC Physiology. 9, 11 (2009).
- Mitzutani, R., et al. X-Ray Microtomographic Imaging of Three-Dimensional Structure of Soft Tissues. Tissue Engineering Part C: Methods. 14 (4), 359-363 (2008).
- Pauwels, E., Loo, D. v., Cornillie, P., Brabant, L., Hoorebeke, L. v. An exploratory study of contrast agents for soft tissue visualization by means of high resolution X-ray computed tomography imaging. Journal of Microscopy. 250, 21-31 (2013).
- Degenhardt, K., Wright, A. C., Horng, D., Padmanabhan, A., Epstein, J. A. Rapid 3D phenotyping of cardiovascular development in mouse embryos by micro-CT with iodine staining. Circulation: Cardiovascular Imaging. 3 (3), 314-322 (2010).
- Dullin, C., et al. μCT of ex-vivo stained mouse hearts and embryos enables a precise match between 3D virtual histology, classical histology and immunochemistry. PLOS ONE. 12 (2), 0170597 (2017).
- Jeffrey, N. S., Stephenson, R. S., Gallagher, J. A., Cox, P. Micro-computed tomography with iodine staining resolves the arrangement of muscle fibres. Journal of Biomechanics. 44, 189-192 (2011).
- Johnson, J. T., et al. Virtual Histology of Transgenic Mouse Embryos for High-Throughput Phenotyping. PLOS Genetics. 2, 61 (2006).
- Leszczyński, B., et al. Visualization and Quantitative 3D Analysis of Intraocular Melanoma and Its Vascularization in a Hamster Eye. International Journal of Molecular Sciences. 19 (2), 332 (2018).
- Mizutani, R., Suzuki, Y. X-ray microtomography in biology. Mircon. 43, 104-115 (2012).
- Busse, M., et al. Three-dimensional virtual histology enabled through cytoplasm-specific X-ray stain for microscopic and nanoscopic computed tomography. Proceedings of the National Academy of Sciences of the United States of America. 115 (10), 2293-2298 (2018).
- Müller, M., et al. Non-destructive high-resolution 3D virtual histology enabled through a cell nucleus-specific stain for X-ray computed tomography. Scientific Reports. 8, 17855 (2018).
- . ZEISS product information: ZEISS Xradia 510 Versa Available from: https://www.zeiss.com/microscopy/int/products/x-ray-microscopy/zeiss-xradia-510-versa.html (2019)
- Nachtrab, F., et al. Development of a Timepix based detector for the NanoXCT project. Journal of Instrumentation. 10 (11), 11009 (2015).
- Kraft, P., et al. Performance of single-photon-counting PILATUS detector modules. Journal of Synchrotron Radiation. 16 (3), 368-375 (2009).
- Kraft, P., et al. Characterization and calibration of PILATUS detectors. IEEE Transactions on Nuclear Science. 56 (3), 758-764 (2009).
- . ZEISS product information: ZEISS Xradia 810 Ultra Available from: https://www.zeiss.com/microscopy/int/products/x-ray-microscopy/zeiss-xradia-510-versa.html (2019)
- Anderson, T. F. Techniques for the preservation of three-dimensional structure in preparing specimens for the electron microscope. Transactions of the New York Academy of Sciences. 13 (4), 130-134 (1951).
- Bray, D., Williams, J. R., Clifford, A. A. . Supercritical Fluid Methods and Protocols. Methods in Biotechnology. 13, 235-243 (2000).
- Lucy, L. B. An iterative technique for the rectification of observed distributions. The Astronomical Journal. 79, 745-765 (1974).
- Richardson, W. H. Bayesian-Based Iterative Method of Image Restoration. The Journal of the Optical Society of America. 62 (1), 55-59 (1972).
- Paganin, F., Mayo, S. C., Gureyev, T. E., Miller, P. R., Wilkins, S. W. Simultaneous phase and amplitude extraction from a single defocused image of a homogeneous object. Journal of Microscopy. 206, 33-40 (2002).
- Riedelsheimer, B., Büchl-Zimmermann, S., Mulisch, M., Welsch, U. . Mikroskopische Technik. , 193-194 (2015).
- Hubbell, J. H., Seltzer, S. M. Tables of Xray mass attenuation coefficients and mass energy-absorption coefficients from 1 keV to 92 keV and 48 additional substances of dosimetric interest, Table 3. National Institute of Standards and Technology. , (1995).
- Hong, H. Y., Yoo, G. S., Choi, J. K. An Eosin Y Method for Protein Determination in Solution. Analytical Letters. 32 (12), 2427-2442 (1999).
- . GE product information: Phoenix nanotom m Available from: https://www.gemeasurement.com/sites/gemc.dev/files/geit-31344en_nanotom_m_0517.pdf (2019)
- Dierick, M., et al. Recent Micro-CT Scanner Developments at UGCT. Nuclear Instruments and Methods in Physics Research Section B. 324, 35-40 (2014).
- Kastner, J., Plank, B., Heinzl, C. Advanced X computed tomography methods: High resolution CT, quantitative CT, 4DCT and phase contrast CT. Proceedings of Digital Industrial Radiology and Computed Tomography. , 120-132 (2015).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten