Method Article
Imagerie 3D d'échantillons de tissus mous à l'aide d'une méthode de coloration spécifique aux rayons X et de tomographie nanoscopique par calcul
* Ces auteurs ont contribué à parts égales
Dans cet article
Résumé
Un protocole pour la visualisation 3D des structures microscopiques de tissu en utilisant une méthode spécifique de coloration de rayon X conçue pour la tomographie calculée de rayon X est présenté.
Résumé
Nous démontrons une méthode basée en laboratoire combinant le microCT de rayon X et le nanoCT avec une tache spécifique de rayon X, qui cible le cytoplasme cellulaire. Le protocole décrit est facile à appliquer, rapide et adapté pour les échantillons de tissus mous plus grands. La méthodologie présentée permet la caractérisation des structures tissulaires cruciales en trois dimensions et est démontrée sur un rein entier de souris. L'approche multi-échelle permet d'imager l'ensemble du rein de la souris et soutient la sélection d'autres volumes d'intérêt, qui sont acquis avec des résolutions plus élevées allant dans la gamme nanométrique. Par conséquent, la morphologie des tissus mous avec un niveau de détail similaire à celui des images de microscopie de lumière histologique correspondante est reproduite. Des aperçus plus profonds dans la configuration 3D des structures de tissu sont réalisés sans empêcher d'autres investigations par des méthodes histologiques.
Introduction
La caractérisation complète des spécimens de tissus mous nécessite des informations sur la microstructure des tissus 3D. L'étalon-or actuel pour les analyses d'échantillons de tissus mous est l'histopathologie. Le tissu et la morphologie cellulaire du spécimen sont explorés en 2D dans des régions d'intérêt sélectionnées (ROI) à l'aide de la microscopie optique1. Cette méthode, cependant, a plusieurs inconvénients. La préparation de l'échantillon est longue, compliquée, destructrice et sujette aux artefacts. Les diapositives microscopiques produites ne fournissent que des informations 2D parallèles au plan de sectionnement. Souvent, le nombre de sections histologiques qui sont étudiées, est limité en raison de contraintes de temps2,3.
Ces dernières années, le domaine de l'histologie 3D a évolué. Ici, des tranches de tissu virtuelles de n'importe quel plan spatial désiré sont accessibles. Cela permet le suivi des structures dans l'ensemble de l'échantillon, ce qui conduit à une meilleure compréhension de l'architecture des tissus 3D et des changements structurels associés à différentes pathologies. Diverses méthodes ont été développées pour atteindre la génération de données de volume 3D. Elles vont d'approches basées sur la section série, qui utilisent la microscopie de lumière ou d'électrons4,5,6,7,8, aux méthodes d'imagerie par blocs, telles que l'imagerie 3D épiscopique ou microscopie électronique à balayage de bloc-face7,8,9. Toutes les méthodes mentionnées, cependant, impliquent soit la section ou la destruction complète de l'échantillon, ce qui ne permet pas d'enquêtes plus approfondies. La résolution obtenue dépend fortement du processus de sectionnement étant sujet aux artefacts tels que décrits dans l'histologie conventionnelle. Ces méthodes souffrent aussi d'artefacts d'alignement.
Les techniques d'imagerie par rayons X 3D telles que la tomographie calculée microscopique et nanoscopique (microCT et nanoCT) aspirent à générer des données 3D à haute résolution sans destruction de l'échantillon tissulaire. Jusqu'à présent, le faible contraste d'atténuation des rayons X des tissus mous et l'accès limité aux hautes résolutions dans un environnement de laboratoire ont nui à leur utilisation pour la visualisation 3D des structures tissulaires microscopiques. Les progrès récents vers la CT à rayons X haute résolution en laboratoire permettent des résolutions bien en dessous de 1 m10,11,12,13.
L'absence de contraste dans les tissus mous dans l'imagerie conventionnelle à base d'atténuation des rayons X est compensée par des agents de coloration, qui améliorent le contraste d'atténuation des rayons X. Les agents de coloration connus d'autres techniques d'imagerie telles que le tétroxide d'osmium (OsO4), l'iodure de potassium d'iode (IKI) ou l'acide phosphotungstic (PTA) sont souvent utilisés14,15,16,17, 18,19,20,21,22,23,24,25. Agents de coloration qui permettent (i) un ciblage biologique spécifique, (ii) une coloration homogène et complète, (iii) une manipulation facile, (iv) une pénétration rapide du tissu sans créer d'artefacts tels que des anneaux de diffusion, (v) des taches tissulaires grandes et denses, et (vi) la compatibilité complète avec l'histopathologie sont exigées pour établir le CT de rayon X comme outil pour la visualisation 3D des structures microscopic de tissu. Dans ce travail, nous montrons comment les échantillons de tissus mous sont préparés pour l'imagerie par rayons X Avec une tache de rayons X spécifique au cytoplasme basée sur l'éosine qui répond aux exigences énoncées ci-dessus26.
L'approche d'imagerie à plusieurs échelles assure l'évaluation de la qualité des taches au moyen d'une mesure de microCT d'aperçu et de la sélection des volumes d'intérêt (VOI) pour d'autres enquêtes à haute résolution. La qualité de coloration est analysée en se concentrant sur les paramètres de coloration tels que (i) l'exhaustivité, (ii) l'apparence des anneaux de diffusion, (iii) l'amélioration de contraste, (iv) l'aspect des artefacts de CT tels que des stries et (v) homogénéité. La configuration nanoCT en laboratoire, qui utilise le grossissement géométrique pour atteindre des résolutions jusqu'à 100 nm, visualise la morphologie des tissus mous sur le niveau (sous)-cellulaire10,27. Une analyse comparative des tranches nanoCT avec des images de microscopie de lumière histologique correspondanteconfirme la reproduction de l'architecture tissulaire avec des détails similaires sur un niveau microscopique en 2D, permettant la caractérisation histopathologique du tissu échantillon. Ce protocole vidéo détaillé vise à sensibiliser et à mettre en évidence le potentiel de cette méthodologie en tant qu'outil d'imagerie des tissus mous 3D non destructif s'intéressant à une vaste communauté scientifique comme les zoologistes, les biologistes et la santé. Professionnels.
Protocole
Attention : Veuillez consulter toutes les fiches de données pertinentes sur la sécurité des matériaux (SDM) avant d'être utilisées. Plusieurs des produits chimiques utilisés dans le protocole sont extrêmement toxiques et cancérigènes. Veuillez utiliser toutes les pratiques de sécurité appropriées lors de l'exécution du protocole de coloration, y compris l'utilisation de commandes techniques (capuchon de fumée, boîte à gants) et d'équipement de protection individuelle (lunettes de sécurité, gants, blouse de laboratoire, pantalon sain à pleine longueur, chaussures à bouts fermés).
Animaux utilisés:
Le logement des animaux a été réalisé au Klinikum rechts der Isar, Université technique de Munich, conformément aux directives de l'Union européenne 2010/63. L'enlèvement des organes a été approuvé par un comité interne de protection des animaux de Klinikum rechts der Isar, Munich, Allemagne (numéro de référence interne 4-005-09). Toutes les procédures étaient conformes aux lignes directrices et règlements pertinents. Tous les laboratoires sont inspectés conformément aux principes de bonne pratique de l'OCDE.
1. Protocole de coloration Eosin
- Pour fixer des échantillons de tissus mous, remplissez un tube de centrifugeuse de 50 mL d'une solution fixative contenant 9,5 ml de solution de formaldéhyde (FA) de 4 % (v/v) et 0,5 mL d'acide acétique glaciaire (AA).
REMARQUE: Préparer la solution FA fraîchement à partir d'une solution FA sans acide de 37% stabilisée avec environ 10% de méthanol. Diluer davantage la solution FA avec la solution tamponnée de phosphate (DPBS) de Dulbecco. Choisissez DPBS sans calcium et magnésium. Gardez la solution FA diluée pas plus d'un mois. Pendant l'acidification, le pH de la solution fixative passe de neutre à environ 3.
CAUTION: Parce que FA est un organe aigu toxique, corrosif et cancérigène, l'utilisation d'un capot de fumée est obligatoire et l'équipement personnel de protection approprié doit être utilisé.- Ajouter l'échantillon de tissus mous fraîchement enlevé dans un tube de centrifugeuse de 50 ml et réfrigérer le tube de centrifugeuse de 50 ml pendant 24 à 72 h.
REMARQUE: Le protocole peut être mis en pause ici. - Laver l'échantillon de tissus mous avec la solution DPBS pendant 1h.
- Ajouter l'échantillon de tissus mous fraîchement enlevé dans un tube de centrifugeuse de 50 ml et réfrigérer le tube de centrifugeuse de 50 ml pendant 24 à 72 h.
- Pour tacher l'échantillon de tissus mous fixes (p. ex., un rein entier de souris), placez le tissu mou dans 2 ml de solution de coloration Eosin Y et incubez l'échantillon pendant 24 h. Gardez l'échantillon sur une plaque horizontale secouant pour un basculement lisse (environ 60 tr/min) pendant l'incubation processus.
REMARQUE : La solution de coloration Y d'éosine a une concentration de 30% (w/v) dans l'eau distillée. Choisissez le volume de la solution de coloration de manière à ce que l'échantillon soit entièrement couvert par la solution de coloration et permettez à l'échantillon de se déplacer librement dans le récipient de l'échantillon. Le temps d'incubation peut différer pour d'autres échantillons et doit être ajusté en conséquence. - Après la coloration, retirer soigneusement l'échantillon de tissus mous du contenant de l'échantillon.
- Enlever soigneusement l'excès d'agent de coloration avec du papier de tissu de cellulose.
- Placez l'échantillon de tissus mous dans un contenant d'échantillon conique au-dessus d'une phase de vapeur d'éthanol pour le stockage et l'utilisation ultérieure.
REMARQUE : Le contenant d'échantillon conique doit toujours contenir quelques gouttes d'éthanol de 70 % (v/v) au fond du tube pour garder l'échantillon de tissus mous humide et prévenir les artefacts.
2. Imagerie microCT à rayons X
REMARQUE : Les mesures de microCT à rayons X ont été effectuées à l'effile microCT, qui offre des mesures de CT (la capacité d'imager l'ensemble de l'échantillon dans le champ de vision (FOV)) et la performance des mesures de CT à haute résolution (la capacité de se concentrer dans sur un volume d'intérêt souhaité (VOI) du même échantillon) jusqu'à 1 m.
- Montez l'échantillon de tissus mous à un porte-échantillon approprié. Assurer un ajustement serré de l'échantillon sur le support de l'échantillon afin d'empêcher l'échantillon de se déplacer pendant les mesures de cT aux rayons X.
- Dans le cas du rein de souris taché : Préparer un support d'échantillon avec deux tubes de centrifugeuse, par lequel le fond d'un tube est coupé. Coller les deux tubes de centrifugeuse ensemble à l'aide d'adhésif à deux composants. Assurer un alignement droit des tubes de centrifugeuse autour de l'axe de rotation. Attendez que l'adhésif durcisse.
- Une fois que le porte-échantillon est prêt à l'emploi, transférer le rein de la souris dans le tube de centrifugeuse intact, qui contient quelques gouttes d'éthanol de 70 % (v/v) au fond du tube.
REMARQUE : La stabilité de l'échantillon est cruciale. Prenez le temps de préparer l'échantillon pour les mesures de la toctologie aux rayons X. L'échantillon de tissus mous est conservé au cours d'une phase de vapeur d'éthanol pour garder l'échantillon humide pendant les mesures de cT de rayons X et empêcher l'échantillon de tissu mou du rétrécissement et d'autres artefacts. L'échantillon de tissus mous ne doit pas être en contact avec le solvant pour éviter l'accumulation du solvant autour de l'échantillon pendant la mesure de la tocnotation aux rayons X, ce qui pourrait entraîner un mouvement de l'échantillon pendant la mesure ou causer des problèmes pendant la reconstruction. Si le titulaire de l'échantillon ne permet pas de tenir le solvant au fond, un papier de cellulose humidifié avec 70 % (v/v) d'éthanol peut être placé dans le support de l'échantillon. Il convient de noter que les artefacts de rétrécissement dus à l'éthanol solvant n'ont pas été observés.
REMARQUE: Le protocole peut être mis en pause ici.
- Après un alignement attentif de l'échantillon, choisissez les paramètres d'acquisition pour une meilleure qualité d'image. Dans le cas des données microCT présentées, acquérir l'analyse à une tension de pointe de 50 kV, un courant de 3,5 W à l'aide de 1601 projections réparties également sur 360 degrés.
REMARQUE : Les paramètres d'acquisition de l'analyse CT de vue d'ensemble ont été choisis pour la meilleure qualité d'image. En tant que tel, l'objectif de la caméra 0.39x a été choisi pour couvrir l'ensemble de l'échantillon dans le champ de vision (FOV). Il en est résulté une taille de pixel efficace de 12 m. Le temps d'exposition de 2 s par projection a fourni un bon rapport signal/bruit. Le retour sur investissement pour la tomodensitome haute résolution a été identifié à l'aide des données microCT de l'analyse de la vue d'ensemble. Les scanners MicroCT intègrent souvent un outil logiciel intégré, qui permet de sélectionner avec précision le retour sur investissement déterminé. Pour les données De NtC haute résolution, l'objectif de la caméra 4x a été choisi, ce qui a donné une taille de pixels efficace de 3,3 m. Ici, un temps d'exposition de 15 s par projection était nécessaire.
REMARQUE: Le protocole peut être mis en pause ici. - Après l'acquisition des données de CT à rayons X, traiter les projections en conséquence pour la reconstruction du volume 3D. Dans le cas des données microCT présentées : Reconstruire les données de CT de rayon X avec le logiciel intégré.
REMARQUE : Les rendus de volume des données microCT indiquées dans la figure 1 et la figure 2 ont été générés à l'aide d'un logiciel de visualisation.
REMARQUE: Le protocole peut être mis en pause ici.
3. Imagerie nanoCT à rayons X
REMARQUE : Le scanner nanoCT à rayons X a été développé en interne. L'instrument sans lentille est équipé d'une source de rayons X nanofocus et d'un détecteur de comptage monophoton. Les données 3D avec des résolutions jusqu'à 100 nm peuvent être générées10. En général, les systèmes nanoCT, y compris ceux qui sont à rayons X optiques, sont disponibles dans le commerce et ne se limitent pas au scanner nanoCT décrit.
- Préparation de l'échantillon NanoCT
- Préparer les VOI de l'échantillon de tissus mous. Couper les tissus mous en très petits morceaux d'environ 0,5 mm de longueur de bord à l'aide d'un scalpel et d'un stéréomicroscope. Dans le cas du rein de la souris: Couper le rein de la souris en deux moitiés le long de l'axe le plus long. Prenez la moitié du rein de la souris et préparez différentes régions anatomiques telles que le cortex rénal et la médulle rénale.
REMARQUE : L'autre moitié du rein de la souris a été transférée à l'histopathologie, où l'échantillon a été incorporé dans la paraffine et traité en conséquence pour produire les sections histologiques typiques comme on le voit dans la figure 3c et la figure 3d . - Transférer les petits morceaux avant la première étape de déshydratation dans un nouveau plat Petri, où ils restent pour toutes les étapes suivantes.
- Déshydrater les échantillons à l'aide de concentrations (toutes v/v) en % : 50, 60, 70, 80, 90, 96 et 100 éthanol équilibré avec de l'eau distillée. Effectuez chaque étape de déshydratation pendant 1 h chacune.
REMARQUE: Le protocole peut être mis en pause ici. Gardez les petits morceaux de tissu dans 100% d'éthanol pendant la nuit. - Point critique sec (CPD) les petits morceaux de tissu.
REMARQUE : L'application de la DPC permet la déshydratation complète de l'échantillon de tissu en échangeant le solvant (ici l'éthanol) avec l'agent de séchage (ici CO2). Cela a été nécessaire pour s'assurer que l'échantillon peut être monté sur les supports d'échantillon de la nanoCT, ne se déplace pas pendant la mesure et peut être positionné très près de la source de rayons X pour permettre un meilleur grossissement géométrique. La configuration nanoCT est basée sur un simple grossissement géométrique, le facteur de grossissement étant défini comme la distance source-détecteur sur la distance source-échantillon. La technique de séchage a d'abord été introduite par Anderson pour préserver la structure 3D des spécimens biologiques pour la microscopie électronique28. Un aperçu de la technique est fourni par Bray29.- Préremplir la chambre à vide avec 100% d'éthanol. Transférer les petits morceaux de tissu dans une capsule micro-poreuse et les placer dans la chambre à vide du CPD. Fermer le système.
REMARQUE : Étant donné que la haute pression est impliquée dans le processus de DPC, assurez-vous que toutes les parties de la DPC, en particulier les raccords, sont intactes et que le système est correctement fermé. - Refroidir la chambre à 6- 8oC et remplir avec du LIQUIDE CO2.
- En remuant, attendre 3 min pour permettre un bon mélange des deux composants. Égoutter soigneusement la chambre. Assurez-vous que le porte-échantillon est toujours recouvert de solvant. Répétez cette étape dix fois pour permettre le remplacement complet de l'éthanol par du CO2 dans l'échantillon.
- Après le remplissage final de la chambre avec du CO2,chauffer la machine au point critique du CO2 (31 oC et 73,8 bar) suivie d'une libération très lente du CO2 gazeux sur une période de 30 min.
REMARQUE : Le rejet du gaz doit être entrepris très lentement, car l'eau condensée peut se former sur l'échantillon. Assurez-vous que la température ne descend pas en dessous du point critique du CO2. Ouvrez uniquement la machine CPD lorsque toute la pression a été libérée du système. - Retirez rapidement les morceaux de tissu CPD de la machine et conservez-les dans un nouveau plat Petri stocké dans un dessiccateur avant d'être utilisé.
REMARQUE: Le protocole peut être mis en pause ici.
- Préremplir la chambre à vide avec 100% d'éthanol. Transférer les petits morceaux de tissu dans une capsule micro-poreuse et les placer dans la chambre à vide du CPD. Fermer le système.
- Préparer les VOI de l'échantillon de tissus mous. Couper les tissus mous en très petits morceaux d'environ 0,5 mm de longueur de bord à l'aide d'un scalpel et d'un stéréomicroscope. Dans le cas du rein de la souris: Couper le rein de la souris en deux moitiés le long de l'axe le plus long. Prenez la moitié du rein de la souris et préparez différentes régions anatomiques telles que le cortex rénal et la médulle rénale.
- Montez les morceaux de tissu CPD à un support d'échantillon approprié. Assurer un ajustement serré de l'échantillon sur le titulaire de l'échantillon afin d'empêcher l'échantillon de se déplacer pendant les mesures de CT. Dans le cas des morceaux de tissu rénal de souris de CPD : Collez les morceaux de tissu avec la superglue à un support d'échantillon.
REMARQUE : Tout mouvement indésirable de l'échantillon pendant les mesures de CT peut causer des problèmes pendant la reconstruction de volume - particulièrement en acquérant un jeu de données avec la taille de voxel de nanomètre.
REMARQUE: Le protocole peut être mis en pause ici. - Après un alignement attentif de l'échantillon, choisissez les paramètres d'acquisition pour une meilleure qualité d'image. Dans le cas des données nanoCT présentées : Acquier des projections à une tension de pointe de 60 kV avec 1599 projections réparties également sur 360 degrés et une taille de voxel d'environ 400 nm.
REMARQUE : Une seule mesure cT acquise à une taille voxel de 400 nm a un VVE de 75 m dans la direction de l'axe de rotation (vertical) et d'environ 560 m dans la direction perpendiculaire à l'axe de rotation (horizontal). Pour étudier de plus grands volumes, une extension du FOV le long de l'axe de rotation peut être réalisée en combinant plusieurs scans à différentes positions verticales. En outre, des balayages locaux de tomographie peuvent être exécutés pour mesurer des échantillons avec un plus grand diamètre d'échantillon perpendiculaire à l'axe de rotation que donné par le FOV d'un balayage global de CT. Les données nanoCT ont été acquises avec un temps d'exposition de 4 s par projection. Ainsi, le temps total d'acquisition par ensemble de données était d'environ 3,5 h.
REMARQUE: Le protocole peut être mis en pause ici. - Après l'acquisition des données CT, traiter les projections en conséquence pour la reconstruction du volume 3D.
- Dans le cas des données nanoCT présentées, normalisez les projections acquises avec des images à champ plat. Améliorez la netteté des projections en utilisant un algorithme de déconvolution Richardson-Lucy30,31. Utilisez une fonction gaussienne symétrique en rotation avec une déviation standard d'un pixel comme noyau de déconvolution.
- Appliquez l'algorithme de récupération de phase de Paganin aux images aiguisées pour augmenter le contraste entre les tissus mous. Définiz les paramètres de l'algorithme pour optimiser la qualité d'image32. Reconstruire les projections prétraitées à l'' gêne grâce à un algorithme de rétroprojection filtré.
REMARQUE : La figure 3 évalue les données nanoCT obtenues avec les sections histologiques correspondantes, qui étaient d'environ 7 m d'épaisseur. Par conséquent, des tranches de projection d'intensité minimale de 18 tranches nanoCT adjacentes d'une épaisseur virtuelle d'environ 7 m ont été générées au moyen du calcul de la valeur minimale pour chaque pixel dans les tranches pertinentes. Un logiciel de visualisation a été utilisé pour rendre le volume des données nanoCT, qui est affiché dans la figure 4.
Résultats
La figure 1 montre les tranches de CT et le rendu en volume des données microCT à basse résolution mettant en évidence l'amélioration du contraste après la coloration. La figure 2 montre les tranches de ToC et le rendu en volume des données microCT à haute résolution dérivées d'une tomographie locale de l'ensemble du rein de la souris. La figure 3 montre des tranches de données nanoCT par rapport aux sections histologiques correspondantes. La figure 4 montre le rendu de la tranche et du volume de la tomodensitome des données nanoCT mettant en évidence les détails structurels au niveau cellulaire. La mesure microCT à basse résolution permet une vue d'ensemble de l'organe entier et permet d'identifier les volumes d'intérêt (VOI) pour la mesure microCT à haute résolution. Grâce à cette approche multi-échelle, la VOI pour le nanoCT est déterminée. Le nanoCT permet une vue très détaillée de l'échantillon de tissus mous au niveau cellulaire. L'étude comparative avec la section histologique correspondante accentue la compatibilité complète avec l'histopathologie. Ici, l'approche de l'imagerie multimodale confirme les résultats obtenus avec les deux modalités.
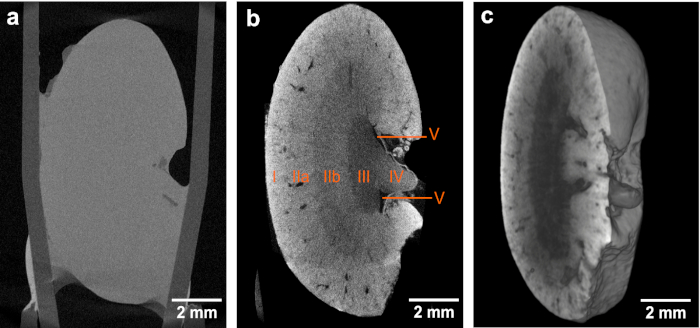
Figure 1. Tranches de CT et rendu en volume des données microCT à basse résolution. (a,b) Aperçu des images du même rein de souris avant et après la coloration, respectivement, mettant en évidence l'amélioration du contraste obtenue après l'application du protocole de coloration à base d'éosine. Les deux ensembles de données microCT ont été acquis à l'aide de paramètres d'acquisition identiques. La taille du voxel dans les deux ensembles de données est de 12 m. L'amélioration du contraste obtenue dans (b) permet l'identification des régions structurelles anatomiques suivantes: Cortex (I), medulla externe (II) avec une distinction supplémentaire dans les bandes extérieures de médulle externe (IIa) et les bandes intérieures de la médulle externe (IIb), médulle intérieure (III), papille (IV) et bassin rénal (V). (c) Rendu en volume des données microCT montrant une section sagittale virtuelle à travers le rein entier de la souris. Ce chiffre a été modifié à partir de Busse et M'ller et al.26S'il vous plaît cliquez ici pour voir une version plus grande de ce chiffre.

Figure 2. CT tranche et le rendu de volume des données microCT à haute résolution dérivées du même rein de souris après application du protocole développé de coloration à base d'éosine. (a) Le coin gauche affiche l'image microCT d'aperçu mettant en évidence le retour sur investissement (boîte bleue) pour l'image haute résolution affichée. Les régions structurelles anatomiques suivantes sont identifiables : Cortex (I), médulle externe (II) avec une distinction supplémentaire dans les rayures extérieures de la médulle externe (IIa) et les bandes intérieures de médulla externe (IIb), la médulle intérieure (III), le calice mineur (IV) et les vaisseaux (V et VI). ( b) Volume de rendu d'intérêt des données microCT haute résolution acquises d'une taille de voxel de 3,3 m. La région de medulla et une section virtuelle par un récipient dérivé d'une tomographie locale du rein entier est montrée. Ce chiffre a été modifié à partir de Busse et de M. Etmiller et d'autres26. Veuillez cliquer ici pour voir une version plus grande de ce chiffre.

Figure 3. Tranches de CT de données nanoCT (a,b) par rapport aux sections histologiques (c,d) dérivées du même rein de souris après application du protocole de coloration à base d'éosine développé. (a) L'image nanoCT du même échantillon de rein de souris après coloration, dissection et DPC montre des structures détaillées de la région (IIb) vues dans la figure 1 et la figure 2. Ceux-ci sont connus sous le nom de membres ascendants épais de la boucle de Henle. (b) Tranche de projection d'intensité minimale dérivée du même ensemble de données nanoCT indiquédans (a) avec une épaisseur de tranche virtuelle d'environ 7 m, ce qui permet une visualisation claire des noyaux cellulaires. (c) Section histologique représentative affichant d'épais membres ascendants de la boucle de Henle avec la visualisation claire des noyaux cellulaires et de la bordure de brosse. La section histologique a une épaisseur approximative de 7 m et a été obtenue à partir du même échantillon de rein de souris après la coloration et l'intégration appliquées à base d'éosine dans un bloc de paraffine. (d) Section histologique représentative avec application de l'hématoxylin de tache de compteur soulignant les noyaux de cellules en pourpre. Préparation de la section histologique proche de la section indiquée dans (c) avec une épaisseur approximative de 7 m. Ce chiffre a été modifié à partir de Busse et M'ller et al.26S'il vous plaît cliquez ici pour voir une version plus grande de ce chiffre.

Figure 4. CT tranche et le rendu en volume des données nanoCT. (a) L'image nanoCT du même échantillon de rein de souris montrant les structures connues sous le nom de membres ascendants épais de la boucle de Henle. Il s'agit d'une vue détaillée de la région (IIb) vue dans la figure 1 et la figure 2 acquises à partir d'un petit morceau du rein d'une taille de voxel d'environ 400 nm. La préparation de l'échantillon impliquait la coloration,la dissection et le rendu de volume des données nanoCT visualisant la structure 3D des membres ascendants épais des boucles de Henle. Ce chiffre a été modifié à partir de Busse et M'ller et al.26S'il vous plaît cliquez ici pour voir une version plus grande de ce chiffre.
Discussion
Actuellement, l'éosine est utilisée comme protocole histologique standard pour étiqueter le cytoplasme cellulaire. L'agent de coloration est appliqué comme une solution aqueuse de 0,1 % (w/v) sur des tranches microscopiques de tissus mous (généralement coupées avec une épaisseur de 2 à 10 m)33. L'application de ce protocole histologique normalisé aux échantillons de tissu 3D tels qu'un rein entier de souris n'a pas comme conséquence un contraste d'atténuation a augmenté l'image de CT. D'une part, cela peut être attribué aux faibles propriétés intrinsèques d'atténuation des tissus mous pour les énergies de rayons X généralement utilisées des systèmes de microCT en laboratoire. Habituellement, les tissus mous sont composés principalement de carbone, d'hydrogène, d'oxygène et d'azote34, et donc, ne se traduit pas par l'amélioration du contraste. D'autre part, la faible concentration d'éosine utilisée pour la coloration était le facteur limitant. Même si une molécule d'éosine contient quatre atomes de bromure (bromine à haut nombre atomique avec Z 3534), les niveaux de sensibilité requis pour l'imagerie par rayons X n'ont pas été atteints.
Pour surmonter ce défi du contraste de basse atténuation, plusieurs concentrations d'éosin ont été étudiées. Une limitation est ici la solubilité maximale de l'éosine dans l'eau, qui est de 30% (w/v) dans une solution aqueuse. La meilleure amélioration de contraste d'atténuation dans le tissu mou a été observée avec la concentration la plus élevée d'éosin, qui était prévue selon la loi de Lambert-Bière. Par conséquent, le protocole final de coloration a été effectué avec la plus forte concentration.
La question de savoir comment préparer le tissu mou de façon optimale au niveau moléculaire pour la procédure de coloration pour améliorer davantage l'amélioration du contraste a été répondue par un ajustement du pH. Ici, l'acidification de l'échantillon de tissu mou pendant la fixation ou avant la coloration s'est avérée cruciale. Hong et coll.35l'ont également montré. L'accumulation plus élevée de l'agent de coloration dans le cytoplasme cellulaire par l'acide a été réalisée par des interactions ioniques améliorées, qui étaient le résultat de la protonation des chaînes latérales d'acide aminé des protéines et des peptides présents dans le cytoplasme cellulaire. Un résultat représentatif soulignant l'amélioration du contraste par rapport à un échantillon de tissus mous non tachés est présenté à la figure 1a,b. Ici, un aperçu structurel d'un rein entier de souris visualisant les régions anatomiques cruciales telles que le cortex, la médulle, la papille et le bassin rénal a été réalisé.
Le protocole de coloration présenté est simple à appliquer et ne contient que trois étapes. Les réactifs requis sont facilement accessibles. Le temps de coloration global de 24 heures est rapide pour une coloration d'organes entiers, ce qui permet la visualisation 3D d'échantillons de tissus mous(figure 1c, Figure 2b et Figure 4b) dans un environnement de laboratoire à échelles multiples jusqu'au niveau cellulaire. Il convient de noter que le temps de coloration global et le volume de la solution de coloration nécessaire pourraient demander certaines adaptations en fonction de la nature de l'échantillon. Néanmoins, le protocole de coloration à base d'éosine convient à la coloration d'organes entiers, ce qui permet ensuite une imagerie microCT à haute résolution d'organes entiers. Les artefacts de rétrécissement dus à l'éthanol solvant, qui a été utilisé pour garder l'échantillon humide pendant les mesures de microCT, n'ont pas été observés. Des étapes de préparation supplémentaires sont nécessaires pour l'imagerie nanoCT, ce qui permet d'enquêter sur les morceaux de tissus plus petits extraits de l'échantillon d'origine. En ce qui concerne les applications histopathologiques futures, les analyses de vue d'ensemble fourniront des renseignements précieux sur les régions et les structures anatomiques modifiées, ce qui permettra de déterminer les IA comme le démontre la figure 2a. Ceux-ci peuvent être étudiés en 3D par microCT (Figure 1c et Figure 2b) ou nanoCT (Figure 4b) et évalués en 2D avec histologie ( Figure3).
Une autre force du protocole est vue dans la compatibilité complète avec l'histopathologie en ce qui concerne la procédure de coloration de H et E. L'application de la procédure de coloration à base d'éosine aux échantillons en vrac n'empêche pas d'autres investigations histologiques (figure 3), même si la concentration d'éosine appliquée est beaucoup plus élevée par rapport à la solution de coloration histologique. La tranche nanoCT d'une épaisseur virtuelle d'environ 400 nm (Figure 3a) se compare déjà très bien à la section histologique (Figure 3c), qui a été dérivée de l'échantillon de tissus mous correspondant. Compte tenu de l'épaisseur approximative d'une section histologique de 7 à 10 m, la génération de tranches de projection d'intensité minimale des données nanoCT (Figure 3b), qui correspondent à une épaisseur virtuelle d'environ 7 m, permet une meilleure comparaison avec la section histologique (Figure 3c). Ici, les noyaux cellulaires sont clairement révélés comme zone de non-atténuation que l'éosine tache spécifiquement les protéines et les peptides dans le cytoplasme cellulaire33.
L'application d'autres colorations de compteur avec des méthodes histologiques standard est possible, même si l'ordre de la coloration par rapport à la procédure de coloration histologique standard a été inversée. En commençant d'abord par le protocole de coloration à base d'éosin développé pour la tomodensitome, suivi d'une coloration contre de ces sections histologiques à base d'éosine avec de l'hématoxylin, permet une compatibilité complète et des résultats dans une coloration de haute qualité affichant la forme attendue d'apparence. La coloration spécifique aux noyaux cellulaires avec l'hématoxylin aigre de Mayer a été appliquée à la section histologique mettant en évidence les noyaux cellulaires en violet (Figure 3d). L'application de la coloration de compteur histologique est actuellement limitée à la tache H. D'autres colorations histologiques standard telles que la base périodique de Schiff acide, Elastica van Gieson ou Gomori argent doivent être évaluées aussi bien que la compatibilité avec des techniques immunohistologiques doit être examinée.
Le protocole de coloration à base d'éosine permet (i) le ciblage cellulaire spécifique au cytoplasme, (ii) la coloration homogène et complète, (iii) la mise en œuvre facile, (iv) la pénétration rapide du tissu sans créer d'artefacts tels que les anneaux de diffusion, (v) la coloration de d'échantillons de tissus mous importants et denses, et (vi) une compatibilité complète avec l'histopathologie à l'égard de la tache de H et E. Ces exigences sont importantes pour permettre la visualisation à haute résolution de la tordeuse à rayons X des tissus mous jusqu'au niveau cellulaire. En combinaison avec les dispositifs nanoCT récemment développés12,36,37, génération non destructive de tranches histologiques virtuelles qui sont comparables en contraste et en résolution aux données histologiques conventionnelles est rendu possible. Cette approche combinée permettra l'établissement de la tordelée à rayons X comme outil précieux pour la visualisation 3D des structures tissulaires microscopiques.
Déclarations de divulgation
Les auteurs n'ont rien à révéler.
Remerciements
Nous remercions le Dr Enken Drecoll pour ses discussions histologiques et l'équipe extrêmement utile d'Excillum AB, en Suède. Nous reconnaissons le soutien financier par l'intermédiaire du DFG Cluster of Excellence Munich Center for Advanced Photonics (MAP) et du programme DFG Gottfried Wilhelm Leibniz. En outre, ce projet de recherche a reçu un financement du programme européen de recherche et d'innovation Union Horizon 2020 dans le cadre de l'accord de subvention Marie-Skoodowska-Curie No. H2020-MSCA-IF-2015-703745-CONSALT.
matériels
| Name | Company | Catalog Number | Comments |
| 50-ml centrifuge tube by Falcon | VWR | 734-0453 | |
| Formaldehyde solution, 37% | Carl Roth | CP10.2 | acid-free, stabilized with ~10% MeOH |
| Glacial acetic acid | Alfa Aesar | 36289.AP | |
| Eosin Y disodium salt | Sigma-Aldrich | E4382 | certified by Biological Stain Commission |
| Phosphate Buffered Saline (PBS) | Merck | L1825 | Dulbecco's formualtion, w/o calcium and magnesium |
| Sample Tubes by Nalgene | Carl Roth | ATK5.1 | |
| Rocking Shaker ST5 | CAT | 60281-0000 | |
| Cellulose tissue paper | VWR | 115-0600 | |
| Forceps, by USBECK Laborgeräte | VWR | 232-0096 | |
| Microcentrifuge tubes by Eppendorf | VWR | 211-2120 | safe-lock, 2.0 ml |
| Ethanol absolute by Baker Analyzed | VWR | 80252500 | |
| Disposable safety scalpel by Aesculap | VWR | AESCBA210 | |
| Petri dish by Sterilin | VWR | 391-2019 | |
| Plastic pasteur pipette | Carl Roth | EA68.1 | graduated, 1 ml |
| Desiccator by Duran | VWR | SCOT247826954 | |
| Silicone grease by Bayer | Sigma-Aldrich | 85404 | high-vacuum |
| Carbon dioxide cylinder with standpipe | Linde | 3700113 | 10 kg, short |
| micro-porous treatment capsule | PLANO GmbH | 4614 | pore size 78 µm (B) |
| Bal-Tec CPD 030 | Bal-Tec AG | CO2 as drying agent | |
| Stemi 2000-C stereomicroscope with KL 1500 LCD | Zeiss | this stereomicroscope has been updated(1) | |
| Zeiss Xradia Versa 500 | Zeiss | this microCT scanner has been updated(2) | |
| Avizo Fire 8.1 | Thermo Fisher Scientific | ||
| PILATUS detector as part of the nanoCT scanner | Dectris | single-photon counting detector(4,5); there are commercially availble nanoCT systems available (6,7) | |
| nanofocus X-ray source as part of the nanoCT scanner | Excillum | high-flux nanofocus X-ray transmission tube(3); there are commercially availble nanoCT systems available(6,7) | |
| (1) Germany, Z. ZEISS product information: ZEISS stereomicroscopes https://www.micro-shop.zeiss.com/de/de/system/Stereomikroskope/1006> (September 06, 2019). | |||
| (2) Germany, Z. ZEISS product information: ZEISS Xradia 510 Versa https://www.zeiss.com/microscopy/int/products/x-ray-microscopy/zeiss-xradia-510-versa.html> (April 10, 2019). | |||
| (3) Nachtrab, F. et al. Development of a Timepix based detector for the NanoXCT project. Journal of Instrumentation 10 (11), C11009, (2015). | |||
| (4) Kraft, P. et al. Performance of single-photon-counting PILATUS detector modules. Journal of Synchrotron Radiation 16 (3), 368-375, (2009). | |||
| (5) Kraft, P. et al. Characterization and calibration of PILATUS detectors. IEEE Transactions on Nuclear Science 56 (3), 758-764, (2009). | |||
| (6) Germany, Z. ZEISS product information: ZEISS Xradia 810 Ultra https://www.zeiss.com/microscopy/int/products/x-ray-microscopy/xradia-810-ultra.html> (April 9 2019). | |||
| (7) Company, G. E. GE product information: Phoenix nanotom m, https://www.gemeasurement.com/sites/gemc.dev/files/geit-31344en_nanotom_m_0517.pdf> (April 10, 2019). |
Références
- Suvarna, S. K., Layton, C., Bancroft, J. D. . Theory and Practice of Histological Techniques. 7th edn. , (2013).
- Chatterjee, S. Artefacts in histopathology. Journal of Oral and Maxillofacial Pathology. 18 (4), 111-116 (2014).
- McInnes, E. Artefacts in histopathology. Comparative Clinical Pathology. 13 (3), 100-108 (2005).
- Andreasen, A., Drewes, A., Assentoft, J., Larsen, N. Computer-assisted alignment of standard serial sections without use of artificial land-marks. A practical approach to the utilization of incomplete information in 3-d reconstruction of the hippocampal region. Journal of Neuroscience Methods. 45 (3), 199-207 (1992).
- Braverman, M. S., Braverman, I. M. Three-dimensional reconstructions of objects from serial sections using a microcomputer graphics system. Journal of Investigative Dermatology. 86 (3), 290-294 (1986).
- Denk, W., Hortsmann, H. Serial block-face scanning electron microscopy to reconstruct three-dimensional tissue nanostructure. PLoS Biology. 2 (11), 329 (2004).
- Mohun, T. J., Weninger, J. W. Imaging heart development using high-resolution episcopic microscopy. Current Opinion in Genetics and Development. 21, 573-578 (2011).
- Weninger, J. W., Meng, S., Streicher, J., Müller, G. B. A new episcopic method for rapid 3-d reconstruction: applications in anatomy and embryology. Anatomy and Embryology. 197 (5), 341-348 (1998).
- Odgaard, A., Andersen, K., Melsen, F., Gundersen, H. J. G. A Direct Method for Fast 3-Dimensional Serial Reconstruction. Journal of Microscopy. 159, 335-342 (1990).
- Müller, M., et al. Myoanatomy of the velevt worm leg revealed by labratory-based nanofocus X-ray source tomography. Proceedings of the National Academy of Sciences of the United States of America. 114 (47), 12378-12383 (2017).
- Salomon, M., Hanke, R., Krüger, P., Uhlmann, N., Voland, V. Realization of a computed tomography setup to achieve resolutions below 1 µm. Nuclear Instruments and Methods in Physics Research Section A. 591, 50-53 (2008).
- Tkachuk, A., et al. X-ray computed tomography in Zernike phase contrast mode at 8 keV with 50-nm resolution using Cu rotating anode X-ray source. Zeitschrift für Kristallographie. 222, 650-655 (2007).
- Withers, P. J. X-ray nanotomography. Materials Today. 10, 26-34 (2007).
- Jahn, H., et al. Evaluation of contrasting techniques for X-ray imaging of velvet worms (Onychophora). Journal of Microscopy. 270 (3), 343-358 (2018).
- Martins de S. e Silva, J., et al. Three-dimensional non-destructive soft-tissue visualization with X-ray staining micro-tomography. Scientific Reports. 5, 14088 (2015).
- Metscher, B. D. MicroCT for Developmental Biology: A Versatile Tool for High-Contrast 3D Imaging at Histological Resolutions. Developmental Dynamics. 238, 632-640 (2009).
- Metscher, B. D. MicroCT for comparative morphology: simple staining methods allow high-contrast 3D imaging of diverse non-mineralized animal tissues. BMC Physiology. 9, 11 (2009).
- Mitzutani, R., et al. X-Ray Microtomographic Imaging of Three-Dimensional Structure of Soft Tissues. Tissue Engineering Part C: Methods. 14 (4), 359-363 (2008).
- Pauwels, E., Loo, D. v., Cornillie, P., Brabant, L., Hoorebeke, L. v. An exploratory study of contrast agents for soft tissue visualization by means of high resolution X-ray computed tomography imaging. Journal of Microscopy. 250, 21-31 (2013).
- Degenhardt, K., Wright, A. C., Horng, D., Padmanabhan, A., Epstein, J. A. Rapid 3D phenotyping of cardiovascular development in mouse embryos by micro-CT with iodine staining. Circulation: Cardiovascular Imaging. 3 (3), 314-322 (2010).
- Dullin, C., et al. μCT of ex-vivo stained mouse hearts and embryos enables a precise match between 3D virtual histology, classical histology and immunochemistry. PLOS ONE. 12 (2), 0170597 (2017).
- Jeffrey, N. S., Stephenson, R. S., Gallagher, J. A., Cox, P. Micro-computed tomography with iodine staining resolves the arrangement of muscle fibres. Journal of Biomechanics. 44, 189-192 (2011).
- Johnson, J. T., et al. Virtual Histology of Transgenic Mouse Embryos for High-Throughput Phenotyping. PLOS Genetics. 2, 61 (2006).
- Leszczyński, B., et al. Visualization and Quantitative 3D Analysis of Intraocular Melanoma and Its Vascularization in a Hamster Eye. International Journal of Molecular Sciences. 19 (2), 332 (2018).
- Mizutani, R., Suzuki, Y. X-ray microtomography in biology. Mircon. 43, 104-115 (2012).
- Busse, M., et al. Three-dimensional virtual histology enabled through cytoplasm-specific X-ray stain for microscopic and nanoscopic computed tomography. Proceedings of the National Academy of Sciences of the United States of America. 115 (10), 2293-2298 (2018).
- Müller, M., et al. Non-destructive high-resolution 3D virtual histology enabled through a cell nucleus-specific stain for X-ray computed tomography. Scientific Reports. 8, 17855 (2018).
- . ZEISS product information: ZEISS Xradia 510 Versa Available from: https://www.zeiss.com/microscopy/int/products/x-ray-microscopy/zeiss-xradia-510-versa.html (2019)
- Nachtrab, F., et al. Development of a Timepix based detector for the NanoXCT project. Journal of Instrumentation. 10 (11), 11009 (2015).
- Kraft, P., et al. Performance of single-photon-counting PILATUS detector modules. Journal of Synchrotron Radiation. 16 (3), 368-375 (2009).
- Kraft, P., et al. Characterization and calibration of PILATUS detectors. IEEE Transactions on Nuclear Science. 56 (3), 758-764 (2009).
- . ZEISS product information: ZEISS Xradia 810 Ultra Available from: https://www.zeiss.com/microscopy/int/products/x-ray-microscopy/zeiss-xradia-510-versa.html (2019)
- Anderson, T. F. Techniques for the preservation of three-dimensional structure in preparing specimens for the electron microscope. Transactions of the New York Academy of Sciences. 13 (4), 130-134 (1951).
- Bray, D., Williams, J. R., Clifford, A. A. . Supercritical Fluid Methods and Protocols. Methods in Biotechnology. 13, 235-243 (2000).
- Lucy, L. B. An iterative technique for the rectification of observed distributions. The Astronomical Journal. 79, 745-765 (1974).
- Richardson, W. H. Bayesian-Based Iterative Method of Image Restoration. The Journal of the Optical Society of America. 62 (1), 55-59 (1972).
- Paganin, F., Mayo, S. C., Gureyev, T. E., Miller, P. R., Wilkins, S. W. Simultaneous phase and amplitude extraction from a single defocused image of a homogeneous object. Journal of Microscopy. 206, 33-40 (2002).
- Riedelsheimer, B., Büchl-Zimmermann, S., Mulisch, M., Welsch, U. . Mikroskopische Technik. , 193-194 (2015).
- Hubbell, J. H., Seltzer, S. M. Tables of Xray mass attenuation coefficients and mass energy-absorption coefficients from 1 keV to 92 keV and 48 additional substances of dosimetric interest, Table 3. National Institute of Standards and Technology. , (1995).
- Hong, H. Y., Yoo, G. S., Choi, J. K. An Eosin Y Method for Protein Determination in Solution. Analytical Letters. 32 (12), 2427-2442 (1999).
- . GE product information: Phoenix nanotom m Available from: https://www.gemeasurement.com/sites/gemc.dev/files/geit-31344en_nanotom_m_0517.pdf (2019)
- Dierick, M., et al. Recent Micro-CT Scanner Developments at UGCT. Nuclear Instruments and Methods in Physics Research Section B. 324, 35-40 (2014).
- Kastner, J., Plank, B., Heinzl, C. Advanced X computed tomography methods: High resolution CT, quantitative CT, 4DCT and phase contrast CT. Proceedings of Digital Industrial Radiology and Computed Tomography. , 120-132 (2015).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon