Method Article
Análisis de SSHHPS Viral del Grupo IV utilizando métodos In Vitro y In Silico
En este artículo
Resumen
Presentamos un protocolo general para identificar tramos cortos de secuencias de proteínas homolopégicos de patógenos de host (SSHHPS) incrustadas en la poliproteína viral. SSHHPS son reconocidos por proteasas virales y dirigen la destrucción dirigida de proteínas huésped específicas por varios virus del Grupo IV.
Resumen
Las enzimas alfavirales se sintetizan en un solo polipéptido. La poliproteína no estructural (nsP) es procesada por su nsP2 cisteína proteasa para producir enzimas activas esenciales para la replicación viral. Las proteasas virales son muy específicas y reconocen secuencias de motivos de sitios de escote conservadas (6-8 aminoácidos). En varios virus del Grupo IV, las secuencias de motivos del sitio de escisión de la proteasa nsP se pueden encontrar en proteínas de huésped específicas implicadas en la generación de las respuestas inmunitarias innatas y, en algunos casos, las proteínas dirigidas parecen estar vinculadas al fenotipo inducido por el virus. Estos virus utilizan tramos cortos de secuencias de proteínas homolopégicos de patógenos del huésped (SSHHPS) para la destrucción específica de proteínas huésped. Para identificar SSHHPS, se pueden introducir secuencias de motivos de sitios de proteasa viral en BLAST y se pueden buscar los genomas del huésped. El escote inicialmente se puede probar utilizando los sustratos purificados de proteasa viral nsP y transferencia de energía por resonancia de fluorescencia (FRET) fabricados en E. coli. Los sustratos de FRET contienen proteína fluorescente cian y amarilla y la secuencia del sitio de escisión (CFP-sequence-YFP). Este ensayo de proteasa se puede utilizar continuamente en un lector de placas o de forma discontinua en geles SDS-PAGE. Los modelos de los sustratos peptídicos unidos se pueden generar en silico para guiar la selección del sustrato y los estudios de mutagénesis. Los sustratos de CFP/YFP también se han utilizado para identificar inhibidores de la proteasa. Estos métodos in vitro e in sálico se pueden utilizar en combinación con ensayos basados en células para determinar si la proteína huésped dirigida afecta la replicación viral.
Introducción
La evidencia de la transferencia horizontal de genes del virus al huésped, o del huésped al virus, se puede encontrar en una variedad de genomas1,2,3,4. Ejemplos de endogenización viral son las secuencias espaciadoras CRISPR que se encuentran en los genomas del huésped bacteriano4. Recientemente, hemos encontrado evidencia de secuencias de proteínas huésped incrustadas en las poliproteínas no estructurales de virus (+)ssRNA Group IV. Estas secuencias dentro de las regiones codificacionales del genoma viral se pueden propagar generacionalmente. Los tramos cortos de secuencias de proteínas homólogas-patógenas (SSHHPS) se encuentran en el virus y el host5,6. SSHHPS son las secuencias de motivos del sitio de escisión conservadas reconocidas por las proteasas virales que tienen homología con proteínas anfitrionas específicas. Estas secuencias dirigen la destrucción de proteínas huésped específicas.
En nuestra publicación anterior6, compilamos una lista de todas las proteínas anfitrionas que fueron objetivo de las proteasas virales y encontramos que la lista de objetivos no era aleatoria(Tabla 1). Dos tendencias eran evidentes. En primer lugar, la mayoría de las proteasas virales que cortan las proteínas huésped pertenecían a virus del Grupo IV (24 de los 25 casos implicados en proteasas virales del Grupo IV), y una proteasa pertenecía a los retrovirus del Grupo VI (VIH, virus de inmunodeficiencia humana)7. En segundo lugar, las dianas de proteína huésped cortadas por las proteasas virales generalmente estaban involucradas en la generación de las respuestas inmunitarias innatas que sugerían que los escotes estaban destinados a antagonizar las respuestas inmunitarias del huésped. La mitad de las proteínas huésped atacadas por las proteasas virales eran componentes conocidos de las cascadas de señalización que generan interferón (IFN) y citoquinas proinflamatorias (Tabla 1). Otros participaron en la transcripción de la célula anfitriona8,9,10 o traducción11. 4han demostrado que muchas secuencias de protoespaciales CRISPR corresponden a genes implicados en la conjugación o replicación del plásmido4.
El Grupo IV incluye, entre otros, Flaviviridae, Picornaviridae, Coronaviridae, Calciviridae y Togaviridae. Varios patógenos nuevos y emergentes pertenecen al Grupo IV, como el virus del Zika (ZIKV), el Nilo Occidental (WNV), el Chikungunya (CHIKV), el virus del síndrome respiratorio agudo grave (SARS) y el virus del síndrome respiratorio de Oriente Medio (MERS). El genoma (+)ssRNA es esencialmente un pedazo de ARNm. Para producir las enzimas necesarias para la replicación del genoma, primero debe traducirse el genoma (+)ssRNA. En alfavirus y otros virus del Grupo IV, las enzimas necesarias para la replicación se producen en una sola poliproteína (es decir, nsP1234 para el VEEV). La poliproteína no estructural (nsP) se procesa proteolíticamente (nsP1234 nsP1, nsP2, nsP3, nsP4) por la proteasa nsP2 para producir enzimas activas12 (Figura 1). El escote de la poliproteína por la proteasa nsP2 es esencial para la replicación viral; esto se ha demostrado mediante la eliminación y mutagénesis dirigida por el sitio de la cisteína del sitio activo de la proteasa nsP213,14. En particular, la traducción de proteínas virales precede a los eventos de replicación del genoma. Por ejemplo, nsP4 contiene la ARN polimerasa dependiente del ARN necesaria para replicar el genoma del ARN (+). La replicación del genoma puede producir intermedios dsRNA; estos intermedios pueden desencadenar las respuestas inmunitarias innatas del huésped. Por lo tanto, estos virus pueden separar las proteínas de respuesta inmune innatas del huésped al principio de la infección con el fin de suprimir sus efectos15,16,17.
El silenciamiento puede ocurrir a nivel de ADN, ARN y proteína. Lo que es común a cada uno de los mecanismos de silenciamiento mostrados en la Figura 1 es que se utilizan secuencias cortas de ADN extraño, ARN o proteínas para guiar la destrucción de objetivos específicos para antagonizar su función. Los mecanismos de silenciamiento son análogos a los programas de "búsqueda y eliminación" que se han escrito en tres idiomas diferentes. La secuencia de sitio de escisión corta es análoga a una "palabra clave". Cada programa tiene una enzima que reconoce la coincidencia entre la secuencia corta (la "palabra clave") y una palabra en el "archivo" que se va a eliminar. Una vez que se encuentra una coincidencia, la enzima corta ("elimina") la secuencia de destino más grande. Los tres mecanismos que se muestran en la Figura 1 se utilizan para defender al huésped de virus o para defender un virus del sistema inmunitario de un huésped.
Las proteasas virales reconocen secuencias de motivos cortos del sitio de escisión entre los aminoácidos de 2 a 11 años; en nucleótidos, esto correspondería a 6-33 bases. A modo de comparación, las secuencias de espaciadores CRISPR son de 26-72 nucleótidos y los ARNi son de 20 a 22 nucleótidos18,19. Aunque estas secuencias son relativamente cortas, se pueden reconocer específicamente. Dada la mayor diversidad de aminoácidos, la probabilidad de un evento de escisión aleatoria es relativamente baja para una proteasa viral que reconoce secuencias de proteínas de 6-8 aminoácidos o más. La predicción de SSHHPS en proteínas huésped dependerá en gran medida de la especificidad de la proteasa viral que se está examinando. Si la proteasa tiene requisitos estrictos de especificidad de secuencia, la posibilidad de encontrar una secuencia de sitio de escisión es de 1/206 a 1 en 64 millones o 1/208 a 1 en 25.600 millones; sin embargo, la mayoría de las proteasas tienen tolerancias de subsitio variables (por ejemplo, R o K pueden tolerarse en el sitio S1). Por lo tanto, no hay ningún requisito para la identidad de secuencia entre las secuencias encontradas en el host frente al virus. Para las proteasas virales que tienen requisitos de secuencia más flexibles (como los que pertenecen a Picornaviridae) la probabilidad de encontrar un sitio de escisión en una proteína huésped puede ser mayor. Muchas de las entradas de la Tabla 1 son de la familia Picornaviridae.
La notación Schechter & Berger20 se utiliza comúnmente para describir los residuos en un sustrato de proteasa y los subsitios a los que se unen, utilizamos esta notación en todo. Los residuos en el sustrato que son N-terminal de la unión scissile se indican como P3-P2-P1, mientras que los que son C-terminal se indican como P1'-P2'-P3'. Los subsitios correspondientes en la proteasa que unen estos residuos de aminoácidos son S3-S2-S1 y S1'-S2'-S3', respectivamente.
Para determinar qué proteínas huésped están siendo atacadas, podemos identificar SSHHPS en los sitios de escisión de poliproteína viral y buscar las proteínas huésped que las contienen. En este documento, delineamos procedimientos para identificar SSHHPS utilizando secuencias de sitios de escisión de proteasa viral conocidas. Los métodos bioinformáticos, los ensayos de proteasa y los métodos de silico descritos están destinados a utilizarse junto con ensayos basados en células.
Las alineaciones secuenciales de las proteínas huésped a las que se dirigen las proteasas virales han revelado diferencias específicas de cada especie dentro de estas secuencias cortas del sitio de escisión. Por ejemplo, se encontró que la proteasa del virus de la encefalitis equina venezolana (VEEV) nsP2 cortó la proteína humana TRIM14, un motivo tripartito (TRIM)6. Algunas proteínas TRIM son factores de restricción viral (por ejemplo,TRIM5-21), la mayoría se cree que son ligasas de ubiquitina E3. TRIM14 carece de un dominio RING (realmente interesante nuevo gen) y no se cree que sea una ligasa E322. SE ha propuesto que TRIM14 sea un adaptador en el mechondriés antiviral mitocondrial (MAVS)22, pero puede tener otras funciones antivirales23. La alineación de las secuencias TRIM14 de varias especies muestra que el equino carece del sitio de escisión y alberga una versión truncada de TRIM14 que le falta el dominio PRY/SPRY del terminal C. Este dominio contiene un sitio de poliubiquitinación(Figura 2). En equino, estos virus son altamente letales (20-80% de mortalidad) mientras que en los seres humanos sólo el 1% muere a causa de las infecciones por VEEV24. El escote del dominio PRY/SPRY puede cortocircuitar transitoriamente la cascada de señalización MAVS. Esta cascada puede ser desencadenada por dsRNA y conduce a la producción de interferón y citoquinas proinflamatorias. Por lo tanto, la presencia del SSHHPS puede ser útil para predecir qué especies tienen sistemas de defensa contra virus específicos del Grupo IV.
En los virus del Grupo IV, se cree que los mecanismos de antagonismo IFN multiplican redundantes25. El escote de proteína huésped puede ser transitorio durante la infección y las concentraciones pueden recuperarse con el tiempo. Encontramos en las células que los productos de escisión TRIM14 podrían detectarse muy temprano después de la transfección (6 h) con un plásmido que codifica la proteasa (promotor del citomegalovirus). Sin embargo, en períodos más largos, no se detectaron los productos de escisión. En las células infectadas por virus, la cinética era diferente y los productos de escisión podían detectarse entre 6-48 h6. Otros han reportado la aparición de productos de escote de proteína huésped tan pronto como 3-6 h después de la infección9,11.
La actividad proteolítica en las células es a menudo difícil de detectar; los productos de escote pueden variar en su solubilidad, concentración, estabilidad y vida útil. En los ensayos a base de células, no se puede suponer que los productos de escote se acumularán en una célula o que las intensidades de la banda de proteínas cortadas y no cortadas mostrarán aumentos y disminuciones compensatorias, ya que la proteína cortada puede degradarse muy rápidamente y no puede ser detectable en una mancha occidental a un peso molecular esperado (MW) (por ejemplo, la región que contiene el epitope podría ser cortada por otros proteases del huésped o podría ser ubinada). Si el sustrato de la proteasa viral es una proteína de respuesta inmune innata, su concentración puede variar durante la infección. Por ejemplo, algunas proteínas de respuesta inmune innatas están presentes antes de la infección viral y son inducidas aún más por el interferón26. Por lo tanto, la concentración de la proteína diana puede fluctuar durante la infección y la comparación de los lysates celulares infectados no infectados puede ser difícil de interpretar. Además, es posible que todas las células no estén transfiguradas o infectadas de forma uniforme. Los ensayos de proteasa in vitro que utilizan proteínas purificadas de E. coli, por otro lado, tienen menos variables para controlar y tales ensayos se pueden hacer usando SDS-PAGE en lugar de inmunoblots. Las proteasas contaminantes pueden inhibirse en los primeros pasos de la purificación proteica del sustrato de CFP/YFP, y las proteasas virales mutadas se pueden purificar y probar como controles para determinar si el escote se debe a la proteasa viral o a una proteasa bacteriana contaminante.
Una limitación de los ensayos de proteasa in vitro es que carecen de la complejidad de una célula de mamífero. Para que una enzima corte su sustrato, los dos deben ser colocalizados. Las proteasas virales del grupo IV difieren en la estructura y la localización. Por ejemplo, la proteasa ZIKV está incrustada en la membrana de retículo endoplasmático (ER) y se enfrenta al citosol, mientras que la proteasa VEEV nsP2 es una proteína soluble en el citoplasma y el núcleo27. Algunas de las secuencias de sitios de escote encontradas en el análisis DE ZIKV SSHHPS estaban en péptidos de señal que sugieren que el escote podría ocurrir de forma co-traduccional para algunos objetivos. Por lo tanto, la ubicación de la proteasa y el sustrato en la célula también debe ser considerado en estos análisis.
Los ensayos a base de células pueden ser valiosos para establecer un papel para las proteínas huésped identificadas en la infección. Los métodos que tienen como objetivo detener la escisión de la proteasa viral de proteínas huésped como la adición de un inhibidor de la proteasa6 o una mutación en la dianahuésped 16 se pueden utilizar para examinar sus efectos en la replicación viral. La sobreexpresión de la proteína dirigida también puede afectar la replicación viral28. Los ensayos de placa u otros métodos se pueden utilizar para cuantificar la replicación viral.
Protocolo
1. Bioinformática: Identificación de SSHHPS en el Genoma Anfitrión Usando BLAST
NOTA: La proteína BLAST se puede encontrar en blast.ncbi.nlm.nih.gov/Blast.cgi.
- Introduzca 20 aminoácidos que rodean el enlace de escisil en la poliproteína viral. Seleccione secuencias de proteínas no redundantes y escriba en el genoma del huésped que se va a buscar (por ejemplo, Homo sapiens).
- Si es necesario, seleccione PHI-BLAST. Escriba una secuencia de patrones (por ejemplo, para los 25 residuos de V12 que se muestran a continuación, introduzca el patrón "AG" sin comillas).

NOTA: En PHI-BLAST, los corchetes [XY] indican que el aminoácido X o Y pueden estar en la posición del subsitio (por ejemplo, AG[AC][GAY]). - Inspeccionar los resultados de BLAST e identificar los impactos que tienen una identidad de secuencia alta a los residuos que se conservan en los sitios de escisión de poliproteína (por ejemplo, proteína de motivo tripartito 14)(Figura 3).
NOTA: Para las proteasas de la serina se espera una mayor conservación del residuo P1, mientras que para las proteasas de cisteína se espera una mayor conservación del residuo P2. - Coloree los residuos que son idénticos a una secuencia de sitio de escisión y que están en orden secuencial (sin huecos). Colorea los residuos tolerados en el subsitio, pero presente en un sitio de escote diferente en un segundo color.
NOTA: Los residuos que representan sustituciones conservadoras (por ejemplo, Leu vs Val) que no están presentes en un sitio de escisión viral también pueden ser encontrados y pueden o no ser reconocidos por la proteasa viral. - Orden de clasificación de los golpes BLAST basados en el número de residuos idénticos o tolerados consecutivos que coinciden con una secuencia de sitio de escisión. En la lista, seleccione las proteínas que contienen 6 residuos idénticos o similares para su análisis en ensayos de proteasa.
- Repita el procedimiento para los otros sitios de escisión (nsP2/3, nsP3/4, etc.) y fortalezca gradualmente la predicción añadiendo más residuos altamente conservados al patrón PHI-BLAST.
- Si es necesario, seleccione PHI-BLAST. Escriba una secuencia de patrones (por ejemplo, para los 25 residuos de V12 que se muestran a continuación, introduzca el patrón "AG" sin comillas).
2. Ensayos In Vitro: Diseño y preparación de sustratos de proteasa
- Construir un plásmido que codifica la proteína fluorescente cian (CFP), 25 aminoácidos de la secuencia del sitio de escisión, seguido de la proteína fluorescente amarilla (YFP, también conocida como Venus29).
NOTA: El plásmido se puede construir utilizando clonación independiente de secuencia y ligadura (SLIC)30 o síntesis de genes comerciales. Un plásmido pet15b que contiene la secuencia mostrada en la Figura 4 fue sintetizado comercialmente y se utilizó aquí.- Para optimizar la longitud del sustrato, construya sustratos FRET de longitud variable adicionales que contengan 12-25 aminoácidos de las secuencias de sitio de escisión de poliproteína viral natural utilizando una reacción SLIC de 2 fragmentos. Analice el escote utilizando el ensayo basado en gel SDS-PAGE o midiendo los parámetros cinéticos en estado estacionario utilizando los métodos siguientes.
NOTA: En algunos casos, los sitios de escote se pueden identificar por homología a sitios de escisión conocidos31. Si no se observa el escote de los sustratos que contienen las secuencias de unión de poliproteínas, puede haber un requisito de residuos adicionales o un motivo estructural (por ejemplo, una hélice alfa32). Alternativamente, la proteasa viral purificada puede estar inactiva. Confirmar el escote de las secuencias de poliproteínas virales antes de seguir el análisis SSHHPS. El número de residuos en el sustrato se optimizó para la proteasa VEEV utilizando sustratos de longitud variable (12 a 25 aminoácidos) seguido según el análisis de Vmax y Km32,33. Los sitios de escisión de proteasa viral zika ns2B/nsB utilizados en los ejemplos se han publicado34,35.
- Para optimizar la longitud del sustrato, construya sustratos FRET de longitud variable adicionales que contengan 12-25 aminoácidos de las secuencias de sitio de escisión de poliproteína viral natural utilizando una reacción SLIC de 2 fragmentos. Analice el escote utilizando el ensayo basado en gel SDS-PAGE o midiendo los parámetros cinéticos en estado estacionario utilizando los métodos siguientes.
- Preparar los sustratos de CFP/YFP transformando recién 8-20 l de BL-21(DE3)E. colicélulas competentes con el plásmido CFP-V12-YFP de acuerdo con las instrucciones del fabricante y la placa de las placas de agar Luria Bertani (LB) que contienen 50 g/ml de ampicilina (37 oC).
- Autoclave cuatro matraces de 4 L que contienen medios LB (1,5 L por matraz) y 100 ml de LB en un matraz de 250 ml. Tapar cada matraz con papel de aluminio.
- Inocular el cultivo de 100 ml con una colonia de bacterias recién transformadas y crecer a 37 oC con temblores (200 rpm) durante la noche.
- Para hacer el sustrato CFP/YFP, inocular cuatro matraces de 4 L con 25 ml de un cultivo nocturno. Comience a agitar los cultivos a 37 oC y supervise el crecimiento mediante espectroscopia UV-vis a 600 nm cada hora.
- Cuando las bacterias alcanzan una absorbancia de 1,0 a 600 nm (aproximadamente 3-4 h de crecimiento) inducen la expresión de proteínas añadiendo 0,5 ml de 1 M isopropyl-D-tiogalactoside (IPTG) por matraz. Después de añadir IPTG, baje la temperatura de la incubadora de agitación a 17 oC y permita que la expresión continúe durante la noche durante 17-20 h.
- Peletizar las bacterias utilizando una centrífuga de alta velocidad a 7.000 x g durante 10 min (4 oC) y retener los pellets. Retire y deseche los medios líquidos. Almacene los pellets a -80 oC inmediatamente.
- Preparar 100 ml de tampón de lisis que contenga 50 mM Tris pH 7.6, 500 mM NaCl, 35 ml de reactivo de extracción de proteína bacteriana, 30 mg de lisozyma, 25 U de DNase y 1 comprimido inhibidor de la proteasa. Resuspenda los pellets en el tampón de lisis con una pipeta y transfiera 25-35 ml a tubos cónicos desechables de 50 ml.
- Coloque los tubos en un vaso de plástico que contenga agua helada. Inserte la punta del sonicador en los tubos de modo que la punta esté a 1 cm de la parte inferior del tubo y sonice las lysates 10-20 veces en el nivel 5 durante intervalos de 15 s hasta que el lisado se vuelva fluido y licuado.
NOTA: Utilice protección auditiva durante la sonicación. - Transfiera el lisado a tubos centrífugos de alta velocidad y centrífuga a 20.500 x g durante 30 minutos a 4 oC. Después del giro, retenga el sobrenadante (100 ml) y transfieralo a una botella limpia. Deseche los pellets.
- Preparar 1 L de Tampón A (50 mM Tris pH 7.6, 500 mM NaCl). Preparar 300 mL de Tampón B (50 mM Tris pH 7.6, 500 mM NaCl, 300 mM Imidazol).
- Equilibre una columna de níquel de 100 ml utilizando volúmenes de 3 columnas de Tampón A y un caudal de 5 ml/min.
- Cargue el lisado en la columna de níquel usando un caudal de 2 a 5 ml/min. Lavar la columna con 2 volúmenes de columna de Tampón A, seguido de volúmenes de columna de 5 o5 de 20% de tampón B. Durante el lavado del 20% del tampón B, la absorbancia a 280 nm (A280) aumentará a medida que los contaminantes se eluyen de la columna durante el lavado. Continúe lavando la columna hasta que el A280 del eluido haya vuelto a los valores de referencia.
- Eluir la proteína con 2-3 volúmenes de columna de 100% Tampón B usando un caudal de 2-5 mL/min y recoger fracciones de 10 ml. Mida el A280 de cada fracción.
- Combine y concentre fracciones que contengan A280 > 0,1 utilizando una unidad de ultrafiltración centrífuga de 15 ml. Gire las unidades de ultrafiltración a 5.000 x g durante 15 minutos y continúe agregando fracciones hasta que el volumen se haya reducido a 50-75 ml.
- Corte una pieza de 14 pulgadas de tubería de diálisis con un corte de peso molecular (MWCO) de 6-8 kDa. Hidratar el tubo de diálisis hirviendo completamente sumergido en 300 ml de agua durante 10 min. Ate un nudo seguro en un extremo de la membrana. Llene la bolsa con tampón de diálisis para asegurarse de que no haya grietas o fugas. Retire el tampón de la bolsa y mantenga la bolsa sumergida en el búfer de diálisis.
- Transfiera la proteína concentrada de 2.2.13 a la bolsa de diálisis con una pipeta de plástico. Retire las burbujas de aire de la bolsa. Cierre la bolsa con un segundo nudo o un clip de diálisis. Dializar la proteína contra 500 mL de 50 mM Tris pH 7.6, 5 mM EDTA (ácido etilendiaminetetraacético), 250 mM naCl en un cilindro graduado de 500 ml durante la noche a 4 oC.
- Dializar la proteína por segunda vez contra 500 mL de 50 mM Tris pH 7.6 a 4 oC durante 2 h.
- Para la columna de intercambio de aniones prepare 500 mL de Tampón A (50 mM Tris pH 7.6) y 500 mL Buffer B (50 mM Tris pH 7.6, 1.0 M NaCl). Equilibrar una columna de intercambio de aniones de 30 ml con 3 volúmenes de columna de búfer A (2-5 ml/min).
- Retire la proteína de la bolsa de diálisis y transfiera a un frasco. Mantenga la botella sobre hielo. Cargue la proteína dializada en la columna (2-5 ml/min).
NOTA: La proteína CFP/YFP unirá la columna y será amarilla en apariencia. - Lave la columna con el búfer A hasta que el A280 vuelva a la línea base (5 ml/min). Eluir la proteína usando un gradiente (0-50% Tampón B, 100 mL) y recoger fracciones de 10 ml.
- Inspeccione las fracciones de columna utilizando SDS-PAGE. Combina aquellos que sean >95% puros.
- Concentrar la proteína en un A280 a 10-20 utilizando una unidad de ultrafiltración centrífuga de 15 ml. Gire el concentrador a 4.500 x g durante 10 min a 4 oC y continúe agregando proteína hasta que se hayan combinado todas las fracciones que contienen proteínas.
- Retire la proteína de la bolsa de diálisis y transfiera a un frasco. Mantenga la botella sobre hielo. Cargue la proteína dializada en la columna (2-5 ml/min).
- Retire cuidadosamente la proteína del concentrador con una pipeta. Alícuota la proteína en tubos de microcentrífuga de 1,5 ml y congelación de flash en nitrógeno líquido para almacenamiento a largo plazo a -80 oC. Intercambio de búfer es la proteína a temperatura ambiente utilizando una columna PD-10 equilibrada con el tampón de ensayo adecuado antes de su uso.
- Utilizando la ley de la cerveza calcular la concentración de proteínas utilizando el A280 y un coeficiente de extinción calculado (por ejemplo, para el sustrato V12 el á 47.790 M-1 cm-1).
NOTA: El coeficiente de extinción se puede calcular a partir de la secuencia proteica de la Figura 4 utilizando el programa Expasy ProtParam (https://web.expasy.org/protparam/).
3. Preparación de la alfaviral nsP2 Cisteína Proteasa
- Diseñar y construir un plásmido que codifica la proteasa. Para las proteasas de cisteína, utilice el plásmido pet32 para construir una proteína de fusión de tioredoxina (Trx).
Nota:El plásmido pet32 codifica un sitio de escote de trombina (LVPRGS) para la eliminación de la tioredoxina y su etiqueta (Figura 5). Tioredoxina ayudará a mantener la cisteína del sitio activo en un estado reducido durante la expresión. Para las proteasas de la serina, la tioredoxina no es necesaria y se pueden omitir los pasos que implican su eliminación por trombina. La secuencia de proteasa VEEV nsP2 se incorporó a un plásmido pet32b que se preparó comercialmente para evitar el manejo de agentes Select.- Transforma de forma nueva el ADN plásmido en BL21(DE3)pLysS E. coli según las instrucciones del fabricante. Placar las bacterias en placas de agar LB que contienen ampicilina.
NOTA: El cloramphenicol solo se utiliza para cepas de E. coli que transportan el plásmido pLysS y se omite si se utilizan células BL21(DE3). No es necesario incluir cloramphenicol en la placa de agar LB en este paso. - Autoclave cuatro matraces de 4 L de 1,5 L de medios LB (6 L de volumen total) y 100 ml de LB en un matraz de 250 ml. Tapar cada matraz con papel de aluminio.
- Inocular un cultivo nocturno de 100 ml de LB/Ampicilina con una colonia de la placa y crecer en una incubadora de temblores (200 rpm) a 37 oC.
- Inocular los matraces de 4 L con 25 ml del cultivo nocturno y añadir los antibióticos adecuados.
NOTA: Los medios para las células de pLysS BL21(DE3) que transportan el plásmido pet32 deben tener concentraciones finales de 25 g/ml de cloramphenicol y 50 g/ml de ampicilina. - Inducir la expresión de proteína añadiendo 0,5 ml de IPTG al cultivo cuando la absorbancia a 600 nm alcance 1,0. Bajar la temperatura de la incubadora de agitación a 17 oC. Permita que la expresión continúe durante la noche (17 h).
- Peletizar las células por centrifugación (7.000 x g durante 10 min a 4oC). Retire y deseche el medio líquido.
NOTA: Los pellets pueden almacenarse a -80 oC durante meses o lysed inmediatamente. - Preparar 100 ml de tampón de lisis (50 mM Tris pH 7.6, 500 mL NaCl, 2 mM beta mercaptoetanol (BME), 30 mg de lisozyma, 5% glicerol, 25 U DNase, 35 mL de reactivo de extracción de proteína bacteriana). Abra las botellas de BME en una campana química al agregar. Mantener el lisado bacteriano en hielo o a 4 oC para esto y todos los pasos subsiguientes.
NOTA: Para las proteasas de cisteína, 2 mM BME se incluye para mantener la cisteína nucleófila reducida. Las columnas se pueden ejecutar a temperatura ambiente utilizando búferes refrigerados. Los tampones deben hacerse con agua desionizada en frío enfriada a 4oC. - Resuspender los gránulos bacterianos en 25 ml de tampón de lisis y transferir 25 ml del lisado en tubos cónicos desechables de 4 x 50 ml. Coloque los tubos en vasos de plástico que contengan agua helada. Sonicar el lysate 10 veces en el nivel 5 para intervalos de 15 s.
- Transfiera el lisado a tubos centrífugos de alta velocidad. Aclarar el lisado por centrifugación (30 min, 20.500 x g a 4oC).
- Preparar 0,5 L de Tampón A (50 mM Tris pH 7,6, 500 mM NaCl, 5% glicerol, 2 mM BME) y enfriar a 4oC.
- Preparar 250 mL de Tampón B (50 mM Tris pH 7.6, 500 mM NaCl, 5% glicerol, 2 mM BME, 300 mM imidazol) y enfriar a 4oC.
- Equilibrar una columna de níquel de 50 ml con 3 volúmenes de columna de Tampón A. Cargue el lisado clarificado en la columna a 2-5 ml/min y deseche los pellets.
- Lave la columna (2-5 mL/min) con 2 volúmenes de columna de Tampón A seguidode según 5 volúmenes de columna de Tampón A que contienen 20% de Tampón B (60 mM Imidazol). Elute la proteína (5 ml/min) con 100% Tampón B y recoge fracciones de 10 ml.
- Combinar y concentrar fracciones que contengan la proteasa que tienen A280 o 0,1 utilizando una unidad de ultrafiltración centrífuga de 15 ml y giros de 15 minutos a 5.000 x g a 4 oC. Después de que el volumen se haya reducido a 5 ml, cambie la proteína en la unidad de concentración añadiendo un tampón de diálisis fresco a la proteína (50 mM Tris pH 7,6, 250 mM NaCl, 5 mM de ditiothreitol (TDT), 1 mM EDTA, 5% de glicerol). Vuelva a girar a 5.000 x g a 4 oC durante 15 min; repetir el paso de intercambio de búfer 2-3 veces. Añadir trombina a la proteína (20 l de 1 unidad/L) antes de la diálisis para eliminar la tioredoxina y su etiqueta.
- Transfiera la proteína a una bolsa de diálisis y marque y marque dos contra 500 ml del tampón de diálisis (4 oC) en un cilindro graduado de 500 ml durante la noche.
NOTA: El sistema FPLC (cromatografía líquida de proteína rápida) y la columna de níquel deben limpiarse a fondo con tampón de desmontaje (2 M NaCl, 50 mM EDTA) antes de proceder a la columna de intercambio de aniones. Cualquier níquel residual en las líneas FPLC convertirá las soluciones tampón que contienen DTT marrón cuando se mezclan. Lave la columna de níquel y el sistema FPLC con 4 volúmenes de columna de agua. La bomba lave bien el sistema FPLC con agua. La columna de níquel se puede regenerar fluyendo 2 volúmenes de columna de 0,2 M de sulfato de níquel sobre la resina para purificaciones posteriores.
- Transforma de forma nueva el ADN plásmido en BL21(DE3)pLysS E. coli según las instrucciones del fabricante. Placar las bacterias en placas de agar LB que contienen ampicilina.
- Para la columna de intercambio de aniones prepare 1 L de TA TM de TM de 50 mM (50 mM Tris pH 7,6, DTT de 5 mM, glicerol al 5%).
- Preparar 0,5 L de Tampón B (50 mM Tris pH 7,6, 5 mM de TDT, 5% glicerol, 1,25 M NaCl).
- Equilibrar una columna de intercambio de aniones de 30 ml con búfer A (3 volúmenes de columna, 2-5 ml/min). Coloque los tubos en el colector de fracciones para la recolección del flujo a través.
NOTA: La proteasa VEEV tiene un punto isoeléctrico calculado (pI) de 8.7 y enlazará columnas de intercambio catiónico, pero fluirá a través de columnas de intercambio de aniones. El pI se puede calcular a partir de la secuencia de proteínas utilizando el programa Expasy ProtParam (https://web.expasy.org/protparam/). - Diluir la proteína dializada 1:3 con Tampón A, y luego cargar la proteína (5 ml/min). Recoja el flujo a través en fracciones de 10 mL.
- Retire la columna de intercambio de aniones del sistema FPLC. Conecte una columna de intercambio catiónico al sistema FPLC. Equilibre una columna de intercambio catiónico de 30 ml con 3 volúmenes de columna de búfer A (5 ml/min).
- Cargue el flujo a través de la columna de intercambio de aniones en la columna de intercambio catiónico a 2-5 ml/min. Lave la columna con el búfer A hasta que el A280 vuelva al nivel de línea base. Elute la proteína con un gradiente de 100 ml (0-50% Tampón B) y recoge fracciones de 10 ml.
NOTA: La proteasa VEEV eluirá alrededor de 0,6 M NaCl. - Inspeccione las fracciones de columna utilizando SDS-PAGE. Combine fracciones que sean >95% puras y concentre en un A280 s 2 usando unidades centrífugas de 15 ml de ultrafiltración centrífuga. La enzima puede congelarse con flash en nitrógeno líquido y almacenarse a -80 oC.
- Cargue el flujo a través de la columna de intercambio de aniones en la columna de intercambio catiónico a 2-5 ml/min. Lave la columna con el búfer A hasta que el A280 vuelva al nivel de línea base. Elute la proteína con un gradiente de 100 ml (0-50% Tampón B) y recoge fracciones de 10 ml.
4. Decir la enzima continuamente usando un lector de placas
- Preparar 50 ml del tampón de ensayo (50 mM HEPES pH 7.0).
- Como las proteasas alfavirales tienen valores degato k relativamente bajos, diluir la enzima en el tampón de ensayo a 4,7 m (esto corresponderá aproximadamente a una A280 a 0,2 para la proteasa VEEV sin Trx).
- Para medir la actividad de la enzima, preparar un stock de sustrato en el tampón de ensayo con una concentración de 185 M; esto corresponderá aproximadamente a un A280 x 9. En 8 tubos de microcentrífuga, prepare las mezclas de reacción que se muestran en la Tabla 2 combinando los volúmenes apropiados del stock de sustrato de 185 m y el tampón. En una tubería de placa de media superficie negra de 96 pocillos de 45 ol de la reacción se mezcla en 3 pocillos (columnas 1, 2, 3). La fila A debe contener la mezcla de reacción [S] a 5 M, y la fila H debe contener la mezcla de reacción [S] a 140 m.
- Ajuste el lector de placas para que detecte simultáneamente fluorescencia a dos longitudes de onda con un ajuste de tubo fotomultiplicador fijo (PMT) (por ejemplo, bajo):
Longitud de onda 1 excitación á 434 nm, emisión a 527 nm
Longitud de onda 2 excitación á 434 nm, emisión a 470 nm - Establezca el tiempo de lectura en 20 min (mide 1 lectura por minuto) y seleccione los pozos que desea leer. Inserte la placa en el lector de placas y mida la velocidad espontánea de hidrólisis durante 20 minutos.
- Ejecute una lectura de punto final de la placa que contiene el sustrato "UNCUT".
NOTA: Estos valores se utilizarán en cálculos de datos posteriores. El promedio de las relaciones de emisión de 3 pozos serán los valores del sustrato "UNCUT" en t a 0 en la Tabla 3. - Retire la placa y la tubería de 5 ml de enzima en cada poca. Lea la placa de nuevo durante 20 minutos con 1 lectura por minuto. Ajuste el lector de placas a los valores absolutos de salida.
NOTA: Para este ensayo, las pendientes serán negativas. Cada pozo contendrá un volumen total de 50 l. - Al final de la lectura, sellar la placa con película para evitar la evaporación. Deje la placa a temperatura ambiente durante la noche para permitir que la enzima corte el sustrato por completo.
- Después de 24 h, retire la película de sellado y realice una lectura de punto final de la placa utilizando la misma PMT que en las placas anteriores. Promedio de estos coeficientes de emisión y entrada en el Cuadro 3 en "CUT". Confirme el escote del sustrato utilizando el ensayo discontinuo SDS-PAGE que se describe a continuación (Paso 5.1.).
- Exporte los datos a una hoja de cálculo. Salida de las unidades de fluorescencia en cada punto de tiempo para las 2 longitudes de onda (Tabla 4).
- Calcular el nmol de sustrato que se ha cortado en el momento t utilizando la ecuación (1) donde X es la relación de emisión (527 nm/470 nm) en un momento dado, neg es la relación de emisión del sustrato "UNCUT" en t a 0, y pos es la relación de emisión del sustrato completamente "CUT" medido después de 24 h de corte (Tabla 3).
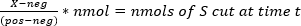
NOTA: Los datos representativos de fluorescencia se muestran para un pozo (bien E7) que contiene un sustrato de 80 m (4 nmol de S por pozo) en la Tabla 4. Los cálculos se realizaron para cada pozo de la placa. - Para cada pocto, trace nmol frente a tiempo (min) y obtenga las velocidades iniciales (pendientes) ajustando los datos a y á mx + b. Para los datos recogidos en 4.1.5, trazar nmol frente a tiempo (min) para cada pozo. La pendiente será igual al producto nmol producido por minuto. Restar las tasas espontáneas de hidrólisis medidas en 4.1.5 de las tasas de reacción catalizadas por enzimas (Tabla 5).
NOTA: La primera lectura se puede recortar a partir de los datos si es artísticamente alta debido al movimiento de la placa en el lector de placas. - Calcular la cantidad de enzima en mg que se añadió a cada pozo (por ejemplo, 0,0009 mg). Una unidad se define como un "mol de producto producido por minuto" (mol/min). Dividir el nmol/min por el mg de enzima presente en el pozo para obtener mU/mg; dividir por 1.000 para obtener U/mg.
- Trazar [S] -M en el eje X y U/mg en el eje Y y ajustar los datos a la ecuación Michaelis-Menten para obtener Vmax y Km. Esto se puede hacer en el software (por ejemplo, GraFit).
5. Decir la enzima de forma discontinua mediante el análisis SDS-PAGE
- Preparar una reacción de 50 l que contenga un sustrato de 10 m y un tampón en lugar de enzimas y etiquetar como "UNCUT."
NOTA: Los volúmenes de sustrato y tampón se muestran en la Tabla 2. Si se ha ejecutado el ensayo continuo, las muestras se pueden utilizar directamente desde la placa de 96 pocillos.- Preparar una reacción de 50 l que contenga un sustrato de 10 m y una enzima de 5 ml y etiquetarla como "CUT". Inicie el temporizador cuando la enzima se añada al sustrato.
NOTA: Los inhibidores se pueden añadir a tubos adicionales que contienen enzima y sustrato. Ajuste el volumen del búfer añadido para compensar el volumen añadido del inhibidor. Las concentraciones de DMSO no deben exceder el 2%. - Incubar las reacciones durante 15-24 h a temperatura ambiente (22 o3 oC). Detenga las reacciones añadiendo 50 s de 2x tampón Laemelli. Después de detener la reacción hierve, cada tubo durante 3-10 min.
- Montar el tanque de gel de acuerdo con las instrucciones del fabricante. Inserte un cassette de gel de poliacrilamida de 17 poveros prefabricado y una presa tampón en el otro lado. Llene el depósito interior de la celda con 1 búfer de funcionamiento SDS hasta que el búfer alcance la parte superior del cassette. Llene el depósito externo medio lleno con el mismo búfer.
- Para analizar el escote utilizando el ensayo discontinuo, cargue 5 ml de cada mezcla de reacción en un carril de un gel SDS-PAGE que comience con la reacción "UNCUT". Incluya un marcador de peso molecular en el primer o último carril.
- Fije los electrodos del tanque de gel a la fuente de alimentación y separe los productos a 110 V durante 60 minutos. Retire el gel del cassette insertando la herramienta de agrietamiento entre las placas. Colocar el gel en una bandeja de plástico y sumergir el gel en 5-10 ml de solución de tinción de gel; bandas serán visibles en un plazo de 30 minutos. Después de 1-24 horas eliminar el exceso de manchas, sumergir el gel en agua y utilizar un imager de gel para tomar una foto del gel.
- Preparar una reacción de 50 l que contenga un sustrato de 10 m y una enzima de 5 ml y etiquetarla como "CUT". Inicie el temporizador cuando la enzima se añada al sustrato.
6. Acoplar los péptidos de sustrato a la proteasa de cisteína VEEV-nsP2
- Descargue el archivo de coordenadas de la proteasa de cisteína VEEV desde el PDB (https://www.rcsb.org/). El código PDB es 2HWK. Guarde el archivo como 2HWK.pdb.
- Preparar la estructura proteica utilizando MOE (https://www.chemcomp.com/). Cargue el archivo PDB de proteína en MOE. Haga clic en Seleccionar y disolvente en la barra lateral derecha y elimine el disolvente.
- Abra el panel Preparación de estructura en la barra de menú superior Proteína. Corrija automáticamente todos los elementos estructurales haciendo clic en Corregir y protonate la estructura haciendo clic en Protonate3D. Agregue cargas parciales a la proteína abriendo el panel Cargas parciales y seleccionando Amber 99 y Ajustar hidrógenos y pares solitarios según sea necesario. Por último, guarde el archivo de estructura como "2HWK_dock.pdb".
- Construir la estructura de los péptidos de sustrato (nsP12, nsP23, nsP34) y TRIM14 utilizando MOE. Abra el panel Constructor de proteínas, introduzca la secuencia de sustrato, establezca la Geometría como Extendida y haga clic en Construir. La estructura se mostrará en la ventana MOE.
- Minimice la estructura del péptido haciendo clic en Minimizar en el panel. Guarde la estructura como un archivo PDB (Figura 6).
- Acople los péptidos de sustrato a VEEV-nsP2 utilizando PyRx/AutoDock 4.2 (http://autodock.scripps.edu/). Abra la herramienta PyRx, edite el ajuste de preferencia, desactive todas las torsiones para preparación de ligando. Cargue la molécula de sustrato, haga clic con el botón derecho en el nombre de la molécula en el panel Navegador, seleccione Hacer ligando para preparar el archivo de acoplamiento de ligando. Cargue la proteína 2HWK_clean.pdb y seleccione Crear macromolécula para preparar el archivo de acoplamiento pdbqt (Figura 7).
- Inicie el Asistente de AutoDock en el panel de acoplamiento en la parte inferior. Seleccione los archivos de ligando y proteína sprepared. Defina el bolsillo de unión a proteínas ajustando manualmente la dimensión de la rejilla que se centra en el residuo catalítico Cys-477. Usando el parámetro de espaciado predeterminado 0.375 . Haga clic en Ejecutar AutoGrid para generar mapas de cuadrícula.
- Ejecute AutoDock y seleccione el método Lamarckian Genetic Algorithm (LGA). Haga clic en los parámetros de acoplamiento y establezca el número de ejecuciones de GA en 50. Utilice los parámetros predeterminados para otros. Haga clic en Reenviar para iniciar la ejecución de acoplamiento.
- Abra el panel Analizar resultados. Inspeccione todas las posturas de enlace previstas. Seleccione el mejor modelo con la energía de unión más baja pronosticada y las interacciones de unión razonables entre el Cys-477 y el sustrato en el sitio de escisión. Guarde el modelo de enlace como archivo PDB para obtener más simulaciones de MD.
7. Simulaciones MD de complejos de sustrato VEEV acoplados
- Prepare los archivos de entrada utilizando Amber (http://ambermd.org/). Siguiendo el protocolo estándar, se realizan simulaciones MD para los modelos de unión de sustrato s–esp. previstos utilizando el paquete AMBER y el campo de fuerza ff99SB.
NOTA: Los sistemas solvados se someten a una minimización de energía exhaustiva antes de las simulaciones de MD. Las condiciones de contorno periódicas se aplican para simular un sistema continuo. El método de malla de partículas Ewald (PME) se empleó para calcular las interacciones electrostáticas de largo alcance. El sistema simulado fue sometido primero a un aumento gradual de la temperatura de 0 K a 300 K sobre 100 ps, y luego equilibrado durante 500 ps a 300 K, seguido de tiradas de producción de 2 ns de longitud en total.- Ejecute el trabajo de simulación en una instalación informática de alto rendimiento. Nuestras simulaciones se ejecutaron en el clúster de Biowulf (https://hpc.nih.gov/) (Figura 8).
- Visualice la salida de trayectoria mediante el programa VMD (http://www.ks.uiuc.edu/Research/vmd/). Analizar las interacciones de unión y los cambios de conformación de los sustratos y TRIM14 dentro del sitio activo de nsP2(Figura 9).
Resultados
El análisis SSHHPS de la proteasa ZIKV ns2B/3 identificó 4 dianas de proteína huésped: FOXG1, SFRP1, una subunidad alfa Gs de una biblioteca de ADNc de retina, y la midtocondrial NT5M 5',3'-nucleotidase (Figura 10)6. En particular, ningún otro método predijo estas proteínas como objetivos potenciales de la proteasa ZIKV. Las mutaciones en el gen FOXG1 se han relacionado con un síndrome congénito caracterizado por deterioro del desarrollo y anomalías cerebrales estructurales como la microcefalia. SFRP1 es una proteína secreta relacionada con el encrespado (SFRP); estos receptores solubles pueden unir competitivamente los ligandos Wnt para antagonizar e inhibir la señalización Wnt. La vía de señalización Wnt participa en la regulación de la respuesta IFN durante la infección por Flavivirus36. Se espera que el escote de SFRP1 mejore la replicación de flavivirus. SFRP1 también participa en la diferenciación de células Th1737. Las alineaciones de secuencia del SSHHPS mostraron diferencias específicas de especies en las secuencias de sitios de escisión(Figura 10D). La secuencia del sitio de escisión en SFRP1 era idéntica en humanos y pollos; ZIKV puede inducir mortalidad y microcefalia en embriones de pollo38. En los roedores, el residuo P1 altamente conservado (K/R)R-G se sustituye por una glicina (RGG). Las cepas inmunocompetentes de ratones son generalmente resistentes a la infección por ZIKV y a la enfermedad39.
Los parámetros cinéticos en estado estacionario y las constantes de inhibición se pueden medir para las secuencias de poliproteínas virales y para las secuencias de proteínas anfitrionas utilizando el ensayo continuo en un lector de placas31,40,41 (Figura 11A). Para obtener información cualitativa sobre el escote, como el escote de una secuencia determinada o la inhibición de la proteasa por diversos compuestos, se puede utilizar el ensayo discontinuo(Figura 11B).
Puede ser necesaria la optimización del número de residuos entre CFP y YFP. Se puede hacer un modelo ligado al sustrato utilizando los métodos in silico. En la Figura 9se muestra un modelo acoplado representativo del cruce nsP1/nsP2. Para la proteasa VEEV nsP2, se había notificado el escote de una secuencia del virus del bosque de Semliki (SFV) de 12 aminoácidos (Km a 0,58 mM)33. Alargar la secuencia del sustrato a 19, 22 y 25 residuos y reducir la resistencia iónica del tampón condujo a una reducción significativa de Km. El examen de la estructura cristalina y el embalaje de cristal ESEV nsP2 también mostró que una parte de una de las uniones estaba empaquetada contra el dominio de la proteasa y era helicoidal. Por lo tanto, los sustratos VEEV más largos pueden unirse mejor debido al reconocimiento de un motivo estructural secundario.
Para TRIM14, obtuvimos un Km a 21 M6,33. La Km para el sustrato que transporta la secuencia de proteína sornte fue comparable a los valores DeK m de los sustratos que contienen las secuencias de sitio de escisión de poliproteína viral (Km(V12) a 12 m y Km(V34) a 21 oM). Las secuencias de sitio de escote en las uniones nsP1/nsP2, nsP2/nsP3 y nsP3/nsP4 se cortaron con diferentes eficiencias. En la célula, esto se piensa para permitir el escote secuencial de la poliproteína42.
Se debe tener precaución al interpretar los resultados negativos. Si no se produce escote, el sitio de escote puede ser demasiado corto o la proteasa purificada puede estar inactiva. Para los sustratos que se cortan, se necesitan experimentos adicionales para confirmar la escisión de la proteína de longitud completa o el escote en las células infectadas por virus. Deben elegirse los experimentos de seguimiento adecuados. También se pueden probar los efectos de la sobreexpresión o silenciamiento de la proteína diana en la replicación viral.
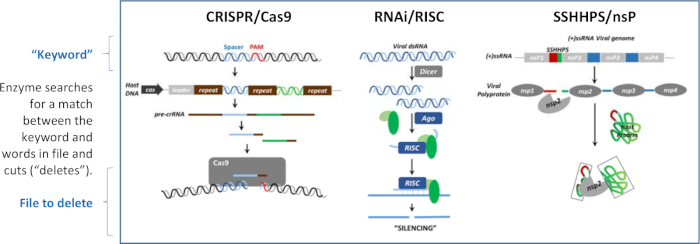
Figura 1: Tres mecanismos de silenciamiento. El silenciamiento puede ocurrir a nivel de ADN, ARN o proteína. Estos algoritmos de "búsqueda y eliminación" utilizan cada uno una "palabra clave" para dirigir el escote de un archivo que contiene la palabra. Esta cifra ha sido modificada de Morazzani et al.32 y sus referencias. Haga clic aquí para ver una versión más grande de esta figura.

Figura 2: Diferencias específicas de especies en secuencias de sitios de escote. Los dominios PRY/SPRY del terminal C de los homólogos TRIM14 se muestran en la alineación. El dominio PRY/SPRY se puede identificar por los motivos conservados resaltados en gris. Human TRIM14 se corta en QEAGA-G por la proteasa de cisteína VEEV nsP2. La secuencia SSHHP se muestra en color. El residuo en verde es el residuo P1'; en azul es el residuo P4, y en rojo son otros residuos conservados dentro de la secuencia de motivos del sitio de escote. Equine harbor una versión truncada de TRIM14 que carece del dominio PRY/SPRY. La lisina resaltada en cian es poli-ubiquitinada y es importante para el montaje del signalosoma MAVS. El dominio C-terminal PRY/SPRY puede ser cortado transitoriamente por la proteasa nsP2 para afectar la respuesta antiviral del huésped intracelularmente durante una infección viral aguda. En equino, este dominio siempre está ausente. Esto sugiere que el dominio PRY/SPRY de TRIM14 puede tener una función protectora contra las infecciones por EVE. Esta figura ha sido reproducida de Morazanni et al.6Haga clic aquí para ver una versión más grande de esta figura.

Figura 3: Identificación SSHHPS mediante BLAST. La secuencia de motivos del sitio de escote en la unión VEEV nsP1/nsP2 se alinea con la secuencia SSHHP en la proteína huésped TRIM14. El residuo coloreado en verde es el residuo P1'; en azul es el residuo P4 y en rojo son otros residuos conservados de la secuencia de motivos del sitio de escote. La mayoría de las alineaciones contenían homología con regiones fuera del motivo del sitio de escisión conservado o no incluían los residuos de la unión escisil del P1/P1'. TRIM14 mostró una coincidencia con 6 residuos en orden secuencial que incluía N1 y P1'. Haga clic aquí para ver una versión más grande de esta figura.

Figura 4: Secuencias de proteínas y ADN del sustrato CFP-V12-YFP para la proteasa de cisteína vev nsP2. Los sitios de restricción NdeI (CATATG) y XhoI (CTCGAG) se muestran en mayúsculas. En rojo es la secuencia del sitio de escote de la poliproteína viral que está entre nsP1 y nsP2. El residuo en verde es el residuo P1' y en azul es el residuo P4 del sitio de escote. Haga clic aquí para ver una versión más grande de esta figura.

Figura 5: Secuencia de proteínas de la construcción de proteasa de cisteína Trx-VEEV-nsP2. La tioredoxina (Trx) se muestra en amarillo. El sitio de escote de trombina y su etiqueta se muestran en cian. Los Cys-Su dyad están etiquetados en rojo. Haga clic aquí para ver una versión más grande de esta figura.
Figura 6: Estructuras de péptidos en MOE. Haga clic aquí para ver una versión más grande de esta figura.
Figura 7: Acoplamiento del péptido de sustrato utilizando PyRx/AutoDock. Haga clic aquí para ver una versión más grande de esta figura.
Figura 8: Trabajos que se ejecutan en el clúster de Biowulf. Haga clic aquí para ver una versión más grande de esta figura.
Figura 9: Modelo del sustrato VEEV P12 que contiene la secuencia del sitio de escisión en la unión nsP1/nsP2. El diadido catalítico Cys-477/His-546 se muestra en azul. Figura se hizo usando Pymol (https://pymol.org). Haga clic aquí para ver una versión más grande de esta figura.
Figura 10: Análisis SSHHPS del virus del Zika ns2B/ns3 proteasa. (A) Objetivos de proteína huésped previstas de la proteasa ZIKV ns2B/ns3. Los residuos en rojo coinciden con una sola secuencia de sitio de escote. Los residuos en verde se toleran en el subsitio y coinciden con los residuos en otros sitios de escote. SFRP1 tenía el mayor número de residuos idénticos en orden consecutivo. (B) las proteínas CFP-sustrato-YFP (50-60 kDa) se expresaron y purificaron con teniendo la secuencia SSHHP pronosticada de cada proteína huésped (humana). La proteasa ZIKV corta la subunidadalfade corte de proteasa DE ZIKV a isla aislada de una biblioteca de ADNc. Los productos de escote son de aproximadamente 28-30 kDa. Las secuencias de sustrato están disponibles en Morazzani et al.6 (C) Mientras que el corte proteasa ns2B/ns3 SFRP1, no cortó sus homólogos (SFRP2 y SFRP4). (D) La alineación de los sitios de escisión de diferentes especies animales puede ser útil para seleccionar un modelo animal para un virus del Grupo IV. Tenga en cuenta que la secuencia R-G conservada difiere entre humanos y roedores en SFRP1. 6Haga clic aquí para ver una versión más grande de esta figura.
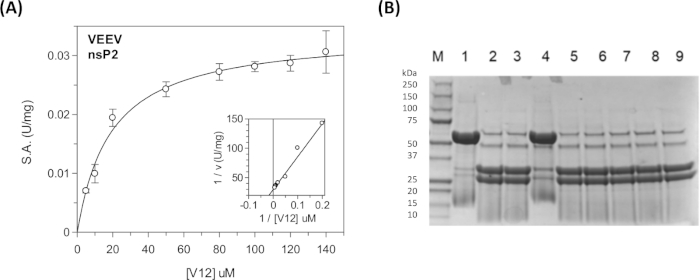
Figura 11: Análisis cinético en estado estacionario utilizando los ensayos continuos y discontinuos. (A) Los datos cinéticos mostrados en la Tabla 5 se trazaron en GraFit. El recuadro muestra la gráfica Lineweaver-Burk. (B) Gel SDS-PAGE que muestra los productos de escisión del sustrato CFP-V12-YFP. En el carril 1 se encuentra el sustrato "UNCUT" (48 kDa). En el carril 2 se encuentra el sustrato "CUT" (31 kDa y 27 kDa). En los carriles 3-9 diferentes compuestos fueron incluidos para probar su actividad inhibitoria. El carril 4 contiene el inhibidor covalente E64d. Estas reacciones se ejecutaron durante la noche durante 17 h a temperatura ambiente. La ebullición de las muestras era necesaria para lograr el patrón de bandas afiladas. La proteasa nsP2 es visible (56 kDa) en las reacciones que contienen enzima, pero no en el carril 1. El carril 1 es el control "sin enzimas". Haga clic aquí para ver una versión más grande de esta figura.
Discusión
La destrucción específica de una proteína o un ácido nucleico guiado por una secuencia extraña sólo se ve en unos pocos casos en biología. Los mecanismos que se muestran en la Figura 1 son mecanismos defensivos que protegen a un host de un virus o un virus de un host.
Utilizando métodos bioinformáticos podemos identificar los objetivos que son destruidos por estos sistemas. En nuestros análisis de secuencias SSHHP, descubrimos que muchas de ellas se podían encontrar en las proteínas necesarias para generar respuestas inmunitarias innatas. Algunos tenían funciones obvias como MAVS y TRIF (TIR-domain-containing adapter-inducing interferon-- , mientras que otros estaban relacionados con la inmunidad a través de mecanismos más complejos (por ejemplo, Histone H3, SFRP1, FOXG1)8,9. La información de destino almacenada en la secuencia SSHHP tiene el potencial de identificar vías que tienen efectos antivirales contra estos virus. Las respuestas antivirales in vivo suelen ser específicas del virus26,43. Por ejemplo, subconjuntos de proteínas TRIM tienen efectos antivirales en diferentes virus43,44,45, algunos son factores de restricción viral (por ejemplo, VIH y TRIM5). La especificidad de las proteínas TRIM (se han identificado 70 euros) todavía se está examinando44,45. La información dentro de SSHHPS puede contribuir a nuestra comprensión de cómo estos virus evaden las respuestas inmunitarias innatas. Otros patrones y correlaciones pueden ser descubiertos a medida que se examinan más SSHHPS.
Las diferencias específicas de las especies fueron evidentes en nuestros análisis(Figura 2, Figura 10). Se sabe que estos virus afectan a algunas especies más que a otras. La información sobre el rango de host, la susceptibilidad del host y las defensas del host puede estar presente en SSHHPS. Por ejemplo, equino, la especie más susceptible a los virus de la encefalitis equina, carecía de la región de TRIM14 humano que fue cortada transitoriamente por la proteasa VEEV nsP2. Los seres humanos rara vez mueren a causa de infecciones por VEEV, pero pueden estar infectados24. La proteína humana TRIM14 llevaba una secuencia de escote de proteasa nsP26. La presencia del sitio de escisión sugiere que los seres humanos tienen un mecanismo de defensa contra estos virus. Se ha pensado que las aves son posibles reservorios de estos virus46. La secuencia SSHHP correspondiente en la proteína TRIM14 de los pollos difería de las secuencias encontradas en humanos y otras especies. Las diferencias sutiles como estas pueden hacer que una proteína huésped objetivo ativable o más fácilmente se cierre. 16 mostraron que una proteína STRING mutada inducible indujo niveles más altos de IFN después de la infección por el virus del dengue y que los ratones llevan naturalmente una versión de STING que no es cortada por la proteasa dengue ns2B3. La proteína murina STING no fue cortada por la proteasa ZIKV47. En nuestro análisis SSHHPS, también observamos diferencias en las secuencias de sitio de escisión de proteasa ZIKV cuando comparamos las proteínas humanas con las de los roedores6 (Figura 10D). La reproducción de las escotes proteolíticas específicas de la especie de proteínas huésped puede ser importante en los modelos animales utilizados para los virus del Grupo IV. La inhibición del escote de proteína huésped también tiene implicaciones con respecto al desarrollo de inhibidores de la proteasa del Grupo IV. En nuestra publicación anterior, mostramos que podríamos inhibir el escote TRIM14 por la proteasa VEEV nsP2 usando el éster metilo CA0746. Este resultado sugiere que los inhibidores de moléculas pequeñas de estas proteasas pueden ser capaces de modular las respuestas inmunitarias innatas que son capaces de suprimir la infección6,31.
La variación genética dentro de una especie también tiene el potencial de producir diferencias en el escote proteolítico. Diferencias sutiles en el uso de codón podrían afectar a la pausa ribosoma48. Dado que algunas proteasas virales del Grupo IV están incrustadas en la membrana er, las diferencias en estas pausas podrían afectar a la escisión de un objetivo si la escisión se produce de forma co-traduccional. Algunos de los sitios de escisión que identificamos estaban en secuencias de péptidos de señal pronosticadas (por ejemplo, SFRP1) mientras que otros eran internos.
El análisis SSHHPS puede producir información que difiere de otros métodos de análisis de proteínas huésped. El análisis SSHHPS era barato y fácil de emplear. El uso de un sistema de expresión bacteriana permitió la prueba de segmentos cortos (25 aminoácidos) de secuencias de mamíferos sin el uso del cultivo celular de mamíferos. Encontramos que los sustratos de CFP-YFP eran capaces de tolerar todas las secuencias de proteínas humanas probadas; sin embargo, los rendimientos variaron. En ensayos similares, sustratos que contienen secuencias de proteínas humanas hasta 63 aminoácidos fueron expresados, purificados y utilizados con éxito para análisis cinéticos y cribado de inhibidores49,50,51. Dado que sólo se necesitan pequeñas cantidades del sustrato para el ensayo discontinuo, se puede explorar un gran número de objetivos. Una ventaja del sistema es que los sustratos CFP/YFP se pueden utilizar para análisis SDS-PAGE y para análisis cinéticos más elaborados (es decir, IC50,Ki, Km, Vmáx.). Para el descubrimiento de fármacos, los compuestos inhibitorios pueden producir artefactos en ensayos fluorescentes. Por lo tanto, el ensayo discontinuo en combinación con un ensayo continuo permite confirmar el escote o la inhibición del escote. Las muestras para el ensayo discontinuo SDS-PAGE se pueden sacar directamente de las placas de 96 pocillos. Los sustratos CFP/YFP se han utilizado para el cribado de bibliotecas compuestas52. Sin embargo, se requieren análisis adicionales para determinar si un sustrato es adecuado para la detección de alto rendimiento, como el cálculo de un factor Z53.
Un desafío en el diseño de un sustrato es identificar la región alrededor de la unión escisil que está unida y reconocida por la proteasa. En los ejemplos que se muestran aquí, comenzamos con 12 secuencias de residuos que se centraron alrededor de la unión escisil. Después de analizar las alineaciones de secuencia de los sitios de escote se encontró la homología a los residuos N-terminal de la unión escisil para la proteasa VEEV, mientras que para la homología proteasa ZIKV a varios de los residuos C-terminal se encontró. Un modelo in silico del sustrato acoplado se puede utilizar para diseñar experimentos de mutagénesis dirigidos por el sitio que sondean los sitios de unión del sustrato. Dado que las secuencias de sustrato y enzimas están en plásmidos, cualquiera de los dos puede ser mutado para probar los modelos in silico o tolerancias subsitio. Esto puede ser ventajoso si una estructura cristalina del sustrato (s) unido (s) no está disponible.
El análisis SSHHPS también puede producir nueva información sobre los mecanismos por los cuales los fenotipos inducidos por virus son producidos por enzimas virales. Uno de los objetivos de ZIKV, SFRP1, es parte de la vía de señalización Wnt y tiene funciones tanto en el desarrollo del cerebro como de los ojos y en las respuestas inmunitarias36,37,54,55,56,57. Encontramos que las otras secuencias de proteínas que podrían ser cortadas por la proteasa ZIKV ns2B/ns3 también estaban en proteínas involucradas en el desarrollo del cerebro y los ojos; se han observado anomalías en ambos en el síndrome de Zika congénito y se cree que forman parte del fenotipo58inducido por el virus.
La previsibilidad de las interacciones huésped-patógeno podría ser aprovechada para una variedad de aplicaciones: terapias virales oncolíticas específicas de objetivos; desariesgo rinde riesgo a las vacunas contra los virus vivos; perfeccionamiento, predicción o selección de modelos animales; predicción del alcance del host o susceptibilidad; predicción de eventos zoonóticos; y la predicción de las defensas de los anfitriones. Dado que los métodos descritos se basan en secuencias, pueden ser de valor para incorporar en el software en el futuro.
Divulgaciones
Las opiniones expresadas aquí son las de los autores y no representan las de la Marina de los Estados Unidos, el Ejército de los Estados Unidos, el Departamento de Defensa de los Estados Unidos o el gobierno de los Estados Unidos.
Agradecimientos
Este trabajo fue apoyado por los números de proyecto de la Agencia de Reducción de Amenazas de Defensa (DTRA) CB-SEED-SEED09-2-0061 y CBCall4-CBM-05-2-0019, y en parte por el programa de investigación intramuros/extramuros de los fondos base NCATS, NIH (XH) y del Laboratorio de Investigación Naval.
Materiales
| Name | Company | Catalog Number | Comments |
| 250 mL Erlenmeyer Flask | VWR | 89000-362 | |
| 2-mercaptoethanol | Acros Organics (Fisher) | 125472500 | Danger: Acutely Toxic. Open bottle in hood to avoid inhaling the fumes. |
| 4 L Pyrex wide-mouth graduated Erlenmeyer flask with screw-cap | Millipore Sigma | CLS49954L-1EA | |
| AKTA Prime Plus | GE Healthcare | 17-0729-01 | |
| AKTA XK 16/20 Column | GE Healthcare | 28988937 | |
| Amicon Ultra-0.5 Centrifugal Filter Unit | Millipore Sigma | UFC501096 | |
| Amicon Ultra-15 Centrifugal Filter Unit | Millipore Sigma | UFC901096 | |
| Amicon Ultra-4 Centrifugal Filter Unit | Millipore Sigma | UFC801024 | |
| Ampicillin | Sigma | A0166 | Danger: Allergic reactions (skin or breathing). |
| Chelating Sepharose Fast Flow | GE Healthcare | 17-0575-02 | Once the resin is equilibrated with 0.2 M Nickel Sulfate it is refered to as a Nickel Column in the text. Column will have a green color after washing with water. The column will have a blue color after equilibrating with buffer. |
| Chloramphenicol | RPI | C61000 | Danger: May cause cancer. |
| Corning 50 mL centrifuge tubes | Corning | 430828 | Suggestion: Polypropylene tubes are less likely to crack during sonication than Polyethylene tubes |
| Corning 96 Well Half-Area Microplate, Non-Binding Surface | Corning | 3993 | |
| Dialysis Tubing Clips | Fisher Scientific | PI68011 | |
| Disposable PD-10 Desalting Column | GE Healthcare | 17-0851-01 | |
| DNAse | Sigma | DN25-1G | |
| DTT (DL-Dithiothreitol) | RPI | D11000-50.0 | Warning: Acute Oral Toxicity; skin and eye irritation |
| EDTA | Fisher Scientific | S311-500 | |
| Fisherbrand Petri Dishes with Clear Lid | Fisher Scientific | FB0875712 | |
| Glycerol | Acros Organics (Fisher) | 15892-0010 | |
| HEPES | Millipore Sigma | H4034-1KG | |
| Imidazole | Acros Organics | 301870010 | Danger: Toxic, Irritant |
| IPTG (Isopropyl β-D-thiogalactopyranoside) | Calbiochem (Millipore Sigma) | 420291 | Do not breathe dust. Avoid contact with eyes and skin. |
| Laemmli Sample Buffer | BIO-RAD | 1610737 | |
| Luria Bertani Agar | Fluka (Millipore Sigma) | L3027-1KG | Suggestion: Autoclave with magnetic stirrer in the liquid, and stir while cooling. Wait to add antibiotic until you can hold your hands on the bottle without pain for 30 seconds. |
| Luria Bertani Media | Fisher Bioreagents | BP1426-2 | |
| Lysozyme | Sigma | L4919-5g | |
| Mini-PROTEAN Tetra Vertical Electrophoresis CellGel Box | BIO-RAD | ||
| Nalgene Oak Ridge High-Speed PPCO Centrifuge Tubes | Nalgene (Thermo Scientific) | 3119-0050 | |
| Nanodrop | Thermo Fisher | ||
| New Brunswick Innova 42R Shaker Incubator | Eppendorf | M1335 | |
| Nickel Sulfate Hexahydrate (Crystalline/Certified ACS), Fisher Chemical | Fisher Scientific | N73-500 | Danger: Harmful if swallowed or inhaled, skin and eye irritation |
| One Shot BL21(DE3) Chemically Competent E. coli | Invitrogen (Thermo Fisher) | C600003 | May be harmful if inhaled or swallowed. May cause skin and eye irritation with susceptible people. |
| One Shot BL21(DE3) pLysS Chemically Competent E. coli | Invitrogen (Thermo Fisher) | C606003 | May be harmful if inhaled or swallowed. May cause skin and eye irritation with susceptible people. |
| pet15b plasmid DNA | Novagen (Millipore Sigma) | 69661 | GenScript Inc. was used for commerical DNA synthesis. The pet15b plasmid was used for the CFP/YFP substrates. |
| pet32b | Novagen (Millipore Sigma) | 69016-3 | The pet32b plasmid was used for the cysteine protease construct. |
| Pierce Protease Inhibitor Mini Tablets, EDTA-free | Thermo Fisher | A32955 | Warning: Skin corrosion/irriation; eye damage |
| Plate Reader | Molecular Devices | Model M5 | |
| Precision Plus Protein All Blue Prestained Protein Standard | BIO-RAD | 161-0373 | |
| Protein Extraction Reagent | Novagen (Millipore Sigma) | 70584-4 | BugBuster or Bper (Catalog # 78248, ThermoFisher) |
| Q-Sepharose Fast Flow | G.E. Healthcare | 17-0510-01 | Anion exchange resin |
| RunBlue (12%) 17-well PAGE gels | Expedeon | BCG01227 | Any 12% pre-cast polyacrylamide gel can be used |
| RunBlue 20x SDS Running Buffer | Expedeon | NXB50500 | Dilute 50 mL with 950 mL deionized water to obtain 1x |
| RunBlue Instant Blue Gel Stain | Expedeon | ISB1L | Do not dilute, use as directed |
| Sodium Chloride | Fisher Chemical | S271-10 | |
| Sonifier Cell Disrupter 450 Sonicator | Branson Ultrasonics (VWR) | Model No. 101-063-346R | Sonicator was used on level 5 |
| Spectra/Por 6-8 kD MWCO | Spectrum Labs | 132645T | Dialysis Tubing |
| SP-Sepharose Fast Flow | G.E. Healthcare | 17-0729-01 | Cation exchange resin |
| Thrombin from bovine plasma | Sigma | T6634-500UN | |
| Tris Base | Fisher Scientific | BP152-500 | Caution: Eye/Skin Irritant |
Referencias
- Liu, H., et al. Widespread Horizontal Gene Transfer from Double-Stranded RNA Viruses to Eukaryotic Nuclear Genomes. Journal of Virology. 84 (22), 11876-11887 (2010).
- Hagai, T., Azia, A., Babu, M. M., Andino, R. Use of host-like peptide motifs in viral proteins is a prevalent strategy in host-virus interactions. Cell Reports. 7 (5), 1729-1739 (2014).
- Gorbalenya, A. E. Host-related sequences in RNA viral genomes. Seminars in Virology. 3, 359-371 (1992).
- Shmakov, S. A., et al. The CRISPR Spacer Space Is Dominated by Sequences from Species-Specific Mobilomes. MBio. 8 (5), 1-18 (2017).
- Legler, P. M., Morazzani, E., Glass, P. J., Compton, J. R. Proteome Editing System and A Biomarker of Veev Infection. United States patent application. , (2018).
- Morazzani, E. M., et al. Proteolytic cleavage of host proteins by the Group IV viral proteases of Venezuelan equine encephalitis virus and Zika virus. Antiviral Research. 164, 106-122 (2019).
- Alvarez, E., Castello, A., Menendez-Arias, L., Carrasco, L. HIV protease cleaves poly(A)-binding protein. Biochemical Journal. 396 (2), 219-226 (2006).
- Falk, M. M., et al. Foot-and-mouth disease virus protease 3C induces specific proteolytic cleavage of host cell histone H3. Journal of Virology. 64 (2), 748-756 (1990).
- Grigera, P. R., Tisminetzky, S. G. Histone H3 modification in BHK cells infected with foot-and-mouth disease virus. Virology. 136 (1), 10-19 (1984).
- Li, W., Ross-Smith, N., Proud, C. G., Belsham, G. J. Cleavage of translation initiation factor 4AI (eIF4AI) but not eIF4AII by foot-and-mouth disease virus 3C protease: identification of the eIF4AI cleavage site. FEBS Letters. 507 (1), 1-5 (2001).
- Kuyumcu-Martinez, M., et al. Calicivirus 3C-like proteinase inhibits cellular translation by cleavage of poly(A)-binding protein. Journal of Virology. 78 (15), 8172-8182 (2004).
- Pietila, M. K., Hellstrom, K., Ahola, T. Alphavirus polymerase and RNA replication. Virus Research. 234, 44-57 (2017).
- Hardy, W. R., Strauss, J. H. Processing the nonstructural polyproteins of sindbis virus: nonstructural proteinase is in the C-terminal half of nsP2 and functions both in cis and in trans. Journal of Virology. 63 (11), 4653-4664 (1989).
- Strauss, E. G., De Groot, R. J., Levinson, R., Strauss, J. H. Identification of the active site residues in the nsP2 proteinase of Sindbis virus. Virology. 191 (2), 932-940 (1992).
- Wang, D., et al. Foot-and-mouth disease virus 3C protease cleaves NEMO to impair innate immune signaling. Journal of Virology. 86 (17), 9311-9322 (2012).
- Aguirre, S., et al. DENV inhibits type I IFN production in infected cells by cleaving human STING. PLoS Pathogens. 8 (10), e1002934(2012).
- Barral, P. M., Sarkar, D., Fisher, P. B., Racaniello, V. R. RIG-I is cleaved during picornavirus infection. Virology. 391 (2), 171-176 (2009).
- Elbashir, S. M., Lendeckel, W., Tuschl, T. RNA interference is mediated by 21- and 22-nucleotide RNAs. Genes & Development. 15 (2), 188-200 (2001).
- Deveau, H., Garneau, J. E., Moineau, S. CRISPR/Cas system and its role in phage-bacteria interactions. Annual Review of Microbiology. 64, 475-493 (2010).
- Schechter, I., Berger, A. On the size of the active site in proteases I. Papain. Biochemical and Biophysical Research Communications. 27 (2), 157-162 (1967).
- Bieniasz, P. D. Intrinsic immunity: a front-line defense against viral attack. Nature Immunology. 5 (11), 1109-1115 (2004).
- Zhou, Z., et al. TRIM14 is a mitochondrial adaptor that facilitates retinoic acid-inducible gene-I-like receptor-mediated innate immune response. Proceedings of the National Academy of Sciences of the U S A. 111 (2), E245-E254 (2014).
- Wang, S., et al. TRIM14 inhibits hepatitis C virus infection by SPRY domain-dependent targeted degradation of the viral NS5A protein. Scientific Reports. 6, 32336(2016).
- Zacks, M. A., Paessler, S. Encephalitic alphaviruses. Veterinary Microbiology. 140 (3-4), 281-286 (2010).
- Hollidge, B. S., Weiss, S. R., Soldan, S. S. The role of interferon antagonist, non-structural proteins in the pathogenesis and emergence of arboviruses. Viruses. 3 (6), 629-658 (2011).
- Carthagena, L., et al. Human TRIM gene expression in response to interferons. PLoS One. 4 (3), e4894(2009).
- Montgomery, S. A., Johnston, R. E. Nuclear import and export of Venezuelan equine encephalitis virus nonstructural protein 2. Journal of Virology. 81 (19), 10268-10279 (2007).
- Nenasheva, V. V., et al. Enhanced expression of trim14 gene suppressed Sindbis virus reproduction and modulated the transcription of a large number of genes of innate immunity. Immunologic Research. 62 (3), 255-262 (2015).
- Nagai, T., et al. A variant of yellow fluorescent protein with fast and efficient maturation for cell-biological applications. Nature Biotechnology. 20 (1), 87-90 (2002).
- Li, M. Z., Elledge, S. J. SLIC: a method for sequence- and ligation-independent cloning. Methods in Molecular Biology. 852, 51-59 (2012).
- Hu, X., et al. Kinetic, Mutational, and Structural Studies of the Venezuelan Equine Encephalitis Virus Nonstructural Protein 2 Cysteine Protease. Biochemistry. 55 (21), 3007-3019 (2016).
- Morazzani, E. M., et al. Proteolytic cleavage of host proteins by the Group IV viral proteases of Venezuelan equine encephalitis virus and Zika virus. Antiviral Research. 164, 106-122 (2019).
- Zhang, D., Tozser, J., Waugh, D. S. Molecular cloning, overproduction, purification and biochemical characterization of the p39 nsp2 protease domains encoded by three alphaviruses. Protein Expression and Purification. 64 (1), 89-97 (2009).
- Lei, J., et al. Crystal structure of Zika virus NS2B-NS3 protease in complex with a boronate inhibitor. Science. 353 (6298), 503-505 (2016).
- Shiryaev, S. A., et al. Characterization of the Zika virus two-component NS2B-NS3 protease and structure-assisted identification of allosteric small-molecule antagonists. Antiviral Research. 143, 218-229 (2017).
- Smith, J. L., Jeng, S., McWeeney, S. K., Hirsch, A. J. A MicroRNA Screen Identifies the Wnt Signaling Pathway as a Regulator of the Interferon Response during Flavivirus Infection. Journal of Virology. 91 (8), (2017).
- Lee, Y. S., et al. The Wnt inhibitor secreted Frizzled-Related Protein 1 (sFRP1) promotes human Th17 differentiation. European Journal of Immunology. 42 (10), 2564-2573 (2012).
- Goodfellow, F. T., et al. Zika Virus Induced Mortality and Microcephaly in Chicken Embryos. Stem Cells and Development. 25 (22), 1691-1697 (2016).
- Morrison, T. E., Diamond, M. S. Animal Models of Zika Virus Infection, Pathogenesis, and Immunity. Journal of Virology. 91 (8), (2017).
- Morazzani, E. M., et al. Books of Abstracts, 254th American Chemical Society National Meeting. , Washington, D.C. (2017).
- Compton, J. R., Mickey, M. J., Hu, X., Marugan, J. J., Legler, P. M. Mutation of Asn-475 in the Venezuelan Equine Encephalitis Virus nsP2 Cysteine Protease Leads to a Self-Inhibited State. Biochemistry. 56 (47), 6221-6230 (2017).
- Vasiljeva, L., et al. Regulation of the sequential processing of Semliki Forest virus replicase polyprotein. Journal of Biological Chemistry. 278 (43), 41636-41645 (2003).
- Uchil, P. D., Quinlan, B. D., Chan, W. T., Luna, J. M., Mothes, W. TRIM E3 ligases interfere with early and late stages of the retroviral life cycle. PLoS Pathogens. 4 (2), e16(2008).
- Ozato, K., Shin, D. M., Chang, T. H., Morse, H. C. 3rd TRIM family proteins and their emerging roles in innate immunity. Nature Reviews Immunology. 8 (11), 849-860 (2008).
- van Tol, S., Hage, A., Giraldo, M. I., Bharaj, P., Rajsbaum, R. The TRIMendous Role of TRIMs in Virus-Host Interactions. Vaccines (Basel). 5 (3), (2017).
- Molaei, G., et al. Dynamics of Vector-Host Interactions in Avian Communities in Four Eastern Equine Encephalitis Virus Foci in the Northeastern U.S. PLoS Neglected Tropical Diseases. 10 (1), e0004347(2016).
- Ding, Q., et al. Species-specific disruption of STING-dependent antiviral cellular defenses by the Zika virus NS2B3 protease. Proceedings of the National Academy of Sciences of the U S A. 115 (27), E6310-E6318 (2018).
- Angov, E., Legler, P. M., Mease, R. M. Adjustment of codon usage frequencies by codon harmonization improves protein expression and folding. Methods in Molecular Biology. 705, 1-13 (2011).
- Ruge, D. R., et al. Detection of six serotypes of botulinum neurotoxin using fluorogenic reporters. Analytical Biochemistry. 411 (2), 200-209 (2011).
- Hu, X., et al. Structural insight into exosite binding and discovery of novel exosite inhibitors of botulinum neurotoxin serotype A through in silico screening. Journal of Computer-Aided Molecular Design. 28 (7), 765-778 (2014).
- Dunning, F. M., et al. Detection of botulinum neurotoxin serotype A, B, and F proteolytic activity in complex matrices with picomolar to femtomolar sensitivity. Applied and Environmental Microbiology. 78 (21), 7687-7697 (2012).
- Nguyen, T. G., et al. Development of fluorescent substrates and assays for the key autophagy-related cysteine protease enzyme ATG4B. Assay and Drug Development Technologies. 12 (3), 176-189 (2014).
- Zhang, J. H., Chung, T. D., Oldenburg, K. R. A Simple Statistical Parameter for Use in Evaluation and Validation of High Throughput Screening Assays. Journal of Biomolecular Screening. 4 (2), 67-73 (1999).
- Bovolenta, P., Esteve, P., Ruiz, J. M., Cisneros, E., Lopez-Rios, J. Beyond Wnt inhibition: new functions of secreted Frizzled-related proteins in development and disease. Journal of Cell Science. 121 (Pt 6), 737-746 (2008).
- Esteve, P., et al. SFRPs act as negative modulators of ADAM10 to regulate retinal neurogenesis. Nature Neuroscience. 14 (5), 562-569 (2011).
- Garcia-Hoyos, M., et al. Evaluation of SFRP1 as a candidate for human retinal dystrophies. Molecular Vision. 10, 426-431 (2004).
- Marcos, S., et al. Secreted frizzled related proteins modulate pathfinding and fasciculation of mouse retina ganglion cell axons by direct and indirect mechanisms. Journal of Neuroscience. 35 (11), 4729-4740 (2015).
- Moore, C. A., et al. Characterizing the Pattern of Anomalies in Congenital Zika Syndrome for Pediatric Clinicians. JAMA Pediatrics. 171 (3), 288-295 (2017).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados