Method Article
Analyse von viralen SSHHPS der Gruppe IV mit In-vitro- und Silico-Methoden
In diesem Artikel
Zusammenfassung
Wir präsentieren ein allgemeines Protokoll zur Identifizierung kurzer Strecken homologe Wirt-Pathogen-Proteinsequenzen (SSHHPS), die in das virale Polyprotein eingebettet sind. SSHHPS werden durch virale Proteasen erkannt und leiten die gezielte Zerstörung bestimmter Wirtsproteine durch mehrere Viren der Gruppe IV.
Zusammenfassung
Alphavirale Enzyme werden in einem einzigen Polypeptid synthetisiert. Das nichtstrukturelle Polyprotein (nsP) wird von seiner nsP2-Cysteinprotease verarbeitet, um aktive Enzyme zu produzieren, die für die Virusreplikation unerlässlich sind. Virale Proteasen sind hochspezifisch und erkennen konservierte Spalt-Standort-Motivsequenzen (ca. 6-8 Aminosäuren). In mehreren Virusarten der Gruppe IV finden sich die Motivsequenzen der nsP-Protease(s) In bestimmten Wirtsproteinen, die an der Erzeugung der angeborenen Immunantworten beteiligt sind, und in einigen Fällen scheinen die zielgerichteten Proteine mit dem virusinduzierten Phänotyp in Verbindung zu stehen. Diese Viren nutzen kurze Strecken homologe Wirt-Pathogen-Proteinsequenzen (SSHHPS) zur gezielten Zerstörung von Wirtsproteinen. Zur Identifizierung von SSHHPS können die viralen Proteasespaltungs-Standort-Motivsequenzen in BLAST eingegeben und die Wirtsgenome durchsucht werden. Cleavage kann zunächst mit den gereinigten nsP-Virusprotease- und Fluoreszenzresonanz-Energieübertragungssubstraten (FRET) aus E. coligetestet werden. Die FRET-Substrate enthalten Cyan- und Gelbfluoreszenzprotein und die Spaltungs-Standortsequenz (CFP-Sequenz-YFP). Dieser Protease-Assay kann kontinuierlich in einem Plattenleser oder diskontinuierlich in SDS-PAGE Gelen eingesetzt werden. Modelle der gebundenen Peptidsubstrate können in Silico erzeugt werden, um Substratauswahl- und Mutagenesestudien zu leiten. GFP/YFP-Substrate wurden auch verwendet, um Proteasehemmer zu identifizieren. Diese In-vitro- und Silico-Methoden können in Kombination mit zellbasierten Assays verwendet werden, um festzustellen, ob das Ziel-Wirtsprotein die Virusreplikation beeinflusst.
Einleitung
Beweise für einen horizontalen Gentransfer vom Virus zum Wirt oder wirt zu Virus, können in einer Vielzahl von Genomengefundenwerden 1,2,3,4. Beispiele für virale Endogenisation sind die CRISPR-Abstandssequenzen, die in bakteriellen Wirtsgenomen gefunden werden4. Kürzlich haben wir Hinweise auf Wirtsproteinsequenzen gefunden, die in die nichtstrukturellen Polyproteine von (+)ssRNA Group IV-Viren eingebettet sind. Diese Sequenzen innerhalb der kodierenden Bereiche des viralen Genoms können generationell vermehrt werden. Die kurzen Strecken homologe Wirt-Pathogen-Proteinsequenzen (SSHHPS) finden sich im Virus und Wirt5,6. SSHHPS sind die konservierten Spalt-Standort-Motivsequenzen, die von viralen Proteasen erkannt werden, die Homologie zu bestimmten Wirtsproteinen haben. Diese Sequenzen leiten die Zerstörung bestimmter Wirtsproteine.
In unserer vorherigen Publikation6haben wir eine Liste aller Wirtsproteine zusammengestellt, die von viralen Proteasen angegriffen wurden, und festgestellt, dass die Liste der Ziele nicht zufällig war (Tabelle 1). Zwei Trends waren offensichtlich. Erstens gehörte die Mehrheit der viralen Proteasen, die Wirtsproteine schneiden, zu Denkowerviren der Gruppe IV (24 von 25 Fällen betrafen virale Proteasen der Gruppe IV), und eine Protease gehörte zur (+)ssRNA-Gruppe VI-Retroviren (HIV, Human Immunodeficiency Virus)7. Zweitens waren die Wirtsproteinziele, die von den viralen Proteasen geschnitten wurden, im Allgemeinen an der Generierung der angeborenen Immunantworten beteiligt, was darauf hindeutet, dass die Spaltungen dazu bestimmt waren, die Immunantworten des Wirts zu verankern. Die Hälfte der von den viralen Proteasen ins Visier genommenen Wirtsproteine waren bekannte Bestandteile von Signalkaskaden, die Interferon (IFN) und proinflammatorische Zytokine erzeugen (Tabelle 1). Andere waren an der Hostzellentranskription8,9,10 oder Translation11beteiligt. Interessanterweise haben Shmakov et al.4 gezeigt, dass viele CRISPR Protospacer-Sequenzen Genen entsprechen, die an Derplasmakonjugation oder Replikation beteiligt sind4.
Gruppe IV umfasst unter anderem Flaviviridae, Picornaviridae, Coronaviridae, Calciviridae und Togaviridae. Mehrere neue und neu auftretende Krankheitserreger gehören zur Gruppe IV wie das Zika-Virus (ZIKV), West-Nil (WNV), Chikungunya (CHIKV), das schwere akute Atemwegssyndrom (SARS) und das Atemwegssyndrom -virus (MERS) im Nahen Osten. Das (+)ssRNA-Genom ist im Wesentlichen ein Stück mRNA. Um die für die Genomreplikation notwendigen Enzyme zu produzieren, muss zunächst das (+)ssRNA-Genom übersetzt werden. Bei Alphaviren und anderen Viren der Gruppe IV werden die für die Replikation notwendigen Enzyme in einem einzigen Polyprotein (d. h. nsP1234 für VEEV) hergestellt. Das nichtstrukturelle Polyprotein (nsP) wird proteolytisch verarbeitet (nsP1234 nsP1, nsP2, nsP3, nsP4) durch die nsP2-Protease zur Herstellung aktiver Enzyme12 (Abbildung 1). Die Spaltung des Polyproteins durch die nsP2-Protease ist für die Virusreplikation unerlässlich; Dies wurde durch Deletion und standortgesteuerte Mutagenese der aktiven Stelle Cystein der nsP2-Protease13,14demonstriert. Insbesondere die Translation viraler Proteine geht genomischen Replikationsereignissen voraus. Beispielsweise enthält nsP4 die RNA-abhängige RNA-Polymerase, die zur Replikation des (+)ssRNA-Genoms erforderlich ist. Die Genomreplikation kann dsRNA-Zwischenprodukte erzeugen; Diese Zwischenprodukte können die angeborenen Immunreaktionen des Wirts auslösen. So können diese Viren Wirt angeborene Immunantwort Proteine früh in der Infektion spalten, um ihre Wirkung zu unterdrücken15,16,17.
Silencing kann auf der Ebene von DNA, RNA und Protein auftreten. Was jedem der in Abbildung 1 gezeigten Silencing-Mechanismen gemeinsam ist, ist, dass kurze fremde DNA-, RNA- oder Proteinsequenzen verwendet werden, um die Zerstörung bestimmter Ziele zu steuern, um ihre Funktion zu veranschlagen. Die Silencing-Mechanismen sind analog zu "Suchen und Löschen" von Programmen, die in drei verschiedenen Sprachen geschrieben wurden. Die kurze Spaltungs-Site-Sequenz ist analog zu einem "Schlüsselwort". Jedes Programm hat ein Enzym, das die Übereinstimmung zwischen der kurzen Sequenz (das "Schlüsselwort") und einem Wort in der "Datei" erkennt, das gelöscht werden soll. Sobald eine Übereinstimmung gefunden ist, schneidet das Enzym die größere Zielsequenz ("löscht"). Die drei in Abbildung 1 gezeigten Mechanismen werden verwendet, um den Wirt vor Viren zu schützen oder um ein Virus vor dem Immunsystem eines Hosts zu schützen.
Virale Proteasen erkennen kurze Spalt-Standort-Motivsequenzen zwischen 2-11 Aminosäuren; in Nukleotiden würde dies 6-33 Basen entsprechen. Zum Vergleich: CrispR-Abstandssequenzen sind Nukleotide von 26-72 und RNAi sind Nukleotide18,19. Obwohl diese Sequenzen relativ kurz sind, können sie spezifisch erkannt werden. Angesichts der höheren Vielfalt an Aminosäuren ist die Wahrscheinlichkeit eines zufälligen Spaltungsereignisses für eine virale Protease, die Proteinsequenzen von 6-8 Aminosäuren oder länger erkennt, relativ gering. Die Vorhersage von SSHHPS in Wirtsproteinen wird weitgehend von der Spezifität der untersuchten viralen Protease abhängen. Wenn die Protease strenge Sequenzspezifitätsanforderungen hat, beträgt die Wahrscheinlichkeit, eine Spaltungs-Standortsequenz zu finden, 1/206 = 1 in 64 Millionen oder 1/208 = 1 in 25,6 Milliarden; Die meisten Proteasen weisen jedoch variable Untersitetoleranzen auf (z. B. können R oder K am Standort S1 toleriert werden). Folglich ist keine Sequenzidentität zwischen den Sequenzen, die auf dem Host gefunden werden, und dem Virus erforderlich. Bei viralen Proteasen, die lockerere Sequenzanforderungen haben (z. B. bei Picornaviridae), kann die Wahrscheinlichkeit, eine Spaltungsstelle in einem Wirtsprotein zu finden, höher sein. Viele der Einträge in Tabelle 1 stammen aus der Familie der Picornaviridae.
Schechter & Berger Notation20 wird häufig verwendet, um die Rückstände in einem Proteasesubstrat und die Untersits, an die sie binden, zu beschreiben, verwenden wir diese Notation durchgehend. Die Rückstände im Substrat, die N-Terminal der Sissile-Bindung sind, werden als P3-P2-P1 bezeichnet, während die Rückstände, die C-Terminal sind, als P1'-P2'-P3' bezeichnet werden. Die entsprechenden Untersiten in der Protease, die diese Aminosäurerückstände binden, sind S3-S2-S1 bzw. S1'-S2'-S3'.
Um zu bestimmen, welche Wirtsproteine ins Visier genommen werden, können wir SSHHPS in den viralen Polyproteinspaltungsstellen identifizieren und nach den Wirtsproteinen suchen, die sie enthalten. Hierin skizzieren wir Verfahren zur Identifizierung von SSHHPS mit bekannten viralen Proteasespaltungs-Site-Sequenzen. Die bioinformatischen Methoden, Protease-Assays und in silico-Methoden sollen in Verbindung mit zellbasierten Assays verwendet werden.
Sequenzausrichtungen der Wirtsproteine, die von viralen Proteasen angegriffen werden, haben artspezifische Unterschiede innerhalb dieser kurzen Spalt-Standortsequenzen aufgedeckt. Zum Beispiel wurde festgestellt, dass das venezolanische Pferdeenzephalitisvirus (VEEV) nsP2-Protease das menschliche TRIM14, ein dreigliedriges Motiv (TRIM) Protein6, schneidet. Einige TRIM-Proteine sind virale Restriktionsfaktoren (z.B. TRIM5-21), die meisten gelten vermutlich als Ubiquitin E3-Ligasen. TRIM14 verfügt nicht über eine RING-Domain (wirklich interessantes neues Gen) und wird nicht als E3-Ligase22angesehen. TRIM14 wurde als Adapter in der mitochondrialen antiviralen Signalososome (MAVS)22vorgeschlagen, kann aber andere antivirale Funktionen haben23. Die Ausrichtung der TRIM14-Sequenzen verschiedener Arten zeigt, dass Pferde nicht die Spaltungsstelle haben und eine abgeschnittene Version von TRIM14 beherbergen, die die C-Terminal PRY/SPRY-Domäne vermisst. Diese Domäne enthält eine Polyubiquitinationsstelle (Abbildung 2). Bei Pferden sind diese Viren hochgradig tödlich (20-80% Mortalität), während beim Menschen nur 1% an VEEV-Infektionen sterben24. Die Cleavage der PRY/SPRY-Domäne kann die MAVS-Signalkaskade vorübergehend kurzschließen. Diese Kaskade kann durch dsRNA ausgelöst werden und führt zur Produktion von Interferon und pro-inflammatorischen Zytokinen. Daher kann das Vorhandensein der SSHHPS nützlich sein, um vorherzusagen, welche Arten Abwehrsysteme gegen bestimmte Viren der Gruppe IV haben.
In der Gruppe IV viren werden IFN-Antagonismusmechanismen als multipliziert redundant25angenommen. Die Dekolleté des Wirtskann während der Infektion vorübergehend sein und die Konzentrationen können sich im Laufe der Zeit erholen. Wir fanden in Zellen, dass TRIM14-Spaltungsprodukte sehr früh nach Transfektion (6 h) mit einem Plasmid nachgewiesen werden konnten, das die Protease (Cytomegalievirus-Promotor) kodiert. Länger wurden die Spaltprodukte jedoch nicht nachgewiesen. In virusinfizierten Zellen war die Kinetik unterschiedlich und Spaltprodukte konnten zwischen 6-48 h6nachgewiesen werden. Andere haben das Auftreten von Wirtprotein-Spaltungsprodukte bereits 3-6 h nach der Infektion9,11berichtet.
Proteolytische Aktivität in Zellen ist oft schwer zu fangen; die Spaltprodukte können in ihrer Löslichkeit, Konzentration, Stabilität und Lebensdauer variieren. In zellbasierten Assays kann nicht davon ausgegangen werden, dass sich Spaltprodukte in einer Zelle ansammeln oder dass die Bandintensitäten von geschnittenem und ungeschnittenem Protein kompensatorische Zu- und Abnahmen aufweisen, da das geschnittene Protein sehr schnell abgebaut werden kann und möglicherweise nicht in einem westlichen Fleck bei einem erwarteten Molekulargewicht (MW) nachweisbar ist (z. B. könnte die Region, die das Epitop enthält, von anderen Wirtsproteasen geklammert werden oder ubiiniert werden könnten). Wenn das Substrat der viralen Protease ein angeborenes Immunantwortprotein ist, kann seine Konzentration während der Infektion variieren. Zum Beispiel sind einige angeborene Immunantwortproteine vor einer Virusinfektion vorhanden und werden durch Interferon26weiter induziert. Die Konzentration des Zielproteins kann daher während der Infektion schwanken, und der Vergleich von nicht infizierten vs. infizierten Zelllysaten kann schwer zu interpretieren sein. Darüber hinaus dürfen nicht alle Zellen gleichmäßig transfiziert oder infiziert sein. In-vitro-Protease-Assays mit gereinigten Proteinen aus E. coli haben dagegen weniger Variablen, für die sie zu kontrollieren sind, und solche Assays können mit SDS-PAGE statt mit Immunoblots durchgeführt werden. Kontaminierende Proteasen können in den frühen Schritten der Proteinreinigung des CFP/YFP-Substrats gehemmt werden, und mutierte virale Proteasen können gereinigt und als Kontrollen getestet werden, um festzustellen, ob die Spaltung auf die virale Protease oder eine kontaminierende bakterielle Protease zurückzuführen ist.
Eine Einschränkung der In-vitro-Protease-Assays ist, dass ihnen die Komplexität einer Säugetierzelle fehlt. Damit ein Enzym sein Substrat schneidet, müssen die beiden kolokalisiert sein. Virale Proteasen der Gruppe IV unterscheiden sich in Struktur und Lokalisierung. Zum Beispiel ist die ZIKV-Protease in die endoplasmatische Retikulum -Membran (ER) eingebettet und steht dem Zytosol gegenüber, während die VEEV nsP2-Protease ein lösliches Protein im Zytoplasma und Kern27ist. Einige der in der ZIKV-SSHHPS-Analyse gefundenen Spaltungs-Standortsequenzen waren in Signalpeptiden, die darauf hindeuten, dass Spaltung für einige Ziele ko-translational auftreten könnte. Daher muss bei diesen Analysen auch die Lage der Protease und des Substrats in der Zelle berücksichtigt werden.
Zellbasierte Assays können wertvoll sein, um eine Rolle für das identifizierte Wirtsprotein(e) bei Infektionen zu etablieren. Methoden, die darauf abzielen, die virale Proteasespaltung von Wirtsproteinen zu stoppen, wie die Zugabe eines Proteasehemmers6 oder eine Mutation im Wirtsziel16, können verwendet werden, um ihre Auswirkungen auf die Virusreplikation zu untersuchen. Eine Überexpression des Zielproteins kann auch die Virusreplikation beeinträchtigen28. Plaque-Assays oder andere Methoden können verwendet werden, um die Virusreplikation zu quantifizieren.
Protokoll
1. Bioinformatik: Identifizierung von SSHHPS im Wirtsgenom mit BLAST
HINWEIS: Protein BLAST finden Sie unter blast.ncbi.nlm.nih.gov/Blast.cgi.
- Geben Sie 20 Aminosäuren um die sissile Bindung in das virale Polyprotein. Wählen Sie nicht redundante Proteinsequenzen aus und geben Sie das zu durchsuchende Wirtsgenom ein (z. B. Homo sapiens).
- Wählen Sie bei Bedarf PHI-BLAST aus. Geben Sie eine Mustersequenz ein (z. B. geben Sie für die unten gezeigten 25 Rückstände von V12 das Muster "AG" ohne Anführungszeichen ein).

HINWEIS: In PHI-BLAST weisen eckige Klammern [XY] darauf hin, dass sich die Aminosäure X oder Y an der Untersesite befinden kann (z. B. AG[AC][GAY]). - Überprüfen Sie die BLAST-Ergebnisse und identifizieren Sie die Treffer mit hoher Sequenzidentität zu Rückständen, die in den Polyproteinspaltungsstellen konserviert werden (z. B. dreigliedriges Motivprotein 14) (Abbildung 3).
HINWEIS: Für Serinproteasen wird eine höhere Konservierung des P1-Rückstandes erwartet, während für Cysteinproteasen eine höhere Konservierung des P2-Rückstandes erwartet wird. - Färben Sie die Rückstände, die mit einer Spaltungs-Standortsequenz identisch sind und in sequenzieller Reihenfolge (keine Lücken) sind. Färben Sie die Rückstände, die an der Unterwebsite toleriert werden, aber an einer anderen Spaltstelle in einer zweiten Farbe vorhanden sind.
HINWEIS: Rückstände, die konservative Substitutionen darstellen (z.B. Leu vs. Val), die nicht an einer viralen Spaltungsstelle vorhanden sind, können ebenfalls gefunden werden und können von der viralen Protease erkannt werden oder auch nicht. - Ordnen Sie die BLAST-Treffer basierend auf der Anzahl aufeinander folgender identischer oder tolerierter Rückstände an, die einer Spalten-Standortsequenz entsprechen. Wählen Sie aus der Liste die Proteine aus, die 6 identische oder ähnliche Rückstände enthalten, um sie in Protease-Assays zu analysieren.
- Wiederholen Sie den Vorgang für die anderen Spaltungsstellen (nsP2/3, nsP3/4 usw.) und verstärken Sie die Vorhersage schrittweise, indem Sie dem PHI-BLAST-Muster stärker erhaltene Rückstände hinzufügen.
- Wählen Sie bei Bedarf PHI-BLAST aus. Geben Sie eine Mustersequenz ein (z. B. geben Sie für die unten gezeigten 25 Rückstände von V12 das Muster "AG" ohne Anführungszeichen ein).
2. In Vitro Assays: Entwerfen und Vorbereiten von Proteasesubstraten
- Konstruieren Sie ein Plasmid, das für das Cyan-Fluoreszenzprotein (CFP) kodiert, 25 Aminosäuren der Spalten-Standortsequenz, gefolgt vom gelben fluoreszierenden Protein (YFP, auch bekannt als Venus29).
HINWEIS: Das Plasmid kann mit sequenz- und ligationsunabhängigem Klonen (SLIC)30 oder kommerzieller Gensynthese konstruiert werden. Ein pet15b-Plasmid, das die in Abbildung 4 dargestellte Sequenz enthält, wurde kommerziell synthetisiert und hier verwendet.- Um die Substratlänge zu optimieren, konstruieren Sie zusätzliche FRET-Substrate mit 12-25 Aminosäuren der natürlichen viralen Polyproteinspaltungs-Site-Sequenzen mit einer 2-Fragment-SLIC-Reaktion. Analysieren Sie die Spaltung mit dem SDS-PAGE-Gel-basierten Assay oder durch Messung der konstanten kinetischen Parameter mit den folgenden Methoden.
HINWEIS: In einigen Fällen können Spaltungsstellen durch Homologie zu bekannten Spaltungsstellen31identifiziert werden. Wird keine Spaltung der Substrate beobachtet, die die Polyprotein-Knotensequenzen enthalten, kann es eine Anforderung an zusätzliche Rückstände oder ein strukturelles Motiv (z.B. eine Alpha-Helix32) geben. Alternativ kann die gereinigte virale Protease inaktiv sein. Bestätigen Sie die Spaltung der viralen Polyproteinsequenzen, bevor Sie die SSHHPS-Analyse durchführen. Die Anzahl der Rückstände im Substrat wurde für die VEEV-Protease mit substängenvariablen Substraten (12 bis 25 Aminosäuren) optimiert, gefolgt von der Analyse von Vmax und Km32,33. Die zika viralen ns2B/nsB Protease-Spaltungsstellen, die in den Beispielen verwendet werden, wurdenveröffentlicht 34,35.
- Um die Substratlänge zu optimieren, konstruieren Sie zusätzliche FRET-Substrate mit 12-25 Aminosäuren der natürlichen viralen Polyproteinspaltungs-Site-Sequenzen mit einer 2-Fragment-SLIC-Reaktion. Analysieren Sie die Spaltung mit dem SDS-PAGE-Gel-basierten Assay oder durch Messung der konstanten kinetischen Parameter mit den folgenden Methoden.
- Bereiten Sie die CFP/YFP-Substrate vor, indem Sie 8-20 L BL-21(DE3) frisch transformierenE. colimit dem CFP-V12-YFP-Plasmid gemäß Herstellerbeschreibung und Platte auf Luria Bertani (LB) Agarplatten, die 50 g/ml Ampicillin (37 °C) enthalten.
- Autoklav vier 4 L-Flaschen mit LB-Medien (1,5 l Medien pro Kolben) und 100 ml LB in einem 250 ml Kolben. Kappen Sie jeden Kolben mit Aluminiumfolie.
- Die 100 ml Kultur mit einer Kolonie der frisch transformierten Bakterien impfen und bei 37 °C mit Schütteln (200 U/min) über Nacht wachsen.
- Um das CFP/YFP-Substrat herzustellen, impfen Sie vier 4-L-Flaschen mit 25 ml einer Nachtkultur. Beginnen Sie, die Kulturen bei 37 °C zu schütteln und überwachen Sie das Wachstum durch UV-Vis-Spektroskopie bei 600 nm pro Stunde.
- Wenn die Bakterien bei 600 nm (etwa 3-4 h Wachstum) eine Absorption von 1,0 nm erreichen, induzieren sie die Proteinexpression, indem sie 0,5 ml von 1 M Isopropyl-D-Thiogalactosid (IPTG) pro Kolben hinzufügen. Nach Zugabe von IPTG die Temperatur des Schüttelinkubators auf 17 °C senken und die Expression über Nacht für 17-20 h fortsetzen können.
- Pellet die Bakterien mit einer Hochgeschwindigkeitszentrifuge bei 7.000 x g für 10 min (4 °C) und behalten Sie die Pellets. Entfernen und entsorgen Sie flüssige Medien. Bewahren Sie die Pellets bei -80 °C oder Lyse sofort auf.
- Bereiten Sie 100 ml Lysepuffer mit 50 mM Tris pH 7,6, 500 mM NaCl, 35 ml bakterielles Proteinextraktionsreagenz, 30 mg Lysozym, 25 U DNase und 1 Protease-Hemmer-Tablette vor. Die Pellets im Lysepuffer mit einer Pipette wieder aufhängen und 25-35 ml in 50 ml Einwegkonusrohre übertragen.
- Legen Sie die Rohre in einen Kunststoffbecher mit Eiswasser. Setzen Sie die Beschallungsspitze in die Rohre ein, so dass die Spitze 1 cm von der Unterseite des Rohres entfernt ist, und beschallen Sie die Lysate 10-20-mal auf Ebene 5 für 15 s Intervalle, bis das Lysat flüssig und verflüssigt wird.
HINWEIS: Verwenden Sie den Gehörschutz während der Beschallung. - Das Lysat bei 20.500 x g 30 min bei 4 °C auf Hochgeschwindigkeitszentrifugenrohre und Zentrifuge übertragen. Halten Sie nach der Drehung den Überstand (ca. 100 ml) auf und geben Sie ihn in eine saubere Flasche. Entsorgen Sie die Pellets.
- Bereiten Sie 1 L Puffer A (50 mM Tris pH 7,6, 500 mM NaCl) vor. Bereiten Sie 300 ml Puffer B (50 mM Tris pH 7,6, 500 mM NaCl, 300 mM Imidazol) vor.
- Equilibrate eine 100 ml Nickelsäule mit 3 Spaltenvolumen von Puffer A und einer Durchflussrate von 5 ml/min.
- Laden Sie das Lysat mit einer Durchflussrate von 2 bis 5 ml/min auf die Nickelsäule. Waschen Sie die Spalte mit 2 Spaltenvolumen von Puffer A, gefolgt von einem Spaltenvolumen von 20 % Puffer B. Während der 20% Puffer B-Waschanlage erhöht sich die Absorption bei 280 nm (A280) als Verunreinigungen aus der Säule während der Wäsche. Fahren Sie mit dem Waschen der Säule fort, bis die A280 des Eluats wieder zu den Basiswerten zurückgekehrt ist.
- Das Protein mit 2-3 Säulenvolumen von 100% Puffer B mit einer Durchflussrate von 2-5 ml/min und 10 ml Brüche sammeln. Messen Sie die A280 jeder Fraktion.
- Kombinieren und konzentrieren Sie Fraktionen, die A280 > 0,1 enthalten, mit einer 15 ml Zentrifugal-Ultrafiltrationseinheit. Drehen Sie die Ultrafiltrationseinheiten 15 min bei 5.000 x g und fügen Sie weiterhin Fraktionen hinzu, bis das Volumen auf 50-75 ml reduziert wurde.
- Schneiden Sie ein 14-Zoll-Stück Dialyseschläuche mit einem Molekulargewichts-Cut-off (MWCO) von 6-8 kDa. Hydratisieren Sie die Dialyseschläuche, indem Sie sie vollständig in 300 ml Wasser für 10 min. binden Sie einen sicheren Knoten an einem Ende der Membran. Füllen Sie den Beutel mit Dialysepuffer, um sicherzustellen, dass keine Risse oder Lecks vorhanden sind. Entfernen Sie den Puffer aus dem Beutel und halten Sie den Beutel im Dialysepuffer unter Wasser.
- Das konzentrierte Protein ab 2.2.13 mit einer Kunststoffpipette in den Dialysebeutel geben. Entfernen Sie alle Luftblasen aus dem Beutel. Schließen Sie die Tasche mit einem zweiten Knoten oder einem Dialyseclip. Dialyse das Protein gegen 500 ml 50 ml Tris pH 7,6, 5 mM EDTA (Ethylendiamintetraessigsäure), 250 mM NaCl in einem 500 ml abgestuften Zylinder über Nacht bei 4 °C.
- Dialyse das Protein ein zweites Mal gegen 500 ml 50 mM Tris pH 7,6 bei 4 °C für 2 h.
- Für die Anionenaustauschsäule 500 ml Puffer A (50 mM Tris pH 7.6) und 500 ml Puffer B (50 mM Tris pH 7.6, 1.0 M NaCl) vorbereiten. Equilibrate eine 30 ml Anionenaustauschsäule mit 3 Spaltenvolumes von Puffer A (2-5 ml/min).
- Entfernen Sie das Protein aus dem Dialysebeutel und geben Sie es in eine Flasche. Die Flasche auf Eis halten. Laden Sie das dialysierte Protein auf die Säule (2-5 ml/min).
HINWEIS: Das CFP/YFP-Protein bindet die Säule und ist gelb im Aussehen. - Waschen Sie die Spalte mit Puffer A, bis die A280 zur Baseline zurückkehrt (5 ml/min). Das Protein mit einem Gradienten (0-50% Puffer B, 100 ml) zu löschen und 10 ml-Fraktionen zu sammeln.
- Überprüfen Sie die Spaltenfraktionen mit SDS-PAGE. Kombinieren Sie diejenigen, die >95% rein sind.
- Konzentrieren Sie das Protein mit einer 15 ml Zentrifugal-Ultrafiltrationseinheit auf eine A 280-10-20-Konzentration. Drehen Sie den Konzentrator bei 4.500 x g für 10 min bei 4 °C und fügen Sie Protein hinzu, bis alle proteinhaltigen Fraktionen kombiniert sind.
- Entfernen Sie das Protein aus dem Dialysebeutel und geben Sie es in eine Flasche. Die Flasche auf Eis halten. Laden Sie das dialysierte Protein auf die Säule (2-5 ml/min).
- Entfernen Sie das Protein vorsichtig mit einer Pipette aus dem Konzentrator. Aliquot das Protein in 1,5 ml Mikrozentrifugenröhren und Blitz einfrieren in flüssigem Stickstoff für die langfristige Lagerung bei -80 °C. Puffertauschen Sie das Protein bei Raumtemperatur mit einer PD-10-Säule, die vor der Verwendung mit dem entsprechenden Assaypuffer ausgeglichen wird.
- Berechnen Sie anhand des Beer-Gesetzes die Proteinkonzentration anhand des A280 und eines berechneten Aussterbekoeffizienten (z.B. für das V12-Substrat das n = 47.790 M-1 cm-1).
HINWEIS: Der Aussterbekoeffizient () kann aus der Proteinsequenz in Abbildung 4 mit dem Expasy ProtParam-Programm (https://web.expasy.org/protparam/ )berechnet werden.
3. Herstellung des Alphaviral nsP2 Cystein Protease
- Entwerfen und konstruieren Sie ein Plasmid, das die Protease kodiert. Für Cystein-Proteasen verwenden Sie das pet32-Plasmid, um ein Thioredoxin (Trx) Fusionsprotein zu konstruieren.
Hinweis:Das pet32 Plasmid kodiert eine Thrombinspaltungsstelle (LVPRGS) zur Entfernung des Thioredoxins und seines His-Tags (Abbildung 5). Thioredoxin wird dazu beitragen, die aktive Stelle Cystein in einem reduzierten Zustand während der Expression zu halten. Bei Serinproteasen wird das Thioredoxin nicht benötigt und Schritte, die seine Entfernung durch Thrombin beinhalten, können weggelassen werden. Die VEEV nsP2 Protease-Sequenz wurde in ein pet32b-Plasmid integriert, das kommerziell hergestellt wurde, um den Umgang mit Select-Agenten zu vermeiden.- Verwandeln Sie die Plasmid-DNA nach Herstellerangaben frisch in BL21(DE3)pLysS E. coli. Die Bakterien auf LB-Agarplatten, die Ampicillin enthalten, verplatten.
HINWEIS: Chloramphenicol wird nur für E. coli-Stämme verwendet, die das PLysS-Plasmid tragen, und wird weggelassen, wenn BL21(DE3)-Zellen verwendet werden. Es ist nicht notwendig, Chloramphenicol auf der LB Agarplatte in diesen Schritt aufzunehmen. - Autoklav vier 4 L-Flaschen mit 1,5 l LB-Medien (6 l Gesamtvolumen) und 100 ml LB in einem 250 ml Kolben. Kappen Sie jeden Kolben mit Aluminiumfolie.
- Eine 100 ml Nachtkultur von LB/Ampicillin mit einer Kolonie vom Teller impfen und in einem Schüttelbrutkasten (200 U/min) bei 37 °C wachsen.
- Die 4 L-Flaschen mit 25 ml der Nachtkultur impfen und die entsprechenden Antibiotika hinzufügen.
HINWEIS: Die Medien für die BL21(DE3) pLysS-Zellen, die das pet32-Plasmid tragen, sollten Endkonzentrationen von 25 g/ml Chloramphenicol und 50 g/ml Ampicillin haben. - Induzieren Sie die Proteinexpression, indem Sie der Kultur 0,5 ml IPTG hinzufügen, wenn die Absorption bei 600 nm 1,0 erreicht. Senken Sie die Temperatur des Schüttelinkubators auf 17 °C. Lassen Sie den Ausdruck über Nacht fortsetzen (ca. 17 h).
- Pellet der Zellen durch Zentrifugation (7.000 x g für 10 min bei 4 °C). Entfernen und entsorgen Sie das flüssige Medium.
HINWEIS: Die Pellets können monatelang bei -80 °C gelagert oder sofort lysiert werden. - Bereiten Sie 100 ml Lysepuffer (50 mM Tris pH 7,6, 500 ml NaCl, 2 mM Beta-Mercaptoethanol (BME), 30 mg Lysozym, 5% Glycerin, 25 U DNase, 35 ml bakterielles Proteinextraktionsreagenz) vor). Öffnen Sie Flaschen von BME in einer chemischen Haube beim Hinzufügen. Halten Sie das bakterielle Lysat für diese und alle nachfolgenden Schritte auf Eis oder bei 4 °C.
HINWEIS: Bei Cysteinproteasen sind 2 mM BME enthalten, um das nucleophile Cystein reduziert zu halten. Die Säulen können bei Raumtemperatur mit gekühlten Puffern ausgeführt werden. Puffer sollten mit kalt entionisiertem Wasser hergestellt werden, das auf 4 °C gekühlt wird. - Die bakteriellen Pellets in einem Lysepuffer von 25 ml wieder aufhängen und 25 ml des Lysats in 4 x 50 ml Einwegkonusröhren übertragen. Legen Sie die Rohre in Kunststoffbecher mit Eiswasser. Beschallen Sie das Lysat 10 Mal auf Ebene 5 für 15 s Intervalle.
- Übertragen Sie das Lysat in Hochgeschwindigkeitszentrifugenrohre. Klären Sie das Lysat durch Zentrifugation (30 min, 20.500 x g bei 4 °C).
- 0,5 l Puffer A (50 mM Tris pH 7,6, 500 mM NaCl, 5% Glycerin, 2 mM BME) vorbereiten und auf 4 °C abkühlen lassen.
- 250 ml Puffer B (50 mM Tris pH 7,6, 500 mM NaCl, 5% Glycerin, 2 mM BME, 300 mM Imidazol) vorbereiten und auf 4 °C abkühlen lassen.
- Equilibrate eine 50 ml Nickelsäule mit 3 Säulenvolumen von Puffer A. Laden Sie das geklärte Lysat bei 2-5 ml/min auf die Säule und entsorgen Sie die Pellets.
- Waschen Sie die Spalte (2-5 ml/min) mit 2 Spaltenvolumen von Puffer A, gefolgt von 5 Spaltenvolumes von Puffer A, die 20% Puffer B (60 mM Imidazole) enthalten. Das Protein (5 ml/min) mit 100% Puffer B elute und 10 ml Fraktionen sammeln.
- Kombinieren und konzentrieren Sie Fraktionen, die die Protease enthalten, die A280 x 0,1 mit einer 15 ml Zentrifugal-Ultrafiltrationseinheit und 15 min Spins bei 5.000 x g bei 4 °C haben. Nachdem das Volumen auf 5 ml reduziert wurde, tauschen Sie das Protein in der Konzentrationseinheit durch Zugabe eines frischen Dialysepuffers in das Protein (50 mM Tris pH 7,6, 250 mM NaCl, 5 mM Dithiothreitol (DTT), 1 mM EDTA, 5% Glycerol) aus. Drehen Sie wieder bei 5.000 x g bei 4 °C für 15 min; den Pufferaustauschschritt 2-3 Mal wiederholen. Fügen Sie dem Protein (20 l von 1 Einheit/L) vor der Dialyse Thrombin hinzu, um Thioredoxin und His-Tag zu entfernen.
- Das Protein in einen Dialysebeutel geben und gegen 500 ml des Dialysepuffers (4 °C) in einem 500 ml abgestuften Zylinder über Nacht dialysieren.
HINWEIS: Das FPLC-System (Fast Protein Liquid Chromatography) und die Nickelsäule sollten mit abisolierendem Puffer (2 M NaCl, 50 mM EDTA) gründlich gereinigt werden, bevor Sie zur Anionenaustauschsäule übergehen. Jegliches Restnickel in den FPLC-Linien verwandelt die Pufferlösungen, die DTT-Braun enthalten, wenn es gemischt wird. Waschen Sie die Nickelsäule und das FPLC-System mit 4 Säulenwasservolumen. Pumpe waschen das FPLC-System gründlich mit Wasser. Die Nickelsäule kann durch fließendes 2-Säulen-Volumen von 0,2 M Nickelsulfat über das Harz für nachfolgende Reinigungen regeneriert werden.
- Verwandeln Sie die Plasmid-DNA nach Herstellerangaben frisch in BL21(DE3)pLysS E. coli. Die Bakterien auf LB-Agarplatten, die Ampicillin enthalten, verplatten.
- Für die Anionenaustauschsäule 1 L Puffer A (50 mM Tris pH 7,6, 5 mM DTT, 5% Glycerin) vorbereiten.
- 0,5 L Puffer B vorbereiten (50 mM Tris pH 7,6, 5 mM DTT, 5% Glycerin, 1,25 M NaCl).
- Equilibrate eine 30 ml Anionenaustauschsäule mit Puffer A (3 Spaltenvolumen, 2-5 ml/min). Legen Sie die Rohre in den Fraktionskollektor für die Erfassung des Durchflusses.
HINWEIS: Die VEEV-Protease hat einen berechneten isoelektrischen Punkt (pI) von 8,7 und bindet Kation-Austausch-Säulen, fließt aber durch Anionenaustauschsäulen. Der pI kann aus der Proteinsequenz mit dem Expasy ProtParam-Programm (https://web.expasy.org/protparam/ )berechnet werden. - Das Dialyseprotein 1:3 mit Puffer A verdünnen und dann das Protein (5 ml/min) laden. Sammeln Sie den Durchfluss in 10 ml Fraktionen.
- Entfernen Sie die Anionenaustauschsäule aus dem FPLC-System. Verbinden Sie eine Kationenaustauschsäule mit dem FPLC-System. Equilibrate eine 30 ml Kationenaustauschsäule mit 3 Spaltenvolumes von Puffer A (5 ml/min).
- Laden Sie den Durchfluss der Anionenaustauschsäule auf die Kationenaustauschsäule bei 2-5 ml/min. Waschen Sie die Spalte mit Puffer A, bis die A280 auf Basisebene zurückkehrt. Das Protein mit einem Gradienten von 100 ml (0-50% Puffer B) vereuten und 10 ml-Fraktionen sammeln.
HINWEIS: Die VEEV-Protease wird bei ca. 0,6 M NaCl. - Überprüfen Sie die Spaltenfraktionen mit SDS-PAGE. Kombinieren Sie Fraktionen, die >95% rein sind, und konzentrieren Sie sich mit 15 ml Zentrifugal-Ultrafiltrationseinheiten auf ein A280 bei 2. Das Enzym kann in flüssigem Stickstoff blitzgefroren und bei -80 °C gelagert werden.
- Laden Sie den Durchfluss der Anionenaustauschsäule auf die Kationenaustauschsäule bei 2-5 ml/min. Waschen Sie die Spalte mit Puffer A, bis die A280 auf Basisebene zurückkehrt. Das Protein mit einem Gradienten von 100 ml (0-50% Puffer B) vereuten und 10 ml-Fraktionen sammeln.
4. Assaying das Enzym kontinuierlich mit einem Plattenleser
- 50 ml Assaypuffer (50 mM HEPES pH 7.0) vorbereiten.
- Da die alphaviralen Proteasen relativ niedrigek-Katzenwerte aufweisen, verdünnen Sie das Enzym im Assaypuffer auf 4,7 m (dies entspricht in etwa einem A280 = 0,2 für die VEEV-Protease ohne Trx).
- Um die Aktivität des Enzyms zu messen, bereiten Sie einen Vorrat an Substrat im Assaypuffer mit einer Konzentration von 185 M vor; dies entspricht in etwa einem A280 = 9. In 8 Mikrozentrifugenrohren bereiten Sie die in Tabelle 2 dargestellten Reaktionsmischungen vor, indem Sie die entsprechenden Volumina des 185-M-Substratbestands und puffern kombinieren. In einer schwarzen Halbfläche 96-Well-Platte Pipet45 l der Reaktion mischt sich in 3 Brunnen (Säulen 1, 2, 3). Zeile A sollte den Reaktionsmix [S] = 5 'M' enthalten, und Zeile H sollte den Reaktionsmix [S] = 140 'M' enthalten.
- Stellen Sie den Plattenleser so ein, dass die Fluoreszenz bei zwei Wellenlängen mit einer festen Photomultiplier-Röhre (PMT) gleichzeitig erkannt wird (z. B. niedrig):
Wellenlänge 1 Anregung = 434 nm, Emission = 527 nm
Wellenlänge 2 Anregung = 434 nm, Emission = 470 nm - Stellen Sie die Lesezeit auf 20 min (Messung 1 Lesemenge pro Minute) und wählen Sie die zu lesenden Brunnen aus. Setzen Sie die Platte in den Plattenleser ein und messen Sie die spontane Hydrolyserate für 20 min. Überwachen Sie die Emissionsverhältnisse (Emission bei 527/Emission bei 470) im Zeitverlauf.
- Führen Sie einen Endpunkt aus, der die Platte liest, die das Substrat "UNCUT" enthält.
HINWEIS: Diese Werte werden in nachfolgenden Datenberechnungen verwendet. Der Durchschnitt der Emissionsverhältnisse aus 3 Bohrguts wird in Tabelle 3mit t = 0 des Substrats "UNCUT" liegen. - Entfernen Sie die Platte und Pipette 5 l Enzym in jeden Brunnen. Lesen Sie die Platte erneut für 20 min mit 1 Lesung pro Minute. Stellen Sie den Plattenleser so ein, dass absolute Werte ausgegeben werden.
HINWEIS: Für diesen Test werden die Pisten negativ sein. Jeder Brunnen wird ein Gesamtvolumen von 50 l enthalten. - Versiegeln Sie die Platte am Ende der Lesemitfolie mit Folie, um Verdunstung zu verhindern. Lassen Sie die Platte über Nacht bei Raumtemperatur, damit das Enzym das Substrat vollständig schneiden kann.
- Entfernen Sie nach 24 h die Dichtfolie und führen Sie einen Endpunktablesen der Platte mit dem gleichen PMT wie in den vorherigen Platten durch. Durchschnitt dieser Emissionsverhältnisse und Desinputs in Tabelle 3 unter "CUT". Bestätigen Sie die Spaltung des Substrats mit dem unten beschriebenen Diskontinuierlichen Test SDS-PAGE (Schritt 5.1.).
- Exportieren Sie die Daten in eine Kalkulationstabelle. Ausgabe der Fluoreszenzeinheiten zu jedem Zeitpunkt für die 2 Wellenlängen (Tabelle 4).
- Berechnen Sie den Nmol von Substrat, der zum Zeitpunkt t geschnitten wurde, mit Gleichung (1), wobei X das Emissionsverhältnis (527 nm/470 nm) zu einem bestimmten Zeitpunkt ist, neg ist das Emissionsverhältnis des "UNCUT"-Substrats bei t = 0 und pos das Emissionsverhältnis des vollständig "CUT"-Substrats, das nach 24 h Schneiden gemessen wird (Tabelle 3).
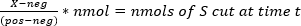
HINWEIS: Repräsentative Fluoreszenzdaten werden in Tabelle 4für einen Brunnen (gut E7) mit 80 M Substrat (4 nmol S pro Brunnen) dargestellt. Die Berechnungen wurden für jeden Brunnen in der Platte durchgeführt. - Zeichnen Sie für jeden Brunnen nmol vs. Zeit (min) und erhalten Sie die Anfangsgeschwindigkeiten (Pisten), indem Sie die Daten an y = mx + b anpassen. Für die in 4.1.5 gesammelten Daten plot nmol vs. zeit (min) für jeden Brunnen. Die Steigung entspricht dem nmol Produkt pro Minute produziert. Subtrahieren Sie die spontanen Hydrolyseraten, gemessen in 4.1.5, von den enzymkatalysierten Reaktionsraten (Tabelle 5).
HINWEIS: Der erste Lesewert kann aus den Daten abgeschnitten werden, wenn er durch die Bewegung der Platte in den Plattenleser artifisch hoch ist. - Berechnen Sie die Menge des Enzyms in mg, das jedem Brunnen zugesetzt wurde (z. B. 0,0009 mg). Eine Einheit ist definiert als ein pro Minute produziertes Produkt. Dividieren Sie die nmol/min durch die mg des Enzyms im Brunnen vorhanden, um mU/mg zu erhalten; dividieren durch 1.000, um U/mg zu erhalten.
- Plot [S] 'M auf der x-Achse und U/mg auf der y-Achse und passen Sie die Daten an die Michaelis-Menten-Gleichung an, um Vmax und Kmzu erhalten. Dies kann in der Software (z.B. GraFit) erfolgen.
5. Assaying des Enzyms Diskontinuierlich mit SDS-PAGE-Analyse
- Bereiten Sie eine 50-L-Reaktion vor, die 10 M Substrat und Puffer anstelle von Enzym und Etikett als "UNCUT" enthält.
HINWEIS: Die Mengen an Substrat und Puffer sind in Tabelle 2dargestellt. Wenn der kontinuierliche Test durchgeführt wurde, können die Proben direkt von der 96-Well-Platte verwendet werden.- Bereiten Sie eine 50-L-Reaktion vor, die ein 10-M-Substrat und ein 5-L-Enzym enthält und als "CUT" gekennzeichnet ist. Starten Sie den Timer, wenn das Enzym dem Substrat zugesetzt wird.
HINWEIS: Inhibitoren können zusätzlichen Röhren zugesetzt werden, die Enzym und Substrat enthalten. Passen Sie das Volumen des zusätzlichen Puffers an, um das zusätzliche Volumen des Inhibitors auszugleichen. Die DMSO-Konzentrationen sollten 2 % nicht überschreiten. - Inkubieren Sie die Reaktionen für 15-24 h bei Raumtemperatur (22 x 3 °C). Beenden Sie die Reaktionen, indem Sie 50 l 2x Laemelli-Puffer hinzufügen. Nach dem Anhalten der Reaktion kochen, jede Röhre für 3-10 min.
- Montieren Sie den Geltank gemäß den Anweisungen des Herstellers. Legen Sie auf der anderen Seite eine 17-Well-vorgegossene 12% Polyacrylamid-Gelkassette und einen Pufferdamm ein. Füllen Sie das innere Reservoir der Zelle mit 1x SDS-Laufpuffer, bis der Puffer die Oberseite der Kassette erreicht. Füllen Sie das externe Reservoir halbvoll mit dem gleichen Puffer.
- Um die Spaltung mit dem diskontinuierlichen Assay zu analysieren, laden Sie 5 l jedes Reaktionsgemisches in eine Spur eines SDS-PAGE-Gels, beginnend mit der "UNCUT"-Reaktion. Fügen Sie eine Molekulargewichtsmarkierung in die erste oder letzte Spur ein.
- Befestigen Sie die Elektroden des Geltanks an der Stromversorgung und trennen Sie die Produkte bei 110 V für 60 min. Entfernen Sie das Gel aus der Kassette, indem Sie das Risswerkzeug zwischen den Platten einlegen. Legen Sie das Gel in eine Kunststoffschale und tauchen Sie das Gel in 5-10 ml Gelfärbung Lösung; Bänder werden innerhalb von 30 min sichtbar sein. Nach 1-24 Stunden entfernen Sie den überschüssigen Fleck, tauchen Sie das Gel in Wasser und verwenden Sie eine Gel-Imager, um ein Bild des Gels zu machen.
- Bereiten Sie eine 50-L-Reaktion vor, die ein 10-M-Substrat und ein 5-L-Enzym enthält und als "CUT" gekennzeichnet ist. Starten Sie den Timer, wenn das Enzym dem Substrat zugesetzt wird.
6. Docking Substrat Peptide an die VEEV-nsP2 Cystein Protease
- Laden Sie die Koordinatendatei für die VEEV-Cystein-Protease aus der PDB (https://www.rcsb.org/ ) herunter. Der PDB-Code ist 2HWK. Speichern Sie die Datei als 2HWK.pdb.
- Bereiten Sie die Proteinstruktur mit MOE (https://www.chemcomp.com/ )vor. Laden Sie die PDB-Datei des Proteins in MOE. Klicken Sie auf die Taste "Auswählen" und "Lösungsmittel" auf der rechten Seitenleiste, und löschen Sie das Lösungsmittel.
- Öffnen Sie das Structure-Vorbereitungs-Bedienfeld in der oberen Menüleiste Protein. Korrigieren Sie automatisch alle Strukturelemente, indem Sie auf Korrigieren klicken und die Struktur protonieren, indem Sie auf Protonate3Dklicken. Fügen Sie dem Protein Teilladungen hinzu, indem Sie das Bedienfeld "Partial Charges" öffnen und Amber 99 und die Wasserstoff- und Einlone-Paare nach Bedarfauswählen. Speichern Sie schließlich die Strukturdatei als "2HWK_dock.pdb".
- Bauen Sie die Struktur für die Substratpeptide (nsP12, nsP23, nsP34) und TRIM14 mit MOE. Öffnen Sie das Protein Builder-Bedienfeld, geben Sie die Substratsequenz ein, legen Sie die Geometrie als Erweitert fest, und klicken Sie auf Erstellen. Die Struktur wird im MOE-Fenster angezeigt.
- Minimieren Sie die Peptidstruktur, indem Sie auf Minimieren im Bedienfeld klicken. Speichern Sie die Struktur als PDB-Datei (Abbildung 6).
- Docken Sie die Substratpeptide mit PyRx/AutoDock 4.2 (http://autodock.scripps.edu/) an VEEV-nsP2 an. Öffnen Sie das PyRx-Tool, bearbeiten Sie die Einstellung für die Voreinstellung, deaktivieren Sie alle Torsions für Ligand Preparation. Laden Sie das Substratmolekül, klicken Sie mit der rechten Maustaste auf den Namen des Moleküls im Navigator-Bedienfeld, wählen Sie Liganden herstellen aus, um die Liganden-Docking-Datei vorzubereiten. Laden Sie das Protein 2HWK_clean.pdb, und wählen Sie Makromolekül erstellen aus, um die pdbqt-Dockingdatei vorzubereiten (Abbildung 7).
- Starten Sie den AutoDock-Assistenten auf dem Docking-Panel unten. Wählen Sie die vorbereiteten Liganden- und Proteindateien aus. Definieren Sie die Proteinbindungstasche, indem Sie die Gitterdimension manuell anpassen, die am katalytischen Rückstand Cys-477 zentriert ist. Verwenden Sie den Standardabstandsparameter 0.375 . Klicken Sie auf AutoGrid ausführen, um Rasterkarten zu generieren.
- Führen Sie AutoDock aus, und wählen Sie die LGA-Methode (Lamarckian Genetic Algorithm)aus. Klicken Sie auf die Docking-Parameter und legen Sie die Anzahl der GA-Läufe auf 50 fest. Verwenden Sie die Standardparameter für andere Benutzer. Klicken Sie auf Vorwärts, um den Docking-Lauf zu starten.
- Öffnen Sie das Bedienfeld Ergebnisse analysieren. Überprüfen Sie alle vorhergesagten Bindungsposen. Wählen Sie das beste Modell mit der niedrigsten vorhergesagten Bindungsenergie und vernünftigen Bindungswechselwirkungen zwischen dem Cys-477 und dem Substrat auf der Spaltstelle aus. Speichern Sie das Bindungsmodell als PDB-Datei für weitere MD-Simulationen.
7. MD Simulationen von Docked VEEV-Substrat-Komplexen
- Bereiten Sie die Eingabedateien mit Amber (http://ambermd.org/ )vor. Nach dem Standardprotokoll werden MD-Simulationen für die vorhergesagten Substratbindungsmodelle mit dem AMBER-Paket und dem Kraftfeld ff99SB durchgeführt.
HINWEIS: Die gelösten Systeme werden vor MD-Simulationen einer gründlichen Energieminimierung unterzogen. Regelmäßige Randbedingungen werden angewendet, um ein kontinuierliches System zu simulieren. Zur Berechnung der elektrostatischen Wechselwirkungen mit großer Reichweite wurde das Partikelnetz Ewald (PME) verwendet. Das simulierte System wurde zunächst einem allmählichen Temperaturanstieg von 0 K auf 300 K über 100 ps unterzogen und dann für 500 ps bei 300 K ausgeglichen, gefolgt von Produktionsläufen von insgesamt 2 ns Länge.- Führen Sie den Simulationsauftrag in einer Hochleistungs-Computing-Anlage aus. Unsere Simulationen wurden auf dem Biowulf-Cluster (https://hpc.nih.gov/) (Abbildung 8) durchgeführt.
- Visualisieren Sie die Trajektorieausgabe mit dem VMD-Programm (http://www.ks.uiuc.edu/Research/vmd/). Analysieren Sie die Bindungswechselwirkungen und Konformationsänderungen der Substrate und TRIM14 innerhalb der aktiven Stelle von nsP2 (Abbildung 9).
Ergebnisse
Die SSHHPS-Analyse der ZIKV ns2B/3-Protease identifizierte 4 Wirtproteinziele: FOXG1, SFRP1, eine G s-Alpha-Untereinheit aus einer retinalen cDNA-Bibliothek und die NT5M-Mitochondrial-5',3'-Nukleotidase (Abbildung 10)6. Bemerkenswert ist, dass keine andere Methode diese Proteine als potenzielle Ziele der ZIKV-Protease vorhersagte. Mutationen im FOXG1-Gen wurden mit einem angeborenen Syndrom in Verbindung gebracht, das durch eine eingeschränkte Entwicklung und strukturelle Hirnanomalien wie Mikrozephalie gekennzeichnet ist. SFRP1 ist ein abgesondertes Frizzled-protein (SFRP); Diese löslichen Rezeptoren können Wnt-Liganden wettbewerbsfähig binden, um die Wnt-Signalisierung zu antagonisieren und zu hemmen. Der Wnt-Signalweg ist an der Regulierung der IFN-Reaktion während der Flavivirus-Infektionbeteiligt 36. Die Spaltung von SFRP1 würde erwartet, um die Flavivirus-Replikation zu verbessern. SFRP1 ist auch an der Th17-Zelldifferenzierung37beteiligt. Sequenzausrichtungen des SSHHPS zeigten artspezifische Unterschiede in den Spalt-Standortsequenzen (Abbildung 10D). Die Spaltungs-Standortsequenz in SFRP1 war bei Menschen und Hühnern identisch; ZIKV kann Mortalität und Mikrozephalie bei Hühnerembryonen induzieren38. Bei Nagetieren wird der hochkonservierte P1-Rückstand (K/R)R-G durch ein Glycin (RGG) ersetzt. Immunkompetente Stämme von Mäusen sind in der Regel resistent gegen ZIKV-Infektion und Krankheit39.
Stetige Zustandskinetische Parameter und Hemmungskonstanten können für die viralen Polyproteinsequenzen und für die Wirtsproteinsequenzen mit dem kontinuierlichen Assay in einem Plattenleser31,40,41 ( Abbildung11A) gemessen werden. Für qualitative Spaltungsinformationen, wie z.B. Spaltung einer bestimmten Sequenz oder die Hemmung der Protease durch verschiedene Verbindungen, kann der diskontinuierliche Assay verwendet werden (Abbildung 11B).
Es kann eine Optimierung der Anzahl der Rückstände zwischen GFP und YFP erforderlich sein. Ein substratgebundenes Modell kann mit den in silico-Methoden hergestellt werden. Abbildung 9zeigt ein repräsentatives dockiertes Modell der nsP1/nsP2-Kreuzung . Für die VEEV nsP2-Protease wurde eine Spaltung einer 12-Aminosäure-Sequenz semliki Forest Virus (SFV) berichtet (Km = 0,58 mM)33. Die Verlängerung der Substratsequenz auf 19, 22 und 25 Rückstände und die Verringerung der Ionenstärke des Puffers führten zu einer signifikanten Reduktion von Km. Die Untersuchung der VEEV nsP2 Kristallstruktur und Kristallverpackung zeigte auch, dass ein Teil einer der Knoten gegen die Protease-Domäne verpackt war und helical war. So können die längeren VEEV-Substrate durch die Erkennung eines sekundären Strukturmotivs besser binden.
Für TRIM14 haben wir ein Km = 21 m6,33erhalten. Das Km für das Substrat, das die Wirtsproteinsequenz trägt, war vergleichbar mit den Km-Werten der Substrate, die die viralen Polyproteinspaltungs-Standortsequenzen (Km(V12) = 12 m und Km(V34) = 21 m enthalten. Die Spalten-Standortsequenzen an den Knoten nsP1/nsP2, nsP2/nsP3 und nsP3/nsP4 wurden mit unterschiedlichen Effizienzen geschnitten. In der Zelle wird gedacht, dass dies eine sequenzielle Spaltung des Polyproteins42ermöglicht.
Bei der Interpretation negativer Ergebnisse ist Vorsicht geboten. Wenn keine Spaltung auftritt, kann die Spaltstelle zu kurz oder die gereinigte Protease inaktiv sein. Für Substrate, die geschnitten werden, sind zusätzliche Experimente erforderlich, um die Spaltung des Voll-Proteins oder die Spaltung in virusinfizierten Zellen zu bestätigen. Es sollten geeignete Folgeexperimente ausgewählt werden. Die Auswirkungen von Überexpression oder Silencing des Zielproteins auf die Virusreplikation können ebenfalls getestet werden.
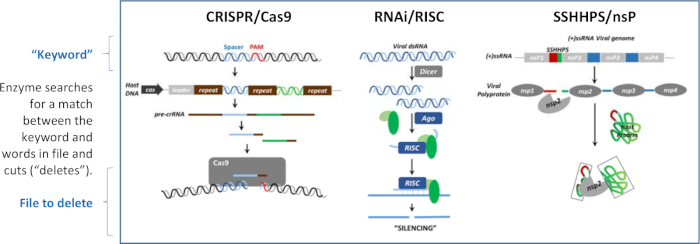
Abbildung 1: Drei Mechanismen des Silencings. Silencing kann auf der Ebene von DNA, RNA oder Protein auftreten. Diese "Suchen und Löschen" Algorithmen verwenden jeweils ein "Schlüsselwort", um die Spaltung einer Datei, die das Wort enthält, zu lenken. Diese Zahl wurde von Morazzani et al.32 und den darin enthaltenen Referenzen geändert. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 2: Artenspezifische Unterschiede in Spalt-Standortsequenzen. Die C-Terminal PRY/SPRY-Domänen der TRIM14-Homologe werden in der Ausrichtung dargestellt. Die PRY/SPRY-Domäne kann durch die konservierten Motive identifiziert werden, die grau hervorgehoben sind. Human TRIM14 wird bei QEAGA-G von der VEEV nsP2 Cystein-Protease geschnitten. Die SSHHP-Sequenz wird in Farbe angezeigt. Der Rückstand in Grün ist der Rückstand von P1; in blau ist der P4-Rückstand, und in Rot sind andere konservierte Rückstände innerhalb der Spaltstellen-Motivsequenz. Equine beherbergen eine abgeschnittene Version von TRIM14, die nicht die PRY/SPRY-Domain hat. Das in Cyan hervorgehobene Lysin ist polyubiquitiniert und wichtig für die Montage des MAVS-Signalossomens. Die C-terminale PRY/SPRY-Domäne kann vorübergehend durch die nsP2-Protease geschnitten werden, um die antivirale Reaktion des Wirts intrazellulär während einer akuten Virusinfektion zu beeinträchtigen. Bei Pferden ist diese Domäne immer nicht vorhanden. Dies deutet darauf hin, dass die PRY/SPRY-Domäne von TRIM14 eine Schutzfunktion gegen VEEV-Infektionen haben kann. Diese Figur wurde von Morazanni et al.6 reproduziert.

Abbildung 3: SSHHPS-Identifikation mit BLAST. Die Spalt-Standort-Motivsequenz am VEEV nsP1/nsP2-Knoten ist an der SSHHP-Sequenz im Wirtsprotein TRIM14 ausgerichtet. Der grün gefärbte Rückstand ist der Rückstand von P1; in blau ist der P4-Rückstand und in rot weitere konservierte Rückstände der Spalt-Standort-Motivsequenz. Die meisten Achsen enthielten Homologie n. Außerhalb des konservierten Spaltstellenmotivs oder enthielten nicht die sissile Bindungsrückstände von P1/P1. TRIM14 zeigte eine Übereinstimmung mit 6 Rückständen in sequenzieller Reihenfolge, die P1 und P1' enthielten. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 4: Protein- und DNA-Sequenzen des CFP-V12-YFP-Substrats für die VEEV nsP2-Cysteinprotease. Die Beschränkungsstandorte NdeI (CATATG) und XhoI (CTCGAG) werden in Großbuchstaben dargestellt. In rot ist die Spaltungs-Standortsequenz aus dem viralen Polyprotein, das sich zwischen nsP1 und nsP2 befindet. Der Rückstand in Grün ist der Rückstand p1 und in blau der P4-Rückstand der Spaltungsstelle. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 5: Proteinsequenz des Trx-VEEV-nsP2-Cystein-Proteasekonstrukts. Thioredoxin (Trx) ist gelb dargestellt. Die Thrombin-Spaltungsstelle und sein His-Tag werden in Cyan dargestellt. Die Cys-His Dyade sind rot beschriftet. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
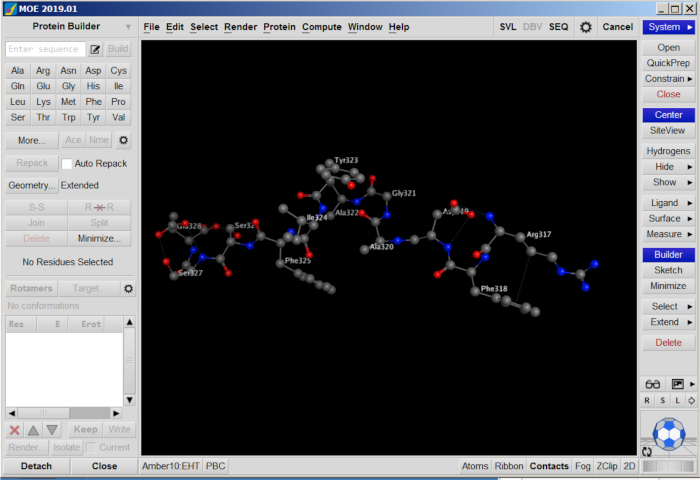
Abbildung 6: Peptidstrukturen in MOE. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Abbildung 7: Andocken von Substratpeptid mit PyRx/AutoDock. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Abbildung 8: Aufträge, die im Biowulf-Cluster ausgeführt werden. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
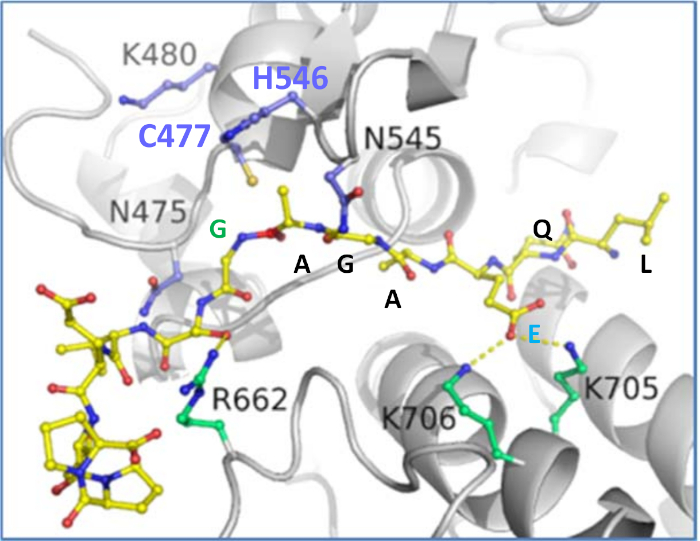
Abbildung 9: Modell des VEEV P12 Substrats, das die Spaltungs-Standortsequenz an der nsP1/nsP2-Kreuzung enthält. Die Katalyse-Dyade Cys-477/His-546 ist blau dargestellt. Die Abbildung wurde mit Pymol (https://pymol.org) gemacht. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
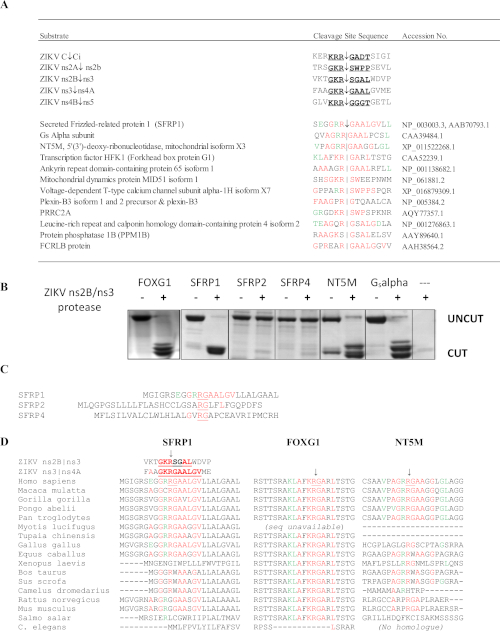
Abbildung 10: SSHHPS Analyse des Zika-Virus ns2B/ns3 Protease. (A) Voraussichtliche Wirtproteinziele der ZIKV ns2B/ns3-Protease. Rückstände in Rot entsprechen einer einzelnen Spalt-Standortsequenz. Rückstände in Grün werden an der Unterstelle toleriert und passen Rückstände an anderen Spaltungsstellen ab. SFRP1 hatte die höchste Anzahl identischer Rückstände in aufeinander folgender Reihenfolge. (B) CFP-Substrat-YFP-Proteine (50-60 kDa) wurden exprimiert und gereinigt, die die vorhergesagte SSHHP-Sequenz aus jedem Wirtsprotein (Mensch) enthielten. Die ZIKV-Protease schnitt menschliche FOXG1, SFRP1, NT5M und eine GsAlpha-Untereinheit isoliert aus einer retinalen cDNA-Bibliothek. Die Dekolleté-Produkte sind ca. 28-30 kDa. Die Substratsequenzen sind in Morazzani et al.6 (C) verfügbar, während die ns2B/ns3 Protease SFRP1 schnitt, schnitt sie ihre Homologe (SFRP2 und SFRP4) nicht ab. (D) Die Ausrichtung der Spaltungsgebiete von verschiedenen Tierarten kann bei der Auswahl eines Tiermodells für ein Virus der Gruppe IV nützlich sein. Beachten Sie, dass sich die konservierte R-G-Sequenz zwischen Menschen und Nagetieren in SFRP1 unterscheidet. Abbildung reproduziert von Morazzani et al.6Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
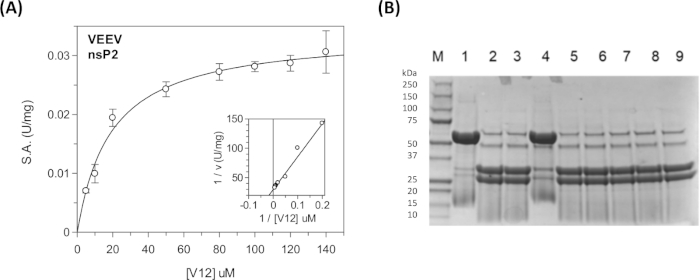
Abbildung 11: Stetige Zustandskinetik analyse mit den kontinuierlichen und diskontinuierlichen Assays. (A) Die in Tabelle 5 dargestellten kinetischen Daten wurden in GraFit dargestellt. Der Einset zeigt das Lineweaver-Burk-Plot. (B) SDS-PAGE Gel mit den Spaltprodukten des CFP-V12-YFP Substrats. In Spur 1 befindet sich das Substrat "UNCUT" (48 kDa). In Spur 2 befindet sich das Substrat "CUT" (31 kDa und 27 kDa). In den Bahnen wurden 3-9 verschiedene Verbindungen aufgenommen, um ihre hemmende Aktivität zu testen. Lane 4 enthält den kovalenten E64d-Inhibitor. Diese Reaktionen wurden über Nacht für 17 h bei Raumtemperatur durchgeführt. Das Kochen der Samples war erforderlich, um das scharfe Banding-Muster zu erreichen. Die nsP2-Protease ist sichtbar (56 kDa) in den Enzym-Haltigen Reaktionen, jedoch nicht in Spur 1. Lane 1 ist die "kein Enzym"-Kontrolle. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Diskussion
Die sequenzspezifische Zerstörung eines Proteins oder einer Nukleinsäure, die von einer fremden Sequenz geleitet wird, ist nur in wenigen Fällen in der Biologie zu beobachten. Die in Abbildung 1 dargestellten Mechanismen sind Abwehrmechanismen, die einen Host vor einem Virus oder einen Virus von einem Host schützen.
Mit bioinformatischen Methoden können wir die Ziele identifizieren, die durch diese Systeme zerstört werden. In unseren Analysen von SSHHP-Sequenzen entdeckten wir, dass viele von ihnen in Proteinen gefunden werden konnten, die benötigt werden, um angeborene Immunantworten zu erzeugen. Einige hatten offensichtliche Rollen wie MAVS und TRIF (TIR-domain-containing adapter-inducing interferon-'), während andere mit Immunität durch komplexere Mechanismen (z.B. Histone H3, SFRP1, FOXG1)8,9zusammenhängen. Die in der SSHHP-Sequenz gespeicherten Zielinformationen haben das Potenzial, Wege zu identifizieren, die antivirale Auswirkungen gegen diese Viren haben. Antivirale Reaktionen in vivo sind oft virusspezifisch26,43. Zum Beispiel haben Teilmengen von TRIM-Proteinen antivirale Wirkungen auf verschiedene Viren43,44,45, einige sind virale Restriktionsfaktoren (z. B. HIV und TRIM5). Die Spezifität von TRIM-Proteinen (70 wurden identifiziert) wird noch untersucht44,45. Die Informationen innerhalb von SSHHPS können zu unserem Verständnis beitragen, wie diese Viren den angeborenen Immunreaktionen entgehen. Andere Muster und Korrelationen können aufgedeckt werden, wenn mehr SSHHPS untersucht werden.
In unseren Analysen zeigten sich artspezifische Unterschiede (Abbildung 2, Abbildung 10). Es ist bekannt, dass diese Viren einige Arten stärker betreffen als andere. Informationen über Hostbereich, Hostanfälligkeit und Hostverteidigung können in SSHHPS vorhanden sein. Zum Beispiel fehlte Pferde, der anfälligsten Art für Pferdeenzephalitisviren, die Region des menschlichen TRIM14, die vorübergehend von der VEEV nsP2 Protease geschnitten wurde. Menschen sterben selten an VEEV-Infektionen, können aber infiziert werden24. Das humane TRIM14-Protein trug eine nsP2-Proteasespaltungssequenz6. Das Vorhandensein der Spaltungsstelle deutet darauf hin, dass Menschen einen Abwehrmechanismus gegen diese Viren haben. Vögel wurden als potenzielle Reservoirs dieser Viren gedacht46. Die entsprechende SSHHP-Sequenz im TRIM14-Protein von Hühnern unterschied sich von den Sequenzen, die beim Menschen und anderen Arten gefunden wurden. Subtile Unterschiede wie diese können ein Ziel-Wirt-Protein uncleavable oder leichter spalten. Aguirre et al.16 zeigten, dass ein onkelwältbar esmutiertes STRING-Protein nach der Dengue-Virusinfektion höhere IFN-Konzentrationen induzierte und dass Mäuse natürlicherweise eine Version von STING tragen, die nicht von der Dengue ns2B3 Protease geschnitten wird. Das murine STING-Protein wurde von der ZIKV-Protease47nicht geschnitten. In unserer SSHHPS-Analyse beobachteten wir auch Unterschiede in den ZIKV-Proteasespaltungs-Standortsequenzen, wenn wir die menschlichen Proteine mit denen von Nagetieren verglichen6 (Abbildung 10D). Die Reproduktion der artspezifischen proteolytischen Spaltungen von Wirtsproteinen kann in Tiermodellen, die für Viren der Gruppe IV verwendet werden, von Bedeutung sein. Die Hemmung der Dekolleté-Spaltung des Wirtsproteins hat auch Auswirkungen auf die Entwicklung von Proteasehemmern der Gruppe IV. In unserer vorherigen Publikation haben wir gezeigt, dass wir trim14 Spaltung durch die VEEV nsP2 Protease mit CA074 Methylester6hemmen können. Dieses Ergebnis legt nahe, dass kleine Molekülinhibitoren dieser Proteasen in der Lage sein könnten, die angeborenen Immunantworten zu modulieren, die in der Lage sind, die Infektion zu unterdrücken6,31.
Genetische Variation innerhalb einer Art hat auch das Potenzial, Unterschiede in der proteolytischen Spaltung zu produzieren. Subtile Unterschiede in der Codon-Nutzung könnten ribosome Pausieren48beeinflussen. Da einige virale Proteasen der Gruppe IV in die ER-Membran eingebettet sind, können Unterschiede in diesen Pausen die Spaltung eines Ziels beeinflussen, wenn die Spaltung ko-translational auftritt. Einige der von uns identifizierten Spaltungsstellen befanden sich in vorhergesagten Signalpeptidsequenzen (z. B. SFRP1), während andere intern waren.
Die SSHHPS-Analyse kann Informationen liefern, die sich von anderen Methoden der Wirtsproteinanalyse unterscheiden. Die SSHHPS-Analyse war kostengünstig und einfach zu verwenden. Die Verwendung eines bakteriellen Expressionssystems ermöglichte das Testen von kurzen Segmenten (25 Aminosäuren) von Säugetiersequenzen ohne die Verwendung von Säugetierzellkultur. Wir fanden heraus, dass die CFP-YFP-Substrate in der Lage waren, alle getesteten menschlichen Proteinsequenzen zu tolerieren; die Erträge jedoch unterschiedlich. In ähnlichen Assays wurden Substrate, die menschliche Proteinsequenzen enthalten, solange 63 Aminosäuren erfolgreich exprimiert, gereinigt und für kinetische Analysen und Inhibitor-Screening49,50,51verwendet. Da für den diskontinuierlichen Assay nur geringe Mengen des Substrats benötigt werden, kann eine große Anzahl von Zielen untersucht werden. Ein Vorteil des Systems ist, dass die CFP/YFP-Substrate für SDS-PAGE-Analysen und für aufwendigere kinetische Analysen (z.B. IC50, Ki, Km, Vmax)verwendet werden können. Für die Entdeckung von Arzneimitteln können hemmende Verbindungen Artefakte in fluoreszierenden Assays produzieren. So ermöglicht der diskontinuierliche Assay in Kombination mit einem kontinuierlichen Assay, die Spaltung oder Hemmung der Spaltung zu bestätigen. Die Proben für den diskontinuierlichen SDS-PAGE-Assay können direkt aus den 96-Well-Platten entnommen werden. CFP/YFP-Substrate wurden für das Screening von Verbundbibliotheken52verwendet. Es sind jedoch zusätzliche Analysen erforderlich, um festzustellen, ob ein Substrat für ein Hochdurchsatzscreening geeignet ist, wie z.B. die Berechnung eines Z-Faktors53.
Eine Herausforderung bei der Gestaltung eines Substrats besteht darin, die Region um die sissile Bindung zu identifizieren, die von der Protease gebunden und erkannt wird. In den hier gezeigten Beispielen begannen wir mit 12 Rückstandssequenzen, die um die sissile Bindung zentriert waren. Nach der Analyse der Sequenzausrichtungen der Spaltungsstellen wurde für die VEEV-Protease eine Homologie zu den Rückständen gefunden, N-Terminal der Sissile-Bindung, während für die ZIKV-Proteasehomologie mehrere der C-Terminal-Rückstände gefunden wurden. Ein in silico Modell des angedockten Substrats kann verwendet werden, um standortgesteuerte Mutageneseexperimente zu entwerfen, die die Bindungsstellen des Substrats untersuchen. Da sich die Substrat- und Enzymsequenzen auf Plasmiden befinden, können entweder die in silico-Modelle oder die Subsite-Toleranzen mutiert werden. Dies kann von Vorteil sein, wenn eine Kristallstruktur des gebundenen Substrats nicht verfügbar ist.
Die SSHHPS-Analyse kann auch neue Informationen über die Mechanismen liefern, mit denen virusinduzierte Phänotypen durch virale Enzyme erzeugt werden. Eines der ZIKV-Ziele, SFRP1, ist Teil des Wnt-Signalwegs und hat Rollen sowohl in der Gehirn- und Augenentwicklung als auch in den Immunantworten36,37,54,55,56,57. Wir fanden heraus, dass die anderen Proteinsequenzen, die durch die ZIKV ns2B/ns3 Protease geschnitten werden konnten, auch in Proteinen an der Entwicklung von Gehirn und Auge beteiligt waren; Anomalien in beiden wurden beim angeborenen Zika-Syndrom beobachtet und gelten als Teil des virusinduzierten Phänotyps58.
Die Vorhersehbarkeit von Wirts-Pathogen-Wechselwirkungen könnte für eine Vielzahl von Anwendungen genutzt werden: zielspezifische onkolytische virale Therapien; Entrisiko von Impfstoffen gegen lebende Viren; Verfeinerung, Vorhersage oder Auswahl von Tiermodellen; Vorhersage des Wirtsbereichs oder der Anfälligkeit; Vorhersage von Zoonose-Ereignissen; und Vorhersage der Host-Verteidigung. Da die beschriebenen Methoden sequenzbasiert sind, können sie von Nutzen sein, um sie in Zukunft in Software zu integrieren.
Offenlegungen
Die hier zum Ausdruck gebrachten Meinungen sind die der Autoren und repräsentieren nicht die der U. S. Navy, der U. S. Army, des U. S. Department of Defense oder der US-Regierung.
Danksagungen
Diese Arbeit wurde von den Projektnummern CB-SEED-SEED09-2-0061 und CBCall4-CBM-05-2-0019 und teilweise vom Intramural/Extramural-Forschungsprogramm der Basisfonds NCATS, NIH (XH) und Naval Research Laboratory unterstützt.
Materialien
| Name | Company | Catalog Number | Comments |
| 250 mL Erlenmeyer Flask | VWR | 89000-362 | |
| 2-mercaptoethanol | Acros Organics (Fisher) | 125472500 | Danger: Acutely Toxic. Open bottle in hood to avoid inhaling the fumes. |
| 4 L Pyrex wide-mouth graduated Erlenmeyer flask with screw-cap | Millipore Sigma | CLS49954L-1EA | |
| AKTA Prime Plus | GE Healthcare | 17-0729-01 | |
| AKTA XK 16/20 Column | GE Healthcare | 28988937 | |
| Amicon Ultra-0.5 Centrifugal Filter Unit | Millipore Sigma | UFC501096 | |
| Amicon Ultra-15 Centrifugal Filter Unit | Millipore Sigma | UFC901096 | |
| Amicon Ultra-4 Centrifugal Filter Unit | Millipore Sigma | UFC801024 | |
| Ampicillin | Sigma | A0166 | Danger: Allergic reactions (skin or breathing). |
| Chelating Sepharose Fast Flow | GE Healthcare | 17-0575-02 | Once the resin is equilibrated with 0.2 M Nickel Sulfate it is refered to as a Nickel Column in the text. Column will have a green color after washing with water. The column will have a blue color after equilibrating with buffer. |
| Chloramphenicol | RPI | C61000 | Danger: May cause cancer. |
| Corning 50 mL centrifuge tubes | Corning | 430828 | Suggestion: Polypropylene tubes are less likely to crack during sonication than Polyethylene tubes |
| Corning 96 Well Half-Area Microplate, Non-Binding Surface | Corning | 3993 | |
| Dialysis Tubing Clips | Fisher Scientific | PI68011 | |
| Disposable PD-10 Desalting Column | GE Healthcare | 17-0851-01 | |
| DNAse | Sigma | DN25-1G | |
| DTT (DL-Dithiothreitol) | RPI | D11000-50.0 | Warning: Acute Oral Toxicity; skin and eye irritation |
| EDTA | Fisher Scientific | S311-500 | |
| Fisherbrand Petri Dishes with Clear Lid | Fisher Scientific | FB0875712 | |
| Glycerol | Acros Organics (Fisher) | 15892-0010 | |
| HEPES | Millipore Sigma | H4034-1KG | |
| Imidazole | Acros Organics | 301870010 | Danger: Toxic, Irritant |
| IPTG (Isopropyl β-D-thiogalactopyranoside) | Calbiochem (Millipore Sigma) | 420291 | Do not breathe dust. Avoid contact with eyes and skin. |
| Laemmli Sample Buffer | BIO-RAD | 1610737 | |
| Luria Bertani Agar | Fluka (Millipore Sigma) | L3027-1KG | Suggestion: Autoclave with magnetic stirrer in the liquid, and stir while cooling. Wait to add antibiotic until you can hold your hands on the bottle without pain for 30 seconds. |
| Luria Bertani Media | Fisher Bioreagents | BP1426-2 | |
| Lysozyme | Sigma | L4919-5g | |
| Mini-PROTEAN Tetra Vertical Electrophoresis CellGel Box | BIO-RAD | ||
| Nalgene Oak Ridge High-Speed PPCO Centrifuge Tubes | Nalgene (Thermo Scientific) | 3119-0050 | |
| Nanodrop | Thermo Fisher | ||
| New Brunswick Innova 42R Shaker Incubator | Eppendorf | M1335 | |
| Nickel Sulfate Hexahydrate (Crystalline/Certified ACS), Fisher Chemical | Fisher Scientific | N73-500 | Danger: Harmful if swallowed or inhaled, skin and eye irritation |
| One Shot BL21(DE3) Chemically Competent E. coli | Invitrogen (Thermo Fisher) | C600003 | May be harmful if inhaled or swallowed. May cause skin and eye irritation with susceptible people. |
| One Shot BL21(DE3) pLysS Chemically Competent E. coli | Invitrogen (Thermo Fisher) | C606003 | May be harmful if inhaled or swallowed. May cause skin and eye irritation with susceptible people. |
| pet15b plasmid DNA | Novagen (Millipore Sigma) | 69661 | GenScript Inc. was used for commerical DNA synthesis. The pet15b plasmid was used for the CFP/YFP substrates. |
| pet32b | Novagen (Millipore Sigma) | 69016-3 | The pet32b plasmid was used for the cysteine protease construct. |
| Pierce Protease Inhibitor Mini Tablets, EDTA-free | Thermo Fisher | A32955 | Warning: Skin corrosion/irriation; eye damage |
| Plate Reader | Molecular Devices | Model M5 | |
| Precision Plus Protein All Blue Prestained Protein Standard | BIO-RAD | 161-0373 | |
| Protein Extraction Reagent | Novagen (Millipore Sigma) | 70584-4 | BugBuster or Bper (Catalog # 78248, ThermoFisher) |
| Q-Sepharose Fast Flow | G.E. Healthcare | 17-0510-01 | Anion exchange resin |
| RunBlue (12%) 17-well PAGE gels | Expedeon | BCG01227 | Any 12% pre-cast polyacrylamide gel can be used |
| RunBlue 20x SDS Running Buffer | Expedeon | NXB50500 | Dilute 50 mL with 950 mL deionized water to obtain 1x |
| RunBlue Instant Blue Gel Stain | Expedeon | ISB1L | Do not dilute, use as directed |
| Sodium Chloride | Fisher Chemical | S271-10 | |
| Sonifier Cell Disrupter 450 Sonicator | Branson Ultrasonics (VWR) | Model No. 101-063-346R | Sonicator was used on level 5 |
| Spectra/Por 6-8 kD MWCO | Spectrum Labs | 132645T | Dialysis Tubing |
| SP-Sepharose Fast Flow | G.E. Healthcare | 17-0729-01 | Cation exchange resin |
| Thrombin from bovine plasma | Sigma | T6634-500UN | |
| Tris Base | Fisher Scientific | BP152-500 | Caution: Eye/Skin Irritant |
Referenzen
- Liu, H., et al. Widespread Horizontal Gene Transfer from Double-Stranded RNA Viruses to Eukaryotic Nuclear Genomes. Journal of Virology. 84 (22), 11876-11887 (2010).
- Hagai, T., Azia, A., Babu, M. M., Andino, R. Use of host-like peptide motifs in viral proteins is a prevalent strategy in host-virus interactions. Cell Reports. 7 (5), 1729-1739 (2014).
- Gorbalenya, A. E. Host-related sequences in RNA viral genomes. Seminars in Virology. 3, 359-371 (1992).
- Shmakov, S. A., et al. The CRISPR Spacer Space Is Dominated by Sequences from Species-Specific Mobilomes. MBio. 8 (5), 1-18 (2017).
- Legler, P. M., Morazzani, E., Glass, P. J., Compton, J. R. Proteome Editing System and A Biomarker of Veev Infection. United States patent application. , (2018).
- Morazzani, E. M., et al. Proteolytic cleavage of host proteins by the Group IV viral proteases of Venezuelan equine encephalitis virus and Zika virus. Antiviral Research. 164, 106-122 (2019).
- Alvarez, E., Castello, A., Menendez-Arias, L., Carrasco, L. HIV protease cleaves poly(A)-binding protein. Biochemical Journal. 396 (2), 219-226 (2006).
- Falk, M. M., et al. Foot-and-mouth disease virus protease 3C induces specific proteolytic cleavage of host cell histone H3. Journal of Virology. 64 (2), 748-756 (1990).
- Grigera, P. R., Tisminetzky, S. G. Histone H3 modification in BHK cells infected with foot-and-mouth disease virus. Virology. 136 (1), 10-19 (1984).
- Li, W., Ross-Smith, N., Proud, C. G., Belsham, G. J. Cleavage of translation initiation factor 4AI (eIF4AI) but not eIF4AII by foot-and-mouth disease virus 3C protease: identification of the eIF4AI cleavage site. FEBS Letters. 507 (1), 1-5 (2001).
- Kuyumcu-Martinez, M., et al. Calicivirus 3C-like proteinase inhibits cellular translation by cleavage of poly(A)-binding protein. Journal of Virology. 78 (15), 8172-8182 (2004).
- Pietila, M. K., Hellstrom, K., Ahola, T. Alphavirus polymerase and RNA replication. Virus Research. 234, 44-57 (2017).
- Hardy, W. R., Strauss, J. H. Processing the nonstructural polyproteins of sindbis virus: nonstructural proteinase is in the C-terminal half of nsP2 and functions both in cis and in trans. Journal of Virology. 63 (11), 4653-4664 (1989).
- Strauss, E. G., De Groot, R. J., Levinson, R., Strauss, J. H. Identification of the active site residues in the nsP2 proteinase of Sindbis virus. Virology. 191 (2), 932-940 (1992).
- Wang, D., et al. Foot-and-mouth disease virus 3C protease cleaves NEMO to impair innate immune signaling. Journal of Virology. 86 (17), 9311-9322 (2012).
- Aguirre, S., et al. DENV inhibits type I IFN production in infected cells by cleaving human STING. PLoS Pathogens. 8 (10), e1002934(2012).
- Barral, P. M., Sarkar, D., Fisher, P. B., Racaniello, V. R. RIG-I is cleaved during picornavirus infection. Virology. 391 (2), 171-176 (2009).
- Elbashir, S. M., Lendeckel, W., Tuschl, T. RNA interference is mediated by 21- and 22-nucleotide RNAs. Genes & Development. 15 (2), 188-200 (2001).
- Deveau, H., Garneau, J. E., Moineau, S. CRISPR/Cas system and its role in phage-bacteria interactions. Annual Review of Microbiology. 64, 475-493 (2010).
- Schechter, I., Berger, A. On the size of the active site in proteases I. Papain. Biochemical and Biophysical Research Communications. 27 (2), 157-162 (1967).
- Bieniasz, P. D. Intrinsic immunity: a front-line defense against viral attack. Nature Immunology. 5 (11), 1109-1115 (2004).
- Zhou, Z., et al. TRIM14 is a mitochondrial adaptor that facilitates retinoic acid-inducible gene-I-like receptor-mediated innate immune response. Proceedings of the National Academy of Sciences of the U S A. 111 (2), E245-E254 (2014).
- Wang, S., et al. TRIM14 inhibits hepatitis C virus infection by SPRY domain-dependent targeted degradation of the viral NS5A protein. Scientific Reports. 6, 32336(2016).
- Zacks, M. A., Paessler, S. Encephalitic alphaviruses. Veterinary Microbiology. 140 (3-4), 281-286 (2010).
- Hollidge, B. S., Weiss, S. R., Soldan, S. S. The role of interferon antagonist, non-structural proteins in the pathogenesis and emergence of arboviruses. Viruses. 3 (6), 629-658 (2011).
- Carthagena, L., et al. Human TRIM gene expression in response to interferons. PLoS One. 4 (3), e4894(2009).
- Montgomery, S. A., Johnston, R. E. Nuclear import and export of Venezuelan equine encephalitis virus nonstructural protein 2. Journal of Virology. 81 (19), 10268-10279 (2007).
- Nenasheva, V. V., et al. Enhanced expression of trim14 gene suppressed Sindbis virus reproduction and modulated the transcription of a large number of genes of innate immunity. Immunologic Research. 62 (3), 255-262 (2015).
- Nagai, T., et al. A variant of yellow fluorescent protein with fast and efficient maturation for cell-biological applications. Nature Biotechnology. 20 (1), 87-90 (2002).
- Li, M. Z., Elledge, S. J. SLIC: a method for sequence- and ligation-independent cloning. Methods in Molecular Biology. 852, 51-59 (2012).
- Hu, X., et al. Kinetic, Mutational, and Structural Studies of the Venezuelan Equine Encephalitis Virus Nonstructural Protein 2 Cysteine Protease. Biochemistry. 55 (21), 3007-3019 (2016).
- Morazzani, E. M., et al. Proteolytic cleavage of host proteins by the Group IV viral proteases of Venezuelan equine encephalitis virus and Zika virus. Antiviral Research. 164, 106-122 (2019).
- Zhang, D., Tozser, J., Waugh, D. S. Molecular cloning, overproduction, purification and biochemical characterization of the p39 nsp2 protease domains encoded by three alphaviruses. Protein Expression and Purification. 64 (1), 89-97 (2009).
- Lei, J., et al. Crystal structure of Zika virus NS2B-NS3 protease in complex with a boronate inhibitor. Science. 353 (6298), 503-505 (2016).
- Shiryaev, S. A., et al. Characterization of the Zika virus two-component NS2B-NS3 protease and structure-assisted identification of allosteric small-molecule antagonists. Antiviral Research. 143, 218-229 (2017).
- Smith, J. L., Jeng, S., McWeeney, S. K., Hirsch, A. J. A MicroRNA Screen Identifies the Wnt Signaling Pathway as a Regulator of the Interferon Response during Flavivirus Infection. Journal of Virology. 91 (8), (2017).
- Lee, Y. S., et al. The Wnt inhibitor secreted Frizzled-Related Protein 1 (sFRP1) promotes human Th17 differentiation. European Journal of Immunology. 42 (10), 2564-2573 (2012).
- Goodfellow, F. T., et al. Zika Virus Induced Mortality and Microcephaly in Chicken Embryos. Stem Cells and Development. 25 (22), 1691-1697 (2016).
- Morrison, T. E., Diamond, M. S. Animal Models of Zika Virus Infection, Pathogenesis, and Immunity. Journal of Virology. 91 (8), (2017).
- Morazzani, E. M., et al. Books of Abstracts, 254th American Chemical Society National Meeting. , Washington, D.C. (2017).
- Compton, J. R., Mickey, M. J., Hu, X., Marugan, J. J., Legler, P. M. Mutation of Asn-475 in the Venezuelan Equine Encephalitis Virus nsP2 Cysteine Protease Leads to a Self-Inhibited State. Biochemistry. 56 (47), 6221-6230 (2017).
- Vasiljeva, L., et al. Regulation of the sequential processing of Semliki Forest virus replicase polyprotein. Journal of Biological Chemistry. 278 (43), 41636-41645 (2003).
- Uchil, P. D., Quinlan, B. D., Chan, W. T., Luna, J. M., Mothes, W. TRIM E3 ligases interfere with early and late stages of the retroviral life cycle. PLoS Pathogens. 4 (2), e16(2008).
- Ozato, K., Shin, D. M., Chang, T. H., Morse, H. C. 3rd TRIM family proteins and their emerging roles in innate immunity. Nature Reviews Immunology. 8 (11), 849-860 (2008).
- van Tol, S., Hage, A., Giraldo, M. I., Bharaj, P., Rajsbaum, R. The TRIMendous Role of TRIMs in Virus-Host Interactions. Vaccines (Basel). 5 (3), (2017).
- Molaei, G., et al. Dynamics of Vector-Host Interactions in Avian Communities in Four Eastern Equine Encephalitis Virus Foci in the Northeastern U.S. PLoS Neglected Tropical Diseases. 10 (1), e0004347(2016).
- Ding, Q., et al. Species-specific disruption of STING-dependent antiviral cellular defenses by the Zika virus NS2B3 protease. Proceedings of the National Academy of Sciences of the U S A. 115 (27), E6310-E6318 (2018).
- Angov, E., Legler, P. M., Mease, R. M. Adjustment of codon usage frequencies by codon harmonization improves protein expression and folding. Methods in Molecular Biology. 705, 1-13 (2011).
- Ruge, D. R., et al. Detection of six serotypes of botulinum neurotoxin using fluorogenic reporters. Analytical Biochemistry. 411 (2), 200-209 (2011).
- Hu, X., et al. Structural insight into exosite binding and discovery of novel exosite inhibitors of botulinum neurotoxin serotype A through in silico screening. Journal of Computer-Aided Molecular Design. 28 (7), 765-778 (2014).
- Dunning, F. M., et al. Detection of botulinum neurotoxin serotype A, B, and F proteolytic activity in complex matrices with picomolar to femtomolar sensitivity. Applied and Environmental Microbiology. 78 (21), 7687-7697 (2012).
- Nguyen, T. G., et al. Development of fluorescent substrates and assays for the key autophagy-related cysteine protease enzyme ATG4B. Assay and Drug Development Technologies. 12 (3), 176-189 (2014).
- Zhang, J. H., Chung, T. D., Oldenburg, K. R. A Simple Statistical Parameter for Use in Evaluation and Validation of High Throughput Screening Assays. Journal of Biomolecular Screening. 4 (2), 67-73 (1999).
- Bovolenta, P., Esteve, P., Ruiz, J. M., Cisneros, E., Lopez-Rios, J. Beyond Wnt inhibition: new functions of secreted Frizzled-related proteins in development and disease. Journal of Cell Science. 121 (Pt 6), 737-746 (2008).
- Esteve, P., et al. SFRPs act as negative modulators of ADAM10 to regulate retinal neurogenesis. Nature Neuroscience. 14 (5), 562-569 (2011).
- Garcia-Hoyos, M., et al. Evaluation of SFRP1 as a candidate for human retinal dystrophies. Molecular Vision. 10, 426-431 (2004).
- Marcos, S., et al. Secreted frizzled related proteins modulate pathfinding and fasciculation of mouse retina ganglion cell axons by direct and indirect mechanisms. Journal of Neuroscience. 35 (11), 4729-4740 (2015).
- Moore, C. A., et al. Characterizing the Pattern of Anomalies in Congenital Zika Syndrome for Pediatric Clinicians. JAMA Pediatrics. 171 (3), 288-295 (2017).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten