Method Article
Grup IV Viral SSHHPS In Vitro ve In Silico Yöntemleri Kullanılarak Analizi
Bu Makalede
Özet
Viral poliproteine gömülü homolog konak-patojen protein dizilerinin (SSHHPS) kısa uzantılarını belirlemek için genel bir protokol sayılmiyoruz. SSHHPS viral proteazlar tarafından tanınan ve çeşitli Grup IV virüsler tarafından belirli konak proteinlerin hedefli imha doğrudan.
Özet
Alfaviral enzimler tek bir polipeptit içinde sentezlenir. Yapısal olmayan poliprotein (nsP) viral replikasyon için gerekli aktif enzimler üretmek için onun nsP2 sistein proteaz tarafından işlenir. Viral proteazlar son derece spesifiktir ve korunmuş dekolte yeri motif dizilerini tanırlar (~6-8 amino asitler). Bazı Grup IV virüslerinde, nsP proteaz(lar) dekolte yeri motif dizileri doğuştan gelen immün yanıtların oluşmasında rol oynayan belirli konak proteinlerde bulunabilir ve bazı durumlarda hedeflenen proteinler virüse bağlı fenotipile bağlantılı görünür. Bu virüsler, konak proteinlerin hedefli imhası için homolog konak-patojen protein dizilerinin (SSHHPS) kısa uzantılarını kullanırlar. SSHHPS'i tanımlamak için viral proteaz dekolte sit motif dizileri BLAST'a girilebilir ve konak genom(lar) aranabilir. Dekolte başlangıçta saflaştırılmış nsP viral proteaz ve floresan rezonans enerji transferi (FRET) substratları E. coliyapılan kullanılarak test edilebilir. FRET yüzeyleri siyan ve sarı floresan protein ve dekolte alanı dizisi (CFP-sequence-YFP) içerir. Bu proteaz tsay sürekli bir plaka okuyucu veya sds-PAGE jelleri sürekli olarak kullanılabilir. Bağlı peptid substrat modelleri substrat seçimi ve mutagenez çalışmaları rehberlik silico oluşturulabilir. CFP/YFP substratları da proteaz inhibitörlerini tanımlamak için kullanılmıştır. Bu in vitro ve in silico yöntemleri, hedeflenen konak proteininin viral replikasyonu etkileyip etkilemediğini belirlemek için hücre bazlı tahlillerle birlikte kullanılabilir.
Giriş
Virüsten konakveya virüse ev sahipliği yapmak için yatay gen transferi kanıtı, çeşitli genomlarda bulunabilir1,2,3,4. Viral endojenizasyona örnek olarak bakteriyel konak genomlarında bulunan CRISPR spacer dizileri4. Son zamanlarda,(+)ssRNA Grup IV virüslerinin yapısal olmayan poliproteinlerine gömülü konak protein dizilerinin kanıtlarını bulduk. Viral genomun kodlama bölgelerindeki bu diziler nesilsel olarak yayılabilir. Homolog konak-patojen protein dizilerinin (SSHHPS) kısa uzantıları virüste bulunur ve5,6. SSHHPS, spesifik konak proteinlere homolojisi olan viral proteazlar tarafından tanınan korunmuş dekolte yeri motif dizileridir. Bu diziler belirli konak proteinlerinin yok oluşuna yönlendirir.
Bir önceki yayınımızda6,viral proteazlar tarafından hedeflenen tüm konak proteinlerin bir listesini derledik ve hedef listesinin rastgele olmadığını tespit ettik(Tablo 1). İki eğilim belirgindi. İlk olarak, konak proteinleri kesen viral proteazların çoğu Grup IV virüslere (24'ü Grup IV viral proteazlar dahil 25 vakadan 24'ü) ve bir proteaz (+)ssRNA Group VI retroviruses (HIV, insan immün yetmezlik virüsü)7'yeaitti. İkinci olarak, viral proteazlar tarafından kesilen konak protein hedefleri genellikle dekoltelerin konakçının bağışıklık yanıtlarını antagonize etmek için tasarlandığını düşündüren doğuştan gelen bağışıklık yanıtlarının oluşmasında rol almıştır. Viral proteazların hedef açtığı konak proteinlerin yarısı interferon (IFN) ve proinflamatuar sitokinler üreten sinyal basamaklarının bilinen bileşenleridir(Tablo 1). Diğerleri ev sahibi hücre transkripsiyon8,9,10 veya çeviri11dahil edildi. İlginçtir, Shmakov ve ark.4 birçok CRISPR protospacer dizileri plazmid konjugasyon veya çoğaltma4dahil genlere karşılık olduğunu göstermiştir.
Grup IV, diğerleri arasında, Flaviviridae, Picornaviridae, Coronaviridae, Calciviridae ve Togaviridaeiçerir. Zika virüsü (ZIKV), Batı Nil (WNV), Chikungunya (CHIKV), şiddetli akut solunum sendromu virüsü (SARS) ve Orta Doğu solunum sendromu virüsü (MERS) gibi birçok yeni ve yeni ortaya çıkan patojen Grup IV'e aittir. (+)ssRNA genomu aslında bir mRNA parçasıdır. Genom replikasyonu için gerekli enzimleri üretmek için önce (+)ssRNA genomu tercüme edilmelidir. Alfavirüsler ve diğer Grup IV virüslerde, replikasyon için gerekli enzimler tek bir poliproteinde (yani VEEV için nsP1234) üretilir. Yapısal olmayan poliprotein (nsP) proteolyically işlenir (nsP1234 nsP1, nsP2, nsP3, nsP4) nsP2 proteaz tarafından aktif enzimler üretmek için12 (Şekil 1). NsP2 proteaz ile poliprotein bölünmesi viral çoğaltma için gereklidir; bu nsP2 proteaz13,14aktif site sistein delemesi ve site yönelimli mutagenez ile gösterilmiştir. Özellikle, viral proteinlerin çevirisi genom replikasyon olaylarından önce gelir. Örneğin, nsP4 (+)ssRNA genomunu çoğaltmak için gereken RNA'ya bağımlı RNA polimeraz içerir. Genom replikasyonu dsRNA ara üretebilir; bu ara lar konakçının doğuştan gelen bağışıklık yanıtlarını tetikleyebilir. Böylece, Bu virüsler etkilerini bastırmak için enfeksiyon erken konak doğuştan gelen bağışıklık yanıtı proteinleri cleave olabilir15,16,17.
Susturma DNA, RNA ve protein düzeyinde oluşabilir. Şekil 1'de gösterilen susturma mekanizmalarının her birinde yaygın olan şey, işlevlerini antagonize etmek için belirli hedeflerin yok edilmesine rehberlik etmek için kısa yabancı DNA, RNA veya protein dizilerinin kullanılmasıdır. Susturma mekanizmaları, üç farklı dilde yazılmış "arama ve silme" programlarına benzerdir. Kısa dekolte site sırası bir "anahtar kelime" benzer. Her program kısa sıra ("anahtar kelime") ve silinecek olan "dosya" bir kelime arasındaki eşleşmeyi tanıyan bir enzim vardır. Bir eşleşme bulunduğunda, enzim daha büyük hedef sırasını keser ("siler"). Şekil 1'de gösterilen üç mekanizma, konakçıyı virüslerden korumak veya bir virüsü bir ev sahibinin bağışıklık sisteminden korumak için kullanılır.
Viral proteazlar ~ 2-11 amino asitler arasında kısa bölünme site motif dizileri tanımak; nükleotitlerde bu 6-33 baza karşılık gelir. Karşılaştırma için, CRISPR spacer dizileri ~ 26-72 nükleotit ve RNAi ~ 20-22 nükleotitler18,19vardır. Bu diziler nispeten kısa olsa da, özellikle tanınabilir. Amino asitlerin yüksek çeşitliliği göz önüne alındığında, rastgele bir bölünme olayı olasılığı nispeten düşük bir viral proteaz 6-8 amino asit veya daha uzun protein dizileri tanıyan için. Konak proteinlerde SSHHPS'nin tahmini büyük ölçüde incelenen viral proteazın özgüllüğüne bağlıdır. Proteaz ın kesin dizi özgüllük gereksinimleri varsa, dekolte site dizisi bulma şansı 1/206 = 1 64 milyonda veya 1/208 = 1'dir 25,6 milyarda; ancak proteazların çoğunda değişken alt alan toleransları vardır (örneğin, R veya K S1 bölgesinde tolere edilebilir). Sonuç olarak, ana bilgisayarda bulunan diziler ile virüs arasında dizi kimliği gereksinimi yoktur. Daha gevşek dizi gereksinimleri olan viral proteazlar için (Picornaviridae'ye ait olanlar gibi) bir konak proteinde dekolte bölgesi bulma olasılığı daha yüksek olabilir. Tablo 1'deki girişlerin çoğu Picornaviridae ailesindendir.
Schechter & Bergergösterimi 20 yaygın bir proteaz substrat ve onlar bağlamak alt siteleri kalıntılar tanımlamak için kullanılır, biz boyunca bu gösterim kullanmak. Makas bağının N-terminali olan substrattaki kalıntılar P3-P2-P1 olarak gösterilirken, C-terminali olanlar P1'-P2'-P3' olarak gösterilir. Bu amino asit kalıntıları bağlamak proteaz karşılık gelen alt siteleri Sırasıyla S3-S2-S1 ve S1'-S2'-S3', vardır.
Hangi ana proteinlerin hedeflendiğini belirlemek için viral poliprotein bölünme bölgelerindeki SSHHPS'yi belirleyebilir ve bunları içeren konak proteinleri arayabiliyoruz. Burada, bilinen viral proteaz dekolte site dizilerini kullanarak SSHHPS'nin tanımlanmasına yönelik prosedürleri ana hatlar. Biyoinformatik yöntemler, proteaz testleri ve tanımlanan silico metodlarında hücre bazlı tahlillerle birlikte kullanılması amaçlanmıştır.
Viral proteazların hedef açtığı konak proteinlerin dizi dizileri bu kısa bölünme alanı dizileri içinde türe özgü farklılıklar ortaya koymuştur. Örneğin, Venezuela at ensefaliti virüsü (VEEV) nsP2 proteaz insan TRIM14, üçlü motif (TRIM) protein6kesmek bulundu. Bazı TRIM proteinleri viral restriksiyon faktörleridir (örneğin, TRIM5α21),çoğu ubiquitin E3 ligazları olduğu düşünülmektedir. TRIM14 bir RING yoksun (gerçekten ilginç yeni gen) etki alanı ve bir E3 ligase olduğu düşünülmemaktadır22. TRIM14 mitokondriyal antiviral sinyalozom bir adaptör olduğu ileri sürülmuştur (MAVS)22, ancak diğer antiviral fonksiyonları olabilir23. Trim14 dizilerinin çeşitli türlerden hizalanması, atların dekolte alanından yoksun olduğunu ve C-terminal PRY/SPRY etki alanını eksik olan TRIM14'ün kesilmiş bir sürümünü barındırdığını göstermektedir. Bu etki alanı bir poliubiquitination sitesi içerir (Şekil 2). At, bu virüsler son derece ölümcül (~ 20-80% mortalite) insanlarda ise sadece ~ 1% VEEV enfeksiyonları24ölür. PRY/SPRY etki alanının bölünmesi, MAVS sinyalajı basamaklama da geçici olarak kısa devre yapabilir. Bu basamak dsRNA tarafından tetiklenebilir ve interferon ve pro-inflamatuar sitokinüretimine yol açar. Bu nedenle, SSHHPS varlığı belirli Grup IV virüslere karşı hangi türlerin savunma sistemleri olduğunu tahmin etmek için yararlı olabilir.
Grup IV virüslerde IFN antagonizm mekanizmalarının25'içarpın. Konak protein bölünmesi enfeksiyon sırasında geçici olabilir ve konsantrasyonlar zamanla düzelebilir. Hücrelerde TRIM14 dekolte ürünlerinin transfeksiyondan sonra çok erken tespit edilebildiğini (6 saat) proteazı (sitomegalovirüs organizatörü) kodlayan bir plazmid ile bulduk. Ancak, daha uzun sürelerde, dekolte ürünleri tespit edilmedi. Virüs enfekte hücrelerde kinetik ler farklıydı ve dekolte ürünleri 6-48 h6arasında tespit edilebilir. Diğerleri erken 3-6 h sonrası enfeksiyon9,11gibi erken konak protein dekolte ürünlerinin görünümünü bildirdin.
Hücrelerde proteolitik aktivite genellikle yakalamak zordur; dekolte ürünleri çözünürlüğü, konsantrasyonu, stabilitesi ve kullanım ömrü açısından değişebilir. Hücre bazlı tahlillerde, dekolte ürünlerinin bir hücrede birikeceği veya kesilen ve kesilmemiş proteinin bant yoğunluklarının kompansatuar artışlar göstereceği ve kesilen proteinin çok hızlı bir şekilde bozulabileceği ve beklenen molekül ağırlığında (MW) batı lekesinde tespit edilemeyeceği varsayılamaz (örneğin, epite içeren bölge diğer konakçı proteasesveya bedide verilebilir). Viral proteaz substrat doğuştan gelen bir bağışıklık yanıtı proteiniseniz, konsantrasyonu enfeksiyon sırasında değişebilir. Örneğin, bazı doğuştan gelen immün yanıt proteinleri viral enfeksiyon öncesinde mevcut ve interferon tarafından daha fazla indüklenen26. Bu nedenle hedef proteinin konsantrasyonu enfeksiyon sırasında dalgalanabilir ve enfekte olmayan hücre lisatlarının karşılaştırılması zor olabilir. Ayrıca, tüm hücreler düzgün transfected veya enfekte olmayabilir. Öte yandan E. coli saflaştırılmış proteinler kullanılarak in vitro proteaz tahlilleri kontrol etmek için daha az değişken var ve bu tür tahliller immünoblots yerine SDS-PAGE kullanılarak yapılabilir. Kontamine proteazlar CFP /YFP substrat protein saflaştırma erken aşamalarında inhibe edilebilir, ve mutasyona uğramış viral proteazlar saflaştırılmış ve dekolte viral proteaz veya kontamine bakteriyel proteaz nedeniyle olup olmadığını belirlemek için kontrol olarak test edilebilir.
In vitro proteaz tahlillerinin bir sınırlaması, bir memeli hücresinin karmaşıklığından yoksun olmalarıdır. Bir enzimin substratını kesmesi için ikisinin birlikte lokalize olması gerekir. Grup IV viral proteazlar yapı ve lokalizasyon açısından farklılık gösterir. Örneğin, ZIKV proteazendilazmik retikulum gömülü ve sitosol yüzleri, VEEV nsP2 proteaz sitoplazma ve çekirdek27çözünür bir protein ise . ZIKV SSHHPS analizinde bulunan bazı dekolte site dizileri sinyal peptidlerinde olup dekoltenin bazı hedefler için birlikte çevrim selamiyet olabileceğini düşündürmektedir. Bu nedenle bu analizlerde proteazın ve hücredeki substratların yeri de göz önünde bulundurulmalıdır.
Hücre bazlı tahliller, enfeksiyonda tanımlanan konak protein(ler) için bir rol oluşturmak için değerli olabilir. Konak proteinlerin viral proteaz dekoltesini durdurmayı amaçlayan yöntemler, örneğin proteaz inhibitörü6 veya konak hedef16'da mutasyon gibi viral çoğalma üzerindeki etkilerini incelemek için kullanılabilir. Hedeflenen proteinin aşırı ekspresyonu da viral replikasyonu etkileyebilir28. Plak tahlilleri veya diğer yöntemler viral replikasyon ölçmek için kullanılabilir.
Protokol
1. Biyoinformatik: BLAST Kullanarak Konak Genomunda SSHHPS'nin Tanımlanması
NOT: Protein BLAST blast.ncbi.nlm.nih.gov/Blast.cgi bulunabilir.
- Viral poliproteinde makas bağını çevreleyen ~20 amino asit girişi. Aranacak yedeksiz protein dizilerini seçin ve konak genomundaki tipini seçin (örn. Homo sapiens).
- Gerekirse PHI-BLAST'ı seçin. Bir desen dizisi yazın (örneğin, aşağıda gösterilen V12'nin 25 kalıntısı için tırnak işareti olmadan "AG" desenini girin).

NOT: PHI-BLAST'da kare parantez [XY] amino asit X veya Y'nin alt konumda olabileceğini gösterir (örneğin, AG[AC][GAY]). - BLAST sonuçlarını inceleyin ve poliprotein bölünme bölgelerinde korunan kalıntılara yüksek sıralı kimliğe sahip isabetleri tanımlayın (örn. üçlü motif protein iprotein 14)(Şekil 3).
NOT: Serine proteazlar için P1 kalıntısının daha yüksek korunması beklenirken, sistein lar için P2 kalıntısının daha yüksek korunması beklenmektedir. - Bir dekolte site dizisiile aynı olan ve sıralı sırayla (boşluk yok) kalıntıları renklendirin. Alt sitede tolere edilen ancak farklı bir dekolte yerinde ikinci bir renkte bulunan kalıntıları renklendirin.
NOT: Viral bir dekolte bölgesinde bulunmayan konservatif ikameleri (örneğin, Leu vs. Val) temsil eden kalıntılar da bulunabilir ve viral proteaz tarafından tanınabilir veya tanınmayabilir. - Blast vuruşsırasını, bir dekolte alanı dizisiyle eşleşen ardışık özdeş veya tolere edilen kalıntısayısına göre sıralayın. Listeden, proteaz tahlillerinde analiz için ≥6 özdeş veya benzer kalıntılar içeren proteinleri seçin.
- Diğer dekolte bölgeleri (nsP2/3, nsP3/4, vb.) için prosedürü tekrarlayın ve PHI-BLAST desenine daha yüksek korunmuş kalıntılar ekleyerek tahmini kademeli olarak güçlendirin.
- Gerekirse PHI-BLAST'ı seçin. Bir desen dizisi yazın (örneğin, aşağıda gösterilen V12'nin 25 kalıntısı için tırnak işareti olmadan "AG" desenini girin).
2. In Vitro Tahlilleri: Proteaz Yüzeylerin Tasarımı ve Hazırlanması
- Siyan floresan protein (CFP), ≤25 amino asitler dekolte site dizisi, sarı floresan protein (YFP, ayrıca Venüs29olarak da bilinir) takip kodlama bir plazmid oluşturmak.
NOT: Plazmid dizi ve ligasyon bağımsız klonlama (SLIC)30 veya ticari gen sentezi kullanılarak inşa edilebilir. Şekil 4'te gösterilen diziyi içeren pet15b plazmid ticari olarak sentezlenmiş ve burada kullanılmıştır.- Substrat uzunluğunu optimize etmek için, 2 parçalı SLIC reaksiyonu kullanarak doğal viral poliprotein bölünme bölgesi dizilerinin 12-25 amino asitlerini içeren ek değişken uzunlukta FRET yüzeyleri inşa edin. SDS-PAGE jel bazlı analizi kullanarak veya aşağıdaki yöntemleri kullanarak sabit durum kinetik parametrelerini ölçerek dekolteyi analiz edin.
NOT: Bazı durumlarda, dekolte siteleri bilinen dekolte siteleri31homoloji ile tespit edilebilir. Poliprotein kavşak dizilerini içeren yüzeylerin bölünmesi gözlenmezise, ek kalıntılar veya yapısal bir motif (örneğin, alfa sarmal32)için bir gereklilik olabilir. Alternatif olarak, saflaştırılmış viral proteaz inaktif olabilir. SSHHPS analizini yapmadan önce viral poliprotein dizilerinin bölünmesini doğrulayın. Substrattaki kalıntı sayısı değişken uzunlukta substratlar (12-25 amino asitler) kullanılarak VEEV proteazı için optimize edilmiş ve ardından Vmax ve Km32,33analiz leri izlemiştir. Örneklerde kullanılan Zika viral ns2B/nsB proteaz dekolte siteleri34,35yayınlanmıştır.
- Substrat uzunluğunu optimize etmek için, 2 parçalı SLIC reaksiyonu kullanarak doğal viral poliprotein bölünme bölgesi dizilerinin 12-25 amino asitlerini içeren ek değişken uzunlukta FRET yüzeyleri inşa edin. SDS-PAGE jel bazlı analizi kullanarak veya aşağıdaki yöntemleri kullanarak sabit durum kinetik parametrelerini ölçerek dekolteyi analiz edin.
- 8-20 μL BL-21(DE3) yeni dönüştürerek CFP/YFP yüzeyleri hazırlayınE. coli50 g/mL Ampicillin (37 °C) içeren Luria Bertani (LB) agar plakaları üzerinde üreticinin talimatlarına ve plakasına göre CFP-V12-YFP plazmidli yetkili hücreler.
- Otoklav dört 4 L şişelb medya içeren (şişe başına 1.5 L medya) ve 250 mL şişe lb 100 mL. Kapak alüminyum folyo ile her şişe.
- 100 mL kültürünü, yeni dönüştürülmüş bakterilerin kolonisi ile aşılamak ve bir gecede sallayarak (200 rpm) 37 °C'de büyüyün.
- CFP/YFP alt tabakasını yapmak için, bir gecede 25 mL'lik dört 4 L şişeyi aşındırın. 37 °C'de kültürleri sallamaya başlayın ve uv-vis spektroskopisi ile büyümeyi saatte 600 nm hızla izleyin.
- Bakteriler 600 nm'de ~1.0 (yaklaşık ~3-4 h büyüme) bir absorbasyona ulaştığında şişe başına 0.5 mL 1 mopropil-β-D-tiogalaktoside (IPTG) ekleyerek protein ekspresyonuna neden olur. IPTG ekledikten sonra, sallayarak kuvöz sıcaklığını 17 °C'ye düşürün ve ifadenin bir gecede 17-20 saat boyunca devam etmesini bekleyin.
- 10 dk (4 °C) için 7.000 x g yüksek hızlı santrifüj kullanarak bakteri pelet ve pelet korumak. Sıvı ortamı çıkarın ve atın. Peletleri -80 °C'de veya lyse'de hemen saklayın.
- 50 mM Tris pH 7.6, 500 mM NaCl, 35 mL bakteriyel protein ekstraksiyonretisu, 30 mg lizozim, 25 U DNase ve 1 proteaz inhibitörü tablet içeren 100 mL lizis tamponu hazırlayın. Bir pipet ile lysis tamponpeletre resuspend ve 50 mL tek kullanımlık konik tüpler içine ~ 25-35 mL aktarın.
- Tüpleri buzlu su içeren plastik bir kabın içine yerleştirin. Uçları tüpün altından ~1 cm olacak şekilde tüplere sonicator ucunu yerleştirin ve lysate sıvı hale gelene ve sıvılaştırılana kadar 15 s aralıklarla seviye 5'te 10-20 kez sonicate olun.
NOT: Sonication sırasında işitme koruması kullanın. - Lysat'ı yüksek hızlı santrifüj tüplerine ve santrifüje 20.500 x g'de 4 °C'de 30 dk'ya aktarın. Döndükten sonra, supernatant (~ 100 mL) korumak ve temiz bir şişe aktarın. Peletleri atın.
- 1 L Arabellek A (50 mM Tris pH 7.6, 500 mM NaCl) hazırlayın. 300 mL Tampon B (50 mM Tris pH 7.6, 500 mM NaCl, 300 mM Imidazol) hazırlayın.
- 3 sütun hacimli Tampon A ve 5 mL/dk akış hızı kullanarak 100 mL nikel kolonu dengeleyin.
- Lysate'yi 2 ila 5 mL/dk akış hızı kullanarak nikel sütununa yükleyin. Kolonda 2 sütun hacmi ile Arabellek A, ardından ~5 sütun hacimleri %20 Tampon B'yi yıkayın. %20 Tampon B yıkama sırasında, yıkama sırasında kolondan kirletici madde olarak 280 nm (A280)absorbesyanı artacaktır. Eluate'ninA 280'i temel değerlere dönene kadar sütunu yıkamaya devam edin.
- 2-5 mL/dk akış hızı kullanarak proteini %100 Tampon B'ye sahip 2-3 kolon hacmiile yakınve 10 mL fraksiyonu toplayın. Her kesirin A280'ini ölçün.
- 15 mL santrifüj ultrafiltrasyon ünitesi kullanarak280 > 0.1 içeren kesirleri birleştirin ve konsantre edin. Ultrafiltrasyon ünitelerini 15 dakika boyunca 5.000 x g'da döndürün ve hacim ~50-75 mL'ye düşürülene kadar kesireklemeye devam edin.
- 6-8 kDa moleküler ağırlık kesme (MWCO) ile diyaliz boru 14 inç parça kesin. Diyaliz borusu kaynatılır ve 10 dk. Herhangi bir çatlak veya sızıntı olmadığından emin olmak için çantayı diyaliz tamponuyla doldurun. Tamponu torbadan çıkarın ve çantayı diyaliz tamponuna batırın.
- Konsantre proteini 2.2.13'ten plastik pipetle diyaliz torbasına aktarın. Çantadan hava kabarcıkları çıkarın. Çantayı ikinci bir düğüm veya diyaliz klibiyle kapatın. 50 mM Tris pH 7.6, 5 mM EDTA (etilendiamintetraasetik asit), 250 mM NaCl 500 mL'lik silindire karşı proteini bir gecede 4 °C'de diyalize alın.
- Proteini 50 mL 50 mL'lik 50 mM Tris pH 7.6'ya karşı 4 °C'de 2 saat diyalize alın.
- Aniyon değişim sütunu için 500 mL Tampon A (50 mM Tris pH 7.6) ve 500 mL Tampon B (50 mM Tris pH 7.6, 1.0 M NaCl) hazırlayın. 30 mL'lik bir anion değişim sütununu 3 sütun hacmine (2-5 mL/dk) dengeleyin.
- Diyaliz torbasından proteini çıkarın ve bir şişeye aktarın. Şişeyi buzda tut. Diyaliz proteinini kolona (2-5 mL/dk) yükleyin.
NOT: CFP/YFP proteini kolonu bağlar ve görünüşte sarı olur. - A280 taban çizgisine (5 mL/dk) dönene kadar sütunu Arabellek A ile yıkayın. Proteini bir degrade kullanarak (0-50% Tampon B, 100 mL) kullanarak elute ve 10 mL fraksiyonları toplamak.
- SDS-PAGE kullanarak sütun kesirlerini inceleyin. Bu birleştirin >95% saf.
- 15 mL santrifüj ultrafiltrasyon ünitesi kullanarak proteini A280 ~10-20'ye yoğunlaştırın. 4 °C'de 10 dakika boyunca 4.500 x g'de konsantratörü döndürün ve protein içeren fraksiyonların tümü birleştirilene kadar protein eklemeye devam edin.
- Diyaliz torbasından proteini çıkarın ve bir şişeye aktarın. Şişeyi buzda tut. Diyaliz proteinini kolona (2-5 mL/dk) yükleyin.
- Bir pipet ile yoğunlaştırıcıdan proteini dikkatlice çıkarın. Aliquot protein 1.5 mL mikrosantrifüj tüpler ve -80 °C'de uzun süreli depolama için sıvı nitrojen içinde flaş dondurma içine. Tampon, kullanmadan önce uygun analiz arabelleği ile dengelendirilen bir PD-10 sütunu kullanarak proteini oda sıcaklığında değiştirin.
- Bira yasası kullanılarak Protein konsantrasyonu A280 ve hesaplanan yok olma katsayısı (örneğin, V12 substratı için = 47.790 M -1 cm-1) kullanılarak hesaplanır.
NOT: Yok olma katsayısı (https://web.expasy.org/protparam/) Expasy ProtParam programı kullanılarak Şekil 4'teki protein dizisinden hesaplanabilir.
3. Alphaviral nsP2 Sistein Proteaz hazırlanması
- Proteazı kodlayan bir plazmid tasarlayıp inşa edin. Sistein proteazları için, bir thioredoxin oluşturmak için pet32 plazmid kullanın (Trx) füzyon proteini.
Not:Pet32 plazmid thioredoxin ve His-tag kaldırılması için bir trombin dekolte sitesi (LVPRGS) kodlar (Şekil 5). Thioredoxin ifade sırasında azaltılmış bir durumda aktif site sistein korumaya yardımcı olacaktır. Serin proteazlar için, tioredoksin gerekli değildir ve trombin ile çıkarılması içeren adımlar atlanabilir. VEEV nsP2 proteaz sekansı, Select ajanlarının kullanımından kaçınmak için ticari olarak hazırlanan pet32b plazmidiniçine dahil edilmiştir.- Plazmid DNA'sını üreticinin talimatlarına göre BL21(DE3)pLysS E. coli'ye yeni dönüştürün. Plaka LB agar plakaları ampisilin içeren bakteri.
NOT: Kloramfenikol sadece pLysS plazmid taşıyan E. coli suşları için kullanılır ve BL21 (DE3) hücreleri kullanılırsa atlanır. Bu adımda LB agar plakaüzerinde kloramfenikol dahil etmek gerekli değildir. - Otoklav dört 4 L şişe 1.5 L LB medya (6 L toplam hacim) ve 100 mL LB bir 250 mL şişe. Kapak alüminyum folyo ile her şişe.
- LB/Ampicillin'in 100 mL'lik bir gecede kültürünü tabaktan bir koloniyle aşılamak ve 37 °C'de sallayarak bir kuluçka makinesinde (200 rpm) büyüyün.
- 4 L şişeyi gece kültürünün 25 mL'si ile aşılamak ve uygun antibiyotiklerekleyin.
NOT: Pet32 plazmidtaşıyan BL21(DE3) pLysS hücrelerinin medyası 25 μg/mL kloramfenikol ve 50 μg/mL Ampicillin son konsantrasyonlara sahip olmalıdır. - 600 nm'deki absorbans 1.0'a ulaştığında kültüre 0,5 mL IPTG ekleyerek protein ekspresyonunu indükleyin. Sallayarak inkübatörün sıcaklığını 17 °C'ye düşürün. İfadenin bir gecede devam etmesine izin verin (~17 saat).
- Hücreleri santrifüjile pelet (4 °C'de 10 dk için 7.000 x g). Sıvı ortamı çıkarın ve atın.
NOT: Peletler -80 °C'de aylarca saklanabilir veya hemen lysed. - 100 mL lizis tamponu hazırlayın (50 mM Tris pH 7.6, 500 mL NaCl, 2 mM beta merkaptoetanol (BME), 30 mg lizozim, %5 gliserol, 25 U DNase, 35 mL bakteriyel protein ekstraksiyon remuarı). Eklerken kimyasal bir başlık içinde BME şişeleri açın. Bakteriyel lizatı buzda veya 4 °C'de saklayın.
NOT: Sistein proteazları için nükleofilik sisteinı azalmış tutmak için 2 mM BME bulunur. Sütunlar soğutulmuş arabellekler kullanılarak oda sıcaklığında çalıştırılabilir. Tamponlar 4 °C'ye soğutulmuş soğuk deiyonize su ile yapılmalıdır. - ~25 mL lysis tamponundaki bakteriyel peletleri yeniden askıya alın ve lizatın ~25 mL'sini 4 x 50 mL tek kullanımlık konik tüplere aktarın. Tüpleri buzlu su içeren plastik gagalara yerleştirin. 15 s aralıklarla seviye 5'te 10 kez lysate sonicate.
- Lysate'yi yüksek hızlı santrifüj tüplerine aktarın. Santrifüj (30 dk, 4 °C'de 20.500 x g) lysate'yi netleştirin.
- 0,5 L Tampon A (50 mM Tris pH 7.6, 500 mM NaCl, %5 gliserol, 2 mM BME) hazırlayın ve 4 °C'ye kadar soğutun.
- 250 mL Tampon B (50 mM Tris pH 7.6, 500 mM NaCl, %5 gliserol, 2 mM BME, 300 mM imidazol) hazırlayın ve 4 °C'ye kadar soğutun.
- 50 mL'lik nikel kolonu 3 kolon hacmi nezle satarak Dengeleyin A. Açıklığa kavuşturulan lysate'yi 2-5 mL/dk'da kolona yükleyin ve peletleri atın.
- Sütunu (2-5 mL/dk) 2 sütun hacmi yle birlikte %20 Buffer B (60 mM Imidazol) içeren 5 sütun hacmi Yle Arabellek A'yı yıkayın. Proteini (5 mL/dk) %100 Tampon B ile yakınve 10 mL fraksiyonu toplayın.
- 15 mL santrifüj ultrafiltrasyon ünitesi ve 4 °C'de 5.000 x g'de 15 dk dönüş kullanarak280 ≥ 0.1'e sahip proteaz içeren fraksiyonları birleştirin ve konsantre edin. Hacim ~5 mL'ye düşürüldükten sonra, proteine taze diyaliz tamponu (50 mM Tris pH 7.6, 250 mM NaCl, 5 mM dithiothreitol (DTT), 1 mM EDTA, %5 Gliserol) ekleyerek konsantrasyon ünitesindeki proteini tampon değişimi ile değiştirin. 15 dakika boyunca 4 °C'de 5.000 x g'de tekrar döndürün; tampon değişim adımını 2-3 kez tekrarlayın. Tioredoksin ve His-tag kaldırmak için diyaliz öncesi protein (20 μL 1 birim / μL) trombin ekleyin.
- Proteini bir diyaliz torbasına aktarın ve bir gecede 500 mL'lik silindirde 500 mL diyaliz tamponuna (4 °C) karşı diyaliz etiği.
NOT: FPLC (hızlı protein sıvı kromatografisi) sistemi ve nikel kolonu, aniyon değişim sütununa geçmeden önce sıyırma tamponu (2 M NaCl, 50 mM EDTA) ile iyice temizlenmelidir. FPLC hatlarındaki artık nikel, dtt içeren tampon çözeltilerini karıştırıldığında kahverengiye çevirir. Nikel kolon ve FPLC sistemini 4 kolon hacimli su ile yıkayın. Pompa su ile iyice FPLC sistemi yıkayın. Nikel kolon sonraki arıtmalar için reçine üzerinde 0,2 M nikel sülfat 2 kolon hacimleri akan tarafından rejenere edilebilir.
- Plazmid DNA'sını üreticinin talimatlarına göre BL21(DE3)pLysS E. coli'ye yeni dönüştürün. Plaka LB agar plakaları ampisilin içeren bakteri.
- Anyon değişim sütunu için 1 L Tampon A (50 mM Tris pH 7.6, 5 mM DTT, %5 gliserol) hazırlayın.
- Hazırlayın 0.5 L Tampon B (50 mM Tris pH 7.6, 5 mM DTT, 5% gliserol, 1.25 M NaCl).
- Arabellek A (3 sütun hacmi, 2-5 mL/dk) ile 30 mL'lik bir anion değişim sütununu dengeleyin. Akış yoluyla toplama için fraksiyon toplayıcı tüpler yerleştirin.
NOT: VEEV proteaz8.7 hesaplanan izoelektrik noktası (pI) vardır ve katyon değişim sütunları bağlayacak ama anyon değişim sütunları üzerinden akacaktır. PI Expasy ProtParam programı(https://web.expasy.org/protparam/)kullanılarak protein dizisinden hesaplanabilir. - Aracıklı proteini 1:3'ü Tampon A ile seyreltin ve sonra proteini (5 mL/dk) yükleyin. 10 mL kesirlerde akış-through toplamak.
- Aiyon değişim sütununu FPLC sisteminden kaldırın. Bir katyon değişim sütununu FPLC sistemine bağlayın. 30 mL katyon değişim sütununu 3 sütun hacmi ile Arabellek A (5 mL/dk) dengeleyin.
- Anyon değişim sütunundaki akışı 2-5 mL/dk'daki katyon değişim sütununa yükleyin. A280 taban çizgisine dönene kadar sütunu Arabellek A ile yıkayın. Proteini 100 mL degrade (%0-50 Tampon B) ile elute ve 10 mL fraksiyonları toplayın.
NOT: VEEV proteazı yaklaşık 0,6 M NaCl'de geçecektir. - SDS-PAGE kullanarak sütun kesirlerini inceleyin. 15 mL santrifüj ultrafiltrasyon üniteleri kullanarak %gt;95 saflıkta olan kesirleri birleştirin ve280 ° 2'ye konsantre olun. Enzim sıvı nitrojende yanıp sönebilir ve -80 °C'de depolanabilir.
- Anyon değişim sütunundaki akışı 2-5 mL/dk'daki katyon değişim sütununa yükleyin. A280 taban çizgisine dönene kadar sütunu Arabellek A ile yıkayın. Proteini 100 mL degrade (%0-50 Tampon B) ile elute ve 10 mL fraksiyonları toplayın.
4. Enzimin Sürekli Olarak Levha Okuyucu Kullanmasını Atama
- 50 mL çıkma arabelleği (50 mM HEPES pH 7.0) hazırlayın.
- Alfaviral proteazlar nispeten düşük kkedi değerlerine sahip olduğu için, tsay tamponundaki enzimi 4,7 μM'ye seyreltin (bu, TRX'siz VEEV proteazı için kabaca280 = 0,2'ye karşılık gelecektir).
- Enzimin aktivitesini ölçmek için, 185 μM konsantrasyonu ile assay tamponunda bir substrat stoku hazırlayın; bu kabaca bir A280 = 9 karşılık gelecektir. 8 mikrosantrifüj tüplerinde, 185 μM substrat stok ve tamponun uygun hacimlerini birleştirerek Tablo 2'de gösterilen reaksiyon karışımlarını hazırlayın. Siyah yarım alan 96 kuyulu bir plaka borusunda reaksiyonun 45°L'si 3 kuyuya karışır (sütun 1, 2, 3). A satırı [S] = 5 μM reaksiyon karışımını içermelidir ve H satırı [S] = 140 μM reaksiyon karışımını içermelidir.
- Plaka okuyucuyu sabit fotoçarpan tüpü (PMT) ayarı (örn. düşük) ile aynı anda iki dalga boyunda floresans algılayacaktır:
Dalga boyu 1 uyarma = 434 nm,emisyon = 527 nm
Dalga boyu 2 uyarma = 434 nm,emisyon = 470 nm - Okuma süresini 20 dakikaya (dakikada 1 okuma ölçmeye) ayarlayın ve okunacak kuyuları seçin. Plakayı plaka okuyucuya yerleştirin ve 20 dk. Zaman içinde emisyon oranlarını (470'te 527/emisyonda emisyon) izleyin.
- "UNCUT" alt katmanını içeren plakanın bitiş noktası okumasını çalıştırın.
NOT: Bu değerler sonraki veri hesaplamalarında kullanılacaktır. 3 kuyudaki emisyon oranlarının ortalaması Tablo 3'tekit = 0'daki "UNCUT" alt tabakasının değerleri olacaktır. - Her kuyuya 5°L'lik bir enzimin plakasını ve pipetini çıkarın. Dakikada 1 okuma ile 20 dakika boyunca tekrar plaka okuyun. Plaka okuyucuyu mutlak değerlere göre ayarlayın.
NOT: Bu tisme için, yamaçlar negatif olacaktır. Her kuyu toplam 50 μL hacim içerecektir. - Okumanın sonunda, buharlaşmayı önlemek için plakayı filmle kapatın. Enzimin substratı tamamen kesmesini sağlamak için tabağı bir gece boyunca oda sıcaklığında bırakın.
- ~24 saat sonra sızdırmazlık filmini çıkarın ve önceki plakalarda olduğu gibi aynı PMT'yi kullanarak plakanın bitiş noktası okumasını gerçekleştirin. Bu emisyon oranlarını ve "CUT" altında Tablo 3'e girdiyi ortalama. Aşağıda açıklanan SDS-PAGE kesintili testini kullanarak substrat bölünmesini doğrulayın (Adım 5.1.).
- Verileri elektronik tabloya dışa aktarın. 2 dalga boyu için her zaman noktasında floresan birimlerinin çıkışı(Tablo 4).
- Belirli bir zaman noktasında X'in emisyon oranının (527 nm/470 nm) olduğu , neg'in t = 0'daki "UNCUT" substratının emisyon oranı olduğu ve pos'un 24 saat kesmeden sonra ölçülen tamamen "CUT" substratının emisyon oranı olduğu (1) denklemi (1) kullanılarak kesilen substratın nmol'u hesaplayAbilirsiniz (Tablo 3).
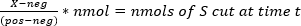
NOT: Tablo 4'te80 μM substrat (kuyu başına 4 nmol) içeren bir kuyu (kuyu E7) için temsili floresan verileri gösterilmiştir. Hesaplamalar plaka her kuyu için yapıldı. - Her kuyu için, nmol vs. zaman (min) çizin ve verileri y = mx + b'ye sığdırarak ilk hızları (eğimleri) elde edin. 4.1.5'te toplanan veriler için, her kuyu için nmol vs. zaman (min) çizin. Eğim, dakikada üretilen nmol ürüne eşit olacaktır. Enzim katalize reaksiyon oranlarından 4.1.5 ile ölçülen hidroliz in spontan oranlarını çıkartın(Tablo 5).
NOT: Plakanın plaka okuyucuya hareketi nedeniyle artifactually yüksekse, ilk okuma verilerden kırpılabilir. - Mg'daki enzim miktarını hesaplayın ve her kuyuya (örn. 0.0009 mg) eklenir. Bir birim dakikada üretilen ürünün bir μmol (μmol/dk) olarak tanımlanır. MU/mg elde etmek için nmol/min'i kuyuda bulunan enzimin mg'ına bölün; U/ mg elde etmek için 1.000 bölün.
- X ekseninde [S] μM ve y ekseninde U/mg'ı çizin ve Vmax ve Kmelde etmek için verileri Michaelis-Menten denklemine sığdırın. Bu yazılım (örneğin, GraFit) yapılabilir.
5. SDS-PAGE Analizini Kullanarak Enzimin Kesilmesi
- Enzim ve etiket yerine 10 μM substrat ve tampon içeren 50 μL'lik bir reaksiyon hazırlayın.
NOT: Alt tabaka ve arabellek hacimleri Tablo 2'degösterilmiştir. Sürekli teşp çalıştırıldıysa, numuneler doğrudan 96 kuyulu plakadan kullanılabilir.- 10 μM substrat ve 5 μL enzim ve etiket içeren 50 μL reaksiyon "CUT" olarak hazırlayın. Enzim substrata eklendiğinde zamanlayıcıyı başlatın.
NOT: Inhibitörler enzim ve substrat içeren ek tüplerek eklenebilir. Eklenen arabellek hacmini, eklenen inhibitör hacmini dengelemek için ayarlayın. DMSO konsantrasyonları %2'yi geçmemelidir. - Oda sıcaklığında ~15-24 saat (22 ± 3 °C) reaksiyonları kuluçkaya yatırın. 2x Laemelli tampon 50 μL ekleyerek reaksiyonları durdurun. Reaksiyon uzerine durduktan sonra kaynatın, her tüp 3-10 dk.
- Jel tankı üreticinin talimatlarına göre monte edin. 17-iyi ön döküm% 12 poliakrilamid jel kaset ve diğer tarafta bir tampon baraj ekleyin. Arabellek kasetin üst bölümüne ulaşana kadar hücrenin iç haznesini 1x SDS çalışan arabellek ile doldurun. Dış hazneyi yarı dolu olarak aynı arabellekle doldurun.
- Kesintisiz testis kullanarak dekolteanaliz etmek için, "UNCUT" reaksiyonu ile başlayan bir SDS-PAGE jelin in bir şeridine her reaksiyon karışımının 5 μL'sini yükleyin. İlk veya son şeride bir molekül ağırlık belirteci ekleyin.
- Jel tankının elektrotlarını güç kaynağına takın ve ürünleri 110 V'de 60 dk. Jeli plastik bir tepsiye yerleştirin ve jeli 5-10 mL jel boyama çözeltisine batırın; bantları 30 dakika içinde görünür olacaktır. 1-24 saat sonra fazla lekekaldırmak, suya jel batırın ve jel bir resim çekmek için bir jel görüntüleyici kullanın.
- 10 μM substrat ve 5 μL enzim ve etiket içeren 50 μL reaksiyon "CUT" olarak hazırlayın. Enzim substrata eklendiğinde zamanlayıcıyı başlatın.
6. Substrat Peptidlerinin VEEV-nsP2 Sistein Proteaz'ına Kenetlenme
- PDB(https://www.rcsb.org/)VEEV sistein proteaz için koordinat dosyasını indirin. PDB kodu 2HWK'dır. Dosyayı 2HWK.pdb olarak kaydedin.
- MOE(https://www.chemcomp.com/)kullanarak protein yapısını hazırlayın. Protein PDB dosyasını MOE'ye yükleyin. Sağ taraftaki çubuktaki Seç ve Çözücü'ye tıklayın ve çözücüyu silin.
- Yapı Hazırlama panelini üst menü çubuğundanaçın Protein . Protonate3D'ye tıklayarak Düzelt'e tıklayarak tüm yapısal öğeleri otomatik olarak düzeltin ve yapıyı protonate'yetıklayın. Kısmi Yükler panelini açarak ve Amber 99'u seçerek proteine kısmi yükler ekleyin ve hidrojen ve Lone çiftlerini gerektiği gibi ayarlayın. Son olarak, yapı dosyasını "2HWK_dock.pdb" olarak kaydedin.
- MOE kullanarak substrat peptidleri (nsP12, nsP23, nsP34) ve TRIM14 için yapı oluşturun. Protein Oluşturucu panelini açın, alt katman sırasını girin, Geometriyi Genişletilmiş olarak ayarlayın ve Build'e tıklayın. Yapı MOE penceresinde gösterilir.
- Panelde Simgedurumuna Küçült'e tıklayarak peptit yapısını en aza indirin. Yapıyı PDB dosyası olarak kaydet (Şekil 6).
- PyRx/AutoDock 4.2(http://autodock.scripps.edu/)kullanarak substrat peptidleri VEEV-nsP2'ye sabitle. PyRx Aracı'nı açın, tercih ayarını düzenlemeyin, Ligand Hazırlığı için tüm torsiyonları inaktive edin. Substrat molekülünü yükleyin, Navigator panelindeki molekül adını sağ tıklatın, ligand yerleştirme dosyasını hazırlamak için ligand yap'ı seçin. Protein 2HWK_clean.pdb'yi yükleyin ve pdbqt yerleştirme dosyasını hazırlamak için makromolekül yap'ı seçin (Şekil 7).
- AutoDock Sihirbazı'nı en alttaki yerleştirme panelinde başlatın. Hazırlanan ligand ve protein dosyalarını seçin. Katalitik kalıntı Kis-477 merkezli ızgara boyutunu manuel olarak ayarlayarak protein bağlama cebini tanımlayın. Varsayılan aralık parametresini kullanarak 0.375 Å. Kılavuz eşlemleri oluşturmak için Otomatik Grid'i Çalıştır'ı tıklatın.
- AutoDock çalıştırın ve Lamarckian Genetik Algoritma (LGA) yöntemiseçin. Yerleştirme Parametrelerini tıklatın ve GA sayısını 50 olarak ayarlayın. Başkaları için varsayılan parametreleri kullanın. Yerleştirme çalışmasını başlatmak için İleri'yi tıklatın.
- Sonuçları Analiz et panelini açın. Öngörülen tüm bağlayıcı pozları inceleyin. Cys-477 ve dekolte yerindeki substrat arasında en düşük öngörülen bağlama enerjisi ve makul bağlama etkileşimleri ile en iyi modeli seçin. Bağlama modelini daha fazla MD simülasyonu için PDB dosyası olarak kaydedin.
7. MD Kenetlenmiş VEEV-substrat Komplekslerinin Simülasyonları
- Amber(http://ambermd.org/)kullanarak giriş dosyalarını hazırlayın. Standart protokolü takiben AMBER paketi ve ff99SB kuvvet alanı kullanılarak öngörülen substrat bağlama modelleri için MD simülasyonları gerçekleştirilir.
NOT: Çözünen sistemler, MD simülasyonları öncesinde kapsamlı bir enerji en aza izasyonuna tabi tutulur. Sürekli bir sistemin simüle sisini için periyodik sınır koşulları uygulanır. Parçacık örgü Ewald (PME) yöntemi uzun menzilli elektrostatik etkileşimleri hesaplamak için kullanılmıştır. Simüle sistem ilk olarak 0 K'den 300 K'ye 100 ps'nin üzerinde kademeli bir sıcaklık artışına maruz kaldı ve ardından 300 K'da 500 ps'ye dengelendi ve ardından toplam 2 ns uzunluğunda üretim devam etti.- Simülasyon işini yüksek performanslı bir bilgi işlem tesisinde çalıştırın. Simülasyonlarımız Biowulf kümesinde(https://hpc.nih.gov/)(Şekil 8)üzerinde çalıştırıldı.
- VMD programı(http://www.ks.uiuc.edu/Research/vmd/)kullanarak yörünge çıktısını görselleştirin. NsP2 aktif alanı içindeki substratların ve TRIM14'ün bağlayıcı etkileşimlerini ve konformasyonel değişikliklerini analiz edin (Şekil 9).
Sonuçlar
ZikV ns2B/3 proteazS analizi 4 konak protein hedefleri tespit: FOXG1, SFRP1, retinal cDNA kütüphanesinden bir Gs alfa alt birimi, ve NT5M mitokondriyal 5',3'-nükleotidaz (Şekil 10)6. Özellikle, başka hiçbir yöntem zikv proteaz potansiyel hedefleri olarak bu proteinlertahmin. FOXG1 genindeki mutasyonlar, mikrosefali gibi gelişme bozukluğu ve yapısal beyin anormallikleri ile karakterize konjenital sendromla bağlantılıdır. SFRP1 salgılanan kıvırcık ilişkili bir proteindir (SFRP); bu çözünür reseptörler, Wnt sinyalini antagonize etmek ve inhibe etmek için Wnt ligandlarını rekabetçi bir şekilde bağlayabilir. Wnt sinyal yolu Flavivirus enfeksiyonu sırasında IFN yanıtının düzenlenmesinde yer almaktadır36. SFRP1 dekoltesi flavivirüs replikasyongeliştirmek için beklenebilir. SFRP1 ayrıca Th17 hücre farklılaşması nda da rol oynar37. SSHHPS dizi hizalamaları dekolte alanı dizilerinde türe özgü farklılıklar göstermiştir(Şekil 10D). SFRP1'deki dekolte yeri dizisi insanlarda ve tavuklarda aynıydı; ZIKV tavuk embriyolarında mortalite ve mikrosefali neden olabilir38. Kemirgenlerde, yüksek oranda korunmuş P1 kalıntısı (K/R)R(R)G bir glisin (RGG) ile ikame edilir. Farelerin immünyonsi genellikle ZIKV enfeksiyonuna ve hastalığa karşı dirençlidir39.
Sabit durum kinetik parametreleri ve inhibisyon sabitleri viral poliprotein dizileri ve bir plaka okuyucu31,40,41 (Şekil 11A)sürekli tsömede kullanılarak konak protein dizileri için ölçülebilir. Belirli bir dizinin bölünmesi veya proteazın çeşitli bileşikler tarafından inhibisyonu gibi nitel bölünme bilgileri için, sürekli olmayan test kullanılabilir(Şekil 11B).
CFP ve YFP arasındaki kalıntı sayısının optimizasyonu gerekebilir. Substrata bağlı bir model in silico yöntemleri kullanılarak yapılabilir. Şekil 9'dansP1/nsP2 bağlantısının temsili bir docked modeli gösterilmiştir. VEEV nsP2 proteazı için, 12-amino asit Semliki Orman Virüsü (SFV) dizisinin dekoltesi bildirilmiştir (Km = 0.58 mM)33. Substrat dizisini 19, 22 ve 25'e kadar uzatmak ve tamponun iyonik mukavemetini azaltmak Km'deönemli bir azalmaya yol açmıştır. VEEV nsP2 kristal yapısı ve kristal ambalajı incelenmesi de kavşaklardan birinin bir kısmının proteaz etki alanına karşı paketlenmiş ve sarmal olduğunu göstermiştir. Böylece, ikincil bir yapısal motifin tanınması nedeniyle DAHA uzun VEEV yüzeyleri daha iyi bağlanabilir.
TRIM14 için Km = 21 μM6,33elde ettik. Konak protein dizisini taşıyan substrat için Km, viral poliprotein bölünme site dizilerini (Km(V12) = 12 μM ve Km(V34) = 21 μM içeren substratların Km değerleri ile karşılaştırılabilirdi. NsP1/nsP2, nsP2/nsP3 ve nsP3/nsP4 kavşaklarında dekolte yeri dizileri farklı verimliliklerle kesildi. Hücrede, bu poliprotein in sıralı dekolte için izin düşünülmektedir42.
Olumsuz sonuçların yorumlanmasında dikkatli olunmalıdır. Bölünme meydana gelmezse, dekolte bölgesi çok kısa olabilir veya saflaştırılmış proteaz etkin olmayabilir. Kesilen substratlar için, virüs bulaşmış hücrelerde tam uzunlukta protein veya dekolte bölünmesi onaylamak için ek deneyler gereklidir. Uygun takip deneyleri seçilmelidir. Hedef proteinin aşırı ekspresyonu veya susturulması nın viral replikasyon üzerindeki etkileri de test edilebilir.
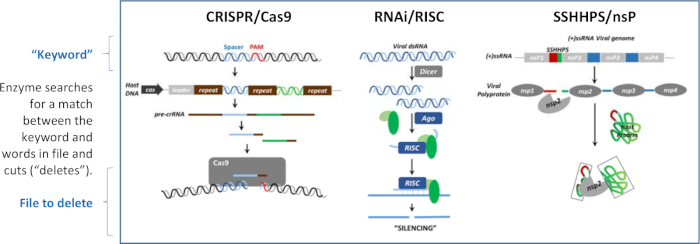
Şekil 1: Susturma üç mekanizma. Susturma DNA, RNA veya protein düzeyinde oluşabilir. Bu "arama ve silme" algoritmalarının her biri, sözcüğü içeren bir dosyanın bölünmesini yönlendirmek için bir "anahtar kelime" kullanır. Bu rakam Morazzani ve ark.32 ve buradaki kaynaklardan değiştirilmiştir. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.

Şekil 2: Bölünme alanı dizilerinde türe özgü farklılıklar. TRIM14 homologlarının C-terminal PRY/SPRY etki alanları hizada gösterilir. PRY/SPRY etki alanı, gri renkle vurgulanan korunmuş motiflerle tanımlanabilir. İnsan TRIM14 QEAGA-G'de VEEV nsP2 sistein proteazı ile kesilir. SSHHP dizisi renkli olarak gösterilir. Yeşilkin kalıntısı P1' kalıntısıdır; mavi P4 kalıntısı, ve kırmızı dekolte site motif dizisi içinde diğer korunmuş kalıntılar vardır. AT, PRY/SPRY etki alanından yoksun TRIM14'ün kesilmiş bir sürümünü barındırıyor. Siyan'da vurgulanan lizin poli-ubiquitinated ve MAVS signalozom montajı için önemlidir. C-terminal PRY/SPRY etki alanı, akut viral enfeksiyon sırasında hücre içi olarak konakçının antiviral yanıtını bozacak şekilde nsP2 proteazı tarafından geçici olarak kesilebilir. At, bu etki alanı her zaman yok. Bu, TRIM14'ün PRY/SPRY etki alanının VEEV enfeksiyonlarına karşı koruyucu bir fonksiyona sahip olabileceğini düşündürmektedir. Bu rakam Morazanni ve ark.6'dançoğaltılmıştır.

Şekil 3: BLAST kullanılarak SSHHPS tanımlaması. VEEV nsP1/nsP2 kavşağındaki bölünme yeri motif ilü, ev sahibi protein TRIM14'teki SSHHP dizisi ile uyumludur. Yeşil renkli kalıntı P1' kalıntısıdır; mavi P4 kalıntısı ve kırmızı dekolte site motif dizisinin diğer korunmuş kalıntılar vardır. Çoğu hizalama korunmuş dekolte alanı motifi dışındaki bölgelere homoloji içeriyordu veya P1/P1'in makas bağı kalıntılarını içermiş. TRIM14, P1 ve P1'i içeren sırayla 6 kalıntıya bir eşleşme gösterdi. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.

Şekil 4: VEEV nsP2 sistein proteazı için CFP-V12-YFP substratının protein ve DNA dizileri. NdeI (CATATG) ve XhoI (CTCGAG) kısıtlama siteleri büyük harflerle gösterilir. Kırmızı nsP1 ve nsP2 arasında olan viral poliproteinden dekolte site dizisidir. Yeşilkin kalıntısı P1' kalıntısı, mavi renkte ise dekolte bölgesinin P4 kalıntısıdır. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.

Şekil 5: Trx-VEEV-nsP2 sistein proteaz yapısının protein dizisi. Thioredoxin (Trx) sarı olarak gösterilir. Trombin dekolte sit alanı ve His-tag siyan içinde gösterilir. Cys-His dyad kırmızı etiketli. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
Şekil 6: MOE'deki peptit yapıları. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
Şekil 7: PyRx/AutoDock kullanılarak substrat peptidin yanaşma. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
Şekil 8: Biowulf kümesinde çalışan işler. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
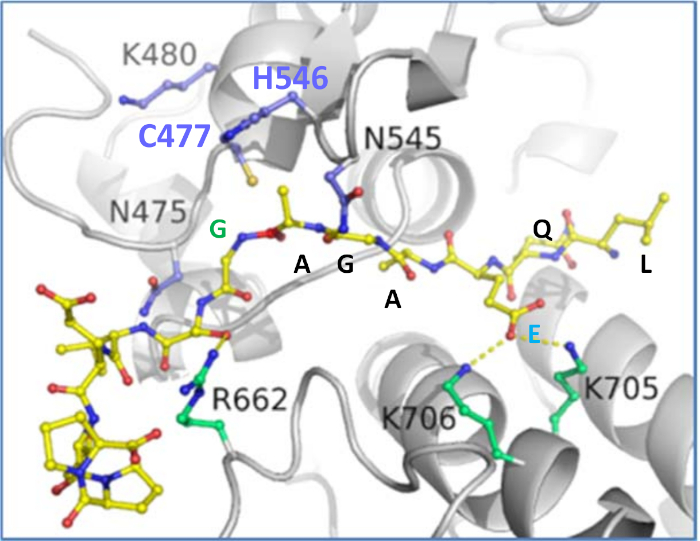
Şekil 9: NsP1/nsP2 kavşağında dekolte yeri dizisini içeren VEEV P12 substrat modeli. Cys-477/His-546 katalitik dyad mavi gösterilir. Şekil Pymol (https://pymol.org) kullanılarak yapılmıştır. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
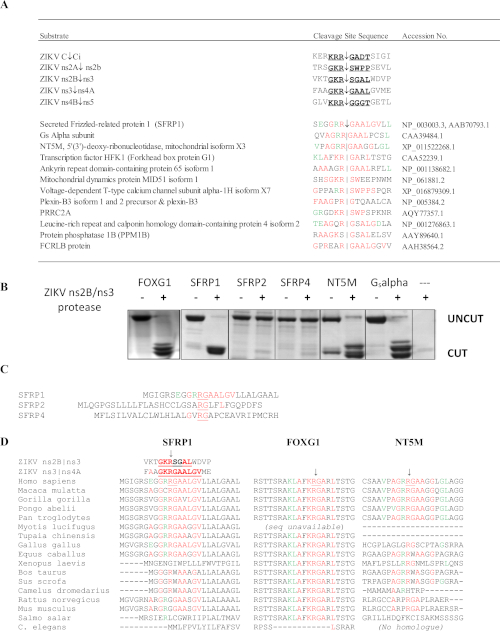
Şekil 10: SSHHPS Zika virüsü ns2B/ns3 proteaz analizi. (A) ZIKV ns2B/ns3 proteazının öngörülen konak protein hedefleri. Kırmızı daki kalıntılar tek bir dekolte alanı dizisiyle eşleşir. Yeşil kalıntılar alt bölgede tolere edilir ve diğer dekolte bölgelerindeki kalıntılarla eşleşir. SFRP1 ardışık sırayla en yüksek özdeş kalıntı sayısına sahipti. (B) CFP-substrat-YFP proteinleri (~50-60 kDa) her konak proteinden (insan) öngörülen SSHHP dizisini içeren ifade edildi ve saflaştırılmıştır. ZIKV proteaz insan FOXG1, SFRP1, NT5M ve bir Retina cDNA kütüphaneden izole bir Gsalfa alt birimi kesti. Dekolte ürünleri yaklaşık 28-30 kDa'dır. Substrat dizileri Morazzani et al.6 (C) ns2B/ns3 proteaz SFRP1 keserken, homologlarını kesmedi (SFRP2 ve SFRP4). (D) Farklı hayvan türlerinin dekolte alanlarının hizalanması, Grup IV virüsü için bir hayvan modeli nin seçilmesinde yararlı olabilir. Korunmuş RG dizisinin SFRP1'deki insanlar ve kemirgenler arasında farklılık olduğunu unutmayın. Morazzani ve ark.6'dançoğaltılanşekil, bu rakamın daha büyük bir versiyonunu görmek için lütfen buraya tıklayınız.
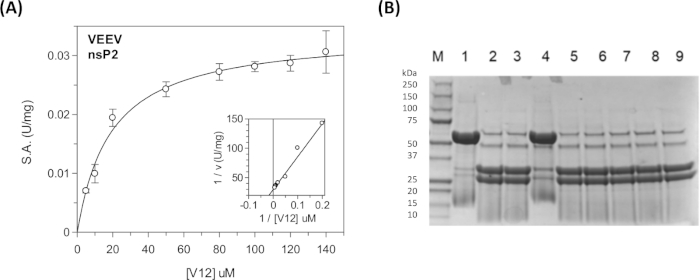
Şekil 11: Sürekli ve sürekli tahliller kullanılarak kararlı durum kinetik analizi. (A) Tablo 5'te gösterilen kinetik veriler GraFit'te çizilmiştir. Inset Lineweaver-Burk arsa gösterir. (B) CFP-V12-YFP substratının dekolte ürünlerini gösteren SDS-PAGE jeli. Şerit 1'de "UNCUT" substratı (48 kDa) bulunur. Şerit 2 "CUT" substrat (31 kDa ve 27 kDa) olduğunu. Şeritlerde 3-9 farklı bileşikler onların inhibitör aktivitesini test etmek için dahil edildi. Lane 4, E64d kovalent inhibitörü içerir. Bu reaksiyonlar oda sıcaklığında ~ 17 saat için bir gecede çalıştırıldı. Keskin bantlama desenine ulaşmak için örneklerin kaynatması gerekiyordu. NSP2 proteazenzim içeren reaksiyonlarda görünür (56 kDa), ancak şerit 1 değil. Lane 1 "enzim yok" kontrolüdür. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
Tartışmalar
Bir proteinin veya yabancı bir dizinin yönlendirilen nükleik asitin diziye özgü yıkımı biyolojide sadece birkaç vakada görülür. Şekil 1'de gösterilen mekanizmalar, bir ana bilgisayarı virüsten veya bir virüsten koruyan savunma mekanizmalarıdır.
Biyoinformatik yöntemleri kullanarak bu sistemler tarafından yok edilen hedefleri belirleyebiliriz. SSHHP dizileri üzerinde yaptığımız analizlerde, bunların çoğunun doğuştan gelen bağışıklık yanıtları üretmek için gerekli proteinlerde bulunabileceğini keşfettik. Bazı MAVS ve TRIF (TIR-etki alanı içeren adaptör-interferon-β indükleyici) gibi bariz rolleri vardı, diğerleri daha karmaşık mekanizmalar olsa bağışıklık ile ilgili iken (örneğin, Histone H3, SFRP1, FOXG1)8,9. SSHHP dizisinde depolanan hedef bilgiler, bu virüslere karşı antiviral etkiye sahip yolları belirleme potansiyeline sahiptir. Vivo antiviral yanıtlar genellikle virüse özgü26,43. Örneğin, TRIM proteinlerinin alt kümelerinin farklı virüsler üzerinde antiviral etkileri vardır43,44,45, bazıları viral kısıtlama faktörleridir (örneğin, HIV ve TRIM5α). TRIM proteinlerinin özgüllüğü (~70 tespit edilmiştir) halen44,45incelenmektedir. SSHHPS içindeki bilgiler, bu virüslerin doğuştan gelen bağışıklık yanıtlarından nasıl kaçtığını anlamamıza katkıda bulunabilir. Daha fazla SSHHPS incelendiğinde diğer desenler ve korelasyonlar ortaya çıkarılabilir.
Analizlerimizde türe özgü farklılıklar belirgindi(Şekil 2, Şekil 10). Bu virüslerin bazı türleri diğerlerinden daha fazla etkilediği bilinmektedir. SSHHPS'de ana bilgisayar aralığı, ana bilgisayar duyarlılığı ve ev sahibi savunması hakkında bilgiler bulunabilir. Örneğin, at ensefalit iliklerine en yatkın türler olan at, VEEV nsP2 proteazı tarafından geçici olarak kesilen insan TRIM14 bölgesinden yoksundu. İnsanlar nadiren VEEV enfeksiyonlarından ölürler ancak24enfeksiyon kapılabilir. İnsan TRIM14 protein bir nsP2 proteaz dekolte dizisi6taşıdı. Dekolte alanının varlığı insanların bu virüslere karşı bir savunma mekanizması olduğunu göstermektedir. Kuşlar bu virüslerin potansiyel rezervuarları olduğu düşünülmektedir46. TRIM14 proteinindeki karşılık gelen SSHHP dizisi tavuklardan insanlarda ve diğer türlerde bulunan dizilerden farklıydı. Bu gibi ince farklılıklar bir hedef konak protein amcaavable veya daha kolay cleaved yapabilir. Aguirre ve ark.16 bir amcaavable mutasyona uğramış STRING protein Dang virüs enfeksiyonu sonra IFN yüksek düzeyde indüklenen ve fareler doğal Olarak Dang ns2B3 proteaz tarafından kesilmez STING bir sürümünü taşımak gösterdi. Murine STING proteini ZIKV proteaz47tarafından kesilmedi. SSHHPS analizimizde, zikv proteaz bölünme alanı dizilerinde insan proteinlerini kemirgen6(Şekil 10 D)ile karşılaştırdığımızda farklılıklar gözlemledik. Grup IV virüsleriçin kullanılan hayvan modellerinde konak proteinlerin türlerine özgü proteolitik dekoltelerinin çoğaltılması önemli olabilir. Konak protein bölünmesiinin inhibisyonu da Grup IV proteaz inhibitörlerinin gelişimi ile ilgili etkileri vardır. Bir önceki yayınımızda, CA074 metil ester6kullanarak VEEV nsP2 proteazı ile TRIM14 dekoltesini inhibe edebileceğimizi gösterdik. Bu sonuç, bu proteazların küçük molekül inhibitörleri enfeksiyonu bastırma yeteneğine sahip doğuştan gelen bağışıklık yanıtları modüle etmek mümkün olabileceğini düşündürmektedir6,31.
Bir tür içindeki genetik varyasyon da proteolitik bölünme farklılıkları üretmek için potansiyele sahiptir. Kodon kullanımındaki ince farklılıklar ribozom duraklamaetkileyebilir 48. Bazı Grup IV viral proteazlar ER membranına gömülü olduğundan, bölünme eş-çeviri selami oluşursa bu duraklamalarda farklılıklar hedefin bölünmesini etkileyebilir. Tespit ettiğimiz dekolte bölgelerinin bazıları tahmin edilen sinyal peptid dizilerinde (örn. SFRP1) iken diğerleri dahiliidi.
SSHHPS analizi, diğer konak protein analiz yöntemlerinden farklı bilgiler üretebilir. SSHHPS analizi ucuz ve kullanımı kolaydı. Bakteriyel ekspresyon sisteminin kullanılması, memeli hücre kültürünü kullanmadan memeli dizilerinin kısa segmentlerinin (~25 amino asitleri) test edilmesine olanak sağladı. CFP-YFP substratlarının test edilen tüm insan protein dizilerine tahammül edebildiğini bulduk; ancak, verimleri çeşitlidir. Benzer tahlillerde, 63 amino asit kadar insan protein dizileri içeren substratlar başarıyla ifade edildi, saflaştırılmış, ve kinetik analizler ve inhibitör tarama için kullanılan49,50,51. Kesintili titrek için sadece küçük miktarlarda substrat gerektiğinden, çok sayıda hedef araştırılabilir. Sistemin bir avantajı CFP /YFP yüzeyleri SDS-PAGE analizleri ve daha ayrıntılı kinetik analizler için kullanılabilir (yani, IC50, Ki, Km, Vmax). İlaç keşfi için, inhibitör bileşikler floresan tahliller eserler üretebilir. Böylece, sürekli bir test ile birlikte sürekli test bir dekolte veya dekolte inhibisyonu onaylamak için izin verir. Kesintili SDS-PAGE teşrisi örnekleri doğrudan 96 kuyulu plakalardan alınabilir. CFP/YFP yüzeyleri bileşik kütüphane taraması için kullanılmıştır52. Ancak, bir alt tabakazın Z-faktör53'ünhesaplanması gibi yüksek iş elde taraması için uygun olup olmadığını belirlemek için ek analizler gereklidir.
Bir substrat tasarımında bir sorun bağlı ve proteaz tarafından tanınan makas bağ etrafında bölge tespit etmektir. Burada gösterilen örneklerde, makas bağı etrafında ortalanmış 12 kalıntı dizisi ile başladık. Veev proteazı için dekolte bölgelerinin homolojisi kalıntılarına homolojinin dizi hizalamaları analiz edildikten sonra, ZIKV proteaz homolojisinde ise C-terminal kalıntılarının bir çoğuna rastlandı. Kenetlenmiş substrat bir silico modeli substrat bağlayıcı siteleri araştırmak site yönettiği mutagenez deneyleri tasarlamak için kullanılabilir. Substrat ve enzim dizileri plazmidler üzerinde olduğundan, silico modellerinde veya alt sektör toleranslarında test etmek için mutasyona uğrayabilir. Bağlı substrat(lar)ın kristal yapısı mevcut değilse bu avantajlı olabilir.
SSHHPS analizi, virüse bağlı fenotiplerin viral enzimler tarafından üretilme mekanizmaları hakkında da yeni bilgiler verebilir. ZikV hedeflerinden biri, SFRP1, Wnt sinyal yolunun bir parçasıdır ve beyin ve göz gelişiminde ve bağışıklık yanıtlarında rolleri vardır36,37,54,55,56,57. ZIKV ns2B/ns3 proteazı ile kesilebilen diğer protein dizilerinin beyin ve göz gelişiminde yer alan proteinlerde de bulunduğunu bulduk; her ikisinde de konjenital Zika sendromunda anormallikler gözlenmiştir ve virüse bağlı fenotip58'inbir parçası olduğu düşünülmektedir.
Konak-patojen etkileşimlerinin öngörülebilirliği çeşitli uygulamalar için kullanılabilir: hedefe özgü onkolitik viral tedaviler; canlı virüs aşılarının risksiz; hayvan modellerinin iyileştirilmesi, tahmin edilmesi veya seçilmesi; konak aralığı veya duyarlılık tahmini; zoonotik olayların tahmini; ve ev sahibi savunma tahmini. Açıklanan yöntemler sıra tabanlı olduğundan, gelecekte yazılıma dahil edilmeleri açısından değerli olabilir.
Açıklamalar
Burada ifade edilen görüşler yazarların görüşleridir ve ABD Donanması, ABD Ordusu, ABD Savunma Bakanlığı veya ABD hükümetinin görüşlerini temsil etmemaktadır.
Teşekkürler
Bu çalışma, Savunma Tehdit Azaltma Ajansı (DTRA) proje numaraları CB-SEED-SEED09-2-0061 ve CBCall4-CBM-05-2-0019 ve kısmen NCATS, NIH (XH) ve Deniz Araştırma Laboratuvarı baz fonlarının Intramural/Extramural araştırma programı tarafından desteklenmiştir.
Malzemeler
| Name | Company | Catalog Number | Comments |
| 250 mL Erlenmeyer Flask | VWR | 89000-362 | |
| 2-mercaptoethanol | Acros Organics (Fisher) | 125472500 | Danger: Acutely Toxic. Open bottle in hood to avoid inhaling the fumes. |
| 4 L Pyrex wide-mouth graduated Erlenmeyer flask with screw-cap | Millipore Sigma | CLS49954L-1EA | |
| AKTA Prime Plus | GE Healthcare | 17-0729-01 | |
| AKTA XK 16/20 Column | GE Healthcare | 28988937 | |
| Amicon Ultra-0.5 Centrifugal Filter Unit | Millipore Sigma | UFC501096 | |
| Amicon Ultra-15 Centrifugal Filter Unit | Millipore Sigma | UFC901096 | |
| Amicon Ultra-4 Centrifugal Filter Unit | Millipore Sigma | UFC801024 | |
| Ampicillin | Sigma | A0166 | Danger: Allergic reactions (skin or breathing). |
| Chelating Sepharose Fast Flow | GE Healthcare | 17-0575-02 | Once the resin is equilibrated with 0.2 M Nickel Sulfate it is refered to as a Nickel Column in the text. Column will have a green color after washing with water. The column will have a blue color after equilibrating with buffer. |
| Chloramphenicol | RPI | C61000 | Danger: May cause cancer. |
| Corning 50 mL centrifuge tubes | Corning | 430828 | Suggestion: Polypropylene tubes are less likely to crack during sonication than Polyethylene tubes |
| Corning 96 Well Half-Area Microplate, Non-Binding Surface | Corning | 3993 | |
| Dialysis Tubing Clips | Fisher Scientific | PI68011 | |
| Disposable PD-10 Desalting Column | GE Healthcare | 17-0851-01 | |
| DNAse | Sigma | DN25-1G | |
| DTT (DL-Dithiothreitol) | RPI | D11000-50.0 | Warning: Acute Oral Toxicity; skin and eye irritation |
| EDTA | Fisher Scientific | S311-500 | |
| Fisherbrand Petri Dishes with Clear Lid | Fisher Scientific | FB0875712 | |
| Glycerol | Acros Organics (Fisher) | 15892-0010 | |
| HEPES | Millipore Sigma | H4034-1KG | |
| Imidazole | Acros Organics | 301870010 | Danger: Toxic, Irritant |
| IPTG (Isopropyl β-D-thiogalactopyranoside) | Calbiochem (Millipore Sigma) | 420291 | Do not breathe dust. Avoid contact with eyes and skin. |
| Laemmli Sample Buffer | BIO-RAD | 1610737 | |
| Luria Bertani Agar | Fluka (Millipore Sigma) | L3027-1KG | Suggestion: Autoclave with magnetic stirrer in the liquid, and stir while cooling. Wait to add antibiotic until you can hold your hands on the bottle without pain for 30 seconds. |
| Luria Bertani Media | Fisher Bioreagents | BP1426-2 | |
| Lysozyme | Sigma | L4919-5g | |
| Mini-PROTEAN Tetra Vertical Electrophoresis CellGel Box | BIO-RAD | ||
| Nalgene Oak Ridge High-Speed PPCO Centrifuge Tubes | Nalgene (Thermo Scientific) | 3119-0050 | |
| Nanodrop | Thermo Fisher | ||
| New Brunswick Innova 42R Shaker Incubator | Eppendorf | M1335 | |
| Nickel Sulfate Hexahydrate (Crystalline/Certified ACS), Fisher Chemical | Fisher Scientific | N73-500 | Danger: Harmful if swallowed or inhaled, skin and eye irritation |
| One Shot BL21(DE3) Chemically Competent E. coli | Invitrogen (Thermo Fisher) | C600003 | May be harmful if inhaled or swallowed. May cause skin and eye irritation with susceptible people. |
| One Shot BL21(DE3) pLysS Chemically Competent E. coli | Invitrogen (Thermo Fisher) | C606003 | May be harmful if inhaled or swallowed. May cause skin and eye irritation with susceptible people. |
| pet15b plasmid DNA | Novagen (Millipore Sigma) | 69661 | GenScript Inc. was used for commerical DNA synthesis. The pet15b plasmid was used for the CFP/YFP substrates. |
| pet32b | Novagen (Millipore Sigma) | 69016-3 | The pet32b plasmid was used for the cysteine protease construct. |
| Pierce Protease Inhibitor Mini Tablets, EDTA-free | Thermo Fisher | A32955 | Warning: Skin corrosion/irriation; eye damage |
| Plate Reader | Molecular Devices | Model M5 | |
| Precision Plus Protein All Blue Prestained Protein Standard | BIO-RAD | 161-0373 | |
| Protein Extraction Reagent | Novagen (Millipore Sigma) | 70584-4 | BugBuster or Bper (Catalog # 78248, ThermoFisher) |
| Q-Sepharose Fast Flow | G.E. Healthcare | 17-0510-01 | Anion exchange resin |
| RunBlue (12%) 17-well PAGE gels | Expedeon | BCG01227 | Any 12% pre-cast polyacrylamide gel can be used |
| RunBlue 20x SDS Running Buffer | Expedeon | NXB50500 | Dilute 50 mL with 950 mL deionized water to obtain 1x |
| RunBlue Instant Blue Gel Stain | Expedeon | ISB1L | Do not dilute, use as directed |
| Sodium Chloride | Fisher Chemical | S271-10 | |
| Sonifier Cell Disrupter 450 Sonicator | Branson Ultrasonics (VWR) | Model No. 101-063-346R | Sonicator was used on level 5 |
| Spectra/Por 6-8 kD MWCO | Spectrum Labs | 132645T | Dialysis Tubing |
| SP-Sepharose Fast Flow | G.E. Healthcare | 17-0729-01 | Cation exchange resin |
| Thrombin from bovine plasma | Sigma | T6634-500UN | |
| Tris Base | Fisher Scientific | BP152-500 | Caution: Eye/Skin Irritant |
Referanslar
- Liu, H., et al. Widespread Horizontal Gene Transfer from Double-Stranded RNA Viruses to Eukaryotic Nuclear Genomes. Journal of Virology. 84 (22), 11876-11887 (2010).
- Hagai, T., Azia, A., Babu, M. M., Andino, R. Use of host-like peptide motifs in viral proteins is a prevalent strategy in host-virus interactions. Cell Reports. 7 (5), 1729-1739 (2014).
- Gorbalenya, A. E. Host-related sequences in RNA viral genomes. Seminars in Virology. 3, 359-371 (1992).
- Shmakov, S. A., et al. The CRISPR Spacer Space Is Dominated by Sequences from Species-Specific Mobilomes. MBio. 8 (5), 1-18 (2017).
- Legler, P. M., Morazzani, E., Glass, P. J., Compton, J. R. Proteome Editing System and A Biomarker of Veev Infection. United States patent application. , (2018).
- Morazzani, E. M., et al. Proteolytic cleavage of host proteins by the Group IV viral proteases of Venezuelan equine encephalitis virus and Zika virus. Antiviral Research. 164, 106-122 (2019).
- Alvarez, E., Castello, A., Menendez-Arias, L., Carrasco, L. HIV protease cleaves poly(A)-binding protein. Biochemical Journal. 396 (2), 219-226 (2006).
- Falk, M. M., et al. Foot-and-mouth disease virus protease 3C induces specific proteolytic cleavage of host cell histone H3. Journal of Virology. 64 (2), 748-756 (1990).
- Grigera, P. R., Tisminetzky, S. G. Histone H3 modification in BHK cells infected with foot-and-mouth disease virus. Virology. 136 (1), 10-19 (1984).
- Li, W., Ross-Smith, N., Proud, C. G., Belsham, G. J. Cleavage of translation initiation factor 4AI (eIF4AI) but not eIF4AII by foot-and-mouth disease virus 3C protease: identification of the eIF4AI cleavage site. FEBS Letters. 507 (1), 1-5 (2001).
- Kuyumcu-Martinez, M., et al. Calicivirus 3C-like proteinase inhibits cellular translation by cleavage of poly(A)-binding protein. Journal of Virology. 78 (15), 8172-8182 (2004).
- Pietila, M. K., Hellstrom, K., Ahola, T. Alphavirus polymerase and RNA replication. Virus Research. 234, 44-57 (2017).
- Hardy, W. R., Strauss, J. H. Processing the nonstructural polyproteins of sindbis virus: nonstructural proteinase is in the C-terminal half of nsP2 and functions both in cis and in trans. Journal of Virology. 63 (11), 4653-4664 (1989).
- Strauss, E. G., De Groot, R. J., Levinson, R., Strauss, J. H. Identification of the active site residues in the nsP2 proteinase of Sindbis virus. Virology. 191 (2), 932-940 (1992).
- Wang, D., et al. Foot-and-mouth disease virus 3C protease cleaves NEMO to impair innate immune signaling. Journal of Virology. 86 (17), 9311-9322 (2012).
- Aguirre, S., et al. DENV inhibits type I IFN production in infected cells by cleaving human STING. PLoS Pathogens. 8 (10), e1002934 (2012).
- Barral, P. M., Sarkar, D., Fisher, P. B., Racaniello, V. R. RIG-I is cleaved during picornavirus infection. Virology. 391 (2), 171-176 (2009).
- Elbashir, S. M., Lendeckel, W., Tuschl, T. RNA interference is mediated by 21- and 22-nucleotide RNAs. Genes & Development. 15 (2), 188-200 (2001).
- Deveau, H., Garneau, J. E., Moineau, S. CRISPR/Cas system and its role in phage-bacteria interactions. Annual Review of Microbiology. 64, 475-493 (2010).
- Schechter, I., Berger, A. On the size of the active site in proteases I. Papain. Biochemical and Biophysical Research Communications. 27 (2), 157-162 (1967).
- Bieniasz, P. D. Intrinsic immunity: a front-line defense against viral attack. Nature Immunology. 5 (11), 1109-1115 (2004).
- Zhou, Z., et al. TRIM14 is a mitochondrial adaptor that facilitates retinoic acid-inducible gene-I-like receptor-mediated innate immune response. Proceedings of the National Academy of Sciences of the U S A. 111 (2), E245-E254 (2014).
- Wang, S., et al. TRIM14 inhibits hepatitis C virus infection by SPRY domain-dependent targeted degradation of the viral NS5A protein. Scientific Reports. 6, 32336 (2016).
- Zacks, M. A., Paessler, S. Encephalitic alphaviruses. Veterinary Microbiology. 140 (3-4), 281-286 (2010).
- Hollidge, B. S., Weiss, S. R., Soldan, S. S. The role of interferon antagonist, non-structural proteins in the pathogenesis and emergence of arboviruses. Viruses. 3 (6), 629-658 (2011).
- Carthagena, L., et al. Human TRIM gene expression in response to interferons. PLoS One. 4 (3), e4894 (2009).
- Montgomery, S. A., Johnston, R. E. Nuclear import and export of Venezuelan equine encephalitis virus nonstructural protein 2. Journal of Virology. 81 (19), 10268-10279 (2007).
- Nenasheva, V. V., et al. Enhanced expression of trim14 gene suppressed Sindbis virus reproduction and modulated the transcription of a large number of genes of innate immunity. Immunologic Research. 62 (3), 255-262 (2015).
- Nagai, T., et al. A variant of yellow fluorescent protein with fast and efficient maturation for cell-biological applications. Nature Biotechnology. 20 (1), 87-90 (2002).
- Li, M. Z., Elledge, S. J. SLIC: a method for sequence- and ligation-independent cloning. Methods in Molecular Biology. 852, 51-59 (2012).
- Hu, X., et al. Kinetic, Mutational, and Structural Studies of the Venezuelan Equine Encephalitis Virus Nonstructural Protein 2 Cysteine Protease. Biochemistry. 55 (21), 3007-3019 (2016).
- Morazzani, E. M., et al. Proteolytic cleavage of host proteins by the Group IV viral proteases of Venezuelan equine encephalitis virus and Zika virus. Antiviral Research. 164, 106-122 (2019).
- Zhang, D., Tozser, J., Waugh, D. S. Molecular cloning, overproduction, purification and biochemical characterization of the p39 nsp2 protease domains encoded by three alphaviruses. Protein Expression and Purification. 64 (1), 89-97 (2009).
- Lei, J., et al. Crystal structure of Zika virus NS2B-NS3 protease in complex with a boronate inhibitor. Science. 353 (6298), 503-505 (2016).
- Shiryaev, S. A., et al. Characterization of the Zika virus two-component NS2B-NS3 protease and structure-assisted identification of allosteric small-molecule antagonists. Antiviral Research. 143, 218-229 (2017).
- Smith, J. L., Jeng, S., McWeeney, S. K., Hirsch, A. J. A MicroRNA Screen Identifies the Wnt Signaling Pathway as a Regulator of the Interferon Response during Flavivirus Infection. Journal of Virology. 91 (8), (2017).
- Lee, Y. S., et al. The Wnt inhibitor secreted Frizzled-Related Protein 1 (sFRP1) promotes human Th17 differentiation. European Journal of Immunology. 42 (10), 2564-2573 (2012).
- Goodfellow, F. T., et al. Zika Virus Induced Mortality and Microcephaly in Chicken Embryos. Stem Cells and Development. 25 (22), 1691-1697 (2016).
- Morrison, T. E., Diamond, M. S. Animal Models of Zika Virus Infection, Pathogenesis, and Immunity. Journal of Virology. 91 (8), (2017).
- Morazzani, E. M., et al. . Books of Abstracts, 254th American Chemical Society National Meeting. , (2017).
- Compton, J. R., Mickey, M. J., Hu, X., Marugan, J. J., Legler, P. M. Mutation of Asn-475 in the Venezuelan Equine Encephalitis Virus nsP2 Cysteine Protease Leads to a Self-Inhibited State. Biochemistry. 56 (47), 6221-6230 (2017).
- Vasiljeva, L., et al. Regulation of the sequential processing of Semliki Forest virus replicase polyprotein. Journal of Biological Chemistry. 278 (43), 41636-41645 (2003).
- Uchil, P. D., Quinlan, B. D., Chan, W. T., Luna, J. M., Mothes, W. TRIM E3 ligases interfere with early and late stages of the retroviral life cycle. PLoS Pathogens. 4 (2), e16 (2008).
- Ozato, K., Shin, D. M., Chang, T. H., Morse, H. C. TRIM family proteins and their emerging roles in innate immunity. Nature Reviews Immunology. 8 (11), 849-860 (2008).
- van Tol, S., Hage, A., Giraldo, M. I., Bharaj, P., Rajsbaum, R. The TRIMendous Role of TRIMs in Virus-Host Interactions. Vaccines (Basel). 5 (3), (2017).
- Molaei, G., et al. Dynamics of Vector-Host Interactions in Avian Communities in Four Eastern Equine Encephalitis Virus Foci in the Northeastern U.S. PLoS Neglected Tropical Diseases. 10 (1), e0004347 (2016).
- Ding, Q., et al. Species-specific disruption of STING-dependent antiviral cellular defenses by the Zika virus NS2B3 protease. Proceedings of the National Academy of Sciences of the U S A. 115 (27), E6310-E6318 (2018).
- Angov, E., Legler, P. M., Mease, R. M. Adjustment of codon usage frequencies by codon harmonization improves protein expression and folding. Methods in Molecular Biology. 705, 1-13 (2011).
- Ruge, D. R., et al. Detection of six serotypes of botulinum neurotoxin using fluorogenic reporters. Analytical Biochemistry. 411 (2), 200-209 (2011).
- Hu, X., et al. Structural insight into exosite binding and discovery of novel exosite inhibitors of botulinum neurotoxin serotype A through in silico screening. Journal of Computer-Aided Molecular Design. 28 (7), 765-778 (2014).
- Dunning, F. M., et al. Detection of botulinum neurotoxin serotype A, B, and F proteolytic activity in complex matrices with picomolar to femtomolar sensitivity. Applied and Environmental Microbiology. 78 (21), 7687-7697 (2012).
- Nguyen, T. G., et al. Development of fluorescent substrates and assays for the key autophagy-related cysteine protease enzyme ATG4B. Assay and Drug Development Technologies. 12 (3), 176-189 (2014).
- Zhang, J. H., Chung, T. D., Oldenburg, K. R. A Simple Statistical Parameter for Use in Evaluation and Validation of High Throughput Screening Assays. Journal of Biomolecular Screening. 4 (2), 67-73 (1999).
- Bovolenta, P., Esteve, P., Ruiz, J. M., Cisneros, E., Lopez-Rios, J. Beyond Wnt inhibition: new functions of secreted Frizzled-related proteins in development and disease. Journal of Cell Science. 121 (Pt 6), 737-746 (2008).
- Esteve, P., et al. SFRPs act as negative modulators of ADAM10 to regulate retinal neurogenesis. Nature Neuroscience. 14 (5), 562-569 (2011).
- Garcia-Hoyos, M., et al. Evaluation of SFRP1 as a candidate for human retinal dystrophies. Molecular Vision. 10, 426-431 (2004).
- Marcos, S., et al. Secreted frizzled related proteins modulate pathfinding and fasciculation of mouse retina ganglion cell axons by direct and indirect mechanisms. Journal of Neuroscience. 35 (11), 4729-4740 (2015).
- Moore, C. A., et al. Characterizing the Pattern of Anomalies in Congenital Zika Syndrome for Pediatric Clinicians. JAMA Pediatrics. 171 (3), 288-295 (2017).
Yeniden Basımlar ve İzinler
Bu JoVE makalesinin metnini veya resimlerini yeniden kullanma izni talebi
Izin talebiThis article has been published
Video Coming Soon
JoVE Hakkında
Telif Hakkı © 2020 MyJove Corporation. Tüm hakları saklıdır