Method Article
Cuantificación de la resistencia a los antibióticos mediada por plásmido en un enfoque de evolución experimental
* Estos autores han contribuido por igual
En este artículo
Resumen
Nuestro enfoque experimental proporciona una estrategia para seguir la abundancia de plásmidos y la resistencia a los antibióticos a lo largo del tiempo en las poblaciones bacterianas.
Resumen
Los plásmidos desempeñan un papel importante en la ecología microbiana y la evolución como vehículos de transferencia lateral de genes y reservorios de funciones genéticas accesorias en poblaciones microbianas. Este es especialmente el caso en entornos que cambian rápidamente, como la exposición fluctuante de antibióticos. Recientemente mostramos que los plásmidos mantienen genes de resistencia a los antibióticos en Escherichia coli sin selección positiva para la presencia de plásmido. Aquí describimos un sistema experimental que permite seguir tanto el genotipo plásmido como el fenotipo en experimentos de evolución a largo plazo. Utilizamos técnicas moleculares para diseñar un plásmido modelo que posteriormente se introduce en un enfoque de sistema de procesamiento por lotes experimental en un huésped de E. coli. Seguimos la frecuencia del plásmido a lo largo del tiempo mediante la aplicación de replicación de las poblaciones de E. coli mientras se cuantifica la persistencia de la resistencia a los antibióticos. Además, monitoreamos la conformación de plásmidos en las células huésped mediante el análisis de la extensión de la formación de muládicos plásmidos mediante el mordete de plásmido y la electroforesis de gel de agarosa. Este enfoque nos permite visualizar no sólo el tamaño del genoma de los plásmidos en evolución, sino también su conformación topológica, un factor muy importante para la herencia de plásmidos. Nuestro sistema combina estrategias moleculares con enfoques microbiológicos tradicionales y proporciona una configuración para seguir plásmidos en poblaciones bacterianas durante mucho tiempo. El enfoque presentado se puede aplicar para estudiar una amplia gama de elementos genéticos móviles en el futuro.
Introducción
Los plásmidos son elementos genéticos circulares y autorreplicantes que son omnipresentes en los prokaryotes. Son agentes de transferencia lateral de genes, ya que pueden transferir rasgos entre poblaciones microbianas, y por lo tanto se consideran un papel importante en la evolución microbiana. Los plásmidos son factores que contribuyen rápidamente a las condiciones que limitan el crecimiento en poco tiempo (por ejemplo, en presencia de antibióticos o pesticidas1) y son responsables de la transición a largo plazo a otros modos de estilo de vida (por ejemplo, la aparición de la patogenicidad2). Los ejemplos más llamativos del impacto de los plásmidos en la transferencia de genes se documentan en ecosistemas expuestos a niveles fluctuantes de antibióticos, como clínicas médicas o en granjas industriales3. Debido a la fuerte selección positiva, muchos plásmidos codifican para los genes de resistencia a los antimicrobianos y a menudo se encuentra que confieren multirresistencia a su huésped bacteriano. Los plásmidos permiten la migración entre poblaciones o especies bacterianas, lo que resulta en una rápida propagación de la resistencia a múltiples antimicrobianos. En condiciones no selectivas, los plásmidos no son esenciales para la célula y a menudo incluso se conocen como elementos parásitos. Sin embargo, los plásmidos son omnipresentes en la naturaleza y su evolución está muy entrelazada con la de los cromosomas bacterianos. La persistencia plásmida en entornos naturales (fluctuantes y no selectivos) sigue siendo poco entendida, pero es de gran importancia para nuestra comprensión de la persistencia de genes de resistencia a los antibióticos en la naturaleza.
La evolución experimental es una poderosa herramienta para el estudio de poblaciones microbianas4. La evolución experimental demostró que la imposición de una fuerte selección para el mantenimiento del plásmido conduce a una evolución compensatoria (es decir, adaptativa) del plásmido o del cromosoma huésped que reduce el coste de la aptitud plásmida y, a su vez, facilita la abundancia de plásmidos (es decir, la persistencia del plásmido)5,6,7. Por lo tanto, después de la interacción plásmido-anfitrión a lo largo del tiempo puede revelar mecanismos importantes de adaptación de ambos elementos. Además, la evolución experimental permite cuantificar la abundancia de células portadoras de plásmidos a lo largo del tiempo en diversas condiciones8,9,10.
La persistencia de plásmidos en los experimentos de evolución puede ser monitoreada por varias estrategias, incluyendo citometría de flujo por clasificación de células fluorescentes activadas (FACS)11, PCR cuantitativo (qPCR)11,o en métodos basados en el cultivo. La citometría de flujo requiere una máquina FACS y la introducción de un gen marcador detectable (fluorescente), como la proteína fluorescente verde (GFP), en el plásmido. Sin embargo, la expresión gFP puede alterar varias propiedades celulares y, además, influir en la ubicación del plásmido en la celda12,lo que a su vez puede influir en la herencia de plásmidos durante la división celular. Un enfoque qPCR para medir la abundancia de plásmido puede ser muy sesgado por el número de copia de plásmido, que puede variar mucho a lo largo de la fase de crecimiento bacteriano y con el tiempo13. Por último, un enfoque basado en la cultura y el revestimiento requiere la introducción de un gen marcador seleccionable. Esto puede ser un gen de resistencia a los antibióticos, que a menudo se codifica en plásmidos naturales; por lo tanto, no es necesaria ninguna manipulación genética. La resistencia a los antibióticos puede ir seguida de un enfoque tradicional de replicación. Por lo tanto, para estudiar la dinámica de plásmido natural, el revestimiento de réplicas es adecuado para monitorear la resisencia de antibióticos codificadas por plásmidos14.
Para visualizar moléculas de plásmido (por ejemplo, para evaluar el tamaño del plásmido), se pueden aplicar varios métodos. Los plásmidos enteros se pueden amplificar utilizando un enfoque basado en PCR. Sin embargo, esto requiere el diseño de imprimaciones específicas, que pueden ser desafiantes durante un experimento de evolución, porque la secuencia de plásmidos puede cambiar con el tiempo. Además, es difícil amplificar los multimers de plásmido en un enfoque basado en PCR debido a múltiples sitios de enlace para los imprimadores PCR. Las moléculas de plásmido multimérico pueden aparecer después de la terminación de la replicación del plásmido o a través de la recombinación de moléculas de plásmido y están orientadas principalmente de cabeza a cola15. Otro enfoque de la visualización de plásmidos combina la digestión enzimática de moléculas de plásmidos por endonucleasas de ADN que cortao o nick una cadena de ADN plásmido con el análisis de electroforesis de gel de agarosa. El mismo plásmido de diferentes tamaños (por ejemplo, monómeros frente a multimers) da lugar a diferentes movilizaciones de gel que se pueden observar al visualizar las moléculas de plásmido. Este enfoque permite la visualización y cuantificación de diferentes conformaciones plásmidas (es decir, estados de multimerización). La conformación plásmido puede utilizarse como indicador de estabilidad plásmido, ya que los multimeros plásmidos se pierden con frecuencia durante la división celular16.
En un trabajo reciente, seguimos la persistencia del plásmido en condiciones que no eran selectivas para la abundancia de plásmidos (es decir, sin selección de antibióticos). Comparamos la persistencia de plásmidos a dos temperaturas diferentes (20 oC y 37 oC) y tres tamaños de población (es decir, tasas de dilución). La aplicación de diversas tasas de dilución, o cuellos de botella de la población, permite la investigación de la influencia del tamaño de la población en la evolución bacteriana y plásmida. En base a nuestros resultados, proponemos que los plásmidos puedan ser neutros para su huésped bacteriano y puedan evolucionar la estabilidad sin ninguna presión de selección8. La estabilidad del plásmido evolucionado se confiere por la reducción de la formación multimer plásmido8.
Aquí, presentamos un protocolo para la cuantificación de la persistencia del plásmido y la investigación de la evolución del plásmido en relación con el mantenimiento de genes de resistencia a antibióticos. El método tiene varios pasos, incluyendo la inserción de un gen de resistencia a antibióticos en un plásmido modelo (que se puede omitir cuando se utiliza un plásmido de resistencia natural), seguido por el uso de la evolución experimental para evaluar el potencial del plásmido para persistir en condiciones no selectivas mientras se determina la dinámica de frecuencia plásmido a lo largo del tiempo utilizando el revestimiento de réplicas, y el análisis del genoma plásmido por visualización. El protocolo descrito aquí fue diseñado para investigar la evolución y persistencia de los plásmidos, pero también se puede aplicar para seguir la evolución de los genes de resistencia cromosómica (u otros genes marcadores) a lo largo del tiempo.
Protocolo
1. Construcción de un plásmido modelo que lleve un gen de resistencia a los antibióticos
NOTA: La cepa Escherichia coli K-12 MG1655 fue utilizada como organismo modelo en todos los experimentos (DSM No. 18039, Colección Alemana de Microorganismos y Cultivos Celulares, DSMZ). Durante la construcción del plásmido se utilizó la cepa E. coli DH5-17.
- PCR amplificar la columna vertebral plásmido de su elección y amplificar el gen de resistencia incluyendo la región promotora por PCR (Figura 1).
- PCR amplifica la columna vertebral del plásmido utilizando una polimerasa de alta fidelidad y los oligonucleótidos pBBR1_for (5'-GCGGCCACCGGCTCTCT-3') y pBBR1_rev (5'-TACGCGCGCCAGCGTGACCC-3') en la plantilla de plásmido pLC (GenBank acc. no. MH238456)18.
- PCR amplificar el gen de resistencia nptII incluyendo el promotor nativo Tn5 19 utilizando una polimerasa de alta fidelidad y oligonucleótidos nptII_gib_for (5'-GCGCCGCCCCCCCCCCCCCCTCCTTGAAGCTGCGCGcTGCCGCGCA-3') y nptII_gib_rev (5'-CGGTCCCCGCCAAAACCCCATCCGTGGTACCGCACACACACACACA-3'). El gen nptII codifica para una fosfotransferasa de neomicina y confiere resistencia a la kanamicina.
NOTA: Diseñe las imprimaciones para el gen de resistencia con aproximadamente 20 bp de secuencia complementaria a la columna vertebral del plásmido a la que se fusionará.
- Limpie ambos fragmentos de PCR con un kit de su elección.
- Unir el producto PCR del gen de resistencia a antibióticos purificados (incluida su región promotora) a la columna vertebral del plásmido purificado y fusionar las regiones homólogas utilizando el ensamblaje isotérmico20, a 50 oC durante 60 minutos.
- Electroportar el producto fusionado en la cepa E. coli DH5.
- Introducir 2 ml del producto desde el paso 1,3 hasta 40 l de células electrocompetentes en cubetas de 2 mm a 4 oC y 2,5 kV. Resuspender las células en 1 ml de caldo de lisógenia (LB) medio.
- Transfiera el volumen total a un tubo de microfúctelo e incuba1 h a 37 oC agitando a 250 rpm en un agitador orbital para permitir la expresión del marcador de resistencia en el plásmido.
- Placa 100 l de las células en placas de agar LB que contienen el antibiótico adecuado (kanamicina 25 g/ml) para seleccionar el gen de resistencia a los antibióticos y así seleccionar para las células portadoras de plásmido. Gire hacia abajo el resto, retire el sobrenadante, resuspenda las células en 100 l lb y placa en una placa de agar selectiva. Incubar las placas a 37oC durante 24 h.
- Para verificar los clones, extraiga los plásmidos construidos a través de la lisis alcalina utilizando un kit de mini-preparación comercial.
- Cosecha de 5 ml del cultivo nocturno estacionario centrifugando a 12.000 x g a temperatura ambiente. Resuspenda las células en la solución de resuspensión, luego lise y neutralice la solución celular. Centrífuga durante 5 min a 12.000 x g.
- Transfiera el sobrenadante a la columna de unión al ADN incluida en el kit y lave la columna dos veces con una centrifugadora de solución de lavado de 500 l 2 veces antes de eluir la membrana de la columna con tampón de elución.
- Realice la secuenciación de Sanger del plásmido para confirmar que la secuencia es correcta.
- Una vez verificada la validez del plásmido, electroporteeele el plásmido (ahora pCON) en la cepa E. coli MG1655 como se describió anteriormente. Esto produce cepa E. coli MG1655 pCON.
2. Monitoreo de bacterias portadoras de plásmidos bajo diversas condiciones a lo largo del tiempo
NOTA: El experimento de evolución se lleva a cabo con cepas portadoras de plásmidos en condiciones no selectivas (medios LB) en dos temperaturas (37 oC y 20 oC) y tres tamaños de cuello de botella de población. El diseño experimental se utiliza para estudiar la persistencia plásmido bajo diversas condiciones.
- Diseño de un experimento de evolución para seguir la frecuencia del plásmido a lo largo del tiempo (Figura 2)
- Placar la cepa de plásmido construida(E. coli MG1655 pCON) en placas de agar LB suplementadas con antibióticos (kanamicina 25 g/ml) e incubar durante la noche a 37 oC.
- Prepare 96 placas de pozo profundo con 1 ml de medio LB en cada poca. Como ancestros bacterianos, recoger ocho colonias aisladas al azar de la placa de agar en pozos independientes. Incubar las placas a 37 oC y 450 rpm en una coctelera durante 24 h. Preparar un stock de glicerol congelado de los clones ancestrales.
- Al día siguiente transferir las ocho poblaciones de réplica en nuevas placas de pozo profundo de acuerdo con el diseño experimental (Figura 2). Los cultivos se diluyen 1:100 (cuello de botella grande, L), 1:1,000 (cuello de botella medio, M), o 1:10,000 (pequeño cuello de botella, S) en un volumen total de 1 ml LB usando PBS para la dilución. Los cultivos diluidos se incuban a 37 oC y 20 oC.
NOTA: Es muy importante controlar la contaminación cruzada en la placa de 96 pocillos profundos. Por lo tanto, utilice un diseño de placa de tablero de ajedrez intercalando pozos inoculados con medio LB libre de bacterias. Utilice este patrón a través de todo el experimento de evolución. - Los cultivos incubados a 37oC se transfieren cada 12 h, mientras que los cultivos a 20oC se transfieren cada 24 h.
NOTA: Durante cada evento de transferencia se aplica el tratamiento de tamaño del cuello de botella y la transferencia en serie se repite en un total de 98 transferencias. El número de transferencias dependerá del diseño experimental de los lectores. - Preparar una población de glicerol congelado de todas las poblaciones regularmente, 2 veces a la semana.
- Supervisión de la frecuencia del plásmido mediante chapado de réplica (Figura 3)
NOTA: Durante el experimento de evolución, la frecuencia de las células portadoras de plásmidos en la población se estima a partir de la proporción de huéspedes. El protocolo de chapado de réplica se muestra en la Figura 3.- Para determinar la frecuencia de plásmido en la población durante el experimento de evolución, los cultivos estacionarios se diluyen en serie y se chapan en placas de agar LB no selectivas. Ajuste la dilución según un rendimiento de 250-500 colonias por placa.
NOTA: Prepare placas de agar LB gruesas antes del revestimiento (agar de 30 ml). - Las poblaciones chapadas son incubadas para el crecimiento nocturno de acuerdo con su temperatura de crecimiento en el experimento.
NOTA: Las colonias deben ser pequeñas, por lo tanto incuban <24 h a 37 oC. - Después del crecimiento nocturno, cuente todas las colonias utilizando una estación de recuento manual o automatizada de colonias y calcule el tamaño total de la población bacteriana en los cultivos.
NOTA: Las colonias en el borde de la placa de agar no deben incluirse en el recuento total de células bacterianas. - Esterilice las piezas cuadradas (20 x 20 cm) de terciopelo de algodón mediante autoclave.
NOTA: El paño de terciopelo debe ser 100% algodón para ser autoclavable. Es importante secar el paño después de la esterilización. - Después de la esterilización del paño de terciopelo, coloque el paño en un bloque redondo y fíjelo con un anillo de metal. Es crucial evitar las arrugas. Coloque cuidadosamente el plato con las colonias cultivadas (agar mirando hacia abajo) en la superficie fija del paño de terciopelo. Asegúrese de que todas las colonias toquen la superficie de terciopelo tocando cuidadosamente el plato Petri de una manera circular.
- Retire cuidadosamente la placa de agar LB y coloque una placa selectiva suplementada con antibióticos (kanamicina 25 g/ml) sobre el paño de terciopelo. Asegúrese de que el plato está tocando todo el terciopelo tocando cuidadosamente en el plato Petri como se describió anteriormente. Después, retire la placa. Deje las placas a temperatura ambiente para el crecimiento nocturno.
- Al día siguiente, evalúe tanto la placa de agar LB como la placa selectiva. Las colonias que crecen en los medios selectivos se cuentan como huéspedes de plásmido (es decir, resistentes a los antibióticos), mientras que las manchas libres de colonias son colonias que no tenían plásmidos y, por lo tanto, no son resistentes al antibiótico (es decir, pierden el plásmido). Esto se hace colocando las placas una sobre la otra y comparando el crecimiento (es decir, marcar las colonias que faltan) y contando el número de colonia en ambas placas. Esto produce el número de células que perdieron el plásmido durante el experimento de evolución.
- Repita este procedimiento a lo largo de todo el experimento de evolución de manera regular (por ejemplo, cada 14 transferencias).
- Para determinar la frecuencia de plásmido en la población durante el experimento de evolución, los cultivos estacionarios se diluyen en serie y se chapan en placas de agar LB no selectivas. Ajuste la dilución según un rendimiento de 250-500 colonias por placa.
3. Visualización de multimers de plásmido utilizando electroforesis de gel
NOTA: La extracción de plásmidos de plásmidos de copia baja a menudo conduce a la contaminación con ADN cromosómico del huésped que necesita ser digerido enzimáticamente antes de la visualización.
- Extraiga ADN plásmido de un cultivo celular inmóvil de 5 ml utilizando lisis alcalina como se describe en el paso 1.5.
- Después, tratar el ADN plásmido extraído con una DNase dependiente de ATP que sólo corta el ADN cromosómico para eliminar la contaminación del ADN cromosómico (ver Tabla de Materiales). Incubar a 37 oC durante 30 minutos después, limpiar el ADN utilizando un kit de su elección.
- Para crear moléculas de círculo abierto de todas las conformaciones de plásmidos (monómeros o multimers), incubar las muestras de ADN plásmido con una enzima de mordida (Nb.BsrDI) e incubar durante 30 min a 65 oC.
- Paralelamente, para crear moléculas de plásmido lineal (es decir, para la comparación de tamaño plásmido), utilice una enzima de restricción de su elección (por ejemplo, HindIII) que corta el plásmido una vez.
NOTA: Esto da como resultado monómeros de plásmido de ADN lineal. Las moléculas de círculo abierto no migran de manera lineal. - Para visualizar el tamaño y la conformación del plásmido, electrophorese las muestras de ADN plásmido y lineal estallado durante 120 min a 4,3 V/cm en un gel de agarosa del 1% (p/v) y un tampón TAE de 1o. Las muestras se tiñen con Verde Midori y el gel se visualiza en un sistema de imágenes de gel (ver Tabla de Materiales). Utilice una escalera de 1 kbp.
Resultados
Aquí, presentamos un enfoque para estudiar la evolución del plásmido mediante la cuantificación de la persistencia del plásmido en una población. En primer lugar, mostramos cómo construir el plásmido llevando cepa De. coli MG1655 pCON que posteriormente se introduce en un experimento de evolución. En segundo lugar, presentamos un método sencillo para seguir la abundancia de plásmido en las poblaciones bacterianas en evolución. Por último, mostramos cómo visualizar el tamaño y la conformación de la molécula de plásmido.
En nuestro trabajo anterior8 utilizando el enfoque presentado llevamos a cabo un experimento de evolución después de la persistencia de plásmidos de resistencia a antibióticos en E. coli en ausencia de antibióticos (Figura 4). Nuestros resultados representativos muestran la evolución de las poblaciones a 37oC y una tasa de dilución de 10-4. Después de la abundancia de células portadoras de plásmidos, observamos una disminución en la frecuencia de las células huésped portadoras de plásmido a lo largo del tiempo (Figura 4). Nuestro enfoque nos permitió descubrir que la pérdida de plásmido fue el resultado de la arquitectura del genoma del plásmido condensado, que llevó a la inestabilidad plásmida causada por conflictos introducidos por la transcripción del gen de la resistencia y la replicación del plásmido en sí. Visualizar las moléculas de plásmido nos permitió descubrir que estos conflictos condujeron a una conformación de plásmido inestable (es decir, formación multimer plásmido, Figura 5). No obstante, observamos la evolución de la estabilidad del plásmido sin la exposición a antibióticos(Figura 4). La estabilidad evolucionada fue conferida por una duplicación intrínseca plásmida que abolió los conflictos de transcripción-replicación y condujo a la formación de plásmidos establemente heredados. Nuestros resultados demuestran así la importancia de la recombinación y la amplificación del genoma en la evolución adaptativa de los elementos genéticos.
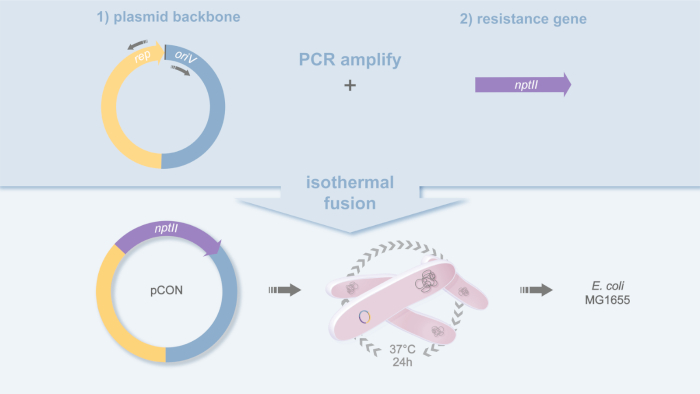
Figura 1: Diseño plásmido de pCON. Representación esquemática de la estrategia de clonación utilizada para construir el plásmido pCON. La columna vertebral del plásmido (pBBR1) y el gen de resistencia a los antibióticos(nptII)se amplifican y fusionan PCR por fusión isotérmica20. Esto produce el plásmido pCON y la cepa MG1655 pCON. Haga clic aquí para ver una versión más grande de esta figura.
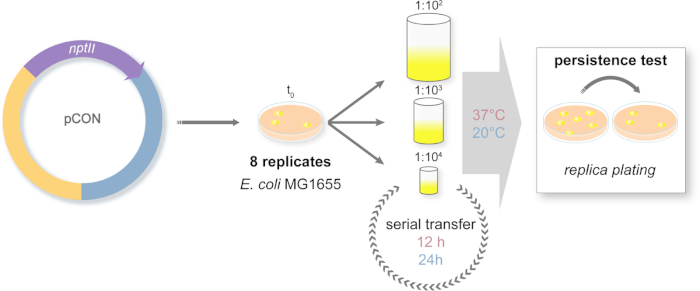
Figura 2: Diseño del experimento de evolución a largo plazo. Representación esquemática del experimento de transferencia en serie. Las poblaciones de plásmido (pCON) están chapadas en soportes selectivos. Las colonias ancestrales se eligen aleatoriamente de la placa y se introducen en un sistema de transferencia en serie. Las transferencias se realizan con tres enfoques de dilución diferentes para simular cuellos de botella de población de diferentes tamaños. Las diluciones se repiten en serie. El experimento se lleva a cabo en dos regímenes de temperatura. La frecuencia de host plásmido se mide a lo largo del experimento a través de la replicación. Haga clic aquí para ver una versión más grande de esta figura.
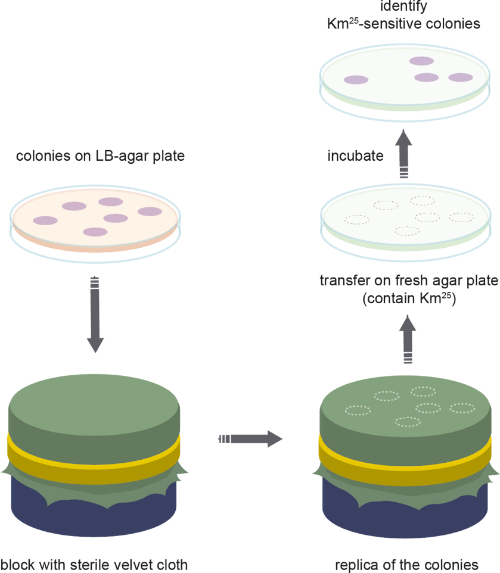
Figura 3: Replicación. Representación esquemática de los pasos utilizados en el revestimiento de réplicas de poblaciones bacterianas. Haga clic aquí para ver una versión más grande de esta figura.
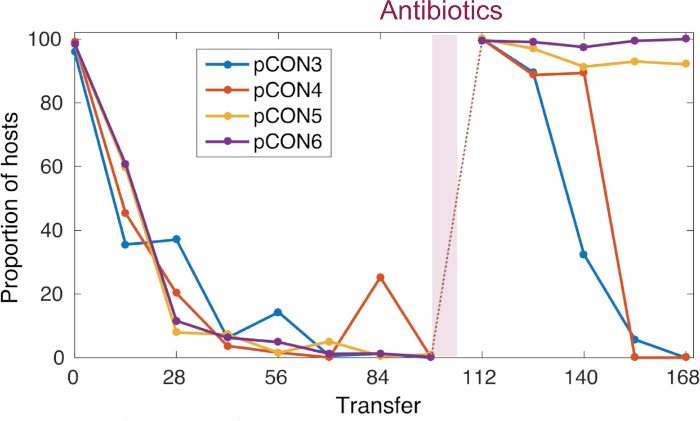
Figura 4: Frecuencia pCON representativa a lo largo del tiempo. La persistencia de pCON se muestra como la proporción de hosts (poblaciones representativas de réplicas) durante el experimento de evolución. Para 98 transferencias, las poblaciones de plásmidos pCON evolucionaron en condiciones no selectivas con un factor de dilución de 10-4. Todas las réplicas portadoras del plásmido pCON disminuyeron en la población. Posteriormente, las poblaciones fueron expuestas a antibióticos para la incubación durante la noche y se cultivaron de nuevo en condiciones no selectivas para probar la evolución de la estabilidad del plásmido. Esta figura ha sido modificada de Wein et al.8Haga clic aquí para ver una versión más grande de esta figura.
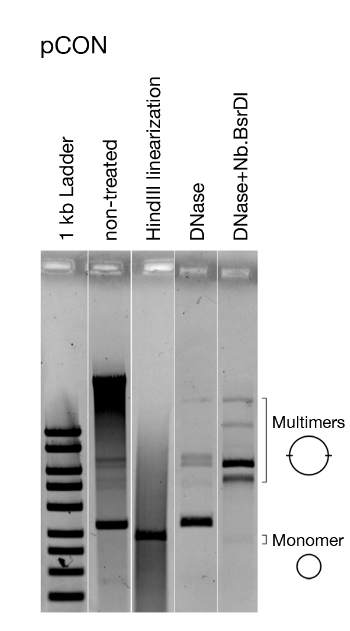
Figura 5: Análisis representativo de la conformación del plásmido. Visualización del plásmido modelo pCON. Visualizado es el ADN plásmido no tratado directamente después de la extracción, el ADN de plásmido linealizado, y tratado con DNase que sólo corta el ADN cromosómico, así como el ADN enzimáticamente nicked (es decir, el ADN de plásmido de círculo abierto). La linealización del plásmido muestra que todos los plásmidos son del mismo tamaño. La eliminación del ADN cromosómico y el pCON de mordida revela la presencia de dimers y otros multimers. Esta cifra ha sido modificada de Wein et al.8. Haga clic aquí para ver una versión más grande de esta figura.
Discusión
En este protocolo, presentamos un enfoque que combina técnicas en biología molecular, evolución experimental y visualización del ADN para investigar el papel de la evolución del plásmido para la persistencia de la resistencia a los antibióticos en las bacterias. Aunque el enfoque presentado combina métodos de diferentes áreas de investigación, todas las técnicas aplicadas son sencillas y se pueden realizar en un laboratorio de microbiología estándar.
Los pasos más críticos en el protocolo incluyen la construcción de la cepa del sistema modelo que incluye la validación genética del genotipo portador del plásmido. En particular, muchos plásmidos codifican naturalmente genes de resistencia a los antibióticos. Por lo tanto, el lector puede omitir el paso 1 del protocolo y proceder directamente con el paso 2. A continuación, los experimentos de evolución deben incluir un diseño aleatorio de las poblaciones de réplica para que los resultados no estén sesgados por la posición de las poblaciones de réplica en la placa de pozo profundo. Además, es especialmente importante llevar a cabo cuidadosamente los pasos de transferencia y dilución en serie en el experimento de evolución, ya que la contaminación falsificaría los resultados. Por último, el revestimiento de réplicas debe llevarse a cabo con mucho cuidado. El tamaño de colonia grande puede ser un problema, pero se puede evitar incubando las placas durante menos de 24 h. Del mismo modo, el número de colonias en una placa podría sesgar los resultados de la réplica. Por lo tanto, las poblaciones deben diluirse antes del revestimiento y la replicación.
Una de las mayores ventajas de nuestro enfoque es que se puede reproducir fácilmente sin necesidad de equipos pesados. Además, otra ventaja de la replicación para seguir los genes marcadores es que solo se evalúan las células vivas, en contraste con la citometría de flujo o qPCR en la que las células muertas pueden evaluarse como vivas. Por lo tanto, el revestimiento de réplica introduce menos sesgo en el recuento de células portadoras de plásmidos. No obstante, una limitación del revestimiento de réplica puede ser el tamaño de la población (es decir, el número de celda) que es posible evaluar en una ejecución experimental.
Utilizando nuestro enfoque, recientemente hemos demostrado que la evolución de la estabilidad del plásmido potencia la persistencia de genes de resistencia a los antibióticos en bacterias. Por lo tanto, desarrollamos un enfoque como herramienta para seguir la persistencia de resistencia mediada por plásmidos que es de gran importancia para seguir la resistencia en el tiempo, especialmente en condiciones sin la presencia de antibióticos.
Divulgaciones
Los autores no tienen nada que revelar.
Agradecimientos
Agradecemos a Gor Margaryan por su apoyo creativo y asistencia técnica. Este trabajo fue apoyado por la ZMB Young Scientist Grant 2017/2018 (premiada a TW) y el programa de enfoque DFG 1819 (Grant No. DA1202/2-1 otorgado a TD).
Materiales
| Name | Company | Catalog Number | Comments |
| 96-deep-well plates | Starlab | ||
| 96-deep-well plates | Roth | EN07.1 | 2 ml, square |
| 96-deep-well plates (cryo) | Starlab | E1702-8400 | Micro-Dilution Tube System |
| Colony counter | Stuart | SC6+ | |
| Cotton velvet | drapery shop | 100 % cotton required | |
| Electrophoresis chamber | BioRad | Agarose gel electrophoresis | |
| Electrophoresis power supply | BioRad | 1645070 | Agarose gel electrophoresis |
| Electroporation cuvettes | BioRad | 1652089 | 0.1 cm |
| Electroporator | BioRad | 1652660 | |
| GeneJet Gel Extraction kit | Thermo Fisher Scientific | K0832 | PCR fragment clean-up |
| GeneJet Plasmid Miniprep kit | Thermo Fisher Scientific | K0503 | Plasmid extraction kit |
| Gibson Assembly | New England Biolabs | E2611S | |
| Incubator | Thermo Fisher Scientific | 50125852 | |
| Incubator (plate shaker) | Heidolph | 1000 | |
| Incubator (shaker) | New Brunswick Scientific | Innova 44 | |
| Inoculating loops | Sigma-Aldrich | ||
| Multi-channel pippetes | Eppendorf | 3125000052, 3125000028 | |
| Multi-channel pippetes | Capp | ME8-1250R | |
| NanoDrop 2000/2000c | Thermo Fisher Scientific | ND2000 | |
| Oligonucleotides | Eurofines | ||
| Petri dishes | Sigma-Aldrich | ||
| Phusion Polymerase | Thermo Fisher Scientific | F533S | |
| Pipettes | Eppendorf | 3123000012, 3123000098, 3123000055, 3123000063, | |
| PlasmidSafe enzyme | Epicentre | 10059400 | |
| Reaction tubes | Eppendorf | 30125150 | |
| Replica block & metal ring | VWR | 601-3401 | PVC cylinder 69 mm; ring 102cm |
| Resctriction enzymes | New England Biolabs | ||
| Thermocycler | BioRad | T100 |
Referencias
- San Millan, A. Evolution of plasmid-mediated antibiotic resistance in the clinical context. Trends in Microbiology. 26 (12), 978-985 (2018).
- Bruto, M., James, A., et al. Vibrio crassostreae, a benign oyster colonizer turned into a pathogen after plasmid acquisition. The ISME Journal. 11 (4), 1043-1052 (2017).
- von Wintersdorff, C. J. H., et al. Dissemination of antimicrobial resistance in microbial ecosystems through horizontal gene transfer. Frontiers in Microbiology. 7 (110), 305-310 (2016).
- Lenski, R. E. Experimental evolution and the dynamics of adaptation and genome evolution in microbial populations. ISME Journal. 11 (10), 2181-2194 (2017).
- De Gelder, L., Williams, J. J., Ponciano, J. E. M., Sota, M., Top, E. M. Adaptive plasmid evolution results in host-range expansion of a broad-host-range plasmid. Genetics. 178 (4), 2179-2190 (2008).
- Harrison, E., Guymer, D., Spiers, A. J., Paterson, S., Brockhurst, M. A. Parallel compensatory evolution stabilizes plasmids across the parasitism-mutualism continuum. Current Biology. 25 (15), 2034-2039 (2015).
- Bouma, J. E., Lenski, R. E. Evolution of a bacteria/plasmid association. Nature. 335 (6188), 351-352 (1988).
- Wein, T., Hülter, N. F., Mizrahi, I., Dagan, T. Emergence of plasmid stability under nonselective conditions maintains antibiotic resistance. Nature Communications. 10 (1), 2595(2019).
- Loftie-Eaton, W., et al. Compensatory mutations improve general permissiveness to antibiotic resistance plasmids. Nature Ecology & Evolution. 1 (9), 1354-1363 (2017).
- Millan, A. S., et al. Positive selection and compensatory adaptation interact to stabilize non-transmissible plasmids. Nature Communications. 5 (1), 1-11 (2014).
- Loftie-Eaton, W., Tucker, A., Norton, A., Top, E. M. Flow cytometry and real-time quantitative PCR as tools for assessing plasmid persistence. Applied and Environmental Microbiology. 80 (17), 5439-5446 (2014).
- Münch, K. M., et al. Polar fixation of plasmids during recombinant protein production in Bacillus megaterium results in population heterogeneity. Applied and Environmental Microbiology. 81 (17), 5976-5986 (2015).
- Jahn, M., Vorpahl, C., Hübschmann, T., Harms, H., Müller, S. Copy number variability of expression plasmids determined by cell sorting and Droplet Digital PCR. Microbial Cell Factories. 15 (1), 211(2016).
- Lederberg, J., Lederberg, M. E. Replica plating and indirect selection of bacterial mutants. Journal of Bacteriology. 63, 399-406 (1952).
- Summers, D. K., Sherratt, D. J. Multimerization of high copy number plasmids causes instability: ColE1 encodes a determinant essential for plasmid monomerization and stability. Cell. 36 (4), 1097-1103 (1984).
- Summers, D. K., Beton, C. W., Withers, H. L. Multicopy plasmid instability: the dimer catastrophe hypothesis. Molecular Microbiology. 8 (6), 1031-1038 (1993).
- Hanahan, D. Studies on transformation of Escherichia coli with plasmids. Journal of Molecular Biology. 166 (4), 557-580 (1983).
- Ilhan, J., et al. Segregational drift and the interplay between plasmid copy number and evolvability. Molecular Biology and Evolution. 36 (3), 472-486 (2019).
- Beck, E., Ludwig, G., Auerswald, E. A., Reiss, B., Schaller, H. Nucleotide sequence and exact localization of the neomycin phosphotransferase gene from transposon Tn5. Gene. 19 (3), 327-336 (1982).
- Gibson, D. G., et al. Enzymatic assembly of DNA molecules up to several hundred kilobases. Nature Methods. 6 (5), 343-345 (2009).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados