Method Article
Análisis de la hipermutación somática en el intron JH4 de células germinales del centro B de parches de Mouse Peyer
En este artículo
Resumen
Aquí se presenta un ensayo para cuantificar la hipermutación somática dentro del locus genético de cadena pesada de inmunoglobulina utilizando células del centro germinal B de los parches del ratón Peyer.
Resumen
Dentro de los centros germinales de los órganos linfoides, las células B maduras alteran su inmunoglobulina expresada (Ig) mediante la introducción de mutaciones no atenuadas en los exones de codificación variable de la Ig pesada y ligera cadena genética loci. Este proceso de hipermutación somática (SHM) requiere la enzima desaminasa de citidina inducida por activación (AID), que convierte deoxycytidinas (C), en desoxiuridinas (U). El procesamiento de los desajustes U:G generados por AID en mutaciones por la escisión de la base y las vías de reparación de desajustes introduce nuevas secuencias de codificación Ig que pueden producir una afinidad más alta. Las mutaciones en los genes de ayuda o reparación del ADN pueden bloquear o alterar significativamente los tipos de mutaciones observadas en el Ig loci. Describimos un protocolo para cuantificar mutaciones intrones JH4 que utiliza clasificación celular activada por fluorescencia (FACS), PCR y secuenciación sanger. Aunque este ensayo no mide directamente la maduración de la afinidad Ig, es indicativo de mutaciones en secuencias de codificación variables de Ig. Además, estos métodos utilizan técnicas comunes de biología molecular que analizan mutaciones en secuencias Ig de múltiples clones de células B. Por lo tanto, este ensayo es una herramienta invaluable en el estudio de la diversificación de SHM e Ig.
Introducción
Las células B, miembros del sistema inmunitario adaptativo, reconocen y eliminan los antígenos mediante la producción de anticuerpos, también conocidos como inmunoglobulinas (Ig). Cada Ig se compone de dos polipéptidos de cadena pesados (IgH) y dos ligeros (IgL), que se mantienen unidos por enlaces de disulfuro para formar la estructura característica de forma "Y" de la Ig1. Los N-termini de IgH e IgL comprenden la región variable (V) de cada polipéptido y juntos forman el sitio de unión de antígenos de la Ig, mientras que la región constante de IgH imparte la función efectora de la Ig. El desarrollo de células B en la médula ósea reorganiza los exones de codificación V de IgH e IgL en un proceso conocido como recombinación V(D)J2,3,4. La transcripción de los exones V recombinados, junto con los respectivos exones de la región constante, forma el ARNM que se traduce al Ig.
Las células B maduras que expresan una Ig ligada a la membrana, también conocida como receptor de células B (BCR), circulan a órganos linfoides secundarios, como el bazo, el ganglio linfático o los parches de Peyer, donde examinan el medio ambiente en busca de antígenos e interactúan con otras células del sistema inmunitario1. Dentro de los centros germinales (GC) de órganos linfoides secundarios, se activan las células B que reconocen el antígeno a través del BCR. Ayudados por células dendríticas foliculares y células T auxiliares foliculares, las células B activadas pueden entonces proliferar y diferenciarse en células plasmáticas y de memoria, que son factores importantes de una respuesta inmune robusta5,6,7,8,9. Además, estas células B activadas pueden someterse a procesos secundarios de diversificación del gen Ig: recombinación de interruptores de clase (RSC) e hipermutación somática (SHM). Durante la RSC, las células B intercambian la región μ constante predeterminada del polipéptido IgH con otra región constante (γ, α, ε) a través de una reacción de recombinación de eliminación de ADN (Figura 1). Esto permite la expresión de un exon constante diferente y la traducción de un nuevo Ig. La célula B pasará de expresar IgM a otro isotipo (IgG, IgA, IgE). CSR cambia la función de efector de la Ig sin alterar su especificidad de antígeno10,11,12. Sin embargo, durante SHM, las células B mutan las regiones de codificación V de IgH e IgL para permitir la producción y selección de Igs de mayor afinidad, lo que puede eliminar más eficazmente un antígeno13,14,15 ( Figura1). Es importante destacar que tanto la RSC como la SHM dependen de la función de una enzima: deaminasa de citidina inducida por activación (AID)16,17,18. Los seres humanos y ratones deficientes en AID no pueden completar RSC o SHM y presentan titers séricos IgM elevados o Hyper-IgM17,19.
En RSC, AID deamina las desoxicitodinas (C) en las regiones de conmutación repetitivas que preceden a cada exones de codificación constante, convirtiéndolas en desoxicitadas (U)20,21, lo que crea un emparejamiento de base desajustado entre desoxiuridinas y desoxiguanosinas (U:G). que son necesarias para la recombinación del ADN, ya sea por la reparación de la escisión base (BER) o la vía de reparación de desajuste (MMR)22,23,24,25,26,27,28,29. En SHM, AID deamina C dentro de los exones de codificación V. La replicación a través de la discordancía U:G genera mutaciones de transición de C:G a T:A, mientras que la eliminación de la base de uracil por la proteína BER, glicelasa de ADN uracil (UNG), antes de la replicación del ADN produce mutaciones de transición y transversión16. Las mutaciones nulas en ung aumentan significativamente C:G a T:A mutaciones de transición21,22. Al igual que CSR, SHM requiere las funciones complementarias de MMR y BER. Durante SHM, MMR genera mutaciones en pares base A:T. La desactivación de mutaciones en la homología MutS 2(MSH2)o la polimerasa de ADN η(Polε)reduce significativamente las mutaciones en bases A:T y mutaciones compuestas en MSH2 y Polε prácticamente elimina mutaciones en las bases A:T21,30,31. De acuerdo con el papel crítico de BER y MMR en la conversión de U generada por AID en mutaciones de transición o transversión, los ratones deficientes tanto para MSH2 como para UNG (MSH2-/-UNG-/-) muestran solo mutaciones de transición de C:G a T:A resultantes de la replicación en el discordancía U:G21.
El análisis de SHM en regiones de codificación V sigue siendo complicado porque el desarrollo de células B puede recombinar cualquiera de los exones de codificación V(D)J en los IgH e IgL loci1,2,4. El análisis preciso de estas regiones V recombinadas y somáticamente mutadas requiere la identificación y aislamiento de clones de células B o el ARN11,13de Ig. El intron JH4, que es 3' del último exon de codificación J en el locus IgH, alberga mutaciones somáticas debido a la propagación de mutaciones 3' del promotor V32,33,34 y por lo tanto se utiliza con frecuencia como marcador sustituto para SHM en V regiones31,35 ( Figura1). Para dilucidar experimentalmente cómo genes específicos o mutaciones genéticas alteran los patrones o tasas de SHM, el intron JH4 se puede secuenciar a partir de las células germinales B (GCBCs) de Peyer, que experimentan altas tasas de SHM36,37,38. Los GCBC se pueden identificar y aislar fácilmente con anticuerpos conjugados fluorescentemente contra marcadores de superficie celular (B220+PNAHI)17,39.
Se presenta un protocolo detallado para caracterizar mutaciones de intrones JH4 en GCBC PP de ratones utilizando una combinación de FACS (clasificación celular activada por fluorescencia), secuenciación de PCR y Sanger (Figura 2).
Protocolo
Todos los ratones mutantes se mantuvieron en un fondo C57BL/6. Los ratones machos y hembras de 2 a 5 meses de edad fueron utilizados para todos los experimentos. La cría y los experimentos con ratones se llevaron a cabo de acuerdo con los protocolos aprobados por el Comité Institucional de Cuidado y Uso de Animales del City College of New York.
1. Disección de los parches de Peyer
- Eutanasia el ratón con 100% CO2 a 3 L/min durante 5 minutos seguido de dislocación cervical para confirmar la muerte. Esterilizar herramientas de disección (tijeras, fórceps, fórceps finos) y manos con guantes con 70% etanol.
- Poner el ratón en la almohadilla de disección con el abdomen expuesto. Rocíe generosamente el cuerpo del ratón con un 70% de etanol antes de hacer cualquier incisión para esterilizar el área de disección.
- Haga una incisión en la piel a través del abdomen y retire la piel del abdomen tirando simultáneamente a ambos lados de la incisión hacia la cabeza y la cola usando fórceps (o manos esterilizadas y con guantes).
- Fija las extremidades delanteras y traseras del ratón.
- Corta la cavidad peritoneal con tijeras para exponer los órganos internos.
- Localice el intestino delgado entre el estómago y el caecum ("J" estructura en forma de cerca del colon). Retire el intestino delgado cortando por debajo del estómago y por encima del caecum.
- Retire cualquier tejido conectivo y grasa que vincule los pliegues del intestino delgado.
NOTA: La grasa tendrá un color blanco distintivo, a diferencia del color rosa del intestino delgado. - Examine la superficie externa del intestino delgado en busca de parches (PPs) del Peyer, que son pequeñas (~1 mm), estructuras ovaladas que aparecen blancas debajo de una fina capa de células epiteliales translúcidas.
- Excita cuidadosamente a todos los PP visibles con tijeras.
NOTA: Un ratón de tipo salvaje C57BL/6 (WT) puede producir 4-8 PPs, mientras que un ratón AID-/- tendrá 6-10 PPs. - Recoja los PPs en un tubo de microcentrífuga de 1,5 ml que contenga 1 ml de amortiguador FACS en hielo.
NOTA: El PP debe hundirse, mientras que la grasa flotará en la superficie y se puede quitar.
2. Aislamiento celular para FACS
- Coloque un filtro de 40 μm en un plato de 6 pozos con 1 ml de amortiguador FACS frío (4 °C).
- Vierta los PPs del tubo de 1,5 ml sobre el filtro.
- Lave los PPs con 1 ml de amortiguador FACS frío, asegurándose de que siempre estén en líquido y en hielo.
- Utilice el extremo plano del émbolo de una jeringa de 1 ml como peste para aplastar los PPs en el filtro hasta que solo quede tejido conectivo en el filtro.
- Lave el filtro y el émbolo con 1 ml de amortiguador FACS frío para liberar las células en el plato de 6 pozos.
- Recoge el ~4 ml de células en el búfer FACS frío y filtrarlas a través de un tubo FACS de 40 μm de tapa de colador.
- Lave la tapa del colador con 1 ml de amortiguador FACS frío.
- Peletizar las celdas a 600 x g a 4 °C durante 5 minutos en una centrífuga de cubo oscilante.
- Decantar al sobrenadante.
- Resuspend las células en 0,4 ml de búfer FACS frío.
- Retire 10 μL para que el recuento de celdas verifique el rendimiento (espere ~5 x 106 celdas/ratón, consulte la Figura 3A)
- Filtre las células restantes a través de una tapa de colador de 40 μm en un tubo FACS y proceda a la tinción para FACS.
3. Tinción de GCBC para FACS
- Añadir 1 bloque Fc de μL (anti-ratón sin etiqueta CD16/CD32) a la suspensión celular de 400 μL y colocar las células en hielo durante 15 minutos.
- Agregue 2 ml de búfer FACS frío para lavar las celdas.
- Células de pellets a 600 x g a 4 °C durante 5 min y desechar el sobrenadante.
- Resuspend las células en 80 μL de búfer FACS frío.
- Retire 10 μL de células del WT PP para cada control de tinción (4 en total, incluidos 3 controles de manchas individuales y 1 control sin manchas). Deje 40 μL del WT PP para el siguiente paso. Como alternativa, utilice cuentas de compensación para los controles de tinción.
- Mancha cada una de las muestras experimentales (por ejemplo, WT y AID-/-) en 500 μL de tampón FACS frío con 2,5 μL de aglutinina de cacahuete (PNA)-biotina durante 15 minutos en hielo.
- Agregue 2 ml de búfer FACS frío para lavar las celdas.
- Células de pellets a 600 x g a 4 °C durante 5 min y desechar el sobrenadante.
- Mancha cada muestra experimental con 500 μL del cóctel en la oscuridad, sobre hielo, durante 15 min (Tabla 1). Asegúrese de que las células estén completamente resuspended en el cóctel de tinción.
- Prepare controles de una sola mancha para la matriz de compensación.
- Manchar las celdas en 500 μL de amortiguador FACS frío utilizando las diluciones especificadas en la Tabla 2.
- Incubar los controles de tinción en la oscuridad, en el hielo, durante 15 min.
- Agregue 2 mL de amortiguador FACS frío a todos los tubos en los pasos 3.7 y 3.8, pegue las células y deseche el sobrenadante para lavar anticuerpos sin consolidar o DAPI.
- Resuspend las células en 500 μL de amortiguador FACS frío y colocar en el hielo.
- Usando un clasificador de celdas, recoge B220+PNAHI de cada muestra experimental manchada. La Figura 3B muestra los porcentajes típicos de B220+ PNAHI obtenidos de WT y AID-/- PPs. La Figura 3C muestra la estrategia de gating FACS.
4. Extracción de ADN de GCBC
- Células ordenadas de pellets a 600 x g a 4 °C durante 5 minutos y desechar el sobrenadante.
- Resuspend las células en 1 ml de búfer FACS frío y transferir las células a un tubo de microcentrífuga de 1,5 ml.
- Peletizar las células a 600 x g a 4 °C durante 5 minutos y desechar el sobrenadante.
- Resuspend las células en 500 μL de tampón de extracción de ADN y 5 μL de 20 mg/ml de Proteinasa K.
- Incubar a 56 °C durante la noche.
- Precipitar el ADN con 500 μL de isopropanol y 1 μL de glucógeno de 20 mg/ml. Mezcle bien el tubo invirtiendo 5-6x.
- Incubar a temperatura ambiente durante 10 minutos.
- Centrífuga en microcentrífuga durante 15 min a 25 °C a 21.000 x g.
- Deseche el sobrenadante y conserve el pellet, que contiene el ADN precipitado y el glucógeno.
- Lave el pellet de ADN con 1 ml de etanol al 70%.
- Peletizar el ADN en una microcentrífuga durante 10 min a 25 °C a 21.000 x g.
- Retire el 70% de etanol y seque al aire el pellet de ADN durante 5-10 min.
NOTA: Evite el secado excesivo, ya que es posible que el ADN no se rehidrate por completo.
- Resuspend el ADN en 30 μL TE buffer e incubar durante la noche a 56 °C.
5. Amplificación y análisis de secuencia intrones JH4
- Cuantifique el ADN midiendo la absorbancia a una longitud de onda de 260 nm (A260).
NOTA: La concentración típica de ADN recuperada de B220+PNAHI GCBCs de un ratón C57BL/6 es de 20-40 ng/μL. - Realice el PCR anidado para el intron JH4 (Tabla 3, Tabla 4). Normalice la cantidad total de ADN genómico utilizado en el primer PCR a la muestra menos concentrada. (por ejemplo, si la muestra menos concentrada es de 5 ng/μL, utilice 58,75 ng de ADN para todas las muestras en el volumen máximo de agua (11,75 μL) en pcr #1).
- Resuelva el producto PCR en un gel de agarose del 1,5% a 200 V durante 20 minutos. El tamaño esperado del amplificador es de 580 bp.
- Excisa el amplificador del gel y extrae el ADN usando un kit de extracción de gel de acuerdo con las instrucciones del fabricante (ver Figura Suplementaria 1).
- Elute el ADN con 30 μL de agua y cuantificar la cantidad de ADN midiendo el A260.
NOTA: La concentración típica del producto PCR purificado es de 3-10 ng/μL.
- Elute el ADN con 30 μL de agua y cuantificar la cantidad de ADN midiendo el A260.
- Ligar el producto PCR purificado en un plásmido con extremos contundentes. Estandarizar la cantidad total de producto PCR utilizado en cada reacción de ligadura (Tabla 5).
- Incubar la reacción de ligadura a temperatura ambiente durante 5 min o durante la noche a 16 °C.
- Transforme las células bacterianas electrocompetentntes con 2 μL de la reacción de ligadura.
- Electropolar a 1,65 kV.
- Rescate en 600 μL de soporte SOC durante 1 h a 37 °C en una incubadora de temblores a 225 rpm.
- Placa 100 μL de bacterias transformadas en LB complementada con placas de agar ampicilina (100 μg/ml) e incuba durante la noche a 37 °C.
- Someta la placa de colonias bacterianas para la secuenciación de Sanger usando la imprimación delantera T7. Alternativamente, cultivar cultivos nocturnos de cada colonia bacteriana y realizar una purificación de plásmidos.
- Si es necesario, repita el PCR, la ligadura y/o la transformación para optimizar el rendimiento de las colonias bacterianas
NOTA: Se debe recoger un mínimo de 30 colonias de cada plato.
- Si es necesario, repita el PCR, la ligadura y/o la transformación para optimizar el rendimiento de las colonias bacterianas
- Estandarizar los datos de secuencia en los archivos de .txt
- Elimine la secuencia de plásmidos.
- Asegúrese de que cada secuencia esté orientada de 5' a 3' según la secuencia de referencia del intron JH4 (NG_005838). Genere el complemento inverso de cualquier secuencia, según sea necesario.
- Alinee las secuencias obtenidas para cada PCR con la secuencia de referencia de intrones JH4 (NG_005838) utilizando un software Clustal Omega (Figura 4A).
- Identificar las diferencias de la secuencia de referencia como mutaciones
- Verifique que todas las mutaciones sean mutaciones puntuales verdaderas examinando el electroferograma de la secuenciación de Sanger. Repita la secuenciación si es necesario. (Figura 4B,C).
- Tabular y cuantificar mutaciones únicas en el intron JH4 para cada genotipo (Figura 5).
- Contar secuencias con mutaciones idénticas sólo una vez
NOTA: No es posible determinar si las secuencias idénticas se generaron durante el PCR o eventos SHM idénticos en diferentes celdas B. - Cuente cada caso de secuencias de intrones JH4 de línea germinosa WT (es decir, aquellas sin mutaciones) como una secuencia única.
- Contar secuencias con mutaciones idénticas sólo una vez
Resultados
Citometría de flujo
Las células B maduras circulan a centros germinales donde sufren maduración de afinidad, expansión clonal y diferenciación en células plasmáticas o de memoria40,41,42,43,44. Estos GCBC pueden ser identificados por numerosos marcadores de superficie celular, incluyendo alta expresión del receptor CD45R/B220 y unión de aglutinina de cacahuete (PNA)45,46. Para aislar los GCBC activados, las células PP fueron manchadas con anticuerpos anti-B220 conjugados a ficoeritro (PE) y biotinilado-PNA, seguidos de estreptococodos conjugados a APC-eFluor780. Las células muertas fueron eliminadas usando el tinte fluorescente 4',6-Diamidino-2-Phenylindole (DAPI), que mancha el ácido nucleico de las células moribundas o muertas47,48. Las células manchadas fueron posteriormente analizadas y ordenadas a través de la citometría de flujo. Los PPs consistían en ~80% B220+ celdas49,50. Wt PPs contienen en promedio 4 x 106 celdas por ratón (Figura 3A). Aproximadamente el 8% de las células WT PP eran B220+PNAHI, que es la mitad del número observado en AID-/ - ( Figura3B). Así, 0,3-0,6 x 106 B220+PNAHI GCBC se obtuvieron después de la clasificación, que fueron suficientes para analizar mutaciones en el intron JH4.
Análisis de secuencias JH4
El intron JH4 fue amplificado por un PCR anidado utilizando imprimaciones comunes de la familia VHJ558 (J558FR3Fw y VHJ558.2), seguido de JH4 intron que abarca los imprimaciones VHJ558.3 y VHJ558.435,37. De las 105 secuencias únicas obtenidas de los GCBC wt, se encontraron un total de 226 mutaciones (Figura 5A). El análisis del espectro de mutaciones gcbc en los ratones WT mostró una gama de transversiones y transversiones a una velocidad de 4 x 10-3 mutaciones/bp, que se calculó dividiendo el número total de bases mutadas por el número total de bases que se secuenciaron32,36,37,38. Además, cada producto JH4 PCR de WT GCBC contenía 1-25 mutaciones(Figura 5B),donde se encontraron con frecuencia múltiples mutaciones en una secuencia33,36. Sólo se identificaron dos mutaciones en 113 secuencias AID-/- (Figura 5A). AID-/- Las células B mostraron 1,66 x 10-5 mutaciones/bp, que fue significativamente inferior a las células WT B (p <0,05)36 y se compara con la tasa de error de la polimerasa de alta fidelidad (5,3 x 10-7 sub/base/duplicación)51,52. Por lo tanto, las células AID-/- B sirvieron como un control negativo útil para este ensayo.
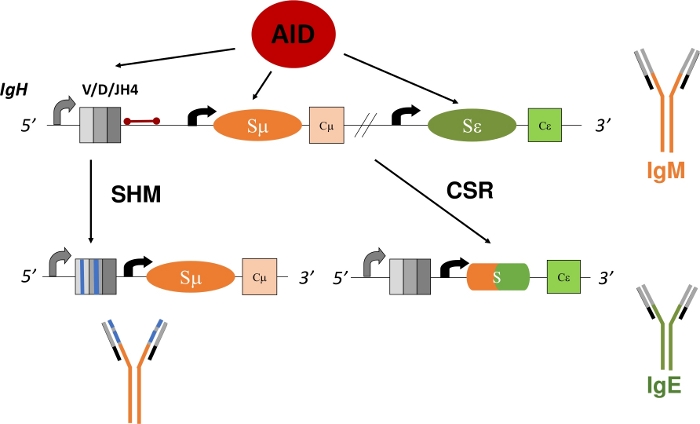
Figura 1: Esquema del locus genético IgH y las regiones objetivo de AID durante la RSC y el SHM. La barra roja indica el intron JH4 de 580 bp que es 3' de reordenamientos VDJH4 y se analiza en este protocolo. En la RSC, la desaminación de las regiones de conmutadores intronic (Sμ y Sε) dependientes de AID promueve la formación de DSB que permite la recombinación de la supresión y la expresión de un nuevo isotipo de anticuerpos (IgM a IgE). Durante SHM, las regiones V (cajas grises) acumulan mutaciones (líneas azules) que pueden conducir a una mayor afinidad. Haga clic aquí para ver una versión más grande de esta figura.
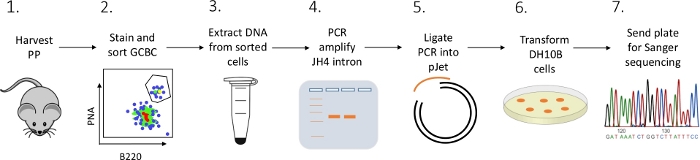
Figura 2: Flujo de trabajo para analizar SHM del intron JH4 en GCBC aislados de PPs. Haga clic aquí para ver una versión más grande de esta figura.
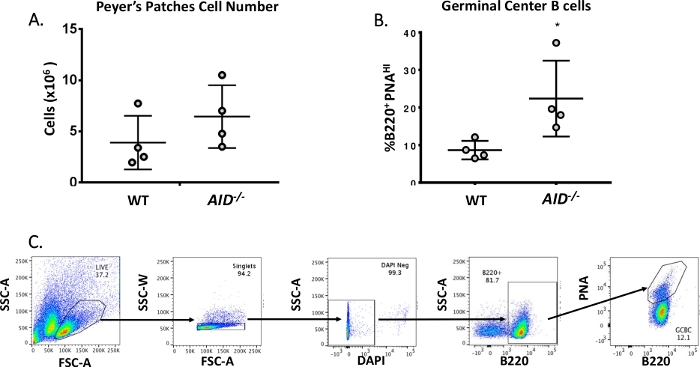
Figura 3: Caracterización de GCBC PP. (A) Número total de células PP de ratones WT y AID-/- (n = 4 por genotipo). Las barras de error representan una desviación estándar de la media. (B) Porcentaje de B220+PNAHI GCBC obtenidos de PPs de WT y AID-/- ratones (n = 4 por genotipo)36. Las barras de error representan una desviación estándar de la media, *p<0.05 mediante la prueba t del alumno. (C) El representante FACS traza ordenar B220+PNAHI GCBCs de los PC. Haga clic aquí para ver una versión más grande de esta figura.
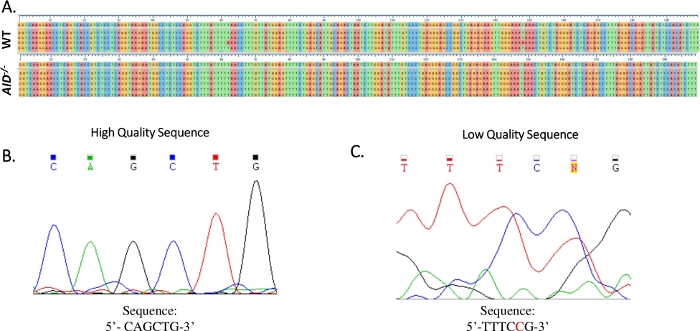
Figura 4: Análisis de datos de secuencia JH4 Sanger. (A) Alineaciones de secuencias de muestra de datos de secuencia Sanger del producto JH4 PCR de WT (superior) y AID-/- (inferior) GCBC a la secuencia genómica de referencia (NG_005838), que es la secuencia inmediatamente por debajo de las marcas de graduación numeradas. Las alineaciones se generaron utilizando Clustal Omega. (B) Electroferograma de datos de secuencia Sanger de alta calidad, que mostraban picos distintos para cada base. (C) Electroferograma de datos de secuencia de baja calidad, que mostraron picos ambiguos y bases no especificadas (N). El nucleótido que se muestra en rojo debe anotarse manualmente en el archivo de texto de secuencia. Haga clic aquí para ver una versión más grande de esta figura.
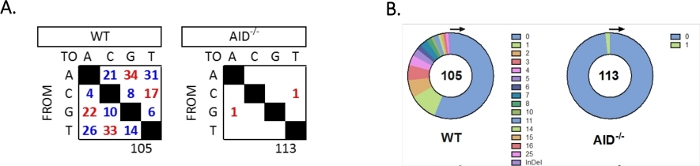
Figura 5: Análisis de mutaciones en el intron JH4 en WT y AID-/- GCBC. (A) El número total de mutaciones de transición (rojo) y transversión (azul) en bases A, C, G y T para cada genotipo se resume en las tablas. El número total de secuencias analizadas se indica debajo de la tabla. (B) El número de mutaciones por amplificador DEP para cada genotipo se representa en los gráficos circulares. Esta cifra ha sido modificada a partir de36 Copyright 2020. La Asociación Americana de Inmunólogos, Inc. Haga clic aquí para ver una versión más grande de esta figura.
| Cóctel de tinción para GCBC | Volumen: 500 μL | ||
| Anticuerpo o tinte | Flúor | dilución | μL |
| B220 | pei | 1000 | 0.5 |
| Streptavidin | APC-eFluor780 | 500 | 1 |
| DAPI | N/A | 500 | 1 |
Tabla 1: Cócteles de tinción para GCBC. El cóctel de los anticuerpos o colorantes indicados (indicados en cursalics) en las diluciones especificadas se utilizó para manchar las células PP en 500 μL para la citometría de flujo.
| Manchas individuales para la compensación | Volumen: 500 μL | ||
| Anticuerpo o tinte | Flúor | dilución | μL |
| B220 | pei | 1000 | 0.5 |
| B220 | APC-eFluor780 | 750 | 0.67 |
| DAPI | N/A | 500 | 1 |
Tabla 2: Controles de manchas individuales para compensación. Los anticuerpos B220 conjugados a los fluoróforos indicados se utilizaron para controles de una sola mancha para compensar la superposición espectral.
| #1 pcr | ||||
| Reactivo | volumen | Condiciones del termociclo | ||
| 5x Buffer | 4 μL | 1 | 95 °C | 3 min |
| 10 mM dNTP | 2 μL | 2 | 94 °C | 30 seg |
| 10 μM J558FR3Fw | 1 μL | 3 | 55 °C | 30 seg |
| 10 μM VHJ558.2 | 1 μL | 4 | 72 °C | 1:30 min |
| Polimerasa de ADN de alta fidelidad | 0,25 μL | Ciclo 2-4 9x | ||
| ADN | x (estandarizar a la muestra menos concentrada) | |||
| H2O | a 20 μL | 5 | 72 °C | 5 min |
| Diluir el producto PCR 1:5 en H2O antes de proceder a pcr #2 | ||||
Tabla 3: PCR anidado del intron JH4. Componentes PCR y condiciones de termociclo para la primera reacción de amplificación. Diluir el primer producto PCR 1:5 con agua y utilizar 1 μL de esta dilución para el segundo PCR.
| #2 PCR | ||||
| Reactivo | volumen | Condiciones del termociclo #2 | ||
| 5x Buffer | 4 μL | 1 | 94 °C | 3 min |
| 10 mM dNTP | 2 μL | 2 | 94 °C | 30 seg |
| 10 μM VHJ558.3 | 1 μL | 3 | 55 °C | 30 seg |
| 10 μM VHJ558.4 | 1 μL | 4 | 72 °C | 30 seg |
| Polimerasa de ADN de alta fidelidad | 0,25 μL | Ciclo 2-4 21x | ||
| PCR diluido#1 | 1 μL | |||
| H2O | a 20 μL | 5 | 72 °C | 5 min |
Tabla 4: Componentes de PCR y condiciones de termociclo para el segundo PCR.
| Reactivo | volumen |
| 2x Buffer | 10 μL |
| PCR purificado | x (estandarizar a la muestra menos concentrada) |
| Plásmido con extremos contundentes | 1 μL |
| Ligamento de ADN T4 | 1 μL |
| H2O | a 20 μL |
| Incubar a temperatura ambiente durante 5 minutos o durante la noche a 16ºC | |
Tabla 5: Reacción de ligadura. Componentes para la ligadura del producto purificado JH4 intron PCR en el plásmido.
| Búfer FACS |
| El calor inactiva FBS a 56 °C durante una hora antes de su uso. Suplemento PBS, pH 7.4 (Gibco, #10010049) con 2.5% (v/v) de FBS inactivado por calor. Conservar a 4°C. |
| Búfer de extracción de ADN (100 mM Tris pH 8.0, 0.1 M EDTA, 0.5% (w/v) SDS) |
| Añadir 50 mL de 1 M Tris pH 8.0, 100mL de 0.5 M EDTA, y 12.5 mL de 20% SDS. Añadir agua destilada a 500 ml. Conservar a temperatura ambiente. |
| Tampón TE (Tris pH de 10 mM 8.0, 1 mM EDTA) |
| Añadir 2,5 ml de 1 M Tris pH 8,0 y 500 mL de 0,5 M EDTA. Añadir agua destilada a 250 ml. Conservar a temperatura ambiente. |
Tabla 6: Recetas de búfer.
| Lista de oligonucleótidos | ||
| J558FR3Fw | 5'-GCCTGACATCTGAGGACTCTGC-3' | |
| VHJ558.2 | 5'-CTGGACTTTCGGTTTGGTG-3' | |
| VHJ558.3 | 5'-GGTCAAGGAACCTCAGTCA-3' | |
| VHJ558.4 | 5'-TCTCTAGACAGCAACTAC-3' | |
Tabla 7: Oligonucleótidos utilizados en el ensayo.
Figura complementaria 1: Imagen de gel de agarose representativa después de completar el paso 5.4. El producto PCR anidado JH4 intron se resolvió en un gel de agarose del 1,5% y el amplificador de 580 bp fue excizado. WT PP indica que el ADN genómico WT PP GCBC se utilizó como plantilla para el primer PCR y AID PP indica que el ADN genómico DE AID-/- PP GCBC se utilizó como plantilla para el primer PCR. ɸ indica el control PCR sin plantilla y - indica que no se cargó nada en el pozo del gel de agarose. El último carril muestra una escalera de ADN de 100 bp. Haga clic aquí para descargar esta figura.
Discusión
Caracterizar SHM dentro de las secuencias de codificación IgH e IgL V de una población heterogénea de células B presenta un desafío, dado que cada célula B reorganiza de forma única los segmentos de codificación V durante la recombinación V(D)J34. En este artículo, describimos un método para identificar mutaciones en el intron JH4 de GCBC. El intron JH4, que se encuentra a 3' del último segmento de codificación J en el locus IgH, se utiliza como sustituto para SHM de regiones V (Figura 1)31,33,34,35. Para catalogar estas mutaciones de intrones JH4 y evaluar cómo los genes específicos afectan a la producción o patrón de mutaciones, se analizan específicamente los GCBC PP. Estas células acumulan mutaciones de intrones JH4 como resultado de la estimulación crónica por microbiota intestinal53. Además, los GCBC B220+PNAHI de los PPs de ratones no inmunizados tienen un espectro de mutación que se compara con los GCBC esplenicos de animales inmunizados54,55. Sin embargo, las mutaciones en el intron JH4 no pueden correlacionarse con la maduración de la afinidad de Ig porque estas mutaciones no son codificantes.
Para determinar si SHM altera la afinidad de Ig, los ratones deben ser inmunizados por vía intraperitoneally con un antígeno, tales como NP (4-hidroxi-3-nitrophenylacetyl) conjugado a CGG (globulina gamma de pollo) o KLH (hemocianina cojera de ojo de cerradura)56. Posteriormente, el ARNM puede purificarse a partir de B220 B220+PNAHI GCBC para examinar SHM dentro de VH186.2, el exon de codificación V que reconoce con mayor frecuencia NP y se muta después de la inmunización NP-CGG o NP-KLH31,57,58,59,60. La mutación del triptófano-33 a una leucina en VH186.2 se ha caracterizado por aumentar la afinidad de Ig hasta 10 veces59,60 y es, por lo tanto, un indicador de que la selección SHM y clonal ha generado alta afinidad Ig. La medición del suero específico NP7 y NP20 Ig titers por ELISA y el cálculo de la relación NP7/NP20 específica de Ig durante el transcurso de la inmunización también documentan la maduración de afinidad Ig resultante de SHM de las regiones V17,21,36. Ambos ensayos se pueden utilizar para correlacionar SHM dentro de las secuencias de codificación VH186.2 con cambios en la maduración de afinidad Ig específica de NP.
Ya sea que se utilicen animales inmunizados o no inmunizados para analizar SHM de VH186.2 o el intron JH4, los GCBC deben identificarse con precisión. Presentamos un enfoque basado en FACS para aislar B220+PNAHI GCBC. Alternativamente, Fas y el antígeno no sulfated α2-6-sialyl-LacNAc, reconocido por el anticuerpo GL761,62,63,64,también se pueden utilizar para aislar GCBCs, que se identifican como B220+Fas+GL7+65 o CD19+Fas+GL7+37. La expresión GL7 refleja estrechamente el PNA en GCBC activados de los ganglios linfáticos64,65,66. Además de utilizar marcadores de anticuerpos específicos para GCBC, los cócteles de tinción deben maximizar la excitación de un flúor y la detección de un biomarcador mientras se minimiza la superposición espectral de la emisión de fluorescencia. Los antígenos expresados a niveles bajos deben detectarse con un anticuerpo que se conjugue a un fluoróforo con una fluorescencia de emisión robusta67. El protocolo de tinción recomendado fue optimizado para su análisis en una clasificadora de celdas equipada con cuatro láseres (405nm, 488nm, 561nm, 633nm) y 12 filtros; sin embargo, las configuraciones de filtro y la disponibilidad del láser varían entre los citómetros. Para modificar el protocolo de acuerdo con la disponibilidad de reactivos y equipos, el lector se refiere a recursos adicionales, visores de espectro en línea y literatura publicada67,68,69,70,71,72,73. El protocolo de tinción multicolor descrito en este documento requiere una compensación de la superposición espectral para garantizar que las poblaciones celulares ordenadas sean GCBC en lugar de la detección inexacta de la emisión de fluorescencia. B220 sirve como un control de tinción útil para el FACS descrito(Cuadro 1B)porque los PPs tendrán poblaciones negativas y positivas distintivas B220(Figura 3C),lo que permite una compensación adecuada de la superposición espectral. La estrategia de gating presentada en la Figura 3C debe utilizarse como guía. Las gráficas de citometría de flujo pueden variar dependiendo de las condiciones de tinción y la configuración del citómetro. Sin embargo, 4-10% de las células vivas deben ser B220+PNAHI 35, 52.
Todas las mutaciones dentro del intron JH4 de los GCBC PP deben validarse para garantizar que las mutaciones observadas reflejen verdaderamente el SHM y no un artefacto de PCR o secuenciación. AID-/- Las células B pueden servir como un control negativo útil al examinar el fenotipo SHM en otros modelos de ratón mutante porque estas células no pueden completar SHM17,19. La tasa de mutación del intron JH4 en el de AID-/- GCBC (1.66x10-5 mutaciones/bp)20,21,36,37,38,50,74 es comparable a la tasa de error de la polimerasa de alta fidelidad (5.3x10-7 sub/base/duplicación)51,52 que se utiliza para amplificar el ADN en el PCR anidado. Si aid-/- ratones no están disponibles, compare el patrón de mutación observado y la frecuencia con la literatura publicada. Las regiones Ig V acumulan 10-3-10-4 mutaciones por división de par base, que es aproximadamente 106-doble que la tasa de mutación de otros genes loci73,75. Los resultados pueden variar con la edad del animal76. Alternativamente, las células B220+PNALO, que marcan no GCBC, pueden utilizarse como control negativo en ausencia de AID-/- ratones52. Si la frecuencia de mutación en los GCBC WT es menor de lo esperado, la secuencia intronic de la línea germinaria WT JH4 puede representarse desproporcionadamente. En este caso, asegúrese de que los GCBC se mancharon y ordenaron adecuadamente y los PCR están libres de contaminación por intrones JH4 de la línea germinosa WT. Además, los datos de secuenciación sin procesar en electroferogramas deben analizarse a fondo para garantizar que las mutaciones en los datos de texto de secuencia no sean artefactos de errores de secuenciación. Por ejemplo, los resultados de secuenciación deficientes de Sanger pueden reducir la fiabilidad de los datos de secuencia (Figura 4). Este control de calidad de los datos de secuencia de Sanger aumentará la precisión y reproducibilidad del análisis de mutación del intron JH4.
Divulgaciones
Los autores no tienen nada que revelar.
Agradecimientos
Agradecemos a Tasuku Honjo por los ratones AID-/-. Este trabajo fue apoyado por el Instituto Nacional de Disparidades de Salud y Salud de las Minorías (5G12MD007603), el Instituto Nacional del Cáncer (2U54CA132378) y el Instituto Nacional de Ciencias Médicas Generales (1SC1GM132035-01).
Materiales
| Name | Company | Catalog Number | Comments |
| 0.2 ml PCR 8-tube FLEX-FREE strip, attached clear flat caps, mixed | USA Scientific | 1402-4708 | |
| Ampicillin sodium salt | Fisher | BP1760-5 | |
| APC-eFluor780 anti-CD45R/B220 | eBioscience | 47-0452-80 | clone RA3-6B2 |
| BD FACSAria II | BD | 643186 | four lasers (405nm, 488nm, 561nm, 633nm) and 12 filters (PacBlue (450/50), AmCyan (502LP; 530/30), SSC (488/10), FITC (502LP; 530/30), PerCP-Cy5.5 (655LP; 695/40), PE (585/15), PE-Texas Red (600LP; 610/20), PE-Cy5 (630LP; 670/14), PE-Cy7 (735LP; 780/60), APC (660/20), Alexa700 (710LP; 730/45), APC-Cy7 (755LP; 780/60)) |
| BD slip tip 1mL syringe | Fisher | 14-823-434 | sterile |
| Biotinylated peanut agglutinin (PNA) | Vector Labs | B-1075-5 | |
| C57BL/6J mice | Jackson Laboratories | 664 | |
| Corning Falcon test tube with cell strainer snap cap | Fisher | 08-771-23 | |
| DAPI (4',6-Diamidino-2-Phenylindole, dihydrochloride) | Fisher | D1306 | 0.5 mg/ml |
| dNTP | NEB | N0447L | 10 mM |
| ElectroMAX DH10B competent cells | Fisher | 18-290-015 | |
| Falcon cell strainer 40mm | Fisher | 08-771-1 | |
| Falcon round-bottom polystyrene tubes (FACS tubes) | Fisher | 14-959-5 | |
| Falcon round-bottom polystyrene tubes (capped) | Fisher | 149591A | |
| Fetal bovine serum | R&D Systems (Atlanta Biologicals) | S11150 | |
| Gibco phosphate buffered saline PBS pH 7.4 | Fisher | 10-010-049 | |
| Glycogen | Sigma | 10901393001 | |
| Lasergene Molecular Biology (MegAlign Pro) | DNA Star | version 15 | |
| PE anti-CD45R/B220 | BD | 553090 | clone RA3-6B2 |
| Proteinase K | Fisher | BP1700-100 | |
| Q5 High-Fidelity DNA Polymerase | NEB | M0491L | |
| QIAquick Gel Extraction Kit | Qiagen | 28706 | |
| Seal-Rite 1.5mL microcentrifuge tubes | USA Scientific | 1615-5500 | |
| Streptavidin APC-eFluor 780 Conjugate | eBioscience | 47-4317-82 | |
| T4 DNA ligase | NEB | M020L | |
| Thermo Scientific CloneJET PCR Cloning Kit | ThermoFisher | FERK1231 | |
| Tissue culture plate 6 well | Fisher | 08-772-1B | sterile |
| Unlabeled anti-mouse CD16/CD32 (Fc block), BD | Fisher | BDB553142 | Clone 2.4G2 |
Referencias
- Murphy, K., Weaver, C. . Janeyway's Immunobiology. , (2016).
- Alt, F. W., et al. VDJ recombination. Immunology Today. 13 (8), 306-314 (1992).
- Schatz, D. G., Ji, Y. Recombination centres and the orchestration of V (D) J recombination. Nature Reviews Immunology. 11 (4), 251-263 (2011).
- Oettinger, M. A., Schatz, D. G., Gorka, C., Baltimore, D. RAG-1 and RAG-2, adjacent genes that synergistically activate V (D) J recombination. Science. 248 (4962), 1517-1523 (1990).
- Berek, C., Berger, A., Apel, M. Maturation of the immune response in germinal centers. Cell. 67 (6), 1121-1129 (1991).
- Linterman, M. A., et al. Foxp3+ follicular regulatory T cells control the germinal center response. Nature Medicine. 17 (8), 975 (2011).
- Shulman, Z., et al. T follicular helper cell dynamics in germinal centers. Science. 341 (6146), 673-677 (2013).
- Good-Jacobson, K. L., et al. PD-1 regulates germinal center B cell survival and the formation and affinity of long-lived plasma cells. Nature Immunology. 11 (6), 535 (2010).
- Kerfoot, S. M., et al. Germinal center B cell and T follicular helper cell development initiates in the interfollicular zone. Immunity. 34 (6), 947-960 (2011).
- Chaudhuri, J., Alt, F. W. Class-switch recombination: interplay of transcription, DNA deamination and DNA repair. Nature Reviews Immunology. 4 (7), 541-552 (2004).
- Alt, F. W., Zhang, Y., Meng, F. L., Guo, C., Schwer, B. Mechanisms of programmed DNA lesions and genomic instability in the immune system. Cell. 152 (3), 417-429 (2013).
- Xu, Z., Zan, H., Pone, E. J., Mai, T., Casali, P. Immunoglobulin class-switch DNA recombination: induction, targeting and beyond. Nature Reviews Immunology. 12 (7), 517-531 (2012).
- Di Noia, J. M., Neuberger, M. S. Molecular mechanisms of antibody somatic hypermutation. Annual Reviews of Biochemistry. 76, 1-22 (2007).
- Peled, J. U., et al. The biochemistry of somatic hypermutation. Annual Review of Immunology. 26, 481-511 (2008).
- Liu, M., Schatz, D. G. Balancing AID and DNA repair during somatic hypermutation. Trends in Immunology. 30 (4), 173-181 (2009).
- Methot, S., Di Noia, J. Molecular Mechanisms of Somatic Hypermutation and Class Switch Recombination. Advances in Immunology. 133, 37-87 (2017).
- Muramatsu, M., et al. Class switch recombination and hypermutation require activation-induced cytidine deaminase (AID), a potential RNA editing enzyme. Cell. 102 (5), 553-563 (2000).
- Petersen-Mahrt, S. K., Harris, R. S., Neuberger, M. S. AID mutates E. coli suggesting a DNA deamination mechanism for antibody diversification. Nature. 418 (6893), 99 (2002).
- Revy, P., et al. Activation-induced cytidine deaminase (AID) deficiency causes the autosomal recessive form of the Hyper-IgM syndrome (HIGM2). Cell. 102 (5), 565-575 (2000).
- Petersen-Mahrt, S. DNA deamination in immunity. Immunological Reviews. 203 (1), 80-97 (2005).
- Rada, C., Di Noia, J. M., Neuberger, M. S. Mismatch recognition and uracil excision provide complementary paths to both Ig switching and the A/T-focused phase of somatic mutation. Molecular Cell. 16 (2), 163-171 (2004).
- Rada, C., et al. Immunoglobulin isotype switching is inhibited and somatic hypermutation perturbed in UNG-deficient mice. Current Biology. 12 (20), 1748-1755 (2002).
- Schrader, C. E., Vardo, J., Stavnezer, J. Role for mismatch repair proteins Msh2, Mlh1, and Pms2 in immunoglobulin class switching shown by sequence analysis of recombination junctions. The Journal of Experimental Medicine. 195 (3), 367-373 (2002).
- Martin, A., et al. Msh2 ATPase activity is essential for somatic hypermutation at AT basepairs and for efficient class switch recombination. The Journal of Experimental Medicine. 198 (8), 1171-1178 (2003).
- Imai, K., et al. Human uracil-DNA glycosylase deficiency associated with profoundly impaired immunoglobulin class-switch recombination. Nature Immunology. 4 (10), 1023-1028 (2003).
- Masani, S., Han, L., Yu, K. Apurinic/apyrimidinic endonuclease 1 is the essential nuclease during immunoglobulin class switch recombination. Molecular and Cellular Biology. 33 (7), 1468-1473 (2013).
- Guikema, J. E., et al. APE1-and APE2-dependent DNA breaks in immunoglobulin class switch recombination. The Journal of Experimental Medicine. 204 (12), 3017-3026 (2007).
- Schrader, C. E., Guikema, J. E., Wu, X., Stavnezer, J. The roles of APE1, APE2, DNA polymerase β and mismatch repair in creating S region DNA breaks during antibody class switch. Philosophical Transactions of the Royal Society B: Biological Sciences. 364 (1517), 645-652 (2009).
- Roa, S., et al. MSH2/MSH6 complex promotes error-free repair of AID-induced dU: G mispairs as well as error-prone hypermutation of A: T sites. PLoS One. 5 (6), 11182 (2010).
- Delbos, F., Aoufouchi, S., Faili, A., Weill, J. C., Reynaud, C. A. DNA polymerase η is the sole contributor of A/T modifications during immunoglobulin gene hypermutation in the mouse. The Journal of Experimental Medicine. 204 (1), 17-23 (2007).
- Maul, R. W., Gearhart, P. J. AID and somatic hypermutation. Advances in Immunology. 105, 159-191 (2010).
- Shen, H. M., Tanaka, A., Bozek, G., Nicolae, D., Storb, U. Somatic hypermutation and class switch recombination in Msh6-/- Ung-/- double-knockout mice. The Journal of Immunology. 177 (8), 5386-5392 (2006).
- Cheng, H. L., et al. Integrity of the AID serine-38 phosphorylation site is critical for class switch recombination and somatic hypermutation in mice. Proceedings of the National Academy of Sciences. 106 (8), 2717-2722 (2009).
- Lebecque, S. G., Gearhart, P. J. Boundaries of somatic mutation in rearranged immunoglobulin genes: 5'boundary is near the promoter, and 3'boundary is approximately 1 kb from V (D) J gene. The Journal of Experimental Medicine. 172 (6), 1717-1727 (1990).
- Jolly, C. J., Klix, N., Neuberger, M. S. Rapid methods for the analysis of immunoglobulin gene hypermutation: application to transgenic and gene targeted mice. Nucleic Acids Research. 25 (10), 1913-1919 (1997).
- Choi, J. E., Matthews, A. J., Michel, G., Vuong, B. Q. AID Phosphorylation Regulates Mismatch Repair-Dependent Class Switch Recombination and Affinity Maturation. The Journal of Immunology. 204 (1), 13-22 (2020).
- McBride, K. M., et al. Regulation of class switch recombination and somatic mutation by AID phosphorylation. The Journal of Experimental Medicine. 205 (11), 2585-2594 (2008).
- Liu, M., et al. Two levels of protection for the B cell genome during somatic hypermutation. Nature. 451 (7180), 841-845 (2008).
- Ross, M., Birbeck, M., Wills, V., Forrester, J., Davis, A. Peanut lectin binding properties of germinal centers of mouse lymphoid tissues. Nature. 284, 364-366 (1980).
- Zhang, J., MacLennan, I. C., Liu, Y. J., Lane, P. J. Is rapid proliferation in B centroblasts linked to somatic mutation in memory B cell clones. Immunology Letters. 18 (4), 297-299 (1988).
- Nieuwenhuis, P., Opstelten, D. Functional anatomy of germinal centers. American Journal of Anatomy. 170 (3), 421-435 (1984).
- Lau, A. W., Brink, R. Selection in the germinal center. Current Opinion in Immunology. 63, 29-34 (2020).
- Victora, G. D., Nussenzweig, M. C. Germinal centers. Annual Review of Immunology. 30, 429-457 (2012).
- Mesin, L., Ersching, J., Victora, G. D. Germinal center B cell dynamics. Immunity. 45 (3), 471-482 (2016).
- Reichert, R. A., Gallatin, W. M., Weissman, I. L., Butcher, E. C. Germinal center B cells lack homing receptors necessary for normal lymphocyte recirculation. The Journal of Experimental Medicine. 157 (3), 813-827 (1983).
- Rose, M., Birbeck, M., Wills, V., Forrester, J., Davis, A. Peanut lectin binding properties of germinal centers of mouse lymphoid tissues. Nature. 284, 364-366 (1980).
- Hamada, S., Fujita, S. DAPI staining improved for quantitative cytofluorometry. Histochemistry. 79 (2), 219-226 (1983).
- Otto, F. DAPI staining of fixed cells for high-resolution flow cytometry of nuclear DNA. Methods in Cell Biology. 33, 105-110 (1990).
- Butcher, E., et al. Surface phenotype of Peyer's patch germinal center cells: implications for the role of germinal centers in B cell differentiation. The Journal of Immunology. 129 (6), 2698-2707 (1982).
- Rogerson, B. J., Harris, D. P., Swain, S. L., Burgess, D. O. Germinal center B cells in Peyer's patches of aged mice exhibit a normal activation phenotype and highly mutated IgM genes. Mechanisms of Ageing and Development. 124 (2), 155-165 (2003).
- Potapov, V., Ong, J. L. Examining sources of error in PCR by single-molecule sequencing. PloS One. 12 (1), 0169774 (2017).
- Gonzalez-Fernandez, A., Milstein, C. Analysis of somatic hypermutation in mouse Peyer's patches using immunoglobulin kappa light-chain transgenes. Proceedings of the National Academy of Sciences. 90 (21), 9862-9866 (1993).
- Reboldi, A., Cyster, J. G. Peyer's patches: organizing B-cell responses at the intestinal frontier. Immunological Reviews. 271 (1), 230-245 (2016).
- Betz, A. G., Rada, C., Pannell, R., Milstein, C., Neuberger, M. S. Passenger transgenes reveal intrinsic specificity of the antibody hypermutation mechanism: clustering, polarity, and specific hot spots. Proceedings of the National Academy of Sciences. 90 (6), 2385-2388 (1993).
- Rada, C., Gupta, S. K., Gherardi, E., Milstein, C. Mutation and selection during the secondary response to 2-phenyloxazolone. Proceedings of the National Academy of Sciences. 88 (13), 5508-5512 (1991).
- Heise, N., Klein, U. Somatic Hypermutation and Affinity Maturation Analysis Using the 4-Hydroxy-3-Nitrophenyl-Acetyl (NP) System. Methods in Molecular Biology. 1623, 191-208 (2017).
- Smith, F., Cumano, A., Licht, A., Pecht, I., Rajewsky, K. Low affinity of kappa chain bearing (4-hydroxy-3-nitrophenyl) acetyl (NP)-specific antibodies in the primary antibody repertoire of C57BL/6 mice may explain lambda chain dominance in primary anti-NP responses. Molecular Immunology. 22 (10), 1209-1216 (1985).
- Takahashi, Y., Dutta, P. R., Cerasoli, D. M., Kelsoe, G. In situ studies of the primary immune response to (4-hydroxy-3-nitrophenyl) acetyl. V. Affinity maturation develops in two stages of clonal selection. The Journal of Experimental Medicine. 187 (6), 885-895 (1998).
- Allen, D., Simon, T., Sablitzky, F., Rajewsky, K., Cumano, A. Antibody engineering for the analysis of affinity maturation of an anti-hapten response. The EMBO Journal. 7 (7), 1995-2001 (1988).
- Cumano, A., Rajewsky, K. Clonal recruitment and somatic mutation in the generation of immunological memory to the hapten NP. The EMBO Journal. 5 (10), 2459-2468 (1986).
- Smith, K., Nossal, G., Tarlinton, D. M. FAS is highly expressed in the germinal center but is not required for regulation of the B-cell response to antigen. Proceedings of the National Academy of Sciences. 92 (25), 11628-11632 (1995).
- Hao, Z., et al. Fas receptor expression in germinal-center B cells is essential for T and B lymphocyte homeostasis. Immunity. 29 (4), 615-627 (2008).
- Cervenak, L., Magyar, A., Boja, R., László, G. Differential expression of GL7 activation antigen on bone marrow B cell subpopulations and peripheral B cells. Immunology Letters. 78 (2), 89-96 (2001).
- Naito, Y., et al. Germinal center marker GL7 probes activation-dependent repression of N-glycolylneuraminic acid, a sialic acid species involved in the negative modulation of B-cell activation. Molecular and Cellular Biology. 27 (8), 3008-3022 (2007).
- Olson, W. J., et al. Orphan Nuclear Receptor NR2F6 Suppresses T Follicular Helper Cell Accumulation through Regulation of IL-21. Cell Reports. 28 (11), 2878-2891 (2019).
- Dorsett, Y., et al. MicroRNA-155 suppresses activation-induced cytidine deaminase-mediated Myc-Igh translocation. Immunity. 28 (5), 630-638 (2008).
- Goetz, C., Hammerbeck, C., Bonnevier, J. . Flow Cytometry Basics for the Non-Expert. , (2018).
- Hawley, T. S., Herbert, D. J., Eaker, S. S., Hawley, R. G. . Flow Cytometry Protocols. , (2004).
- Costa, E., et al. A new automated flow cytometry data analysis approach for the diagnostic screening of neoplastic B-cell disorders in peripheral blood samples with absolute lymphocytosis. Leukemia. 20 (7), 1221-1230 (2006).
- McKinnon, K. M. Flow cytometry: An overview. Current Protocols in Immunology. 120 (1), 1-11 (2018).
- McKinnon, K. M. Multiparameter Conventional Flow Cytometry. Methods in Molecular Biology. , 139-150 (2018).
- Lucchesi, S., et al. Computational Analysis of Multiparametric Flow Cytometric Data to Dissect B Cell Subsets in Vaccine Studies. Cytometry Part A. 97, 259-267 (2019).
- Longerich, S., Tanaka, A., Bozek, G., Nicolae, D., Storb, U. The very 5' end and the constant region of Ig genes are spared from somatic mutation because AID does not access these regions. The Journal of Experimental Medicine. 202 (10), 1443-1454 (2005).
- Retter, I., et al. Sequence and characterization of the Ig heavy chain constant and partial variable region of the mouse strain 129S1. The Journal of Immunology. 179 (4), 2419-2427 (2007).
- Shen, H. M., Peters, A., Baron, B., Zhu, X., Storb, U. Mutation of BCL-6 gene in normal B cells by the process of somatic hypermutation of Ig genes. Science. 280 (5370), 1750-1752 (1998).
- Richter, K., et al. Altered pattern of immunoglobulin hypermutation in mice deficient in Slip-GC protein. Journal of Biological Chemistry. 287 (38), 31856-31865 (2012).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados