Method Article
마우스 페이어의 패치에서 발아 센터 B 세포의 JH4 인트론에서 체증 의 분석
요약
여기에 제시된 마우스 페이어의 패치에서 발아 센터 B 세포를 사용하여 면역 글로불린 무거운 사슬 유전자 궤적 내의 체세포 초열을 정량화하는 분석이다.
초록
림프구의 발아 센터 내에서 성숙한 B 세포는 Ig 무거운 및 광 사슬 유전자 loci의 가변 코딩 엑소에 템플릿되지 않은 돌연변이를 도입하여 발현 면역 글로불린 (Ig)을 변경합니다. 체세포 초순 (SHM)의 이 과정은 탈옥시티딘 (C)을 탈옥시티딘 (U)으로 변환하는 효소 활성화 유도 cytidine deaminase (AID)를 필요로한다. AID 생성 된 U:G 불일치를 기본 절제 및 불일치 수리 경로에 의해 돌연변이로 처리하는 것은 더 높은 친화성 Ig를 생성 할 수있는 새로운 Ig 코딩 서열을 소개합니다. AID 또는 DNA 수리 유전자에 있는 돌연변이는 Ig loci에서 관찰된 돌연변이의 모형을 막거나 현저하게 바꿀 수 있습니다. 우리는 형광 활성화 세포 분류 (FACS), PCR 및 Sanger 시퀀싱을 사용하는 JH4 인트론 돌연변이를 정량화하는 프로토콜을 설명합니다. 이 분석이 Ig 선호도 성숙을 직접 측정하지는 않지만 Ig 변수 코딩 서열에서 돌연변이를 나타냅니다. 추가적으로, 이 방법은 다중 B 세포 클론의 Ig 순서에 있는 돌연변이를 분석하는 일반적인 분자 생물학 기술을 이용합니다. 따라서, 이 분석은 SHM과 Ig 다양화의 연구에 귀중한 도구입니다.
서문
B 세포, 적응 성 면역 계통의 구성원은 면역 글로불린 (Ig)이라고도하는 항체를 생산하여 항원을 인식하고 제거합니다. 각 Ig는 Ig1의특징적인 "Y" 형상 구조를 형성하기 위해 이황화물 결합에 의해 함께 개최되는 두 개의 무거운 (IgH)와 두 개의 빛 (IgL) 체인 폴리 펩티드로 구성됩니다. IgH 및 IgL의 N-termini는 각 폴리펩티드의 가변(V) 영역을 구성하고 함께 Ig의 항원 결합 부위를 형성하지만 IgH의 일정 영역은 Ig의 이펙터 기능을 부여한다. 골수에서 B 세포를 개발하면 V(D)J 재조합2,3,4로알려진 공정에서 IgH 및 IgL의 V 코딩엑온을재배열한다. 재결합된 V exons의 전사는 각각의 일정한 영역 엑슨과 결합되어 Ig로 변환되는 mRNA를 형성합니다.
B세포 수용체(BCR)라고도 하는 멤브레인 바운드 Ig를 발현하는 성숙한 B 세포는 비장, 림프절 또는 페이어의 패치와 같은 이차 림프구 기관으로 순환하여 항원 환경을 조사하고 면역 계통1의다른 세포와 상호 작용한다. 이차 림프구의 발아 센터(GC) 내에서 BCR을 통해 항원을 인식하는 B 세포가 활성화된다. 여포 수지상 세포 및 여포 도우미 T 세포에 의해 원조, 활성화 된 B 세포는 다음 증식 및 강력한 면역 반응의 중요한 이펙터 플라즈마 및 메모리 세포로 분화 할 수있다5,6,7,8,9. 추가적으로, 이 활성화된 B 세포는 이차 Ig 유전자 다양화 프로세스를 겪을 수 있습니다 - 클래스 스위치 재조합 (CSR) 및 체세포 초열 (SHM). CSR 동안, B 세포는 DNA 삭제 재조합반응(도 1)을통해 다른 일정한 영역(γ, α, ε)과 IgH 폴리펩티드의 기본 μ 일정한 영역을 교환한다. 이렇게 하면 다른 일정한 엑소를 표현하고 새 Ig를 번역할 수 있습니다. B 셀은 IgM을 발현하는 것에서 다른 동종유형(IgG, IgA, IgE)으로 전환됩니다. CSR은항원 특이성(10,11,12)을변경하지 않고 Ig의 이펙터 기능을 변경한다. 그러나, SHM 동안, B 세포는 보다 효과적으로 항원13,14,15 (도 1)를제거할 수 있는 더 높은 친화성 Igs의 생산 및 선택을 가능하게 하기 위하여 IgH 와 IgL의 V 코딩영역을돌연변이한다. 중요하게도, CSR과 SHM 은 하나의 효소의 기능에 의존한다: 활성화 유도 된 cytidine deaminase (AID)16,17,18. AID에서 결핍된 인간과 마우스는 CSR 또는 SHM을 완료할 수 없으며 높은 IgM 혈청 티터 또는 하이퍼-IgM17,19로존재한다.
CSR에서 AID는 각 일정한 코딩 엑온보다 먼저 반복적인 스위치 영역에서 데옥시티딘(C)을 제거하여 데옥시리딘과 데옥시구아노신(U:G)과 일치하는 베이스 페어링을 생성하는 데옥시리딘(U)20,21로변환합니다. 이러한 U:G 불일치는 DNA 재조합에 필요한 이중 좌초 DNA 브레이크로 변환되며, 이는 기본 절제 수리(BER) 또는 불일치 수리(MMR)경로(22,23,24,25, 26,27,28,29)중 하나에 의해 된다. SHM에서 AID는 V 코딩 엑온 내에서 C를 분해합니다. U:G 불일치를 통해 복제는 C:G 내지 T:A 전이 돌연변이를 생성하는 반면, BER 단백질에 의한 우라실 베이스를 제거하는 반면, 우라실 DNA 글리코슬라제(UNG)는 DNA 복제 전에 전환 및 전환돌연변이(16)를생성한다. UNG에서 Null 돌연변이는 C:G에서 T:A 전이 돌연변이21,22로크게 증가한다. CSR과 마찬가지로 SHM은 MMR 및 BER의 상호 보완적인 역할이 필요합니다. SHM 동안 MMR은 A:T 염기 쌍에서 돌연변이를 생성합니다. 돌연변이 부호2(MSH2) 또는 DNA중합효소η(Polθ)에서돌연변이를 현저히 감소시키며 MSH2의 A:T 염기 및 복합 돌연변이및 극중에서 돌연변이를 사실상 폐지한다21,30,31. AID 생성 된 U를 전환 또는 전환 돌연변이로 변환하는 BER 및 MMR에 대한 중요한 역할과 일치하며, MSH2 및 UNG(MSH2/- UNG-/-)디스플레이 C:G ~ T:A 전이 돌연변이는 U:G불일치(21)를통해 복제하여 발생하는 전이 돌연변이만 을 나타낸다.
V 코딩 영역에서 SHM의 분석은 개발 B 세포가 IgH 및 IgL loci1,2,4에서V(D)J 코딩 엑온 중 어느 것을 재결합할 수 있기 때문에 복잡합니다. 이러한 고유하게 재결합되고 체세포적으로 돌연변이된 V 영역의 정확한 분석은 B 세포 또는 Ig mRNA11,13의클론의 식별 및 격리를 필요로 한다. 이그H 궤적에서 마지막 J 코딩 엑손의 3'인 JH4 인트론은 V 프로모터32,33,34의 돌연변이 3'의 확산으로 인한 체질 돌연변이를 항만하므로 V 영역31, 35 (도 1)에서SHM에 대한 대리 마커로 자주 사용된다. 특정 유전자 또는 유전 돌연변이가 SHM 패턴 또는 비율을 변경하는 방법을 실험적으로 해명하기 위해 JH4 인트론은 SHM36,37,38의높은 비율을 겪는 Peyer의 패치 (PP) 발아센터 B 세포 (GCBC)로부터 서열 될 수 있습니다. GCBC는 세포 표면 마커(B220+PNAHI)17,39에대하여 형광으로 공주된 항체로 쉽게 식별되고 격리될 수 있다.
상세 프로토콜은 FACS(형광 활성 세포 선별), PCR 및 생거 시퀀싱(도2)의조합을 사용하여 마우스로부터 PP GCBCs에서 JH4 인트론 돌연변이를 특성화하기 위해 제시된다.
프로토콜
모든 돌연변이 마우스는 C57BL/6 배경에서 유지되었다. 나이 일치 (2-5 개월) 남성과 여성 마우스는 모든 실험에 사용되었다. 쥐를 사용한 축산 및 실험은 뉴욕 시 대학 기관 동물 관리 및 사용 위원회가 승인한 프로토콜에 따라 수행되었습니다.
1. 페이어 패치의 해부
- 5분 동안 3L/min에서 100% CO2로 마우스를 안락사시키고 자궁 경부 탈구로 사망을 확인합니다. 해부 도구 (가위, 집게, 미세 집게)와 장갑을 낀 손과 70 %의 에탄올로 살균하십시오.
- 복부가 노출된 해부 패드에 마우스를 놓습니다. 해부 영역을 살균하기 위해 절개를 하기 전에 마우스 의 몸을 70 % 에탄올로 넉넉하게 분사하십시오.
- 복부를 가로질러 피부에 절개를 하고 집게 (또는 살균, 장갑을 낀 손)를 사용하여 머리와 꼬리쪽으로 절개의 양쪽에 동시에 당겨 복부에서 피부를 제거합니다.
- 마우스의 앞다리와 뒷다리를 고정합니다.
- 내부 장기를 노출하는 가위로 복막 구멍을 잘라냅니다.
- 위장과 케이쿰 사이의 소장을 찾습니다 (결장 근처의 "J"모양의 구조). 위장 아래와 caecum 위에 절단하여 소장을 제거합니다.
- 소장의 주름을 함께 연결하는 결합 조직과 지방을 제거하십시오.
참고 : 지방은 소장의 분홍색 색상과는 달리 독특한 흰색을 가질 것입니다. - 작은 (~1mm) 작은 Peyer의 패치(PP)에 대한 소장의 외부 표면을 검사하여 반투명 상피 세포의 얇은 층 아래에 흰색으로 보이는 타원형 구조입니다.
- 가위로 눈에 보이는 모든 PP를 조심스럽게 소비합니다.
참고: C57BL/6 와일드 타입(WT) 마우스 1개에는 4-8P를 얻을 수 있는 반면 AID-/-마우스는 6-10P를 사용할 수 있습니다. - 얼음에 FACS 버퍼의 1 mL을 포함하는 1.5 mL 미세 원심 분리기 튜브로 P를 수집합니다.
참고 : 지방이 표면에 떠 있고 제거 할 수있는 반면 PP는 가라 앉어야합니다.
2. FACS에 대한 세포 격리
- 40 μm 필터를 6웰 접시에 놓고 1mL의 차가운 (4 °C) FACS 버퍼를 놓습니다.
- 1.5mL 튜브에서 PP를 필터에 붓습니다.
- 차가운 FACS 버퍼 1mL로 PP를 세척하여 항상 액체와 얼음 위에 있는지 확인합니다.
- 결합 조직만 필터에 남아있을 때까지 필터에 PP를 분쇄하는 유봉으로 1 mL 주사기에서 플런저의 평평한 끝을 사용합니다.
- 필터와 플런저를 1mL의 차가운 FACS 버퍼로 세척하여 세포를 6 웰 접시로 방출합니다.
- 차가운 FACS 버퍼에서 세포의 ~4 mL을 수집하고 40 μm 스트레이너 캡 FACS 튜브를 통해 필터링합니다.
- 차가운 FACS 버퍼의 1 mL로 스트레이너 캡을 세척합니다.
- 스윙 버킷 원심분리기에서 5분 동안 4°C에서 600 x g의 세포를 펠렛합니다.
- 상체를 장식합니다.
- 콜드 FACS 버퍼의 0.4 mL에서 세포를 다시 중단합니다.
- 셀 카운트에 대한 10 μL을 제거하여 수율을 확인하십시오(예상 ~5 x 106 셀/마우스, 그림 3A참조)
- 40 μm 스트레이너 캡을 통해 남은 세포를 FACS 튜브로 필터링하고 FACS에 대한 염색을 진행합니다.
3. FACS에 대한 GCB를 염색
- 400 μL 셀 서스펜션에 1 μL Fc 블록(레이블이 없는 안티 마우스 CD16/CD32)을 추가하고 세포를 얼음 위에 15분 간 배치합니다.
- 차가운 FACS 버퍼 2mL을 추가하여 세포를 세척합니다.
- 펠릿 셀600 x g에서 4°C에서 5분 동안 및 상복부셀을 폐기하십시오.
- 차가운 FACS 버퍼의 80 μL에서 세포를 다시 중단합니다.
- 각 염색 제어에 대해 WT PP에서 세포의 10 μL을 제거합니다(단일 염색 컨트롤 3개 및 스테인드 컨트롤 1개를 포함). 다음 단계로 WT PP의 40 μL을 둡니다. 또는 염색 컨트롤에 보정 구슬을 사용합니다.
- 각 실험 샘플(예: WT 및 AID-/-)을얼음 위에 15분 동안 땅콩 아글루틴(PNA)-비오틴2.5μL의 차가운 FACS 버퍼500μL에 스테인합니다.
- 차가운 FACS 버퍼 2mL을 추가하여 세포를 세척합니다.
- 펠릿 셀600 x g에서 4°C에서 5분 동안 및 상복부셀을 폐기하십시오.
- 어둠 속에서 칵테일의 500 μL을 가진 각 실험 샘플을 얼음 에 15 분 동안 얼룩지게합니다 (표 1). 염색 칵테일에서 세포가 완전히 재주중단되었는지 확인합니다.
- 보정 행렬에 대한 단일 얼룩 컨트롤을 준비합니다.
- 표 2에지정된 희석을 사용하여 차가운 FACS 버퍼의 500 μL에 세포를 얼룩지게 한다.
- 어둠 속에서, 얼음 위에 15분 동안 스테인딩 컨트롤을 배양합니다.
- 3.7 및 3.8 단계의 모든 튜브에 2 mL 콜드 FACS 버퍼를 추가하고, 세포를 펠릿하고, 무한항체 또는 DAPI를 씻어 내도록 슈퍼나탄을 버린다.
- 차가운 FACS 버퍼의 500 μL에서 세포를 다시 중단하고 얼음에 놓습니다.
- 셀 선별기를 사용하여 각 염색 된 실험 샘플에서 B220+PNAHI를 수집하십시오. 도 3B는 WT 및AID-/-PP에서 얻은 B220+ PNAHI의 일반적인 백분율을 보여 주며, 도 3C는 FACS 게이팅 전략을 표시합니다.
4. GCBC에서 DNA 추출
- 펠릿은 세포를 4°C에서 600 x g에서 5분 동안 분류하고 상체를 폐기합니다.
- 감기 FACS 버퍼의 1 mL에서 세포를 다시 중단하고 1.5 mL 미세 원심 분리기 튜브로 세포를 전송.
- 셀을 4°C에서 600 x g로 5분 동안 펠릿하고 상체를 폐기합니다.
- DNA 추출 버퍼 500 μL 및 20 mg/mL Proteinase K의 5 μL에서 세포를 다시 중단합니다.
- 하룻밤 사이에 56 °C에서 배양하십시오.
- 500 μL 이소프로판올과 20 mg/mL 글리코겐의 1 μLDNA를 침전시합니다. 5-6x를 반전하여 튜브를 완전히 섞습니다.
- 실온에서 10분 동안 배양하십시오.
- 21,000 x g에서25°C에서 15분 동안 미세원심분리기의 원심분리기.
- 상체를 버리고 침전된 DNA와 글리코겐을 포함하는 펠릿을 유지합니다.
- DNA 펠릿을 70% 에탄올 1mL로 씻으십시오.
- 21,000 x g에서 25°C에서 10분 동안 마이크로센트심분리기에서 DNA를 펠렛합니다.
- 70% 에탄올을 제거하고 DNA 펠릿을 5-10분 동안 공기 건조시하십시오.
참고: DNA가 완전히 수분을 보충하지 않을 수 있기 때문에 과다 건조를 피하십시오.
- 30 μL TE 버퍼에서 DNA를 다시 중단하고 56°C에서 하룻밤 동안 배양한다.
5. JH4 인트론 서열 증폭 및 분석
- 260nm(A260)의 파장에서 흡광도를 측정하여 DNA를 정량화한다.
참고: C57BL/6 마우스의 정렬된 B220+PNAHI GCB에서 회수된 DNA의 전형적인 농도는 20-40 ng/μL입니다. - JH4 인트론(표3, 표 4)에대해 중첩된 PCR을 수행한다. 제1 PCR에서 사용되는 총 유전체 DNA양을 가장 적게 농축된 시료로 정규화한다. (예를 들어, 가장 농축된 시료가 5 ng/μL인 경우, PCR #1 최대 물 부피(11.75 μL)의 모든 시료에 대해 DNA의 58.75 ng를 사용하십시오.).
- PCR 제품을 20분 동안 200V에서 1.5% 아가로즈 젤로 해결합니다. 예상 앰플리카 크기는 580 bp입니다.
- 젤에서 앰플리콘을 소비하고 제조업체의 지침에 따라 젤 추출 키트를 사용하여 DNA를 추출합니다(보충 도 1참조).
- 30 μL의 물로 DNA를 엘테우고 A260을 측정하여 DNA의 양을 정량화합니다.
참고: 정제된 PCR 제품의 일반적인 농도는 3-10 ng/μL입니다.
- 30 μL의 물로 DNA를 엘테우고 A260을 측정하여 DNA의 양을 정량화합니다.
- 정제 된 PCR 제품을 무딘 끝으로 플라스미드로 리게이트합니다. 모든 결찰반응(표 5)에사용되는 PCR 제품의 총량을 표준화합니다.
- 16°C에서 5분 또는 하룻밤 동안 실온에서 결찰 반응을 배양한다.
- 결찰 반응의 2 μL로 전기 유능한 세균 세포를 변환합니다.
- 1.65 kV에서 전기 전극.
- SOC 매체의 600 μL에서 37°C에서 225 rpm의 흔들리는 인큐베이터에서 1시간 동안 구조합니다.
- 변형된 박테리아의 플레이트 100 μL은 암피실린(100 μg/mL) 한천 판을 보충하고 37°C에서 하룻밤 동안 배양한다.
- T7 포워드 프라이머를 사용하여 Sanger 시퀀싱을 위해 세균 성 식민지의 접시를 제출하십시오. 또는, 각 세균 식민지의 하룻밤 문화를 성장하고 플라스미드 정화를 수행.
- 필요한 경우 PCR, 결찰 및/또는 변환을 반복하여 세균 성 식민지의 수율을 최적화합니다.
참고: 각 플레이트에서 최소 30개의 콜로니를 골라야 합니다.
- 필요한 경우 PCR, 결찰 및/또는 변환을 반복하여 세균 성 식민지의 수율을 최적화합니다.
- .txt 파일의 시퀀스 데이터 표준화
- 플라스미드 시퀀스를 삭제합니다.
- JH4 인트론 참조 서열(NG_005838)에 따라 모든 시퀀스가 5'에서 3'으로 방향인지 확인합니다. 필요에 따라 모든 시퀀스의 역보수를 생성합니다.
- Clustal 오메가소프트웨어(도 4A)를사용하여 JH4 인트론 기준 서열(NG_005838)에 대해 각 PCR에 대해 얻은 서열을 정렬합니다.
- 참조 서열의 차이점을 돌연변이로 식별합니다.
- 모든 돌연변이가 Sanger 시퀀싱의 전극체를 검사하여 진정한 점 돌연변이인지 확인합니다. 필요한 경우 연속 순서를 반복합니다. (그림4B,C).
- 각 유전자형에 대해 JH4 인트론에서 고유 돌연변이를 태피화하고 정량화한다(도5).
- 동일한 돌연변이가 있는 서열을 한 번만 계산합니다.
참고: PCR 또는 다른 B 셀에서 동일한 SHM 이벤트 중에 동일한 서열이 생성되었는지 확인할 수 없습니다. - WT 생식선 JH4 인트론 서열의 모든 인스턴스를 고유 시퀀스로 계산합니다(즉, 돌연변이가 없는 인스턴스).
- 동일한 돌연변이가 있는 서열을 한 번만 계산합니다.
결과
유동 세포측정
성숙한 B 세포는 친화성 성숙, 복제 팽창 및 혈장 또는 메모리 세포로 분화되는 발아 센터로 순환하여40,41,42,43,44로분화한다. 이들 GCBC는 CD45R/B220 수용체의 높은 발현과 땅콩 아글루티닌(PNA)45,46의결합을 포함한 수많은 세포 표면 마커에 의해 식별될 수 있다. 활성화된 GCBC를 분리하기 위하여는, PP 세포는 물리에리트린 (PE) 및 biotinylated-PNA에 공각된 반대로 B220 항체로 염색되었고, 그 다음에 Streptavidin이 APC-eFluor780에 공주하였다. 죽은 세포는 형광 4',6-디아미드노-2-페닐린돌(DAPI) 염료를 사용하여 제거되었으며, 이는 죽거나 죽은 세포의 핵산을 염색하는47,48. 스테인드 세포는 이후에 유동 세포측정을 통해 분석및 분류하였다. PP는 ~80% B220+ 셀49,50으로구성하였다. WT PP는 마우스 당 평균 4 x 106 셀을 함유하고 있습니다(그림3A). WT PP 세포의 약 8%는 B220+PNAHI였으며,이는 AID에서관찰된 수의절반인 -/ -(그림 3B)였다. 따라서, 0.3-0.6 x 106 B220+PNAHI GCBCs는 분류 후 얻어졌으며, 이는 JH4 인트론에서 돌연변이를 분석하기에 충분하였다.
JH4 서열 분석
JH4 인트론은 일반적인 VHJ558 패밀리 프라이머(J558FR3Fw 및 VHJ558.2)를 사용하여 중첩PCR에 의해 증폭되었으며, 그 다음으로 JH4 인트론이 프라이머 VHJ558.3 및 VHJ558.435,37을아우르는 것으로 그 뒤를 이었다. WT GCBC로부터 얻은 105개의 고유 서열 중 총 226개의 돌연변이가발견되었다(그림 5A). WT 마우스에서 의 GCBC 돌연변이 스펙트럼의 분석은 4 x10-3 돌연변이/bp의 비율로 전환 및 전이의 범위를 보였으며, 이는32,36,37,38을서열화한 총 염기 수에 의해 돌연변이된 염기의 총 수를 분할하여 계산하였다. 또한, WT GCBC로부터의 각 JH4 PCR 제품에는 1-25돌연변이(도 5B)가포함되어 있으며, 여기서 다중 돌연변이는 하나의 서열33,36에서자주 발견되었다. 만 두 돌연변이는 113 AID-/--서열(도 5A)에서확인되었다. AID-/-B 세포는 WT B 세포(p&0.05)보다 현저히 낮았던 1.66 x 10-5 돌연변이/bp를 나타내었으며, 높은 충실도 폴리머라제(5.3 x10-7 서브/베이스/두 배)의 오차율과 비교하였다51,52. 따라서 AID-/-B 세포는 이 분석에 유용한 음수 대조군으로 작용하였다.
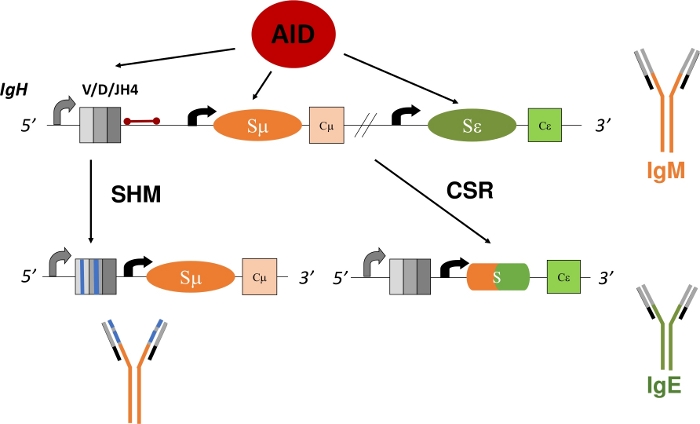
그림 1: CSR 및 SHM 도중 AID에 의해 표적으로 한 IgH 유전자 궤적 및 지구의 회로도. 빨간색 막대는 VDJH4 재배열의 3'이며 이 프로토콜에서 분석되는 580 bp JH4 인트론을 나타냅니다. CSR에서, 내성 스위치 영역(Sμ 및 Sθ)의 AID 의존적 탈환은 새로운 항체 동위형(IgM to IgE)의 삭제 재조합 및 발현을 허용하는 DSB 형성을 촉진한다. SHM 동안, V 영역(회색 상자)은 더 높은 친화성 Ig로 이어질 수 있는 돌연변이(파란색 선)를 축적한다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
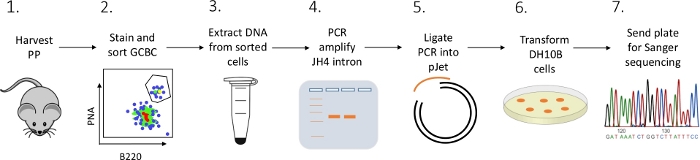
그림 2: PC로부터 격리된 GCBC에서 JH4 인트론의 SHM을 분석하는 워크플로. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
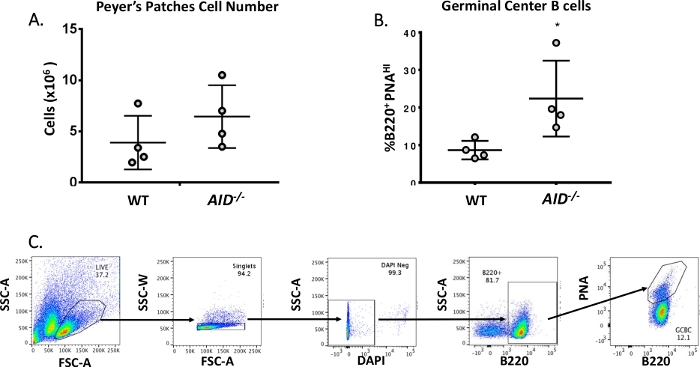
그림 3: PP GCBC의 특성화. (A)WT 및AID-/-마우스로부터의 PP 세포의 총 수(n = 4 유전자형당). 오류 막대는 평균과 표준 편차를 나타냅니다. (B)WT 및AID-/-마우스(n= 4 유전자형당 4개) PP로부터 얻은 B220+PNAHI GCB의백분율(36). 오류 막대는 학생의 t-테스트를 사용하여 평균 *p&0.05와 표준 편차를 나타냅니다. (C)대표 FACS는 PPs에서 B220+PNAHI GCB를 정렬플롯.
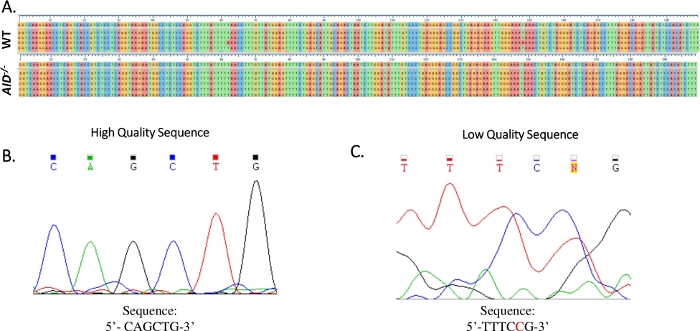
그림 4: JH4 Sanger 서열 데이터의 분석. (A)WT(위) 및AID-/---(아래) GCBCs로부터 JH4 PCR 제품의 Sanger 서열 데이터의 샘플 서열 정렬은 번호가 매겨진 진드기 마크 바로 아래에 있는 서열인 참조 게놈 서열(NG_005838)에 있다. 정렬은 클루스탈 오메가를 사용하여 생성되었습니다. (B)각 베이스에 대해 뚜렷한 피크를 표시 고품질 Sanger 서열 데이터의 일렉트로페로그램. (C)저품질 서열 데이터의 일렉트로페로그램은 모호한 봉우리와 불특정 염기(N)를 나타냈다. 빨간색으로 표시된 뉴클레오티드는 서열 텍스트 파일에 수동으로 추가로 추가되어야 합니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
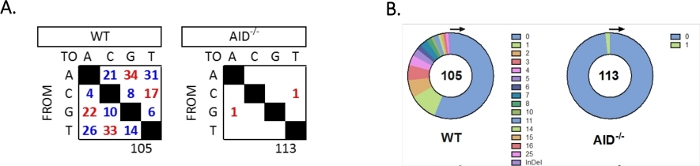
그림 5: WT 및 AID-/-GCBCs에서 JH4 인트론에서의 돌연변이 분석. (A)각 유전자형에 대한 A, C, G 및 T 염기에서의 전환 수(red) 및 트랜스버전(blue) 돌연변이가 표에 요약된다. 분석된 총 시퀀스 수는 표 아래에 표시됩니다. (B)각 유전자형에 대한 PCR 앰플리턴당 돌연변이의 수는 원형 차트에 묘사된다. 이 수치는 최외36 저작권 2020에서 수정되었습니다. 미국 면역학자 협회, Inc.는 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
| GCBC용 염색 칵테일 | 부피: 500 μL | ||
| 항체 또는 염료 | 플루오로포레 | 희석 | μL |
| B220 | PE | 1000 | 0.5 |
| 스트렙타비딘 | APC-eFluor780 | 500 | 1 |
| DAPI | N/A | 500 | 1 |
표 1: GCBC용 염색 칵테일. 지정된 희석제에서 지정된 항체 또는 염료(기울임꼴로 표시)의 칵테일은 유동 세포측정을 위해 500 μL에서 PP 세포를 염색하는 데 사용되었다.
| 보상을 위한 단일 얼룩 | 부피: 500 μL | ||
| 항체 또는 염료 | 플루오로포레 | 희석 | μL |
| B220 | PE | 1000 | 0.5 |
| B220 | APC-eFluor780 | 750 | 0.67 |
| DAPI | N/A | 500 | 1 |
표 2: 보상을 위한 단일 얼룩 컨트롤. 표시된 형광에 공인된 B220 항체는 스펙트럼 중첩을 보상하기 위해 단일 얼룩 조절에 사용되었다.
| PCR #1 | ||||
| 시약 | 음량 | 열순환기 조건 | ||
| 5x 버퍼 | 4 μL | 1 | 95°C | 3분 |
| 10mM dNTP | 2 μL | 2 | 94 °C | 30초 |
| 10 μM J558FR3Fw | 1 μL | 3 | 55 °C | 30초 |
| 10 μM VHJ558.2 | 1 μL | 4 | 72 °C | 1분 30분 |
| 높은 충실도 DNA 폴리머라제 | 0.25 μL | 사이클 2-4 9x | ||
| DNA | x(가장 집중된 시료로 표준화) | |||
| H2O | ~ 20 μL | 5 | 72 °C | 5분 |
| PCR #2 진행하기 전에 H2O에서 PCR 제품을 희석 | ||||
표 3: JH4 인트론의 중첩 된 PCR. 첫 번째 증폭 반응을 위한 PCR 구성 요소 및 열순환조건. 첫 번째 PCR 제품을 1:5물로 희석하고 두 번째 PCR에 이 희석의 1 μL을 사용합니다.
| PCR #2 | ||||
| 시약 | 음량 | 온도 사이클러 조건 #2 | ||
| 5x 버퍼 | 4 μL | 1 | 94 °C | 3분 |
| 10mM dNTP | 2 μL | 2 | 94 °C | 30초 |
| 10 μM VHJ558.3 | 1 μL | 3 | 55 °C | 30초 |
| 10 μM VHJ558.4 | 1 μL | 4 | 72 °C | 30초 |
| 높은 충실도 DNA 폴리머라제 | 0.25 μL | 사이클 2-4 21x | ||
| 희석 PCR #1 | 1 μL | |||
| H2O | ~ 20 μL | 5 | 72 °C | 5분 |
표 4: 두 번째 PCR용 PCR 구성 요소 및 열순환기 조건.
| 시약 | 음량 |
| 2x 버퍼 | 10 μL |
| 정제 된 PCR | x(가장 집중된 시료로 표준화) |
| 무딘 끝이있는 플라스미드 | 1 μL |
| T4 DNA 리개세 | 1 μL |
| H2O | ~ 20 μL |
| 16ºC에서 5분 또는 하룻밤 동안 객실 온도에서 배양 | |
표 5: 결찰 반응. 정제된 JH4 인트론 PCR 제품의 계합을 위한 성분이 플라스미드로 한다.
| FACS 버퍼 |
| 열은 사용하기 전에 1 시간 동안 56 °C에서 FBS를 비활성화합니다. 가열 불활성화 FBS의 2.5 %(v / v)와 PBS, pH 7.4 (Gibco, #10010049)를 보충합니다. 4°C에 보관하십시오. |
| DNA 추출 버퍼 (100 mM Tris pH 8.0, 0.1 M EDTA, 0.5 % (w / v) SDS) |
| 1M Tris pH 8.0, 0.5M EDTA의 100mL, 20% SDS의 12.5mL의 50mL를 추가합니다. 증류수를 500mL에 추가합니다. 실온에서 보관하십시오. |
| TE 버퍼 (10m M Tris pH 8.0, 1 mM EDTA) |
| 1 M Tris pH 8.0의 2.5mL, 0.5 M EDTA의 500 mL을 추가하십시오. 증류수를 250mL에 넣습니다. 실온에서 보관하십시오. |
표 6: 버퍼 레시피.
| 올리고뉴클레오티드 목록 | ||
| J558FR3Fw | 5'-GCCTGACATCTGACTCTCTGC-3' | |
| VHJ558.2 | 5'-CTGGACTTTCGGTGTGTG-3' | |
| VHJ558.3 | 5'-GGTCAAGGAACCTCAGTCA-3' | |
| VHJ558.4 | 5'-TCTC타카카카가액타크-3' | |
표 7: 분석에 사용되는 올리고뉴클레오티드.
보충도 1: 5.4단계 완료 후 대표적인 아가로즈 젤 이미지. JH4 인트론 중첩 PCR 제품은 1.5% 아가로즈 젤로 해결되었고 580 bp 앰플리콘이 절제되었다. WT PP는 WT PP GCBC 게놈 DNA가 제1 PCR 및 AID PP를 위한 템플릿으로 사용되었다는 것을 나타내며, AID-/-PP GCBC 게놈 DNA가 제1 PCR의 템플릿으로 사용되었다는 것을 나타낸다. ɸ 템플릿 PCR 제어를 나타내지 않으며 아가로즈 젤의 우물에 로드된 것이 없음을 나타냅니다. 마지막 차선은 100 bp DNA 사다리를 보여줍니다. 이 그림을 다운로드하려면 여기를 클릭하십시오.
토론
이종성 B 세포 집단의 IgH 및 IgL V 코딩 서열 내에서 SHM을 특성화하는 것은 각 B 셀이 V(D)J 재조합34동안 V 코딩 세그먼트를 고유하게 재구성한다는 점을 감안할 때 도전한다. 이 논문에서, 우리는 GCBCs의 JH4 intron에 있는 돌연변이를 확인하는 방법을 기술합니다. IGH 궤적에서 마지막 J 코딩 세그먼트의 3'에 위치한 JH4 인트론은 V 영역의 SHM에 대한 대리로 사용된다(도1)31,33,34,35. 이러한 JH4 인트론 돌연변이를 분류하고 특정 유전자가 돌연변이의 생산 또는 패턴에 미치는 영향을 평가하기 위해 PP GCBC는 구체적으로 분석됩니다. 이러한 세포는 장 내미생물제53에의한 만성 자극의 결과로 JH4 인트론 돌연변이를 축적한다. 더욱이, 면역되지 않은 마우스의 PP로부터의 B220+ PNAHI GCBC는 면역화된 동물로부터의 비장 GCBCC와 비교되는 돌연변이스펙트럼을갖는다(54,55). 그러나, JH4 인트론의 돌연변이는 이러한 돌연변이가 비코딩이기 때문에 Ig 선호도 성숙과 상관될 수 없다.
SHM이 Ig 선호도를 바꾸는지 여부를 결정하기 위해 마우스는 Np(4-하이드록시-3-니트로페닐라실)와 같은 항원을 통해 회귀적으로 접종되어야 하며 CGG(치킨 감마 글로불린) 또는 KLH(키홀 림펫혈청)에공인한다. 이어서, mRNA는 비장 B220+PNAHI GCBC로부터 정제되어 VH186.2 내에서 SHM을 검사할 수 있으며, NP-CGG 또는 NP-KLH 예방 접종31,57,58,59,60에따라 돌연변이되는 V 코딩 엑온은 가장 빈번하게 인식한다. VH186.2에서 류신에 대한 트립토판-33의 돌연변이는 10배59,60까지 Ig 선호도를 증가시키는 것을 특징으로 하며, 따라서 SHM 및 클로날 선택이 높은 친화성 Ig를 생성했다는 하나의 지표가 있다. ELISA에 의한 NP7 및 NP20 특이적 혈청 이그 티터를 측정하고 예방 접종 과정에서 Ig 특이적 NP7/NP20 비율을 계산하는 것도 V영역의 SHM(17,21,36)의SHM에서 인한 Ig 선호도 성숙을 문서화한다. 이 두 가지 모두 VH186.2 코딩 시퀀스 내에서 NP 특이적 Ig 선호도 성숙의 변화와 SHM을 상호 연관시키는 데 사용할 수 있습니다.
예방 접종 또는 예방 접종되지 않은 동물이 VH186.2또는 JH4 인트론의 SHM을 분석하는 데 사용되든 GCBC를 정확하게 식별해야합니다. 우리는 B220+PNAHI GCB를 분리하는 FACS 기반의 접근 방식을제시합니다. 대안적으로, GL7 항체61,62,63,64에의해 인식되는 Fas 및 비 황지방 α2-6-sialyl-LacNAc 항원은 B220+Fas+GL7+65 또는 CD19+Fas + GL7+37 +37로식별되는 GCCC를 분리하는 데 사용할 수 있습니다. GL7발현은 림프절64,65,66의활성화된 GCBC에서 PNA를 밀접하게 미러로 한다. GCBC에 특정 한 항체 마커를 사용 하 여, 염색 칵테일 형광의 흥분을 극대화 하 고 형광 방출의 스펙트럼 중복을 최소화 하는 동안 바이오 마커의 검출을 극대화 한다. 낮은 수준에서 발현된 항원은 강력한 방출형광(67)을가진 형광에 컨쥬게이트되는 항체로 검출되어야 한다. 권장 염색 프로토콜은 4개의 레이저(405nm, 488nm, 561nm, 633nm) 및 12개의 필터를 갖춘 세포 선별기의 분석에 최적화되었다. 그러나 필터 구성과 레이저 가용성은 사이토미터마다 다릅니다. 시약 및 장비 가용성에 따라 프로토콜을 개정하기 위해 독자는 추가 자원, 온라인 스펙트럼 시청자 및 출판 된 문학67,68,69,70,71,72, 73을지칭합니다. 본 원에 설명된 다중 색 염색 프로토콜은 분류된 세포 집단이 형광 방출의 부정확한 검출보다는 GCBC인지 확인하기 위해 스펙트럼 중첩의 보상을 필요로 한다. B220은 PP가 특유의 B220 음성 및 양성집단(도 3C)을가지기 때문에 설명된 FACS(표1B)에대한 유용한 염색 제어역할을 하며, 이는 스펙트럼 중첩에 대한 적절한 보상을 허용한다. 그림 3C에 제시된 게이팅 전략은 지침으로 사용해야 합니다. 유동 세포측정플롯은 염색 조건 및 사이토미터 설정에 따라 달라질 수 있습니다. 그럼에도 불구하고, 살아있는 세포의 4-10%는 B220+PNAHI 35, 52여야한다.
PP GCBC의 JH4 인트론 내의 모든 돌연변이는 관찰된 돌연변이가 PCR 또는 시퀀싱의 유물이 아니라 SHM을 진정으로 반사하도록 검증되어야 합니다. AID-/-b 세포는 이러한 세포가 SHM17,19를완료할 수 없기 때문에 다른 돌연변이 마우스 모델에서 SHM 표현형을 검사할 때 유용한 음수 대조군역할을 할 수 있다. AID-/-GCBC(1.66x10-5 돌연변이/bp)의 JH4 인트론 돌연변이율은20,21, 36,37,38,50,74의 오차율(5.3x10-7 서브/베이스/두 배)의 오차율(5.3x10-7 서브/베이스/두배)의 오차율(5.3x10-7 서브/베이스/두배)와 비교하여 DNA를 증폭시키는 데 사용되고 있다. AID-/-마우스를 사용할 수 없는 경우, 관찰된 돌연변이 패턴 및 주파수를 출판된 문헌과 비교한다. Ig V 영역은 기본 쌍 분단당 10-3-10-4 돌연변이를 축적하며, 이는 다른 유전자 loci 73,75의돌연변이 율보다 약 106배더 높다. 결과는 동물의 나이에 따라 다를 수 있습니다76. 대안적으로, 비-GCBC를 표시하는 B220+ PNALO 세포는AID-/--마우스(52)의 부재 시 음수 대조군으로 사용될 수 있다. WT GCBC의 돌연변이 주파수가 예상보다 낮으면 WT 생식선 JH4 내성 서열이 불균형적으로 표현될 수 있다. 이 경우 GCBC가 염색되어 적절하게 분류되고 PcR이 WT 생식선 JH4 인트론 오염으로부터 무료인지 확인하십시오. 또한, 일열 텍스트 데이터의 돌연변이가 시퀀싱 오류의 아티팩트가 아님을 확인하기 위해 전자프로그램의 원시 염기서열 분석 데이터를 철저히 분석해야 합니다. 예를 들어, Sanger 시퀀싱 결과가 좋지 않은 경우 시퀀스 데이터의 신뢰성을 감소시킬 수있다(도 4). Sanger 서열 데이터의 이러한 품질 관리는 JH4 인트론 돌연변이 분석의 정확성과 재현성을 증가시킬 것이다.
공개
저자는 공개 할 것이 없습니다.
감사의 말
우리는 AID-/-- 마우스에 대한 다스쿠 혼조 에게 감사드립니다. 이 작품은 소수 민족 건강 불균형에 국립 연구소에 의해 지원되었다 (5G12MD007603), 국립 암 연구소 (2U54CA132378), 일반 의학의 국립 연구소 (1SC1GM132035-01).
자료
| Name | Company | Catalog Number | Comments |
| 0.2 ml PCR 8-tube FLEX-FREE strip, attached clear flat caps, mixed | USA Scientific | 1402-4708 | |
| Ampicillin sodium salt | Fisher | BP1760-5 | |
| APC-eFluor780 anti-CD45R/B220 | eBioscience | 47-0452-80 | clone RA3-6B2 |
| BD FACSAria II | BD | 643186 | four lasers (405nm, 488nm, 561nm, 633nm) and 12 filters (PacBlue (450/50), AmCyan (502LP; 530/30), SSC (488/10), FITC (502LP; 530/30), PerCP-Cy5.5 (655LP; 695/40), PE (585/15), PE-Texas Red (600LP; 610/20), PE-Cy5 (630LP; 670/14), PE-Cy7 (735LP; 780/60), APC (660/20), Alexa700 (710LP; 730/45), APC-Cy7 (755LP; 780/60)) |
| BD slip tip 1mL syringe | Fisher | 14-823-434 | sterile |
| Biotinylated peanut agglutinin (PNA) | Vector Labs | B-1075-5 | |
| C57BL/6J mice | Jackson Laboratories | 664 | |
| Corning Falcon test tube with cell strainer snap cap | Fisher | 08-771-23 | |
| DAPI (4',6-Diamidino-2-Phenylindole, dihydrochloride) | Fisher | D1306 | 0.5 mg/ml |
| dNTP | NEB | N0447L | 10 mM |
| ElectroMAX DH10B competent cells | Fisher | 18-290-015 | |
| Falcon cell strainer 40mm | Fisher | 08-771-1 | |
| Falcon round-bottom polystyrene tubes (FACS tubes) | Fisher | 14-959-5 | |
| Falcon round-bottom polystyrene tubes (capped) | Fisher | 149591A | |
| Fetal bovine serum | R&D Systems (Atlanta Biologicals) | S11150 | |
| Gibco phosphate buffered saline PBS pH 7.4 | Fisher | 10-010-049 | |
| Glycogen | Sigma | 10901393001 | |
| Lasergene Molecular Biology (MegAlign Pro) | DNA Star | version 15 | |
| PE anti-CD45R/B220 | BD | 553090 | clone RA3-6B2 |
| Proteinase K | Fisher | BP1700-100 | |
| Q5 High-Fidelity DNA Polymerase | NEB | M0491L | |
| QIAquick Gel Extraction Kit | Qiagen | 28706 | |
| Seal-Rite 1.5mL microcentrifuge tubes | USA Scientific | 1615-5500 | |
| Streptavidin APC-eFluor 780 Conjugate | eBioscience | 47-4317-82 | |
| T4 DNA ligase | NEB | M020L | |
| Thermo Scientific CloneJET PCR Cloning Kit | ThermoFisher | FERK1231 | |
| Tissue culture plate 6 well | Fisher | 08-772-1B | sterile |
| Unlabeled anti-mouse CD16/CD32 (Fc block), BD | Fisher | BDB553142 | Clone 2.4G2 |
참고문헌
- Murphy, K., Weaver, C. Janeyway's Immunobiology. , Garland science. (2016).
- Alt, F. W., et al. VDJ recombination. Immunology Today. 13 (8), 306-314 (1992).
- Schatz, D. G., Ji, Y. Recombination centres and the orchestration of V (D) J recombination. Nature Reviews Immunology. 11 (4), 251-263 (2011).
- Oettinger, M. A., Schatz, D. G., Gorka, C., Baltimore, D. RAG-1 and RAG-2, adjacent genes that synergistically activate V (D) J recombination. Science. 248 (4962), 1517-1523 (1990).
- Berek, C., Berger, A., Apel, M. Maturation of the immune response in germinal centers. Cell. 67 (6), 1121-1129 (1991).
- Linterman, M. A., et al. Foxp3+ follicular regulatory T cells control the germinal center response. Nature Medicine. 17 (8), 975(2011).
- Shulman, Z., et al. T follicular helper cell dynamics in germinal centers. Science. 341 (6146), 673-677 (2013).
- Good-Jacobson, K. L., et al. PD-1 regulates germinal center B cell survival and the formation and affinity of long-lived plasma cells. Nature Immunology. 11 (6), 535(2010).
- Kerfoot, S. M., et al. Germinal center B cell and T follicular helper cell development initiates in the interfollicular zone. Immunity. 34 (6), 947-960 (2011).
- Chaudhuri, J., Alt, F. W. Class-switch recombination: interplay of transcription, DNA deamination and DNA repair. Nature Reviews Immunology. 4 (7), 541-552 (2004).
- Alt, F. W., Zhang, Y., Meng, F. L., Guo, C., Schwer, B. Mechanisms of programmed DNA lesions and genomic instability in the immune system. Cell. 152 (3), 417-429 (2013).
- Xu, Z., Zan, H., Pone, E. J., Mai, T., Casali, P. Immunoglobulin class-switch DNA recombination: induction, targeting and beyond. Nature Reviews Immunology. 12 (7), 517-531 (2012).
- Di Noia, J. M., Neuberger, M. S. Molecular mechanisms of antibody somatic hypermutation. Annual Reviews of Biochemistry. 76, 1-22 (2007).
- Peled, J. U., et al. The biochemistry of somatic hypermutation. Annual Review of Immunology. 26, 481-511 (2008).
- Liu, M., Schatz, D. G. Balancing AID and DNA repair during somatic hypermutation. Trends in Immunology. 30 (4), 173-181 (2009).
- Methot, S., Di Noia, J. Molecular Mechanisms of Somatic Hypermutation and Class Switch Recombination. Advances in Immunology. 133, 37-87 (2017).
- Muramatsu, M., et al. Class switch recombination and hypermutation require activation-induced cytidine deaminase (AID), a potential RNA editing enzyme. Cell. 102 (5), 553-563 (2000).
- Petersen-Mahrt, S. K., Harris, R. S., Neuberger, M. S. AID mutates E. coli suggesting a DNA deamination mechanism for antibody diversification. Nature. 418 (6893), 99(2002).
- Revy, P., et al. Activation-induced cytidine deaminase (AID) deficiency causes the autosomal recessive form of the Hyper-IgM syndrome (HIGM2). Cell. 102 (5), 565-575 (2000).
- Petersen-Mahrt, S. DNA deamination in immunity. Immunological Reviews. 203 (1), 80-97 (2005).
- Rada, C., Di Noia, J. M., Neuberger, M. S. Mismatch recognition and uracil excision provide complementary paths to both Ig switching and the A/T-focused phase of somatic mutation. Molecular Cell. 16 (2), 163-171 (2004).
- Rada, C., et al. Immunoglobulin isotype switching is inhibited and somatic hypermutation perturbed in UNG-deficient mice. Current Biology. 12 (20), 1748-1755 (2002).
- Schrader, C. E., Vardo, J., Stavnezer, J. Role for mismatch repair proteins Msh2, Mlh1, and Pms2 in immunoglobulin class switching shown by sequence analysis of recombination junctions. The Journal of Experimental Medicine. 195 (3), 367-373 (2002).
- Martin, A., et al. Msh2 ATPase activity is essential for somatic hypermutation at AT basepairs and for efficient class switch recombination. The Journal of Experimental Medicine. 198 (8), 1171-1178 (2003).
- Imai, K., et al. Human uracil-DNA glycosylase deficiency associated with profoundly impaired immunoglobulin class-switch recombination. Nature Immunology. 4 (10), 1023-1028 (2003).
- Masani, S., Han, L., Yu, K. Apurinic/apyrimidinic endonuclease 1 is the essential nuclease during immunoglobulin class switch recombination. Molecular and Cellular Biology. 33 (7), 1468-1473 (2013).
- Guikema, J. E., et al. APE1-and APE2-dependent DNA breaks in immunoglobulin class switch recombination. The Journal of Experimental Medicine. 204 (12), 3017-3026 (2007).
- Schrader, C. E., Guikema, J. E., Wu, X., Stavnezer, J. The roles of APE1, APE2, DNA polymerase β and mismatch repair in creating S region DNA breaks during antibody class switch. Philosophical Transactions of the Royal Society B: Biological Sciences. 364 (1517), 645-652 (2009).
- Roa, S., et al. MSH2/MSH6 complex promotes error-free repair of AID-induced dU: G mispairs as well as error-prone hypermutation of A: T sites. PLoS One. 5 (6), 11182(2010).
- Delbos, F., Aoufouchi, S., Faili, A., Weill, J. C., Reynaud, C. A. DNA polymerase η is the sole contributor of A/T modifications during immunoglobulin gene hypermutation in the mouse. The Journal of Experimental Medicine. 204 (1), 17-23 (2007).
- Maul, R. W., Gearhart, P. J. AID and somatic hypermutation. Advances in Immunology. 105, 159-191 (2010).
- Shen, H. M., Tanaka, A., Bozek, G., Nicolae, D., Storb, U. Somatic hypermutation and class switch recombination in Msh6-/- Ung-/- double-knockout mice. The Journal of Immunology. 177 (8), 5386-5392 (2006).
- Cheng, H. L., et al. Integrity of the AID serine-38 phosphorylation site is critical for class switch recombination and somatic hypermutation in mice. Proceedings of the National Academy of Sciences. 106 (8), 2717-2722 (2009).
- Lebecque, S. G., Gearhart, P. J. Boundaries of somatic mutation in rearranged immunoglobulin genes: 5'boundary is near the promoter, and 3'boundary is approximately 1 kb from V (D) J gene. The Journal of Experimental Medicine. 172 (6), 1717-1727 (1990).
- Jolly, C. J., Klix, N., Neuberger, M. S. Rapid methods for the analysis of immunoglobulin gene hypermutation: application to transgenic and gene targeted mice. Nucleic Acids Research. 25 (10), 1913-1919 (1997).
- Choi, J. E., Matthews, A. J., Michel, G., Vuong, B. Q. AID Phosphorylation Regulates Mismatch Repair-Dependent Class Switch Recombination and Affinity Maturation. The Journal of Immunology. 204 (1), 13-22 (2020).
- McBride, K. M., et al. Regulation of class switch recombination and somatic mutation by AID phosphorylation. The Journal of Experimental Medicine. 205 (11), 2585-2594 (2008).
- Liu, M., et al. Two levels of protection for the B cell genome during somatic hypermutation. Nature. 451 (7180), 841-845 (2008).
- Ross, M., Birbeck, M., Wills, V., Forrester, J., Davis, A. Peanut lectin binding properties of germinal centers of mouse lymphoid tissues. Nature. 284, 364-366 (1980).
- Zhang, J., MacLennan, I. C., Liu, Y. J., Lane, P. J. Is rapid proliferation in B centroblasts linked to somatic mutation in memory B cell clones. Immunology Letters. 18 (4), 297-299 (1988).
- Nieuwenhuis, P., Opstelten, D. Functional anatomy of germinal centers. American Journal of Anatomy. 170 (3), 421-435 (1984).
- Lau, A. W., Brink, R. Selection in the germinal center. Current Opinion in Immunology. 63, 29-34 (2020).
- Victora, G. D., Nussenzweig, M. C. Germinal centers. Annual Review of Immunology. 30, 429-457 (2012).
- Mesin, L., Ersching, J., Victora, G. D. Germinal center B cell dynamics. Immunity. 45 (3), 471-482 (2016).
- Reichert, R. A., Gallatin, W. M., Weissman, I. L., Butcher, E. C. Germinal center B cells lack homing receptors necessary for normal lymphocyte recirculation. The Journal of Experimental Medicine. 157 (3), 813-827 (1983).
- Rose, M., Birbeck, M., Wills, V., Forrester, J., Davis, A. Peanut lectin binding properties of germinal centers of mouse lymphoid tissues. Nature. 284, 364-366 (1980).
- Hamada, S., Fujita, S. DAPI staining improved for quantitative cytofluorometry. Histochemistry. 79 (2), 219-226 (1983).
- Otto, F. DAPI staining of fixed cells for high-resolution flow cytometry of nuclear DNA. Methods in Cell Biology. 33, 105-110 (1990).
- Butcher, E., et al. Surface phenotype of Peyer's patch germinal center cells: implications for the role of germinal centers in B cell differentiation. The Journal of Immunology. 129 (6), 2698-2707 (1982).
- Rogerson, B. J., Harris, D. P., Swain, S. L., Burgess, D. O. Germinal center B cells in Peyer's patches of aged mice exhibit a normal activation phenotype and highly mutated IgM genes. Mechanisms of Ageing and Development. 124 (2), 155-165 (2003).
- Potapov, V., Ong, J. L. Examining sources of error in PCR by single-molecule sequencing. PloS One. 12 (1), 0169774(2017).
- Gonzalez-Fernandez, A., Milstein, C. Analysis of somatic hypermutation in mouse Peyer's patches using immunoglobulin kappa light-chain transgenes. Proceedings of the National Academy of Sciences. 90 (21), 9862-9866 (1993).
- Reboldi, A., Cyster, J. G. Peyer's patches: organizing B-cell responses at the intestinal frontier. Immunological Reviews. 271 (1), 230-245 (2016).
- Betz, A. G., Rada, C., Pannell, R., Milstein, C., Neuberger, M. S. Passenger transgenes reveal intrinsic specificity of the antibody hypermutation mechanism: clustering, polarity, and specific hot spots. Proceedings of the National Academy of Sciences. 90 (6), 2385-2388 (1993).
- Rada, C., Gupta, S. K., Gherardi, E., Milstein, C. Mutation and selection during the secondary response to 2-phenyloxazolone. Proceedings of the National Academy of Sciences. 88 (13), 5508-5512 (1991).
- Heise, N., Klein, U. Somatic Hypermutation and Affinity Maturation Analysis Using the 4-Hydroxy-3-Nitrophenyl-Acetyl (NP) System. Methods in Molecular Biology. 1623, 191-208 (2017).
- Smith, F., Cumano, A., Licht, A., Pecht, I., Rajewsky, K. Low affinity of kappa chain bearing (4-hydroxy-3-nitrophenyl) acetyl (NP)-specific antibodies in the primary antibody repertoire of C57BL/6 mice may explain lambda chain dominance in primary anti-NP responses. Molecular Immunology. 22 (10), 1209-1216 (1985).
- Takahashi, Y., Dutta, P. R., Cerasoli, D. M., Kelsoe, G. In situ studies of the primary immune response to (4-hydroxy-3-nitrophenyl) acetyl. V. Affinity maturation develops in two stages of clonal selection. The Journal of Experimental Medicine. 187 (6), 885-895 (1998).
- Allen, D., Simon, T., Sablitzky, F., Rajewsky, K., Cumano, A. Antibody engineering for the analysis of affinity maturation of an anti-hapten response. The EMBO Journal. 7 (7), 1995-2001 (1988).
- Cumano, A., Rajewsky, K. Clonal recruitment and somatic mutation in the generation of immunological memory to the hapten NP. The EMBO Journal. 5 (10), 2459-2468 (1986).
- Smith, K., Nossal, G., Tarlinton, D. M. FAS is highly expressed in the germinal center but is not required for regulation of the B-cell response to antigen. Proceedings of the National Academy of Sciences. 92 (25), 11628-11632 (1995).
- Hao, Z., et al. Fas receptor expression in germinal-center B cells is essential for T and B lymphocyte homeostasis. Immunity. 29 (4), 615-627 (2008).
- Cervenak, L., Magyar, A., Boja, R., László, G. Differential expression of GL7 activation antigen on bone marrow B cell subpopulations and peripheral B cells. Immunology Letters. 78 (2), 89-96 (2001).
- Naito, Y., et al. Germinal center marker GL7 probes activation-dependent repression of N-glycolylneuraminic acid, a sialic acid species involved in the negative modulation of B-cell activation. Molecular and Cellular Biology. 27 (8), 3008-3022 (2007).
- Olson, W. J., et al. Orphan Nuclear Receptor NR2F6 Suppresses T Follicular Helper Cell Accumulation through Regulation of IL-21. Cell Reports. 28 (11), 2878-2891 (2019).
- Dorsett, Y., et al. MicroRNA-155 suppresses activation-induced cytidine deaminase-mediated Myc-Igh translocation. Immunity. 28 (5), 630-638 (2008).
- Goetz, C., Hammerbeck, C., Bonnevier, J. Flow Cytometry Basics for the Non-Expert. , Springer. (2018).
- Hawley, T. S., Herbert, D. J., Eaker, S. S., Hawley, R. G. Flow Cytometry Protocols. , Springer. (2004).
- Costa, E., et al. A new automated flow cytometry data analysis approach for the diagnostic screening of neoplastic B-cell disorders in peripheral blood samples with absolute lymphocytosis. Leukemia. 20 (7), 1221-1230 (2006).
- McKinnon, K. M. Flow cytometry: An overview. Current Protocols in Immunology. 120 (1), 1-11 (2018).
- McKinnon, K. M. Multiparameter Conventional Flow Cytometry. Methods in Molecular Biology. , 139-150 (2018).
- Lucchesi, S., et al. Computational Analysis of Multiparametric Flow Cytometric Data to Dissect B Cell Subsets in Vaccine Studies. Cytometry Part A. 97, 259-267 (2019).
- Longerich, S., Tanaka, A., Bozek, G., Nicolae, D., Storb, U. The very 5' end and the constant region of Ig genes are spared from somatic mutation because AID does not access these regions. The Journal of Experimental Medicine. 202 (10), 1443-1454 (2005).
- Retter, I., et al. Sequence and characterization of the Ig heavy chain constant and partial variable region of the mouse strain 129S1. The Journal of Immunology. 179 (4), 2419-2427 (2007).
- Shen, H. M., Peters, A., Baron, B., Zhu, X., Storb, U. Mutation of BCL-6 gene in normal B cells by the process of somatic hypermutation of Ig genes. Science. 280 (5370), 1750-1752 (1998).
- Richter, K., et al. Altered pattern of immunoglobulin hypermutation in mice deficient in Slip-GC protein. Journal of Biological Chemistry. 287 (38), 31856-31865 (2012).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유