Method Article
Microscopía de Fuerza Atómica Combinada con Espectroscopía Infrarroja como Herramienta para Sondear Química de Bacteria Única
En este artículo
Resumen
La microscopía de fuerza atómica-espectroscopía infrarroja (AFM-IR) proporciona una poderosa plataforma para estudios bacterianos, lo que permite lograr una resolución a nanoescala. Tanto el mapeo de los cambios subcelulares (por ejemplo, en la división celular) como los estudios comparativos de la composición química (por ejemplo, derivados de la resistencia a los medicamentos) se pueden realizar a nivel de una sola célula en bacterias.
Resumen
La Microscopía de Fuerza Atómica-Espectroscopía Infrarroja (AFM-IR) es una novedosa técnica combinatoria, que permite la caracterización simultánea de las propiedades físicas y la composición química de la muestra con resolución a nanoescala. Al combinar AFM con IR, se supera la limitación de resolución espacial de IR convencional, lo que permite lograr una resolución de 20-100 nm. Esto abre la puerta a una amplia gama de nuevas aplicaciones de IR para sondear muestras más pequeñas que varios micrómetros, previamente inalcanzables por medio de la microscopía IR convencional. AFM-IR es eminentemente adecuado para la investigación bacteriana, proporcionando información espectral y espacial a nivel de célula única e intracelular. Las crecientes preocupaciones de salud mundial y la predicción futura desfavorable con respecto a las infecciones bacterianas, y especialmente, el rápido desarrollo de la resistencia a los antimicrobianos, han creado una necesidad urgente de una herramienta de investigación capaz de sondear fenotípicamente a nivel unicelular y subcelular. AFM-IR ofrece el potencial de abordar esta necesidad, al permitir la caracterización detallada de la composición química de una sola bacteria. Aquí, proporcionamos un protocolo completo para la preparación de muestras y la adquisición de datos de espectros individuales y modalidad de mapeo, para la aplicación de AFM-IR a estudios bacterianos.
Introducción
Las bacterias son organismos procariotas unicelulares, que ocurren en varias formas y tamaños, típicamente en el rango de varios cientos de nanómetros a micrómetros. Existen en una variedad de hábitats y son esenciales para la existencia de la vida. Dentro del cuerpo humano, la mayoría de las bacterias presentes en el intestino son inofensivas y muchas son de hecho beneficiosas1. Sin embargo, varias especies bacterianas son patógenas y causan una variedad de enfermedades infecciosas. Las infecciones bacterianas pueden conducir al desarrollo de sepsis y shock séptico: una afección potencialmente mortal, resultante de la respuesta del cuerpo a una infección2. La sepsis es una importante amenaza para la salud a nivel mundial, con una alta prevalencia en todo el mundo y tasas de mortalidad graves. Solo en 2017, se registraron aproximadamente 50 millones de casos de sepsis en todo el mundo, de los que 11 millones resultaron en la muerte (aproximadamente el 20%)2. Además, se demostró que una disminución en las posibilidades de supervivencia del paciente, debido al retraso en la terapia, ocurre de manera cada hora3,4.
Las infecciones bacterianas se tratan con antibióticos. La gravedad de las posibles consecuencias de las infecciones bacterianas del torrente sanguíneo (BSI), junto con una clara importancia del inicio rápido de la terapia antimicrobiana, provocan la necesidad de la administración inmediata de antibióticos. Sin embargo, como los enfoques diagnósticos actuales utilizados en la práctica clínica (por ejemplo, el cultivo de sangre) requieren un tiempo relativamente largo, la administración de antibióticos a menudo ocurre antes del diagnóstico positivo de BSI5. Este factor conduce a un uso excesivo extensivo de antibióticos, lo que, junto con el uso excesivo de antibióticos en otros sectores como la agricultura, crea una severa presión evolutiva hacia el desarrollo de resistencia a los antimicrobianos (AMR)6,7. La TMM es actualmente uno de los problemas de salud mundial más apremiantes7,8 y, para 2050, se prevé que se convierta en la principal causa de muerte9. El desarrollo de resistencia, junto con la propagación de cepas de AMR está ocurriendo a un ritmo alarmante7,8,9 y supera, con creces, la tasa de descubrimiento de nuevos antibióticos10. Nuevos fenotipos resistentes están surgiendo continuamente en todo el mundo, mientras que
la investigación dedicada a comprender los cambios relacionados con la TMM es a menudo lenta y limitada por los enfoques disponibles11. Además, los métodos comúnmente utilizados, como la reacción en cadena de la polimerasa (PCR) y la secuenciación de genes completos (WGS), se centran solo en los cambios genotípicos. Estos no son suficientes para revelar los mecanismos de resistencia11,lo que provoca una necesidad urgente de una herramienta de investigación que permita comprender la composición química de las bacterias.
La espectroscopia infrarroja (IR) proporciona una caracterización molecular de la muestra y, por lo tanto, es un candidato prometedor para el sondeo bacteriano fenotípico. Desde sus primeras aplicaciones12,una gran magnitud de ejemplos de su uso se demostró en la literatura13,14. Estos incluyen la identificación fenotípica de bacterias en el género15,la especie16y el nivel de la cepa17,18. Sin embargo, la resolución espacial de la RI convencional está restringida a varias micras debido al límite de resolución espacial de difracción de longitud de onda19. Dado que el tamaño de la mayoría de las bacterias se encuentra por debajo de ese límite (por ejemplo, Staphylococcus aureus ≈ de 400 nm de diámetro), la RI convencional no es aplicable para sondear a nivel unicelular o intracelular.
La limitación de la resolución espacial se superó recientemente mediante la combinación de la espectroscopia IR con la microscopía de fuerza atómica (AFM-IR). En este caso, la absorción IR se detecta indirectamente, a través de la expansión térmica del material19,20,21,22. En resumen, la absorción de la radiación IR resulta en un aumento de la temperatura local. Esto se puede medir directamente23 o a través de la medición de la oscilación de la sonda en voladizo AFM, resultante del impulso de fuerza creado por la absorción IR20,21. La técnica combinatoria AFM-IR permite alcanzar una resolución espacial que se acerca a los 20 nm, proporcionando información simultánea sobre las propiedades físicas locales de una muestra (AFM) y su composición química (AFM-IR). Es posible la recopilación de ambos espectros individuales de puntos seleccionados y el mapeo de la intensidad de los valores de número de onda seleccionados dentro de un área elegida.
Considerando la resolución espacial alcanzable de AFM-IR, es evidente que la técnica abre la posibilidad de sondeo químico/fenotípico de células de bacterias individuales y su composición intracelular24. Hasta ahora, varios ejemplos de la aplicación de AFM-IR para bacterias individuales se demostraron en la literatura19,20,21,22,25,26,27,28. Estos implican análisis espectral único19,21,22 y mapeo a nivel subcelular19,22,25,26,27,28. Por ejemplo, se ha descrito la capacidad de detectar vesículas lipídicas intracelulares27 y virus28 dentro de una sola bacteria. Estos resultados demuestran la utilidad de AFM-IR para estudios a nanoescala de bacterias individuales y patógenos clínicamente relevantes19.
Por lo tanto, presentamos un método de preparación y recolección de muestras para datos AFM-IR de muestras bacterianas multicapa, monocapa y unicelular. El protocolo aquí descrito se aplicó para estudiar diferentes especies de bacterias22 y los cambios en su composición química. En particular, se investigó el desarrollo in vivo de resistencia a la vancomicina y no susceptibilidad a la daptomicina en pares clínicos de S. aureus19. Tanto la resistencia intermitente a la vancomicina como la no susceptibilidad a la daptomicina en S. aureus (VISA y DpR) surgieron relativamente recientemente, tras el aumento del uso e introducción de estos antibióticos en las clínicas, constituyendo un problema médico significativo. Además, en particular, el mecanismo de no susceptibilidad a la daptomicina sigue siendo difícil de alcanzar, lo que impide el desarrollo de fármacos alternativos19,29. El protocolo presentado se centra en la provisión de espectros AFM-IR confiables de bacterias individuales, que pueden analizarse más a fondo utilizando una variedad de enfoques quimiométricos, de acuerdo con los objetivos experimentales. Además, incluye el enfoque de mapeo, que es aplicable para estudios intracelulares.
Protocolo
Todo el trabajo realizado con bacterias patógenas debe llevarse a cabo con las medidas de seguridad adecuadas. Estos incluyen trabajar en un laboratorio con un nivel de bioseguridad adecuado y en una cabina de bioseguridad (PC2), así como una descontaminación cuidadosa del área de trabajo con un desinfectante adecuado, por ejemplo, solución de etanol al 80%. Se debe usar el EPP apropiado todo el tiempo.
1. Preparación de disolventes y materiales
- Disolventes: Utilice agua ultrapura como disolvente. Use agua purificada, en autoclave antes del experimento para evitar cualquier posible contaminación cruzada.
- Sustrato: Utilice cualquiera de estos sustratos para AFM-IR, por ejemplo, ZnSe, CaF2,BaF2,etc. Dado que AFM-IR es, en principio, una técnica no destructiva, se puede aplicar una variedad de otras herramientas de investigación a la misma muestra después del análisis AFM-IR. Por ejemplo, la correlación de los resultados con la espectroscopia Raman se puede realizar si se utilizan diapositivas De Grado RamanCaF 2 o BaF2.
- Use viales de vidrio en lugar de tubos de plástico, ya que el plástico puede contaminar la muestra.
2. Preparación de muestras para AFM-IR
- Crecimiento/incubación de la muestra
- Cultive bacterias en medios líquidos o en placas sólidas. Seleccione el tipo de medio, las condiciones de crecimiento (por ejemplo, temperatura, disponibilidad de oxígeno) y el tiempo de crecimiento de acuerdo con los requisitos específicos de las especies de bacterias bajo investigación. Por ejemplo, para S. aureus se pueden utilizar placas de agar de infusión cardíaca (HI), con crecimiento durante 16 h a 37 ° C en condiciones aeróbicas.
NOTA: Para lograr los mejores resultados, el crecimiento / incubación debe producir suficientes bacterias que permitan la recolección de un micro-pellet de muestra. El número específico de unidades formadoras de colonias o células bacterianas depende del tipo y tamaño de la bacteria.
- Cultive bacterias en medios líquidos o en placas sólidas. Seleccione el tipo de medio, las condiciones de crecimiento (por ejemplo, temperatura, disponibilidad de oxígeno) y el tiempo de crecimiento de acuerdo con los requisitos específicos de las especies de bacterias bajo investigación. Por ejemplo, para S. aureus se pueden utilizar placas de agar de infusión cardíaca (HI), con crecimiento durante 16 h a 37 ° C en condiciones aeróbicas.
- Deposición de muestras
- Usando un lazo estéril, recoja cuidadosamente las bacterias de las colonias en la placa de agar y transfiéralas a un tubo de vidrio. Recolecte bacterias solo de la parte superior de las colonias. Si recolecta muestras de un cultivo líquido, usando una pipeta, transfiera aproximadamente 1 ml de la suspensión bacteriana a un tubo de vidrio. El volumen se puede modificar dependiendo de la carga bacteriana.
NOTA: Es importante intentar no recolectar (o minimizar tanto como sea posible la recolección de) ningún medio de debajo de la colonia. Los pasos posteriores de la preparación de la muestra tienen como objetivo eliminar cualquier residuo potencial de medios. La minimización del medio potencial residual desde el principio permite la adquisición espectral de datos de células bacterianas purificadas. Los pasos 2.2.2 y 2.2.3 se aplican a las muestras preparadas a partir de placas de agar. Para las muestras preparadas a partir de medios líquidos, vaya al paso 2.2.4. - Agregue 1 ml de agua ultrapura al tubo. Vórtice hasta que el gránulo bacteriano recolectado ya no sea visible en la parte inferior del tubo (generalmente 1-2 min).
- Estimar la turbidez aproximada de la solución utilizando, por ejemplo, los estándares de McFarland mediante la comparación visual30 entre la solución preparada y los estándares de McFarland. Si la turbidez de la suspensión bacteriana parece ser muy baja, agregue más bacterias de la placa usando un asa estéril y un vórtice nuevamente. Repita hasta que la turbidez aproximada de la solución sea comparable a los estándares mcFarland 0.5 y 1. Esto generalmente producirá una buena cantidad de pellet bacteriano.
- Centrifugar la suspensión bacteriana a 3.000 x g durante 5 min para obtener un pellet.
NOTA: Los parámetros de centrifugación se pueden modificar para obtener pellet bacteriano. Se debe tener precaución si aumenta la fuerza g, para no inducir la rotura de bacterias (especialmente en el caso de bacterias Gram-negativas). - Con una pipeta, retire suavemente el sobrenadante de la parte superior del pellet. Agregue 1 ml de agua ultrapura al tubo y al vórtice para volver a suspender el pellet. Posteriormente, centrifugar la muestra como se hizo en el paso 2.2.4.
- Repita el procedimiento de lavado (pasos 2.2.2 y 2.2.4) al menos tres veces. En caso de recolección de la muestra inicial de medios líquidos, repita el procedimiento al menos cuatro veces (extracción de medios seguida de tres lavados).
- Después del lavado final, retire el sobrenadante, agregue agua ultrapura y vórtice durante al menos 2 minutos. Posteriormente, deposite 5 μL de la muestra sobre el sustrato (por ejemplo, CaF2de grado Raman).
- Si el espesor deseado de la muestra es una multicapa de bacterias, deje que la muestra se seque al aire.
- Si el espesor deseado es monocapa o bacterias individuales, inmediatamente después de depositar la muestra (paso 2.2.7) agregue entre 20 y 100 μL de agua ultrapura y mezcle suavemente con una punta de pipeta. Dejar secar al aire.
NOTA: El volumen exacto de agua puede variar entre experimentos, ya que depende de muchos factores (por ejemplo, el tamaño del organismo, la densidad del pellet, etc.) y, por lo tanto, se determina mejor empíricamente. La preparación de una serie de muestras con diferentes volúmenes de agua ultrapura agregada permite seleccionar una muestra con el espesor / densidad deseada de bacterias. El grosor / densidad de las bacterias se puede visualizar fácilmente a través de AFM en las etapas posteriores. Ejemplos de imágenes AFM de muestras monocapa y de una sola célula se muestran en la Figura 1A-H. - Monte el sustrato en un disco de muestra de metal AFM con cinta adhesiva de doble cara.
- Usando un lazo estéril, recoja cuidadosamente las bacterias de las colonias en la placa de agar y transfiéralas a un tubo de vidrio. Recolecte bacterias solo de la parte superior de las colonias. Si recolecta muestras de un cultivo líquido, usando una pipeta, transfiera aproximadamente 1 ml de la suspensión bacteriana a un tubo de vidrio. El volumen se puede modificar dependiendo de la carga bacteriana.
3. Preparación del instrumento
NOTA: Los procedimientos instrumentales descritos aquí son para el instrumento enumerado en la Tabla de Materiales. El procedimiento instrumental detallado puede diferir ligeramente del descrito aquí si se utiliza un modelo más nuevo del instrumento AFM-IR.
- Encienda e inicialice el instrumento pulsando el botón Inicializar. Asegúrese de que el obturador láser esté en la posición Abierta para la prueba láser.
- Si se configura un sistema de purga, purgue el instrumento con N2 encendiendo el flujo de N2. Ajuste la purga de nitrógeno para lograr un nivel de humedad estable (por ejemplo, 20%). Asegúrese de que la humedad no fluctúe durante las mediciones y entre la recolección de datos de fondo y de muestra. Se recomienda permitir aproximadamente 20 minutos para que los niveles de humedad se estabilicen.
- Cargue la muestra en la cámara de muestra pulsando el botón Cargar. La carga de muestras se realiza a través del asistente de software. Mientras opera el asistente de software, primero concéntrese en la punta, use flechas para mover la etapa del microscopio en la dirección Z y haga clic en Siguiente. En segundo lugar, ajuste el punto de recopilación de datos, utilizando flechas que guían el movimiento en el plano y alinee el láser AFM y el detector AFM utilizando las perillas en la parte superior del cabezal AFM. Posteriormente, concéntrese en la superficie de la muestra moviendo la etapa del microscopio en dirección Z.
NOTA: En el manual de software31se proporcionan ilustraciones detalladas de cada paso de la carga de muestras. Centrarse en la muestra debe llevarse a cabo con cuidado. Cuando se acerque a la superficie de la muestra en la dirección Z, utilice una velocidad de motor lenta. - Acérquese a la muestra sin participar haciendo clic en el botón Enfoque.
4. Recopilación de datos
- Fondo
- Antes de la adquisición de datos, recopile los antecedentes. Para la recolección de fondo, asegúrese de que el obturador láser esté en la posición Abierta. Seleccione el rango espectral y la resolución (dependiendo del objetivo del análisis) y el número de exploraciones y el número de medias cruzadas de fondo. Por lo general, se recomienda que sean altos (por ejemplo, 1024 exploraciones y 3 como promedios).
NOTA: En general, se recomienda una resolución espectral de4 cm -1 u 8 cm-1 y rangos espectrales de3.200 cm -1–2.800 cm-1 y 1.800 cm-1–900 cm-1. - Después de la adquisición del fondo, guarde el archivo de fondo. El archivo no se almacena automáticamente. Cambie la posición del obturador láser a Cerrar.
- Antes de la adquisición de datos, recopile los antecedentes. Para la recolección de fondo, asegúrese de que el obturador láser esté en la posición Abierta. Seleccione el rango espectral y la resolución (dependiendo del objetivo del análisis) y el número de exploraciones y el número de medias cruzadas de fondo. Por lo general, se recomienda que sean altos (por ejemplo, 1024 exploraciones y 3 como promedios).
- Muestra – espectros únicos
- Pulse el botón Engage para activar la muestra. El sistema comenzará a acercarse a la superficie de la muestra, hasta que se detecte el contacto directo.
NOTA: El punto de set utilizado en este trabajo oscilaba entre 0,15 y 2 V y las ganancias de retroalimentación (ganancia I y ganancia P) normalmente se establecerían en 3 y 10. Las sondas de contacto NIR2 se utilizan comúnmente con el sistema nanoIR2 (modelo: PR-EX-nIR2-10, frecuencia de resonancia (kHz): 13 +/−4 kHz, constante de resorte (N/m): 0.07−0.4 Nm-1). - Recopile una imagen AFM para visualizar la superficie. En primera instancia, escanee un área más grande (por ejemplo, 50 x 50 μm) con una resolución espacial más baja (por ejemplo, 200 x 200 puntos)(Figura 1I).
NOTA: Los datos de AFM-IR siempre se recopilan en modo de contacto, sin embargo, los datos de AFM se pueden recopilar en modo de contacto o de toque. - A partir de la imagen de altura/deflexión AFM, seleccione un área de interés específica y vuelva a obtener una imagen con mayor resolución espacial(Figura 1J–K). Asegúrese de que la velocidad de recopilación de datos sea adecuada, con un movimiento lento de la punta (por ejemplo, velocidad de escaneo de 0,2 a 0,4 Hz).
- Seleccione el punto de medición (por ejemplo, una sola bacteria) y mueva la punta al punto.
- Alinee el láser IR. Para este propósito, use un número de onda en el que la muestra se absorberá. Para materiales biológicos esto puede ser, por ejemplo, amida I (1655 cm-1). Asegúrese de que el filtro de paso de banda esté desactivado y haga clic en Iniciar IR. El gráfico derecho en el medidor nanoIR (FFT de la deflexión mostrada como amplitud vs. frecuencia) debe mostrar al menos un pico claro y el gráfico izquierdo (deflexión vs. tiempo) debe tener una forma de onda periódica. Si este no es el caso, proceda a optimizar los puntos IR.
NOTA: Incluso si la transformada rápida de Fourier (FFT) y la deflexión muestran el perfil esperado, se recomienda realizar la optimización de los puntos IR para al menos varios números de onda, donde se esperan bandas. - Optimice los puntos calientes para la recopilación de datos IR utilizando los valores de número de onda seleccionados. Puede ser útil utilizar un espectro IR convencional de las bacterias (por ejemplo, espectro ATR de pellet bacteriano) para identificar las posiciones de las bandas y usarlas para optimizar los puntos calientes. Seleccione varios valores de número de onda (por ejemplo, 8–10) de varias regiones espectrales.
NOTA: Si el espectro IR convencional de bacterias de interés no se pudo recopilar antes de la recopilación de datos AFM-IR, los espectros bacterianos disponibles en la literatura se pueden utilizar como una guía aproximada. El resultado de la optimización de un punto IR es una imagen, que presenta un mapa de la señal de magnitud FFT en cada ubicación x e y. La ubicación con la señal más grande se selecciona automáticamente. Ejemplos de tales imágenes se dan en el manual de software31. - Después de optimizar los puntos IR para los valores de número de onda seleccionados, defina los parámetros de la recopilación de datos espectrales: región espectral, resolución espectral, número de escaneos y potencia aplicada e indíquelos en las ventanas apropiadas en el software. La resolución espectral debe coincidir con la resolución de fondo y la región espectral debe estar dentro de la región espectral para la que se recopiló el fondo.
NOTA: Un conjunto inicial genérico de parámetros podría ser: rango espectral: 3200 cm-1–2800 cm-1 y 1800 cm-1–900 cm-1, resolución espectral: 4 cm-1 u 8 cm-1, número de escaneos: 512–2048. - Si es necesario, ajuste la potencia del láser dependiendo de la señal. En general, los valores entre el 8% y el 10% de la potencia del láser deben ser suficientes para una buena calidad de la señal. Los valores más altos se pueden usar con precaución, ya que pueden provocar daños en la muestra.
NOTA: El porcentaje de potencia del láser puede variar según el tipo de láser IR. Los valores porcentuales dados aquí son para el láser OPO. - Haga clic en Adquirir para recopilar el espectro AFM-IR.
- Vuelva a recopilar los datos de AFM de la misma área después de la recopilación del espectro AFM-IR. Esto es muy recomendable, ya que revelará cualquier deriva potencial y / o influencia destructiva en la muestra.
- Si el espectro AFM-IR es satisfactorio y no se observa ninguna influencia destructiva en la muestra, proceda con la recopilación de datos. Si es necesario, defina una serie de puntos para la recopilación de datos mediante la opción Matriz y la altura de AFM recopilada o la imagen de deflexión. Esta opción permite recoger espectros consecutivamente de cada punto con los mismos parámetros espectrales definidos para un solo espectro.
- Si la imagen AFM recolectada después de la recolección del espectro AFM-IR revela una influencia destructiva en la muestra (típicamente un punto quemado), reduzca la potencia; seleccione un punto diferente y repita los pasos 4.3.8–4.3.11.
- Si la señal en el espectro AFM-IR no es satisfactoria, compruebe la exactitud de la optimización de los puntos IR (paso 4.3.6). Si es correcto, aumente ligeramente la potencia del láser y repita los pasos 4.3.7–4.3.11. Esto se puede repetir hasta que se logre una señal satisfactoria.
- Pulse el botón Engage para activar la muestra. El sistema comenzará a acercarse a la superficie de la muestra, hasta que se detecte el contacto directo.
- Muestra– enfoque de imagen
NOTA: Se recomienda encarecidamente registrar un solo espectro AFM-IR de la bacteria antes de recopilar una imagen de distribución de intensidad para un valor de número de onda seleccionado.- Grabe una imagen AFM del área de muestra elegida. Para hacer esto, primero recopile una imagen AFM de un área más grande con una resolución espacial más baja (por ejemplo, 50 x 50 μm, 200 x 200 puntos), luego seleccione una región de interés y recopile una imagen AFM con mayor resolución espacial (como se ilustra en la Figura 1I-K).
- Seleccione los valores de número de onda para las imágenes AFM-IR.
- Asegúrese de que el punto IR del láser esté optimizado para los valores de número de onda seleccionados (paso 4.3.6). Si el punto IR no está optimizado para algunos números de onda (sin un máximo claro), optimítelo para ellos.
- Defina los parámetros del área fotografiada: el ancho y el alto, el número de puntos de datos en dirección X e Y.
NOTA: Si se aplica la selección consecutiva de puntos de las imágenes AFM anteriores (como se demuestra en la Figura 1I-K),los campos de ancho y alto se rellenarán automáticamente, al marcar el área. - Defina los parámetros de adquisición de señal espectral: la longitud de onda, el número de escaneos y la potencia del láser.
NOTA: El número de escaneos debe mantenerse dentro de lo razonable. 64 o 32 escaneos generalmente permitirán una cantidad suficiente de señal. - Defina los parámetros del movimiento de la punta AFM haciendo clic en la velocidad de escaneo. Cuanto mayor sea el número de escaneos en el paso anterior y el número de puntos de datos en la dirección X, más lentos serán los movimientos de la punta. La falta de ajuste entre estos parámetros resultará en un movimiento demasiado rápido de la punta, lo que impedirá la adquisición real del número definido de escaneos de cada punto.
NOTA: Por ejemplo, para una colección adecuada de señal IR con 64 co-adiciones y 200 puntos, establezca la velocidad de escaneo como 0.07 kHz. - Asegúrese de que la casilla Habilitar imágenes IR esté marcada.
- Comience a obtener imágenes. AFM-IR de la intensidad de la señal en el número de onda seleccionado se recopilará simultáneamente con los datos AFM de esa área.
NOTA: Cuando se utiliza el láser OPO, es posible recopilar adicionalmente simultáneamente la imagen de frecuencia máxima de resonancia de contacto. Esto se puede utilizar para obtener información sobre la rigidez relativa de la muestra en diferentes lugares. - Utilice la ventana Secuencia de captura para establecer una colección consecutiva de datos AFM-IR de la misma área con los mismos parámetros, pero para diferentes valores de número de onda. Para ello, abra la ventana Secuencia de captura, escriba cada número de onda y defina la potencia láser aplicada (para cada número de onda).
- Exportar los datos recopilados (AFM y AFM-IR, espectros individuales e imágenes) a varios formatos y analizarlos utilizando métodos adecuados para objetivos de investigación específicos.
Resultados
El protocolo descrito permite obtener una gama de tipos de distribuciones celulares de bacterias en el sustrato, dependiendo de la concentración inicial de la muestra y la cantidad de agua añadida. La Figura 1 ilustra los ejemplos de imágenes AFM (altura y deflexión) registradas a partir de monocapas y muestras unicelulares preparadas utilizando el protocolo descrito de bacterias Gram-positivas (S. aureus) y Gram-negativas (Escherichia coli).
El protocolo descrito aquí se puede utilizar para imágenes AFM-IR de estructuras intra y extracelulares de bacterias individuales. Un ejemplo de esta aplicación se muestra en la Figura 2,que muestra los resultados del monitoreo de los cambios químicos localizados espacialmente que ocurren durante la división de una célula de S. aureus. Aunque el secado al aire se considera comúnmente como un enfoque de fijación para la preparación bacteriana, las bacterias, por naturaleza, muestran una resistencia muy alta a factores externos como la temperatura y se informó que sobrevivieron a la deshidratación32. Los resultados presentados aquí fueron adquiridos a partir de una muestra seca al aire. La formación de un tabique, que ocurre antes de la división celular, fue observada y monitoreada a través de imágenes AFM(Figura 2A-D)mediante la recolección de 12 imágenes de la misma área consecutivamente (recolección de una sola imagen ≈ 20 min). La Figura 2A-D muestra 4 imágenes AFM seleccionadas, con un tiempo entre la recopilación de cada imagen de aproximadamente 40 min. La estructura formada (tabique) es de 45 nm de altura. El tabique formado es claramente visible en las imágenes de altura y deflexión de AFM(Figura 2E-F). Los espectros AFM-IR registrados desde el área celular y del tabique(Figura 2G,puntos de origen marcados en la Figura 2F)se normalizaron a la banda de amida I antes de la comparación, para minimizar la influencia de la variación del grosor de la muestra entre los puntos de recolección de datos. El espectro AFM-IR del tabique se caracteriza por una mayor intensidad relativa de bandas a 1240 y 1090 cm-1 en comparación con el espectro AFM-IR recogido del área celular. Estos se atribuyen a los grupos de carbohidratos y fosfodiéster de los componentes de la pared celular (incluyendo, por ejemplo, peptidoglicano y ácido teicoico)22.
El protocolo descrito también se puede utilizar para la comparación de un solo espectro entre un número de muestras diferentes. Un ejemplo de esta aplicación junto con los resultados se muestra en la Figura 3 y la Figura 4. El objetivo del estudio es determinar los cambios químicos que se producen como resultado del desarrollo in vivo de resistencia intermitente a la vancomicina en S. aureus (VISA). Para ello, se recogieron pares clínicos de muestras de los pacientes, con la cepa madre aislada al ingreso en el hospital y antes de la terapia antibiótica (S. aureussusceptible a la vancomicina, VSSA) y la cepa hija aislada del mismo paciente después del ingreso de antibióticos y el fracaso clínico. Las muestras se cultivaron en medio de agar y se prepararon de acuerdo con el protocolo(Figura 3A-B). Los espectros AFM-IR se recolectaron de múltiples bacterias individuales (y múltiples muestras) para VSSA y VISA y posteriormente se analizaron utilizando varios enfoques quimiométricos(Figura 3C).
No se observaron diferencias morfológicas entre las células VSSA y VISA(Figura 4A-C). Sin embargo, los espectros AFM-IR(Figura 4D,F)y sus segundos derivados(Figura 4E,G)demostraron una clara diferencia en la composición química entre cepas resistentes y susceptibles. La intensidad relativa de las bandas asociadas con los grupos de carbohidratos y fosfodiéster de los componentes de la pared celular (en particular, la banda a 1088 cm-1)aumentó claramente en la cepa resistente, en comparación con la contraparte susceptible. Cabe destacar el hecho de que todos los espectros recodificados (VISA: 81, VSSA: 88) muestran una pequeña desviación estándar. Esto demuestra una buena reproducibilidad de los datos registrados de varias muestras preparadas a partir de la misma cepa, ya que no fue posible discriminar entre los espectros registrados a partir de diferentes muestras de la misma cepa. Las diferencias observadas indicaron un aumento del grosor de la pared celular en cepas resistentes, en comparación con la contraparte susceptible, que se mantiene en concordancia con otros informes de la literatura33,34.

Figura 1: Imágenes representativas de AFM de varias muestras bacterianas para mediciones de AFM-IR. Dependiendo de la dilución sobre el sustrato, el protocolo permite obtener multicapas y monocapas de bacterias, así como muestras unicelulares. Imágenes representativas de AFM de: (A–D) monocapa y (E–H) muestra unicelular para (A,B,E,F) Gram-positiva (S. aureus) y (C,D,G,H) Gram negativa (E. coli). (A, C, E, G) muestran imágenes de altura y (B, D, F, H) muestran las imágenes de deflexión correspondientes. Tamaño de las áreas fotografiadas: (A-D,G,H) 20 x 20 μm, (E,F) 50 x 50 μm. (I-K) selección consecutiva de un área para mapeo AFM-IR. Esto se logra utilizando imágenes AFM con resolución espacial creciente en el ejemplo de una sola célula de S. aureus. Cada imagen se recoge mediante muestreo de 200 x 200 puntos, con una resolución espacial creciente debido a la reducción del tamaño del área fotografiada. Tamaño de las áreas fotografiadas: (I) 40 x 40 μm, (J) 20 x 20 μm y (K) 2,24 x 2,24 μm. El cuadrado negro en (I) marca el área fotografiada en (J). El cuadrado negro en (J) marca el área fotografiada en (K). Haga clic aquí para ver una versión más grande de esta figura.
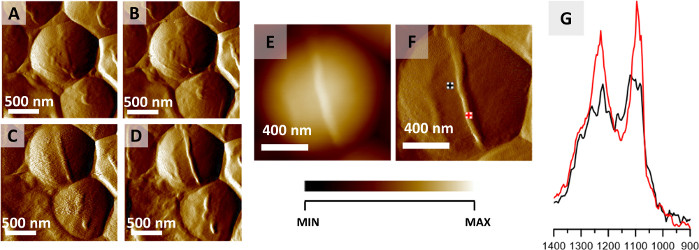
Figura 2: Monitoreo de la división celular de S. aureus a través de AFM-IR. (A-D) Imágenes AFM de la célula de S. aureus que muestran la formación del tabique que precede a la división celular. Tamaño del área fotografiada: 2 x 2 μm. Las imágenes se seleccionaron de una serie más grande (12 imágenes grabadas cada 20 min) y representan datos registrados cada 40 min.(E-F)AFM altura e imagen de deflexión registrada al final de la formación del tabique celular con puntos marcados de recolección de espectros AFM-IR. Tamaño del área fotografiada 1,17 x 1,15 μm. La altura de la estructura recién formada es de 45 nm. (G) Espectros AFM-IR registrados desde el área celular (negro) y el área del tabique (rojo) (marcado en (F)), en el rango 1400–900 cm-1. Ambos espectros se normalizaron a la banda de amida I y demuestran un aumento en la intensidad relativa de los componentes de la pared celular del tabique. Esta cifra ha sido modificada a partir de K. Kochan et al.22. Haga clic aquí para ver una versión más grande de esta figura.
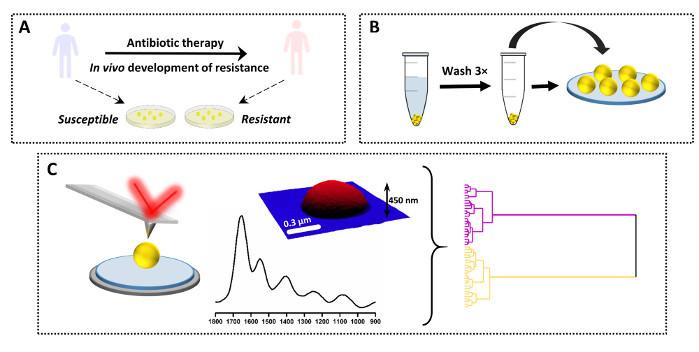
Figura 3: Una visión general del diseño experimental para el estudio AFM-IR de la resistencia a los antimicrobianos. (A) Origen de la muestra y preparación inicial: la cepa parental susceptible se recogió de un paciente antes de la terapia con antibióticos y la cepa resistente a la hija se obtuvo del mismo paciente después de la terapia con antibióticos y el fracaso clínico (desarrollo de resistencia in vivo). Las bacterias se aislaron y cultivaron en agar Heart Infusion (HI) durante 16 h a 37 °C. (B) Preparación posterior de la muestra para AFM-IR, incluida la recogida de la muestra seguida del lavado de la paleta bacteriana (3×) y la deposición de la muestra. (C) Recopilación y análisis de datos AFM-IR: altura AFM y espectro AFM-IR (1800–900 cm-1). Tamaño del área de la imagen AFM: 1,7 x 1,4 μm. El espectro AFM-IR se recogió de la mitad de la célula. Los datos se analizaron posteriormente utilizando enfoques quimiométricos, incluido el análisis de conglomerados jerárquicos. Esta cifra ha sido modificada a partir de K. Kochan et al.19. Haga clic aquí para ver una versión más grande de esta figura.
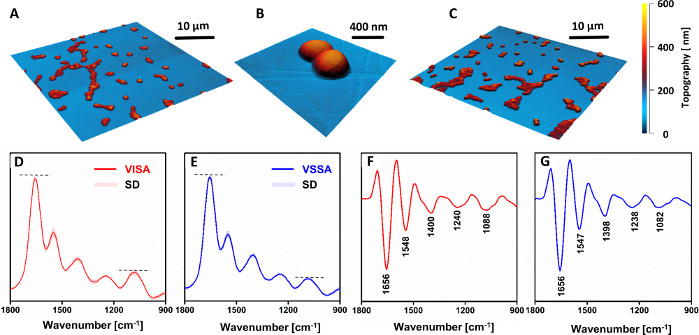
Figura 4: Resultados de AFM y AFM-IR del estudio de cambios químicos en vancomicina intermedia S. aureus (VISA) en comparación con S. aureus susceptible a vancomicina (VSSA) en pares clínicos. Imágenes AFM de muestras de una sola célula(A-B)VISA y(C)VSSA. Tamaño de las áreas fotografiadas: (A,C) 40 x 40 μm, (B) 2,56 x 2,45 μm. (D–E) Espectros AFM-IR medios y sus segundas derivadas (F–G) para: (D,F) VISA y (E,G) células VSSA, en el rango espectral 1800–900 cm-1. Los espectros presentados son un promedio de 81 (VISA) y 88 (VSSA) espectros individuales y se presentan junto con la desviación estándar (DE). El promedio se realizó después de la normalización de todos los espectros individuales juntos. Las bandas principales están marcadas en (F-G). Esta cifra ha sido modificada a partir de K. Kochan et al.19. Haga clic aquí para ver una versión más grande de esta figura.

Figura 5: Deriva de la imagen durante el registro consecutivo de mapas AFM-IR a valores de número de onda seleccionados para la celda de S. aureus. (Fila superior): Las imágenes AFM se grabaron simultáneamente con los correspondientes mapas AFM-IR(fila inferior)basados en la intensidad de la señal IR a los valores de número de onda seleccionados. Los valores de número de onda (966, 1055, 1079, 1106, 1234, 1398, 1454, 1540, 1656, 1740 cm-1) están anotados sobre la fila inferior. Cada conjunto (imagen AFM y mapa AFM-IR) se registró directamente después de la imagen anterior (aproximadamente 40 minutos por conjunto). El tamaño del área fotografiada/mapeada: 1,54 x 1,57 μm. Una clara deriva es visible entre las imágenes. Haga clic aquí para ver una versión más grande de esta figura.
Discusión
La utilidad de la espectroscopia IR para la caracterización de una amplia gama de muestras biológicas en el contexto de su composición química está bien establecida. Durante la última década, la espectroscopia IR se ha convertido en una herramienta prometedora para los estudios bacterianos12,13,14,15,16,17. Sigue atrayendo un interés sustancial en el campo de la microbiología, como una de las pocas técnicas que permiten una caracterización fenotípica a través de la composición química. En este contexto, el principal inconveniente de la microscopía FTIR convencional radica en la limitada resolución espacial, lo que impide los estudios unicelulares y subcelulares de bacterias. De hecho, el pequeño tamaño de las bacterias representa un impedimento no solo para la RI, sino para la gran mayoría de las técnicas. Por lo tanto, las herramientas de investigación disponibles para estudios unicelulares y subcelulares de bacterias son significativamente limitadas. La combinación de AFM con IR permite superar la limitación de resolución espacial de la espectroscopia IR, proporcionando una herramienta novedosa para la investigación bacteriana, capaz de sondear a nanoescala la composición química.
La técnica no se limita a estudios unicelulares y permite sondear una variedad de muestras, que varían en grosor. Sin lugar a dudas, la preparación limpia y cuidadosa de la muestra es fundamental para lograr imágenes de alta calidad. El protocolo aquí presente proporciona un método para preparar muestras multicapa, monocapa y / o unicelular de varias bacterias(Figura 1). La muestra preparada depende de varios factores, incluida la carga bacteriana inicial, la dilución posterior al lavado y la dilución adicional en el sustrato. La cantidad de muestra obtenida después de diluir los gránulos lavados y antes de la deposición en el sustrato generalmente permite la preparación de numerosas muestras. Por lo tanto, para obtener la distribución deseada de la muestra en el sustrato, a menudo es beneficioso preparar una serie de muestras, que varían en su dilución. Para los estudios que apuntan a la recolección de espectros AFM-IR en lugar de imágenes subcelulares, modificar la cantidad de muestra (por ejemplo, de monocapa a multicapa) puede ser beneficioso para aumentar la intensidad de la señal.
Otro aspecto crítico en la preparación de muestras es la eliminación adecuada de los residuos medios. Dependiendo de los métodos de cultivo de muestra seleccionados, la muestra se recoge de un medio líquido o de una placa de agar. En ambos casos, es probable que el residuo medio esté presente en la muestra, aunque en mucha menor medida tras la recolección de placas de agar. Como los medios de crecimiento bacteriano contienen una gran cantidad de varios componentes biológicos, es fundamental garantizar la eliminación adecuada del medio. Recomendamos tres lavados con agua ultrapura para muestras de placas de agar y al menos cuatro lavados para muestras recolectadas de medio. El número de lavados se puede aumentar, si es necesario; sin embargo, para la comparación entre varias muestras, es importante mantenerla consistente entre muestras. El protocolo demostrado utiliza agua, en lugar de disolventes como la solución tampón de fosfato (PBS) o solución salina. Tanto el PBS como la solución salina conducen a la formación de cristales al secarse al aire, lo que puede dañar las bacterias. Además, ambos son una fuente de bandas IR intensas, con PBS, en particular, que contiene múltiples bandas en la región de huellas dactilares. La falta de capacidad para el uso de solución salina o PBS, actualmente representa una limitación importante para la técnica. Por lo general, el uso de agua para el lavado no causa ninguna influencia destructiva en las bacterias; sin embargo, se debe tener cuidado y, si es posible, se debe limitar el tiempo de exposición al agua. Si el protocolo de preparación de la muestra debe detenerse en la etapa de lavado, se recomienda dejar la muestra en forma peletizada después de eliminar el agua. Esto es de particular importancia para las bacterias Gram-negativas, que contienen una pared celular más delgada ya que son más propensas a la ruptura.
Para garantizar datos AFM-IR adecuados y de alta calidad, varios aspectos en el protocolo de recopilación de datos son de importancia crítica. En primer lugar, la recopilación correcta de antecedentes es esencial para la adquisición de datos. En particular, es necesario mantener niveles de humedad estables durante toda la recolección de fondo, así como entre el fondo y la recolección de muestras. Para garantizar esto, recomendamos purgar el instrumento con nitrógeno y mantener los niveles de humedad no superiores al 25%. La falta de purga puede imponer una limitación significativa, particularmente en lugares con alta humedad. En segundo lugar, se debe destacar la importancia de una optimización adecuada de los puntos IR. Para obtener los mejores resultados, el conocimiento a priori sobre la posición de los máximos de banda puede ser beneficioso. Por ejemplo, se puede utilizar un espectro IR convencional de pellet bacteriano para determinar las posiciones de las bandas esperadas de una muestra. Si eso no es posible de adquirir, como un enfoque alternativo, el usuario puede utilizar los espectros IR disponibles en la literatura o comenzar la optimización utilizando una posición de banda que sea razonable esperar en la bacteria (por ejemplo, amida I y amida II). En tercer lugar, para la recopilación de datos, es importante destacar la importancia de una cuidadosa selección de potencia (que permita lograr una buena relación S / N), ya que puede tener un efecto destructivo. La potencia aconsejada depende del grosor de la muestra, con una guía aproximada disponible en el manual del instrumento31. Recomendamos probar empíricamente el estado de la muestra después de la medición mediante la recolección de una imagen AFM, ya que revelará cualquier influencia destructiva. Además, la recopilación de imágenes AFM de la misma área antes y después de la recolección de espectros AFM-IR sirve como una buena confirmación de que no se ha producido ninguna deriva y que los espectros se originan en el punto seleccionado en la celda. La posibilidad de deriva es particularmente importante cuando se aplica la modalidad de imagen, a través de imágenes consecutivas de intensidad IR a valores de número de onda seleccionados. Un ejemplo de esto se ilustra en la Figura 5. El área fotografiada se definió al comienzo del experimento y está destinada a ser consistente para todos los valores de número de onda. Sin embargo, una clara deriva es visible entre cada imagen de altura AFM (y la intensidad de número de onda IR correspondiente), con un tiempo de adquisición de cada mapa de aproximadamente 40 min. Debido a esto, para los usuarios que recopilan datos de imágenes, recomendamos seleccionar siempre un área ligeramente más grande que la muestra de interés, para garantizar que incluso después de la existencia de deriva, la muestra de interés permanezca dentro del área fotografiada.
Las limitaciones potenciales del protocolo incluyen la falta de capacidad para recopilar datos en un estado hidratado en soluciones fisiológicas (por ejemplo, solución salina o PBS) descritas anteriormente. Además, especialmente en áreas de alta humedad, a menudo hay una necesidad de purga de nitrógeno. Además, el protocolo permite sondear organismos de hasta 100 nm de tamaño, excluyendo la posibilidad de su uso para estructuras más pequeñas. Aunque esto se puede superar utilizando un láser diferente (por ejemplo, láser de cascada cuántica que permite alcanzar la resolución espacial de 20 nm), también se asocia con un rango espectral limitado, así como con dificultades para obtener una buena relación señal/ruido. Finalmente, el sondeo de superficies blandas puede presentar un desafío con la punta no detectando la superficie correctamente y procediendo más allá del punto de contacto, hasta la rotura. Aunque esto generalmente no es un problema con las muestras bacterianas, puede ocurrir en las mediciones de muestras más suaves. En tales casos, se recomienda intentar enganchar en la superficie limpia del sustrato cerca de la muestra.
El protocolo descrito se puede utilizar para numerosos tipos de investigación bacteriana, incluidos estudios comparativos entre varias muestras, así como el examen subcelular. Los datos pueden ser analizados utilizando enfoques quimiométricos para espectros únicos y modalidades de imagen35,dependiendo del objetivo de la investigación. Además, el protocolo también se puede modificar para su aplicación a otro material biológico (como hongos, levaduras, células, etc.), mediante la adición de fijación.
Divulgaciones
Nos gustaría agradecer a Bruker por el pago de la tarifa de publicación. KK, BRW, AP y PH son inventores de una patente internacional (PCTIB2020/052339), que describe algunos de los aspectos fundamentales del enfoque.
Agradecimientos
Nos gustaría agradecer a Bruker por su apoyo. Este trabajo fue apoyado por Monash University Advancing Women's Success Grant (K. Kochan). A.Y.P reconoce el apoyo de una beca profesional del Consejo Nacional de Salud e Investigación Médica de Australia (APP1117940). Este trabajo fue financiado por un Proyecto de Descubrimiento del Consejo Australiano de Investigación DP180103484. Quisiéramos dar las gracias al Sr. Finlay Shanks por su apoyo instrumental y a la Sra. Xenia Kostoulias por su asistencia técnica con las muestras.
Materiales
| Name | Company | Catalog Number | Comments |
| AFM metal specimen disc | PST ProSciTech Pty Ltd | GA530-15 | Recommended 15 mm |
| Anasys AFM-IR nanoIR2 | Anaysys Instruments | model: nanoIR2 | |
| Contact mode NIR2 Probes for nanoIR 2 | Bruker / Anasys Instruments | - | Model: Model: PR-EX-NIR2 |
| Heraeus Pico 17 Microcentrifuge | Thermo Scientific | - | - |
| Matlab | Mathworks Inc | - | Multivariate data analysis software |
| Micro-centrifuge tubes, 1.5 mL | Heathrow Scientific | HEA4323 | Can be replaced with any other micro-centrifuge tube |
| NanoIR 2 instrument | Bruker / Anasys Instruments | - | - |
| PLS toolbox | Mathworks Inc | - | GUI for Matlab |
| Selected bacterial medium (e.g. HBA Columbia Plates) | Thermo Fisher | PP2001 | Provided type of medium is an example and can be replaced by others, depending on the type of experiment |
| Selected bacterial strain | - | - | The source depends on the aim of research (patient isolates, ATCC strains, etc.) |
| Substrate (e.g. Raman grade CaF2) | Crystran | CAFP13-2R | Recommended size: 13 mm Ø x 2.0 mm |
| Tip pipette 1000 µl | Axygen | T-1000-B | - |
| Tip pipette 200 µl | Axygen | T-200-C | - |
| Tip pipette 0.5-10 µl | Axygen | T-300-R | - |
| Ultrapure water | - | - | - |
Referencias
- Sears, C. L. A dynamic partnership: celebrating our gut flora. Anaerobe. 11 (5), 247-251 (2005).
- Rudd, K. E., et al. regional, and national sepsis incidence and mortality, 1990-2017: analysis for the Global Burden of Disease Study. The Lancet. 395 (10219), 200-211 (2020).
- Seymour, C. W., et al. Time to treatment and mortality during mandated emergency care for sepsis. The New England Journal of Medicine. 376 (23), 2235-2244 (2017).
- Weiss, S. L., et al. Delayed antimicrobial therapy increases mortality and organ dysfunction duration in pediatric sepsis. Critical Care Medicine. 42 (11), 2409-2417 (2014).
- Peker, N., Couto, N., Sinha, B., Rossen, J. W. Diagnosis of bloodstream infections from positive blood cultures and directly from blood samples: recent developments in molecular approaches. Clinical Microbiology and Infection: The Official Publication of the European Society of Clinical Microbiology and Infectious Diseases. 24 (9), 944-955 (2018).
- Aminov, R. I. The role of antibiotics and antibiotic resistance in nature. Environmental Microbiology. 11 (12), 2970-2988 (2009).
- Levy, S. B., Marshall, B. Antibacterial resistance worldwide: causes, challenges and responses. Nature Medicine. 10 (12), 122-129 (2004).
- Roca, I., et al. The global threat of antimicrobial resistance: science for intervention. New Microbes and New Infections. 6, 22-29 (2015).
- O'Neill, J. The review on antimicrobial resistance. Tackling drug-resistant infections globally: final report and recommendations. Wellcome Trust. , (2016).
- Perry, J. A., Westman, E. L., Wright, G. D. The antibiotic resistome: what's new. Current Opinion in Microbiology. 21, 45-50 (2014).
- Piddock, L. J. Assess drug-resistance phenotypes, not just genotypes. Nature Microbiology. 1 (8), 16120 (2016).
- Naumann, D., Helm, D., Labischinski, H. Microbiological characterizations by FT-IR spectroscopy. Nature. 351 (6321), 81-82 (1991).
- Zarnowiec, P., Lechowicz, L., Czerwonka, G., Kaca, W. Fourier Transform Infrared Spectroscopy (FTIR) as a tool for the identification and differentiation of pathogenic bacteria. Current Medicinal Chemistry. 22 (14), 1710-1718 (2015).
- Quintelas, C., Ferreira, E. C., Lopes, J. A., Sousa, C. An overview of the evolution of infrared spectroscopy applied to bacterial typing. Biotechnology Journal. 13 (1), 1700449 (2018).
- San-Blas, E., Cubillán, N., Guerra, M., Portillo, E., Esteves, I. Characterization of xenorhabdus and photorhabdus bacteria by Fourier transform mid-infrared spectroscopy with attenuated total reflection (FT-IR/ATR). Spectrochimica Acta. Part A, Molecular and Biomolecular Spectroscopy. 93, 58-62 (2012).
- Sousa, C., et al. Discrimination of the acinetobacter calcoaceticus-acinetobacter baumannii complex species by Fourier transform infrared spectroscopy. European Journal of Clinical Microbiology & Infectious Diseases. 33 (8), 1345-1353 (2014).
- Rodriguez-Saona, L. E., Khambaty, F. M., Fry, F. S., Calvey, E. M. Rapid detection and identification of bacterial strains by Fourier transform near-infrared spectroscopy. Journal of Agricultural and Food Chemistry. 49 (2), 574-579 (2001).
- Dawson, S. E., et al. Implementation of Fourier transform infrared spectroscopy for the rapid typing of uropathogenic Escherichia coli. European Journal of Clinical Microbiology & Infectious Diseases. 33 (6), 983-988 (2014).
- Kochan, K., et al. Detection of Antimicrobial Resistance-Related Changes in Biochemical Composition of Staphylococcus aureus by Means of Atomic Force Microscopy-Infrared Spectroscopy. Analytical Chemistry. 91 (24), 15397-15403 (2019).
- Dazzi, A., Prater, C. B. AFM-IR: Technology and Applications in Nanoscale Infrared Spectroscopy and Chemical Imaging. Chemical Reviews. 117 (7), 5146-5173 (2017).
- Dazzi, A., et al. AFM-IR: combining atomic force microscopy and infrared spectroscopy for nanoscale chemical characterization. Applied Spectroscopy. 66 (12), 1365-1384 (2012).
- Kochan, K., et al. et al. In vivo atomic force microscopy-infrared spectroscopy of bacteria. Journal of the Royal Society, Interface. 15 (140), (2018).
- Katzenmeyer, A. M., et al. Mid-infrared spectroscopy beyond the diffraction limit via direct measurement of the photothermal effect. Nanoscale. 7 (42), 17637-17641 (2015).
- . Bruker Life Science Applications Available from: https://www.bruker.com/products/surface-and-dimensional-analysis/nanoscale-infrared-spectrometers/nanoscale-ir-spectroscopy-applications/life-sciences.html (2020)
- Mayet, C., Dazzi, A., Prazeres, R., Ortega, J. M., Jaillard, D. In situ identification and imaging of bacterial polymer nanogranules by infrared nanospectroscopy. Analyst. 135 (10), 2540-2545 (2010).
- Baldassarre, L., et al. Mapping the amide I absorption in single bacteria and mammalian cells with resonant infrared nanospectroscopy. Nanotechnology. 27 (7), 075101 (2016).
- Vitry, P., et al. Combining infrared and mode synthesizing atomic force microscopy: Application to the study of lipid vesicles inside Streptomyces bacteria. Nano Research. 9 (6), 1674-1681 (2016).
- Dazzi, A., et al. Chemical mapping of the distribution of viruses into infected bacteria with a photothermal method. Ultramicroscopy. 108 (7), 635-641 (2008).
- Steenbergen, J. N., Alder, J., Thorne, G. M., Tally, F. P. Daptomycin: a lipopeptide antibiotic for the treatment of serious Gram-positive infections. The Journal of Antimicrobial Chemotherapy. 55 (3), 283-288 (2005).
- Garcia, L. S. MacFarlan Standards. Clinical Microbiology Procedures Handbook, 3rd Edition. , (2010).
- NanolR-2 System Manual. Anasys Instruments Available from: https://www.anasysinstruments.com/downloadpr/nanoIR2_s_System_Manual.pdf (2020)
- Whelan, D. R., et al. Detection of an en masse and reversible B- to A-DNA conformational transition in prokaryotes in response to desiccation. Journal of the Royal Society, Interface. 11 (97), 20140454 (2014).
- McGuinness, W. A., Malachowa, N., DeLeo, F. R. Vancomycin Resistance in Staphylococcus aureus. The Yale Journal of Biology and Medicine. 90 (2), 269-281 (2017).
- Howden, B. P., Peleg, A. Y., Stinear, T. P. The evolution of vancomycin intermediate Staphylococcus aureus (VISA) and heterogenous-VISA. Infection, Genetics and Evolution: Journal of Molecular Epidemiology and Evolutionary Genetics in Infectious Diseases. 21, 575-582 (2014).
- Perez-Guaita, D., et al. Multispectral Atomic Force Microscopy-Infrared Nano-Imaging of Malaria Infected Red Blood Cells. Analytical Chemistry. 90 (5), 3140-3148 (2018).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados