Method Article
Monitoreo de las interacciones proteína-ligando en células humanas mediante RMN cuantitativa en células en tiempo real utilizando un biorreactor de alta densidad celular
En este artículo
Resumen
Este protocolo describe la configuración de un biorreactor de RMN para mantener viables las células humanas encapsuladas durante un máximo de 72 h, seguido de la adquisición y el análisis de datos de RMN en las células resueltos en el tiempo. La metodología se aplica para monitorizar las interacciones proteína-ligando intracelular en tiempo real.
Resumen
La RMN en células es un enfoque único para observar las propiedades estructurales y dinámicas de las macromoléculas biológicas a resolución atómica directamente en células vivas. Se puede observar plegamiento de proteínas, modificaciones químicas y cambios conformacionales inducidos por la unión al ligando. Por lo tanto, este método tiene un gran potencial en el contexto del desarrollo de fármacos. Sin embargo, la corta vida útil de las células humanas confinadas en el espectrómetro de RMN limita el rango de aplicación de la RMN en las células. Para superar este problema, se emplean biorreactores de RMN que pueden mejorar en gran medida la estabilidad de la muestra celular a lo largo del tiempo y, lo que es más importante, permitir el registro en tiempo real de los espectros de RMN en la célula. De esta forma, se puede monitorizar en tiempo real la evolución de procesos como la penetración del ligando y la unión a la diana proteica intracelular. Los biorreactores a menudo están limitados por la baja viabilidad celular a un alto número de células, lo que resulta en una compensación entre la sensibilidad general del experimento y la viabilidad celular. Recientemente informamos de un biorreactor de RMN que mantiene un alto número de células humanas metabólicamente activas durante largos períodos de tiempo, hasta 72 h. Esta configuración se aplicó para monitorear las interacciones proteína-ligando y la modificación química de proteínas. También introdujimos un flujo de trabajo para el análisis cuantitativo de los datos de RMN en tiempo real, basado en la resolución de curvas multivariantes. El método proporciona perfiles de concentración de las especies químicas presentes en las células en función del tiempo, que pueden analizarse más a fondo para obtener parámetros cinéticos relevantes. Aquí proporcionamos una descripción detallada de la configuración del biorreactor de RMN y su aplicación para monitorear las interacciones proteína-ligando en células humanas.
Introducción
La espectroscopia de resonancia magnética nuclear (RMN) en células ha surgido recientemente como un enfoque poderoso para investigar las propiedades estructurales y dinámicas de las macromoléculas dentro del entorno celular1,2,3,4,5,6. La RMN en células tuvo éxito en la investigación de procesos funcionalmente relevantes como el plegamiento/plegamiento incorrecto de proteínas7,8,9, la unión a metales7,10, la formación de enlaces disulfuro11,12 y la interacción proteína-proteína13, la interacción proteína-ligando14,15,16 y la interacción ácido nucleico-ligando17 ,18 en células humanas vivas. Uno de los factores limitantes de las aplicaciones de RMN en células es la corta vida útil de las células durante el experimento. La solución a este problema implica el uso de biorreactores de RMN. En estos dispositivos, se aplica un flujo constante de medio de crecimiento a las células, que se mantienen confinadas dentro del espectrómetro de RMN, con el fin de proporcionar oxígeno y nutrientes y eliminar los subproductos tóxicos. Tras el advenimiento de la RMN en las células, se han desarrollado varios diseños de biorreactores de RMN para mejorar la viabilidad celular durante períodos de tiempo más largos, en los que las bacterias o las células de mamíferos se encapsulan en un hidrogel19,20,21,22 o se mantienen en suspensión y se perfunden mediante el uso de una membrana de microdiálisis23 . Tales biorreactores han permitido la adquisición de experimentos de RMN más largos con una mayor relación señal-ruido (S/N)5 y, lo que es aún más importante, podrían emplearse para investigar procesos celulares en tiempo real22,23,24. Gracias a la alta sensibilidad química y conformacional de la RMN, esta última aplicación puede proporcionar información valiosa sobre la cinética de los procesos funcionales dentro de las células vivas a resolución atómica.
En este protocolo, mostramos cómo configurar y operar un biorreactor mejorado recientemente reportado25, que se obtuvo combinando un diseño de biorreactor modular existente23 con el enfoque basado en la encapsulación celular en hidrogel que fueron iniciados por otros grupos19,20,21,22,26,27 . Describimos la aplicación del biorreactor a estudios de RMN en tiempo real en las células de la unión intracelular a ligandos de observación de proteínas en células HEK293T. En el biorreactor, las células se encapsulan a alta densidad en hilos de gel de agarosa y se mantienen altamente viables y metabólicamente activas durante hasta 72 h, durante las cuales se registran experimentos de RMN en las células en tiempo real. El biorreactor está compuesto por un tubo de vidrio que se ajusta a sondas de RMN estándar de 5 mm que es estanca y está conectada a un soporte de tubo para que la cámara de muestra interna tenga un diámetro interno de 4,2 mm, una altura de 38 mm y un volumen de 526 μL. La entrada es un capilar PEEK de 7 metros de largo (o.d. = 1/32", i.d. = 0.5 mm) insertado en la cámara de muestra hasta ~ 6 mm desde la parte inferior, mientras que la salida es un capilar de PTFE de 7 metros de largo (o.d. = 1/32", i.d. = 0.5 mm) unido en la parte superior del soporte del tubo (Figura 1). El tubo se inserta coaxialmente en una línea de temperatura controlada conectada a un baño de agua. La entrada y la salida están conectadas a través de tubos PEEK a una válvula de 4 vías y 2 posiciones conectada a una bomba FPLC para controlar el flujo medio y un contenedor de residuos.
El biorreactor se aplica para estudiar la cinética de la interacción, previamente reportada14,25, entre dos fármacos, acetazolamida (AAZ) y metazolamida (MZA), en células humanas con la segunda isoforma de la anhidrasa carbónica humana (CA II), diana farmacológicamente relevante28,29,30, y la cinética de la formación del enlace disulfuro intramolecular, promovido por la pequeña molécula ebselen25, 31, de la forma libre de cobre y unida al zinc del cobre humano, la superóxido dismutasa de zinc (SOD1), una enzima antioxidante implicada en la aparición de la esclerosis lateral amiotrófica7,8,32. Finalmente, el análisis cuantitativo de los datos de RMN en tiempo real se realiza en MATLAB utilizando el algoritmo Multivariate Curve Resolution-Alternating Least Squares (MCR-ALS)33, a través del cual se obtienen los componentes espectrales puros y los perfiles de concentración en función del tiempo para las especies observadas, que pueden analizarse más a fondo para obtener parámetros cinéticos relevantes.
El protocolo comienza a partir de un matraz T75 de células HEK293T (~ 3 x 107 células por matraz) que sobreexpresa transitoriamente CA II humano (sin etiquetar) o SOD1 humano (marcado con 15N). Las células se cultivaron y mantuvieron en matraces T75 con DMEM alto en glucosa por 1:10 pasajes cada 3-4 días y se transfectaron con el ADNc que codifica la proteína de interés 48 h antes del experimento. Los pasos involucrados en esta fase se informan en detalle en otra parte34.
Protocolo
1. Configuración del reactivo y la solución
- Para preparar dmEM completo, agregue 5 ml de L-glutamina 200 mM, 5 ml de penicilina-estreptomicina 100x y 50 ml de suero fetal bovino (FBS, concentración final de 10% vol/vol) a 440 ml de DMEM.
NOTA: Esta solución se puede almacenar a 4 °C durante 1 mes. - Preparar la solución de agarosa disolviendo 150 mg de agarosa de baja gelificación en 10 ml de solución salina tamponada con fosfato (PBS) a 85 °C para obtener una solución al 1,5% (p/v). Esterilizar por filtración con un filtro de 0,22 μm. Preparar 1 ml de alícuotas de solución de agarosa en tubos tapados de 1,5 ml y conservar a 4 °C.
- Preparar el medio biorreactor.
- Disolver 13,4 g de polvo dmEM en 1 L de H2O ultrapuro.
NOTA: Dependiendo de la aplicación, el volumen final requerido puede diferir (por ejemplo, para un medio de 500 ml, disuelva 6,7 g de polvo en 500 ml de H2O). - Agregue 2% fbS, 10 mM NaHCO3, 1x penicilina-estreptomicina (100x) y 2% D2O (por ejemplo, para 500 ml de medio, agregue 10 ml de FBS, 0.4 g de NaHCO3, 5 ml de penicilina-estreptomicina 100x y 10 ml de D2O).
- Mida el pH con un medidor de pH y, si es necesario, ajuste a 7.4 agregando HCl.
NOTA: Por lo general, el pH inicial es muy cercano a 7.4. - Filtre el medio biorreactor con un filtro estéril accionado por vacío en una botella de vidrio estéril de 250 ml o 500 ml.
- En la campana de flujo laminar, selle la botella con un cabezal de acero estéril con dos boquillas de manguera y conéctelas a un tubo FEP (o.d. = 1/8", es decir, = 1,6 mm) que se conectará a la bomba y a un filtro de jeringa de PTFE de 0,22 μm para la entrada de aire.
- Disolver 13,4 g de polvo dmEM en 1 L de H2O ultrapuro.
2. Configuración del biorreactor
- Ensamble la unidad de flujo utilizando un segundo tubo de RMN de la unidad de flujo, que luego se reemplazará por el que contiene las celdas. Consulte las instrucciones de funcionamiento de la unidad de flujo para el montaje correcto.
NOTA: En este punto, la unidad de flujo ya debe estar limpia (de lo contrario, realice el paso 4.2). - Ajuste el baño de agua conectado al control de temperatura de la unidad de flujo a 37 °C. Coloque la botella del depósito en el baño de agua.
- Conecte el tubo FEP de la botella del depósito a la bomba.
- Gire la válvula del biorreactor para "bypass" y prellene la bomba con medio.
- Gire la válvula del biorreactor para "fluir" y rellene previamente el biorreactor con medio a 0,1 ml/min.
3. Preparación de la muestra celular
- Recoge las células de la incubadora de CO2 .
- Tome un matraz T75 de células HEK293T transfectadas de la incubadora de CO2 y retire el medio gastado.
- Lave las celdas dos veces con 7 ml (cada uno) de PBS a temperatura ambiente (~ 20 ° C).
- Use 2 ml de tripsina/EDTA para separar las células. Después de agregar la solución, incube durante 5 minutos a temperatura ambiente para separar las células.
NOTA: Las células transfectadas pueden tardar un poco más en desprenderse. Si es necesario, incubar las células a 37 °C. - Inactivar tripsina con 20 ml de DMEM completo; Resuspender completamente las células mediante pipeteos hacia arriba y hacia abajo y transferirlas en un tubo centrífugo de 50 ml.
- Centrifugar las células a 800 x g durante 5 min a temperatura ambiente y desechar el sobrenadante.
- Lave las células con 10 ml de PBS a temperatura ambiente para eliminar el medio residual.
- Centrifugar las células a 800 x g durante 5 min a temperatura ambiente y desechar el sobrenadante.
- Transfiera el pellet celular a un tubo de microcentrífuga tapado de 1,5 ml.
- Incrustar células en hilos de agarosa.
- Derretir una alícuota de agarosa solidificada a 85 °C en un baño de agua y posteriormente mantenerla en solución a 37 °C en un calentador de bloque.
- Con una pipeta Pasteur, llene la parte inferior del tubo de RMN de la unidad de flujo con 60-70 μL de gel de agarosa al 1,5% y colóquelo en hielo. Esto creará un tapón inferior de ~ 5 mm de altura que permite colocar la muestra de la celda dentro del volumen activo de la bobina de RMN de 1H.
- Calentar el pellet de células obtenido en el paso 3.1.8 a 37 °C durante 15−20 s en el termobloqueo.
- Resuspend células en 450 μL de solución de agarosa. Tenga cuidado de evitar la formación de burbujas.
- Aspire la suspensión de célula-agarosa en un tubo PEEK de cromatografía de ~30 cm de largo (i.d. = 0,75 mm) conectado a una jeringa de 1 ml.
NOTA: Antes de la aspiración, el tubo y el volumen muerto de la jeringa deben rellenarse previamente con PBS a temperatura ambiente para evitar la formación de burbujas. La longitud del tubo no es crítica. - Deje que el tubo se enfríe a temperatura ambiente durante 2 min.
- Prellene el tubo de RMN de la unidad de flujo con 100 μL de PBS a temperatura ambiente.
- Fundir hilos de células incrustadas en agarosa en el tubo de RMN de la unidad de flujo empujando suavemente la jeringa.
NOTA: Para llenar el tubo de RMN de forma homogénea, comience colocando el extremo del tubo PEEK en la parte inferior del tubo de RMN y continúe hacia la parte superior mientras gira lentamente de izquierda a derecha. - Repita los pasos 3.2.5, 3.2.6 y 3.2.8 hasta que se haya fundido toda la suspensión de células de agarosa.
- Inserte células en el biorreactor.
- Retire el tubo de RMN vacío de la unidad de flujo y aumente el caudal a 2 ml/min durante unos minutos para eliminar las burbujas de gas residuales en el tubo de entrada.
- Ajuste el caudal a 0,2 ml/min e inserte el tubo de RMN que contiene las células empujándolo hacia arriba lenta pero constantemente.
NOTA: El flujo activo del medio evita el reflujo del contenido del tubo a través de la entrada, que de otro modo ocurriría durante la inserción.
4. Operación y limpieza del biorreactor
- Operación del biorreactor durante el experimento de RMN.
- Ajuste la temperatura en el espectrómetro de RMN a 310 K.
- Inserte la unidad de flujo en el espectrómetro.
- Suministrar el medio biorreactor a un caudal de 0,1 ml/min durante toda la duración de los experimentos de RMN en células.
- En el momento deseado durante el experimento, inyecte una solución concentrada de molécula externa a la botella de depósito medio perforando el tubo de silicona con una jeringa estéril de aguja larga.
NOTA: La concentración final de molécula en el medio debe elegirse en función del conocimiento previo de la toxicidad celular y, si está disponible, de la tasa de difusión prevista / estimada a través de la membrana celular. - Al final del experimento de RMN, reemplace el tubo que contiene las células con un tubo vacío y enjuague la unidad de flujo con agua.
- Biorreactor limpio in situ.
- Limpie la unidad de flujo haciendo fluir las siguientes soluciones a 1 ml/min: hidróxido de sodio (NaOH) de 0,2 M; 3 M de ácido cítrico; 0,2 M de NaOH, durante al menos 30 min cada uno, seguido de agua ultrapura filtrada estéril durante >2 h.
- Limpie y autoclave la botella del depósito y el conjunto de tubos después de cada carrera.
5. Experimentos de RMN
- Configuración de los experimentos de RMN.
NOTA: Realice estos pasos de antemano, antes de la preparación de la muestra de RMN en la celda, para evitar retrasos entre la recolección de células y la adquisición de datos.- Cree un nuevo conjunto de datos en el espectrómetro de RMN y establezca los parámetros para los experimentos de RMN deseados.
- Establezca parámetros para experimentos de RMN 1D 1H.
- Centrar la frecuencia portadora 1H a 4,7 ppm en la señal de agua.
- Seleccione el programa de pulso zgesgp, establezca el ancho espectral en 20 ppm y un pulso cuadrado de 1.000 μs y 180 ° para la supresión de agua. Establezca un retardo entre escaneos de 1 s. Adquiere el espectro con 32 escaneos.
- Para las células que expresan CA II sin etiquetar: seleccione el programa de pulso p3919gp, establezca el ancho espectral en 30 ppm para cubrir la región imino del espectro y ajuste el retardo para la supresión binomial de agua de modo que la excitación máxima se centre en los cambios químicos de las señales de interés (d7 = 20 μs a 950 MHz). Establezca un retardo entre escaneos de ≥1 s. Adquiera con 512 escaneos.
- Para las células que expresan SOD1 marcado con 15N: seleccione el programa de pulso sfhmqf3gpph, establezca los anchos espectrales 1H y 15N en 16 y 50 ppm, respectivamente, el desplazamiento de pulso con forma y el ancho de banda de excitación en 8.5 y 6 ppm, respectivamente, y un pulso de 350 μs para el esquema de desacoplamiento (garp4 u otro dependiendo del instrumento). Establezca un retardo entre escaneos de 0,3 s. Adquiera con 16 escaneos y 128 incrementos en la dimensión 15N.
- Adquisición de espectros de RMN en tiempo real.
- Una vez que el biorreactor se inserta en el espectrómetro de RMN, espere unos minutos para permitir el intercambio del medio.
NOTA: Este proceso se monitorea fácilmente desde la apariencia de la señal de bloqueo, ya que el PBS se reemplaza con un medio que contiene 2% de D2O. - Ajuste la coincidencia y el ajuste del canal 1H, ajuste el imán y calcule la longitud del pulso duro 1H 90 °.
- Ajuste los niveles de potencia de 1H en cada secuencia de pulsos de acuerdo con el pulso duro de 1H.
- Registre un primer espectro zgesgp 1H para comprobar el contenido de la muestra y la homogeneidad del campo.
- Copie los experimentos zgesgp y p3919gp/sfhmqcf3gpph al número deseado y póngalos en cola en la cola de adquisición.
NOTA: Los espectros zgesgp sólo se utilizan para controlar el estado de la muestra y la homogeneidad del campo; por lo tanto, se pueden omitir o grabar con menos frecuencia. - Para celdas que expresan CA II sin etiquetar: procese los espectros p3919gp aplicando la función de ventana de llenado cero y ampliación de línea exponencial (LB = 20 Hz).
- Para las celdas que expresan SOD1 marcado con 15N: procese los espectros sfhmqcf3gpph aplicando relleno cero y función de ventana de campana sinusoidal cuadrada (SSB = 2) en ambas dimensiones.
NOTA: El tamaño de los espectros procesados se puede reducir aún más eliminando las regiones libres de señales (en Topspin, esto se hace estableciendo los valores STSR y STSI deseados).
- Una vez que el biorreactor se inserta en el espectrómetro de RMN, espere unos minutos para permitir el intercambio del medio.
6. Análisis MCR-ALS
- Para el análisis de espectros CA II, importe regiones espectrales 1D en MATLAB R2019b.
- En el software, cree una lista de experimentos que se exportarán en el menú Lista de conjuntos de datos de procesos .
- Utilizando una versión modificada del programa au convbin2asc, exporte la región espectral de interés en formato ASCII para cada espectro.
Nota : esto crea un archivo de texto denominado ascii-spec.txt en cada subdirectorio de espectro. - En MATLAB, importe las regiones espectrales mediante el script personalizado Load_ascii_spectra.
NOTA: Este script requiere el directorio del conjunto de datos como entrada y produce un espectro de matriz 2D que contiene los espectros 1D apilados y una matriz 1D cs que contiene los desplazamientos químicos. - Ejecute el script Load_acqus para extraer las marcas de tiempo de los espectros 1D.
NOTA: Este script produce una matriz 1D times_hours que contiene el incremento de tiempo para cada espectro expresado en horas, con el espectro inicial en el tiempo = 0.
- Para el análisis de espectros SOD1, importe espectros 2D en MATLAB R2020b.
- En Topspin, cree una lista de los experimentos que se exportarán en el menú Lista de conjuntos de datos de procesos .
- En MATLAB, importe los espectros 2D mediante el script personalizado Load_2D_spectra.
NOTA: Este script requiere el directorio del conjunto de datos como entrada y produce una matriz 3D Spectra que contiene los espectros 2D apilados y una matriz 1D cs que contiene los desplazamientos químicos. - Ejecute el script Load_acqus para extraer las marcas de tiempo de los espectros 2D.
- Especifique las regiones espectrales de interés en el script personalizado Cut_2D_spectra y ejecute el script para cortar submatrizes 3D [(1H x 15N) intensidades espectrales) x tiempo]; remodelarlos como matrices 2D (puntos de tiempo x intensidades espectrales) y unirlos.
NOTA: Esto produce una matriz 2D JoinSpec_flat que contiene las regiones espectrales reformadas y unidas.
- Ejecute MCR-ALS 2.0 en modo GUI.
- Abra la GUI de MCR-ALS 2.0 ejecutando el script mcr_main.
- En la ficha Selección de datos , cargue los espectros o la matriz JoinSpec_flat. Los datos se pueden trazar para su comprobación.
- Evalúe el número de componentes mediante la descomposición de valores singulares (SVD) o manualmente.
NOTA: El número de componentes debe corresponder al número de especies distintas presentes en el experimento. En este caso n = 2, correspondiente a la proteína libre y unida. - Seleccione un método para la estimación inicial de los espectros puros. Se puede utilizar la detección de variables más pura o el análisis factorial evolutivo (EFA).
- En la ventana Selección del conjunto de datos , seleccione Continuar.
- Establezca las restricciones para las concentraciones en la ventana Restricciones: Modo de fila . Aplique una restricción de no negatividad, seleccione fnnls (Fast nonnegativity-constrained least-squares) como "implementación y 2 especies". Aplique 1 restricción de cierre, establezca la restricción en 1, la condición de cierre como "igual a" y aplíquela a todas las especies.
NOTA: Esto obliga a que la suma de las concentraciones de cada especie sea igual a 1, de modo que los perfiles obtenidos de cada especie se normalicen con respecto a la concentración total de proteínas. - Establezca las restricciones para los espectros en la ventana Restricciones: Modo de columna . Aplique una restricción de no negatividad, seleccione fnnls como "implementación y 2 especies".
NOTA: Esta restricción no debe aplicarse si hay señales negativas presentes en los espectros de RMN. - En la ventana final, establezca 50 iteraciones y un criterio de convergencia de 0.01. Especifique los nombres de salida para Concentraciones, Espectros y Desviación Std. Haga clic en Continuar para ejecutar el ajuste MCR-ALS.
NOTA: Una ventana gráfica mostrará el resultado del ajuste con gráficos de los perfiles de concentración y los espectros de los componentes puros. - Para el conjunto de datos SOD1: utilice el script personalizado Rebuild_2D_spectra para reconstruir las regiones espectrales 2D a partir de la salida 1D de MCR-ALS y trazarlas.
7. Prueba azul de tripano
- Recupere el contenido del tubo de RMN con una pipeta Pasteur y transfiera las roscas de agarosa a un tubo tapado de 1,5 ml.
- Retire el medio residual enjuagando los hilos de agarosa con 600 μL de PBS y centrífuguelos a 4.000 x g durante 1 min a temperatura ambiente. Deseche el sobrenadante.
- Añadir 250 μL de PBS y 50 μL de solución de azul de tripano al 0,4%.
- Incubar durante 2 min con pipeteo continuo.
- Lavar dos veces con 600 μL de PBS descartando el sobrenadante.
- Coloque algunos hilos de agarosa en un portaobjetos de microscopio y córtelos con cuchillas de afeitar para crear pequeñas rebanadas de gel. Seleccione las rodajas más delgadas (espesor < 0,4 mm, idealmente ~0,2 mm) para el análisis.
- Transfiera las rodajas de gel a una cámara de conteo de células de fabricación propia que consta de dos portaobjetos de vidrio espaciados por tres capas de película de parafina (~ 0,4 mm de espesor total) en cada lado.
NOTA: También se podría utilizar una corredera de cámara; sin embargo, las rodajas de gel más gruesas que la altura de la cámara (0,1 mm) se apretarían, rompiendo las celdas incrustadas. - Adquirir imágenes de las células dentro de la agarosa y contar las células blancas y azules.
- Calcule la viabilidad celular como (células totales - celdas azules) / células totales.
Resultados
El protocolo anterior permite la encapsulación de células en hilos de hidrogel para maximizar la viabilidad celular durante largos períodos de tiempo, necesarios para investigar en tiempo real los procesos intracelulares. En el biorreactor, las células se mantienen vivas y metabólicamente activas hasta 72 h, como lo confirma la prueba azul de Tripano (Figura 2a-c). En principio, este protocolo se puede aplicar para observar una proteína intracelular de interés que sufre cualquier cambio conformacional o químico. En la primera aplicación descrita anteriormente, se aplica el biorreactor para monitorizar en tiempo real la unión de dos inhibidores, AAZ y MZA, a CA II sobreexpresados en el citosol de las células HEK293T. El primer espectro de escultura de excitación 1H (zgesgp) registrado se utiliza para evaluar la intensidad general de la señal (que es proporcional al número de células), la presencia de señales de la proteína sobreexpresada y la homogeneidad del campo (Figura 2d). En el caso de CA II, la unión intracelular de los dos inhibidores puede ser monitoreada por WATERGATE 3-9-1935 1D 1H espectros de RMN (p3919gp), mediante la observación de señales 1H en la región entre 11 y 16 ppm. Estas señales surgen de las histidinas coordinadoras de zinc y otros residuos aromáticos de CA II36 y se perturban al unirse al ligando14,15. Las concentraciones de ligandos pueden elegirse en función de la velocidad de difusión o, si se dispone de ellos, de valores de permeabilidad previamente determinados14. El enlace exitoso se confirma visualmente por la aparición de un conjunto adicional de señales en la región espectral de interés, que reemplaza gradualmente las señales originales (Figura 3). Las curvas de unión dependientes del tiempo se obtienen mediante el análisis MCR-ALS, que separa los dos conjuntos de señales de RMN que surgen de la CA II libre y unida (Figura 4a) y proporciona simultáneamente los perfiles de concentración relativa de las dos especies (Figura 4b). En la segunda aplicación, el biorreactor se aplica para monitorear la formación de enlace disulfuro intramolecular SOD1 unido al zinc promovido por ebselen, un mimético de glutatión peroxidasa, en células humanas. Este proceso se monitorea observando cambios en los espectros 1H-15N 2D SOFAST-HMQC37 (que proporciona una huella digital de la conformación de la columna vertebral de la proteína) causados por perturbaciones de la estructura de la proteína inducidas por la formación de enlaces disulfuro. Las señales adicionales que surgen de SOD1 oxidado con disulfuro aparecen en el espectro 1H-15N y reemplazan gradualmente a las de SOD1 reducida por disulfuro. El análisis MCR-ALS en regiones seleccionadas del espectro 2D separa las señales que surgen de las dos especies (Figura 5a) y proporciona sus perfiles de concentración relativa (Figura 5b). Las curvas de concentración obtenidas pueden ser analizadas posteriormente mediante ajuste no lineal para proporcionar información sobre la cinética de los procesos objeto de estudio25.
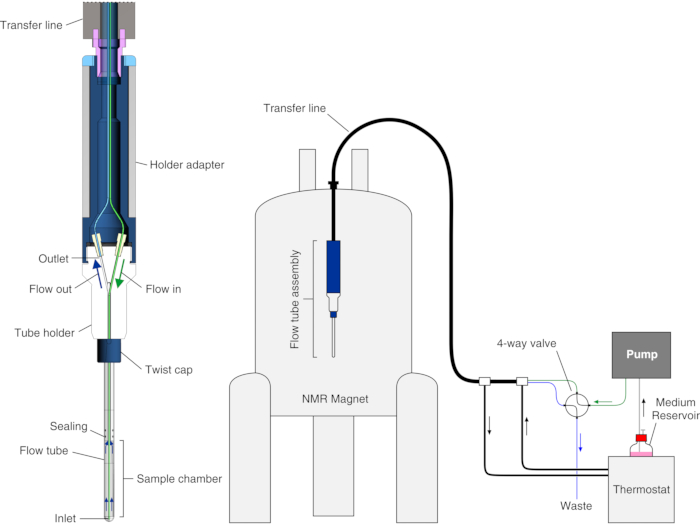
Figura 1: Esquema del biorreactor. Izquierda: vista de sección transversal de la unidad de flujo vacía. Derecha: esquema de la configuración del biorreactor. El tubo de entrada PEEK se muestra en verde; el tubo de salida de PTFE se muestra en azul. El panel izquierdo se reproduce con permiso de Luchinat et al.25. Haga clic aquí para ver una versión más grande de esta figura.
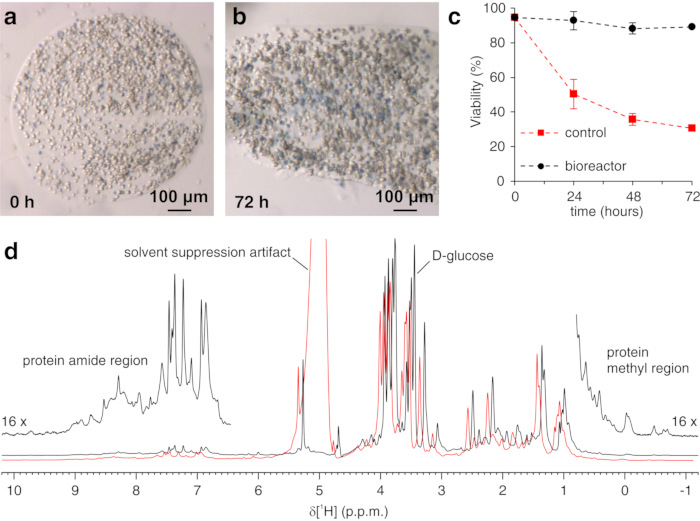
Figura 2: Prueba de trypan blue en células encapsuladas y verificación de muestras por RMN de 1H. Rodajas representativas de agarosa que contienen células incrustadas y teñidas con azul de tripano (a) inmediatamente después de la fundición y (b) después de 72 h en el biorreactor; c) viabilidad celular en función del tiempo en el biorreactor de RMN bajo flujo activo (negro) y en condiciones estáticas (rojo), medida por Trypan Blue Test. d) espectros de RMN zgesgp 1H registrados en células incrustadas en agarosa que sobreexpresan CA II en ausencia (negro) y en presencia (rojo) de burbujas de gas en el biorreactor. En este último caso, la disminución de la homogeneidad del campo provoca el ensanchamiento de la línea y la aparición de artefactos de supresión de disolventes. Se etiquetan características espectrales interesantes. Los paneles (a-c) se reproducen con permiso de Luchinat et al.25. Haga clic aquí para ver una versión más grande de esta figura.
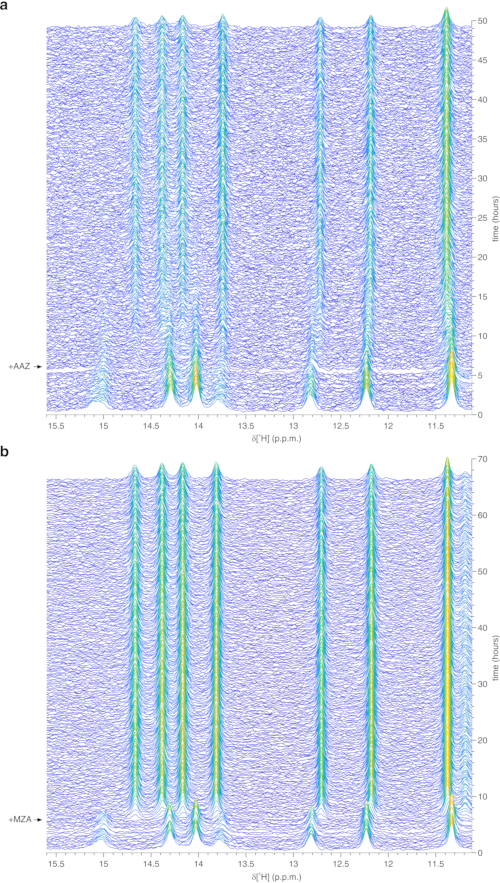
Figura 3: Datos representativos de RMN en tiempo real en la célula 1H obtenidos en células encapsuladas en agarosa en el biorreactor. Diagramas en cascada de espectros de RMN 1D 1H (región entre 15,6 y 11,1 ppm) de células HEK293T que sobreexpresan CA II y posteriormente tratadas con (a) 25 μM AAZ y (b) 10 μM MZA, registradas en función del tiempo en el biorreactor de RMN. El tiempo de tratamiento del ligando se muestra con una flecha. La intensidad espectral (a.u.) está codificada por colores de azul (más bajo) a amarillo (más alto). Esta cifra se reproduce con permiso de Luchinat et al.25. Haga clic aquí para ver una versión más grande de esta figura.
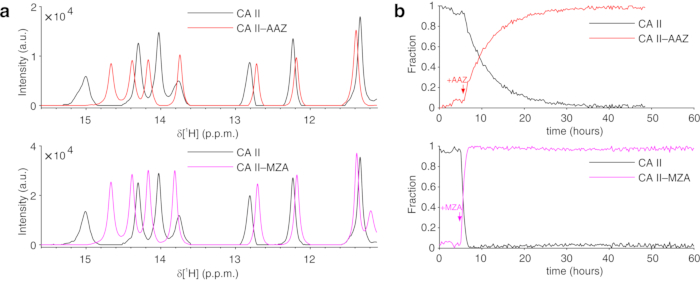
Figura 4: Salida representativa de MCR-ALS a partir de espectros de RMN 1D. (a) Espectros de RMN 1H de los componentes puros reconstruidos por MCR-ALS: CA II en ausencia de ligandos (negro) y en el complejo con AAZ (rojo) o MZA (magenta); b) perfiles de concentración relativa de CA II libre (negro) y unido en función del tiempo tras la adición de AAZ (rojo) o MZA (magenta) obtenidos por MCR-ALS. Los tiempos de tratamiento del ligando están marcados con flechas. Esta cifra se reproduce con permiso de Luchinat et al.25. Haga clic aquí para ver una versión más grande de esta figura.
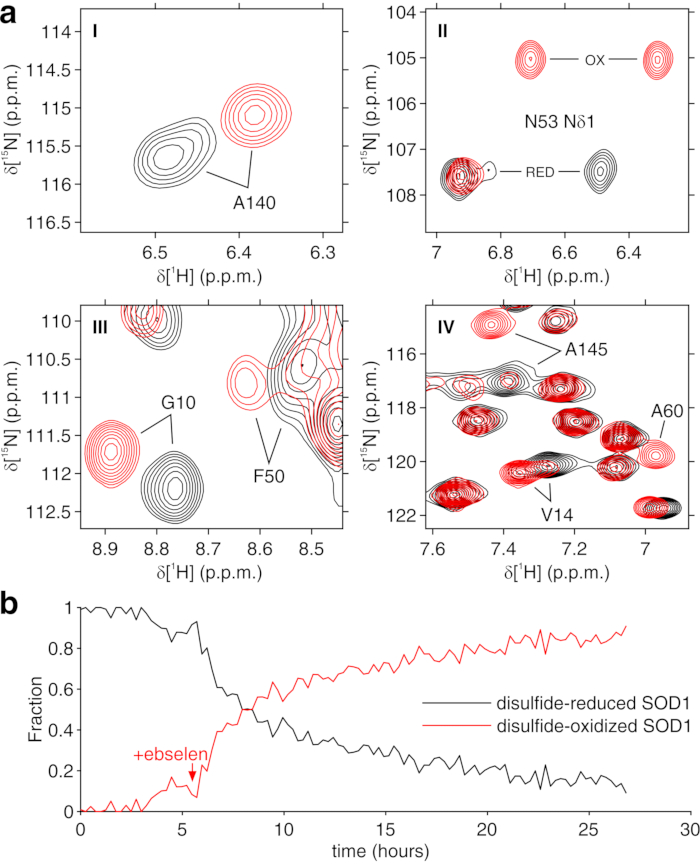
Figura 5: Salida representativa de MCR-ALS a partir de espectros de RMN 2D. (a) Regiones espectrales 1H-15N (etiquetadas I-IV) de los componentes puros reconstruidos por MCR-ALS: SOD1 reducido por disulfuro (negro) y SOD1 oxidado por disulfuro (rojo); b) perfil de concentración relativa de los componentes puros en función del tiempo tras la adición de ebselen (marcado con una flecha) obtenido por MCR-ALS. Esta cifra se reproduce con permiso de Luchinat et al.25. Haga clic aquí para ver una versión más grande de esta figura.
Discusión
El objetivo de utilizar un biorreactor para el experimento de RMN en las células es mantener las células vivas y metabólicamente activas durante un período prolongado de tiempo. Para lograr este objetivo deben tenerse en cuenta una serie de aspectos críticos. En primer lugar, es primordial evitar la contaminación bacteriana al preparar la muestra celular y durante la adquisición de datos de RMN. Si se utilizan cepas de E. coli u otras bacterias comúnmente utilizadas para la clonación de genes y la expresión de proteínas recombinantes en el laboratorio, pueden contaminar las células durante la preparación de la muestra. Una vez en el biorreactor, las bacterias crecerán rápidamente explotando el medio de crecimiento fresco y causarán la muerte celular debido a la producción de endotoxinas. La contaminación bacteriana solo se detecta en una etapa avanzada, cuando convierte el medio de crecimiento en amarillo y turbio. Además, la limpieza incompleta del biorreactor podría causar contaminación de la bomba o el tubo con bacterias, levaduras o mohos comunes.
Un requisito para el éxito del experimento es evitar la formación de burbujas de gas. Las burbujas de gas atrapadas entre las roscas de agarosa en el volumen activo de la bobina de RMN introducirían grandes inhomogeneidades del campo magnético, causando una supresión incompleta de la señal H2O y una pérdida severa de calidad espectral (Figura 2d). Las burbujas pueden ser causadas por el aire atrapado en el sistema o por la formación de CO2 gaseoso. El primero se puede evitar fácilmente enjuagando el sistema con medio antes de insertar la muestra de célula, mientras que para evitar el segundo se recomienda disminuir la concentración de NaHCO3 en el medio de crecimiento y mantener todas las partes del sistema a una temperatura constante para minimizar las diferencias en la solubilidad de CO2 . El metabolismo aeróbico celular también puede causar la formación de CO2 gaseoso, que se puede prevenir aumentando el caudal.
La viabilidad de la celda debe verificarse después de cada ejecución de Trypan Blue Test. Sin embargo, eso no proporciona información sobre la actividad metabólica. Para obtener una imagen más completa del estado metabólico de las células durante la operación del biorreactor, se pueden realizar espectros de RMN 31P para evaluar la producción de ATP en función del tiempo23,25. Sin embargo, a menudo es necesaria una sonda dedicada para esta medición, lo que puede permitir el registro simultáneo con RMN de 1H.
En el caso de la CA II, la presencia de señales reporteras bien resueltas en una región inusual del espectro 1H facilita el análisis a partir de espectros simples de RMN 1D y no requiere enriquecimiento de isótopos durante la expresión de proteínas. En general, otras proteínas podrían dar lugar a señales 1H útiles para monitorizar cambios espectrales en otras regiones, como la típica de la proteína núcleo hidrófobo entre 0 y -1 ppm11; sin embargo, estas regiones tienden a estar abarrotadas de proteínas plegadas mayores de ~ 10 kDa. En este caso, como se muestra para SOD1, es preferible enriquecer la proteína con 15N, proporcionando un medio de crecimiento uniformemente enriquecido con 15N durante la expresión de la proteína, y monitorear los cambios en tiempo real en los espectros de RMN 2D 1H-15N. Los espectros 2D se importan como matrices 2D en MATLAB, se reorganizan en matrices 1D y se apilan antes del análisis MCR-ALS. Este último enfoque es generalmente aplicable a cualquier proteína intracelular que dé lugar a señales detectables, y proporciona información sobre los cambios conformacionales de la proteína a nivel de residuo único. En principio, este último enfoque puede generalizarse a los espectros nD y a otros esquemas de etiquetado de isótopos.
En cuanto a la aplicación a diferentes tipos de células, el protocolo debe adaptarse fácilmente a diferentes líneas celulares y no requiere que la proteína de interés se exprese directamente en las células. Por lo tanto, se pueden combinar otros enfoques de la RMN en la célula con este protocolo, en el que la macromolécula de interés se produce de forma recombinante, o sintetizada, y posteriormente se inserta en las células por electroporación o por otros métodos de administración1,9,38. Cuando se trabaja con diferentes líneas celulares o protocolos de preparación de muestras, es posible que sea necesario optimizar empíricamente parámetros como la concentración de agarosa, el grosor de la rosca y la densidad celular final en las roscas de agarosa. Además, la aplicabilidad del protocolo descrito aquí se limita a las células que toleran la encapsulación de agarosa. Otros tipos de células pueden requerir diferentes formulaciones de hidrogel, mientras que se recomienda una configuración diferente cuando se analizan células que crecen nativamente en suspensión, por ejemplo, haciendo uso de una membrana de microdiálisis coaxial para garantizar la difusión de nutrientes mientras se mantienen las células suspendidas confinadas en el tubo de RMN23.
En comparación con otros diseños de biorreactores de RMN19,20,21,22, el dispositivo descrito aquí se basa en una unidad de flujo disponible comercialmente, adaptada con modificaciones menores. Por lo tanto, el dispositivo se puede replicar fácilmente en diferentes laboratorios con alta reproducibilidad. Además, permite un funcionamiento estandarizado y el pleno cumplimiento de las estrictas normas de seguridad del laboratorio, si es necesario. En general, la flexibilidad y facilidad de operación del biorreactor debe permitir muchas otras aplicaciones de RMN en solución, tanto en células como in vitro, además de las ya reportadas23,25. Eventualmente, el mismo diseño de biorreactor podría aplicarse a muestras que se asemejan más al entorno fisiológico de un tejido, como esferoides u organoides, siempre que se encuentren andamios apropiados para mantener vivas dichas muestras, o incluso para mantener su crecimiento, en el espectrómetro de RMN.
Divulgaciones
Los autores declaran que no hay conflicto de intereses.
Agradecimientos
Este trabajo ha sido apoyado por iNEXT-Discovery, número de subvención 871037, financiado por el programa Horizonte 2020 de la Comisión Europea, por Instruct-ULTRA, número de subvención 731005, un proyecto H2020 de la UE para desarrollar aún más los servicios de Instruct-ERIC, y por Ministero dell'Istruzione, dell'Università e della Ricerca PRIN grant 20177XJCHX. Los autores reconocen el apoyo de Instruct-ERIC, un proyecto Landmark ESFRI, a través del Premio JRA número 815 y el uso de los recursos del Centro CERM /CIRMMP Italia. Agradecemos a Matteo Pennestri (Bruker, Reino Unido) por brindar apoyo para la operación de la unidad de flujo InsightMR.
Materiales
| Name | Company | Catalog Number | Comments |
| Materials | |||
| Citric acid | Sigma-Aldrich | 251275 | |
| D2O | Sigma-Aldrich | 453366 | |
| DMEM, high glucose | Life Technologies | 10313-021 | |
| DMEM, high glucose, powder | Sigma-Aldrich | D5648 | |
| FBS | Life Technologies | 10270 | |
| HCl | Sigma-Aldrich | 30721 | |
| L-glutamine (200 mM) | Life Technologies | 25030 | |
| Low-gelling agarose, powder | Sigma-Aldrich | A4018 | |
| NaHCO3, powder | Carlo Erba | 478537 | |
| PBS | Life Technologies | 10010 | |
| Penicillin–streptomycin (10,000 U/ml) | Life Technologies | 15140-122 | |
| NaOH, pellets | Sigma-Aldrich | 30620 | |
| Trypan Blue solution (0.4% (wt/vol) | Sigma-Aldrich | T8154 | Hazard statement(s): H350 may cause cancer. |
| Trypsin–EDTA (0.05% (wt/vol)) | Life Technologies | 25300-054 | |
| Equipment | |||
| Avance III Spectrometer equipped with a 5 mm CryoProbe | Bruker | n/a | All modern spectrometers and narrow-bore magnets equipped with 5 mm probes are compatible. |
| InsightMR flow unit | Bruker | n/a | |
| P-920 pump module from ÄKTA FPLC | GE Healthcare | n/a | Any FPLC, HPLC peristaltic or syringe pump should be compatible with the flow unit. |
Referencias
- Inomata, K., et al. High-resolution multi-dimensional NMR spectroscopy of proteins in human cells. Nature. 458 (7234), 106-109 (2009).
- Luchinat, E., Banci, L. In-cell NMR: a topical review. IUCrJ. 4, 108-118 (2017).
- Dzatko, S., et al. Evaluation of the stability of DNA i-motifs in the nuclei of living mammalian cells. Angewandte Chemie (International Ed. in English). 57 (8), 2165-2169 (2018).
- Luchinat, E., Banci, L. In-cell NMR in human cells: direct protein expression allows structural studies of protein folding and maturation. Accounts of Chemical Research. 51 (6), 1550-1557 (2018).
- Tanaka, T., et al. High-resolution protein 3D structure determination in living eukaryotic cells. Angewandte Chemie (International Ed. in English). 58 (22), 7284-7288 (2019).
- Siegal, G., Selenko, P. Cells, drugs and NMR. Journal of Magnetic Resonance. 306, 202-212 (1997).
- Banci, L., et al. Atomic-resolution monitoring of protein maturation in live human cells by NMR. Nature Chemical Biology. 9 (5), 297-299 (2013).
- Luchinat, E., et al. In-cell NMR reveals potential precursor of toxic species from SOD1 fALS mutants. Nature Communications. 5, 5502 (2014).
- Theillet, F. -. X., et al. Structural disorder of monomeric α-synuclein persists in mammalian cells. Nature. 530 (7588), 45-50 (2016).
- Barbieri, L., Luchinat, E., Banci, L. Intracellular metal binding and redox behavior of human DJ-1. Journal of biological inorganic chemistry: JBIC: A Publication of the Society of Biological Inorganic Chemistry. 23 (1), 61-69 (2018).
- Banci, L., Barbieri, L., Luchinat, E., Secci, E. Visualization of redox-controlled protein fold in living cells. Chemistry & Biology. 20 (6), 747-752 (2013).
- Mercatelli, E., Barbieri, L., Luchinat, E., Banci, L. Direct structural evidence of protein redox regulation obtained by in-cell NMR. Biochimica Et Biophysica Acta. 1863 (2), 198-204 (2016).
- Barbieri, L., Luchinat, E., Banci, L. Protein interaction patterns in different cellular environments are revealed by in-cell NMR. Scientific Reports. 5, 14456 (2015).
- Luchinat, E., et al. Drug screening in human cells by NMR spectroscopy allows the early assessment of drug potency. Angewandte Chemie (International Ed. in English). 59 (16), 6535-6539 (2020).
- Luchinat, E., et al. Intracellular binding/unbinding kinetics of approved drugs to carbonic anhydrase II observed by in-cell NMR. ACS Chemical Biology. 15 (10), 2792-2800 (2020).
- DeMott, C. M., et al. Potent inhibitors of mycobacterium tuberculosis growth identified by using in-cell NMR-based screening. ACS Chemical Biology. 13 (3), 733-741 (2018).
- Krafcikova, M., et al. Monitoring DNA-ligand interactions in living human cells using NMR spectroscopy. Journal of the American Chemical Society. 141 (34), 13281-13285 (2019).
- Broft, P., et al. In-cell NMR of functional riboswitch aptamers in eukaryotic cells. Angewandte Chemie (International Ed. in English). , (2020).
- Sharaf, N. G., Barnes, C. O., Charlton, L. M., Young, G. B., Pielak, G. J. A bioreactor for in-cell protein NMR. Journal of magnetic resonance. 202 (2), 140-146 (2010).
- Kubo, S., et al. A gel-encapsulated bioreactor system for NMR studies of protein-protein interactions in living mammalian cells. Angewandte Chemie (International Ed. in English). 52 (4), 1208-1211 (2013).
- Inomata, K., Kamoshida, H., Ikari, M., Ito, Y., Kigawa, T. Impact of cellular health conditions on the protein folding state in mammalian cells. Chemical Communications. 53 (81), 11245-11248 (2017).
- Breindel, L., DeMott, C., Burz, D. S., Shekhtman, A. Real-time in-cell nuclear magnetic resonance: ribosome-targeted antibiotics modulate quinary protein interactions. Biochemistry. 57 (5), 540-546 (2018).
- Cerofolini, L., et al. Real-time insights into biological events: in-cell processes and protein-ligand interactions. Biophysical Journal. 116 (2), 239-247 (2019).
- Breindel, L., Burz, D. S., Shekhtman, A. Active metabolism unmasks functional protein-protein interactions in real time in-cell NMR. Communications Biology. 3, (2020).
- Luchinat, E., Barbieri, L., Campbell, T. F., Banci, L. Real-time quantitative in-cell NMR: ligand binding and protein oxidation monitored in human cells using multivariate curve resolution. Analytical Chemistry. 92 (14), 9997-10006 (2020).
- Koczula, K. M., et al. Metabolic plasticity in CLL: adaptation to the hypoxic niche. Leukemia. 30 (1), 65-73 (2016).
- Alshamleh, I., et al. Real-time NMR spectroscopy for studying metabolism. Angewandte Chemie (International Ed. in English). 59 (6), 2304-2308 (2020).
- Supuran, C. T. Carbonic anhydrases: novel therapeutic applications for inhibitors and activators. Nature Reviews. Drug Discovery. 7 (2), 168-181 (2008).
- Neri, D., Supuran, C. T. Interfering with pH regulation in tumours as a therapeutic strategy. Nature Reviews. Drug Discovery. 10 (10), 767-777 (2011).
- Alterio, V., Di Fiore, A., D'Ambrosio, K., Supuran, C. T., De Simone, G. Multiple binding modes of inhibitors to carbonic anhydrases: how to design specific drugs targeting 15 different isoforms. Chemical Reviews. 112 (8), 4421-4468 (2012).
- Capper, M. J., et al. The cysteine-reactive small molecule ebselen facilitates effective SOD1 maturation. Nature Communications. 9 (1), 1693 (2018).
- Trist, B., Hilton, J. B., Crouch, P. J., Hare, D. J., Double, K. L. Superoxide dismutase 1 in health and disease: How a front-line antioxidant becomes neurotoxic. Angewandte Chemie (International Ed. in English). , (2020).
- Tauler, R. Multivariate curve resolution applied to second order data. Chemometrics and Intelligent Laboratory Systems. 30 (1), 133-146 (1995).
- Barbieri, L., Luchinat, E., Banci, L. Characterization of proteins by in-cell NMR spectroscopy in cultured mammalian cells. Nature Protocols. 11 (6), 1101-1111 (2016).
- Piotto, M., Saudek, V., Sklenár, V. Gradient-tailored excitation for single-quantum NMR spectroscopy of aqueous solutions. Journal of biomolecular NMR. 2 (6), 661-665 (1992).
- Vasa, S. K., Singh, H., Grohe, K., Linser, R. Assessment of a large enzyme-drug complex by proton-detected solid-state NMR spectroscopy without deuteration. Angewandte Chemie (International Ed. in English). 58 (17), 5758-5762 (2019).
- Schanda, P., Brutscher, B. Very fast two-dimensional NMR spectroscopy for real-time investigation of dynamic events in proteins on the time scale of seconds. Journal of the American Chemical Society. 127 (22), 8014-8015 (2005).
- Ogino, S., et al. Observation of NMR signals from proteins introduced into living mammalian cells by reversible membrane permeabilization using a pore-forming toxin, streptolysin O. Journal of the American Chemical Society. 131 (31), 10834-10835 (2009).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados