Method Article
Monitoramento de interações proteína-ligantes em células humanas por NMR quantitativo em tempo real usando um bioreator de alta densidade celular
Neste Artigo
Resumo
Este protocolo descreve a configuração de um bioreator NMR para manter as células humanas encapsuladas viáveis por até 72 h, seguida por aquisição e análise de dados NMR resolvidos no tempo. A metodologia é aplicada para monitorar interações proteína-ligantes intracelulares em tempo real.
Resumo
A RMN in-cell é uma abordagem única para observar as propriedades estruturais e dinâmicas das macromoléculas biológicas na resolução atômica diretamente em células vivas. Podem ser observadas dobras proteicas, modificações químicas e alterações conformais induzidas pela ligação de ligantes. Portanto, este método tem grande potencial no contexto do desenvolvimento de drogas. No entanto, a curta vida útil das células humanas confinadas no espectrômetro NMR limita a faixa de aplicação da RMN in-cell. Para superar esse problema, são empregados bioreatores de RN que podem melhorar muito a estabilidade da amostra celular ao longo do tempo e, importante, permitir o registro em tempo real de espectros de RMN em células. Dessa forma, a evolução de processos como penetração de ligantes e vinculação ao alvo da proteína intracelular pode ser monitorada em tempo real. Os bioreatores são frequentemente limitados pela baixa viabilidade celular em números de células altas, o que resulta em uma troca entre a sensibilidade geral do experimento e a viabilidade celular. Recentemente, relatamos um bioreator NMR que mantém um alto número de células humanas metabolicamente ativas por longos períodos de tempo, até 72 h. Esta configuração foi aplicada para monitorar interações proteína-ligantes e modificação química proteica. Também introduzimos um fluxo de trabalho para análise quantitativa dos dados de RMN em tempo real, com base na resolução de curvas multivariadas. O método fornece perfis de concentração das espécies químicas presentes nas células em função do tempo, que podem ser posteriormente analisados para obter parâmetros cinéticos relevantes. Aqui fornecemos uma descrição detalhada da configuração do bioreator NMR e sua aplicação para monitorar interações proteína-ligantes em células humanas.
Introdução
A espectroscopia de ressonância magnética nuclear (NMR) em células surgiu recentemente como uma abordagem poderosa para investigar propriedades estruturais e dinâmicas de macromoléculas no ambiente celular1,2,3,4,5,6. A RMN in-cell conseguiu na investigação de processos funcionalmente relevantes, como dobramento/dobra de proteína7,8,9, ligação metálica7,10, formação de ligação dissulfeto11,12, interação proteína-proteína13, interação proteína-ligante14,15,16 e interação ácido-ligante nucleico17 18 em células humanas vivas. Um dos fatores limitantes das aplicações de RMN na célula é a curta vida útil das células durante o experimento. A solução para este problema envolve o uso de bioreatores de RMN. Nesses dispositivos, é aplicado um fluxo constante de meio de crescimento às células, que são mantidas confinadas dentro do espectrômetro NMR, a fim de fornecer oxigênio e nutrientes e remover subprodutos tóxicos. Após o advento da RMR in-cell, vários projetos de bioreatores de RM Foram desenvolvidos para melhorar a viabilidade celular por períodos mais longos de tempo, nos quais bactérias ou células mamíferas são encapsuladas em um hidrogel19,20,21,22 ou mantidas em suspensão e perfumadas através do uso de uma membrana de microdiálise23 . Tais bioreatores permitiram a aquisição de experimentos de RMM mais longos com maior relação sinal-ruído (S/N)5 e, ainda mais importante, poderiam ser empregados para investigar processos celulares em tempo real22,23,24. Graças à alta sensibilidade química e conformacional da RMN, esta última aplicação pode fornecer insights preciosos sobre a cinética dos processos funcionais dentro das células vivas em resolução atômica.
Neste protocolo, mostramos como configurar e operar um bioreator melhorado recentemente relatado25, que foi obtido pela combinação de um projeto bioreator modular existente23 com a abordagem que conta com o encapsulamento celular em hidrogel que foram pioneiros por outros grupos19,20,21,22,26,27 . Descrevemos a aplicação do bioreator a estudos de RMN em tempo real de proteína intracelular observar ligante em células HEK293T. No bioreator, as células são encapsuladas em alta densidade em fios de gel agarose e são mantidas altamente viáveis e metabolicamente ativas por até 72 h, durante as quais experimentos de RMN em tempo real são registrados. O bioreator é composto por um tubo de vidro que se encaixa em sondas NMR padrão de 5 mm que são impermeáveis e conectadas a um suporte de tubo para que a câmara de amostra interna tenha 4,2 mm de diâmetro interno, 38 mm de altura e um volume de 526 μL. A entrada é um capilar PEEK de 7 metros de comprimento (o.d. = 1/32", i.d. = 0,5 mm) inserido na câmara de amostra até ~6 mm da parte inferior, enquanto a tomada é um capilar PTFE de 7 metros de comprimento (o.d. = 1/32", i.d. = 0,5 mm) anexado na parte superior do suporte do tubo (Figura 1). A tubulação é inserida coaxially em uma linha controlada pela temperatura conectada a um banho de água. A entrada e a saída são conectadas através de tubos PEEK a uma válvula de 4 vias e 2 posições presa a uma bomba FPLC para controlar o fluxo médio e um recipiente de resíduos.
O bioreator é aplicado para estudar a cinética da interação, relatada anteriormente14,25, entre duas drogas, acetazolamida (AAZ) e metanfetamina (MZA), em células humanas com a segunda isoforme da anidrato carbônico humano (CA II), um alvo farmacologicamente relevante28,29,30, e a cinética da formação da ligação de dissulfeto intramolecular, promovida pela pequena molécula ebselen25, 31, da forma livre de cobre, ligada ao zinco de cobre humano, desmutase de superóxido de zinco (SOD1), uma enzima antioxidante implicada no início da esclerose lateral amiotrófica7,8,32. Finalmente, a análise quantitativa dos dados de RMN em tempo real é realizada no MATLAB utilizando o algoritmo Multivariate Curve Resolution-Alternating Least Squares (MCR-ALS)33, através do qual os componentes espectrais puros e os perfis de concentração em função do tempo são obtidos para as espécies observadas, que podem ser ainda mais analisados para obter parâmetros cinéticos relevantes.
O protocolo começa a partir de um frasco T75 de células HEK293T (~3 x 107 células por frasco) transitavelmente superexpressando ca II humano (sem rótulo) ou humano SOD1 (15N-rotulado). As células foram cultivadas e mantidas em frascos T75 com dMEM de alta glicose por 1:10 passagens a cada 3-4 dias e transfeinadas com o cDNA codificando a proteína de interesse 48 h antes do experimento. As etapas envolvidas nesta fase são relatadas detalhadamente em outros lugares34.
Protocolo
1. Configuração de reagente e solução
- Para preparar dMEM completo, adicione 5 mL L-glutamina 200 mM, penicilina-estreptomicina 100x e 50 mL de bovino fetal (FBS, concentração final de 10% vol/vol) a 440 mL DMEM.
NOTA: Esta solução pode ser armazenada a 4 °C por 1 mês. - Prepare a solução agarose dissolvendo 150 mg de agarose de baixa gelagem em 10 mL de salina tamponada de fosfato (PBS) a 85 °C para obter uma solução de 1,5% (w/v). Esterilize por filtragem com um filtro de 0,22 μm. Prepare 1 mL de solução agarose em tubos tampados de 1,5 mL e armazene a 4 °C.
- Prepare o meio bioreator.
- Dissolva 13,4 g de pó DMEM em 1 L de ultrapure H2O.
NOTA: Dependendo da aplicação, o volume final necessário pode diferir (por exemplo, para o meio de 500 mL, dissolver 6,7 g de pó em 500 mL H2O). - Adicionar 2% FBS, 10 mM NaHCO3, 1x penicilina-estreptomicina (100x), e 2% D2O (por exemplo, para 500 mL médio, adicionar 10 mL de FBS, 0,4 g de NaHCO3, 5 mL de penicilina-estreptomicina 100x, e 10 mL de D2O).
- Meça o pH usando um medidor de pH e, se necessário, ajuste para 7,4 adicionando HCl.
NOTA: Normalmente, o pH inicial é muito próximo de 7,4. - Filtre o meio bioreator com um filtro estéril movido a vácuo em uma garrafa de vidro estéril de 250 mL ou 500 mL.
- Na coifa de fluxo laminar, sele a garrafa com um fone de ouvido de aço estéril com dois bicos de mangueira e conecte-os a uma tubulação FEP (o.d. = 1/8", i.d. = 1,6 mm) que será conectado à bomba e a um filtro de seringa PTFE de 0,22 μm para entrada de ar.
- Dissolva 13,4 g de pó DMEM em 1 L de ultrapure H2O.
2. Configuração do bioreator
- Monte a unidade de fluxo usando um tubo NMR de segunda unidade de fluxo, que será posteriormente substituído pelo que contém as células. Consulte as instruções de operação da unidade de fluxo para o conjunto correto.
NOTA: Neste momento, a unidade de fluxo já deve ser limpa (caso não, realize a etapa 4.2). - Coloque o banho de água conectado ao controle de temperatura da unidade de fluxo para 37 °C. Coloque a garrafa do reservatório no banho de água.
- Conecte a tubulação FEP da garrafa do reservatório à bomba.
- Gire a válvula bioreatorial para "contornar" e pré-enchimento da bomba com meio.
- Gire a válvula bioreatorial para "fluxo" e pré-enchia o bioreator com médio a 0,1 mL/min.
3. Preparação da amostra celular
- Recolhe as células da incubadora de CO2 .
- Pegue um frasco T75 de células HEK293T transfectadas da incubadora de CO2 e remova o meio gasto.
- Lave as células duas vezes com 7 mL (cada) de PBS à temperatura ambiente (~20 °C).
- Use 2 mL de trippsina/EDTA para desprender células. Depois de adicionar a solução, incubar por 5 minutos à temperatura ambiente para desprender as células.
NOTA: As células transfeinadas podem demorar um pouco mais para se desprender. Se necessário, incubar as células a 37 °C. - Inativar trippsina com 20 mL de DMEM completo; resuspensar completamente as células pipetando para cima e para baixo e transferi-las em um tubo de centrífuga de 50 mL.
- Centrifugar as células a 800 x g por 5 min à temperatura ambiente e descartar o supernatante.
- Lave as células com 10 mL de PBS à temperatura ambiente para remover o meio residual.
- Centrifugar as células a 800 x g por 5 min à temperatura ambiente e descartar o supernatante.
- Transfira a pelota celular para um tubo de microcentrifus de tampa de 1,5 mL.
- Incorporar células em fios de ágarose.
- Derreta uma alíquota de agarose solidificada a 85 °C em um banho de água e, posteriormente, mantenha-a em solução a 37 °C em um aquecedor de bloco.
- Com uma pipeta Pasteur, encha a parte inferior do tubo NMR da unidade de fluxo com 60-70 μL de gel de 1,5% de agarose e coloque-o no gelo. Isso criará um plugue de fundo de ~5 mm de altura que permite colocar a amostra de célula dentro do volume ativo da bobina 1H NMR.
- Aqueça a pelota de células obtidas na etapa 3.1.8 a 37 °C para 15-20 s no termbloco.
- Células resuspend em 450 μL de solução agarose. Tenha cuidado para evitar a formação de bolhas.
- Aspire a suspensão de célula-agarose em um tubo peek de cromatografia de ~30 cm de comprimento (i.d. = 0,75 mm) conectado a uma seringa de 1 mL.
NOTA: Antes da aspiração, o tubo e o volume morto da seringa devem ser pré-preenchidos com PBS à temperatura ambiente para evitar a formação de bolhas. O comprimento da tubulação não é crítico. - Deixe a tubulação esfriar à temperatura ambiente por 2 minutos.
- Preenche o tubo NMR da unidade de fluxo com 100 μL de PBS à temperatura ambiente.
- Fios fundidos de células embutidas na agarose no tubo NMR da unidade de fluxo empurrando suavemente a seringa.
NOTA: Para encher o tubo NMR de forma homogênea, comece colocando a extremidade da tubulação PEEK na parte inferior do tubo NMR e prossiga em direção ao topo enquanto balança lentamente para a esquerda para a direita. - Repetição de passos 3.2.5, 3.2.6 e 3.2.8 até que toda a suspensão de agarose de célula tenha sido lançada.
- Insira células no bioreator.
- Remova o tubo de RMN vazio da unidade de fluxo e aumente a vazão para 2 mL/min por alguns minutos para remover bolhas residuais de gás na tubulação de entrada.
- Defina a vazão para 0,2 mL/min e insira o tubo NMR contendo as células empurrando-a para cima lentamente, mas de forma constante.
NOTA: O fluxo ativo do meio evita o fluxo de recuo do conteúdo do tubo através da entrada, o que ocorreria de outra forma durante a inserção.
4. Operação e limpeza de bioreatores
- Operação bioreatorial durante o experimento NMR.
- Coloque a temperatura no espectrômetro NMR para 310 K.
- Insira a unidade de fluxo no espectrômetro.
- Forneça o meio bioreator a uma taxa de fluxo de 0,1 mL/min durante toda a duração dos experimentos de RMR na célula.
- No momento desejado durante o experimento, injete uma solução concentrada de molécula externa para a garrafa média do reservatório perfurando a tubulação de silicone com uma seringa de agulha longa estéril.
NOTA: A concentração final da molécula no meio deve ser escolhida com base no conhecimento prévio da toxicidade celular e, se disponível, na taxa de difusão prevista/estimada através da membrana celular. - No final do experimento NMR, substitua o tubo contendo as células por um tubo vazio e enxágue a unidade de fluxo com água.
- Bioreator limpo no local.
- Limpe a unidade de fluxo fluindo as seguintes soluções a 1 mL/min: hidróxido de sódio de 0,2 M (NaOH); Ácido cítrico de 3 M; 0,2 M NaOH, por pelo menos 30 min cada, seguido por água ultrapura filtrada estéril por >2 h.
- Limpe e autoclave a garrafa do reservatório e o conjunto de tubos após cada execução.
5. Experimentos de RMN
- Configuração dos experimentos de RMN.
NOTA: Realize estas etapas com antecedência, antes da elaboração da amostra de RMM celular, para evitar quaisquer atrasos entre a coleta celular e a aquisição de dados.- Crie um novo conjunto de dados no espectrômetro NMR e defina os parâmetros para os experimentos de RMN desejados.
- Defina parâmetros para experimentos 1D 1H NMR.
- Centralizar a frequência portadora 1H a 4,7 ppm no sinal de água.
- Selecione o programa de pulso zgesgp, defina a largura espectral para 20 ppm e um pulso quadrado de 1.000 μs e 180° para supressão de água. Defina um atraso de inter-digitalização de 1 s. Adquira o espectro com 32 varreduras.
- Para células expressando CA II não rotulado: selecione o programa de pulso p3919gp, defina a largura espectral para 30 ppm para cobrir a região imino do espectro e ajuste o atraso para supressão de água binomial para que a máxima excitação esteja centrada nos turnos químicos dos sinais de interesse (d7 = 20 μs a 950 MHz). Defina um atraso de inter-digitalização de ≥1 s. Adquira com 512 exames.
- Para células expressando SOD1 com etiqueta 15N: selecione o programa de pulso sfhmqf3gpph, definir as larguras espectrais de 1H e 15N para 16 e 50 ppm, respectivamente, o deslocamento de pulso moldado e a largura de banda de excitação para 8,5 e 6 ppm, respectivamente, e um pulso de 350 μs para esquema de desacoplamento (garp4 ou outro, dependendo do instrumento). Defina um atraso de inter-digitalização de 0,3 s. Adquira com 16 scans e 128 incrementos na dimensão 15N.
- Aquisição de espectros NMR em tempo real.
- Uma vez que o bioreator esteja inserido no espectrômetro NMR, espere alguns minutos para permitir a troca do meio.
NOTA: Este processo é facilmente monitorado a partir do aparecimento do sinal de bloqueio, pois o PBS é substituído por meio contendo 2% D2O. - Ajuste a correspondência e a sintonia do canal 1H, shim o ímã e calcule o comprimento do pulso duro de 1H 90°.
- Ajuste os níveis de alimentação 1H em cada sequência de pulso de acordo com o pulso rígido 1H.
- Registo um primeiro espectro zgesgp 1H para verificar o conteúdo da amostra e a homogeneidade de campo.
- Copie os experimentos zgesgp e p3919gp/sfhmqcf3gpph para o número desejado e enfie-os no spooler de aquisição.
NOTA: Os espectros zgesgp são usados apenas para controlar o estado da amostra e a homogeneidade de campo; portanto, eles podem ser pulados ou registrados com menos frequência. - Para células expressando CA II não rotulado: processe o espectro p3919gp aplicando zero preenchimento e função de janela de ampliação exponencial da linha (LB = 20 Hz).
- Para células expressas 15N rotuladas DE SOD1: processe o espectro sfhmqcf3gpph aplicando zero preenchimento e função de janela de sino seno quadrado (SSB = 2) em ambas as dimensões.
NOTA: O tamanho do espectro processado pode ser ainda mais reduzido removendo regiões livres de sinais (em Topspin, isso é feito definindo os valores STSR e STSI desejados).
- Uma vez que o bioreator esteja inserido no espectrômetro NMR, espere alguns minutos para permitir a troca do meio.
6. Análise MCR-ALS
- Para a análise dos espectros CA II, importe regiões espectrais 1D no MATLAB R2019b.
- No software, crie uma lista de experimentos a serem exportados no menu Lista de Configuração de Dados do processo .
- Utilizando uma versão modificada do programa au convbin2asc, exporte a região espectral de interesse no formato ASCII para cada espectro.
NOTA: Isso cria um arquivo de texto chamado ascii-spec.txt em cada subdiretório de espectro. - No MATLAB, importe as regiões espectrais usando o script personalizado Load_ascii_spectra.
NOTA: Este script requer o diretório de conjunto de dados como uma entrada e produz um espectro de matriz 2D contendo o espectro 1D empilhado e um conjunto de cs de matriz 1D contendo as mudanças químicas. - Execute o script Load_acqus para extrair os horários do espectro 1D.
NOTA: Este script produz uma matriz 1D times_hours contendo o incremento de tempo para cada espectro expresso em horas, com o espectro inicial no momento = 0.
- Para a análise do espectro SOD1, importe espectros 2D em MATLAB R2020b.
- No Topspin, crie uma lista de experimentos a serem exportados no menu Lista de Configuração de Dados do processo .
- No MATLAB, importe o espectro 2D usando o script personalizado Load_2D_spectra.
NOTA: Este script requer o diretório de conjunto de dados como uma entrada e produz um Spectra de matriz 3D contendo o espectro 2D empilhado e um cs de matriz 1D contendo as mudanças químicas. - Execute o script Load_acqus para extrair os horários do espectro 2D.
- Especifique as regiões espectrais de interesse no script personalizado Cut_2D_spectra e execute o script para cortar sub arrays 3D [(1H x 15N) intensidades espectrais x tempo]; remodelá-los como matrizes 2D (pontos de tempo x intensidades espectrais) e junte-os.
NOTA: Isso produz uma matriz 2D JoinSpec_flat contendo as regiões espectrais remodeladas e unidas.
- Execute o MCR-ALS 2.0 no modo GUI.
- Abra o MCR-ALS 2.0 GUI executando o script mcr_main.
- Na guia Seleção de dados , carregue o espectro ou a matriz JoinSpec_flat. Os dados podem ser plotados para verificação.
- Avalie o número de componentes por Decomposição de Valor Singular (SVD) ou manualmente.
NOTA: O número de componentes deve corresponder ao número de espécies distintas presentes no experimento. Neste caso n = 2, correspondente à proteína livre e vinculada. - Selecione um método para a estimativa inicial do espectro puro. A detecção variável mais pura ou a Análise de Fatores Em Evolução (EFA) podem ser utilizadas.
- Na janela Seleção do Conjunto de dados , selecione Continuar.
- Defina as restrições para as concentrações na janela Restrições: Modo de linha . Aplique uma restrição de não negatividade, selecione fnnls (quadrados menos restritos à nonnegatividade rápida) como "implementação e 2 espécies". Aplique 1 restrição de fechamento, defina a restrição para 1, a condição de fechamento como "igual a" e aplique a todas as espécies.
NOTA: Isso força a soma das concentrações de cada espécie a ser igual a 1, de modo que os perfis obtidos de cada espécie sejam normalizados em relação à concentração total de proteínas. - Defina as restrições para o espectro na janela Restrições: Modo coluna . Aplique uma restrição de não negatividade, selecione fnnls como "implementação e 2 espécies".
NOTA: Esta restrição não deve ser aplicada se sinais negativos estiverem presentes no espectro NMR. - Na janela final, defina 50 iterações e critério de convergência de 0,01. Especifique os nomes de saída para concentrações, espectro e desvio de Dst. Clique em Continuar a executar o encaixe MCR-ALS.
NOTA: Uma janela gráfica mostrará o resultado do encaixe com parcelas dos perfis de concentração e dos espectros dos componentes puros. - Para o conjunto de dados SOD1: use o script personalizado Rebuild_2D_spectra para reconstruir as regiões espectrais 2D a partir da saída 1D do MCR-ALS e plotá-las.
7. Teste azul trypan
- Recupere o conteúdo do tubo NMR com uma tubulação Pasteur e transfira os fios de agarose para um tubo tampado de 1,5 mL.
- Remova o meio residual enxaguando os fios de agarose com 600 μL de PBS e centrifuá-los a 4.000 x g por 1 min à temperatura ambiente. Descarte o supernatante.
- Adicione 250 μL de PBS e 50 μL de solução azul Trypan de 0,4%.
- Incubar por 2 min com tubulação contínua.
- Lave duas vezes com 600 μL de PBS descartando o supernaspetivo.
- Coloque alguns fios de agarose em um slide de microscópio e corte-os com lâminas de barbear para criar pequenas fatias de gel. Selecione as fatias mais finas (espessura < 0,4 mm, idealmente ~0,2 mm) para a análise.
- Transfira as fatias de gel para uma câmara de contagem de células auto-fabricadas composta por dois slides de vidro espaçados por três camadas de filme de parafina (~0,4 mm de espessura total) em cada lado.
NOTA: Um slide de câmara também pode ser usado; no entanto, as fatias de gel mais grossas que a altura da câmara (0,1 mm) seriam espremidas, rompendo as células incorporadas. - Adquira imagens de células dentro da ágarose e conte células brancas e azuis.
- Calcular a viabilidade celular como (células totais - células azuis) / células totais.
Resultados
O protocolo acima permite o encapsulamento de células em fios de hidrogel para maximizar a viabilidade celular por longos períodos de tempo, necessário para investigar em processos intracelulares em tempo real. No bioreator, as células são mantidas vivas e metabolicamente ativas até 72 h, conforme confirmado pelo teste azul trypan (Figura 2a-c). Em princípio, este protocolo pode ser aplicado para observar uma proteína intracelular de interesse submetida a quaisquer alterações conformais ou químicas. Na primeira aplicação descrita acima, o bioreator é aplicado para monitorar em tempo real a ligação de dois inibidores, AAZ e MZA, para CA II superexpresso no citosol das células HEK293T. O primeiro espectro de escultura de excitação 1H (zgesgp) registrado é usado para avaliar a intensidade geral do sinal (que é proporcional ao número de células), a presença de sinais da proteína superexpressa e da homogeneidade de campo (Figura 2d). No caso do CA II, a ligação intracelular dos dois inibidores pode ser monitorada pelo espectro WATERGATE 3-9-1935 1D 1H NMR (p3919gp), observando sinais 1H na região entre 11 e 16 ppm. Esses sinais surgem das histidinações coordenadas pelo zinco e outros resíduos aromáticos do CA II36 e são perturbados com a ligação de ligadura14,15. As concentrações de ligantes podem ser escolhidas com base na taxa de difusão ou, se disponível, a partir de valores de permeabilidade previamente determinados14. A vinculação bem sucedida é confirmada visualmente pelo aparecimento de um conjunto adicional de sinais na região espectral de interesse, que gradualmente substitui os sinais originais (Figura 3). As curvas de ligação dependentes do tempo são obtidas pela análise MCR-ALS, que separa os dois conjuntos de sinais de RMN decorrentes de CA II livre e vinculado (Figura 4a) e fornece simultaneamente os perfis de concentração relativos das duas espécies (Figura 4b). Na segunda aplicação, o bioreator é aplicado para monitorar a formação de ligação de dissulfeto intramolecular ligado ao zinco promovida pela ebselen, um mimético de glutationa peroxidase, em células humanas. Este processo é monitorado observando alterações nos espectros 1H-15N 2D SOFAST-HMQC37 (que fornece uma impressão digital da conformação da coluna vertebral proteica) causada por perturbações da estrutura proteica induzida pela formação de vínculo de dissulfeto. Sinais adicionais decorrentes do SOD1 oxidado de dissulfeto aparecem no espectro 1S-15N e gradualmente substituem os do SOD1 reduzido por dissulfeto. A análise mcr-ALS sobre regiões selecionadas do espectro 2D separa os sinais decorrentes das duas espécies (Figura 5a) e fornece seus perfis de concentração relativos (Figura 5b). As curvas de concentração obtidas podem ser posteriormente analisadas por encaixe não linear para fornecer informações sobre a cinética dos processos em estudo25.
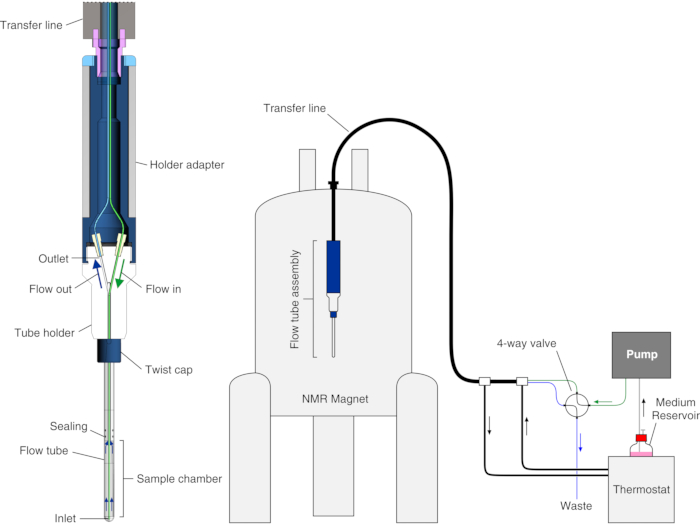
Figura 1: Esquema do bioreator. Esquerda: vista transversal da unidade de fluxo vazia. Direito: esquema da configuração do bioreator. A tubulação de entrada PEEK é mostrada em verde; a tubulação de saída PTFE é mostrada em azul. O painel esquerdo é reproduzido com permissão de Luchinat et al.25. Clique aqui para ver uma versão maior desta figura.
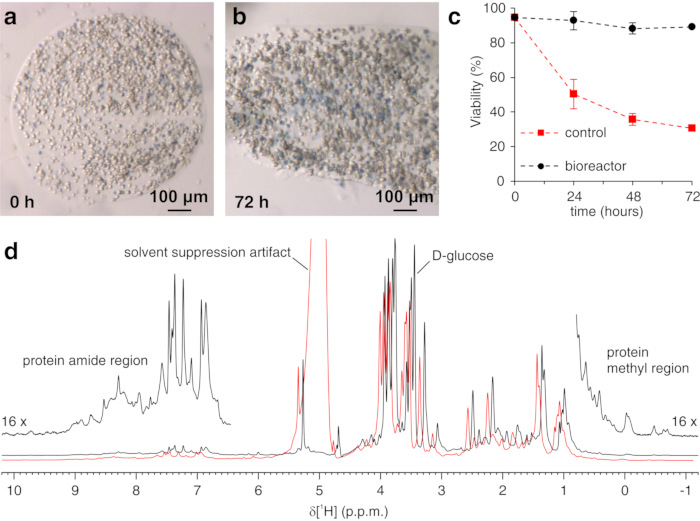
Figura 2: Teste Azul trypan em células encapsuladas e verificação de amostra por 1H NMR. Fatias representativas de agarose contendo células embutidas e manchadas com azul Trypan (a) imediatamente após fundição e (b) após 72 h no bioreator; c Viabilidade celular em função do tempo no bioreator NMR sob fluxo ativo (preto) e em condições estáticas (vermelho), medido pelo Trypan Blue Test. (d) zgesgp 1H NMR espectro registrado em células incorporadas agarose superexpressando CA II na ausência (preto) e na presença (vermelha) de bolhas de gás no bioreator. Neste último caso, a diminuição da homogeneidade de campo causa a ampliação da linha e o aparecimento de artefatos de supressão de solventes. Características espectrais interessantes são rotuladas. Os painéis (a-c) são reproduzidos com permissão de Luchinat et al.25. Clique aqui para ver uma versão maior desta figura.
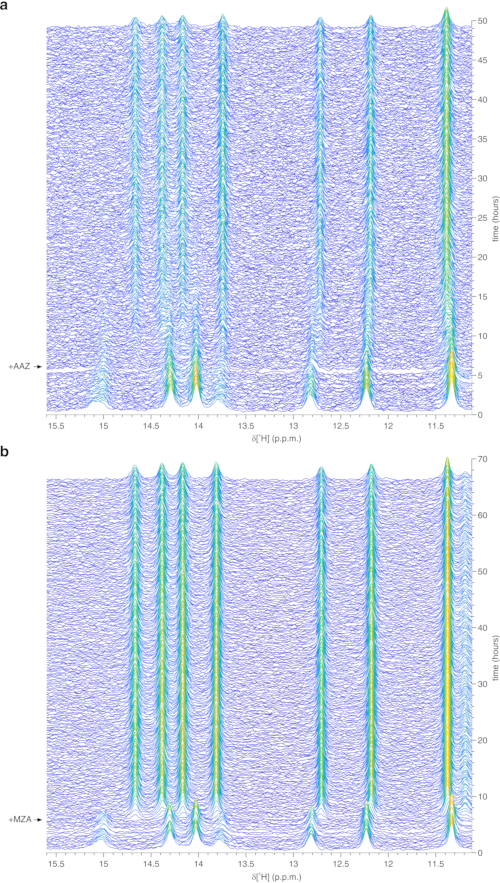
Figura 3: Dados representativos em tempo real de NMR 1H obtidos em células encapsuladas por agarose no bioreator. Parcelas de cachoeira de espectros 1D 1H NMR (região entre 15,6 e 11,1 ppm) de células HEK293T superexpressas CA II e posteriormente tratadas com (a) 25 μM AAZ e (b) 10 μM MZA, registradas em função do tempo no bioretor NMR. O tempo de tratamento de ligante é mostrado com uma flecha. A intensidade espectral (a.u.) é codificada por cores de azul (mais baixo) para amarelo (mais alto). Este valor é reproduzido com permissão de Luchinat et al.25. Clique aqui para ver uma versão maior desta figura.
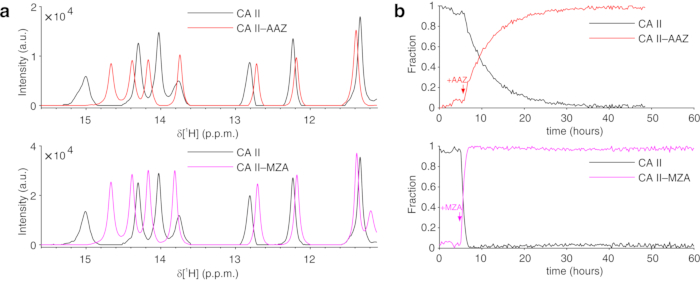
Figura 4: Produção representativa mcr-ALS a partir de espectros 1D NMR. (a) espectros de NMR 1H dos componentes puros reconstruídos pela MCR-ALS: CA II na ausência de ligantes (preto) e no complexo com AAZ (vermelho) ou MZA (magenta); b Perfis de concentração relativa de CA II livre (preto) e vinculados em função do tempo após a adição de AAZ (vermelho) ou MZA (magenta) obtidos pelo MCR-ALS. Os tempos de tratamento de ligante são marcados com flechas. Este valor é reproduzido com permissão de Luchinat et al.25. Clique aqui para ver uma versão maior desta figura.
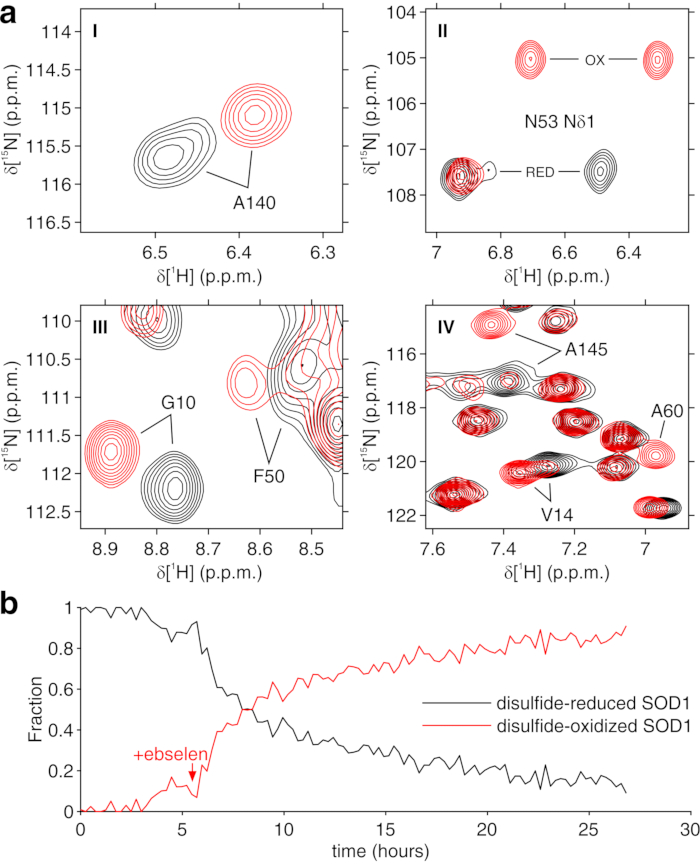
Figura 5: Produção representativa mcr-ALS de espectros 2D NMR. (a) regiões espectrais 1H-15N (rotulada I-IV) dos componentes puros reconstruídos pelo MCR-ALS: SOD1 reduzido por dissulfeto (preto) e SOD1 oxidado por dissulfeto (vermelho); b Perfil de concentração relativa dos componentes puros em função do tempo após a adição de ebselen (marcado com uma seta) obtido por MCR-ALS. Este valor é reproduzido com permissão de Luchinat et al.25. Clique aqui para ver uma versão maior desta figura.
Discussão
O objetivo de usar um bioreator para experimento de RMN em células é manter as células vivas e metabolicamente ativas por um período prolongado de tempo. Uma série de aspectos críticos devem ser levados em consideração para alcançar esse objetivo. Em primeiro lugar, é primordial evitar a contaminação bacteriana ao preparar a amostra celular e durante a aquisição de dados de RMN. Se as cepas de E. coli ou outras bactérias comumente usadas para clonagem genética e expressão de proteína recombinante forem usadas em laboratório, elas podem contaminar as células durante a preparação da amostra. Uma vez no bioreator, as bactérias crescerão rapidamente explorando o meio de crescimento fresco e causarão morte celular devido à produção de endotoxinas. A contaminação bacteriana só é detectada em um estágio avançado, quando transforma o crescimento médio amarelo e turvo. Além disso, a limpeza incompleta do bioreator pode causar contaminação da bomba ou do tubo com bactérias, leveduras ou moldes comuns.
Um requisito para o sucesso do experimento é evitar a formação de bolhas de gás. Bolhas de gás presas entre os fios de agarose no volume ativo da bobina NMR introduziriam grandes inhomogeneidades de campo magnético, causando supressão incompleta do sinal H2O e perda severa de qualidade espectral (Figura 2d). Bolhas podem ser causadas pelo ar preso no sistema ou pela formação de CO2 gasoso. O primeiro pode ser facilmente evitado lavando o sistema com meio antes de inserir a amostra celular, enquanto para evitar o segundo é recomendado diminuir a concentração de NaHCO3 no meio de crescimento, e manter todas as partes do sistema em temperatura constante para minimizar as diferenças na solubilidade do CO2. O metabolismo aeróbico celular também pode causar a formação de CO2 gasoso, que pode ser evitado aumentando a vazão.
A viabilidade celular deve ser verificada após cada execução do Trypan Blue Test. No entanto, isso não fornece insights sobre a atividade metabólica. Para obter um quadro mais completo do estado metabólico das células durante a operação do bioreator, os espectros de RMN 31P podem ser realizados para avaliar a produção de ATP em função do tempo23,25. No entanto, uma sonda dedicada é frequentemente necessária para esta medição, o que pode permitir a gravação simultânea com 1H NMR.
No caso do CA II, a presença de sinais repórteres bem resolvidos em uma região incomum do espectro 1H facilita a análise de espectro 1D simples de NMR e não requer enriquecimento de isótopos durante a expressão proteica. Em geral, outras proteínas poderiam dar origem a sinais 1H úteis para monitorar mudanças espectrais em outras regiões, como a típica do núcleo hidrofóbico proteico entre 0 e -1 ppm11; no entanto, essas regiões tendem a ser lotadas para proteínas dobradas maiores que ~10 kDa. Neste caso, como mostrado para SOD1, é preferível enriquecer a proteína com 15N, fornecendo uniformemente meio de crescimento enriquecido em 15N durante a expressão proteica, e para monitorar as mudanças em tempo real nos espectros 2D 1H-15N NMR. Os espectros 2D são importados como matrizes 2D no MATLAB, reorganizados para matrizes 1D e empilhados antes da análise MCR-ALS. Esta última abordagem é geralmente aplicável a qualquer proteína intracelular que dê origem a sinais detectáveis, e fornece informações sobre alterações conformais de proteínas no nível único de resíduo. Em princípio, esta última abordagem pode ser generalizada para espectros nD e para outros esquemas de rotulagem de isótopos.
Quanto à aplicação a diferentes tipos de células, o protocolo deve ser facilmente adaptado a diferentes linhas celulares e não exige que a proteína de interesse seja diretamente expressa nas células. Portanto, outras abordagens para a RMN in-cell podem ser combinadas com este protocolo, no qual a macromolécula de interesse é produzida recombinantemente, ou sintetizada, e posteriormente inserida nas células por eletroporação ou por outros métodos de entrega1,9,38. Ao trabalhar com diferentes linhas celulares ou protocolos de preparação de amostras, parâmetros como a concentração de agarose, a espessura do fio e a densidade celular final nos fios de agarose podem precisar ser otimizados empiricamente. Além disso, a aplicabilidade do protocolo aqui descrito limita-se às células que toleram o encapsulamento de ágaroses. Outros tipos de células podem exigir formulações diferentes de hidrogel, enquanto uma configuração diferente é recomendada ao analisar células que crescem nativamente em suspensão, por exemplo, fazendo uso de uma membrana de microdiálise coaxial para garantir a difusão de nutrientes, mantendo as células suspensas confinadas no tubo NMR23.
Em comparação com outros projetos bioreator NMR19,20,21,22, o dispositivo descrito aqui conta com uma unidade de fluxo comercialmente disponível, adaptada com pequenas modificações. Portanto, o dispositivo pode ser facilmente replicado em diferentes laboratórios com alta reprodutibilidade. Além disso, permite a operação padronizada e o cumprimento integral das rigorosas normas de segurança laboratorial, se necessário. No geral, a flexibilidade e facilidade de funcionamento do bioreator devem permitir muitas outras aplicações de solução NMR, tanto em células quanto in vitro, além daquelas já relatadas23,25. Eventualmente, o mesmo desenho bioreator poderia ser aplicado a amostras que se assemelham mais ao ambiente fisiológico de um tecido, como esferoides ou organoides, desde que andaimes apropriados sejam encontrados para manter tais amostras vivas ou mesmo sustentar seu crescimento no espectrômetro de NMR.
Divulgações
Os autores declaram que não há conflito de interesses.
Agradecimentos
Este trabalho foi apoiado pelo iNEXT-Discovery, número de subvenção 871037, financiado pelo programa Horizon 2020 da Comissão Europeia, pela Instruct-ULTRA, número de subvenção 731005, um projeto da UE H2020 para desenvolver ainda mais os serviços da Instruct-ERIC, e pelo Ministro dell'Istruzione, dell'Università e della Ricerca PRIN grant 20177XJX. Os autores reconhecem o apoio do Instruct-ERIC, um projeto da Landmark ESFRI, através do Prêmio JRA número 815 e o uso de recursos do Centro Itália CERM/CIRMMP. Agradecemos a Matteo Pennestri (Bruker, Reino Unido) por fornecer suporte para a operação da unidade de fluxo InsightMR.
Materiais
| Name | Company | Catalog Number | Comments |
| Materials | |||
| Citric acid | Sigma-Aldrich | 251275 | |
| D2O | Sigma-Aldrich | 453366 | |
| DMEM, high glucose | Life Technologies | 10313-021 | |
| DMEM, high glucose, powder | Sigma-Aldrich | D5648 | |
| FBS | Life Technologies | 10270 | |
| HCl | Sigma-Aldrich | 30721 | |
| L-glutamine (200 mM) | Life Technologies | 25030 | |
| Low-gelling agarose, powder | Sigma-Aldrich | A4018 | |
| NaHCO3, powder | Carlo Erba | 478537 | |
| PBS | Life Technologies | 10010 | |
| Penicillin–streptomycin (10,000 U/ml) | Life Technologies | 15140-122 | |
| NaOH, pellets | Sigma-Aldrich | 30620 | |
| Trypan Blue solution (0.4% (wt/vol) | Sigma-Aldrich | T8154 | Hazard statement(s): H350 may cause cancer. |
| Trypsin–EDTA (0.05% (wt/vol)) | Life Technologies | 25300-054 | |
| Equipment | |||
| Avance III Spectrometer equipped with a 5 mm CryoProbe | Bruker | n/a | All modern spectrometers and narrow-bore magnets equipped with 5 mm probes are compatible. |
| InsightMR flow unit | Bruker | n/a | |
| P-920 pump module from ÄKTA FPLC | GE Healthcare | n/a | Any FPLC, HPLC peristaltic or syringe pump should be compatible with the flow unit. |
Referências
- Inomata, K., et al. High-resolution multi-dimensional NMR spectroscopy of proteins in human cells. Nature. 458 (7234), 106-109 (2009).
- Luchinat, E., Banci, L. In-cell NMR: a topical review. IUCrJ. 4, 108-118 (2017).
- Dzatko, S., et al. Evaluation of the stability of DNA i-motifs in the nuclei of living mammalian cells. Angewandte Chemie (International Ed. in English). 57 (8), 2165-2169 (2018).
- Luchinat, E., Banci, L. In-cell NMR in human cells: direct protein expression allows structural studies of protein folding and maturation. Accounts of Chemical Research. 51 (6), 1550-1557 (2018).
- Tanaka, T., et al. High-resolution protein 3D structure determination in living eukaryotic cells. Angewandte Chemie (International Ed. in English). 58 (22), 7284-7288 (2019).
- Siegal, G., Selenko, P. Cells, drugs and NMR. Journal of Magnetic Resonance. 306, 202-212 (1997).
- Banci, L., et al. Atomic-resolution monitoring of protein maturation in live human cells by NMR. Nature Chemical Biology. 9 (5), 297-299 (2013).
- Luchinat, E., et al. In-cell NMR reveals potential precursor of toxic species from SOD1 fALS mutants. Nature Communications. 5, 5502 (2014).
- Theillet, F. -. X., et al. Structural disorder of monomeric α-synuclein persists in mammalian cells. Nature. 530 (7588), 45-50 (2016).
- Barbieri, L., Luchinat, E., Banci, L. Intracellular metal binding and redox behavior of human DJ-1. Journal of biological inorganic chemistry: JBIC: A Publication of the Society of Biological Inorganic Chemistry. 23 (1), 61-69 (2018).
- Banci, L., Barbieri, L., Luchinat, E., Secci, E. Visualization of redox-controlled protein fold in living cells. Chemistry & Biology. 20 (6), 747-752 (2013).
- Mercatelli, E., Barbieri, L., Luchinat, E., Banci, L. Direct structural evidence of protein redox regulation obtained by in-cell NMR. Biochimica Et Biophysica Acta. 1863 (2), 198-204 (2016).
- Barbieri, L., Luchinat, E., Banci, L. Protein interaction patterns in different cellular environments are revealed by in-cell NMR. Scientific Reports. 5, 14456 (2015).
- Luchinat, E., et al. Drug screening in human cells by NMR spectroscopy allows the early assessment of drug potency. Angewandte Chemie (International Ed. in English). 59 (16), 6535-6539 (2020).
- Luchinat, E., et al. Intracellular binding/unbinding kinetics of approved drugs to carbonic anhydrase II observed by in-cell NMR. ACS Chemical Biology. 15 (10), 2792-2800 (2020).
- DeMott, C. M., et al. Potent inhibitors of mycobacterium tuberculosis growth identified by using in-cell NMR-based screening. ACS Chemical Biology. 13 (3), 733-741 (2018).
- Krafcikova, M., et al. Monitoring DNA-ligand interactions in living human cells using NMR spectroscopy. Journal of the American Chemical Society. 141 (34), 13281-13285 (2019).
- Broft, P., et al. In-cell NMR of functional riboswitch aptamers in eukaryotic cells. Angewandte Chemie (International Ed. in English). , (2020).
- Sharaf, N. G., Barnes, C. O., Charlton, L. M., Young, G. B., Pielak, G. J. A bioreactor for in-cell protein NMR. Journal of magnetic resonance. 202 (2), 140-146 (2010).
- Kubo, S., et al. A gel-encapsulated bioreactor system for NMR studies of protein-protein interactions in living mammalian cells. Angewandte Chemie (International Ed. in English). 52 (4), 1208-1211 (2013).
- Inomata, K., Kamoshida, H., Ikari, M., Ito, Y., Kigawa, T. Impact of cellular health conditions on the protein folding state in mammalian cells. Chemical Communications. 53 (81), 11245-11248 (2017).
- Breindel, L., DeMott, C., Burz, D. S., Shekhtman, A. Real-time in-cell nuclear magnetic resonance: ribosome-targeted antibiotics modulate quinary protein interactions. Biochemistry. 57 (5), 540-546 (2018).
- Cerofolini, L., et al. Real-time insights into biological events: in-cell processes and protein-ligand interactions. Biophysical Journal. 116 (2), 239-247 (2019).
- Breindel, L., Burz, D. S., Shekhtman, A. Active metabolism unmasks functional protein-protein interactions in real time in-cell NMR. Communications Biology. 3, (2020).
- Luchinat, E., Barbieri, L., Campbell, T. F., Banci, L. Real-time quantitative in-cell NMR: ligand binding and protein oxidation monitored in human cells using multivariate curve resolution. Analytical Chemistry. 92 (14), 9997-10006 (2020).
- Koczula, K. M., et al. Metabolic plasticity in CLL: adaptation to the hypoxic niche. Leukemia. 30 (1), 65-73 (2016).
- Alshamleh, I., et al. Real-time NMR spectroscopy for studying metabolism. Angewandte Chemie (International Ed. in English). 59 (6), 2304-2308 (2020).
- Supuran, C. T. Carbonic anhydrases: novel therapeutic applications for inhibitors and activators. Nature Reviews. Drug Discovery. 7 (2), 168-181 (2008).
- Neri, D., Supuran, C. T. Interfering with pH regulation in tumours as a therapeutic strategy. Nature Reviews. Drug Discovery. 10 (10), 767-777 (2011).
- Alterio, V., Di Fiore, A., D'Ambrosio, K., Supuran, C. T., De Simone, G. Multiple binding modes of inhibitors to carbonic anhydrases: how to design specific drugs targeting 15 different isoforms. Chemical Reviews. 112 (8), 4421-4468 (2012).
- Capper, M. J., et al. The cysteine-reactive small molecule ebselen facilitates effective SOD1 maturation. Nature Communications. 9 (1), 1693 (2018).
- Trist, B., Hilton, J. B., Crouch, P. J., Hare, D. J., Double, K. L. Superoxide dismutase 1 in health and disease: How a front-line antioxidant becomes neurotoxic. Angewandte Chemie (International Ed. in English). , (2020).
- Tauler, R. Multivariate curve resolution applied to second order data. Chemometrics and Intelligent Laboratory Systems. 30 (1), 133-146 (1995).
- Barbieri, L., Luchinat, E., Banci, L. Characterization of proteins by in-cell NMR spectroscopy in cultured mammalian cells. Nature Protocols. 11 (6), 1101-1111 (2016).
- Piotto, M., Saudek, V., Sklenár, V. Gradient-tailored excitation for single-quantum NMR spectroscopy of aqueous solutions. Journal of biomolecular NMR. 2 (6), 661-665 (1992).
- Vasa, S. K., Singh, H., Grohe, K., Linser, R. Assessment of a large enzyme-drug complex by proton-detected solid-state NMR spectroscopy without deuteration. Angewandte Chemie (International Ed. in English). 58 (17), 5758-5762 (2019).
- Schanda, P., Brutscher, B. Very fast two-dimensional NMR spectroscopy for real-time investigation of dynamic events in proteins on the time scale of seconds. Journal of the American Chemical Society. 127 (22), 8014-8015 (2005).
- Ogino, S., et al. Observation of NMR signals from proteins introduced into living mammalian cells by reversible membrane permeabilization using a pore-forming toxin, streptolysin O. Journal of the American Chemical Society. 131 (31), 10834-10835 (2009).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados