Method Article
Überwachung von Protein-Liganden-Interaktionen in menschlichen Zellen durch quantitative In-Zell-NMR in Echtzeit mit einem Bioreaktor mit hoher Zelldichte
In diesem Artikel
Zusammenfassung
Dieses Protokoll beschreibt den Aufbau eines NMR-Bioreaktors, um verkapselte menschliche Zellen bis zu 72 h lebensfähig zu halten, gefolgt von einer zeitaufgelösten NMR-Datenerfassung und -analyse in der Zelle. Die Methodik wird angewendet, um intrazelluläre Protein-Ligand-Interaktionen in Echtzeit zu überwachen.
Zusammenfassung
In-Cell-NMR ist ein einzigartiger Ansatz, um die strukturellen und dynamischen Eigenschaften biologischer Makromoleküle bei atomarer Auflösung direkt in lebenden Zellen zu beobachten. Proteinfaltung, chemische Modifikationen und Konformationsänderungen, die durch Ligandenbindung induziert werden, können beobachtet werden. Daher hat diese Methode ein großes Potenzial im Rahmen der Arzneimittelentwicklung. Die kurze Lebensdauer menschlicher Zellen, die im NMR-Spektrometer eingeschlossen sind, schränkt jedoch den Anwendungsbereich der In-Cell-NMR ein. Um dieses Problem zu lösen, werden NMR-Bioreaktoren eingesetzt, die die Stabilität der Zellproben im Laufe der Zeit erheblich verbessern und vor allem die Echtzeitaufzeichnung von NMR-Spektren in der Zelle ermöglichen können. Auf diese Weise kann die Entwicklung von Prozessen wie Ligandenpenetration und Bindung an das intrazelluläre Proteintarget in Echtzeit überwacht werden. Bioreaktoren sind oft durch eine geringe Zelllebensfähigkeit bei hohen Zellzahlen begrenzt, was zu einem Kompromiss zwischen der Gesamtempfindlichkeit des Experiments und der Zelllebensfähigkeit führt. Wir haben kürzlich über einen NMR-Bioreaktor berichtet, der eine hohe Anzahl menschlicher Zellen über einen längeren Zeitraum von bis zu 72 h metabolisch aktiv hält. Dieses Setup wurde angewendet, um Protein-Ligand-Wechselwirkungen und die chemische Modifikation von Proteinen zu überwachen. Wir haben auch einen Workflow für die quantitative Analyse der Echtzeit-NMR-Daten eingeführt, der auf der multivariaten Kurvenauflösung basiert. Die Methode liefert Konzentrationsprofile der in den Zellen vorhandenen chemischen Spezies in Abhängigkeit von der Zeit, die weiter analysiert werden können, um relevante kinetische Parameter zu erhalten. Hier geben wir eine detaillierte Beschreibung des NMR-Bioreaktor-Aufbaus und seiner Anwendung zur Überwachung von Protein-Ligand-Interaktionen in menschlichen Zellen.
Einleitung
Die In-Cell-Kernspinresonanzspektroskopie (NMR) hat sich kürzlich als leistungsfähiger Ansatz zur Untersuchung struktureller und dynamischer Eigenschaften von Makromolekülen in der zellulären Umgebung herausgestellt1,2,3,4,5,6. In-Cell-NMR gelang die Untersuchung funktionell relevanter Prozesse wie Proteinfaltung/Fehlfaltung7,8,9, Metallbindung7,10, Disulfidbindungsbildung11,12 und Protein-Protein-Interaktion13, Protein-Liganden-Interaktion14,15,16 und Nukleinsäure-Liganden-Interaktion17 ,18 in lebenden menschlichen Zellen. Einer der limitierenden Faktoren von In-Cell-NMR-Anwendungen ist die kurze Lebensdauer der Zellen während des Experiments. Die Lösung dieses Problems beinhaltet den Einsatz von NMR-Bioreaktoren. In diesen Geräten wird ein konstanter Fluss von Wachstumsmedium auf die Zellen aufgebracht, die im NMR-Spektrometer eingeschlossen gehalten werden, um Sauerstoff und Nährstoffe bereitzustellen und toxische Nebenprodukte zu entfernen. Nach dem Aufkommen der zellinternen NMR wurden mehrere NMR-Bioreaktordesigns entwickelt, um die Zelllebensfähigkeit über längere Zeiträume zu verbessern, in denen entweder Bakterien oder Säugetierzellen in einem Hydrogel eingekapselt werden19,20,21,22 oder in Suspension gehalten und durch die Verwendung einer Mikrodialysemembran perfundiert werden23 . Solche Bioreaktoren haben die Aufnahme längerer NMR-Experimente mit erhöhtem Signal-Rausch-Verhältnis (S/N)5 ermöglicht und, was noch wichtiger ist, könnten verwendet werden, um zelluläre Prozesse in Echtzeit zu untersuchen22,23,24. Dank der hohen chemischen und konformationsbedingten Sensitivität der NMR kann die letztgenannte Anwendung wertvolle Erkenntnisse über die Kinetik funktioneller Prozesse in lebenden Zellen bei atomarer Auflösung liefern.
In diesem Protokoll zeigen wir, wie ein verbesserter Bioreaktor eingerichtet und betrieben werden kann, über den kürzlich berichtet wurde25, der durch die Kombination eines bestehenden modularen Bioreaktordesigns23 mit dem Ansatz erhalten wurde, der auf der Zellverkapselung in Hydrogel beruht, die von anderen Gruppen entwickelt wurden19,20,21,22,26,27 . Wir beschreiben die Anwendung des Bioreaktors auf Echtzeit-In-Cell-NMR-Studien der intrazellulären Proteinbeobachtungsligandenbindung in HEK293T-Zellen. Im Bioreaktor werden die Zellen mit hoher Dichte in Agarosegelfäden eingekapselt und bis zu 72 h lang hochlebensfähig und metabolisch aktiv gehalten, wobei Echtzeit-In-Cell-NMR-Experimente aufgezeichnet werden. Der Bioreaktor besteht aus einem Glasröhrchen, in das standardmäßige 5-mm-NMR-Sonden passen, das wasserdicht ist und mit einem Röhrenhalter verbunden ist, so dass die interne Probenkammer einen Innendurchmesser von 4,2 mm, eine Höhe von 38 mm und ein Volumen von 526 μL hat. Der Einlass ist eine 7 Meter lange PEEK-Kapillare (o.d. = 1/32", i.d. = 0,5 mm), die bis zu ~6 mm von unten in die Probenkammer eingeführt wird, während der Auslass eine 7 Meter lange PTFE-Kapillare (o.d. = 1/32", i.d. = 0,5 mm) ist, die an der Oberseite des Röhrchenhalters angebracht ist (Abbildung 1). Der Schlauch wird koaxial in eine temperaturgesteuerte Leitung eingeführt, die mit einem Wasserbad verbunden ist. Der Ein- und Auslass ist über PEEK-Schläuche mit einem 4-Wege-2-Positionen-Ventil verbunden, das an eine FPLC-Pumpe zur Steuerung des Mediumstroms und eines Abfallbehälters angeschlossen ist.
Der Bioreaktor wird verwendet, um die Kinetik der zuvor berichteten14,25 Wechselwirkung zwischen zwei Arzneimitteln, Acetazolamid (AAZ) und Methazolamid (MZA), in menschlichen Zellen mit der zweiten Isoform der humanen Carboanhydrase (CA II), einem pharmakologisch relevanten Ziel28,29,30, und die Kinetik der Bildung der intramolekularen Disulfidbindung, gefördert durch das kleine Molekül Ebselen25, zu untersuchen. 31, der kupferfreien, zinkgebundenen Form von menschlichem Kupfer, Zink-Superoxid-Dismutase (SOD1), einem antioxidativen Enzym, das am Ausbruch der amyotrophen Lateralsklerose beteiligt ist7,8,32. Schließlich wird die quantitative Analyse der Echtzeit-NMR-Daten in MATLAB unter Verwendung des MCR-ALS-Algorithmus (Multivariate Curve Resolution-Alternating Least Squares)33 durchgeführt, durch den die reinen spektralen Komponenten und die Konzentrationsprofile als Funktion der Zeit für die beobachteten Spezies erhalten werden, die weiter analysiert werden können, um relevante kinetische Parameter zu erhalten.
Das Protokoll beginnt mit einem T75-Kolben von HEK293T-Zellen (~ 3 x 107 Zellen pro Kolben), die vorübergehend entweder menschliches CA II (unmarkiert) oder humanes SOD1 (15N-markiert) überexprimieren. Die Zellen wurden in T75-Kolben mit DMEM-hoher Glukose um 1:10 Passagen alle 3-4 Tage gezüchtet und gehalten und 48 h vor dem Experiment mit der cDNA, die für das Protein von Interesse kodiert, transfiziert. Über die Schritte in dieser Phase wird an anderer Stelle ausführlich berichtet34.
Protokoll
1. Reagens- und Lösungseinrichtung
- Um ein vollständiges DMEM herzustellen, fügen Sie 5 ml L-Glutamin 200 mM, 5 ml Penicillin-Streptomycin 100x und 50 ml fetales Rinderserum (FBS, 10% vol / vol Endkonzentration) zu 440 ml DMEM hinzu.
HINWEIS: Diese Lösung kann 1 Monat lang bei 4 °C gelagert werden. - Bereiten Sie Agaroselösung vor, indem Sie 150 mg niedrig gelierende Agarose in 10 ml phosphatgepufferter Kochsalzlösung (PBS) bei 85 °C auflösen, um eine 1,5%ige (w/v) ige Lösung zu erhalten. Sterilisieren Sie durch Filtration mit einem 0,22 μm Filter. Bereiten Sie 1 ml Aliquots Agaroselösung in 1,5 mL verschlossenen Röhrchen vor und lagern Sie sie bei 4 °C.
- Bereiten Sie das Bioreaktormedium vor.
- 13,4 g DMEM-Pulver werden in 1 l hochreinem H2O gelöst.
HINWEIS: Je nach Anwendung kann das erforderliche Endvolumen abweichen (z. B. bei 500 ml Medium 6,7 g Pulver in 500 mL H2O lösen). - Fügen Sie 2% FBS, 10 mM NaHCO3, 1x Penicillin-Streptomycin (100x) und 2% D2O hinzu (z. B. für 500 ml Medium 10 ml FBS, 0,4 g NaHCO3, 5 ml Penicillin-Streptomycin 100x und 10 ml D2O).
- Messen Sie den pH-Wert mit einem pH-Messgerät und stellen Sie ihn bei Bedarf durch Zugabe von HCl auf 7,4 ein.
HINWEIS: Typischerweise liegt der anfängliche pH-Wert sehr nahe bei 7,4. - Filtern Sie das Bioreaktormedium mit einem vakuumbetriebenen Sterilfilter in einer sterilen 250 mL oder 500 mL Glasflasche.
- In der Laminar-Flow-Haube verschließen Sie die Flasche mit einem sterilen Stahlkopfstück mit zwei Schlauchdüsen und schließen Sie sie an einen FEP-Schlauch (o.d. = 1/8", i.d. = 1,6 mm) an, der an die Pumpe angeschlossen wird, und an einen 0,22 μm PTFE-Spritzenfilter für die Luftansaugung.
- 13,4 g DMEM-Pulver werden in 1 l hochreinem H2O gelöst.
2. Aufbau des Bioreaktors
- Montieren Sie die Durchflusseinheit mit einem zweiten NMR-Rohr der Durchflusseinheit, das später durch das die Zellen enthaltende ersetzt wird. Die richtige Montage finden Sie in der Bedienungsanleitung der Durchflusseinheit.
HINWEIS: Zu diesem Zeitpunkt sollte die Durchflusseinheit bereits gereinigt sein (wenn nicht, führen Sie Schritt 4.2 aus). - Stellen Sie das an die Temperaturregelung der Durchflusseinheit angeschlossene Wasserbad auf 37 °C ein. Stellen Sie die Reservoirflasche in das Wasserbad.
- Schließen Sie den FEP-Schlauch der Reservoirflasche an die Pumpe an.
- Drehen Sie das Bioreaktorventil auf "Bypass" und füllen Sie die Pumpe mit Medium vor.
- Drehen Sie das Bioreaktorventil auf "Flow" und füllen Sie den Bioreaktor mit Medium bei 0,1 ml/min vor.
3. Vorbereitung der Zellprobe
- Sammeln Sie die Zellen aus dem CO2-Inkubator.
- Nehmen Sie einen T75-Kolben mit transfizierten HEK293T-Zellen aus dem CO2-Inkubator und entfernen Sie das verbrauchte Medium.
- Waschen Sie die Zellen zweimal mit 7 ml (jeweils) PBS bei Raumtemperatur (~ 20 ° C).
- Verwenden Sie 2 ml Trypsin / EDTA, um Zellen zu lösen. Nach Zugabe der Lösung 5 Minuten bei Raumtemperatur inkubieren, um die Zellen zu lösen.
HINWEIS: Transfizierte Zellen können etwas länger brauchen, um sich zu lösen. Falls erforderlich, inkubieren Sie die Zellen bei 37 °C. - Inaktivieren Sie Trypsin mit 20 ml vollständigem DMEM; Suspendieren Sie die Zellen gründlich durch Pipettieren auf und ab und übertragen Sie sie in einem 50-ml-Zentrifugenröhrchen.
- Zentrifugieren Sie die Zellen bei 800 x g für 5 min bei Raumtemperatur und entsorgen Sie den Überstand.
- Waschen Sie die Zellen mit 10 ml PBS bei Raumtemperatur, um das Restmedium zu entfernen.
- Zentrifugieren Sie die Zellen bei 800 x g für 5 min bei Raumtemperatur und entsorgen Sie den Überstand.
- Übertragen Sie das Zellpellet in ein 1,5 ml gekapptes Mikrozentrifugenröhrchen.
- Betten Sie Zellen in Agarosefäden ein.
- Ein Aliquot erstarrter Agarose bei 85 °C in einem Wasserbad schmelzen und anschließend in einer Blockheizung bei 37 °C in Lösung halten.
- Mit einer Pasteur-Pipette füllen Sie den Boden des NMR-Rohrs der Durchflusseinheit mit 60-70 μL 1,5% Agarosegel und legen Sie es in Eis. Dadurch entsteht ein ~ 5 mm hoher Bodenstecker, der es ermöglicht, die Zellprobe innerhalb des aktiven Volumens der 1H-NMR-Spule zu platzieren.
- Das in Schritt 3.1.8 erhaltene Zellpellet wird bei 37 °C für 15−20 s im Thermoblock erhitzt.
- Resuspendieren Sie Zellen in 450 μL Agaroselösung. Achten Sie darauf, die Bildung von Blasen zu vermeiden.
- Saugen Sie die Zell-Agarose-Suspension in einen ~30 cm langen Chromatographie-PEEK-Schlauch (i.d. = 0,75 mm) ab, der mit einer 1-ml-Spritze verbunden ist.
HINWEIS: Vor dem Absaugen sollten der Schlauch und das tote Volumen der Spritze bei Raumtemperatur mit PBS vorgefüllt werden, um die Bildung von Blasen zu vermeiden. Die Länge des Schlauches ist nicht kritisch. - Lassen Sie den Schlauch bei Raumtemperatur 2 min abkühlen.
- Füllen Sie die Durchflusseinheit NMR-Röhre mit 100 μL PBS bei Raumtemperatur vor.
- Gießen Sie Fäden von Zellen, die in Agarose eingebettet sind, in das NMR-Rohr der Durchflusseinheit, indem Sie die Spritze vorsichtig drücken.
HINWEIS: Um das NMR-Röhrchen homogen zu füllen, platzieren Sie zunächst das Ende des PEEK-Schlauchs an der Unterseite des NMR-Röhrchens und fahren Sie nach oben, während Sie langsam von links nach rechts schwingen. - Wiederholen Sie die Schritte 3.2.5, 3.2.6 und 3.2.8, bis die gesamte Zell-Agarose-Suspension gegossen wurde.
- Fügen Sie Zellen in den Bioreaktor ein.
- Entfernen Sie das leere NMR-Rohr aus der Durchflusseinheit und erhöhen Sie die Durchflussrate für einige Minuten auf 2 ml/min, um Restgasblasen im Einlassschlauch zu entfernen.
- Stellen Sie die Durchflussrate auf 0,2 ml/min ein und führen Sie den NMR-Röhrchen, in dem sich die Zellen befinden, ein, indem Sie es langsam, aber stetig nach oben drücken.
HINWEIS: Der aktive Fluss des Mediums vermeidet den Rückfluss des Schlauchinhalts durch den Einlass, der sonst während des Einführens auftreten würde.
4. Betrieb und Reinigung des Bioreaktors
- Bioreaktorbetrieb während des NMR-Experiments.
- Stellen Sie die Temperatur im NMR-Spektrometer auf 310 K ein.
- Setzen Sie die Durchflusseinheit in das Spektrometer ein.
- Versorgen Sie das Bioreaktormedium mit einer Durchflussrate von 0,1 ml/min für die gesamte Dauer der zellinternen NMR-Experimente.
- Injizieren Sie zum gewünschten Zeitpunkt während des Experiments eine konzentrierte Lösung eines externen Moleküls in die Flasche mit mittlerem Reservoir, indem Sie den Silikonschlauch mit einer sterilen Langnadelspritze durchstechen.
HINWEIS: Die endgültige Konzentration des Moleküls im Medium sollte auf der Grundlage früherer Kenntnisse der Zelltoxizität und, falls verfügbar, der vorhergesagten/geschätzten Diffusionsrate durch die Zellmembran ausgewählt werden. - Ersetzen Sie am Ende des NMR-Experiments das Röhrchen mit den Zellen durch ein leeres Rohr und spülen Sie die Durchflusseinheit mit Wasser ab.
- Bioreaktor clean-in-place.
- Reinigen Sie die Durchflusseinheit, indem Sie die folgenden Lösungen mit 1 ml/min fließen lassen: 0,2 m Natriumhydroxid (NaOH); 3 M Zitronensäure; 0,2 M NaOH, für jeweils mindestens 30 min, gefolgt von steril gefiltertem Reinstwasser für >2 h.
- Reinigen und autoklavieren Sie die Behälterflasche und die Schlauchbaugruppe nach jedem Durchlauf.
5. NMR-Experimente
- Aufbau der NMR-Experimente.
HINWEIS: Führen Sie diese Schritte vor der Vorbereitung der zellinternen NMR-Probe durch, um Verzögerungen zwischen der Zellsammlung und der Datenerfassung zu vermeiden.- Erstellen Sie einen neuen Datensatz am NMR-Spektrometer und legen Sie die Parameter für die gewünschten NMR-Experimente fest.
- Legen Sie Parameter für 1D-1H-NMR-Experimente fest.
- Zentriere die 1H-Trägerfrequenz bei 4,7 ppm auf dem Wassersignal.
- Wählen Sie das zgesgp-Pulsprogramm, stellen Sie die Spektralbreite auf 20 ppm und einen 1.000-μs-180°-Quadratpuls zur Wasserunterdrückung ein. Legen Sie eine Verzögerung zwischen den Scans von 1 s fest. Erfassen Sie das Spektrum mit 32 Scans.
- Für Zellen, die unmarkiertes CA II exprimieren: Wählen Sie das p3919gp-Pulsprogramm, stellen Sie die Spektralbreite auf 30 ppm ein, um den Imino-Bereich des Spektrums abzudecken, und passen Sie die Verzögerung für die binomiale Wasserunterdrückung so an, dass die maximale Anregung auf die chemischen Verschiebungen der interessierenden Signale zentriert ist (d7 = 20 μs bei 950 MHz). Legen Sie eine Verzögerung zwischen den Scans von ≥1 s fest. Erfassen Sie mit 512 Scans.
- Für Zellen, die 15N-markiertes SOD1 exprimieren: Wählen Sie das Pulsprogramm sfhmqf3gpph, stellen Sie die Spektralbreiten 1H und 15N auf 16 bzw. 50 ppm, die Offset und Anregungsbandbreite des geformten Pulses auf 8,5 bzw. 6 ppm und einen Puls von 350 μs für das Entkopplungsschema (Garp4 oder andere je nach Instrument). Legen Sie eine Verzögerung zwischen den Scans von 0,3 s fest. Erfassen Sie mit 16 Scans und 128 Schritten in der 15N-Dimension.
- Echtzeit-NMR-Spektrenerfassung.
- Sobald der Bioreaktor in das NMR-Spektrometer eingeführt wurde, warten Sie einige Minuten, um den Austausch des Mediums zu ermöglichen.
HINWEIS: Dieser Prozess kann leicht vom Auftreten des Sperrsignals aus überwacht werden, da das PBS durch ein Medium ersetzt wird, das 2% D2O enthält. - Passen Sie die Anpassung und Abstimmung des 1H-Kanals an, schleifen Sie den Magneten und berechnen Sie die 1H 90 ° harte Pulslänge.
- Passen Sie die 1H-Leistungsstufen in jeder Pulssequenz entsprechend dem harten 1H-Puls an.
- Nehmen Sie ein erstes zgesgp 1H-Spektrum auf, um den Probengehalt und die Feldhomogenität zu überprüfen.
- Kopieren Sie die Experimente zgesgp und p3919gp/sfhmqcf3gpph auf die gewünschte Anzahl und stellen Sie sie in die Warteschlange im Erfassungsspooler.
HINWEIS: Die zgesgp-Spektren werden nur verwendet, um den Zustand der Probe und die Feldhomogenität zu kontrollieren; Daher können sie entweder übersprungen oder seltener aufgezeichnet werden. - Für Zellen, die unmarkierte CA II exprimieren: Verarbeiten Sie die p3919gp-Spektren durch Anwendung der Nullfüllung und der exponentiellen Linienverbreiterungsfensterfunktion (LB = 20 Hz).
- Für Zellen, die 15N-markiertes SOD1 exprimieren: Verarbeiten Sie die SFHMQCF3gpph-Spektren durch Anwendung der Nullfüllung und der quadratischen Sinusglockenfensterfunktion (SSB = 2) in beiden Dimensionen.
HINWEIS: Die Größe der verarbeiteten Spektren kann weiter reduziert werden, indem signalfreie Bereiche entfernt werden (in Topspin geschieht dies durch Einstellen der gewünschten STSR- und STSI-Werte).
- Sobald der Bioreaktor in das NMR-Spektrometer eingeführt wurde, warten Sie einige Minuten, um den Austausch des Mediums zu ermöglichen.
6. MCR-ALS-Analyse
- Für die Analyse von CA II-Spektren importieren Sie 1D-Spektralbereiche in MATLAB R2019b.
- Erstellen Sie in der Software eine Liste der Experimente, die im Menü Prozess-Dataset-Liste exportiert werden sollen.
- Exportieren Sie mit einer modifizierten Version des au-Programms convbin2asc den interessierenden Spektralbereich im ASCII-Format für jedes Spektrum.
HINWEIS: Dadurch wird in jedem Spektrum-Unterverzeichnis eine Textdatei mit dem Namen ascii-spec.txt erstellt. - Importieren Sie in MATLAB die Spektralbereiche mithilfe des benutzerdefinierten Skripts Load_ascii_spectra.
HINWEIS: Dieses Skript benötigt das Datensatzverzeichnis als Eingabe und erzeugt ein 2D-Array-Spektren mit den gestapelten 1D-Spektren und ein 1D-Array-cs, das die chemischen Verschiebungen enthält. - Führen Sie das Skript Load_acqus aus, um die Zeitstempel aus den 1D-Spektren zu extrahieren.
HINWEIS: Dieses Skript erzeugt ein 1D-Array times_hours das das Zeitinkrement für jedes Spektrum in Stunden enthält, wobei das Anfangsspektrum bei time = 0 ist.
- Für die Analyse von SOD1-Spektren importieren Sie 2D-Spektren in MATLAB R2020b.
- Erstellen Sie in Topspin eine Liste der Experimente, die im Menü " Prozess-Dataset-Liste" exportiert werden sollen.
- Importieren Sie in MATLAB die 2D-Spektren mithilfe des benutzerdefinierten Skripts Load_2D_spectra.
HINWEIS: Dieses Skript benötigt das Datensatzverzeichnis als Eingabe und erzeugt ein 3D-Array-Spektren mit den gestapelten 2D-Spektren und ein 1D-Array cs, das die chemischen Verschiebungen enthält. - Führen Sie das Load_acqus Skript aus, um die Zeitstempel aus den 2D-Spektren zu extrahieren.
- Geben Sie die interessierenden Spektralbereiche im benutzerdefinierten Skript Cut_2D_spectra an und führen Sie das Skript aus, um 3D-Unterarrays [(1H x 15N) spektrale Intensitäten) x Zeit] zu schneiden. Gestalten Sie sie zu 2D-Arrays um (Zeitpunkte x spektrale Intensitäten) und fügen Sie sie zusammen.
HINWEIS: Dadurch entsteht ein 2D-Array JoinSpec_flat das die umgeformten und verbundenen Spektralbereiche enthält.
- Führen Sie MCR-ALS 2.0 im GUI-Modus aus.
- Öffnen Sie die MCR-ALS 2.0 GUI, indem Sie das Skript mcr_main ausführen.
- Laden Sie auf der Registerkarte Datenauswahl die Spektren oder die JoinSpec_flat Matrix. Die Daten können zur Überprüfung aufgetragen werden.
- Bewerten Sie die Anzahl der Komponenten entweder durch SVD (Singular Value Decomposition) oder manuell.
HINWEIS: Die Anzahl der Komponenten sollte der Anzahl der verschiedenen Arten entsprechen, die im Experiment vorhanden sind. In diesem Fall n = 2, entsprechend dem freien und gebundenen Protein. - Wählen Sie eine Methode zur Erstschätzung der reinen Spektren. Es kann entweder die reinste Variablenerkennung oder die Evolving Factor Analysis (EFA) verwendet werden.
- Wählen Sie im Fenster Auswahl des Datensatzes die Option Weiter aus.
- Legen Sie die Einschränkungen für die Konzentrationen im Fenster Einschränkungen: Zeilenmodus fest. Wenden Sie eine Nicht-Negativitäts-Einschränkung an, wählen Sie fnnls (Fast nonnegativity-constrained least-squares) als "Implementierung und 2 Arten". Wenden Sie 1 Schließungseinschränkung an, setzen Sie die Einschränkung auf 1, die Schließbedingung auf "gleich" und gelten Sie für alle Arten.
HINWEIS: Dies zwingt die Summe der Konzentrationen jeder Spezies, gleich 1 zu sein, so dass die erhaltenen Profile jeder Spezies in Bezug auf die Gesamtproteinkonzentration normalisiert werden. - Legen Sie die Einschränkungen für die Spektren im Fenster Einschränkungen: Spaltenmodus fest. Wenden Sie eine Nicht-Negativitätseinschränkung an, wählen Sie fnnls als "Implementierung und 2 Arten".
HINWEIS: Diese Einschränkung sollte nicht angewendet werden, wenn negative Signale in den NMR-Spektren vorhanden sind. - Legen Sie im letzten Fenster 50 Iterationen und das Konvergenzkriterium 0,01 fest. Geben Sie die Ausgabenamen für Konzentrationen, Spektren und Std.-Abweichung an. Klicken Sie auf Weiter , um die MCR-ALS-Anpassung auszuführen.
HINWEIS: Ein grafisches Fenster zeigt das Ergebnis der Anpassung mit Diagrammen der Konzentrationsprofile und den Spektren der reinen Komponenten. - Für das SOD1-Dataset: Verwenden Sie das benutzerdefinierte Skript Rebuild_2D_spectra, um die 2D-Spektralbereiche aus der 1D-Ausgabe von MCR-ALS zu rekonstruieren und darzustellen.
7. Trypan Blau Test
- Gewinnen Sie den NMR-Röhrcheninhalt mit einer Pasteur-Pipette zurück und übertragen Sie die Agarosefäden in ein 1,5-ml-gekapptes Rohr.
- Entfernen Sie das Restmedium, indem Sie die Agarosefäden mit 600 μL PBS spülen und bei Raumtemperatur bei 4.000 x g für 1 min zentrifugieren. Verwerfen Sie den Überstand.
- Fügen Sie 250 μL PBS und 50 μL 0,4% Trypan Blue Solution hinzu.
- Inkubieren Sie für 2 min mit kontinuierlichem Pipettieren.
- Zweimal waschen, wobei 600 μL PBS den Überstand entsorgen.
- Legen Sie ein paar Agarosefäden auf einen Objektträger und hacken Sie sie mit Rasierklingen, um kleine Gelscheiben zu erzeugen. Wählen Sie die dünnsten Scheiben (Dicke < 0,4 mm, idealerweise ~0,2 mm) für die Analyse aus.
- Übertragen Sie die Gelscheiben in eine selbstgebaute Zellzählkammer, die aus zwei Glasobjektträgern besteht, die auf jeder Seite von drei Schichten Paraffinfilm (~ 0,4 mm Gesamtdicke) angeordnet sind.
HINWEIS: Es kann auch ein Kammerschlitten verwendet werden. Die Gelscheiben, die dicker als die Kammerhöhe (0,1 mm) sind, würden jedoch gequetscht, wodurch die eingebetteten Zellen zerreißen würden. - Erfassen Sie Bilder von Zellen in der Agarose und zählen Sie weiße und blaue Zellen.
- Berechnen Sie die Zelllebensfähigkeit als (Gesamtzellen - blaue Zellen) / Gesamtzellen.
Ergebnisse
Das obige Protokoll ermöglicht die Verkapselung von Zellen in Hydrogelfäden, um die Zelllebensfähigkeit für lange Zeiträume zu maximieren, die notwendig sind, um intrazelluläre Prozesse in Echtzeit zu untersuchen. Im Bioreaktor werden die Zellen bis zu 72 h am Leben erhalten und metabolisch aktiv, wie der Trypan-Blue-Test bestätigt (Abbildung 2a-c). Im Prinzip kann dieses Protokoll angewendet werden, um ein intrazelluläres Protein von Interesse zu beobachten, das Konformations- oder chemische Veränderungen durchläuft. In der ersten oben beschriebenen Anwendung wird der Bioreaktor eingesetzt, um in Echtzeit die Bindung von zwei Inhibitoren, AAZ und MZA, an CA II zu überwachen, die im Zytosol von HEK293T-Zellen überexprimiert sind. Das erste aufgezeichnete 1H-Anregungsformungsspektrum (zgesgp) wird verwendet, um die Gesamtsignalintensität (die proportional zur Anzahl der Zellen ist), das Vorhandensein von Signalen des überexprimierten Proteins und die Feldhomogenität zu beurteilen (Abbildung 2d). Im Fall von CA II kann die intrazelluläre Bindung der beiden Inhibitoren durch WATERGATE 3-9-1935 1D 1H NMR-Spektren (p3919gp) überwacht werden, indem 1H-Signale im Bereich zwischen 11 und 16 ppm beobachtet werden. Diese Signale entstehen aus den zinkkoordinierenden Histidinen und anderen aromatischen Rückständen von CA II36 und sind bei der Ligandenbindung14,15 gestört. Die Ligandenkonzentrationen können auf der Grundlage der Diffusionsrate oder, falls verfügbar, anhand zuvor festgelegter Permeabilitätswerte gewählt werden14. Die erfolgreiche Bindung wird visuell durch das Auftreten eines zusätzlichen Satzes von Signalen in der interessierenden Spektralregion bestätigt, der die ursprünglichen Signale allmählich ersetzt (Abbildung 3). Zeitabhängige Bindungskurven werden durch MCR-ALS-Analyse erhalten, die die beiden Sätze von NMR-Signalen, die sich aus freiem und gebundenem CA II ergeben, trennt (Abbildung 4a) und gleichzeitig die relativen Konzentrationsprofile der beiden Spezies liefert (Abbildung 4b). In der zweiten Anwendung wird der Bioreaktor eingesetzt, um die Bildung einer zinkgebundenen intramolekularen SOD1-Disulfidbindung zu überwachen, die durch Ebselen, ein Glutathionperoxidase-Mimetikum, in menschlichen Zellen gefördert wird. Dieser Prozess wird überwacht, indem Veränderungen in 1H-15N 2D-SOFAST-HMQC37-Spektren (die einen Fingerabdruck der Proteinrückgratkonformation liefern) beobachtet werden, die durch Störungen der Proteinstruktur verursacht werden, die durch die Bildung von Disulfidbindungen induziert werden. Zusätzliche Signale, die sich aus disulfidoxidiertem SOD1 ergeben, erscheinen im 1H-15N-Spektrum und ersetzen allmählich diejenigen aus disulfidreduziertem SOD1. Die MCR-ALS-Analyse an ausgewählten Regionen des 2D-Spektrums trennt die von den beiden Spezies ausgehenden Signale (Abbildung 5a) und liefert ihre relativen Konzentrationsprofile (Abbildung 5b). Die erhaltenen Konzentrationskurven können durch nichtlineare Anpassung weiter analysiert werden, um Informationen über die Kinetik der untersuchten Prozesse zu liefern25.
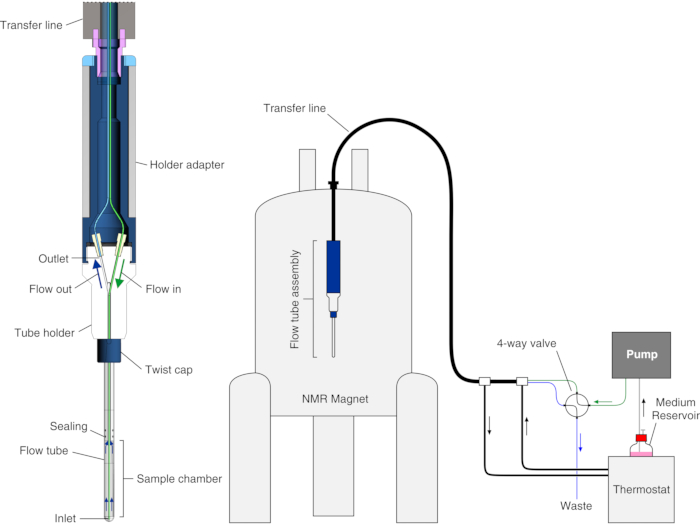
Abbildung 1: Schema des Bioreaktors. Links: Querschnittsansicht der leeren Durchflusseinheit. Rechts: Schema des Bioreaktoraufbaus. Der PEEK-Einlassschlauch ist grün dargestellt; Der PTFE-Auslassschlauch ist blau dargestellt. Die linke Tafel wird mit Genehmigung von Luchinat et al.25 reproduziert. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
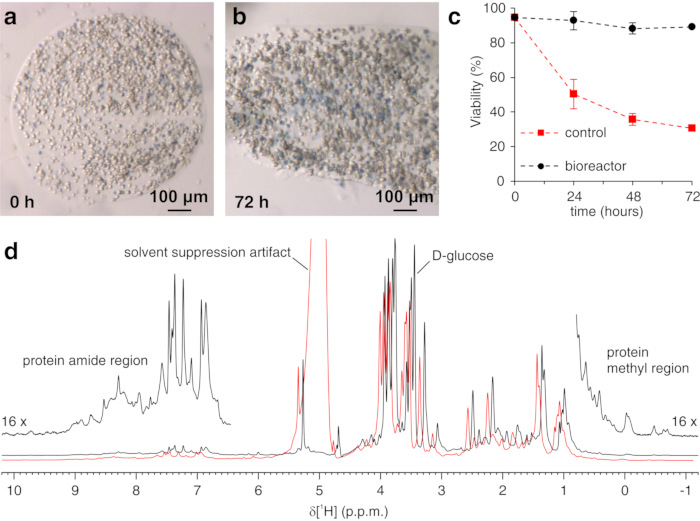
Abbildung 2: Trypan Blue Test an verkapselten Zellen und Probenkontrolle durch 1H NMR. Repräsentative Agarosescheiben, die eingebettete Zellen enthalten und unmittelbar nach dem Gießen und (b) nach 72 h im Bioreaktor mit Trypanblau (a) gefärbt sind; c) Zelllebensfähigkeit als Funktion der Zeit im NMR-Bioreaktor unter aktiver Strömung (schwarz) und unter statischen Bedingungen (rot), gemessen mit dem Trypan-Blue-Test. d) zgesgp 1H-NMR-Spektren, die an in Agarose eingebetteten Zellen aufgezeichnet wurden, die CA II in Abwesenheit (schwarz) und in Gegenwart (rot) von Gasblasen im Bioreaktor überexprimieren. Im letzteren Fall verursacht eine verminderte Feldhomogenität eine Linienverbreiterung und das Auftreten von Lösungsmittelunterdrückungsartefakten. Interessante spektrale Merkmale werden beschriftet. Die Tafeln (a-c) werden mit Genehmigung von Luchinat et al.25 reproduziert. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
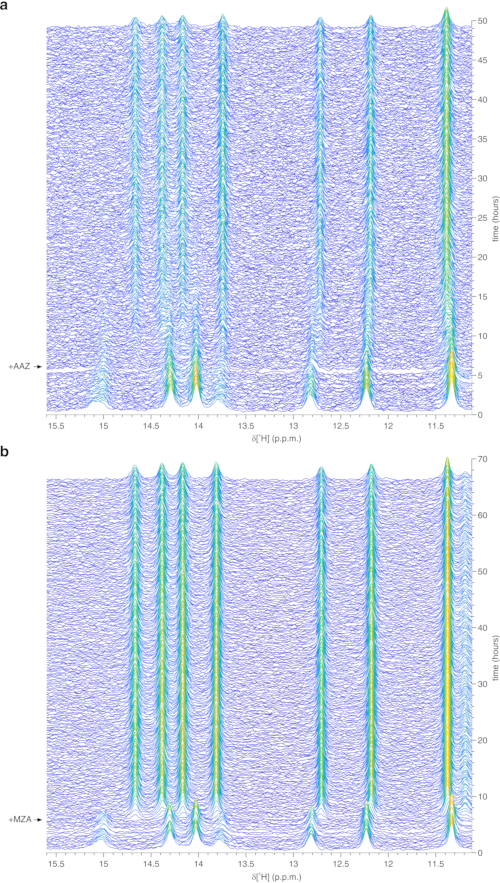
Abbildung 3: Repräsentative Echtzeit-1H-NMR-Daten in der Zelle, die an Agarose-verkapselten Zellen im Bioreaktor gewonnen wurden. Wasserfalldiagramme von 1D-1H-NMR-Spektren (Bereich zwischen 15,6 und 11,1 ppm) von HEK293T-Zellen, die CA II überexprimieren und anschließend mit (a) 25 μM AAZ und (b) 10 μM MZA behandelt wurden, aufgezeichnet als Funktion der Zeit im NMR-Bioreaktor. Der Zeitpunkt der Ligandenbehandlung wird mit einem Pfeil angezeigt. Die spektrale Intensität (a.u.) wird farblich von blau (niedrigste) bis gelb (höchste) codiert. Diese Figur wird mit Genehmigung von Luchinat et al.25 reproduziert. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
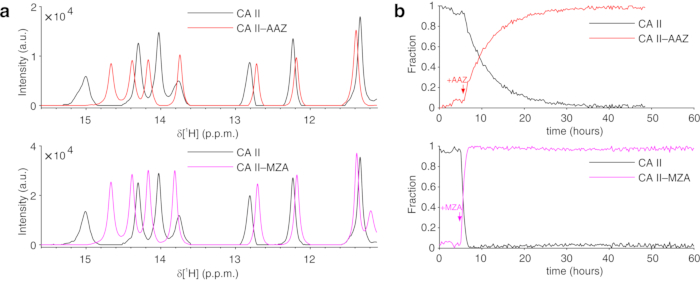
Abbildung 4: Repräsentativer MCR-ALS-Ausgang aus 1D-NMR-Spektren. a) 1H-NMR-Spektren der reinen Komponenten, rekonstruiert durch MCR-ALS: CA II in Abwesenheit von Liganden (schwarz) und im Komplex mit AAZ (rot) oder MZA (Magenta); b) relative Konzentrationsprofile von freiem (schwarz) und gebundenem CA II als Funktion der Zeit bei Zugabe von AAZ (rot) oder MZA (Magenta), erhalten durch MCR-ALS. Die Zeiten der Ligandenbehandlung sind mit Pfeilen gekennzeichnet. Diese Figur wird mit Genehmigung von Luchinat et al.25 reproduziert. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
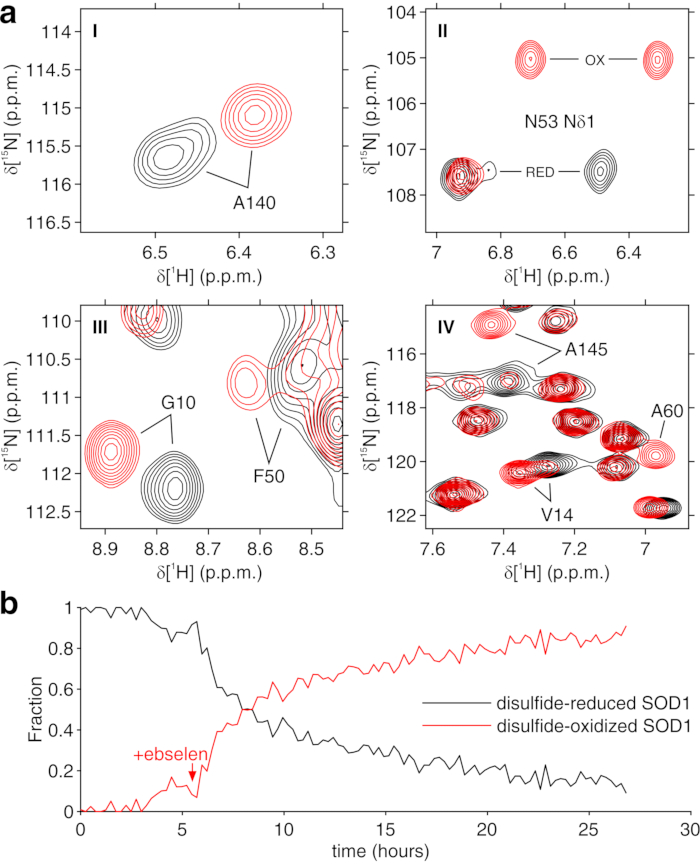
Abbildung 5: Repräsentativer MCR-ALS-Ausgang aus 2D-NMR-Spektren. a) 1H-15N-Spektralbereiche (gekennzeichnet mit I-IV) der durch MCR-ALS rekonstruierten Reinkomponenten: disulfidreduziertes SOD1 (schwarz) und disulfidoxidiertes SOD1 (rot); b) relatives Konzentrationsprofil der reinen Bestandteile in Abhängigkeit von der Zeit bei Zugabe von Ebselen (mit Pfeil gekennzeichnet), das durch MCR-ALS erhalten wird. Diese Figur wird mit Genehmigung von Luchinat et al.25 reproduziert. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
Diskussion
Ziel der Verwendung eines Bioreaktors für In-Cell-NMR-Experimente ist es, Zellen über einen längeren Zeitraum am Leben und metabolisch aktiv zu halten. Um dieses Ziel zu erreichen, müssen eine Reihe kritischer Aspekte berücksichtigt werden. Erstens ist es von größter Bedeutung, eine bakterielle Kontamination bei der Vorbereitung der Zellprobe und während der NMR-Datenerfassung zu vermeiden. Wenn Stämme von E. coli oder anderen Bakterien, die üblicherweise für die Klonierung von Genen und die rekombinante Proteinexpression verwendet werden, im Labor verwendet werden, können sie die Zellen während der Probenvorbereitung kontaminieren. Sobald sie sich im Bioreaktor befinden, wachsen die Bakterien schnell, indem sie das frische Wachstumsmedium ausbeuten und durch die Produktion von Endotoxinen zum Zelltod führen. Bakterielle Kontamination wird erst in einem fortgeschrittenen Stadium entdeckt, wenn sie das Wachstumsmedium gelb und trüb färbt. Darüber hinaus kann eine unvollständige Reinigung des Bioreaktors zu einer Kontamination der Pumpe oder des Schlauches mit Bakterien, Hefen oder gewöhnlichen Schimmelpilzen führen.
Voraussetzung für den Erfolg des Experiments ist die Vermeidung der Gasblasenbildung. Gasblasen, die zwischen den Agarosefäden im aktiven Volumen der NMR-Spule eingeschlossen sind, würden große Magnetfeldinhomogenitäten verursachen, was zu einer unvollständigen Unterdrückung des H2O-Signals und einem schweren Verlust der spektralen Qualität führt (Abbildung 2d). Blasen können durch im System eingeschlossene Luft oder durch die Bildung von gasförmigem CO2 verursacht werden. Ersteres kann leicht vermieden werden, indem das System vor dem Einsetzen der Zellprobe mit Medium gespült wird, während es zur Vermeidung von Letzterem empfohlen wird, die Konzentration von NaHCO3 im Wachstumsmedium zu verringern und alle Teile des Systems auf einer konstanten Temperatur zu halten, um Unterschiede in der CO2-Löslichkeit zu minimieren. Der zelluläre aerobe Stoffwechsel kann auch die Bildung von gasförmigem CO2 verursachen, das durch Erhöhung der Durchflussrate verhindert werden kann.
Die Zelllebensfähigkeit sollte nach jedem Durchlauf von Trypan Blue Test überprüft werden. Das gibt jedoch keine Erkenntnisse über die Stoffwechselaktivität. Um ein vollständigeres Bild des Stoffwechselzustands der Zellen während des Bioreaktorbetriebs zu erhalten, können 31P-NMR-Spektren durchgeführt werden, um die Produktion von ATP in Abhängigkeit von der Zeit zu beurteilen23,25. Für diese Messung ist jedoch häufig eine dedizierte Sonde erforderlich, die eine gleichzeitige Aufzeichnung mit 1H NMR ermöglichen kann.
Im Fall von CA II erleichtert das Vorhandensein von gut aufgelösten Reportersignalen in einem ungewöhnlichen Bereich des 1H-Spektrums die Analyse aus einfachen 1D-NMR-Spektren und erfordert keine Isotopenanreicherung während der Proteinexpression. Im Allgemeinen könnten andere Proteine zu 1H-Signalen führen, die für die Überwachung spektraler Veränderungen in anderen Regionen nützlich sind, wie z. B. das für den hydrophoben Kern des Proteins typische zwischen 0 und -1 ppm11; Diese Regionen neigen jedoch dazu, für gefaltete Proteine, die größer als ~ 10 kDa sind, überfüllt zu sein. In diesem Fall ist es, wie für SOD1 gezeigt, vorzuziehen, das Protein mit 15N anzureichern, indem während der Proteinexpression gleichmäßig 15N-angereichertes Wachstumsmedium bereitgestellt wird, und Echtzeitänderungen in 2D-1H-15N-NMR-Spektren zu überwachen. 2D-Spektren werden als 2D-Arrays in MATLAB importiert, zu 1D-Arrays umarrangiert und vor der MCR-ALS-Analyse gestapelt. Der letztgenannte Ansatz ist im Allgemeinen auf jedes intrazelluläre Protein anwendbar, das nachweisbare Signale hervorruft, und liefert Informationen über Proteinkonformationsänderungen auf der Ebene der einzelnen Rückstände. Im Prinzip kann der letztere Ansatz auf nD-Spektren und auf andere Isotopenmarkierungsschemata verallgemeinert werden.
In Bezug auf die Anwendung auf verschiedene Zelltypen sollte das Protokoll leicht an verschiedene Zelllinien angepasst werden und erfordert nicht, dass das interessierende Protein direkt in den Zellen exprimiert wird. Daher können andere Ansätze zur zellinternen NMR mit diesem Protokoll kombiniert werden, bei dem das interessierende Makromolekül rekombinant hergestellt oder synthetisiert und anschließend durch Elektroporation oder durch andere Abgabemethoden in die Zellen eingefügt wird1,9,38. Bei der Arbeit mit verschiedenen Zelllinien oder Probenvorbereitungsprotokollen müssen Parameter wie die Agarosekonzentration, die Fadendicke und die endgültige Zelldichte in den Agarosefäden möglicherweise empirisch optimiert werden. Darüber hinaus ist die Anwendbarkeit des hier beschriebenen Protokolls auf Zellen beschränkt, die eine Agaroseverkapselung tolerieren. Andere Zelltypen erfordern möglicherweise andere Hydrogelformulierungen, während bei der Analyse von Zellen, die nativ in Suspension wachsen, ein anderer Aufbau empfohlen wird, z. B. unter Verwendung einer koaxialen Mikrodialysemembran, um die Nährstoffdiffusion sicherzustellen und gleichzeitig suspendierte Zellen im NMR-Röhrchen einzuschließen23.
Im Vergleich zu anderen NMR-Bioreaktorkonstruktionen19,20,21,22 beruht die hier beschriebene Vorrichtung auf einer kommerziell erhältlichen Durchflusseinheit, die mit geringfügigen Modifikationen angepasst ist. Daher kann das Gerät problemlos in verschiedenen Laboren mit hoher Reproduzierbarkeit repliziert werden. Darüber hinaus ermöglicht es einen standardisierten Betrieb und bei Bedarf die vollständige Einhaltung strenger Laborsicherheitsvorschriften. Insgesamt dürften die Flexibilität und die einfache Bedienung des Bioreaktors viele andere Anwendungen der Lösungs-NMR sowohl in Zellen als auch in vitro ermöglichen, zusätzlich zu denen, wie bereits berichtet23,25. Schließlich könnte das gleiche Bioreaktordesign auf Proben angewendet werden, die eher der physiologischen Umgebung eines Gewebes ähneln, wie Sphäroide oder Organoide, vorausgesetzt, dass geeignete Gerüste gefunden werden, um solche Proben am Leben zu erhalten - oder sogar ihr Wachstum aufrechtzuerhalten - im NMR-Spektrometer.
Offenlegungen
Die Autoren erklären, dass kein Interessenkonflikt vorliegt.
Danksagungen
Diese Arbeit wurde unterstützt von iNEXT-Discovery, Grant Nummer 871037, finanziert durch das Horizon 2020-Programm der Europäischen Kommission, von Instruct-ULTRA, Grant Number 731005, einem EU-H2020-Projekt zur Weiterentwicklung der Dienstleistungen von Instruct-ERIC, und vom Ministero dell'Istruzione, dell'Università e della Ricerca PRIN Grant 20177XJCHX. Die Autoren würdigen die Unterstützung von Instruct-ERIC, einem wegweisenden ESFRI-Projekt, durch den JRA Award Nummer 815 und die Nutzung von Ressourcen des CERM/CIRMMP Italy Centre. Wir danken Matteo Pennestri (Bruker, UK) für die Unterstützung des Betriebs der InsightMR-Durchflusseinheit.
Materialien
| Name | Company | Catalog Number | Comments |
| Materials | |||
| Citric acid | Sigma-Aldrich | 251275 | |
| D2O | Sigma-Aldrich | 453366 | |
| DMEM, high glucose | Life Technologies | 10313-021 | |
| DMEM, high glucose, powder | Sigma-Aldrich | D5648 | |
| FBS | Life Technologies | 10270 | |
| HCl | Sigma-Aldrich | 30721 | |
| L-glutamine (200 mM) | Life Technologies | 25030 | |
| Low-gelling agarose, powder | Sigma-Aldrich | A4018 | |
| NaHCO3, powder | Carlo Erba | 478537 | |
| PBS | Life Technologies | 10010 | |
| Penicillin–streptomycin (10,000 U/ml) | Life Technologies | 15140-122 | |
| NaOH, pellets | Sigma-Aldrich | 30620 | |
| Trypan Blue solution (0.4% (wt/vol) | Sigma-Aldrich | T8154 | Hazard statement(s): H350 may cause cancer. |
| Trypsin–EDTA (0.05% (wt/vol)) | Life Technologies | 25300-054 | |
| Equipment | |||
| Avance III Spectrometer equipped with a 5 mm CryoProbe | Bruker | n/a | All modern spectrometers and narrow-bore magnets equipped with 5 mm probes are compatible. |
| InsightMR flow unit | Bruker | n/a | |
| P-920 pump module from ÄKTA FPLC | GE Healthcare | n/a | Any FPLC, HPLC peristaltic or syringe pump should be compatible with the flow unit. |
Referenzen
- Inomata, K., et al. High-resolution multi-dimensional NMR spectroscopy of proteins in human cells. Nature. 458 (7234), 106-109 (2009).
- Luchinat, E., Banci, L. In-cell NMR: a topical review. IUCrJ. 4, 108-118 (2017).
- Dzatko, S., et al. Evaluation of the stability of DNA i-motifs in the nuclei of living mammalian cells. Angewandte Chemie (International Ed. in English). 57 (8), 2165-2169 (2018).
- Luchinat, E., Banci, L. In-cell NMR in human cells: direct protein expression allows structural studies of protein folding and maturation. Accounts of Chemical Research. 51 (6), 1550-1557 (2018).
- Tanaka, T., et al. High-resolution protein 3D structure determination in living eukaryotic cells. Angewandte Chemie (International Ed. in English). 58 (22), 7284-7288 (2019).
- Siegal, G., Selenko, P. Cells, drugs and NMR. Journal of Magnetic Resonance. 306, 202-212 (1997).
- Banci, L., et al. Atomic-resolution monitoring of protein maturation in live human cells by NMR. Nature Chemical Biology. 9 (5), 297-299 (2013).
- Luchinat, E., et al. In-cell NMR reveals potential precursor of toxic species from SOD1 fALS mutants. Nature Communications. 5, 5502 (2014).
- Theillet, F. -. X., et al. Structural disorder of monomeric α-synuclein persists in mammalian cells. Nature. 530 (7588), 45-50 (2016).
- Barbieri, L., Luchinat, E., Banci, L. Intracellular metal binding and redox behavior of human DJ-1. Journal of biological inorganic chemistry: JBIC: A Publication of the Society of Biological Inorganic Chemistry. 23 (1), 61-69 (2018).
- Banci, L., Barbieri, L., Luchinat, E., Secci, E. Visualization of redox-controlled protein fold in living cells. Chemistry & Biology. 20 (6), 747-752 (2013).
- Mercatelli, E., Barbieri, L., Luchinat, E., Banci, L. Direct structural evidence of protein redox regulation obtained by in-cell NMR. Biochimica Et Biophysica Acta. 1863 (2), 198-204 (2016).
- Barbieri, L., Luchinat, E., Banci, L. Protein interaction patterns in different cellular environments are revealed by in-cell NMR. Scientific Reports. 5, 14456 (2015).
- Luchinat, E., et al. Drug screening in human cells by NMR spectroscopy allows the early assessment of drug potency. Angewandte Chemie (International Ed. in English). 59 (16), 6535-6539 (2020).
- Luchinat, E., et al. Intracellular binding/unbinding kinetics of approved drugs to carbonic anhydrase II observed by in-cell NMR. ACS Chemical Biology. 15 (10), 2792-2800 (2020).
- DeMott, C. M., et al. Potent inhibitors of mycobacterium tuberculosis growth identified by using in-cell NMR-based screening. ACS Chemical Biology. 13 (3), 733-741 (2018).
- Krafcikova, M., et al. Monitoring DNA-ligand interactions in living human cells using NMR spectroscopy. Journal of the American Chemical Society. 141 (34), 13281-13285 (2019).
- Broft, P., et al. In-cell NMR of functional riboswitch aptamers in eukaryotic cells. Angewandte Chemie (International Ed. in English). , (2020).
- Sharaf, N. G., Barnes, C. O., Charlton, L. M., Young, G. B., Pielak, G. J. A bioreactor for in-cell protein NMR. Journal of magnetic resonance. 202 (2), 140-146 (2010).
- Kubo, S., et al. A gel-encapsulated bioreactor system for NMR studies of protein-protein interactions in living mammalian cells. Angewandte Chemie (International Ed. in English). 52 (4), 1208-1211 (2013).
- Inomata, K., Kamoshida, H., Ikari, M., Ito, Y., Kigawa, T. Impact of cellular health conditions on the protein folding state in mammalian cells. Chemical Communications. 53 (81), 11245-11248 (2017).
- Breindel, L., DeMott, C., Burz, D. S., Shekhtman, A. Real-time in-cell nuclear magnetic resonance: ribosome-targeted antibiotics modulate quinary protein interactions. Biochemistry. 57 (5), 540-546 (2018).
- Cerofolini, L., et al. Real-time insights into biological events: in-cell processes and protein-ligand interactions. Biophysical Journal. 116 (2), 239-247 (2019).
- Breindel, L., Burz, D. S., Shekhtman, A. Active metabolism unmasks functional protein-protein interactions in real time in-cell NMR. Communications Biology. 3, (2020).
- Luchinat, E., Barbieri, L., Campbell, T. F., Banci, L. Real-time quantitative in-cell NMR: ligand binding and protein oxidation monitored in human cells using multivariate curve resolution. Analytical Chemistry. 92 (14), 9997-10006 (2020).
- Koczula, K. M., et al. Metabolic plasticity in CLL: adaptation to the hypoxic niche. Leukemia. 30 (1), 65-73 (2016).
- Alshamleh, I., et al. Real-time NMR spectroscopy for studying metabolism. Angewandte Chemie (International Ed. in English). 59 (6), 2304-2308 (2020).
- Supuran, C. T. Carbonic anhydrases: novel therapeutic applications for inhibitors and activators. Nature Reviews. Drug Discovery. 7 (2), 168-181 (2008).
- Neri, D., Supuran, C. T. Interfering with pH regulation in tumours as a therapeutic strategy. Nature Reviews. Drug Discovery. 10 (10), 767-777 (2011).
- Alterio, V., Di Fiore, A., D'Ambrosio, K., Supuran, C. T., De Simone, G. Multiple binding modes of inhibitors to carbonic anhydrases: how to design specific drugs targeting 15 different isoforms. Chemical Reviews. 112 (8), 4421-4468 (2012).
- Capper, M. J., et al. The cysteine-reactive small molecule ebselen facilitates effective SOD1 maturation. Nature Communications. 9 (1), 1693 (2018).
- Trist, B., Hilton, J. B., Crouch, P. J., Hare, D. J., Double, K. L. Superoxide dismutase 1 in health and disease: How a front-line antioxidant becomes neurotoxic. Angewandte Chemie (International Ed. in English). , (2020).
- Tauler, R. Multivariate curve resolution applied to second order data. Chemometrics and Intelligent Laboratory Systems. 30 (1), 133-146 (1995).
- Barbieri, L., Luchinat, E., Banci, L. Characterization of proteins by in-cell NMR spectroscopy in cultured mammalian cells. Nature Protocols. 11 (6), 1101-1111 (2016).
- Piotto, M., Saudek, V., Sklenár, V. Gradient-tailored excitation for single-quantum NMR spectroscopy of aqueous solutions. Journal of biomolecular NMR. 2 (6), 661-665 (1992).
- Vasa, S. K., Singh, H., Grohe, K., Linser, R. Assessment of a large enzyme-drug complex by proton-detected solid-state NMR spectroscopy without deuteration. Angewandte Chemie (International Ed. in English). 58 (17), 5758-5762 (2019).
- Schanda, P., Brutscher, B. Very fast two-dimensional NMR spectroscopy for real-time investigation of dynamic events in proteins on the time scale of seconds. Journal of the American Chemical Society. 127 (22), 8014-8015 (2005).
- Ogino, S., et al. Observation of NMR signals from proteins introduced into living mammalian cells by reversible membrane permeabilization using a pore-forming toxin, streptolysin O. Journal of the American Chemical Society. 131 (31), 10834-10835 (2009).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten