Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Extracción y visualización de agregados proteicos después del tratamiento de Escherichia coli con un estresor proteotóxico
En este artículo
Resumen
Este protocolo describe la extracción y visualización de proteínas agregadas y solubles de Escherichia coli después del tratamiento con un antimicrobiano proteotóxico. Seguir este procedimiento permite una comparación cualitativa de la formación de agregados proteicos in vivo en diferentes cepas bacterianas y/o entre tratamientos.
Resumen
La exposición de los organismos vivos a tensiones ambientales y celulares a menudo causa interrupciones en la homeostasis de las proteínas y puede resultar en la agregación de proteínas. La acumulación de agregados de proteínas en las células bacterianas puede conducir a alteraciones significativas en el comportamiento fenotípico celular, incluida una reducción en las tasas de crecimiento, resistencia al estrés y virulencia. Existen varios procedimientos experimentales para el examen de estos fenotipos mediados por factores estresantes. Este artículo describe un ensayo optimizado para la extracción y visualización de proteínas agregadas y solubles de diferentes cepas de Escherichia coli después del tratamiento con un antimicrobiano que contiene plata-rutenio. Se sabe que este compuesto genera especies reactivas de oxígeno y causa una agregación generalizada de proteínas.
El método combina una separación basada en centrifugación de agregados de proteínas y proteínas solubles de células tratadas y no tratadas con la posterior separación y visualización por electroforesis en gel de dodecil sulfato de sodio-poliacrilamida (SDS-PAGE) y tinción de Coomassie. Este enfoque es simple, rápido y permite una comparación cualitativa de la formación de agregados proteicos en diferentes cepas de E. coli. La metodología tiene una amplia gama de aplicaciones, incluida la posibilidad de investigar el impacto de otros antimicrobianos proteotóxicos en la agregación de proteínas in vivo en una amplia gama de bacterias. Además, el protocolo se puede utilizar para identificar genes que contribuyen a aumentar la resistencia a las sustancias proteotóxicas. Las bandas de gel se pueden utilizar para la identificación posterior de proteínas que son particularmente propensas a la agregación.
Introducción
Las bacterias están inevitablemente expuestas a una miríada de tensiones ambientales, incluyendo pH bajo (por ejemplo, en el estómago de los mamíferos)1,2, especies reactivas de oxígeno y cloro (ROS / RCS) (por ejemplo, durante el estallido oxidativo en los fagocitos)3,4,5, temperaturas elevadas (por ejemplo, en aguas termales o durante el choque térmico)6,7, y varios antimicrobianos potentes (por ejemplo, AGXX utilizado en este protocolo)8. Las proteínas son particularmente vulnerables a cualquiera de estos factores estresantes, y la exposición puede provocar el desplegamiento de proteínas que luego se agregan las semillas. Todos los organismos emplean sistemas de protección que les permiten hacer frente al mal plegamiento de proteínas9. Sin embargo, el estrés severo puede abrumar la maquinaria de control de calidad de proteínas e interrumpir la estructura secundaria y / o terciaria de las proteínas, que en última instancia inactiva las proteínas. Como consecuencia, los agregados de proteínas pueden afectar gravemente las funciones celulares críticas requeridas para el crecimiento y la supervivencia bacteriana, la resistencia al estrés y la virulencia10. Por lo tanto, la investigación centrada en la agregación de proteínas y sus consecuencias en las bacterias es un tema relevante debido a su impacto potencial en el control de enfermedades infecciosas.
El despliegue y la agregación de proteínas inducidas por el calor son a menudo reversibles7. Por el contrario, otras tensiones proteotóxicas, como el estrés oxidativo, pueden causar modificaciones irreversibles de las proteínas a través de la oxidación de cadenas laterales de aminoácidos específicos, lo que resulta en el desplegamiento / mal de la proteína y, eventualmente, la agregación deproteínas 4. La formación inducida por estrés de agregados de proteínas insolubles ha sido ampliamente estudiada en el contexto de chaperonas moleculares y sus funciones protectoras en levaduras y bacterias11,12,13. Se han publicado varios protocolos que utilizan una variedad de técnicas bioquímicas para el aislamiento y análisis de agregados de proteínas insolubles14,15,16,17. Los protocolos existentes se han utilizado principalmente para estudiar la agregación de proteínas bacterianas tras el choque térmico y/o la identificación de chaperonas moleculares. Si bien estos protocolos ciertamente han sido un avance en el campo, hay algunos inconvenientes importantes en los procedimientos experimentales porque requieren (i) un gran volumen de cultivo bacteriano de hasta 10 L14,17, (ii) procesos complicados de interrupción física, incluido el uso de disruptores celulares, prensa francesa y / o sonicación14,15,17o (iii) procesos repetidos que consumen mucho tiempo pasos de lavado e incubación15,16,17.
Este artículo describe un protocolo modificado que tiene como objetivo abordar las limitaciones de los enfoques anteriores y permite el análisis de la cantidad de agregados de proteínas formados en dos cepas diferentes de Escherichia coli después del tratamiento con un recubrimiento superficial antimicrobiano proteotóxico. El recubrimiento está compuesto por metal-plata (Ag) y rutenio (Ru)-acondicionado con ácido ascórbico, y su actividad antimicrobiana se logra mediante la generación de especies reactivas de oxígeno8,18. Aquí hay una descripción detallada de la preparación del cultivo bacteriano después del tratamiento con el compuesto antimicrobiano y una comparación del estado de agregación de proteínas tras la exposición de dos cepas de E. coli con perfiles de susceptibilidad distintos a la concentración creciente del antimicrobiano. El método descrito es barato, rápido y reproducible y se puede utilizar para estudiar la agregación de proteínas en presencia de otros compuestos proteotóxicos. Además, el protocolo se puede modificar para analizar el impacto que las deleciones genéticas específicas tienen en la agregación de proteínas en una variedad de bacterias diferentes.
Protocolo
1. Tratamiento de estrés de las cepas de E. coli MG1655 y CFT073
- Inocular 5 ml de caldo de lisogenia (LB) medio con una sola colonia de cepa commensal de E. coli MG1655 y cepa uropatógena de E. coli (UPEC) CFT073, respectivamente, e incubar durante 14-16 h (durante la noche) a 37 °C y 300 rpm.
NOTA: Escherichia coli CFT073 es un patógeno humano. El manejo de CFT073 debe realizarse con las medidas de bioseguridad apropiadas en un laboratorio certificado de Bioseguridad Nivel-2. - Diluir cada cepa en un matraz de 500 ml que contenga 70 ml de ácido 3-( N-morfolino)propanosulfónico (MOPS)-glucosa (MOPS-g) (Tabla 1) medio a una densidad óptica a 600 nm (OD600) valor de 0,1. Incubar a 37 °C y 300 rpm hasta que se alcance la fase logarítlica media (OD600 = 0.5-0.55).
- Transfiera 20 ml de cada cultivo a tres matraces precalentados de 125 ml e incube a 37 °C y 300 rpm durante 2 min.
NOTA: Como se requiere el procesamiento oportuno de las muestras, no maneje más de 6 cultivos a la vez. - Preparar una solución de compuesto antimicrobiano en medio MOPS-g a una concentración de 2 mg/ml. Añadir el antimicrobiano a cada cultivo para alcanzar las concentraciones indicadas. Para el control no tratado, agregue el volumen requerido de medio MOPS-g.
NOTA: Vórtice la solución antimicrobiana de 2 mg/ml para permitir una distribución uniforme de las partículas compuestas y evitar la sedimentación. - Incubar los cultivos durante 45 min a 37 °C y 300 rpm.
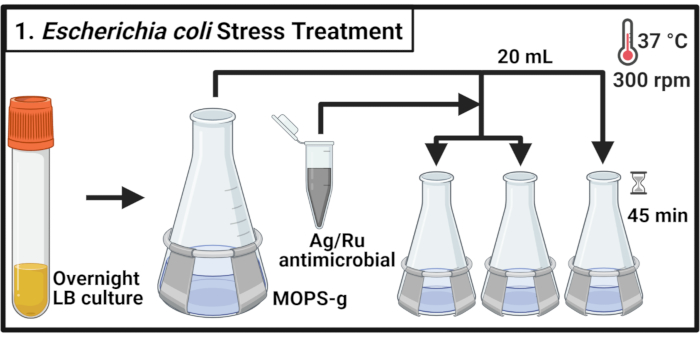
Figura 1: Tratamiento del estrés por Escherichia coli. Los cultivos bacterianos se cultivan en MOPS-g y se tratan con las concentraciones indicadas del antimicrobiano que contiene plata-rutenio cuando se alcanza la fase logarítrica media. Abreviaturas: LB = caldo de lisogenia; Ag-Ru = plata-rutenio; MOPS-g = 3-( N-morfolino)ácido propanosulfónico (MOPS)-glucosa. Haga clic aquí para ver una versión más grande de esta figura.
2. Recolección de muestras de células bacterianas
- Después de 45 min de tratamiento de estrés, determine el OD600 de cada cultivo. Para cada muestra, células de cosecha equivalentes a 4 mL de OD600 = 1 en tubos centrífugo de 15 mL por centrifugación durante 15 min a 3.000 × g y 4 °C.
- Retire completamente el sobrenadante y resuspón los gránulos celulares en 50 μL de tampón de lisis helado (Tabla 1). Incubar las muestras durante 30 min sobre hielo.
NOTA: Este paso de lisis degrada la capa de peptidoglicano. Utilice siempre un tampón de lisis recién preparado. - Transfiera las muestras a tubos de microcentrífuga de 1,7 ml. Congelar a -80 °C hasta su uso posterior.
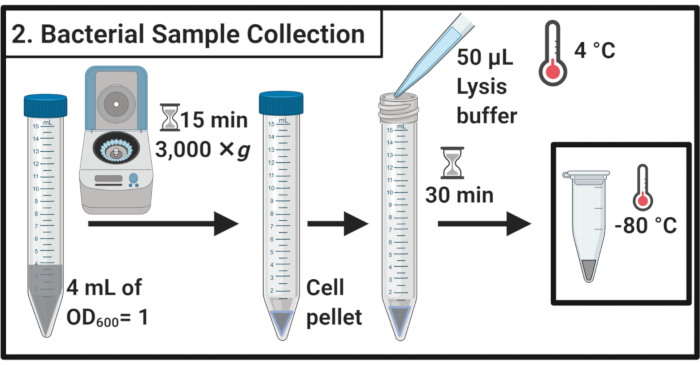
Figura 2: Recolección de muestras bacterianas. Las muestras celulares se cosechan por centrifugación y se resuspenden en tampón de lisis seguido de almacenamiento a -80 ° C. Haga clic aquí para ver una versión más grande de esta figura.
3. Extracción de los agregados de proteínas insolubles
- Descongelar muestras en hielo.
NOTA: El ciclo de congelación-descongelación contribuye a la lisis celular. - Añadir 360 μL de tampón helado A (Tabla 1) y mezclar suavemente mediante pipeteo.
NOTA: El choque osmótico también contribuirá a la lisis celular. - Transfiera la muestra a un tubo de microcentrífuga de 2 ml que contenga ~ 200 μL de perlas de vidrio de 0,5 mm. Incubar durante 30 min a 8 °C en un termomezclador con agitación a 1.400 rpm.
NOTA: Este paso da como resultado la interrupción física de la célula. Se puede usar un inhibidor de la proteasa para minimizar la degradación de proteínas. La interrupción se puede realizar a 4 °C. Tenga en cuenta que se ha informado que el uso de perlas de vidrio induce la agregación de un pequeño subconjunto de proteínas en la levadura19. - Incubar durante 5 min sobre hielo sin agitar para asentar las perlas de vidrio. Transfiera 200 μL del lisato celular a tubos de microcentrífuga de 1,7 ml.
NOTA: Evite la transferencia de las cuentas de vidrio. - Centrifugadora a 16.000 × g y 4 °C durante 20 min. Recoja el sobrenadante, que contiene proteínas solubles, y proceda a la sección 4.
- Resuspend el pellet en 200 μL de tampón helado A (Tabla 1) utilizando la pipeta. Centrifugadora a 16.000 × g y 4 °C durante 20 min. Retire con cuidado el sobrenadante por completo.
- Añadir 200 μL de tampón B helado (véase la Tabla 1 y la Tabla de Materiales)y resuspender cuidadosamente el pellet mediante pipeteo.
NOTA: El detergente no iónico solubiliza la proteína de membrana. - Repetir la centrifugación a 16.000 × g y 4 °C durante 20 min. Retire con cuidado el sobrenadante.
- Resuspend el pellet en 200 μL de tampón frío A (Tabla 1) mediante pipeteo. Centrifugadora a 16.000 × g y 4 °C durante 20 min. Retire completamente el sobrenadante.
- Vuelva a colocar el pellet en 100 μL de tampón de muestra SDS reductor 1x (Tabla 1) y hierva durante 5 min a 95 °C en un termomezclador.
- Guarde la muestra a -20 °C para continuar más tarde o cargarla inmediatamente en un gel de poliacrilamida SDS para su separación.
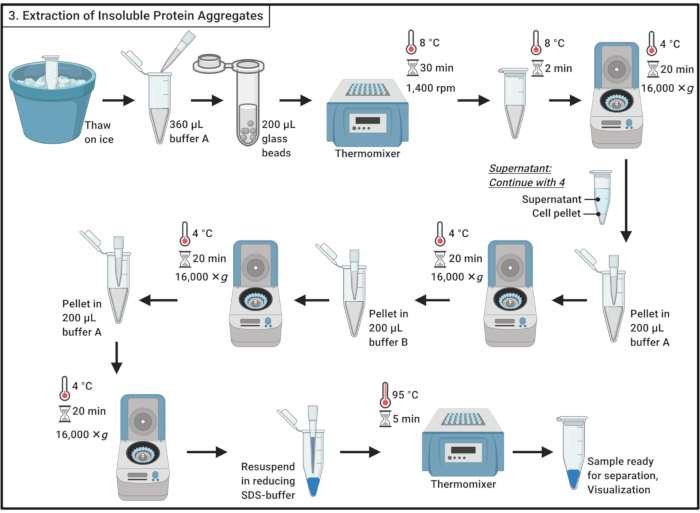
Figura 3: Extracción de agregados proteicos insolubles. La extracción de agregados de proteínas implica una serie de pasos que incluyen la interrupción celular, la separación de agregados de proteínas de proteínas de proteínas solubles, la solubilización de proteínas de membrana y el lavado. Abreviatura: SDS = dodecil sulfato de sodio. Haga clic aquí para ver una versión más grande de esta figura.
4. Preparación de muestras de proteína soluble
- Mezclar 1 volumen de ácido tricloroacético (TCA) 100% con 4 volúmenes de muestra de proteína soluble del paso 3.6.
NOTA: El manejo de TCA requiere una campana extractora de humos y equipo de protección personal y un procedimiento de eliminación de desechos aprobado. - Incubar durante 10 min a 4 °C para permitir la precipitación de proteínas.
NOTA: El precipitado blanco aparecerá muy pronto. - Centrifugar para precipitar a 21.000 × g y 4 °C durante 5 min y retirar el sobrenadante. Lave el pellet con 200 μL de acetona helada para eliminar los desechos celulares. Centrifugar a 21.000 × g y 4 °C durante 5 min y retirar el sobrenadante. Repita estas acciones en el paso 4.3 un total de tres veces.
- Coloque los tubos de la microcentrífuga con tapas abiertas en un termomezclador a 37 °C para eliminar la acetona restante del pellet.
NOTA: La incubación de más de 5 min puede reducir la solubilidad del pellet de proteína. - Añadir 100 μL de tampón SDS reductor 1x (Tabla 1) y disolver completamente el pellet. Hervir la muestra durante 5 min a 95 °C.
- Almacene la muestra a -20 °C para continuar más tarde o cargue inmediatamente en un gel de poliacrilamida SDS para su separación.
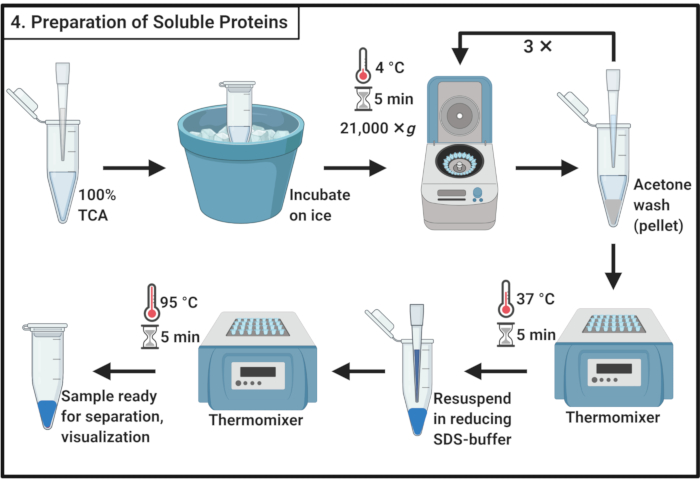
Figura 4: Preparación de proteínas solubles. La preparación de proteína soluble implica un paso de precipitación con ácido tricloroacético y un lavado repetido con acetona helada. Abreviaturas: TCA = ácido tricloroacético; SDS = dodecil sulfato de sodio. Haga clic aquí para ver una versión más grande de esta figura.
5. Separación y visualización de agregados proteicos extraídos utilizando SDS-PAGE
- Prepare un gel de poliacrilamida SDS al 12%.
- Para dos geles separadores, pipetee 5,1 ml de agua de doble destilación (ddH2O), 3,75 ml de solución de Tris-HCl (pH 8,8), 7,5 ml de SDS al 20% (p/v), 6 ml de solución de acrilamida/bisacrilamida al 30 % (29:1), 75 ml de persulfato de amonio al 10% y 10 ml de tetrametilendiamina (TEMED) en un tubo de centrífuga de 15 ml y mezcle suavemente sin introducir burbujas de aire. Vierta el gel con una pipeta de 1 ml dentro de las placas de vidrio, dejando los 2 cm superiores libres de la mezcla. Agregue un 70% de etanol en la parte superior del gel separador y permita una interfaz uniforme entre las dos capas.
- Después de la polimerización del gel separador, prepare el gel de apilamiento pipeteando 1.535 mL de ddH2O, 625 mL de Solución de Tris-HCl (pH 6.8), 12.5 mL de SDS al 20% (p/v), 335 mL de solución de acrilamida/bisacrilamida al 30% (29:1), 12.5 mL de persulfato de amonio al 10% y 2.5 mL de TEMED. Retire el etanol de los geles separadores y agregue la solución de gel de apilamiento. Inserte un peine con el número deseado de bolsillos sin introducir burbujas de aire. Permitir la polimerización durante 20-30 min.
- Cargue 4 μL de cada muestra y escalera de proteínas en pozos separados y ejecute el gel (s) en el tampón tris-Glycine (Tabla 1) a 144 V durante 45 min a temperatura ambiente.
NOTA: Detenga el gel cuando la banda de bromofenoles esté a punto de migrar fuera del gel. - Manche el gel o geles en una solución A de Fairbanks precalenada (Tabla 1) durante 30 min en un balancín.
- Decolore el gel (s) en una solución D de Fairbanks precalentada(Tabla 1)hasta el fondo deseado (por ejemplo, durante la noche) en un balancín.
Figura 5: Separación y visualización de proteínas. Las muestras se separan por SDS-PAGE y se visualizan mediante tinción de Coomassie. Abreviatura: SDS-PAGE = electroforesis en gel de dodecil sulfato de sodio-poliacrilamida. Haga clic aquí para ver una versión más grande de esta figura.
Resultados
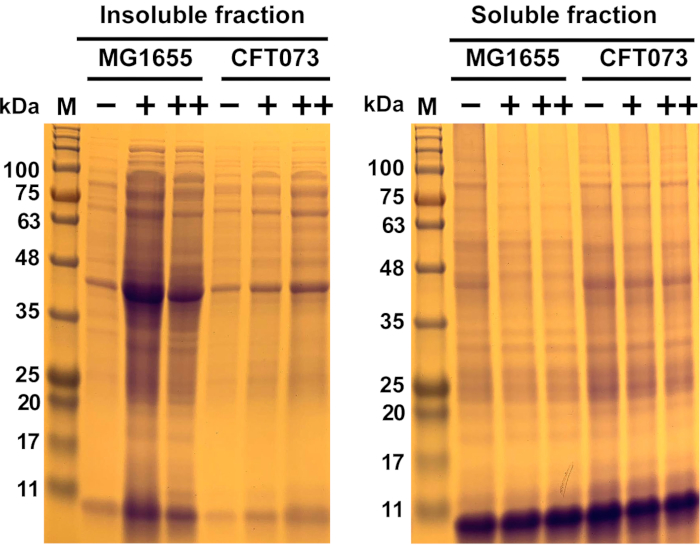
Figura 6: Resultados representativos de la agregación de proteínas inducida por antimicrobianos en la cepa commensal de Escherichia coli MG1655 y la cepa UPEC CFT073. Las cepas de E. coli MG1655 y CFT073 se cultivaron a 37 °C y 300 rpm a OD600= 0,5-0,55 en medios MOPS-g antes de ser tratadas con las concentraciones indicadas (-...
Discusión
Este protocolo describe una metodología optimizada para el análisis de la formación de agregados proteicos después del tratamiento de diferentes cepas de E. coli con un antimicrobiano proteotóxico. El protocolo permite la extracción simultánea de fracciones de proteínas insolubles y solubles de células de E. coli tratadas y no tratadas. En comparación con los protocolos existentes para el aislamiento de agregados de proteínas de las células14,
Divulgaciones
Los autores no tienen nada que revelar.
Agradecimientos
Este trabajo fue apoyado por los fondos de inicio de la Facultad de Ciencias Biológicas de la Universidad Estatal de Illinois, la Subvención de la Iniciativa de Nueva Facultad de la Universidad Estatal de Illinois y la subvención del NIAID R15AI164585 (a J.-U. D.). G.M.A. fue apoyado por el Programa de Apoyo a la Investigación de Pregrado de la Universidad Estatal de Illinois (a G.M.A.). K. P. H. fue apoyado por una beca RISE proporcionada por el Servicio Alemán de Intercambio Académico (DAAD). Los autores agradecen al Dr. Uwe Landau y al Dr. Carsten Meyer de Largentech Vertriebs GmbH por proporcionar el polvo AGXX. Las Figuras 1, Figura 2, Figura 3, Figura 4y Figura 5 se generaron con Biorender.
Materiales
| Name | Company | Catalog Number | Comments |
| Chemicals/Reagents | |||
| Acetone | Fisher Scientific | 67-64-1 | |
| 30% Acrylamide/Bisacrylamide solution 29:1 | Bio-Rad | 1610156 | |
| Ammonium persulfate | Millipore Sigma | A3678-100G | |
| Benzonase nuclease | Sigma | E1014-5KU | |
| Bluestain 2 Protein ladder, 5-245 kDa | GoldBio | P008-500 | |
| β-mercaptoethanol | Millipore Sigma | M6250-100ML | |
| Bromophenol blue | GoldBio | B-092-25 | |
| Coomassie Brilliant Blue R-250 | MP Biomedicals LLC | 821616 | |
| D-Glucose | Millipore Sigma | G8270-1KG | |
| D-Sucrose | Acros Organics | 57-50-1 | |
| Ethylenediamine tetra acetic acid (EDTA) | Sigma-Aldrich | SLBT9686 | |
| Glacial Acetic acid | Millipore Sigma | ARK2183-1L | |
| Glycerol, 99% | Sigma-Aldrich | G5516-1L | |
| Glycine | GoldBio | G-630-1 | |
| Hydrochloric acid, ACS reagent | Sigma-Aldrich | 320331-2.5L | |
| Isopropanol (2-Propanol) | Sigma | 402893-2.5L | |
| LB broth (Miller) | Millipore Sigma | L3522-1KG | |
| LB broth with agar (Miller) | Millipore Sigma | L2897-1KG | |
| Lysozyme | GoldBio | L-040-25 | |
| 10x MOPS Buffer | Teknova | M2101 | |
| Nonidet P-40 | Thomas Scientific | 9036-19-5 | |
| Potassium phosphate, dibasic | Sigma-Aldrich | P3786-1KG | |
| Potassium phosphate, monobasic | Acros Organics | 7778-77-0 | |
| Sodium dodecyl sulfate (SDS) | Sigma-Aldrich | L3771-500G | |
| Tetramethylethylenediamine (TEMED) | Millipore Sigma | T9281-50ML | |
| Thiamine | Sigma-Aldrich | T4625-100G | |
| 100% Trichloroacetic acid | Millipore Sigma | T6399-100G | |
| Tris base | GoldBio | T-400-1 | |
| Material/Equipment | |||
| Centrifuge tubes (15 mL) | Alkali Scientific | JABG-1019 | |
| Erlenmeyer flask (125 mL) | Carolina | 726686 | |
| Erlenmeyer flask (500 mL) | Carolina | 726694 | |
| Freezer: -80 °C | Fisher Scientific | ||
| Glass beads (0.5 mm) | BioSpec Products | 1107-9105 | |
| Microcentrifuge | Hermle | Z216MK | |
| Microcentriguge tubes (1.7 mL) | VWR International | 87003-294 | |
| Microcentriguge tubes (2.0 mL) | Axygen Maxiclear Microtubes | MCT-200-C | |
| Plastic cuvettes | Fischer Scientific | 14-377-012 | |
| Power supply | ThermoFisher Scientific | EC105 | |
| Rocker | Alkali Scientific | RS7235 | |
| Shaking incubator (37 °C) | Benchmark Scientific | ||
| Small glass plate | Bio-Rad | 1653311 | |
| Spacer plates (1 mm) | Bio-Rad | 1653308 | |
| Spectrophotometer | Thermoscientific | 3339053 | |
| Tabletop centrifuge for 15 mL centrifuge tubes | Beckman-Coulter | ||
| Vertical gel electrophoresis chamber | Bio-Rad | 1658004 | |
| Vortexer | Fisher Vortex Genie 2 | 12-812 | |
| Thermomixer | Benchmark Scientific | H5000-HC | |
| 10 well comb | Bio-Rad | 1653359 |
Referencias
- Dahl, J. -. U., et al. HdeB functions as an acid-protective chaperone in bacteria. Journal of Biological Chemistry. 290 (1), 65-75 (2015).
- Foit, L., George, J. S., Zhang, B. W., Brooks, C. L., Bardwell, J. C. A. Chaperone activation by unfolding. Proceedings of the National Academy of Sciences of the United States of America. 110 (14), 1254-1262 (2013).
- Sultana, S., Foti, A., Dahl, J. -. U. Bacterial defense systems against the neutrophilic oxidant hypochlorous acid. Infection and Immunity. 88 (7), 00964 (2020).
- Dahl, J. -. U., Gray, M. J., Jakob, U. Protein quality control under oxidative stress conditions. Journal of Molecular Biology. 427 (7), 1549-1563 (2015).
- Groitl, B., Dahl, J. -. U., Schroeder, J. W., Jakob, U. Pseudomonas aeruginosa defense systems against microbicidal oxidants. Molecular Microbiology. 106 (3), 335-350 (2017).
- Casadevall, A. Thermal restriction as an antimicrobial function of fever. PLoS Pathogens. 12 (5), 1005577 (2016).
- Richter, K., Haslbeck, M., Buchner, J. The heat shock response: life on the verge of death. Molecular Cell. 40 (2), 253-266 (2010).
- Van Loi, V., Busche, T., Preuß, T., Kalinowski, J., Bernhardt, J. The AGXX ® antimicrobial coating causes a thiol-specific oxidative stress response and protein S-bacillithiolation in Staphylococcus aureus. Frontiers in Microbiology. 9, 3037 (2018).
- Anfinsen, C. B., Scheraga, H. A. Experimental and theoretical aspects of protein folding. Advances in Protein Chemistry. 29, 205-300 (1975).
- Schramm, F. D., Schroeder, K., Jonas, K. Protein aggregation in bacteria. FEMS Microbiology Reviews. 44 (1), 54-72 (2020).
- Tomoyasu, T., Mogk, A., Langen, H., Goloubinoff, P., Bukau, B. Genetic dissection of the roles of chaperones and proteases in protein folding and degradation in the Escherichia coli cytosol. Molecular Microbiology. 40 (2), 397-413 (2001).
- Gray, M. J., et al. Polyphosphate is a primordial chaperone. Molecular Cell. 53 (5), 689-699 (2014).
- Weids, A. J., Ibstedt, S., Tamás, M. J., Grant, C. M. Distinct stress conditions result in aggregation of proteins with similar properties. Scientific Reports. 6, 24554 (2016).
- Mogk, A., et al. Identification of thermolabile Escherichia coli proteins: prevention and reversion of aggregation by DnaK and ClpB. EMBO Journal. 18 (24), 6934-6949 (1999).
- Fay, A., Glickman, M. S. An essential nonredundant role for mycobacterial DnaK in native protein folding. PLoS Genetics. 10 (7), 1004516 (2014).
- Schramm, F. D., Heinrich, K., Thüring, M., Bernhardt, J., Jonas, K. An essential regulatory function of the DnaK chaperone dictates the decision between proliferation and maintenance in Caulobacter crescentus. PLoS Genetics. 13 (12), 1007148 (2017).
- Maisonneuve, E., Fraysse, L., Moinier, D., Dukan, S. Existence of abnormal protein aggregates in healthy Escherichia coli cells. Journal of Bacteriology. 190 (3), 887-893 (2008).
- Heiss, A., Freisinger, B., Held-Föhn, E. Enhanced antibacterial activity of silver-ruthenium coated hollow microparticles. Biointerphases. 12 (5), (2017).
- Papnayotou, I., Sun, B., Roth, A. F., Davis, N. G. Protein aggregation induced during glass bead lysis of yeast. Yeast. 27 (10), 801-816 (2010).
- Chuang, S. E., Blattner, F. R. Characterization of twenty-six new heat shock genes of Escherichia coli. Journal of Bacteriology. 175 (16), 5242-5252 (1993).
- Imlay, J. A. The molecular mechanisms and physiological consequences of oxidative stress: Lessons from a model bacterium. Nature Reviews Microbiology. 11 (7), 443-454 (2013).
- Mühlhofer, M., et al. The heat shock response in yeast maintains protein homeostasis by chaperoning and replenishing proteins. Cell Reports. 29 (13), 4593-4607 (2019).
- Chandrangsu, P., Rensing, C., Helmann, J. D. Metal homeostasis and resistance in bacteria. Nature Reviews Microbiology. 15, 338-350 (2017).
- Stevens, M., et al. HSP60/10 chaperonin systems are inhibited by a variety of approved drugs, natural products, and known bioactive molecules. Bioorganic and Medicinal Chemistry Letters. 29 (9), 1106-1112 (2019).
- Schramm, F. D., Schroeder, K., Alvelid, J., Testa, I., Jonas, K. Growth-driven displacement of protein aggregates along the cell length ensures partitioning to both daughter cells in Caulobacter crescentus. Molecular Microbiology. 111 (6), 1430-1448 (2019).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados