É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Extração e Visualização de Agregados proteicos após tratamento de Escherichia coli com um Estressor Proteotóxico
Neste Artigo
Resumo
Este protocolo descreve a extração e visualização de proteínas agregadas e solúveis de Escherichia coli após o tratamento com um antimicrobiano proteotóxico. Após este procedimento permite uma comparação qualitativa da formação de agregado de proteínas in vivo em diferentes cepas bacterianas e/ou entre os tratamentos.
Resumo
A exposição de organismos vivos a estresses ambientais e celulares muitas vezes causa interrupções na homeostase proteica e pode resultar em agregação de proteínas. O acúmulo de agregados proteicos em células bacterianas pode levar a alterações significativas no comportamento fenotípico celular, incluindo uma redução nas taxas de crescimento, resistência ao estresse e virulência. Existem vários procedimentos experimentais para o exame desses fenótipos mediados pelo estressor. Este artigo descreve um ensaio otimizado para a extração e visualização de proteínas agregadas e solúveis de diferentes cepas de Escherichia coli após o tratamento com um antimicrobiano contendo prata-rutênio. Este composto é conhecido por gerar espécies reativas de oxigênio e causa agregação generalizada de proteínas.
O método combina uma separação baseada em centrifugação de agregados proteicos e proteínas solúveis de células tratadas e não tratadas com posterior separação e visualização por eletroforese de gel de sulfato-poliacrilamida de sódio (SDS-PAGE) e coloração coomassie. Esta abordagem é simples, rápida e permite uma comparação qualitativa da formação de proteína agregada em diferentes cepas de E. coli. A metodologia possui uma ampla gama de aplicações, incluindo a possibilidade de investigar o impacto de outros antimicrobianos proteotóicos na agregação de proteínas in vivo em uma ampla gama de bactérias. Além disso, o protocolo pode ser usado para identificar genes que contribuem para o aumento da resistência às substâncias proteotoxias. Bandas de gel podem ser usadas para a identificação subsequente de proteínas particularmente propensas à agregação.
Introdução
As bactérias são inevitavelmente expostas a uma miríade de estresses ambientais, incluindo pH baixo (por exemplo, no estômago dos mamíferos)1,2, oxigênio reativo e espécies de cloro (ROS/RCS) (por exemplo, durante a explosão oxidativa em fagocitos)3,4,5, temperaturas elevadas (por exemplo, em fontes termais ou durante o choque térmico)6,7, e vários antimicrobianos potentes (por exemplo, AGXX usados neste protocolo)8. As proteínas são particularmente vulneráveis a qualquer um desses estressores, e a exposição pode provocar a não-/misfolding de proteínas que, em seguida, a agregação de sementes. Todos os organismos empregam sistemas de proteção que lhes permitem lidar com o erro de proteína9. No entanto, o estresse severo pode sobrecarregar o maquinário de controle de qualidade proteica e interromper a estrutura secundária e/ou terciária das proteínas, o que acaba inativando proteínas. Como consequência, os agregados proteicos podem prejudicar severamente as funções celulares críticas necessárias para o crescimento e sobrevivência bacteriana, resistência ao estresse e virulência10. Portanto, pesquisas com foco na agregação de proteínas e suas consequências nas bactérias são um tema relevante devido ao seu potencial impacto no controle de doenças infecciosas.
O desdobramento e a agregação de proteínas induzidas pelo calor são muitas vezes reversíveis7. Em contraste, outras tensões proteotoxiais, como o estresse oxidativo, podem causar modificações proteicas irreversíveis através da oxidação de cadeias laterais específicas de aminoácidos resultando em proteínas não-/misfolding e, eventualmente, agregação deproteínas 4. A formação induzida pelo estresse de agregados proteicos insolúveis tem sido extensivamente estudada no contexto de acompanhantes moleculares e suas funções protetoras na levedura e bactérias11,12,13. Vários protocolos foram publicados que utilizam uma variedade de técnicas bioquímicas para o isolamento e análise dos agregados de proteínas insolúveis14,15,16,17. Os protocolos existentes têm sido usados principalmente para estudar a agregação de proteínas bacterianas mediante choque térmico e/ou identificação de acompanhantes moleculares. Embora esses protocolos tenham sido certamente um avanço para o campo, há alguns grandes inconvenientes nos procedimentos experimentais porque exigem (i) um grande volume de cultura bacteriana de até 10 L14,17, (ii) complicados processos de interrupção física, incluindo o uso de disruptores celulares, imprensa francesa e/ou sonicação14,15,17, ou (iii) demorado repetidos lalavís e incubação etapas15,16,17.
Este artigo descreve um protocolo modificado que visa abordar as limitações das abordagens anteriores e permite a análise da quantidade de agregados proteicos formados em duas cepas diferentes de Escherichia coli após o tratamento com um revestimento de superfície antimicrobiana proteotoxic. O revestimento é composto de metal-prata (Ag) e rutênio (Ru)-condicionado com ácido ascórbico, e sua atividade antimicrobiana é alcançada pela geração de espécies reativas de oxigênio8,18. Aqui está uma descrição detalhada da preparação da cultura bacteriana após o tratamento com o composto antimicrobiano e uma comparação do estado de agregação de proteínas após a exposição de duas cepas de E. coli com perfis distintos de suscetibilidade ao aumento da concentração do antimicrobiano. O método descrito é barato, rápido e reprodutível e pode ser usado para estudar a agregação de proteínas na presença de outros compostos proteotóxicos. Além disso, o protocolo pode ser modificado para analisar o impacto que exclusões genéticas específicas têm na agregação de proteínas em uma variedade de bactérias diferentes.
Protocolo
1. Tratamento de estresse de E. coli cepas MG1655 e CFT073
- Inocula 5 mL de caldo de lisogenia (LB) médio com uma única colônia de cepa E. coli commensal MG1655 e uropatogenic E. coli (UPEC) cepa CFT073, respectivamente, e incubar por 14-16 h (durante a noite) a 37 °C e 300 rpm.
NOTA: Escherichia coli CFT073 é um patógeno humano. O manuseio do CFT073 deve ser realizado com medidas de biossegurança adequadas em um laboratório certificado de Biossegurança Nível-2. - Diluir cada cepa em um frasco de 500 mL contendo70 mL de ácido propanossulfônico (MOPS)-glicose (MOPS-g)(Tabela 1)médio a uma densidade óptica de 600 nm (OD600) valor de 0,1. Incubar a 37 °C e 300 rpm até a fase de registro médio (OD600 = 0,5-0,55).
- Transfira 20 mL de cada cultura em três frascos pré-armados de 125 mL e incubar a 37 °C e 300 rpm por 2 min.
NOTA: Como o processamento oportuno das amostras é necessário, manuseie não mais do que 6 culturas por vez. - Prepare uma solução composta antimicrobiana em meio MOPS-g a uma concentração de 2 mg/mL. Adicione o antimicrobiano a cada cultura para alcançar as concentrações indicadas. Para o controle não tratado, adicione o volume necessário de meio MOPS-g.
NOTA: Vortex a solução antimicrobiana de 2 mg/mL para permitir uma distribuição uniforme das partículas compostas e evitar a sedimentação. - Incubar as culturas por 45 min a 37 °C e 300 rpm.
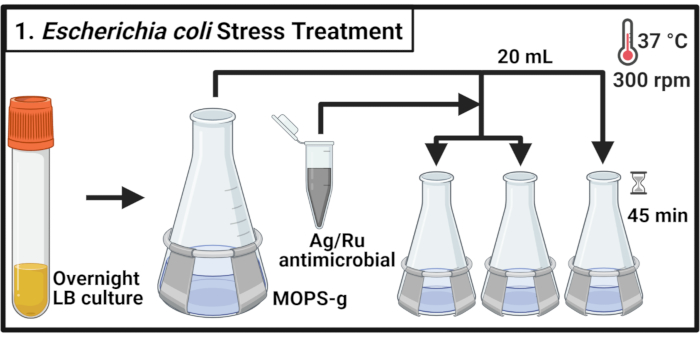
Figura 1: Tratamento de estresse Escherichia coli. As culturas bacterianas são cultivadas em MOPS-g e tratadas com as concentrações indicadas do antimicrobiano contendo rutênio prateado quando a fase de registro médio é atingida. Abreviaturas: LB = caldo de lysogeny; Ag-Ru = prata-rutênio; MOPS-g = 3-(N-morpholino)ácido propanesulfônico (MOPS)-glicose. Clique aqui para ver uma versão maior desta figura.
2. Coleta de amostras de células bacterianas
- Após 45 minutos de tratamento de estresse, determine o OD600 de cada cultura. Para cada amostra, as células de colheita equivalem a 4 mL de OD600 = 1 em tubos de centrífugas de 15 mL por centrifugação por centrifugação por 15 min a 3.000 × g e 4 °C.
- Remova completamente o supernasciente e resuspense as pelotas de célula em 50 μL de tampão de lise gelada(Tabela 1). Incubar as amostras por 30 minutos no gelo.
NOTA: Este passo de lise degrada a camada peptidoglycan. Use sempre o tampão de lise recém-preparado. - Transfira as amostras para tubos de microcentrifuuge de 1,7 mL. Congele a -80 °C até que seja mais útil.
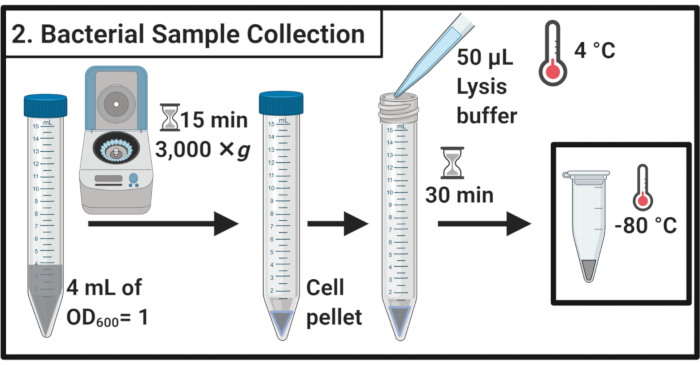
Figura 2: Coleta de amostras bacterianas. As amostras de células são colhidas por centrifugação e resuspensadas no tampão de lise seguido de armazenamento a -80 °C. Clique aqui para ver uma versão maior desta figura.
3. Extrair os agregados proteicos insolúveis
- Descongelar amostras no gelo.
NOTA: O ciclo de congelamento contribui para a lise celular. - Adicione 360 μL de tampão gelado A(Tabela 1) e misture delicadamente por pipetação.
NOTA: O choque osmótico também contribuirá para a lise celular. - Transfira a amostra para um tubo de microcentrifuuge de 2 mL contendo ~200 μL de contas de vidro de 0,5 mm. Incubar por 30 min a 8 °C em um termomixer com agitação a 1.400 rpm.
NOTA: Esta etapa resulta na ruptura física da célula. Um inibidor de protease pode ser usado para minimizar a degradação da proteína. A interrupção pode ser realizada a 4 °C. Note-se que o uso de contas de vidro foi relatado para induzir a agregação de um pequeno subconjunto de proteínas na levedura19. - Incubar por 5 minutos no gelo sem tremer para assar as contas de vidro. Transfira 200 μL do lysato celular em tubos de microcentrifuuge de 1,7 mL.
NOTA: Evite a transferência das contas de vidro. - Centrifugar a 16.000 × g e 4 °C por 20 min. Recolher o supernante, que contém proteínas solúveis, e seguir para a seção 4.
- Resuspenda a pelota em 200 μL de tampão gelado A (Tabela 1) usando a pipeta. Centrifugar a 16.000 × g e 4 °C por 20 min. Remova cuidadosamente o sobrenante completamente.
- Adicione 200 μL de tampão gelado B (ver Tabela 1 e a Tabela de Materiais) e resuspense cuidadosamente a pelota por pipetação.
NOTA: O detergente não iônico solubiliza a proteína da membrana. - Repita a centrifugação a 16.000 × g e 4 °C por 20 min. Remova cuidadosamente o supernatante.
- Resuspenda a pelota em 200 μL de tampão frio A (Tabela 1) por pipetação. Centrifugar a 16.000 × g e 4 °C por 20 min. Remova completamente o supernatante.
- Resuspenda a pelota em 100 μL de 1x reduzindo o tampão de amostra SDS(Tabela 1) e ferva por 5 min a 95 °C em um termomixer.
- Armazene a amostra a -20 °C para prosseguir mais tarde ou carregar imediatamente em um gel de poliacrilamida SDS para separação.
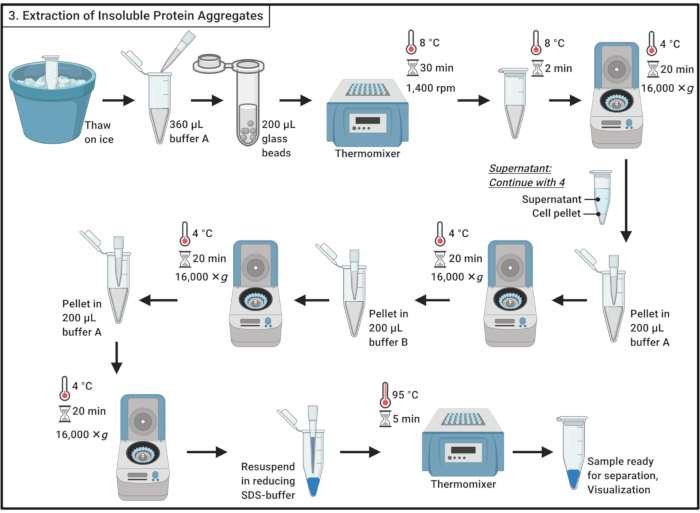
Figura 3: Extração de agregados proteicos insolúveis. A extração de agregados proteicos envolve uma série de etapas, incluindo a ruptura celular, a separação de agregados proteicos de proteínas solúveis, a solubilização de proteínas de membrana e a lavagem. Abreviação: SDS = sulfato de dodecyl de sódio. Clique aqui para ver uma versão maior desta figura.
4. Preparação da amostra de proteína solúvel
- Misture 1 volume de ácido 100% tricloroacético (TCA) com 4 volumes de amostra de proteína solúvel a partir do passo 3.6.
NOTA: O manuseio do TCA requer um capô de fumaça e equipamentos de proteção individual e um procedimento aprovado de descarte de resíduos. - Incubar por 10 min a 4 °C para permitir a precipitação da proteína.
NOTA: O precipitado branco aparecerá muito em breve. - Centrífuga para precipitar a 21.000 × g e 4 °C por 5 min e remover o sobrenante. Lave a pelota com 200 μL de acetona gelada para remover detritos celulares. Centrifugar a 21.000 × g e 4 °C por 5 min e remover o supernatante. Repita essas ações na etapa 4.3 um total de três vezes.
- Coloque os tubos de microcentrifuuge com tampas abertas em um termomixer a 37 °C para remover a acetona restante da pelota.
NOTA: A incubação de mais de 5 min pode reduzir a solubilidade da pelota de proteína. - Adicione 100 μL de 1x reduzindo o buffer SDS(Tabela 1) e dissolva completamente a pelota. Ferva a amostra por 5 min a 95 °C.
- Armazene a amostra a -20 °C para prosseguir mais tarde ou carregar imediatamente em um gel de poliacrilamida SDS para separação.
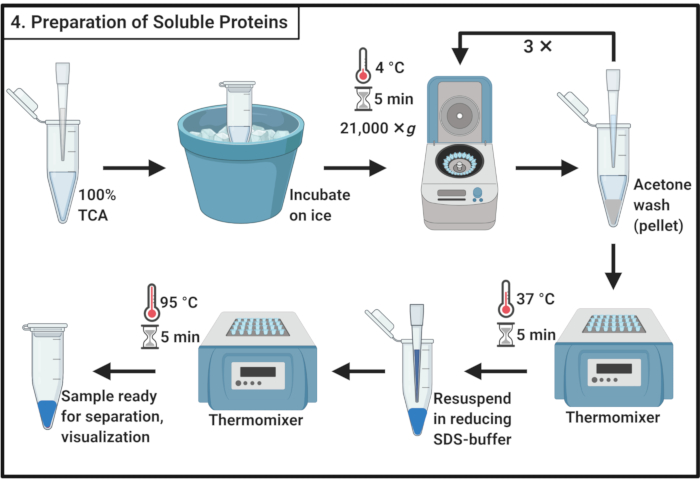
Figura 4: Preparação de proteínas solúveis. A preparação da proteína solúvel envolve um passo de precipitação com ácido tricloroacético e lavagem repetida com acetona gelada. Abreviaturas: TCA = ácido tricloroacético; SDS = sulfato de dodecyl de sódio. Clique aqui para ver uma versão maior desta figura.
5. Separação e visualização de agregados de proteínas extraídas utilizando SDS-PAGE
- Prepare um gel de poliacrilamida SDS de 12%.
- Para dois géis separados, pipeta 5,1 mL de água dupla destilada (ddH2O), 3,75 mL de Tris-HCl (pH 8,8), 7,5 mL de 20% (w/v) SDS, 6 mL de solução de acrilamida/bisacrilamida (29:1), 75 mL de persulgo de amônio de 10% w/v e 10 mL de tetrametetilenodiamina (TEMED) em um tubo de centrífugo de 15 mL e misture suavemente sem introduzir bolhas de ar. Despeje o gel usando uma tubulação de 1 mL dentro das placas de vidro, deixando a parte superior 2 cm livre da mistura. Adicione 70% de etanol na parte superior do gel de separação e permita uma interface uniforme entre as duas camadas.
- Após a polimerização do gel de separação, prepare o gel de empilhamento por pipetação de 1,535 mL de ddH2O, 625 mL de Tris-HCl (pH 6.8), 12,5 mL de 20% (w/v) SDS, 335 mL de solução de acrilamida/bisacrilamida (29:1), 12,5 mL de persulgênio de 10% w/v de amônio e 2,5 mL de TEMED. Retire o etanol dos géis de separação e adicione a solução de gel de empilhamento. Insira um pente com o número desejado de bolsos sem introduzir bolhas de ar. Deixe a polimerização por 20-30 min.
- Carregue 4 μL de cada amostra e escada proteica em poços separados e execute o gel(s) em tampão de corrida Tris-Glycine(Tabela 1) a 144 V por 45 min a temperatura ambiente.
NOTA: Pare o gel quando a banda bromofenol estiver prestes a migrar para fora do gel. - Manche o gel(s) em uma solução de Fairbanks pré-enfracaada A (Tabela 1) por 30 min em um roqueiro.
- Decolor o gel(s) em uma solução de Fairbanks pré-enloucou D(Tabela 1) até o fundo desejado (por exemplo, durante a noite) em um roqueiro.
Figura 5: Separação e visualização de proteínas. As amostras são separadas por SDS-PAGE e visualizadas pela coloração coomassie. Abreviação: SDS-PAGE = sulfato de sulfato de sódio-poliacrilamida gel eletroforese. Clique aqui para ver uma versão maior desta figura.
Resultados
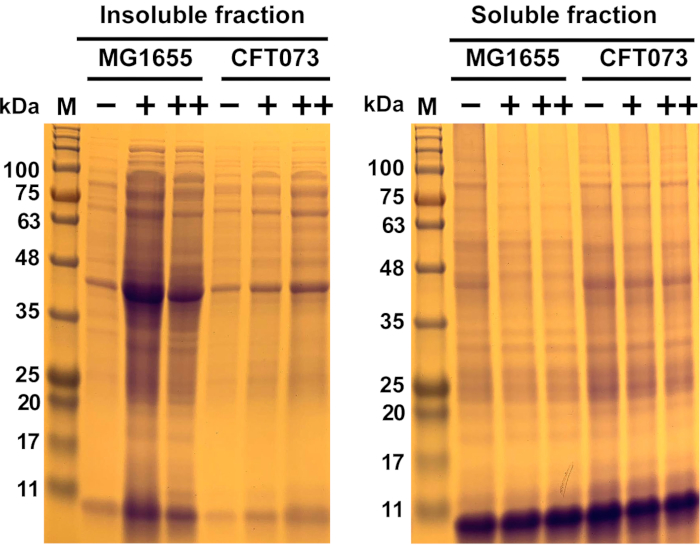
Figura 6: Resultados representativos da agregação de proteínas induzidas por antimicrobianos na cepa Escherichia coli commensal MG1655 e cepa UPEC CFT073. As cepas E. coli MG1655 e CFT073 foram cultivadas a 37 °C e 300 rpm a OD600= 0,5-0,55 na mídia MOPS-g antes de serem tratadas com as concentrações indicadas (-, 0 mg/mL; ...
Discussão
Este protocolo descreve uma metodologia otimizada para a análise da formação de agregado de proteínas após o tratamento de diferentes cepas de E. coli com um antimicrobiano proteotóxico. O protocolo permite a extração simultânea de frações proteicas insolúveis e solúveis de células E. coli tratadas e não tratadas. Em comparação com os protocolos existentes para o isolamento agregado de proteínas das células14,15,
Divulgações
Os autores não têm nada a revelar.
Agradecimentos
Este trabalho foi apoiado pelos fundos de startup da Illinois State University School of Biological Sciences, Illinois State University New Faculty Initiative Grant, e o NIAID grant R15AI164585 (para J.-U. D.). G.M.A. foi apoiado pelo Programa de Apoio à Pesquisa de Graduação da Universidade Estadual de Illinois (para G.M.A.). K. P. H. foi apoiado por uma bolsa rise fornecida pelo Serviço Alemão de Intercâmbio Acadêmico (DAAD). Os autores agradecem ao Dr. Uwe Landau e ao Dr. Carsten Meyer da Largentech Vertriebs GmbH por fornecerem o pó AGXX. Foram geradas as Figuras 1, Figura 2, Figura 3, Figura 4e Figura 5 com Biorender.
Materiais
| Name | Company | Catalog Number | Comments |
| Chemicals/Reagents | |||
| Acetone | Fisher Scientific | 67-64-1 | |
| 30% Acrylamide/Bisacrylamide solution 29:1 | Bio-Rad | 1610156 | |
| Ammonium persulfate | Millipore Sigma | A3678-100G | |
| Benzonase nuclease | Sigma | E1014-5KU | |
| Bluestain 2 Protein ladder, 5-245 kDa | GoldBio | P008-500 | |
| β-mercaptoethanol | Millipore Sigma | M6250-100ML | |
| Bromophenol blue | GoldBio | B-092-25 | |
| Coomassie Brilliant Blue R-250 | MP Biomedicals LLC | 821616 | |
| D-Glucose | Millipore Sigma | G8270-1KG | |
| D-Sucrose | Acros Organics | 57-50-1 | |
| Ethylenediamine tetra acetic acid (EDTA) | Sigma-Aldrich | SLBT9686 | |
| Glacial Acetic acid | Millipore Sigma | ARK2183-1L | |
| Glycerol, 99% | Sigma-Aldrich | G5516-1L | |
| Glycine | GoldBio | G-630-1 | |
| Hydrochloric acid, ACS reagent | Sigma-Aldrich | 320331-2.5L | |
| Isopropanol (2-Propanol) | Sigma | 402893-2.5L | |
| LB broth (Miller) | Millipore Sigma | L3522-1KG | |
| LB broth with agar (Miller) | Millipore Sigma | L2897-1KG | |
| Lysozyme | GoldBio | L-040-25 | |
| 10x MOPS Buffer | Teknova | M2101 | |
| Nonidet P-40 | Thomas Scientific | 9036-19-5 | |
| Potassium phosphate, dibasic | Sigma-Aldrich | P3786-1KG | |
| Potassium phosphate, monobasic | Acros Organics | 7778-77-0 | |
| Sodium dodecyl sulfate (SDS) | Sigma-Aldrich | L3771-500G | |
| Tetramethylethylenediamine (TEMED) | Millipore Sigma | T9281-50ML | |
| Thiamine | Sigma-Aldrich | T4625-100G | |
| 100% Trichloroacetic acid | Millipore Sigma | T6399-100G | |
| Tris base | GoldBio | T-400-1 | |
| Material/Equipment | |||
| Centrifuge tubes (15 mL) | Alkali Scientific | JABG-1019 | |
| Erlenmeyer flask (125 mL) | Carolina | 726686 | |
| Erlenmeyer flask (500 mL) | Carolina | 726694 | |
| Freezer: -80 °C | Fisher Scientific | ||
| Glass beads (0.5 mm) | BioSpec Products | 1107-9105 | |
| Microcentrifuge | Hermle | Z216MK | |
| Microcentriguge tubes (1.7 mL) | VWR International | 87003-294 | |
| Microcentriguge tubes (2.0 mL) | Axygen Maxiclear Microtubes | MCT-200-C | |
| Plastic cuvettes | Fischer Scientific | 14-377-012 | |
| Power supply | ThermoFisher Scientific | EC105 | |
| Rocker | Alkali Scientific | RS7235 | |
| Shaking incubator (37 °C) | Benchmark Scientific | ||
| Small glass plate | Bio-Rad | 1653311 | |
| Spacer plates (1 mm) | Bio-Rad | 1653308 | |
| Spectrophotometer | Thermoscientific | 3339053 | |
| Tabletop centrifuge for 15 mL centrifuge tubes | Beckman-Coulter | ||
| Vertical gel electrophoresis chamber | Bio-Rad | 1658004 | |
| Vortexer | Fisher Vortex Genie 2 | 12-812 | |
| Thermomixer | Benchmark Scientific | H5000-HC | |
| 10 well comb | Bio-Rad | 1653359 |
Referências
- Dahl, J. -. U., et al. HdeB functions as an acid-protective chaperone in bacteria. Journal of Biological Chemistry. 290 (1), 65-75 (2015).
- Foit, L., George, J. S., Zhang, B. W., Brooks, C. L., Bardwell, J. C. A. Chaperone activation by unfolding. Proceedings of the National Academy of Sciences of the United States of America. 110 (14), 1254-1262 (2013).
- Sultana, S., Foti, A., Dahl, J. -. U. Bacterial defense systems against the neutrophilic oxidant hypochlorous acid. Infection and Immunity. 88 (7), 00964 (2020).
- Dahl, J. -. U., Gray, M. J., Jakob, U. Protein quality control under oxidative stress conditions. Journal of Molecular Biology. 427 (7), 1549-1563 (2015).
- Groitl, B., Dahl, J. -. U., Schroeder, J. W., Jakob, U. Pseudomonas aeruginosa defense systems against microbicidal oxidants. Molecular Microbiology. 106 (3), 335-350 (2017).
- Casadevall, A. Thermal restriction as an antimicrobial function of fever. PLoS Pathogens. 12 (5), 1005577 (2016).
- Richter, K., Haslbeck, M., Buchner, J. The heat shock response: life on the verge of death. Molecular Cell. 40 (2), 253-266 (2010).
- Van Loi, V., Busche, T., Preuß, T., Kalinowski, J., Bernhardt, J. The AGXX ® antimicrobial coating causes a thiol-specific oxidative stress response and protein S-bacillithiolation in Staphylococcus aureus. Frontiers in Microbiology. 9, 3037 (2018).
- Anfinsen, C. B., Scheraga, H. A. Experimental and theoretical aspects of protein folding. Advances in Protein Chemistry. 29, 205-300 (1975).
- Schramm, F. D., Schroeder, K., Jonas, K. Protein aggregation in bacteria. FEMS Microbiology Reviews. 44 (1), 54-72 (2020).
- Tomoyasu, T., Mogk, A., Langen, H., Goloubinoff, P., Bukau, B. Genetic dissection of the roles of chaperones and proteases in protein folding and degradation in the Escherichia coli cytosol. Molecular Microbiology. 40 (2), 397-413 (2001).
- Gray, M. J., et al. Polyphosphate is a primordial chaperone. Molecular Cell. 53 (5), 689-699 (2014).
- Weids, A. J., Ibstedt, S., Tamás, M. J., Grant, C. M. Distinct stress conditions result in aggregation of proteins with similar properties. Scientific Reports. 6, 24554 (2016).
- Mogk, A., et al. Identification of thermolabile Escherichia coli proteins: prevention and reversion of aggregation by DnaK and ClpB. EMBO Journal. 18 (24), 6934-6949 (1999).
- Fay, A., Glickman, M. S. An essential nonredundant role for mycobacterial DnaK in native protein folding. PLoS Genetics. 10 (7), 1004516 (2014).
- Schramm, F. D., Heinrich, K., Thüring, M., Bernhardt, J., Jonas, K. An essential regulatory function of the DnaK chaperone dictates the decision between proliferation and maintenance in Caulobacter crescentus. PLoS Genetics. 13 (12), 1007148 (2017).
- Maisonneuve, E., Fraysse, L., Moinier, D., Dukan, S. Existence of abnormal protein aggregates in healthy Escherichia coli cells. Journal of Bacteriology. 190 (3), 887-893 (2008).
- Heiss, A., Freisinger, B., Held-Föhn, E. Enhanced antibacterial activity of silver-ruthenium coated hollow microparticles. Biointerphases. 12 (5), (2017).
- Papnayotou, I., Sun, B., Roth, A. F., Davis, N. G. Protein aggregation induced during glass bead lysis of yeast. Yeast. 27 (10), 801-816 (2010).
- Chuang, S. E., Blattner, F. R. Characterization of twenty-six new heat shock genes of Escherichia coli. Journal of Bacteriology. 175 (16), 5242-5252 (1993).
- Imlay, J. A. The molecular mechanisms and physiological consequences of oxidative stress: Lessons from a model bacterium. Nature Reviews Microbiology. 11 (7), 443-454 (2013).
- Mühlhofer, M., et al. The heat shock response in yeast maintains protein homeostasis by chaperoning and replenishing proteins. Cell Reports. 29 (13), 4593-4607 (2019).
- Chandrangsu, P., Rensing, C., Helmann, J. D. Metal homeostasis and resistance in bacteria. Nature Reviews Microbiology. 15, 338-350 (2017).
- Stevens, M., et al. HSP60/10 chaperonin systems are inhibited by a variety of approved drugs, natural products, and known bioactive molecules. Bioorganic and Medicinal Chemistry Letters. 29 (9), 1106-1112 (2019).
- Schramm, F. D., Schroeder, K., Alvelid, J., Testa, I., Jonas, K. Growth-driven displacement of protein aggregates along the cell length ensures partitioning to both daughter cells in Caulobacter crescentus. Molecular Microbiology. 111 (6), 1430-1448 (2019).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados