Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Extraktion und Visualisierung von Proteinaggregaten nach Behandlung von Escherichia coli mit einem proteotoxischen Stressor
In diesem Artikel
Zusammenfassung
Dieses Protokoll beschreibt die Extraktion und Visualisierung von aggregierten und löslichen Proteinen aus Escherichia coli nach Behandlung mit einem proteotoxischen antimikrobiellen Mittel. Nach diesem Verfahren kann ein qualitativer Vergleich der Proteinaggregatbildung in vivo in verschiedenen Bakterienstämmen und/oder zwischen Behandlungen ermöglicht werden.
Zusammenfassung
Die Exposition lebender Organismen gegenüber Umwelt- und Zellstress verursacht häufig Störungen in der Proteinöostase und kann zu einer Proteinaggregation führen. Die Ansammlung von Proteinaggregaten in Bakterienzellen kann zu signifikanten Veränderungen des zellulären phänotypischen Verhaltens führen, einschließlich einer Verringerung der Wachstumsraten, der Stressresistenz und der Virulenz. Für die Untersuchung dieser stressorvermittelten Phänotypen existieren mehrere experimentelle Verfahren. Dieser Artikel beschreibt einen optimierten Assay für die Extraktion und Visualisierung von aggregierten und löslichen Proteinen aus verschiedenen Escherichia coli-Stämmen nach Behandlung mit einem silber-rutheniumhaltigen antimikrobiellen Mittel. Es ist bekannt, dass diese Verbindung reaktive Sauerstoffspezies erzeugt und eine weit verbreitete Proteinaggregation verursacht.
Die Methode kombiniert eine zentrifugationsbasierte Trennung von Proteinaggregaten und löslichen Proteinen aus behandelten und unbehandelten Zellen mit anschließender Trennung und Visualisierung durch Natriumdodecylsulfat-Polyacrylamid-Gelelektrophorese (SDS-PAGE) und Coomassie-Färbung. Dieser Ansatz ist einfach, schnell und ermöglicht einen qualitativen Vergleich der Proteinaggregatbildung in verschiedenen E. coli-Stämmen. Die Methodik hat eine breite Palette von Anwendungen, einschließlich der Möglichkeit, den Einfluss anderer proteotoxischer antimikrobieller Mittel auf die In-vivo-Proteinaggregation in einer Vielzahl von Bakterien zu untersuchen. Darüber hinaus kann das Protokoll verwendet werden, um Gene zu identifizieren, die zu einer erhöhten Resistenz gegen proteotoxische Substanzen beitragen. Gelbänder können zur anschließenden Identifizierung von Proteinen verwendet werden, die besonders anfällig für Aggregation sind.
Einleitung
Bakterien sind unweigerlich einer Vielzahl von Umweltbelastungen ausgesetzt, darunter ein niedriger pH-Wert (z. B. im Säugetiermagen)1,2, reaktive Sauerstoff- und Chlorspezies (ROS / RCS) (z. B. während des oxidativen Bursts in Phagozyten)3,4,5, erhöhte Temperaturen (z. B. in heißen Quellen oder während des Hitzeschocks)6,7und mehrere starke antimikrobielle Mittel (z. B. AGXX, die in diesem Protokoll verwendet werden)8. Proteine sind besonders anfällig für einen dieser Stressoren, und die Exposition kann zu einer Proteinentfaltung führen, die dann die Aggregation aussaatiert. Alle Organismen verwenden Schutzsysteme, die es ihnen ermöglichen, mit Proteinfehlfaltungen fertig zu werden9. Starker Stress kann jedoch die Proteinqualitätskontrollmaschinerie überfordern und die sekundäre und / oder tertiäre Struktur von Proteinen stören, was letztendlich Proteine inaktiviert. Infolgedessen können Proteinaggregate kritische Zellfunktionen, die für das Wachstum und Überleben von Bakterien, die Stressresistenz und die Virulenz erforderlich sind, stark beeinträchtigen10. Daher ist die Forschung, die sich auf die Proteinaggregation und ihre Folgen in Bakterien konzentriert, aufgrund ihrer möglichen Auswirkungen auf die Kontrolle von Infektionskrankheiten ein relevantes Thema.
Hitzeinduzierte Proteinentfaltung und -aggregation sind oft reversibel7. Im Gegensatz dazu können andere proteotoxische Belastungen, wie oxidativer Stress, irreversible Proteinmodifikationen durch die Oxidation spezifischer Aminosäure-Seitenketten verursachen, was zu einer Proteinentfaltung und schließlich zu einer Proteinaggregation führt4. Die stressinduzierte Bildung unlöslicher Proteinaggregate wurde ausführlich im Zusammenhang mit molekularen Chaperonen und deren Schutzfunktionen in Hefen und Bakterien untersucht11,12,13. Es wurden mehrere Protokolle veröffentlicht, die eine Vielzahl von biochemischen Techniken zur Isolierung und Analyse von unlöslichen Proteinaggregatenverwenden 14,15,16,17. Die bestehenden Protokolle wurden hauptsächlich verwendet, um die bakterielle Proteinaggregation bei Hitzeschock und / oder die Identifizierung molekularer Chaperone zu untersuchen. Während diese Protokolle sicherlich ein Fortschritt auf dem Gebiet waren, gibt es einige große Unannehmlichkeiten in den experimentellen Verfahren, da sie (i) ein großes Bakterienkulturvolumen von bis zu 10 L14,17, (ii) komplizierte physikalische Störungsprozesse, einschließlich der Verwendung von Zelldisruptoren, französischer Presse und / oder Beschallung14,15,17oder (iii) zeitaufwendige wiederholte Wasch- und Inkubationsschritte15,16,17.
Dieser Artikel beschreibt ein modifiziertes Protokoll, das darauf abzielt, die Grenzen der vorherigen Ansätze anzugehen und die Analyse der Menge an Proteinaggregaten ermöglicht, die in zwei verschiedenen Escherichia coli-Stämmen nach der Behandlung mit einer proteotoxischen antimikrobiellen Oberflächenbeschichtung gebildet werden. Die Beschichtung besteht aus Metall-Silber (Ag) und Ruthenium (Ru)-konditioniert mit Ascorbinsäure, und ihre antimikrobielle Aktivität wird durch die Erzeugung von reaktiven Sauerstoffspezies8,18erreicht. Hierin finden Sie eine detaillierte Beschreibung der Herstellung der Bakterienkultur nach Behandlung mit der antimikrobiellen Verbindung und einen Vergleich des Proteinaggregationsstatus bei Exposition von zwei E. coli-Stämmen mit unterschiedlichen Anfälligkeitsprofilen für eine zunehmende Konzentration des antimikrobiellen Wirkstoffs. Die beschriebene Methode ist kostengünstig, schnell und reproduzierbar und kann verwendet werden, um die Proteinaggregation in Gegenwart anderer proteotoxischer Verbindungen zu untersuchen. Darüber hinaus kann das Protokoll modifiziert werden, um die Auswirkungen spezifischer Gendreichungen auf die Proteinaggregation in einer Vielzahl verschiedener Bakterien zu analysieren.
Protokoll
1. Stressbehandlung der E. coli-Stämme MG1655 und CFT073
- Impfen Sie 5 ml Lysogenbrühe (LB) Medium mit einer einzigen Kolonie von kommensalen E. coli Stamm MG1655 bzw. uropathogenen E. coli (UPEC) Stamm CFT073 und inkubieren Sie für 14-16 h (über Nacht) bei 37 °C und 300 U / min.
HINWEIS: Escherichia coli CFT073 ist ein humaner Krankheitserreger. Die Handhabung von CFT073 muss mit geeigneten Biosicherheitsmaßnahmen in einem Biosafety Level-2 zertifizierten Labor durchgeführt werden. - Jeder Stamm wird in einen 500-ml-Kolben mit 70ml 3-(N-morpholino)propanesulfonsäure (MOPS)-Glucose (MOPS-g)(Tabelle 1)mittlerem bis zu einer optischen Dichte bei 600 nm (OD600)Wert von 0,1 verdünnt. Bei 37 °C und 300 U/min bis zur Mittleren Log-Phase (OD600 = 0,5-0,55) inkubieren.
- 20 ml jeder Kultur in drei vorgewärmte 125-ml-Kolben geben und bei 37 °C und 300 U/min für 2 min inkubieren.
HINWEIS: Da eine rechtzeitige Verarbeitung der Proben erforderlich ist, behandeln Sie nicht mehr als 6 Kulturen gleichzeitig. - Herstellung einer antimikrobiellen Verbindungslösung in MOPS-g-Medium in einer Konzentration von 2 mg/ml. Fügen Sie das antimikrobielle Mittel zu jeder Kultur hinzu, um die angegebenen Konzentrationen zu erreichen. Für die unbehandelte Steuerung fügen Sie das erforderliche Volumen des MOPS-g-Mediums hinzu.
HINWEIS: Wirbelt die antimikrobielle Lösung mit 2 mg / ml, um eine gleichmäßige Verteilung der Verbindungspartikel zu ermöglichen und Sedimentation zu vermeiden. - Inkubieren Sie die Kulturen für 45 min bei 37 °C und 300 U/min.
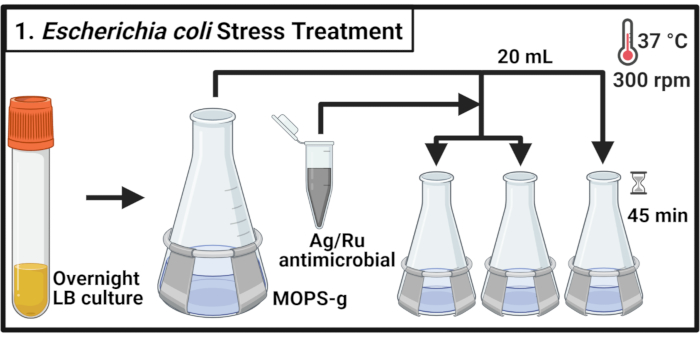
Abbildung 1: Escherichia coli Stressbehandlung. Bakterienkulturen werden in MOPS-g gezüchtet und mit den angegebenen Konzentrationen des silber-rutheniumhaltigen antimikrobiellen Mittels behandelt, wenn die Mid-Log-Phase erreicht ist. Abkürzungen: LB = Lysogeniebrühe; Ag-Ru = Silber-Ruthenium; MOPS-g = 3-( N-Morpholino)propanesulfonsäure (MOPS)-Glucose. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
2. Sammeln von Bakterienzellproben
- Nach 45 Minuten Stressbehandlung bestimmen Sie den OD600 jeder Kultur. Für jede Probe Erntezellen entsprechend 4 mL OD600 = 1 in 15 ml Zentrifugenröhrchen durch Zentrifugation für 15 min bei 3.000 × g und 4 °C.
- Entfernen Sie den Überstand vollständig und entfernen Sie die Zellpellets in 50 μL eiskaltem Lysepuffer (Tabelle 1). Inkubieren Sie die Proben für 30 Minuten auf Eis.
HINWEIS: Dieser Lyseschritt baut die Peptidoglykanschicht ab. Verwenden Sie immer frisch zubereiteten Lysepuffer. - Die Proben werden in 1,7 ml Mikrozentrifugenröhrchen überführen. Bei -80 °C bis zur weiteren Verwendung einfrieren.
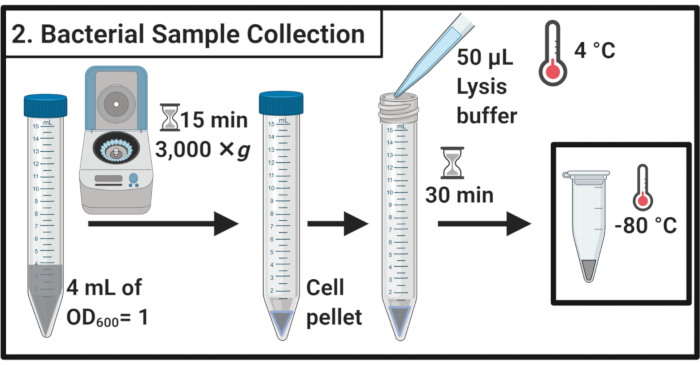
Abbildung 2: Bakterienprobenentnahme. Die Zellproben werden durch Zentrifugation entnommen und im Lysepuffer wieder aufgewärmt, gefolgt von einer Lagerung bei -80 °C. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
3. Extraktion der unlöslichen Proteinaggregate
- Proben auf Eis auftauen.
HINWEIS: Der Frost-Tau-Zyklus trägt zur Zelllyse bei. - 360 μL Eiskaltpuffer A (Tabelle 1) zugeben und durch Pipettieren vorsichtig mischen.
HINWEIS: Der osmotische Schock trägt auch zur Zelllyse bei. - Die Probe wird in ein 2 mL Mikrozentrifugenröhrchen mit ~200 μL 0,5 mm Glasperlen gegeben. 30 min bei 8 °C in einem Thermomixer mit Schütteln bei 1.400 U/min inkubieren.
HINWEIS: Dieser Schritt führt zur physischen Störung der Zelle. Ein Proteasehemmer kann verwendet werden, um den Proteinabbau zu minimieren. Die Störung kann bei 4 °C durchgeführt werden. Beachten Sie, dass die Verwendung von Glasperlen Berichten zufolge die Aggregation einer kleinen Untergruppe von Proteinen in Hefeinduziert 19. - 5 Minuten auf Eis inkubieren, ohne zu schütteln, um die Glasperlen abzulegen. 200 μL des Zelllysats in 1,7 ml Mikrozentrifugenröhrchen überführen.
HINWEIS: Vermeiden Sie die Übertragung der Glasperlen. - Zentrifuge bei 16.000 × g und 4 °C für 20 min. Sammeln Sie den Überstand, der lösliche Proteine enthält, und fahren Sie mit Abschnitt 4 fort.
- Das Pellet wird in 200 μL Eiskaltpuffer A (Tabelle 1) mit der Pipette resuspendiert. Zentrifuge bei 16.000 × g und 4 °C für 20 min. Entfernen Sie den Überstand vorsichtig vollständig.
- Fügen Sie 200 μL Eiskaltpuffer B hinzu (siehe Tabelle 1 und Materialtabelle)und entfernen Sie das Pellet vorsichtig durch Pipettieren.
HINWEIS: Das nichtionische Reinigungsmittel löst Membranprotein. - Wiederholen Sie die Zentrifugation bei 16.000 × g und 4 °C für 20 min. Entfernen Sie vorsichtig den Überstand.
- Das Pellet wird in 200 μL Kaltpuffer A (Tabelle 1) durch Pipettieren wieder aufgebraucht. Zentrifuge bei 16.000 × g und 4 °C für 20 min. Entfernen Sie den Überstand vollständig.
- Das Pellet in 100 μL 1x reduzierendem SDS-Probenpuffer (Tabelle 1) wieder aufbringen und 5 min bei 95 °C in einem Thermomixer kochen lassen.
- Lagern Sie die Probe bei -20 °C, um später fortzufahren oder sofort zur Trennung auf ein SDS-Polyacrylamidgel zu laden.
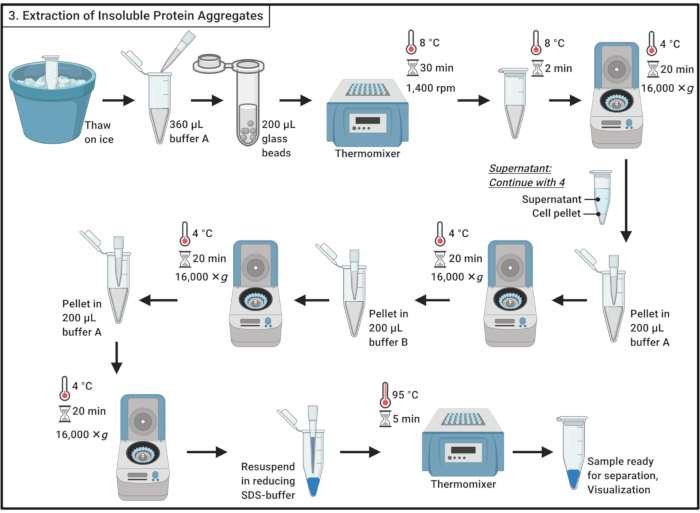
Abbildung 3: Extraktion unlöslicher Proteinaggregate. Die Extraktion von Proteinaggregaten umfasst eine Reihe von Schritten, darunter Zellaufschluss, die Trennung von Proteinaggregaten von löslichen Proteinen, die Solubilisierung von Membranproteinen und das Waschen. Abkürzung: SDS = Natriumdodecylsulfat. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
4. Probenvorbereitung für lösliche Proteine
- Mischen Sie 1 Volumen 100% Trichloressigsäure (TCA) mit 4 Volumina löslicher Proteinprobe aus Schritt 3.6.
HINWEIS: Die Handhabung von TCA erfordert einen Abzug und persönliche Schutzausrüstung sowie ein zugelassenes Abfallentsorgungsverfahren. - 10 min bei 4 °C inkubieren, um eine Proteinfällung zu ermöglichen.
HINWEIS: Weißer Niederschlag wird sehr bald erscheinen. - Zentrifuge, um bei 21.000 × g und 4 °C für 5 min auszuscheiden und den Überstand zu entfernen. Waschen Sie das Pellet mit 200 μL eiskaltem Aceton, um Zellablagerungen zu entfernen. Zentrifuge bei 21.000 × g und 4 °C für 5 min und entfernen Sie den Überstand. Wiederholen Sie diese Aktionen in Schritt 4.3 insgesamt dreimal.
- Legen Sie die Mikrozentrifugenröhrchen mit offenen Deckeln bei 37 °C in einen Thermomixer, um das restliche Aceton aus dem Pellet zu entfernen.
HINWEIS: Eine Inkubation von mehr als 5 min kann die Löslichkeit des Proteinpellets verringern. - Fügen Sie 100 μL 1x reduzierenden SDS-Puffer (Tabelle 1) hinzu und lösen Sie das Pellet vollständig auf. Kochen Sie die Probe 5 min bei 95 °C.
- Lagern Sie die Probe bei -20 °C, um später fortzufahren, oder laden Sie sie sofort zur Trennung auf ein SDS-Polyacrylamidgel.
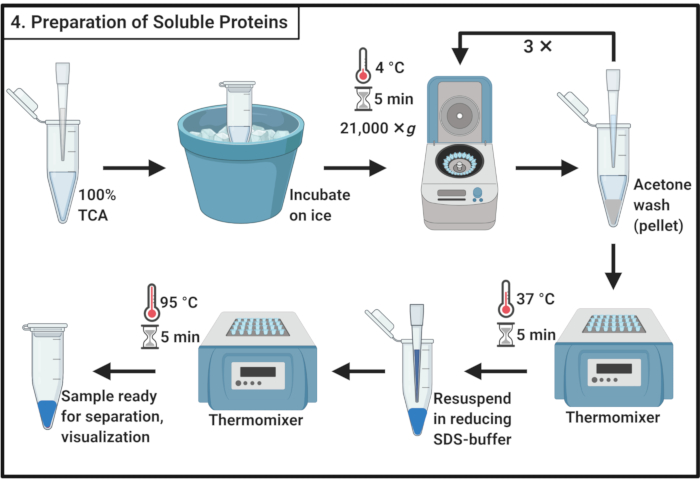
Abbildung 4: Herstellung löslicher Proteine. Die Herstellung von löslichem Protein beinhaltet einen Fällungsschritt mit Trichloressigsäure und wiederholtes Waschen mit eiskaltem Aceton. Abkürzungen: TCA = Trichloressigsäure; SDS = Natriumdodecylsulfat. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
5. Trennung und Visualisierung von extrahierten Proteinaggregaten mittels SDS-PAGE
- Bereiten Sie ein 12% SDS-Polyacrylamid-Gel vor.
- Für zwei Trenngele werden 5,1 mL doppelt destilliertes Wasser (ddH2O), 3,75 ml Tris-HCl (pH 8,8), 7,5 ml 20% (w/v) SDS, 6 mL 30% Acrylamid/Bisacrylamid (29:1)-Lösung, 75 ml 10% w/v Ammoniumpersulfat und 10 mL Tetramethylethylendiamin (TEMED) in ein 15 mL Zentrifugenröhrchen pipetten und vorsichtig ohne Luftblasen gemischt. Gießen Sie das Gel mit einer 1 ml Pipett in die Glasplatten und lassen Sie die oberen 2 cm frei von der Mischung. Fügen Sie 70% Ethanol auf die Oberseite des Trenngels hinzu und ermöglichen Sie eine gleichmäßige Grenzfläche zwischen den beiden Schichten.
- Nach der Polymerisation des Trenngels wird das Stapelgel durch Pipettieren von 1,535 mlddH2O,625 ml Tris-HCl (pH 6,8), 12,5 ml 20 % (w/v) SDS, 335 ml 30 %iger Acrylamid/Bisacrylamid (29:1)-Lösung, 12,5 ml 10 % w/v Ammoniumpersulfat und 2,5 ml TEMED hergestellt. Entfernen Sie das Ethanol aus den Trenngelen und fügen Sie die Stapelgellösung hinzu. Setzen Sie einen Kamm mit der gewünschten Anzahl von Taschen ein, ohne Luftblasen einzuführen. Polymerisation für 20-30 min zulassen.
- Laden Sie 4 μL jeder Probe und Proteinleiter in separate Vertiefungen und führen Sie das Gel (die Gele) in Tris-Glycin-Laufpuffer (Tabelle 1) bei 144 V für 45 min bei Raumtemperatur aus.
HINWEIS: Stoppen Sie das Gel, wenn das Bromphenolband im Begriff ist, aus dem Gel zu migrieren. - Das(n) Gel(e) in einer vorgewarnten Fairbanks-Lösung A (Tabelle 1) für 30 min auf einer Wippe einfärben.
- Entfärben Sie das Gel(e) in einer vorgewarmten Fairbanks-Lösung D (Tabelle 1) bis zum gewünschten Hintergrund (z. B. über Nacht) auf einer Wippe.
Abbildung 5: Proteintrennung und Visualisierung. Die Proben werden durch SDS-PAGE getrennt und durch Coomassie-Färbung visualisiert. Abkürzung: SDS-PAGE = Natriumdodecylsulfat-Polyacrylamid-Gelelektrophorese. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Ergebnisse
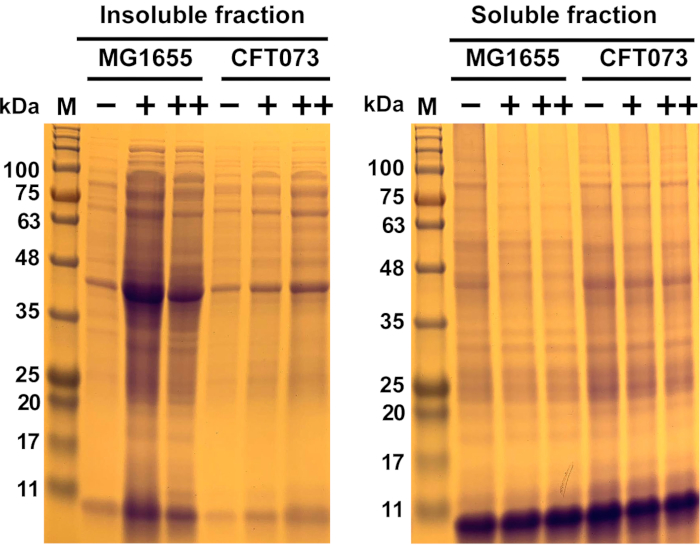
Abbildung 6: Repräsentative Ergebnisse der antimikrobiell induzierten Proteinaggregation im kommsalen Escherichia coli-Stamm MG1655 und UPEC-Stamm CFT073. Die E. coli-Stämme MG1655 und CFT073 wurden bei 37 °C und 300 U/min auf OD600= 0,5-0,55 in MOPS-g-Medien gezüchtet, bevor sie mit den angegebenen Konzentrationen (-, 0 mg/ml...
Diskussion
Dieses Protokoll beschreibt eine optimierte Methodik zur Analyse der Proteinaggregatbildung nach Behandlung verschiedener E. coli-Stämme mit einem proteotoxischen antimikrobiellen Mittel. Das Protokoll ermöglicht die gleichzeitige Extraktion von unlöslichen und löslichen Proteinfraktionen aus behandelten und unbehandelten E. coli-Zellen. Im Vergleich zu bestehenden Protokollen zur Proteinaggregatisolierung aus den Zellen14,15,
Offenlegungen
Die Autoren haben nichts preiszugeben.
Danksagungen
Diese Arbeit wurde durch Startfonds der Illinois State University School of Biological Sciences, den Illinois State University New Faculty Initiative Grant und den NIAID-Zuschuss R15AI164585 (an J.-U. D.). G.M.A. wurde vom Illinois State University Undergraduate Research Support Program (zu G.M.A.) unterstützt. K. P. H. wurde durch ein RISE-Stipendium des Deutschen Akademischen Austauschdienstes (DAAD) unterstützt. Die Autoren danken Dr. Uwe Landau und Dr. Carsten Meyer von der Largentech Vertriebs GmbH für die Bereitstellung des AGXX-Pulvers. Die Abbildungen 1, Abbildung 2, Abbildung 3, Abbildung 4und Abbildung 5 wurden mit Biorender generiert.
Materialien
| Name | Company | Catalog Number | Comments |
| Chemicals/Reagents | |||
| Acetone | Fisher Scientific | 67-64-1 | |
| 30% Acrylamide/Bisacrylamide solution 29:1 | Bio-Rad | 1610156 | |
| Ammonium persulfate | Millipore Sigma | A3678-100G | |
| Benzonase nuclease | Sigma | E1014-5KU | |
| Bluestain 2 Protein ladder, 5-245 kDa | GoldBio | P008-500 | |
| β-mercaptoethanol | Millipore Sigma | M6250-100ML | |
| Bromophenol blue | GoldBio | B-092-25 | |
| Coomassie Brilliant Blue R-250 | MP Biomedicals LLC | 821616 | |
| D-Glucose | Millipore Sigma | G8270-1KG | |
| D-Sucrose | Acros Organics | 57-50-1 | |
| Ethylenediamine tetra acetic acid (EDTA) | Sigma-Aldrich | SLBT9686 | |
| Glacial Acetic acid | Millipore Sigma | ARK2183-1L | |
| Glycerol, 99% | Sigma-Aldrich | G5516-1L | |
| Glycine | GoldBio | G-630-1 | |
| Hydrochloric acid, ACS reagent | Sigma-Aldrich | 320331-2.5L | |
| Isopropanol (2-Propanol) | Sigma | 402893-2.5L | |
| LB broth (Miller) | Millipore Sigma | L3522-1KG | |
| LB broth with agar (Miller) | Millipore Sigma | L2897-1KG | |
| Lysozyme | GoldBio | L-040-25 | |
| 10x MOPS Buffer | Teknova | M2101 | |
| Nonidet P-40 | Thomas Scientific | 9036-19-5 | |
| Potassium phosphate, dibasic | Sigma-Aldrich | P3786-1KG | |
| Potassium phosphate, monobasic | Acros Organics | 7778-77-0 | |
| Sodium dodecyl sulfate (SDS) | Sigma-Aldrich | L3771-500G | |
| Tetramethylethylenediamine (TEMED) | Millipore Sigma | T9281-50ML | |
| Thiamine | Sigma-Aldrich | T4625-100G | |
| 100% Trichloroacetic acid | Millipore Sigma | T6399-100G | |
| Tris base | GoldBio | T-400-1 | |
| Material/Equipment | |||
| Centrifuge tubes (15 mL) | Alkali Scientific | JABG-1019 | |
| Erlenmeyer flask (125 mL) | Carolina | 726686 | |
| Erlenmeyer flask (500 mL) | Carolina | 726694 | |
| Freezer: -80 °C | Fisher Scientific | ||
| Glass beads (0.5 mm) | BioSpec Products | 1107-9105 | |
| Microcentrifuge | Hermle | Z216MK | |
| Microcentriguge tubes (1.7 mL) | VWR International | 87003-294 | |
| Microcentriguge tubes (2.0 mL) | Axygen Maxiclear Microtubes | MCT-200-C | |
| Plastic cuvettes | Fischer Scientific | 14-377-012 | |
| Power supply | ThermoFisher Scientific | EC105 | |
| Rocker | Alkali Scientific | RS7235 | |
| Shaking incubator (37 °C) | Benchmark Scientific | ||
| Small glass plate | Bio-Rad | 1653311 | |
| Spacer plates (1 mm) | Bio-Rad | 1653308 | |
| Spectrophotometer | Thermoscientific | 3339053 | |
| Tabletop centrifuge for 15 mL centrifuge tubes | Beckman-Coulter | ||
| Vertical gel electrophoresis chamber | Bio-Rad | 1658004 | |
| Vortexer | Fisher Vortex Genie 2 | 12-812 | |
| Thermomixer | Benchmark Scientific | H5000-HC | |
| 10 well comb | Bio-Rad | 1653359 |
Referenzen
- Dahl, J. -. U., et al. HdeB functions as an acid-protective chaperone in bacteria. Journal of Biological Chemistry. 290 (1), 65-75 (2015).
- Foit, L., George, J. S., Zhang, B. W., Brooks, C. L., Bardwell, J. C. A. Chaperone activation by unfolding. Proceedings of the National Academy of Sciences of the United States of America. 110 (14), 1254-1262 (2013).
- Sultana, S., Foti, A., Dahl, J. -. U. Bacterial defense systems against the neutrophilic oxidant hypochlorous acid. Infection and Immunity. 88 (7), 00964 (2020).
- Dahl, J. -. U., Gray, M. J., Jakob, U. Protein quality control under oxidative stress conditions. Journal of Molecular Biology. 427 (7), 1549-1563 (2015).
- Groitl, B., Dahl, J. -. U., Schroeder, J. W., Jakob, U. Pseudomonas aeruginosa defense systems against microbicidal oxidants. Molecular Microbiology. 106 (3), 335-350 (2017).
- Casadevall, A. Thermal restriction as an antimicrobial function of fever. PLoS Pathogens. 12 (5), 1005577 (2016).
- Richter, K., Haslbeck, M., Buchner, J. The heat shock response: life on the verge of death. Molecular Cell. 40 (2), 253-266 (2010).
- Van Loi, V., Busche, T., Preuß, T., Kalinowski, J., Bernhardt, J. The AGXX ® antimicrobial coating causes a thiol-specific oxidative stress response and protein S-bacillithiolation in Staphylococcus aureus. Frontiers in Microbiology. 9, 3037 (2018).
- Anfinsen, C. B., Scheraga, H. A. Experimental and theoretical aspects of protein folding. Advances in Protein Chemistry. 29, 205-300 (1975).
- Schramm, F. D., Schroeder, K., Jonas, K. Protein aggregation in bacteria. FEMS Microbiology Reviews. 44 (1), 54-72 (2020).
- Tomoyasu, T., Mogk, A., Langen, H., Goloubinoff, P., Bukau, B. Genetic dissection of the roles of chaperones and proteases in protein folding and degradation in the Escherichia coli cytosol. Molecular Microbiology. 40 (2), 397-413 (2001).
- Gray, M. J., et al. Polyphosphate is a primordial chaperone. Molecular Cell. 53 (5), 689-699 (2014).
- Weids, A. J., Ibstedt, S., Tamás, M. J., Grant, C. M. Distinct stress conditions result in aggregation of proteins with similar properties. Scientific Reports. 6, 24554 (2016).
- Mogk, A., et al. Identification of thermolabile Escherichia coli proteins: prevention and reversion of aggregation by DnaK and ClpB. EMBO Journal. 18 (24), 6934-6949 (1999).
- Fay, A., Glickman, M. S. An essential nonredundant role for mycobacterial DnaK in native protein folding. PLoS Genetics. 10 (7), 1004516 (2014).
- Schramm, F. D., Heinrich, K., Thüring, M., Bernhardt, J., Jonas, K. An essential regulatory function of the DnaK chaperone dictates the decision between proliferation and maintenance in Caulobacter crescentus. PLoS Genetics. 13 (12), 1007148 (2017).
- Maisonneuve, E., Fraysse, L., Moinier, D., Dukan, S. Existence of abnormal protein aggregates in healthy Escherichia coli cells. Journal of Bacteriology. 190 (3), 887-893 (2008).
- Heiss, A., Freisinger, B., Held-Föhn, E. Enhanced antibacterial activity of silver-ruthenium coated hollow microparticles. Biointerphases. 12 (5), (2017).
- Papnayotou, I., Sun, B., Roth, A. F., Davis, N. G. Protein aggregation induced during glass bead lysis of yeast. Yeast. 27 (10), 801-816 (2010).
- Chuang, S. E., Blattner, F. R. Characterization of twenty-six new heat shock genes of Escherichia coli. Journal of Bacteriology. 175 (16), 5242-5252 (1993).
- Imlay, J. A. The molecular mechanisms and physiological consequences of oxidative stress: Lessons from a model bacterium. Nature Reviews Microbiology. 11 (7), 443-454 (2013).
- Mühlhofer, M., et al. The heat shock response in yeast maintains protein homeostasis by chaperoning and replenishing proteins. Cell Reports. 29 (13), 4593-4607 (2019).
- Chandrangsu, P., Rensing, C., Helmann, J. D. Metal homeostasis and resistance in bacteria. Nature Reviews Microbiology. 15, 338-350 (2017).
- Stevens, M., et al. HSP60/10 chaperonin systems are inhibited by a variety of approved drugs, natural products, and known bioactive molecules. Bioorganic and Medicinal Chemistry Letters. 29 (9), 1106-1112 (2019).
- Schramm, F. D., Schroeder, K., Alvelid, J., Testa, I., Jonas, K. Growth-driven displacement of protein aggregates along the cell length ensures partitioning to both daughter cells in Caulobacter crescentus. Molecular Microbiology. 111 (6), 1430-1448 (2019).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten