Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Identificación automática de ramas dendríticas y su orientación
En este artículo
Resumen
Presented es una herramienta computacional que permite la medición automática simple y directa de las orientaciones de las ramas dendríticas neuronales a partir de imágenes de fluorescencia 2D.
Resumen
La estructura de los árboles dendríticos neuronales juega un papel clave en la integración de las entradas sinápticas en las neuronas. Por lo tanto, la caracterización de la morfología de las dendritas es esencial para una mejor comprensión de la función neuronal. Sin embargo, la complejidad de los árboles dendríticos, tanto cuando están aislados como especialmente cuando se encuentran dentro de las redes neuronales, no se ha entendido completamente. Desarrollamos una nueva herramienta computacional, SOA (Segmentation and Orientation Analysis), que permite la medición automática de la orientación de ramas dendríticas a partir de imágenes de fluorescencia de cultivos neuronales 2D. SOA, escrito en Python, utiliza la segmentación para distinguir las ramas dendríticas del fondo de la imagen y acumula una base de datos sobre la dirección espacial de cada rama. La base de datos se utiliza para calcular parámetros morfológicos como la distribución direccional de las ramas dendríticas en una red y la prevalencia del crecimiento paralelo de ramas dendríticas. Los datos obtenidos pueden utilizarse para detectar cambios estructurales en las dendritas en respuesta a la actividad neuronal y a estímulos biológicos y farmacológicos.
Introducción
La morfogénesis dendrítica es un tema central en neurociencia, ya que la estructura del árbol dendrítico afecta a las propiedades computacionales de la integración sináptica en las neuronas1,2,3. Además, las anomalías morfológicas y las modificaciones en las ramas dendríticas están implicadas en trastornos degenerativos y del desarrollo neurológico4,5,6. En cultivos neuronales donde la ramificación dendrítica se puede visualizar más fácilmente, las interacciones entre ramas dendríticas no hermanas regulan los sitios y la extensión de la agrupación sináptica a lo largo de las ramas, un comportamiento que puede afectar la coactividad sináptica y la plasticidad7,8,9. Por lo tanto, la caracterización de los parámetros morfológicos del árbol dendrítico utilizando cultivos neuronales bidimensionales (2D) es ventajosa para comprender la morfogénesis dendrítica y la funcionalidad de neuronas individuales y redes de neuronas. Sin embargo, esta es una tarea desafiante porque las ramas dendríticas forman una malla compleja incluso en cultivos neuronales 2D "simplificados".
Se han desarrollado varias herramientas para rastrear y analizar automáticamente las estructuras dendríticas10,11,12,13. Sin embargo, la mayoría de estas herramientas están diseñadas para redes neuronales 3D y son naturalmente demasiado complejas para usarlas con redes 2D. Por el contrario, las herramientas de análisis morfológico menos avanzadas suelen implicar un componente significativo del trabajo manual asistido por ordenador, que consume mucho tiempo y es susceptible al sesgo del operador14. Las herramientas semiautomáticas existentes, como 'ImageJ'15 (un paquete de procesamiento de imágenes de código abierto de los NIH con una vasta colección de herramientas de análisis de imágenes biológicas desarrolladas por la comunidad), reducen en gran medida el trabajo manual del usuario. Sin embargo, todavía se necesitan algunas intervenciones manuales durante el procesamiento de imágenes, y la calidad de la segmentación puede ser menos que deseable.
En este trabajo se presenta el SOA, una sencilla herramienta automatizada que permite la segmentación directa y el análisis de orientación de ramas dendríticas dentro de redes neuronales 2D. El SOA puede detectar varios objetos en forma de línea en imágenes 2D y caracterizar sus propiedades morfológicas. Aquí, utilizamos el SOA para segmentar ramas dendríticas en imágenes de fluorescencia 2D de redes dendríticas en cultivo. El software identifica las ramas dendríticas y realiza con éxito mediciones de parámetros morfológicos como el paralelismo y la distribución espacial. El SOA se puede adaptar fácilmente para el análisis de procesos celulares de otros tipos de células y para el estudio de redes no biológicas.
Protocolo
NOTA: El Ministerio de Salud de Israel aprobó el uso de ratones bajo el protocolo IL-218-01-21 para el uso ético de animales de experimentación. SOA solo es compatible con Windows 10 y Python 3.9. Está disponible como código abierto: https://github.com/inbar2748/DendriteProject. En este enlace, también hay un README. DM que tiene instrucciones para descargar el software, un enlace al sitio web del software y un archivo de requisitos que contiene información sobre las versiones requeridas de todos los paquetes. También se han proporcionado ejemplos adicionales de análisis realizados con el software.
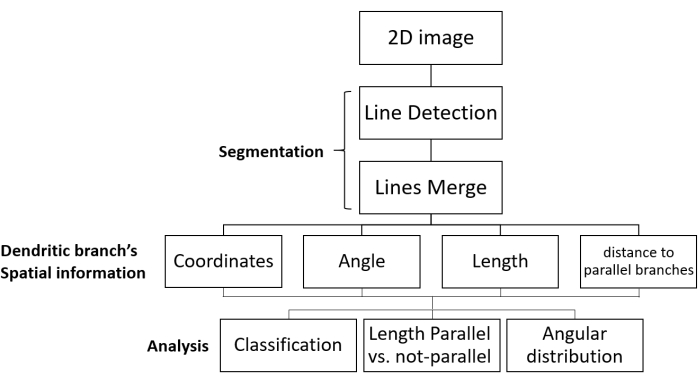
Figura 1: Flujo de trabajo SOA para análisis de segmentación y dirección de crecimiento. Se muestran los pasos de procesamiento de imágenes fluorescentes de redes dendríticas y el análisis de datos. La imagen 2D se carga, se segmenta (en dos pasos: las ramas dendríticas se detectan como líneas, y luego se fusionan las líneas relevantes), y se obtiene la información espacial de cada rama dendrítica. Los datos se recopilan para todas las ramas dendríticas de la imagen. Finalmente, los datos se analizan para dar los parámetros morfológicos deseados. Abreviatura: SOA = análisis de segmentación y orientación. Haga clic aquí para ver una versión más grande de esta figura.
1. Abra la aplicación SOA.
- Abra la dirección URL: https://mega.nz/folder/bKZhmY4I#4WAaec4biiGt4_1lJlL4WA, busque la carpeta COMPRIMIDA SOA.zip y descargue el archivo ZIP haciendo doble clic.
- Descomprima la carpeta haciendo clic derecho en SOA.zip y elija Extraer archivos. Observe la ventana Ruta de extracción y Opciones que se abre y el cuadro de texto Dirección de destino que muestra la ruta de acceso de los archivos extraídos. Para extraer a una ubicación diferente, haga clic en una de las carpetas en el panel derecho de la ventana para convertirla en la carpeta de destino. Haga clic en Aceptar para extraer los archivos a esa carpeta.
- Abra el archivo SOA extraído y haga doble clic en SOA.exe. Espere a que se abra una ventana negra, después de lo cual aparecerá la aplicación.

Figura 2: Ejemplo de un flujo de trabajo utilizando la GUI de SOA. Columna izquierda: secciones de gui del flujo de trabajo. Columna central: imagen de una red dendrítica, procesada durante el flujo de trabajo (barra de escala: 20 μm). Columna derecha: ampliación del área marcada por un rectángulo rojo en las imágenes de la columna central (Barra de escala: 4 μm). Paso 1: Selección y carga de una imagen. Paso 2: La primera etapa de segmentación es la detección de líneas que representan las ramas dendríticas identificadas. Paso 3: La segunda etapa de la segmentación es la fusión basada en la proximidad del revestimiento del segmento en ramas dendríticas individuales. La configuración de todos los pasos se puede modificar. Abreviaturas: SOA = análisis de segmentación y orientación; GUI = interfaz gráfica de usuario. Haga clic aquí para ver una versión más grande de esta figura.
2. Abra una imagen para analizar.
- En la barra de menús Carga de SOA Viewer | seleccione Elegir archivo | Elija una imagen de los archivos del equipo | haga clic en él (solo .png .jpg .tif .bmp archivos) | Abrir | Observe la ruta de acceso del archivo | Próximo.
3. Optimización de la segmentación
NOTA: En la barra de menús Propiedades del visor SOA , cambie los valores de los parámetros seleccionados para ajustar la configuración del proceso de segmentación. Una descripción detallada de los parámetros, como el Umbral, se proporciona en el Material Suplementario.
- En Aristas, ajuste el umbral de la visualización seleccionando el Umbral e introduciendo un número.
NOTA: Cuanto menor sea el número del umbral, más líneas se detectarán. El umbral es un número que oscila entre 0 y 255. El valor predeterminado se ha establecido en 0. - En líneas de combinación:
- Ajuste la distancia mínima a combinar para la pantalla seleccionando la distancia mínima para combinar e introduciendo un número.
NOTA: La distancia mínima para combinar varía de 0 a 30 píxeles. El valor predeterminado se establece en 20. - Ajuste el ángulo mínimo a combinar para la pantalla seleccionando el ángulo mínimo para combinar e introduciendo un número.
NOTA: El ángulo mínimo a fusionar varía de 0 a 30°. El valor predeterminado se establece en 10.
- Ajuste la distancia mínima a combinar para la pantalla seleccionando la distancia mínima para combinar e introduciendo un número.
- Haga clic en Crear imagen de segmentación de vista previa.
NOTA: Se mostrará una imagen de vista previa de los resultados de segmentación de acuerdo con los valores actualizados. Además, se mostrará el número de líneas antes de la fusión y el número de líneas después de la fusión. - Cambiar los parámetros para lograr la máxima identificación de segmentos. Si es necesario cambiar las Propiedades, haga clic en el botón Cerrar ventana y siga los pasos 3.1-3.4.
4. Cree los archivos de salida.
- Pulse OK para visualizar las imágenes de segmentación y los gráficos de análisis. Observe la ventana que aparece para seleccionar una ubicación donde se guardará el archivo .xlsx.
- Inserte un nombre de archivo | Elija Guardar | espere a que se cree y guarde el archivo .xlsx con datos.
NOTA: Además del archivo .xlsx, se mostrarán automáticamente los siguientes archivos: un archivo que presenta la imagen original, la imagen de reconocimiento de línea, la imagen final de la segmentación y tres gráficos de análisis.
5. Barra de herramientas de navegación
NOTA: Se incluye una barra de herramientas de navegación en todas las ventanas de figuras y se puede utilizar para navegar por el conjunto de datos. Cada uno de los botones en la parte inferior de la barra de herramientas se describe a continuación.
- Para navegar hacia adelante y hacia atrás entre las vistas definidas anteriormente, utilice los botones Adelante y Atrás .
NOTA: Los botones Inicio, Adelante y Atrás son similares a los controles Inicio, Adelante y Atrás de un navegador web. Inicio vuelve a la pantalla predeterminada, la imagen original. - Utilice el botón Zoom para desplazarse y hacer zoom. Para activar el movimiento panorámico y el zoom, pulse el botón Zoom y, a continuación, mueva el ratón a la ubicación deseada de la imagen.
- Para desplazar la figura, mantenga pulsado el botón izquierdo del ratón mientras la arrastra a una nueva posición. Suelte el botón del ratón y el punto seleccionado en la imagen aparecerá en la nueva posición. Durante el movimiento panorámico, mantenga presionadas las teclas x o y para restringir el movimiento a los ejes x o y , respectivamente.
- Para hacer zoom, mantenga presionado el botón derecho del mouse y arrástrelo a una nueva ubicación. Desplácese hacia la derecha para acercar el eje x y desplácese hacia la izquierda para alejar el eje x. Haga lo mismo con los movimientos del eje Y y hacia arriba / abajo. Al hacer zoom, tenga en cuenta que el punto debajo del mouse permanece estacionario, lo que permite acercar o alejar alrededor de ese punto. Utilice las teclas modificadoras x, y o CONTROL para limitar el zoom a la conservación x, y o relación de aspecto, respectivamente.
- Para activar el modo Zoom a rectángulo, haga clic en el botón Zoom a rectángulo . Coloque el cursor sobre la imagen y pulse el botón izquierdo del ratón. Arrastre el ratón a una nueva ubicación mientras mantiene pulsado el botón para definir una región rectangular.
NOTA: Los límites de vista de ejes se ampliarán a la región definida cuando se presione el botón izquierdo del mouse. Los límites de la vista de ejes se alejarán cuando se presione el botón derecho del mouse, colocando los ejes originales en la región definida. - Utilice la herramienta de configuración de subtramas para configurar la apariencia de la subtrama.
NOTA: Los lados izquierdo, derecho, superior e inferior de la subtrama, así como el espacio entre filas y columnas, se pueden estirar o comprimir. - Para abrir un cuadro de diálogo de guardar archivo, haga clic en el botón Guardar y guarde el archivo en los siguientes formatos: .png, .ps, .eps, .svg o .pdf.
Resultados
Se realizó un análisis representativo sobre imágenes de redes dendríticas en cultivo. Las células se extrajeron según lo descrito por Baranes et al. 16,17. Brevemente, las células del hipocampo se extrajeron de los cerebros de ratas postnatales y se cultivaron en fundas de vidrio 2D durante 1-2 semanas. Los cultivos se fijaron y se tiñeron a través de inmunofluorescencia indirecta utilizando un anticuerpo contra el marcador de proteína dendrít...
Discusión
Se requieren urgentemente estrategias efectivas para extraer información morfológica de imágenes 2D para mantenerse al día con los datos de imágenes biológicas. Aunque los datos de imágenes se pueden generar en horas, el análisis en profundidad de las imágenes lleva mucho tiempo. Como resultado, el procesamiento de imágenes se ha convertido claramente en un obstáculo importante en muchos campos. Esto se debe en parte a la alta complejidad de los datos, especialmente cuando se trata de muestras biológicas. Ade...
Divulgaciones
Los autores declaran que no tienen intereses financieros contrapuestos.
Agradecimientos
Los autores desean agradecer al Dr. Orly Weiss por la preparación de las imágenes culturales.
Materiales
| Name | Company | Catalog Number | Comments |
| Matplotlib | 2002 - 2012 John Hunter, Darren Dale, Eric Firing, Michael Droettboom and the Matplotlib development team; 2012 - 2021 The Matplotlib development team. | 3.4.2 | a Python 2D plotting library |
| matplotlib-scalebar | Philippe Pinard | 0.7.2 | artist for matplotlib to display a scale bar |
| NumPy | The NumPy community. | 1.20.3 | fundamental package for scientific computing library |
| OpenCV | OpenCV team | 4.5.2.54 | Open Source Computer Vision Library |
| PyCharm | JetBrains | 2020.3.1 (Community Edition) version | Build #PC-203.6682.86, built on December 18, 2020. Runtime version: 11.0.9.1+11-b1145.37 amd64. VM: OpenJDK 64-Bit Server VM by JetBrains s.r.o. Windows 10 10.0. Memory: 978M, Cores: 4 |
| PyQt5 | Riverbank Computing | 5.15.4 | manage the GUI |
| python | Python Software Foundation License | 3.9 version | |
| Qt Designer | The QT Company Ltd. | 5.11.1 version | |
| scipy | Community library project | 1.6.3 | Python-based ecosystem of open-source software for mathematics, science, and engineering |
| Seaborn | Michael Waskom. | 0.11.1 | Python's Statistical Data Visualization Library. |
| Windows 10 | Microsoft | ||
| Xlsxwriter | John McNamara | 1.4.3 | Python module for creating Excel XLSX files |
Referencias
- Ferrante, M., Migliore, M., Ascoli, G. Functional impact of dendritic branch-point morphology. Journal of Neuroscience. 33 (5), 2156-2165 (2013).
- Spruston, N. Pyramidal neurons: dendritic structure and synaptic integration. Nature Reviews Neuroscience. 9 (3), 206-221 (2008).
- Chklovskii, D. Synaptic Connectivity and Neuronal Morphology: Two Sides of the Same Coin. Neuron. 43 (5), 609-617 (2004).
- Chapleau, C., Larimore, J., Theibert, A., Pozzo-Miller, L. Modulation of dendritic spine development and plasticity by BDNF and vesicular trafficking: fundamental roles in neurodevelopmental disorders associated with mental retardation and autism. Journal of Neurodevelopmental Disorders. 1 (3), 185-196 (2009).
- Irwin, S. Dendritic Spine Structural Anomalies in Fragile-X Mental Retardation Syndrome. Cerebral Cortex. 10 (10), 1038-1044 (2000).
- Kaufmann, W. Dendritic anomalies in disorders associated with mental retardation. Cerebral Cortex. 10 (10), 981-991 (2000).
- Pinchas, M., Baranes, D. Dendritic branch intersections are structurally regulated targets for efficient axonal wiring and synaptic clustering. PLoS ONE. 8 (12), 82083 (2013).
- Cove, J., Blinder, P., Baranes, D. Contacts among non-sister dendritic branches at bifurcations shape neighboring dendritic branches and pattern their synaptic inputs. Brain Research. 1251, 30-41 (2009).
- Blinder, P., Cove, J., Foox, M., Baranes, D. Convergence among non-sister dendritic branches: An activity-controlled mean to strengthen network connectivity. PLoS ONE. 3 (11), 3782 (2008).
- Glaser, J., Glaser, E. Neuron imaging with neurolucida - PC-based system for image combining microscopy. Computerized Medical Imaging and Graphics. 14 (5), 307-317 (1990).
- Scorcioni, R., Polavaram, S., Ascoli, G. L-Measure: a web-accessible tool for the analysis, comparison and search of digital reconstructions of neuronal morphologies. Nature Protocols. 3 (5), 866-876 (2008).
- Torben-Nielsen, B. An efficient and extendable python library to analyze neuronal morphologies. Neuroinformatics. 12 (4), 619-622 (2014).
- Parekh, R., Ascoli, G. Neuronal morphology goes digital: A research hub for cellular and system neuroscience. Neuron. 78 (1), 206 (2013).
- heng, J., Zhou, X., Sabatini, B. L., Wong, S. T. C. NeuronIQ: A novel computational approach for automatic dendrite SPINES detection and analysis. 2007 IEEE/NIH Life Science Systems and Applications Workshop. , 168-171 (2007).
- Image processing and analysis in Java. NIH. ImageJ Available from: https://imagej.nih.gov/ij (2021)
- Peretz, H., Talpalar, A. E., Vago, R., Baranes, D. Superior survival and durability of neurons and astrocytes on 3-dimensional aragonite biomatrices. Tissue Engineering. 13, 461-472 (2007).
- Morad, T. I., Hendler, R. M., Weiss, O. E., Canji, E. A., Merfeld, I., Dubinsky, Z., Minnes, R., Francis, Y. I., Baranes, D. Gliosis of astrocytes cultivated on coral skeleton is regulated by the matrix surface topography. Biomedical Materials. 14 (4), 045005 (2019).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoExplorar más artículos
This article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados