このコンテンツを視聴するには、JoVE 購読が必要です。 サインイン又は無料トライアルを申し込む。
Method Article
樹状枝の自動同定とその向き
要約
提示は2D蛍光画像からの神経樹状枝の向きを簡単かつ直接自動で測定できる計算ツールです。
要約
神経樹状樹木の構造は、ニューロンのシナプス入力の統合において重要な役割を果たしています。したがって、樹状突起の形態の特徴付けは、神経機能のより良い理解のために不可欠である。しかし、樹状樹木の複雑さは、孤立した場合、特に神経ネットワーク内に位置する場合の両方で、完全には理解されていません。2D神経細胞培養物の蛍光画像から樹状枝の向きを自動測定できる新しい計算ツール 「SOA (セグメンテーション・オリエンテーション分析)」を開発しました。Python で書かれた SOA は、樹状の枝と画像の背景を区別するためにセグメンテーションを使用し、各ブランチの空間方向にデータベースを蓄積します。次に、ネットワーク内の樹状枝の方向分布や、並列樹状枝成長の有病率などの形態学的パラメータを計算するために使用されます。得られたデータは、神経活性に応答して樹状突起の構造変化を検出し、生物学的および薬理学的刺激に使用することができる。
概要
樹状形態形成は神経科学の中心的な主題であり、樹状樹木の構造はニューロンのシナプス統合の計算特性に影響を及ぼす1,2,3。また、樹状枝の形態異常や変形性は、変性・神経発達障害の4,5,6に関係しています。樹状の影響がより容易に可視化できる神経培養において、非姉妹樹状枝間の相互作用は、分岐に沿ったシナプスクラスタリングの部位および程度を調節し、シナプス共活動および可塑性に影響を及ぼす可能性のある行動7,8,9である。したがって、二次元(2D)ニューロン培養を用いた樹状樹木の形態学的パラメータの特徴づけは、樹状形態形成およびニューロンの単一およびネットワークの機能性を理解するために有利である。しかし、樹状枝は「単純化された」2Dニューロン培養においても複雑なメッシュを形成するため、これは困難な作業です。
樹状構造10,11,12,13を自動的にトレースし解析するツールがいくつか開発されています。しかし、これらのツールのほとんどは3Dニューロンネットワーク用に設計されており、2Dネットワークでは使用するには複雑すぎます。対照的に、あまり高度でない形態学的解析ツールは、通常、非常に時間がかかり、オペレータbias14の影響を受けやすいコンピュータ支援手作業の重要なコンポーネントを含みます。「ImageJ'15(コミュニティ開発の生物学的画像解析ツールの膨大なコレクションを持つNIHオープンソース画像処理パッケージ)」などの既存の半自動ツールは、ユーザーの手作業を大幅に削減します。ただし、画像処理中に手動で介入する必要がある場合があり、セグメンテーションの品質が望ましいものよりも低くなる可能性があります。
本論文では、2Dニューロンネットワーク内の樹状枝を直接セグメンテーション・オリエンテーション解析できるシンプルな自動化ツール「SOA」を紹介する。SOA は、2D 画像内のさまざまな線状オブジェクトを検出し、その形態学的特性を特徴付けることができます。ここでは、培養中の樹状ネットワークの2D蛍光画像で樹状枝をセグメント化するためのSOAを用いた。ソフトウェアは樹状枝を識別し、並列処理や空間分布などの形態学的パラメータの測定を正常に行います。SOAは、他の細胞タイプの細胞プロセスの解析や非生物学的ネットワークの研究に容易に適応することができます。
Access restricted. Please log in or start a trial to view this content.
プロトコル
注:イスラエル保健省は、実験動物の倫理的使用のためのプロトコルIL-218-01-21の下でマウスの使用を承認しました。SOA は、Windows 10 および Python 3.9 とのみ互換性があります。これは、オープンソースコードとして利用可能です: https://github.com/inbar2748/DendriteProject。このリンクには README もあります。ソフトウェアをダウンロードするための指示、ソフトウェアのウェブサイトへのリンク、およびすべてのパッケージの必要なバージョンに関する情報を含む要件ファイルを持つDMファイル。ソフトウェアを使用して実行される分析の追加の例も提供されています。
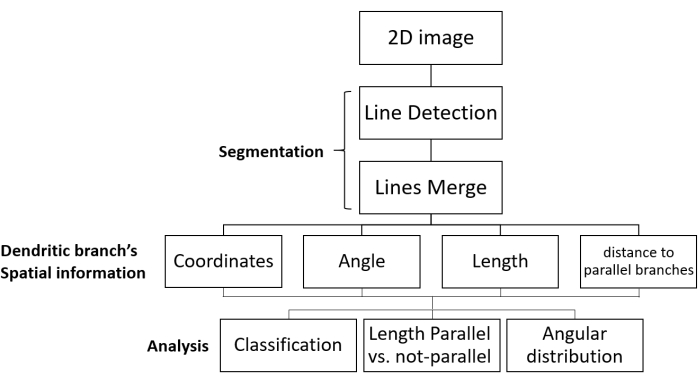
図1:セグメンテーションおよび成長方向解析のSOAワークフロー 樹状ネットワークの蛍光画像の処理ステップとデータ解析を示す。2D画像はアップロードされ、セグメント化され(樹状枝は線分として検出され、関連する線がマージされる)、各樹状枝の空間情報が得られます。データは、画像内のすべての樹状枝に対して収集されます。最後に、データを分析して、所望の形態学的パラメータを与える。略語: SOA = セグメンテーションと方向解析 この図の大きなバージョンを表示するには、ここをクリックしてください。
1. SOA アプリケーションを開きます。
- URL アドレスを開きます: https://mega.nz/folder/bKZhmY4I#4WAaec4biiGt4_1lJlL4WA、 SOA .zip zip 形式のフォルダーを見つけて、ZIP ファイルをダブルクリックしてダウンロードします。
- SOA を右クリックしてフォルダを解凍し.zipファイルの展開を選択します。開いた [抽出パスとオプション] ウィンドウと、抽出されたファイルのパスを表示する [宛先アドレス] テキスト ボックスを確認します。別の場所に展開するには、ウィンドウの右側のパネルでいずれかのフォルダをクリックして、コピー先のフォルダにします。[OK] をクリックして、そのフォルダにファイルを展開します。
- 抽出した SOA ファイルを開き、 SOA .exe をダブルクリックします。黒いウィンドウが開くのを待ち、その後にアプリケーションが表示されます。

図2:SOAのGUIを使用したワークフローの例 左列: ワークフローの GUI セクション。中央の列: 樹状ネットワークの画像, ワークフロー中に処理(スケールバー: 20 μm)。右の列: 中央の列の画像に赤い長方形でマークされた領域の拡大率(スケールバー:4 μm)。ステップ 1: 画像の選択とアップロード。ステップ2:セグメンテーションの第1段階は、識別された樹状枝を表す線の検出です。ステップ3:セグメンテーションの第2段階は、個々の樹状枝のセグメント裏地の近接ベースの合併です。すべてのステップの設定は変更できます。略語: SOA = セグメンテーションと方向分析;GUI = グラフィカル・ユーザー・インターフェース。 この図の大きなバージョンを表示するには、ここをクリックしてください。
2. 画像を開いて分析します。
- SOA ビューアアップロードメニューバー|[ファイルの選択] |コンピュータファイルから画像を選択|クリック (ファイルのみ.png .jpg .tif .bmp) ||を開く ファイルのパスを|次に。
3. セグメンテーションの最適化
注: [SOA ビューアのプロパティ] メニュー バーで、選択したパラメータの値を変更してセグメンテーションプロセスの設定を調整します。 しきい値などのパラメータの詳細な説明は、 補足材料に記載されています。
- [エッジ] で、[しきい値] を選択して数値を入力して、表示のしきい値を調整します。
注: しきい値の数が小さいほど、検出される行数が多くなります。しきい値は、0 から 255 の範囲の数値です。既定値は 0 に設定されています。 - マージ行で:
- 合成する最小距離を選択して数値を入力し、ディスプレイの 最小距離 を調整します。
注: マージする最小距離 は、0 ~ 30 ピクセルの範囲です。デフォルト値は 20 に設定されています。 - 合成する最小角度を選択して数値を入力して、ディスプレイにマージ する最小角度 を調整します。
注: マージする最小角度 の範囲は 0 ~ 30° です。デフォルト値は 10 に設定されています。
- 合成する最小距離を選択して数値を入力し、ディスプレイの 最小距離 を調整します。
- [ プレビュー セグメンテーション イメージの作成] をクリックします。
注: セグメンテーション結果のプレビュー イメージは、更新された値に従って表示されます。また、マージ前の行数とマージ後の行数も表示されます。 - パラメータを変更して、セグメントの識別を最大限に引き出します。プロパティを変更する必要がある場合は、 ウィンドウを閉じる ボタンをクリックして、手順3.1-3.4に従います。
4. 出力ファイルを作成します。
- [OK] を押して、セグメンテーション 画像と分析グラフを視覚化します。.xlsx ファイルを保存する場所を選択するためのウィンドウが表示されることを確認します。
- ファイル名を挿入|[ 保存] |の 選択データを含む.xlsxファイルが作成され、保存されるまで待ちます。
注: .xlsx ファイルに加えて、元の画像を表示するファイル、線の認識イメージ、セグメンテーションの最終イメージ、3 つの解析グラフが自動的に表示されます。
5. ナビゲーションツールバー
注: ナビゲーション ツールバーはすべての図ウィンドウに含まれており、データ セット内を移動するために使用できます。ツールバーの下部にある各ボタンについて、以下に説明します。
- 以前に定義したビュー間を移動するには、[ 進む ] ボタンと [戻る ] ボタンを使用します。
注: ホーム、 進む、および 戻る ボタンは、Web ブラウザーの ホーム、 進む、および 戻る の各コントロールに似ています。 ホーム はデフォルトの画面、元の画像に戻ります。 - [ズーム] ボタンを使用して、画面移動とズームを行います。パンとズームをアクティブにするには、[ズーム]ボタンを押してから、画像内の目的の場所にマウスを移動します。
- 図をパンするには、マウスの左ボタンを押したまま、新しい位置にドラッグします。マウス ボタンを放すと、イメージ内の選択したポイントが新しい位置に表示されます。パン中に 、x キーまたは y キーを押したままにして、モーションを x 軸または y 軸に制限します。
- ズームするには、マウスの右ボタンを押したまま、新しい場所にドラッグします。右に移動して X 軸を拡大表示し、左に移動して X 軸を縮小します。Y 軸と上下の動きに対して同じ操作を行います。ズームする場合、マウスの下の点は静止したままで、そのポイントの周囲を拡大または縮小できます。修飾子キー x、 y、または CONTROL を使用して、それぞれ x、 y、またはアスペクト比の保持にズームを制限します。
- 四角形にズーム モードをアクティブにするには、[ 四角形にズーム ] ボタンをクリックします。イメージの上にカーソルを置き、マウスの左ボタンを押します。ボタンを押したまま新しい位置にマウスをドラッグして、矩形領域を定義します。
注: 軸ビューの制限は、マウスの左ボタンを押すと、定義された領域にズームされます。軸ビューの制限は、マウスの右ボタンを押すと縮小され、定義された領域に元の軸が配置されます。 - サブプロット設定ツールを使用して、サブプロットの外観を設定します。
注: サブプロットの左、右、上、下の辺、行と列の間のスペースは、ストレッチまたは圧縮できます。 - ファイル保存ダイアログを開くには、[ 保存 ] ボタンをクリックし、.png、.ps、.eps、.svg、または.pdf形式でファイルを保存します。
Access restricted. Please log in or start a trial to view this content.
結果
培養中の樹状ネットワークの画像に対して代表的な分析を行った。Baranesらで説明したように細胞を抽出した。16,17.簡単に言えば、海馬細胞を出生後ラットの脳から抽出し、2Dガラスのカバーリップで1〜2週間培養した。培養物を次に固定し、樹状タンパク質マーカー、微小管関連タンパク質2(MAP2)に対する抗体を用いて間接的な免疫蛍光を介?...
Access restricted. Please log in or start a trial to view this content.
ディスカッション
生物学的画像データに追いつくためには、2D画像から形態学的情報を抽出するための効果的な戦略が緊急に必要とされています。画像データは数時間で生成できますが、画像の詳細な分析には長い時間がかかります。その結果、画像処理は明らかに多くの分野で大きな障害となっています。これは、特に生物学的サンプルを扱う場合、データの複雑度が高いためです。また、多くのユーザー?...
Access restricted. Please log in or start a trial to view this content.
開示事項
著者らは、競合する財政的利益はないと宣言している。
謝辞
著者らは、文化イメージの準備のためにオルリー・ワイス博士に感謝したいと思います。
Access restricted. Please log in or start a trial to view this content.
資料
| Name | Company | Catalog Number | Comments |
| Matplotlib | 2002 - 2012 John Hunter, Darren Dale, Eric Firing, Michael Droettboom and the Matplotlib development team; 2012 - 2021 The Matplotlib development team. | 3.4.2 | a Python 2D plotting library |
| matplotlib-scalebar | Philippe Pinard | 0.7.2 | artist for matplotlib to display a scale bar |
| NumPy | The NumPy community. | 1.20.3 | fundamental package for scientific computing library |
| OpenCV | OpenCV team | 4.5.2.54 | Open Source Computer Vision Library |
| PyCharm | JetBrains | 2020.3.1 (Community Edition) version | Build #PC-203.6682.86, built on December 18, 2020. Runtime version: 11.0.9.1+11-b1145.37 amd64. VM: OpenJDK 64-Bit Server VM by JetBrains s.r.o. Windows 10 10.0. Memory: 978M, Cores: 4 |
| PyQt5 | Riverbank Computing | 5.15.4 | manage the GUI |
| python | Python Software Foundation License | 3.9 version | |
| Qt Designer | The QT Company Ltd. | 5.11.1 version | |
| scipy | Community library project | 1.6.3 | Python-based ecosystem of open-source software for mathematics, science, and engineering |
| Seaborn | Michael Waskom. | 0.11.1 | Python's Statistical Data Visualization Library. |
| Windows 10 | Microsoft | ||
| Xlsxwriter | John McNamara | 1.4.3 | Python module for creating Excel XLSX files |
参考文献
- Ferrante, M., Migliore, M., Ascoli, G. Functional impact of dendritic branch-point morphology. Journal of Neuroscience. 33 (5), 2156-2165 (2013).
- Spruston, N. Pyramidal neurons: dendritic structure and synaptic integration. Nature Reviews Neuroscience. 9 (3), 206-221 (2008).
- Chklovskii, D. Synaptic Connectivity and Neuronal Morphology: Two Sides of the Same Coin. Neuron. 43 (5), 609-617 (2004).
- Chapleau, C., Larimore, J., Theibert, A., Pozzo-Miller, L. Modulation of dendritic spine development and plasticity by BDNF and vesicular trafficking: fundamental roles in neurodevelopmental disorders associated with mental retardation and autism. Journal of Neurodevelopmental Disorders. 1 (3), 185-196 (2009).
- Irwin, S. Dendritic Spine Structural Anomalies in Fragile-X Mental Retardation Syndrome. Cerebral Cortex. 10 (10), 1038-1044 (2000).
- Kaufmann, W. Dendritic anomalies in disorders associated with mental retardation. Cerebral Cortex. 10 (10), 981-991 (2000).
- Pinchas, M., Baranes, D. Dendritic branch intersections are structurally regulated targets for efficient axonal wiring and synaptic clustering. PLoS ONE. 8 (12), 82083(2013).
- Cove, J., Blinder, P., Baranes, D. Contacts among non-sister dendritic branches at bifurcations shape neighboring dendritic branches and pattern their synaptic inputs. Brain Research. 1251, 30-41 (2009).
- Blinder, P., Cove, J., Foox, M., Baranes, D. Convergence among non-sister dendritic branches: An activity-controlled mean to strengthen network connectivity. PLoS ONE. 3 (11), 3782(2008).
- Glaser, J., Glaser, E. Neuron imaging with neurolucida - PC-based system for image combining microscopy. Computerized Medical Imaging and Graphics. 14 (5), 307-317 (1990).
- Scorcioni, R., Polavaram, S., Ascoli, G. L-Measure: a web-accessible tool for the analysis, comparison and search of digital reconstructions of neuronal morphologies. Nature Protocols. 3 (5), 866-876 (2008).
- Torben-Nielsen, B. An efficient and extendable python library to analyze neuronal morphologies. Neuroinformatics. 12 (4), 619-622 (2014).
- Parekh, R., Ascoli, G. Neuronal morphology goes digital: A research hub for cellular and system neuroscience. Neuron. 78 (1), 206(2013).
- heng, J., Zhou, X., Sabatini, B. L., Wong, S. T. C. NeuronIQ: A novel computational approach for automatic dendrite SPINES detection and analysis. 2007 IEEE/NIH Life Science Systems and Applications Workshop. , 168-171 (2007).
- Image processing and analysis in Java. NIH. ImageJ. , Available from: https://imagej.nih.gov/ij (2021).
- Peretz, H., Talpalar, A. E., Vago, R., Baranes, D. Superior survival and durability of neurons and astrocytes on 3-dimensional aragonite biomatrices. Tissue Engineering. 13, 461-472 (2007).
- Morad, T. I., Hendler, R. M., Weiss, O. E., Canji, E. A., Merfeld, I., Dubinsky, Z., Minnes, R., Francis, Y. I., Baranes, D. Gliosis of astrocytes cultivated on coral skeleton is regulated by the matrix surface topography. Biomedical Materials. 14 (4), 045005(2019).
Access restricted. Please log in or start a trial to view this content.
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved