A subscription to JoVE is required to view this content. Sign in or start your free trial.
Method Article
זיהוי אוטומטי של ענפים דנדריטיים והתמצאותם
In This Article
Summary
מוצג הוא כלי חישובי המאפשר מדידה אוטומטית פשוטה וישירה של כיוונים של ענפים דנדריטיים עצביים מתמונות פלואורסצנטיות 2D.
Abstract
המבנה של עצים דנדריטיים עצביים ממלא תפקיד מרכזי בשילוב של תשומות סינפטיות בנוירונים. לכן, אפיון המורפולוגיה של דנדריטים חיוני להבנה טובה יותר של תפקוד עצבי. עם זאת, המורכבות של עצים דנדריטיים, הן כאשר מבודדים ובמיוחד כאשר ממוקם בתוך רשתות עצביות, לא הובן לחלוטין. פיתחנו כלי חישובי חדש, SOA (פילוח וניתוח אוריינטציה), המאפשר מדידה אוטומטית של הכיוון של ענפים דנדריטיים מתמונות פלואורסצנטיות של תרביות עצביות דו-ממדיות. SOA, שנכתב בפייתון, משתמש בפילוח כדי להבחין בין ענפים דנדריטיים מרקע התמונה וצובר מסד נתונים בכיוון המרחבי של כל ענף. לאחר מכן משמש מסד הנתונים לחישוב פרמטרים מורפולוגיים כגון התפלגות כיוונית של ענפים דנדריטיים ברשת ושכיחות צמיחת הענף הדנדריטי המקביל. הנתונים המתקבלים יכולים לשמש כדי לזהות שינויים מבניים בדנדריטים בתגובה לפעילות עצבית ולגירויים ביולוגיים ופרמקולוגיים.
Introduction
מורפוגנזה דנדריטית היא נושא מרכזי במדעי המוח, שכן המבנה של העץ הדנדריטי משפיע על המאפיינים החישוביים של שילוב סינפטי בנוירונים1,2,3. יתר על כן, חריגות מורפולוגיות ושינויים בענפים דנדריטיים מעורבים בהפרעות ניווניות נוירו-התפתחותיות4,5,6. בתרבויות עצביות שבהן ניתן לדמיין בקלות רבה יותר את ההשלכות הדנדריטיות, האינטראקציות בין ענפים דנדריטיים שאינם אחות מווסתים את האתרים ואת היקף ההתקבצות הסינפטית לאורך הענפים, התנהגות שעשויה להשפיע על שיתוף פעולה סינפטי ופלסטיות7,8,9. לכן, אפיון הפרמטרים המורפולוגיים של העץ דנדריטי באמצעות תרביות עצביות דו מימדיות (2D) הוא יתרון להבנת מורפוגנזה דנדריטית ופונקציונליות של יחיד ורשתות של נוירונים. עם זאת, זוהי משימה מאתגרת מכיוון שענפים דנדריטיים יוצרים רשת מורכבת אפילו בתרבויות נוירונים "פשוטים" ודו-חמצניים.
מספר כלים פותחו כדי לעקוב ולנתח באופן אוטומטי מבנים דנדריטיים10,11,12,13. עם זאת, רוב הכלים האלה מיועדים לרשתות נוירונים תלת-ממדיות והם באופן טבעי מורכבים מדי לשימוש עם רשתות 2D. לעומת זאת, כלי ניתוח מורפולוגיים פחות מתקדמים כרוכים בדרך כלל ברכיב משמעותי של עבודת כפיים בסיוע מחשב, שהיא גוזלת זמן רב ורגיש להטיית מפעיל14. כלים חצי אוטומטיים קיימים, כגון 'ImageJ'15 (חבילת עיבוד תמונה בקוד פתוח של NIH עם אוסף עצום של כלי ניתוח תמונה ביולוגית שפותחו על ידי הקהילה), מפחיתים במידה רבה את עבודת היד הידנית של המשתמשים. עם זאת, עדיין יש צורך בהתערבויות ידניות מסוימות במהלך עיבוד התמונה, ואיכות הפילוח יכולה להיות פחות רצויה.
מאמר זה מציג את SOA, כלי אוטומטי פשוט המאפשר פילוח ישיר וניתוח כיוון של ענפים דנדריטיים בתוך רשתות נוירונים 2D. SOA יכול לזהות אובייקטים שונים דמויי קו בתמונות 2D ולאפיין את המאפיינים המורפולוגיים שלהם. כאן, השתמשנו ב- SOA לפיזור סניפים דנדריטיים בתמונות פלואורסצנטיות 2D של רשתות דנדריטיות בתרבות. התוכנה מזהה את הענפים הדנדריטיים ומבצעת בהצלחה מדידות של פרמטרים מורפולוגיים כגון מקביליות והפצה מרחבית. ניתן להתאים את SOA בקלות לניתוח תהליכים תאיים מסוגי תאים אחרים ולחקר רשתות לא ביולוגיות.
Access restricted. Please log in or start a trial to view this content.
Protocol
הערה: משרד הבריאות הישראלי אישר שימוש בעכברים על פי פרוטוקול IL-218-01-21 לשימוש אתי בבעלי חיים ניסיוניים. SOA תואם רק ל- Windows 10 ול- Python 3.9. הוא זמין כקוד קוד פתוח: https://github.com/inbar2748/DendriteProject. בקישור זה, יש גם README. קובץ DM בעל הוראות להורדת התוכנה, קישור לאתר האינטרנט של התוכנה וקובץ דרישות המכיל מידע על הגירסאות הנדרשות של כל החבילות. דוגמאות נוספות לניתוח שבוצעו באמצעות התוכנה סופקו גם שם.
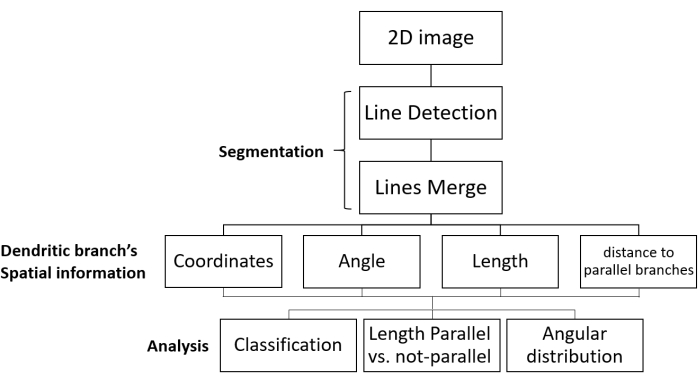
איור 1: זרימת עבודה של SOA עבור פילוח וניתוח כיוון צמיחה. מוצגים הם שלבי העיבוד של תמונות פלואורסצנטיות של רשתות דנדריטיות וניתוח נתונים. התמונה הדו-פעמית מועלית, מפולחת (בשני שלבים: ענפים דנדריטיים מזוהים כשורות, ואז הקווים הרלוונטיים ממוזגים), והמידע המרחבי של כל ענף דנדריטי מתקבל. הנתונים נאספים עבור כל הענפים הדנדריטיים בתמונה. לבסוף, הנתונים מנותחים כדי לתת את הפרמטרים המורפולוגיים הרצויים. קיצור: SOA = פילוח וניתוח כיוון. אנא לחץ כאן כדי להציג גירסה גדולה יותר של איור זה.
1. פתח את יישום SOA.
- פתח את כתובת כתובת ה- URL: https://mega.nz/folder/bKZhmY4I#4WAaec4biiGt4_1lJlL4WA, מצא את התיקיה .zip SOA מכווצת והורד את קובץ ה- ZIP על-ידי לחיצה כפולה.
- בטל את הניצוי של התיקיה על-ידי לחיצה באמצעות לחצן העכבר הימני על SOA.zip ובחר Extract Files. שים לב לחלון נתיב החילוץ והאפשרויות שנפתח ובתיבת הטקסט כתובת יעד המציגה את הנתיב עבור הקבצים שחולצו. כדי לחלץ למיקום אחר, לחץ על אחת התיקיות בחלונית הימנית של החלון כדי להפוך אותו לתיקיית היעד. לחץ על אישור כדי לחלץ את הקבצים לתיקיה זו.
- פתח את קובץ SOA שחולץ ולחץ פעמיים על .EXE SOA. המתן לפתיחת חלון שחור ולאחר מכן יופיע היישום.

איור 2: דוגמה לזרימת עבודה המשתמשת ב- GUI של SOA. עמודה שמאלית: מקטעי GUI של זרימת העבודה. עמודה אמצעית: תמונה של רשת דנדריטית, מעובדת במהלך זרימת העבודה (סרגל קנה מידה: 20 מיקרומטר). עמודה ימנית: הגדלה של האזור המסומן במלבן אדום בתמונות של העמודה האמצעית (סרגל קנה מידה: 4 מיקרומטר). שלב 1: בחירה והעלאה של תמונה. שלב 2: השלב הראשון של הפילוח הוא איתור קווים המייצגים את הענפים הדנדריטיים המזוהים. שלב 3: השלב השני של הפילוח הוא מיזוג מבוסס קרבה של רירית פלח בענפים דנדריטיים בודדים. ניתן לשנות את ההגדרות של כל השלבים. קיצורים: SOA = פילוח וניתוח כיוון; GUI = ממשק משתמש גרפי. אנא לחץ כאן כדי להציג גירסה גדולה יותר של איור זה.
2. פתח תמונה לניתוח.
- בשורת התפריטים 'העלאת מציג SOA ' | בחר בחר | קבצים בחר תמונה מקבצי המחשב | לחץ על זה (קבצי .png .jpg .tif .bmp בלבד) | פתח | התבונן בנתיב הקובץ | הבא.
3. אופטימיזציה של פילוח
הערה: בשורת התפריטים מאפייני מציג SOA , שנה את הערכים של הפרמטרים שנבחרו כדי להתאים את הגדרות תהליך הפילוח. תיאור מפורט של הפרמטרים, כגון הסף, ניתן בחומר המשלים.
- בקצוות, התאם את הסף עבור התצוגה על-ידי בחירת הסף והזנת מספר.
הערה: ככל שהמספר עבור הסף נמוך יותר, כך מזוהים יותר שורות. הסף הוא מספר הנע בין 0 ל- 255. ערך ברירת המחדל הוגדר כ- 0. - בשורות מיזוג:
- התאם את המרחק המינימלי למיזוג עבור התצוגה על-ידי בחירת המרחק Min למיזוג והזנת מספר.
הערה: המרחק בין המינים למיזוג נע בין 0 ל- 30 פיקסלים. ערך ברירת המחדל מוגדר ל- 20. - התאם את הזווית המינימלית למיזוג עבור התצוגה על-ידי בחירת הזווית Min למיזוג והזנת מספר.
הערה: זווית ה-Min למיזוג נעה בין 0 ל- 30°. ערך ברירת המחדל מוגדר ל- 10.
- התאם את המרחק המינימלי למיזוג עבור התצוגה על-ידי בחירת המרחק Min למיזוג והזנת מספר.
- לחץ על יצירת תמונת פילוח תצוגה מקדימה.
הערה: תמונת תצוגה מקדימה של תוצאות הפילוח תוצג בהתאם לערכים המעודכנים. בנוסף, יוצגו מספר השורות לפני המיזוג ומספר השורות לאחר המיזוג. - שנה את הפרמטרים כדי להשיג זיהוי מרבי של מקטעים. אם יש צורך לשנות את המאפיינים, לחץ על לחצן סגור חלון ובצע את שלבים 3.1-3.4.
4. צור את קבצי הפלט.
- לחץ על אישור כדי להציג באופן חזותי את תמונות הפילוח ואת הגרפים המנתחים. שים לב לחלון שמופיע לבחירת מיקום שבו יישמר קובץ .xlsx.
- הוספת שם קובץ | בחר שמור | המתן לקובץ .xlsx עם נתונים שייווצר וישמרו.
הערה: בנוסף לקובץ .xlsx, הקבצים הבאים יוצגו באופן אוטומטי: קובץ המציג את התמונה המקורית, תמונת זיהוי הקו, התמונה הסופית של הפילוח ושלושה גרפי ניתוח.
5. סרגל הכלים לניווט
הערה: סרגל כלים לניווט כלול בכל חלונות האיור וניתן להשתמש בו כדי לנווט בערכת הנתונים. כל אחד מהלחצנים בתחתית סרגל הכלים מתואר להלן.
- כדי לנווט הלוך ושוב בין תצוגות שהוגדרו קודם לכן, השתמש בלחצנים קדימה ואחורה.
הערה: הלחצנים 'בית', 'קדימה' ו'הקודם' דומים לפקדים 'בית', 'קדימה' ו'הקודם' בדפדפן אינטרנט. הבית חוזר למסך ברירת המחדל, התמונה המקורית. - השתמש בלחצן זום כדי לגלול ולהגדיל את התצוגה. כדי להפעיל גלילה רציפה ושינוי גודל תצוגה, לחץ על לחצן זום ולאחר מכן הזז את העכבר למיקום הרצוי בתמונה.
- כדי לגלול את האיור, לחץ והחזק את לחצן העכבר השמאלי תוך כדי גרירתו למיקום חדש. שחרר את לחצן העכבר והנקודה שנבחרה בתמונה תופיע במיקום החדש. בעת גלילה רציפה, החזק את מקשי x או y ל לדחיית התנועה לצירים x או y , בהתאמה.
- כדי להגדיל את התצוגה, החזק את לחצן העכבר הימני לחוץ וגרור אותו למיקום חדש. זוז ימינה כדי להגדיל את ציר ה-x ולנוע שמאלה כדי להקטין את התצוגה בציר ה-x. עשה את אותו הדבר עבור ציר ה- y ותנועות למעלה/למטה. בעת שינוי גודל תצוגה, שים לב שהנקודה מתחת לעכבר נשארת נייחת, ומאפשרת הגדלה או הקטנה של נקודה זו. השתמש במקשי הצירוף x, y או CONTROL כדי להגביל את גודל התצוגה לשימור יחס x, y או רוחב-גובה, בהתאמה.
- כדי להפעיל את מצב מרחק מלבן למלבן, לחצו על הלחצן 'התקרבות למלבן '. מקם את הסמן מעל התמונה ולחץ על לחצן העכבר השמאלי. גרור את העכבר למיקום חדש בעת החזקת הלחצן כדי להגדיר אזור מלבני.
הערה: מגבלות תצוגת הצירים יוגדלו לאזור המוגדר בעת לחיצה על לחצן העכבר השמאלי. מגבלות תצוגת הצירים יוגדלו בעת לחיצה על לחצן העכבר הימני, תוך מיקום הצירים המקוריים באזור המוגדר. - השתמש בכלי תצורת תת-עלילה כדי לקבוע את תצורת המראה של עלילת המשנה.
הערה: ניתן למתוח או לדחוס את הצד השמאלי, הימני, העליון והתחתון של לוח המשנה, כמו גם את הרווח בין שורות ועמודות. - כדי לפתוח תיבת דו-שיח לשמירת קבצים, לחץ על לחצן שמור ושמור את הקובץ בתבניות הבאות: .png, .ps, .eps, .svg או .pdf.
Access restricted. Please log in or start a trial to view this content.
תוצאות
ניתוח מייצג בוצע על תמונות של רשתות דנדריטיות בתרבות. תאים חולצו כמתואר על ידי Baranes ואח '. 16,17. בקצרה, תאי ההיפוקמפוס חולצו ממוחות של חולדות לאחר הולדות וטופחו על כיסויי זכוכית דו-פעמיים במשך שבוע-שבועיים. התרבויות תוקנו והוכתמו אז באמצעות אימונופלואורס?...
Access restricted. Please log in or start a trial to view this content.
Discussion
אסטרטגיות יעילות לחילוץ מידע מורפולוגי מתמונות 2D נדרשות בדחיפות כדי לעמוד בקצב של נתוני הדמיה ביולוגיים. למרות שניתן להפיק נתוני הדמיה בשעות, ניתוח מעמיק של התמונות אורך זמן רב. כתוצאה מכך, עיבוד תמונה הפך בבירור למכשול מרכזי בתחומים רבים. זאת, בין היתר, בשל המורכבות הגבוהה של הנתונים, במי?...
Access restricted. Please log in or start a trial to view this content.
Disclosures
המחברים מצהירים כי אין להם אינטרסים כלכליים מתחרים.
Acknowledgements
המחברים מבקשים להודות לד"ר אורלי וייס על הכנת תמונות התרבות.
Access restricted. Please log in or start a trial to view this content.
Materials
| Name | Company | Catalog Number | Comments |
| Matplotlib | 2002 - 2012 John Hunter, Darren Dale, Eric Firing, Michael Droettboom and the Matplotlib development team; 2012 - 2021 The Matplotlib development team. | 3.4.2 | a Python 2D plotting library |
| matplotlib-scalebar | Philippe Pinard | 0.7.2 | artist for matplotlib to display a scale bar |
| NumPy | The NumPy community. | 1.20.3 | fundamental package for scientific computing library |
| OpenCV | OpenCV team | 4.5.2.54 | Open Source Computer Vision Library |
| PyCharm | JetBrains | 2020.3.1 (Community Edition) version | Build #PC-203.6682.86, built on December 18, 2020. Runtime version: 11.0.9.1+11-b1145.37 amd64. VM: OpenJDK 64-Bit Server VM by JetBrains s.r.o. Windows 10 10.0. Memory: 978M, Cores: 4 |
| PyQt5 | Riverbank Computing | 5.15.4 | manage the GUI |
| python | Python Software Foundation License | 3.9 version | |
| Qt Designer | The QT Company Ltd. | 5.11.1 version | |
| scipy | Community library project | 1.6.3 | Python-based ecosystem of open-source software for mathematics, science, and engineering |
| Seaborn | Michael Waskom. | 0.11.1 | Python's Statistical Data Visualization Library. |
| Windows 10 | Microsoft | ||
| Xlsxwriter | John McNamara | 1.4.3 | Python module for creating Excel XLSX files |
References
- Ferrante, M., Migliore, M., Ascoli, G. Functional impact of dendritic branch-point morphology. Journal of Neuroscience. 33 (5), 2156-2165 (2013).
- Spruston, N. Pyramidal neurons: dendritic structure and synaptic integration. Nature Reviews Neuroscience. 9 (3), 206-221 (2008).
- Chklovskii, D. Synaptic Connectivity and Neuronal Morphology: Two Sides of the Same Coin. Neuron. 43 (5), 609-617 (2004).
- Chapleau, C., Larimore, J., Theibert, A., Pozzo-Miller, L. Modulation of dendritic spine development and plasticity by BDNF and vesicular trafficking: fundamental roles in neurodevelopmental disorders associated with mental retardation and autism. Journal of Neurodevelopmental Disorders. 1 (3), 185-196 (2009).
- Irwin, S. Dendritic Spine Structural Anomalies in Fragile-X Mental Retardation Syndrome. Cerebral Cortex. 10 (10), 1038-1044 (2000).
- Kaufmann, W. Dendritic anomalies in disorders associated with mental retardation. Cerebral Cortex. 10 (10), 981-991 (2000).
- Pinchas, M., Baranes, D. Dendritic branch intersections are structurally regulated targets for efficient axonal wiring and synaptic clustering. PLoS ONE. 8 (12), 82083(2013).
- Cove, J., Blinder, P., Baranes, D. Contacts among non-sister dendritic branches at bifurcations shape neighboring dendritic branches and pattern their synaptic inputs. Brain Research. 1251, 30-41 (2009).
- Blinder, P., Cove, J., Foox, M., Baranes, D. Convergence among non-sister dendritic branches: An activity-controlled mean to strengthen network connectivity. PLoS ONE. 3 (11), 3782(2008).
- Glaser, J., Glaser, E. Neuron imaging with neurolucida - PC-based system for image combining microscopy. Computerized Medical Imaging and Graphics. 14 (5), 307-317 (1990).
- Scorcioni, R., Polavaram, S., Ascoli, G. L-Measure: a web-accessible tool for the analysis, comparison and search of digital reconstructions of neuronal morphologies. Nature Protocols. 3 (5), 866-876 (2008).
- Torben-Nielsen, B. An efficient and extendable python library to analyze neuronal morphologies. Neuroinformatics. 12 (4), 619-622 (2014).
- Parekh, R., Ascoli, G. Neuronal morphology goes digital: A research hub for cellular and system neuroscience. Neuron. 78 (1), 206(2013).
- heng, J., Zhou, X., Sabatini, B. L., Wong, S. T. C. NeuronIQ: A novel computational approach for automatic dendrite SPINES detection and analysis. 2007 IEEE/NIH Life Science Systems and Applications Workshop. , 168-171 (2007).
- Image processing and analysis in Java. NIH. ImageJ. , Available from: https://imagej.nih.gov/ij (2021).
- Peretz, H., Talpalar, A. E., Vago, R., Baranes, D. Superior survival and durability of neurons and astrocytes on 3-dimensional aragonite biomatrices. Tissue Engineering. 13, 461-472 (2007).
- Morad, T. I., Hendler, R. M., Weiss, O. E., Canji, E. A., Merfeld, I., Dubinsky, Z., Minnes, R., Francis, Y. I., Baranes, D. Gliosis of astrocytes cultivated on coral skeleton is regulated by the matrix surface topography. Biomedical Materials. 14 (4), 045005(2019).
Access restricted. Please log in or start a trial to view this content.
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved