È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Identificazione automatica dei rami dendritici e loro orientamento
In questo articolo
Riepilogo
Presentato è uno strumento computazionale che consente la misurazione automatica semplice e diretta degli orientamenti dei rami dendritici neuronali da immagini di fluorescenza 2D.
Abstract
La struttura degli alberi dendritici neuronali svolge un ruolo chiave nell'integrazione degli input sinaptici nei neuroni. Pertanto, la caratterizzazione della morfologia dei dendriti è essenziale per una migliore comprensione della funzione neuronale. Tuttavia, la complessità degli alberi dendritici, sia quando isolati che soprattutto quando si trovano all'interno di reti neuronali, non è stata completamente compresa. Abbiamo sviluppato un nuovo strumento computazionale, SOA (Segmentation and Orientation Analysis), che consente la misurazione automatica dell'orientamento dei rami dendritici da immagini a fluorescenza di colture neuronali 2D. SOA, scritto in Python, utilizza la segmentazione per distinguere i rami dendritici dallo sfondo dell'immagine e accumula un database sulla direzione spaziale di ciascun ramo. Il database viene quindi utilizzato per calcolare parametri morfologici come la distribuzione direzionale dei rami dendritici in una rete e la prevalenza della crescita parallela dei rami dendritici. I dati ottenuti possono essere utilizzati per rilevare cambiamenti strutturali nei dendriti in risposta all'attività neuronale e agli stimoli biologici e farmacologici.
Introduzione
La morfogenesi dendritica è un argomento centrale nelle neuroscienze, poiché la struttura dell'albero dendritico influenza le proprietà computazionali dell'integrazione sinaptica nei neuroni1,2,3. Inoltre, anomalie morfologiche e modificazioni nei rami dendritici sono implicate in disturbi degenerativi e dello sviluppo neurologico4,5,6. Nelle colture neuronali in cui la ramificazione dendritica può essere visualizzata più facilmente, le interazioni tra rami dendritici non fratelli regolano i siti e l'estensione del clustering sinaptico lungo i rami, un comportamento che può influenzare la coattività sinaptica e la plasticità7,8,9. Pertanto, la caratterizzazione dei parametri morfologici dell'albero dendritico utilizzando colture neuronali bidimensionali (2D) è vantaggiosa per comprendere la morfogenesi dendritica e la funzionalità di singoli e reti di neuroni. Tuttavia, questo è un compito impegnativo perché i rami dendritici formano una rete complessa anche in colture neuronali 2D "semplificate".
Sono stati sviluppati diversi strumenti per tracciare e analizzare automaticamente le strutture dendritiche10,11,12,13. Tuttavia, la maggior parte di questi strumenti sono progettati per le reti neuronali 3D e sono naturalmente troppo complessi per essere utilizzati con le reti 2D. Al contrario, gli strumenti di analisi morfologica meno avanzati coinvolgono in genere una componente significativa del lavoro manuale assistito da computer, che richiede molto tempo e suscettibile alla distorsione dell'operatore14. Gli strumenti semi-automatici esistenti, come "ImageJ"15 (un pacchetto di elaborazione delle immagini open source NIH con una vasta collezione di strumenti di analisi delle immagini biologiche sviluppati dalla comunità), riducono in gran parte il lavoro manuale dell'utente. Tuttavia, alcuni interventi manuali sono ancora necessari durante l'elaborazione delle immagini e la qualità della segmentazione può essere inferiore a quella desiderabile.
Questo documento presenta la SOA, un semplice strumento automatizzato che consente la segmentazione diretta e l'analisi dell'orientamento dei rami dendritici all'interno di reti neuronali 2D. La SOA è in grado di rilevare vari oggetti simili a linee in immagini 2D e caratterizzarne le proprietà morfologiche. Qui, abbiamo usato la SOA per segmentare i rami dendritici in immagini di fluorescenza 2D di reti dendritiche in coltura. Il software identifica i rami dendritici ed esegue con successo misurazioni di parametri morfologici come il parallelismo e la distribuzione spaziale. La SOA può essere facilmente adattata per l'analisi di processi cellulari di altri tipi cellulari e per lo studio di reti non biologiche.
Access restricted. Please log in or start a trial to view this content.
Protocollo
NOTA: Il Ministero della Salute israeliano ha approvato l'uso di topi secondo il protocollo IL-218-01-21 per l'uso etico di animali da esperimento. SOA è compatibile solo con Windows 10 e Python 3.9. È disponibile come codice open source: https://github.com/inbar2748/DendriteProject. A questo link, c'è anche un README. DM che contiene le istruzioni per il download del software, un collegamento al sito Web del software e un file dei requisiti contenente informazioni sulle versioni richieste di tutti i pacchetti. Ulteriori esempi di analisi eseguite utilizzando il software sono stati forniti anche lì.
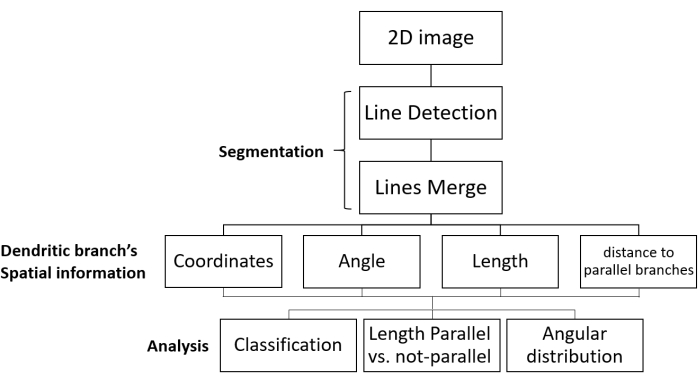
Figura 1: Flusso di lavoro SOA per la segmentazione e l'analisi della direzione di crescita. Sono mostrate le fasi di elaborazione delle immagini fluorescenti delle reti dendritiche e l'analisi dei dati. L'immagine 2D viene caricata, segmentata (in due passaggi: i rami dendritici vengono rilevati come linee e quindi le linee pertinenti vengono unite) e si ottengono le informazioni spaziali di ciascun ramo dendritico. I dati vengono raccolti per tutti i rami dendritici nell'immagine. Infine, i dati vengono analizzati per fornire i parametri morfologici desiderati. Abbreviazione: SOA = analisi di segmentazione e orientamento. Fare clic qui per visualizzare una versione più grande di questa figura.
1. Aprire l'applicazione SOA.
- Apri l'indirizzo URL: https://mega.nz/folder/bKZhmY4I#4WAaec4biiGt4_1lJlL4WA, trova la cartella compressa SOA.zip e scarica il file ZIP facendo doppio clic.
- Decomprimi la cartella facendo clic con il pulsante destro del mouse su SOA.zip e scegli Estrai file. Osservare la finestra Percorso di estrazione e opzioni visualizzata e la casella di testo Indirizzo di destinazione che visualizza il percorso dei file estratti. Per estrarre in una posizione diversa, fare clic su una delle cartelle nel pannello di destra della finestra per renderla la cartella di destinazione. Fare clic su OK per estrarre i file in tale cartella.
- Aprire il file SOA estratto e fare doppio clic su SOA.exe. Attendi che si apra una finestra nera, dopodiché apparirà l'applicazione.

Figura 2: Esempio di flusso di lavoro che utilizza la GUI della SOA. Colonna di sinistra: sezioni GUI del flusso di lavoro. Colonna centrale: immagine di una rete dendritica, elaborata durante il flusso di lavoro (barra di scala: 20 μm). Colonna di destra: ingrandimento dell'area contrassegnata da un rettangolo rosso nelle immagini della colonna centrale (barra della scala: 4 μm). Passo 1: Selezione e caricamento di un'immagine. Fase 2: La prima fase della segmentazione è il rilevamento di linee che rappresentano i rami dendritici identificati. Fase 3: La seconda fase della segmentazione è la fusione basata sulla prossimità del rivestimento del segmento nei singoli rami dendritici. Le impostazioni di tutti i passaggi possono essere modificate. Abbreviazioni: SOA = analisi di segmentazione e orientamento; GUI = interfaccia utente grafica. Fare clic qui per visualizzare una versione più grande di questa figura.
2. Apri un'immagine da analizzare.
- Nella barra dei menu Caricamento visualizzatore SOA | seleziona Scegli file | Scegliere un'immagine dai file del computer | cliccaci sopra (solo .png .jpg .tif .bmp file) | Apri | Osservare il percorso del file | Prossimo.
3. Ottimizzazione della segmentazione
NOTA: nella barra dei menu Proprietà del visualizzatore SOA , modificare i valori dei parametri selezionati per regolare le impostazioni del processo di segmentazione. Una descrizione dettagliata dei parametri, come la Soglia, è fornita nel Materiale supplementare.
- In Bordi,regolate la soglia per la visualizzazione selezionando la soglia e immettendo un numero.
NOTA: più basso è il numero per la soglia, più righe vengono rilevate. Soglia è un numero che varia da 0 a 255. Il valore predefinito è stato impostato su 0. - In Unisci righe:
- Regolate la distanza minima da unire per la visualizzazione selezionando la distanza minima da unire e immettendo un numero.
NOTA: la distanza minima da unire varia da 0 a 30 pixel. Il valore predefinito è impostato su 20. - Regolate l'angolo minimo da unire per la visualizzazione selezionando l'angolo minimo da unire e immettendo un numero.
NOTA: l'angolo minimo di unione varia da 0 a 30°. Il valore predefinito è impostato su 10.
- Regolate la distanza minima da unire per la visualizzazione selezionando la distanza minima da unire e immettendo un numero.
- Fare clic su Crea immagine di segmentazione di anteprima.
NOTA: verrà visualizzata un'immagine di anteprima dei risultati della segmentazione in base ai valori aggiornati. Inoltre, verranno visualizzati il numero di righe prima dell'unione e il numero di righe dopo l'unione. - Modificare i parametri per ottenere la massima identificazione dei segmenti. Se è necessario modificare le Proprietà, fare clic sul pulsante Chiudi finestra e seguire i passaggi 3.1-3.4.
4. Creare i file di output.
- Premere OK per visualizzare le immagini di segmentazione e i grafici di analisi. Osservare la finestra visualizzata per selezionare una posizione in cui verrà salvato il file .xlsx.
- Inserire un nome file | Scegli Salva | attendere che il file .xlsx con i dati venga creato e salvato.
NOTA: oltre al file .xlsx, verranno visualizzati automaticamente i seguenti file: un file che presenta l'immagine originale, l'immagine di riconoscimento delle linee, l'immagine finale della segmentazione e tre grafici di analisi.
5. Barra degli strumenti di navigazione
NOTA: una barra degli strumenti di navigazione è inclusa in tutte le finestre delle figure e può essere utilizzata per spostarsi all'interno del set di dati. Ciascuno dei pulsanti nella parte inferiore della barra degli strumenti è descritto di seguito.
- Per spostarsi avanti e indietro tra le viste definite in precedenza, utilizzare i pulsanti Avanti e Indietro .
NOTA: i pulsanti Home, Avanti e Indietro sono simili ai controlli Home, Avanti e Indietro di un browser Web. Home ritorna alla schermata predefinita, l'immagine originale. - Utilizzare il pulsante Zoom per eseguire la panoramica e lo zoom. Per attivare la panoramica e lo zoom, premere il pulsante Zoom , quindi spostare il mouse nella posizione desiderata nell'immagine.
- Per eseguire una panoramica della figura, tenere premuto il pulsante sinistro del mouse mentre lo si trascina in una nuova posizione. Rilascia il pulsante del mouse e il punto selezionato nell'immagine apparirà nella nuova posizione. Durante la panoramica, tenete premuti i tasti x o y per limitare il movimento rispettivamente agli assi x o y.
- Per ingrandire, tieni premuto il pulsante destro del mouse e trascinalo in una nuova posizione. Spostarsi a destra per ingrandire l'asse x e spostarsi a sinistra per ridurre lo zoom sull'asse x. Fate lo stesso per i movimenti dell'asse y e su/giù. Quando si esegue lo zoom, si noti che il punto sotto il mouse rimane fermo, consentendo di ingrandire o rimpicciolire quel punto. Utilizzate i tasti modificatori x, y o CTRL per limitare lo zoom rispettivamente alla conservazione delle proporzioni x, y o delle proporzioni.
- Per attivare la modalità Zoom su rettangolo, fare clic sul pulsante Zoom su rettangolo . Posizionare il cursore sull'immagine e premere il pulsante sinistro del mouse. Trascinare il mouse in una nuova posizione tenendo premuto il pulsante per definire un'area rettangolare.
NOTA: i limiti di visualizzazione degli assi verranno ingranditi nell'area definita quando si preme il pulsante sinistro del mouse. I limiti di visualizzazione degli assi verranno ridotti quando si preme il pulsante destro del mouse, posizionando gli assi originali nell'area definita. - Utilizzare lo strumento di configurazione del sottotrame per configurare l'aspetto della sottotrama.
NOTA: i lati sinistro, destro, superiore e inferiore della sottotrama, nonché lo spazio tra righe e colonne, possono essere allungati o compressi. - Per aprire una finestra di dialogo di salvataggio file, fare clic sul pulsante Salva e salvare il file nei seguenti formati: .png, ps, .eps, .svg o .pdf.
Access restricted. Please log in or start a trial to view this content.
Risultati
Un'analisi rappresentativa è stata eseguita su immagini di reti dendritiche in coltura. Le cellule sono state estratte come descritto da Baranes et al. 16,17. In breve, le cellule dell'ippocampo sono state estratte dal cervello dei ratti postnatali e coltivate su coperture di vetro 2D per 1-2 settimane. Le colture sono state poi fissate e colorate attraverso immunofluorescenza indiretta utilizzando un anticorpo contro il marcatore proteico dendritico, l...
Access restricted. Please log in or start a trial to view this content.
Discussione
Sono urgentemente necessarie strategie efficaci per estrarre informazioni morfologiche da immagini 2D per tenere il passo con i dati di imaging biologico. Sebbene i dati di imaging possano essere generati in poche ore, l'analisi approfondita delle immagini richiede molto tempo. Di conseguenza, l'elaborazione delle immagini è chiaramente diventata un grosso ostacolo in molti campi. Ciò è dovuto in parte all'elevata complessità dei dati, soprattutto quando si tratta di campioni biologici. Inoltre, poiché molti utenti ...
Access restricted. Please log in or start a trial to view this content.
Divulgazioni
Gli autori dichiarano di non avere interessi finanziari concorrenti.
Riconoscimenti
Gli autori desiderano ringraziare il Dr. Orly Weiss per la preparazione delle immagini della cultura.
Access restricted. Please log in or start a trial to view this content.
Materiali
| Name | Company | Catalog Number | Comments |
| Matplotlib | 2002 - 2012 John Hunter, Darren Dale, Eric Firing, Michael Droettboom and the Matplotlib development team; 2012 - 2021 The Matplotlib development team. | 3.4.2 | a Python 2D plotting library |
| matplotlib-scalebar | Philippe Pinard | 0.7.2 | artist for matplotlib to display a scale bar |
| NumPy | The NumPy community. | 1.20.3 | fundamental package for scientific computing library |
| OpenCV | OpenCV team | 4.5.2.54 | Open Source Computer Vision Library |
| PyCharm | JetBrains | 2020.3.1 (Community Edition) version | Build #PC-203.6682.86, built on December 18, 2020. Runtime version: 11.0.9.1+11-b1145.37 amd64. VM: OpenJDK 64-Bit Server VM by JetBrains s.r.o. Windows 10 10.0. Memory: 978M, Cores: 4 |
| PyQt5 | Riverbank Computing | 5.15.4 | manage the GUI |
| python | Python Software Foundation License | 3.9 version | |
| Qt Designer | The QT Company Ltd. | 5.11.1 version | |
| scipy | Community library project | 1.6.3 | Python-based ecosystem of open-source software for mathematics, science, and engineering |
| Seaborn | Michael Waskom. | 0.11.1 | Python's Statistical Data Visualization Library. |
| Windows 10 | Microsoft | ||
| Xlsxwriter | John McNamara | 1.4.3 | Python module for creating Excel XLSX files |
Riferimenti
- Ferrante, M., Migliore, M., Ascoli, G. Functional impact of dendritic branch-point morphology. Journal of Neuroscience. 33 (5), 2156-2165 (2013).
- Spruston, N. Pyramidal neurons: dendritic structure and synaptic integration. Nature Reviews Neuroscience. 9 (3), 206-221 (2008).
- Chklovskii, D. Synaptic Connectivity and Neuronal Morphology: Two Sides of the Same Coin. Neuron. 43 (5), 609-617 (2004).
- Chapleau, C., Larimore, J., Theibert, A., Pozzo-Miller, L. Modulation of dendritic spine development and plasticity by BDNF and vesicular trafficking: fundamental roles in neurodevelopmental disorders associated with mental retardation and autism. Journal of Neurodevelopmental Disorders. 1 (3), 185-196 (2009).
- Irwin, S. Dendritic Spine Structural Anomalies in Fragile-X Mental Retardation Syndrome. Cerebral Cortex. 10 (10), 1038-1044 (2000).
- Kaufmann, W. Dendritic anomalies in disorders associated with mental retardation. Cerebral Cortex. 10 (10), 981-991 (2000).
- Pinchas, M., Baranes, D. Dendritic branch intersections are structurally regulated targets for efficient axonal wiring and synaptic clustering. PLoS ONE. 8 (12), 82083(2013).
- Cove, J., Blinder, P., Baranes, D. Contacts among non-sister dendritic branches at bifurcations shape neighboring dendritic branches and pattern their synaptic inputs. Brain Research. 1251, 30-41 (2009).
- Blinder, P., Cove, J., Foox, M., Baranes, D. Convergence among non-sister dendritic branches: An activity-controlled mean to strengthen network connectivity. PLoS ONE. 3 (11), 3782(2008).
- Glaser, J., Glaser, E. Neuron imaging with neurolucida - PC-based system for image combining microscopy. Computerized Medical Imaging and Graphics. 14 (5), 307-317 (1990).
- Scorcioni, R., Polavaram, S., Ascoli, G. L-Measure: a web-accessible tool for the analysis, comparison and search of digital reconstructions of neuronal morphologies. Nature Protocols. 3 (5), 866-876 (2008).
- Torben-Nielsen, B. An efficient and extendable python library to analyze neuronal morphologies. Neuroinformatics. 12 (4), 619-622 (2014).
- Parekh, R., Ascoli, G. Neuronal morphology goes digital: A research hub for cellular and system neuroscience. Neuron. 78 (1), 206(2013).
- heng, J., Zhou, X., Sabatini, B. L., Wong, S. T. C. NeuronIQ: A novel computational approach for automatic dendrite SPINES detection and analysis. 2007 IEEE/NIH Life Science Systems and Applications Workshop. , 168-171 (2007).
- Image processing and analysis in Java. NIH. ImageJ. , Available from: https://imagej.nih.gov/ij (2021).
- Peretz, H., Talpalar, A. E., Vago, R., Baranes, D. Superior survival and durability of neurons and astrocytes on 3-dimensional aragonite biomatrices. Tissue Engineering. 13, 461-472 (2007).
- Morad, T. I., Hendler, R. M., Weiss, O. E., Canji, E. A., Merfeld, I., Dubinsky, Z., Minnes, R., Francis, Y. I., Baranes, D. Gliosis of astrocytes cultivated on coral skeleton is regulated by the matrix surface topography. Biomedical Materials. 14 (4), 045005(2019).
Access restricted. Please log in or start a trial to view this content.
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon