Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Microscopía de fluorescencia para internalización de ATP mediada por macropinocitosis en células tumorales humanas y ratones xenoinjertados con tumores
* Estos autores han contribuido por igual
En este artículo
Resumen
Desarrollamos un método reproducible para visualizar la internalización del trifosfato de adenosina fluorescente no hidrolizable (ATP), un sustituto de ATP, con alta resolución celular. Validamos nuestro método utilizando ensayos independientes in vitro e in vivo: líneas celulares tumorales humanas y ratones inmunodeficientes xenoinjertados con tejido tumoral humano.
Resumen
Se ha demostrado que el trifosfato de adenosina (ATP), incluido el ATP extracelular (eATP), desempeña un papel importante en varios aspectos de la tumorigénesis, como la resistencia a los medicamentos, la transición epitelial-mesenquimal (EMT) y la metástasis. La eATP intratumoral es de 10a 3 a 104 veces mayor en concentración que en los tejidos normales. Si bien eATP funciona como un mensajero para activar la señalización purinérgica para la inducción de EMT, también es internalizado por las células cancerosas a través de la macropinocitosis regulada al alza, un tipo específico de endocitosis, para realizar una amplia variedad de funciones biológicas. Estas funciones incluyen proporcionar energía a las reacciones bioquímicas que requieren ATP, donar grupos fosfato durante la transducción de señales y facilitar o acelerar la expresión génica como cofactor transcripcional. El ATP está fácilmente disponible, y su estudio en el cáncer y otros campos sin duda aumentará. Sin embargo, el estudio eATP se encuentra en una etapa temprana, y las preguntas no resueltas siguen sin respuesta antes de que las actividades importantes y versátiles desempeñadas por eATP y ATP intracelular internalizado puedan desentrañarse por completo.
Las contribuciones de los laboratorios de estos autores a estos primeros estudios de eATP incluyen imágenes microscópicas de ATP fluorescente no hidrolizable, junto con dextrans fluorescentes de alto y bajo peso molecular, que sirven como trazadores de macropinocitosis y endocitosis, así como varios inhibidores de endocitosis, para monitorear y caracterizar el proceso de internalización de eATP. Esta modalidad de imagen se aplicó a líneas celulares tumorales y a ratones inmunodeficientes, xenoinjertados con tumores de cáncer humano, para estudiar la internalización de eATP in vitro e in vivo. Este artículo describe estos protocolos in vitro e in vivo, con énfasis en la modificación y el ajuste fino de las condiciones del ensayo para que los ensayos de internalización eATP mediados por macropinocitosis / endocitosis se puedan realizar con éxito en diferentes sistemas.
Introducción
La absorción oportunista de nutrientes extracelulares intratumorales (es decir) ha sido nombrada recientemente un sello distintivo clave para el metabolismo del cáncer1. Uno de estos nutrientes importantes es el ATP, ya que la concentración de ieATP es 103 y 104 veces mayor que la que se encuentra en los tejidos normales, en el rango de varios cientos de μM a mM bajo2,3,4,5. Como molécula clave de energía y señalización, el ATP juega un papel central en el metabolismo celular en células cancerosas y sanas6,7,8. El ATP extracelular no solo está involucrado en el crecimiento de células cancerosas, sino que también promueve la resistencia a los medicamentos9. Recientemente se han identificado funciones previamente no reconocidas del ATP, como la actividad hidrotrópica, lo que implica la participación del ATP en enfermedades como el Alzheimer10. De hecho, parece que nuestra comprensión del ATP y sus funciones en las células cancerosas, las células sanas y otras células enfermas está lejos de ser completa. Sin embargo, debido a la inestabilidad del ATP y las altas tasas de renovación en las células, es técnicamente difícil monitorear el movimiento del ATP a través de la membrana celular y hacia la célula.
Para abordar este problema y satisfacer la necesidad de esta área de investigación, se desarrolló un método en el que se utilizó ATP fluorescente no hidrolizable (NHF-ATP)(Figura 1)como sustituto para visualizar la internalización del ATP y observar la localización espacial intracelular del ATP internalizado, tanto in vitro como in vivo11,12 . Se ha demostrado que el NHF-ATP sustituye al ATP endógeno para investigar el movimiento del ATP a través de las membranas celulares animales, tanto en líneas celulares cancerosas como en tejido tumoral humano xenoinjertado en ratones inmunodeficientes11,12. Además, la administración de inhibidores de la macropinocitosis a las células bloqueó la internalización de eATP, lo que sugiere que la absorción intracelular de eATP implica un mecanismo macropinocitótico9,11,12. Este protocolo permite el colabeling de base inmunocomprimida contra proteínas específicas de la célula y, por lo tanto, la identificación de qué tipo de célula internaliza el NHF-ATP. Utilizando xenoinjertos tumorales in vivo y microscopía de alta resolución, el NHF-ATP se puede visualizar espacialmente a través de la muestra de tejido e incluso dentro de una sola célula. Estos métodos también permiten el análisis cuantitativo, como el porcentaje de absorción celular, el número de vesículas macropinocitóticas y la cinética de internalización. Este artículo describe en detalle cómo el NHF-ATP, trabajando solo o junto con endocitosis-trazador fluorescente dextrans13,14,15,16, se puede utilizar en diferentes entornos experimentales para estudiar la internalización y localización intracelular de ATP, después de la internalización en las células.

Figura 1: Estructuras de ATP fluorescente no hidrolizable y tetrametilrodamina marcadas con dextrano fluorescente de alto peso molecular. (A) Estructura de NHF-ATP. (B) Representación esquemática de HMWFD. Abreviaturas: ATP = trifosfato de adenosina; NHF-ATP = ATP fluorescente no hidrolizable; TMR = tetrametilrodamina; HMWFD = dextrano fluorescente de alto peso molecular. Haga clic aquí para ver una versión más grande de esta figura.
Access restricted. Please log in or start a trial to view this content.
Protocolo
Todos los procedimientos reportados en este documento se realizaron de acuerdo con el IACUC de la Universidad de Ohio y con el NIH.
1. Selección de ATP fluorescente no hidrolizable (NHF-ATP) y dextrans
- Seleccionar un NHF-ATP conjugado con fluoróforos(Figura 1A)y trazadores de endocitosis, dextrans fluorescentes de alto y bajo peso molecular (TMR-HMWFD y TMR-LMWFD)(Figura 1B),en función de las longitudes de onda de emisión preferidas (por ejemplo, sistema de imágenes equipado con filtros apropiados) y el proceso de endocitosis específico a estudiar.
2. Estudios de localización de ATP, in vitro (Figura 2)
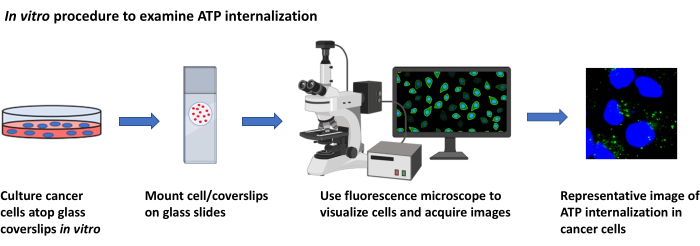
Figura 2: Procedimiento in vitro para examinar la internalización del ATP. Representación esquemática del protocolo para visualizar la internalización de ATP extracelular en células cancerosas cultivadas mediante microscopía de fluorescencia. Haga clic aquí para ver una versión más grande de esta figura.
- Cultivo celular y preparación de células
NOTA: Realizar cultivo celular en condiciones estériles en una campana de cultivo de tejidos.- Prepare el Medio Águila Modificada (DMEM) de Dulbecco, que contiene 10% (v/v) suero fetal bovino (FBS) y 1% (v/v) penicilina/estreptomicina (en adelante, DMEM/FBS), solución salina estéril tamponada con fosfato (PBS) y 0,25% de tripsina/ácido etilendiaminatetraacético (EDTA), en un baño de agua a 37 °C.
- Cultive células cancerosas humanas en DMEM/FBS en una placa de cultivo de tejidos de 100 mm. Mantenga las células en una incubadora a 37 °C con una atmósfera de 5% de CO2.
- Cuando las células alcanzan la confluencia, pase las células eliminando primero el medio de cultivo. A continuación, enjuague el plato con 5 ml de PBS estéril, retire el PBS y agregue 3 ml de tripsina al 0.25%. Incubar a 37 °C en una atmósfera de CO2 al 5% durante 5 min.
- Recupere el plato, luego agregue 6 ml de DMEM / FBS para detener la tripsinización. Transfiera las células en suspensión a un tubo cónico de 15 ml y centrífuga a 800 × g durante 5 minutos para granular las células.
- Después de la centrifugación, aspire el sobrenadante y use 10 ml de DMEM / FBS para resuspendir el pellet celular mediante pipeteo.
- Cuente la densidad celular y la viabilidad utilizando un hemocitómetro. Utilice DMEM/FBS para diluir la suspensión celular a una densidad de ~7.5 × 104 células/ml.
- Preparación de fundas y células de siembra
- Lave las fundas de 12 mm con etanol al 70% y límpielas cuidadosamente con toallitas delicadas. Esterilizar las fundas y un par de fórceps mediante autoclave.
- En una campana de cultivo de tejidos, use fórceps para colocar un cubrehojas en cada pocillo de una placa de cultivo de tejidos de 24 pocillos.
NOTA: Más tarde, el coverslip, con celdas, se montará directamente en un portaobjetos de microscopio para obtener imágenes. - Dispensar 300 μL de la suspensión celular (células en DMEM/FBS), a una densidad de siembra de ~2,5 × 104 células por pozo, en la placa de 24 pocillos que contiene las fundas esterilizadas. Incubar en condiciones estériles a 37 °C con un flujo de CO2 del 5%.
- Inanición de células
- Veinticuatro horas después de la siembra, retire el DMEM / FBS de cada pozo. Agregue inmediatamente 300 μL de DMEM sin suero precalentado en cada pocillo para matar de hambre de suero a las células durante 15-18 h para inducir la absorción de nutrientes extracelulares.
NOTA: El período de inanición de 15-18 h es un parámetro crítico.
- Veinticuatro horas después de la siembra, retire el DMEM / FBS de cada pozo. Agregue inmediatamente 300 μL de DMEM sin suero precalentado en cada pocillo para matar de hambre de suero a las células durante 15-18 h para inducir la absorción de nutrientes extracelulares.
- Preparación de soluciones NHF-ATP y HMWFD/LMWFD
- Utilice una balanza analítica para pesar tmR-dextrano fluorescente de alto peso molecular (70 kDa) (TMR-HMWFD, 1 mg/mL), un trazador para visualizar macropinosomas, o NHF-ATP (10 μmol/L) en DMEM sin suero en un tubo de microcentrífuga de 1,5 ml. Coloque los tubos, protegidos de la luz, en un baño de agua a 37 °C durante 15 min.
- Centrifugadora a 12.000 × g durante 5 min a temperatura ambiente. Transfiera cuidadosamente el sobrenadante transparente a un nuevo tubo de microcentrífuga de 1,5 ml, dejando intacto cualquier gránulo o escombros para eliminar los cristales indisolubles.
- Agregue las soluciones del paso 2.4.1 a las células de cada pozo e incube las células durante 30 min a 37 °C.
NOTA: Si las soluciones HMWFD y NHF-ATP se van a mezclar para la coincubación con las células, prepare ambas soluciones a 2 veces las concentraciones finales. Las soluciones se mezclarán más tarde en una proporción de 1: 1 para lograr las concentraciones de trabajo precisas finales. Evite la luz ya que los reactivos son sensibles a la luz.
- Tratamiento de las células y fijación
- En una placa fresca de 24 pocillos, dispense 500 μL de PBS precalentado en cada uno de los cinco pozos.
- Después de la incubación celular, recoja cuidadosamente cada funda con fórceps. Enjuague cada funda sumergiéndola en 500 μL de PBS precalentado. Repita cinco veces usando los cinco pozos llenos de PBS.
NOTA: El lavado suave de las células en los labios es fundamental para el éxito de este experimento. - Después del lavado final de PBS, toque el cubrecolchas en una delicada toallita de tarea para absorber PBS adicional y transfiera el cubrecolchas inmediatamente a formaldehído frío (4 ° C) al 3.7%, precargado en una placa de 24 pocillos. Fije las celdas durante 15 minutos a temperatura ambiente.
- Mientras se fijan las células, el microscopio se desliza previamente con 70% de etanol. Retire los cubrehojas de los pocillos y móntelos en los portaobjetos, utilizando 5 μL de medio de montaje acuoso que contenga la tinción nuclear 4′,6-diamidino-2-fenilindol (DAPI), por funda. Seque suavemente el exceso de PBS con una toalla de papel o una toallita delicada para tareas.
- Microscopía de fluorescencia y adquisición de imágenes
- De dos a 24 horas después de los pasos anteriores, capture imágenes de células e interiorice HMWFD y / o NHF-ATP utilizando un sistema de imágenes de epifluorescencia y un software de adquisición de datos.
NOTA: En esta subsección se describen los pasos para adquirir imágenes utilizando un microscopio Nikon NiU, equipado con capacidad de imagen de epifluorescencia, y el software Nikon NIS Elements. Sin embargo, se pueden utilizar otros sistemas de imágenes y software de adquisición comparables. Siga las instrucciones de funcionamiento del fabricante.- Coloque el portaobjetos en el escenario de un microscopio de epifluorescencia vertical en modo binocular. Acceda al programa de imágenes.
- Seleccione el objetivo 10x, ajuste el escenario para definir el enfoque y escanee la diapositiva de izquierda a derecha de manera serpenteante para identificar las regiones de interés.
NOTA: La identificación de las regiones de interés variará entre los tipos de células, con algunas líneas celulares / tipos de cáncer que exhiben diversos y distintos grados de absorción de TMR-HMWFD y / o NHF-ATP. - Seleccione el objetivo 40x y cambie del modo binocular al modo de captura de imágenes, utilizando el interruptor del microscopio.
- Haga clic en el icono Calidad en vivo en el programa de imágenes para ver y posteriormente adquirir imágenes.
- Con el panel OC de la barra de herramientas Anotaciones y mediciones, defina los parámetros de exposición para cada cubo de filtro o canal fluorescente.
NOTA: Seleccione el tiempo de exposición adecuado para cada canal, ya que las intensidades de la señal son diferentes. Por ejemplo, seleccione un tiempo de exposición de 200 ms para DAPI, 2 s para HMWFD y 4 s para NHF-ATP. Una vez que se determina el tiempo de exposición por canal, use esta configuración para todas las imágenes, por canal, con diferentes tratamientos o condiciones. - Una vez que se hayan establecido los ajustes de exposición para cada canal, utilice la barra de herramientas de adquisición multicanal para adquirir una imagen de 3 canales con los ajustes de exposición definidos.
NOTA: La adquisición de imágenes a través del modo de adquisición ND multicanal permite la captura automática de imágenes para cada canal del mismo campo de visión. El obturador se cierra automáticamente entre los cambios de torreta. - Alternativamente, adquiera imágenes multicanal manualmente alternando entre cubos de filtro, configurando el tiempo de exposición, cerrando / abriendo el obturador entre la adquisición de imágenes para cada canal y superponiendo cada imagen tomada para canales individuales.
NOTA: El modo de adquisición ND automatiza este proceso y proporciona imágenes combinadas. - Guarde la imagen como archivo .nd2 (el formato Nikon Elements guarda metadatos). Guarde los archivos TIF, incluida la imagen de canal combinada y las imágenes de canal individuales.
NOTA: Los archivos TIF se pueden utilizar con una selección más amplia de aplicaciones de software. - Utilice la función Recuento de objetos de la barra de herramientas Análisis para contar el número de celdas NHF-ATP-, TMR-HMWFD-, y/o TMR-LMWFD-positivas en un archivo de imagen .nd2 guardado.
- Exporte los datos a una hoja de cálculo a través del programa de análisis.
- De dos a 24 horas después de los pasos anteriores, capture imágenes de células e interiorice HMWFD y / o NHF-ATP utilizando un sistema de imágenes de epifluorescencia y un software de adquisición de datos.
- Cuantificación y análisis de datos
- Para cada condición ensayada, imagen de 50 a 100 células para la cuantificación. Utilizando el software de análisis de datos (software incluido en el sistema de imágenes de epifluorescencia u otro software), cuente y calcule el número medio de vesículas fluorescentes por célula.
- Utilizar métodos estadísticos apropiados para analizar los resultados cuantificados.
3. Internalización de ATP en tumores, ex vivo (Figura 3)

Figura 3: Procedimiento in vivo para examinar la internalización del ATP. Representación esquemática del protocolo para visualizar la internalización de ATP extracelular en xenoinjertos tumorales mediante criosecciona y microscopía de fluorescencia. Haga clic aquí para ver una versión más grande de esta figura.
- Preparación de cultivos celulares para implantación
- Cultivar células cancerosas al 80% de confluencia a 37 °C en un matrazde 225 cm 2, utilizando DMEM suplementado con FBS, a una concentración final de 10% (v/v) y penicilina/estreptomicina al 1% (v/v).
- Lave las células dos veces con 10 ml de PBS. Pre-caliente 0.25% tripsina/EDTA a 37 °C. Añadir 8 ml de tripsina/EDTA e incubar a 37 °C durante 2 min.
- Una vez que las células comiencen a desprenderse de la parte inferior del matraz, use una pipeta serológica estéril de 10 ml para agregar 8 ml de DMEM / FBS. Aspirar dos veces para desalojar cualquier célula adherente. Utilice la pipeta para transferir las células desprendidas del matraz a un tubo cónico de 50 ml.
- Agregue 10 ml de DMEM/FBS utilizando una pipeta de 10 ml y recoja todas las celdas flotantes restantes en el mismo tubo cónico de 50 ml.
- Centrifugar la suspensión celular a 600 × g,4 °C durante 4 min. Retire el sobrenadante y vuelva a suspender las células en 1 ml de PBS helado.
- Cuente la densidad celular usando un hemocitómetro. Mantenga la suspensión celular en hielo mientras cuenta.
- Centrifugar la suspensión celular a 600 × g,4 °C durante 4 min. Retire el sobrenadante y suspenda las células en PBS helado de tal manera que la densidad celular se convierta en 5 × 106 células por 100 μL de PBS. Transfiera la suspensión celular a un tubo de microcentrífuga de 1,5 ml.
- Inyección subcutánea de células cancerosas para el desarrollo de tumores de xenoinjerto
- Use una jeringa sin látex (1 ml) con una aguja deslizante de precisión (aguja de 27 G) para la inyección de células cancerosas.
- Transfiera la suspensión celular (5 × 106 en 100 μL de PBS) a un tubo de microcentrífuga de 1,5 ml. Extraiga las células en la jeringa.
- Seleccione un sitio de inyección en el flanco de un ratón inmunodeficiente (desnudo) y limpie suavemente la piel con etanol al 75%. Limpie el exceso de etanol con una delicada limpieza de tareas.
- Para la inyección subcutánea, sostenga la aguja en un ángulo de aproximadamente 10 ° con respecto a la piel. Inserte la punta de la aguja, con el bisel hacia arriba, justo debajo de la piel, de modo que solo 1-2 mm de la aguja sea visible fuera de la piel. Dispense las células de la jeringa lentamente durante aproximadamente 10 s.
- Después de inyectar todo el volumen, continúe manteniendo la aguja en su lugar durante 3-5 s, luego retire la aguja y use un dedo para aplicar una presión suave pero firme en el sitio de inyección durante 3-5 segundos más para evitar fugas del contenido inyectado.
- Monitoree y mida el crecimiento tumoral usando pinzas vernier hasta que los tumores alcancen un volumen de 200-500 mm3.
- Preparación de soluciones HMWFD y NHF-ATP para ser utilizadas después de la resección tumoral
- Disolver 300 μL de 16 mg/ml de HMWFD en DMEM (medio de cultivo) sin suero, incubar en un baño de agua a 37 °C durante 30 min y centrifugar a 12.000 × g durante 5 min como se describió anteriormente. Transfiera la solución a un tubo de microcentrífuga de 1,5 ml.
- Añadir 40 μL de material análogo de NHF-ATP (1 mM) a 160 μL de DMEM sin suero para preparar una solución de NHF-ATP de 0,2 mM.
- Preparación de pozos experimentales
NOTA: Este diseño experimental ensayará la internalización intracelular de HMWFD + NHF-ATP, indicativa de captación por macropinosomas.- Preparar los pocillos de la siguiente manera: Pozo #1, Control: 200 μL de DMEM sin suero; Pozo #2, Control: 100 μL de 16 mg/mL LMWFD + 100 μL de DMEM libre de suero = 200 μL de 8 mg/mL LMWFD; Pozo #3, Control: 100 μL de 0.2 mM NHF-ATP + 100 μL de DMEM libre de suero = 200 μL de 0.1 mM NHF-ATP; Bueno #4; Experimental: 100 μL de 16 mg/mL HMWFD + 100 μL de 0,2 mM NHF-ATP = 200 μL de 0,1 mM NHF-ATP y 8 mg/mL HMWFD.
- Preparación de tejidos tumorales
- Eutanasiar al ratón por luxación cervical o según el protocolo aprobado por la IACUC.
- Use un bisturí de tamaño 10 para cortar los tumores aislados a un grosor de ~ 500-1,000 μm.
- Incubar las rebanadas tumorales en DMEM sin suero suplementado con 100 μM NHF-ATP y/o 8 mg/mL H/LMWFD en tubos de microcentrífuga durante 40 min a 37 °C con un flujo de CO2 al 5%.
NOTA: Después de la incubación, el metabolismo del tejido tumoral hace que el color del medio cambie. - Enjuague los tejidos en PBS precalentado a 37 °C (2 ml por cada enjuague en una placa de 24 pocillos).
- Transfiera el tejido a una nueva placa de 24 pocillos con PBS fresco precalentado, enjuague y repita cuatro veces con un suave agitación.
- Crio-incrustación (preparación de bloques de tejido congelado)
- Prepare las etiquetas de identificación para cada tumor que se va a cosechar. Cortar un trozo de cinta de laboratorio de 2 cm y doblar por la mitad, los lados adhesivos juntos, a lo largo. Use un rotulador para etiquetar la etiqueta, por ejemplo, con un número de identificación de ratón/tumor.
- Prepare moldes de incrustación colocando moldes de tejido de acero inoxidable directamente sobre hielo seco.
NOTA: El hielo seco puede causar congelación, quemaduras y asfixia. Use guantes aislados cuando manipule hielo seco. Use hielo seco en un área bien ventilada. No almacene hielo seco en un recipiente herméticamente sellado. En su lugar, guárdelo en un recipiente (como un enfriador de espuma de poliestireno) que permita que el gas escape. - Mientras el moho se enfría, coloque un pequeño charco de medio de congelación de tejido en una placa de cultivo de tejido de 10 mm. Asegúrese de que el volumen sea suficiente para sumergir el tejido tumoral que se va a cosechar.
- Use una cuchara perforada para recoger el tejido tumoral resecado y coloque inmediatamente el tejido en un medio de congelación, asegurándose de que el tejido esté sumergido. Usando la cuchara perforada, enrolle suavemente el tejido en el medio de congelación, asegurándose de que el medio esté bañando todas las superficies del tejido.
- Mueva cuidadosamente el tejido en el molde de incrustación que contiene el medio de congelación. Coloque la etiqueta de etiqueta correspondiente verticalmente en el medio de congelación / molde para congelar en su lugar. Asegúrese de que la etiqueta escrita sea visible fuera del medio.
- Cuando se complete la congelación (el medio de congelación se vuelve blanco opaco), retire el bloque de tejido del molde, colóquelo sobre hielo seco y repita para cada tumor. Guarde los bloques de tejido a -80 °C durante varios meses antes del procedimiento de crioseccionación.
- Preparación de portaobjetos de muestras de tejidos
- Para maximizar la posibilidad de encontrar células positivas para la internalización y tener regiones tisulares más representativas, recolecte criosecciones en serie a -18 a -20 ° C usando un criostato.
- Prechill herramientas de criostato (cuchilla, hoja de afeitar, placa antivuelco, soporte de mandril de tejido, pincel) y equilibran los bloques de tejido tumoral colocándolos en una cámara de criostato a -18 a -20 ° C. Ajuste el ángulo del soporte de la cuchilla a 5-10 °. Recorte cuidadosamente el bloque de tejido, según sea necesario, con una cuchilla de afeitar y móntelo en el soporte del mandril utilizando un medio de congelación de tejido como "pegamento".
- Bloquee el soporte del mandril en la posición vertical de la unidad de microtomo, que avanza a la distancia establecida (por ejemplo, 10 μm) con cada giro de la manivela. Coloque la placa estabilizadora para que descanse justo por encima de la altura de la cuchilla. Para evitar que el tejido se enrosque antes de avanzar el microtomo, deslice cuidadosamente el pulgar sobre el borde inferior del bloque de tejido.
- A medida que el microtomo avanza y la sección de tejido cae sobre la placa de metal, use un pincel para guiar la sección de tejido y desenrolle el tejido, si es necesario.
- Coloque el portaobjetos del microscopio sobre la sección de tejido sin tocarlo para que la sección se sienta atraída por el portaobjetos.
NOTA: Las cuchillas de criostato (de alto perfil, desechables) son extremadamente afiladas y pueden causar lesiones graves. Tenga cuidado al manipular cuchillas y operar el criostato. Use un protector de cuchillas, si está disponible. Se requiere una capacitación adecuada. - Cortar el tumor en secciones de 10 μm de grosor. Transfiera inmediatamente las secciones cortadas a un portaobjetos de microscopio de vidrio cargado positivamente.
NOTA: Para las secciones en serie, primero recoja una sección de 10 μm de espesor en la esquina superior izquierda de cada una de las ocho diapositivas cargadas positivamente. Avance el criostato a través de los siguientes 100-200 μm de tejido y deseche el tejido. Transfiera inmediatamente todas las secciones cortadas a portaobjetos de microscopio de vidrio. - A continuación, recoja otra sección de 10 μm de espesor, junto a la sección de tejido colocada previamente, para cada una de las ocho diapositivas. Repita este proceso de recolección en serie hasta que cada una de las ocho diapositivas contenga ocho secciones de tejido, cada una separada por 100-200 μm. Mantenga las secciones de tejido en la oscuridad para preservar la fluorescencia.
NOTA: Las secciones de tejido en las diapositivas se pueden almacenar en una caja de diapositivas a -80 ° C durante varios meses.
- Para maximizar la posibilidad de encontrar células positivas para la internalización y tener regiones tisulares más representativas, recolecte criosecciones en serie a -18 a -20 ° C usando un criostato.
- Fijación de portaobjetos de tejido
- PASO CRÍTICO: Fijar las secciones de tejido en etanol al 95% a -18 a -20 °C durante 5 min.
- Lave la sección fija durante 5 minutos con PBS a temperatura ambiente y luego monte las secciones tumorales fijas debajo de una cubierta de vidrio utilizando 10 μL de un medio de montaje acuoso con DAPI.
- De doce a 24 h después del montaje, examine las secciones tumorales fijas mediante microscopía de fluorescencia y adquiera imágenes, como se describe para las células cultivadas anteriormente.
- Microscopía de fluorescencia y adquisición de imágenes
- Identifique las regiones de interés y adquiera imágenes, como se describe en la sección 2.6.
- Cuantificación y análisis de datos
- Cuantificar las células y aplicar los análisis estadísticos adecuados, como en la sección 2.7.
4. Internalización de ATP en tumores, in vivo
- Preparar cultivos celulares para la implantación como se describe en la sección 3.1.
- Inyección subcutánea de células cancerosas para el desarrollo de tumores de xenoinjerto
- Generar tumores xenoinjertados como se describe en la sección 3.2.
- Inyección de ATP y/o dextrano en tumores de xenoinjerto
- Preparar las soluciones de tratamiento de DMEM (vehículo) o 8 mg/ml HMWFD o LMWFD, con o sin NHF-ATP (100 μM) en DMEM, como se describió anteriormente.
- Use una jeringa de 1 ml para recolectar 50 μL de una solución de tratamiento e inyecte la solución directamente en cada tumor de xenoinjerto. Repita el procedimiento para cuatro réplicas biológicas de cada tratamiento.
- Recolección de tejidos y crioincrustación
- Prepare las etiquetas de identificación para cada tumor que se va a cosechar. Cortar un trozo de cinta de laboratorio de 2 cm y doblar por la mitad, los lados adhesivos juntos, a lo largo. Use un rotulador para etiquetar la etiqueta, por ejemplo, con un número de identificación de ratón/tumor.
- Aproximadamente 5 minutos después de la inyección, sacrificar al ratón por luxación cervical o de acuerdo con el protocolo aprobado por la IACUC.
- Usando un bisturí de tamaño 10, haga una incisión adyacente al tumor y aproximadamente perpendicular a la dirección de la inyección con aguja. Use fórceps y tijeras quirúrgicas para resecar el tejido tumoral del tejido circundante.
- Divida el tumor en dos a cuatro piezas de 1 cm2, dependiendo del tamaño total del tumor.
- Preparar los moldes de incrustación e incrustar el tejido, tal y como se describe anteriormente en la sección 3.6. Asegúrese de que el tiempo de cosecha, desde la inyección intratumoral de dextrano hasta la crioincrustación, no sea más de 7-8 min.
- Preparación de portaobjetos de muestras de tejidos
- Recoger las secciones de tumor en serie, como se describe en la sección 3.7.
- Fijación de portaobjetos de tejido
- Fijar el tejido, como se describe en la sección 3.8.
- Microscopía de fluorescencia y adquisición de imágenes
- Identifique las regiones de interés y adquiera imágenes, como se describe en la sección 2.6.
- Cuantificación y análisis de datos
- Cuantificar las células y aplicar los análisis estadísticos adecuados, tal como se describe en la sección 2.7.
Access restricted. Please log in or start a trial to view this content.
Resultados
Estudio in vitro
La internalización intracelular de NHF-ATP se demostró mediante la co-localización de NHF-ATP con HMWFD o LMWFD (Figura 4). El éxito de este procedimiento se basa principalmente en el uso de concentraciones apropiadas de NHF-ATP y dextrans y en la determinación del tipo o tipos apropiados de dextrans (poli-lisina vs. neutro). Por ejemplo, para investigar la macropinocitosis, se eligió HMWFD ya que está internalizado solo...
Access restricted. Please log in or start a trial to view this content.
Discusión
Se desarrolló un método para el análisis espacial, temporal y cuantitativo de la internalización celular de ATP no hidrolizable. Este método es ampliamente aplicable para su uso en diversos sistemas biológicos, incluidos varios modelos tumorigénicos, para los cuales proporcionamos instrucción técnica y datos representativos. Para adquirir datos interpretables durante los estudios de internalización de ATP in vivo (sección 4 del protocolo), es fundamental limitar el tiempo experimental transcurrido des...
Access restricted. Please log in or start a trial to view this content.
Divulgaciones
Los autores no declaran intereses contrapuestos.
Agradecimientos
La crioseccionación se realizó in situ en el Núcleo de Histopatología de la Universidad de Ohio. Este trabajo fue apoyado en parte por fondos de puesta en marcha (Ohio University College of Arts & Sciences) a C Nielsen; Concesión de los NIH R15 CA242177-01 y concesión de RSAC a X Chen.
Access restricted. Please log in or start a trial to view this content.
Materiales
| Name | Company | Catalog Number | Comments |
| A549 cells, human lung epithelial, carcinoma | National Cancer Institute | n/a | Less expensive source |
| Acetone | Fisher Scientific | S25904 | |
| Aluminum foil, Reynolds | Grainger | 6CHG6 | |
| Aqueous Mounting Medium, ProLong Gold Anti-fade Reagent | ThermoFisher | P36930 | |
| ATP analog | Jena Biosciences | NK-101 | |
| Autoclave, sterilizer | Grainger | 33ZZ40 | |
| Blades, cryostat, high profile | C. L. Sturkey, Inc. | DT554550 | |
| Calipers, vernier | Grainger | 4KU77 | |
| Cell medium, Ham's Nutrient Mixture F12, serum-free | Millipore Sigma | 51651C-1000ML | |
| Centrifuge, refrigerated with swinging bucket rotor | Eppendorf | 5810R | |
| Chloroform | Acros Organics | 423555000 | |
| Conical tube, 15 mL | VWR | 21008-216 | |
| Conical tube, 50 mL | VWR | 21008-242 | |
| Coverslips, glass, 12 mm | Corning | 2975-245 | |
| Cryostat, Leica CM1950 | Leica Biosystems | CM1950 | |
| Delicate task wipe, Kim Wipes | Kimberly-Clark | 34155 | |
| Dextran, Lysine fixable, High Molecular Weight (HMW) | Invitrogen | D1818 | MW = 70,000, Tetramethylrhodamine |
| Dextran, Neutral, High Molecular Weight (HMW) | Invitrogen | D1819 | |
| Dulbecco's Modified Eagle Medium (DMEM), serum-free | Fisher Scientific | 11885076 | |
| Dry ice | Local delivery | Custom order | |
| Epifluorescent imaging system, Nikon NiU and Nikon NIS Elements acquisition software | Nikon | Custom order | |
| Ethanol | Fisher Scientific | BP2818-4 | |
| Fetal bovine serum (FBS) | ThermoFisher | 16000044 | |
| Forceps, Dumont #7, curved | Fine Science Tools | 11274-20 | |
| Forceps, Dumont #5, straight | Fine Science Tools | 11254-20 | |
| Gloves (small, medium, large) | Microflex | N191, N192, N193 | |
| Gloves, MAPA Temp-Ice 700 Thermal (for handling dry ice) | Fisher Scientific | 19-046-563 | |
| Hemocytometer | Daigger | EF16034F EA | |
| Incubator, cell culture | Eppendorf | Galaxy 170 S | |
| Labelling tape | Fisher Scientific | 159015R | |
| Marking pen, Sharpie (ultra-fine) | Staples | 642736 | |
| Mice, immunodeficient (Nu/J) | Jackson Laboratory | 2019 | |
| Microcentrifuge, accuSpin Micro17 | Fisher Scientific | 13-100-675 | |
| Microcentrifgue tubes, Eppendorf tubes (1.5 mL) | Axygen | MCT-150-C | |
| Microscope slide box | Fisher Scientific | 50-751-4983 | |
| Needle, 27 gauge | Becton-Dickinson | 752 0071 | |
| Paintbrush | Grainger | 39AL12 | |
| Paper towels | Staples | 33550 | |
| Paraformaldehyde | Acros Organics | 416785000 | |
| Penicillin/Streptomycin | Gibco | 15140122 | |
| Perforated spoon, 15 mm diameter, 135 mm length | Roboz Surgical Instrument Co. | RS-6162 | |
| Phosphate buffered saline (PBS) | Fisher Scientific | BP3991 | |
| Pipet tips (10 μL) | Fisher Scientific | 02-707-438 | |
| Pipet tips (200 μL) | Fisher Scientific | 02-707-411 | |
| Pipet tips (1000 μL) | Fisher Scientific | 02-707-403 | |
| Pipets, serological (10 mL) | VWR | 89130-910 | |
| Pippetor, Gilson P2 | Daigger | EF9930A | |
| Pipettor Starter Kit, Gilson (2-10 μL, 20-200 μL, 200-1000 μL) | Daigger | EF9931A | |
| Platform shaker - orbital, benchtop | Cole-Parmer | EW-51710-23 | |
| Positively-charged microscope slides, Superfrost | Fisher Scientific | 12-550-15 | |
| Scalpel, size 10, Surgical Design, Inc. | Fisher Scientific | 22-079-707 | |
| Scissors, surgical - sharp, curved | Fine Science Tools | 14005-12 | |
| Software for image analysis, Nikon Elements | Nikon | Custom order | |
| Software for image analysis, ImageJ (FIJI) | National Institutes of Health | n/a | Download online (free) |
| Specimen disc 30 mm (chuck holder), cryostat accessory | Leica Biosystems | 14047740044 | |
| Staining tray, 245 mm BioAssay Dish | Corning | 431111 | |
| Syringe, 1 cc | Becton-Dickinson | 309623 | |
| Tape, laboratory, 19 mm width | Fisher Scientific | 15-901-5R | |
| Timer | Fisher Scientific | 14-649-17 | |
| Tissue culture dish, 100 x 15 mm diameter | Fisher Scientific | 08-757-100D | |
| Tissue culture flask, 225 cm2 | ThermoFisher | 159933 | |
| Tissue culture plate, 24-well | Becton-Dickinson | 353226 | |
| Tissue embedding mold, stainless steel | Tissue Tek | 4161 | |
| Tissue Freezing Medium, Optimal Cutting Temperature (OCT) | Fisher Scientific | 4585 | |
| Trypsin-EDTA (ethylenediaminetetraacetic acid), 0.25% | Gibco | 25200072 | |
| Water bath, Precision GP 2S | ThermoFisher | TSGP2S |
Referencias
- Pavlova, N. N., Thompson, C. B. The emerging hallmarks of cancer metabolism. Cell Metabolism. 23 (1), 27-47 (2016).
- Pellegatti, P., et al. Increased level of extracellular ATP at tumor sites: in vivo imaging with plasma membrane luciferase. PLoS ONE. 3, 25992008(2008).
- Falzoni, S., Donvito, G., Di Virgilio, F. Detecting adenosine triphosphate in the pericellular space. Interface Focus. 3 (3), 20120101(2013).
- Michaud, M., et al. Autophagy-dependent anticancer immune responses induced by chemotherapeutic agents in mice. Science. 334, 1573-1577 (2011).
- Wilhelm, K., et al. Graft-versus-host disease is enhanced by extracellular ATP activating P2X7R. Nature Medicine. 16, 1434-1438 (2010).
- Vander Heiden, M. G., Cantley, L. C., Thompson, C. B. Understanding the Warburg effect: the metabolic requirements of cell proliferation. Science. 324, 1029-1033 (2009).
- Cairns, R. A., Harris, I. S., Mak, T. W. Regulation of cancer cell metabolism. Nature Reviews Cancer. 11, 85-95 (2011).
- Chen, X., Qian, Y., Wu, S. The Warburg effect: evolving interpretations of an established concept. Free Radical Biology & Medicine. 79, 253-263 (2015).
- Wang, X., et al. Extracellular ATP, as an energy and phosphorylating molecule, induces different types of drug resistances in cancer cells through ATP internalization and intracellular ATP level increase. Oncotarget. 8 (5), 87860-87877 (2017).
- Patel, A., et al. ATP as a biological hydrotrope. Science. 356, 753-756 (2017).
- Qian, Y., et al. Extracellular ATP is internalized by macropinocytosis and induces intracellular ATP increase and drug resistance in cancer cells. Cancer Letters. 351, 242-251 (2014).
- Qian, Y., Wang, X., Li, Y., Cao, Y., Chen, X. Extracellular ATP a new player in cancer metabolism: NSCLC cells internalize ATP in vitro and in vivo using multiple endocytic mechanisms. Molecular Cancer Research. 14, 1087-1096 (2016).
- Commisso, C., et al. Macropinocytosis of protein is an amino acid supply route in Ras-transformed cells. Nature. 497, 633-637 (2013).
- Li, L., et al. The effect of the size of fluorescent dextran on its endocytic pathway. Cell Biology International. 39, 531-539 (2015).
- Yanagawa, Y., Matsumoto, M., Togashi, H. Enhanced dendritic cell antigen uptake via alpha2 adrenoceptor-mediated PI3K activation following brief exposure to noradrenaline. Journal of Immunology. 185, 5762-5768 (2010).
- Hoppe, H. C., et al. Antimalarial quinolines and artemisinin inhibit endocytosis in Plasmodium falciparum. Antimicrobial Agents & Chemotherapy. 48, 2370-2378 (2004).
- Chaudry, I. H. Does ATP cross the cell plasma membrane. Yale Journal of Biology & Medicine. 55, 1-10 (1982).
- Pant, H. C., Terakawa, S., Yoshioka, T., Tasaki, I., Gainer, H. Evidence for the utilization of extracellular [gamma-32P]ATP for the phosphorylation of intracellular proteins in the squid giant axon. Biochimica et Biophysica Acta. 582, 107-114 (1979).
- Chaudry, I. H., Baue, A. E. Further evidence for ATP uptake by rat tissues. Biochimica et Biophysica Acta. 628, 336-342 (1980).
- Koppenol, W. H., Bounds, P. L., Dang, C. V. Otto Warburg's contributions to current concepts of cancer metabolism. Nature Reviews Cancer. 11, 325-337 (2011).
- Dang, C. V. Links between metabolism and cancer. Genes & Development. 26, 877-890 (2012).
- Israelsen, W. J., Vander Heiden, M. G. ATP consumption promotes cancer metabolism. Cell. 143, 669-671 (2010).
- Koster, J. C., Permutt, M. A., Nichols, C. G. Diabetes and insulin secretion: the ATP-sensitive K+ channel (K ATP) connection. Diabetes. 54, 3065-3072 (2005).
- Szendroedi, J., et al. Muscle mitochondrial ATP synthesis and glucose transport/phosphorylation in type 2 diabetes. PLoS Medicine. 4, 154(2007).
- Miyamoto, S., et al. Mass spectrometry imaging reveals elevated glomerular ATP/AMP in diabetes/obesity and identifies sphingomyelin as a possible mediator. EBioMedicine. 7, 121-134 (2016).
Access restricted. Please log in or start a trial to view this content.
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoExplorar más artículos
This article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados