Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Fluoreszenzmikroskopie für ATP-Internalisierung, vermittelt durch Makropinozytose in menschlichen Tumorzellen und tumor-xenografierten Mäusen
* Diese Autoren haben gleichermaßen beigetragen
In diesem Artikel
Zusammenfassung
Wir haben eine reproduzierbare Methode entwickelt, um die Internalisierung von nicht hydrolysierbarem fluoreszierendem Adenosintriphosphat (ATP), einem ATP-Surrogat, mit hoher zellulärer Auflösung sichtbar zu machen. Wir validierten unsere Methode mit unabhängigen In-vitro- und In-vivo-Assays - humane Tumorzelllinien und immundefiziente Mäuse, die mit menschlichem Tumorgewebe xenotransplantiert wurden.
Zusammenfassung
Es hat sich gezeigt, dass Adenosintriphosphat (ATP), einschließlich extrazellulärer ATP (eATP), eine bedeutende Rolle in verschiedenen Aspekten der Tumorgenese spielt, wie z. B. Arzneimittelresistenz, epithelial-mesenchymaler Übergang (EMT) und Metastasierung. Intratumorales eATP ist 103 bis 104 mal höher in der Konzentration als in normalem Gewebe. Während eATP als Botenstoff fungiert, um die purinerge Signalgebung für die EMT-Induktion zu aktivieren, wird es auch von Krebszellen durch hochregulierte Makropinozytose, eine spezifische Art der Endozytose, internalisiert, um eine Vielzahl von biologischen Funktionen auszuführen. Zu diesen Funktionen gehören die Bereitstellung von Energie für ATP-erfordernde biochemische Reaktionen, die Spende von Phosphatgruppen während der Signaltransduktion und die Erleichterung oder Beschleunigung der Genexpression als transkriptioneller Cofaktor. ATP ist leicht verfügbar, und seine Studie in Krebs und anderen Bereichen wird zweifellos zunehmen. Die eATP-Studie befindet sich jedoch noch in einem frühen Stadium, und ungelöste Fragen bleiben unbeantwortet, bevor die wichtigen und vielseitigen Aktivitäten von eATP und internalisierter intrazellulärer ATP vollständig entschlüsselt werden können.
Die Beiträge der Labors dieser Autoren zu diesen frühen eATP-Studien umfassen die mikroskopische Bildgebung von nicht hydrolysierbarem fluoreszierendem ATP in Verbindung mit hoch- und niedermolekularem fluoreszierendem Dextrans, die als Makropinozytose- und Endozytose-Tracer dienen, sowie verschiedene Endozytoseinhibitoren, um den eATP-Internalisierungsprozess zu überwachen und zu charakterisieren. Diese Bildgebungsmodalität wurde auf Tumorzelllinien und auf immundefiziente Mäuse angewendet, die mit menschlichen Krebstumoren xenotransplantiert wurden, um die eATP-Internalisierung in vitro und in vivozu untersuchen. Dieses Papier beschreibt diese in vitro und in vivo Protokolle, mit einem Schwerpunkt auf der Modifikation und Feinabstimmung der Assay-Bedingungen, so dass die Makropinozytose-/Endozytose-vermittelten eATP-Internalisierungsassays erfolgreich in verschiedenen Systemen durchgeführt werden können.
Einleitung
Die opportunistische Aufnahme von intratumoralen extrazellulären (dh) Nährstoffen wurde kürzlich zu einem Schlüsselmerkmal für den Krebsstoffwechsel ernannt1. Einer dieser wichtigen Nährstoffe ist ATP, da die Konzentration von ieATP 103 und 104 mal höher ist als die in normalem Gewebe, im Bereich von mehreren hundert μM bis zu niedrigem mM2,3,4,5. Als wichtiges Energie- und Signalmolekül spielt ATP eine zentrale Rolle im Zellstoffwechsel in krebsartigen und gesunden Zellen6,7,8. Extrazelluläres ATP ist nicht nur am Wachstum von Krebszellen beteiligt, sondern fördert auch die Arzneimittelresistenz9. Bisher nicht erkannte Funktionen von ATP, wie die hydrotrope Aktivität, wurden kürzlich identifiziert, was eine ATP-Beteiligung an Krankheiten wie Alzheimer10impliziert. Tatsächlich scheint es, dass unser Verständnis von ATP und seinen Funktionen in Krebszellen, gesunden Zellen und anderen erkrankten Zellen bei weitem nicht vollständig ist. Aufgrund der Instabilität von ATP und der hohen Fluktuationsraten in den Zellen ist es jedoch technisch schwierig, die Bewegung von ATP über die Zellmembran und in die Zelle zu überwachen.
Um dieses Problem anzugehen und den Bedarf dieses Forschungsbereichs zu decken, wurde eine Methode entwickelt, bei der nichthydrolysierbare fluoreszierende ATP (NHF-ATP) (Abbildung 1) als Surrogat verwendet wurde, um die Internalisierung von ATP zu visualisieren und die intrazelluläre räumliche Lokalisierung von internalisiertem ATP sowohl in vitro als auch in vivozu beobachten11,12 . Es wurde gezeigt, dass NHF-ATP endogenes ATP ersetzt, um die ATP-Bewegung über tierische Zellmembranen zu untersuchen, sowohl in Krebszelllinien als auch in menschlichem Tumorgewebe, das an immundefizienten Mäusen xenotransplantiert wurde11,12. Darüber hinaus blockierte die Verabreichung von Makropinozytoseinhibitoren an Zellen die eATP-Internalisierung, was darauf hindeutet, dass die intrazelluläre Aufnahme von eATP einen makropinozytotischen Mechanismus beinhaltet9,11,12. Dieses Protokoll erlaubt eine immunbasierte Comarkierung gegen zellspezifische Proteine und damit die Identifizierung, welcher Zelltyp NHF-ATP internalisiert. Mit In-vivo-Tumor-Xenotransplantaten und hochauflösender Mikroskopie kann NHF-ATP räumlich über die Gewebeprobe und sogar innerhalb einer einzelnen Zelle sichtbar gemacht werden. Diese Methoden ermöglichen auch quantitative Analysen, wie z. B. den Prozentsatz der zellulären Aufnahme, die Anzahl der makropinozytotischen Vesikel und die Internalisierungskinetik. Dieser Artikel beschreibt detailliert, wie NHF-ATP allein oder zusammen mit dem endozytose-tracer fluoreszierenden Dextrans13,14,15,16, in verschiedenen experimentellen Umgebungen verwendet werden kann, um die Internalisierung und intrazelluläre Lokalisation von ATP nach Internalisierung in Zellen zu untersuchen.

Abbildung 1: Strukturen von nicht hydrolysierbarem fluoreszierendem ATP und Tetramethylrhodamin markiertem hochmolekularem Fluoreszenzdextran. (A) Struktur von NHF-ATP. (B) Schematische Darstellung der HMWFD. Abkürzungen: ATP = Adenosintriphosphat; NHF-ATP = nicht hydrolysierbare fluoreszierbares ATP; TMR = Tetramethylrhodamin; HMWFD = hochmolekulares fluoreszierendes Dextran. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Access restricted. Please log in or start a trial to view this content.
Protokoll
Alle hierin berichteten Verfahren wurden in Übereinstimmung mit der IACUC der Ohio University und dem NIH durchgeführt.
1. Auswahl von nicht hydrolysierbarem fluoreszierendem ATP (NHF-ATP) und Dextrans
- Wählen Sie einen fluorophor-konjugierten NHF-ATP (Abbildung 1A) und Endozytose-Tracer, hoch- und niedermolekularen fluoreszierenden Dextrans (TMR-HMWFD und TMR-LMWFD) (Abbildung 1B), basierend auf den bevorzugten Emissionswellenlängen (z. B. Bildgebungssystem mit geeigneten Filtern) und dem zu untersuchenden spezifischen Endozytoseprozess.
2. ATP-Lokalisierungsstudien, in vitro (Abbildung 2)
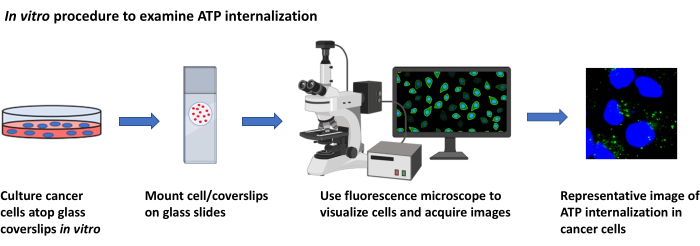
Abbildung 2: In-vitro-Verfahren zur Untersuchung der ATP-Internalisierung. Schematische Darstellung des Protokolls zur Visualisierung der Internalisierung von extrazellulärem ATP in kultivierten Krebszellen mittels Fluoreszenzmikroskopie. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
- Zellkultur und Zellpräparation
HINWEIS: Führen Sie die Zellkultur unter sterilen Bedingungen in einer Gewebekulturhaube durch.- Bereiten Sie Dulbecco's Modified Eagle Medium (DMEM), bestehend aus 10% (v/v) fetalem Rinderserum (FBS) und 1% (v/v) Penicillin/Streptomycin (im Folgenden DMEM/FBS genannt), steriler phosphatgepufferter Kochsalzlösung (PBS) und 0,25% Trypsin/Ethylendiamintetraessigsäure (EDTA) in einem 37 °C wasserbad vor.
- Kultur menschlicher Krebszellen in DMEM/FBS in einer 100 mm Gewebekulturschale. Halten Sie die Zellen in einem Auf 37 °C eingestellten Inkubator mit einer 5%CO2-Atmosphäre.
- Wenn die Zellen den Zusammenfluss erreichen, passieren Sie die Zellen, indem Sie zuerst das Kulturmedium entfernen. Als nächstes spülen Sie die Schale mit 5 ml sterilem PBS ab, entfernen Sie das PBS und fügen Sie 3 ml 0,25% Trypsin hinzu. Bei 37 °C in einer 5% igenCO2-Atmosphäre für 5 min inkubieren.
- Rufen Sie die Schüssel auf und fügen Sie dann 6 ml DMEM / FBS hinzu, um die Trypsinisierung zu stoppen. Die Zellen in Suspension in ein konisches 15-ml-Rohr überführen und bei 800 × g für 5 min zentrifugieren, um die Zellen zu pelletieren.
- Nach der Zentrifugation den Überstand absaugen und 10 ml DMEM/FBS verwenden, um das Zellpellet durch Pipettieren wieder zu versenken.
- Zählen Sie die Zelldichte und Lebensfähigkeit mit einem Hämozytometer. Verwenden Sie DMEM/FBS, um die Zellsuspension auf eine Dichte von ~ 7,5 × 104 Zellen / ml zu verdünnen.
- Vorbereitung von Deckgläsern und Samenzellen
- 12 mm Deckgläser mit 70% Ethanol waschen und vorsichtig mit filigranen Arbeitstüchern abwischen. Sterilisieren Sie die Deckgläser und eine Pinzette durch Autoklavieren.
- Verwenden Sie in einer Gewebekulturhaube eine Pinzette, um einen Deckglas in jede Vertiefung einer 24-Well-Gewebekulturplatte zu legen.
HINWEIS: Später wird der Deckglas mit Zellen zur Bildgebung direkt auf einem Objektträger montiert. - Geben Sie 300 μL der Zellsuspension (Zellen in DMEM/FBS) bei einer Aussaatdichte von ~ 2,5 × 104 Zellen pro Vertiefung in die 24-Well-Platte, die die sterilisierten Deckgläser enthält. Inkubieren Sie unter sterilen Bedingungen bei 37 °C mit 5%CO2-Fluss.
- Aushungern von Zellen
- Entfernen Sie vierundzwanzig Stunden nach der Aussaat das DMEM/FBS aus jeder Vertiefung. Fügen Sie sofort 300 μL vorgewärmtes serumfreies DMEM in jede Vertiefung hinzu, um die Zellen für 15-18 h serumhungern zu lassen, um die Aufnahme extrazellulärer Nährstoffe zu induzieren.
HINWEIS: Die Hungerperiode von 15-18 h ist ein kritischer Parameter.
- Entfernen Sie vierundzwanzig Stunden nach der Aussaat das DMEM/FBS aus jeder Vertiefung. Fügen Sie sofort 300 μL vorgewärmtes serumfreies DMEM in jede Vertiefung hinzu, um die Zellen für 15-18 h serumhungern zu lassen, um die Aufnahme extrazellulärer Nährstoffe zu induzieren.
- Vorbereitung von NHF-ATP und HMWFD/LMWFD Lösungen
- Verwenden Sie eine Analysenwaage, um hochmolekulares (70 kDa) fluoreszierendes TMR-Dextran (TMR-HMWFD, 1 mg/ml), einen Tracer zur Visualisierung von Makropinosomen, oder NHF-ATP (10 μmol/L) in serumfreiem DMEM in einem 1,5 ml Mikrozentrifugenröhrchen zu wiegen. Stellen Sie die Röhren lichtgeschützt für 15 min in ein 37 °C warmes Wasserbad.
- Zentrifuge bei 12.000 × g für 5 min bei Raumtemperatur. Übertragen Sie den klaren Überstand vorsichtig in ein neues 1,5 ml Mikrozentrifugenrohr, wobei alle Pellets oder Ablagerungen intakt bleiben, um unauflösliche Kristalle zu entfernen.
- Die Lösungen aus Schritt 2.4.1 werden den Zellen in jeder Vertiefung zugegeben und die Zellen 30 min bei 37 °C inkubiert.
ANMERKUNG: Wenn HMWFD- und NHF-ATP-Lösungen zur Co-Inkubation mit den Zellen gemischt werden sollen, bereiten Sie beide Lösungen mit dem 2-fachen der Endkonzentrationen vor. Die Lösungen werden später im Verhältnis 1:1 gemischt, um die endgültigen genauen Arbeitskonzentrationen zu erreichen. Vermeiden Sie Licht, da die Reagenzien lichtempfindlich sind.
- Behandlung von Zellen und Fixierung
- Geben Sie in einer frischen 24-Well-Platte 500 μL vorgewärmtes PBS in jede der fünf Vertiefungen.
- Nehmen Sie nach der Zellinkubation jeden Deckglas vorsichtig mit einer Pinzette auf. Spülen Sie jeden Deckglas ab, indem Sie ihn in 500 μL vorgewärmtes PBS tauchen. Wiederholen Sie dies fünfmal mit den fünf PBS-gefüllten Vertiefungen.
HINWEIS: Das schonende Waschen von Zellen auf Deckgläsern ist entscheidend für den Erfolg dieses Experiments. - Klopfen Sie nach der letzten PBS-Wäsche auf den Deckglas auf ein empfindliches Arbeitstuch, um zusätzliches PBS zu absorbieren, und übertragen Sie den Deckglas sofort auf kaltes (4 ° C) 3,7% Formaldehyd, das in einer 24-Well-Platte vorgespannt ist. Fixieren Sie die Zellen für 15 min bei Raumtemperatur.
- Während die Zellen fixiert werden, werden Mikroskopobjektträger mit 70% Ethanol vorgereinigt. Entfernen Sie die Deckgläser aus den Vertiefungen und montieren Sie sie auf den Objektträgern, wobei Sie 5 μL wässriges Montagemedium verwenden, das die Kernfärbung 4 ′,6-Diamidino-2-phenylindol (DAPI) pro Deckglas enthält. Tupfen Sie überschüssiges PBS vorsichtig mit einem Papiertuch oder einem empfindlichen Aufgabenwischer ab.
- Fluoreszenzmikroskopie und Bildaufnahme
- Erfassen Sie zwei bis 24 Stunden nach den oben genannten Schritten Bilder von Zellen und internalisierten HMWFD und / oder NHF-ATP mit einem Epifluoreszenz-Bildgebungssystem und einer Datenerfassungssoftware.
HINWEIS: In diesem Unterabschnitt werden die Schritte zur Aufnahme von Bildern mit einem Nikon NiU-Mikroskop beschrieben, das mit Einer Epifluoreszenz-Bildgebungsfunktion und der Nikon NIS Elements-Software ausgestattet ist. Es können jedoch auch andere vergleichbare Bildgebungssysteme und Erfassungssoftware verwendet werden. Folgen Sie den Bedienungsanleitungen des Herstellers.- Legen Sie den Objektträger im binokularen Modus auf die Bühne eines aufrechten Epifluoreszenzmikroskops. Greifen Sie auf das Imaging-Programm zu.
- Wählen Sie das 10-fache Objektiv aus, passen Sie die Bühne an, um den Fokus zu definieren, und scannen Sie das Dia von links nach rechts in einer serpentinenförmigen Weise, um die interessierenden Bereiche zu identifizieren.
HINWEIS: Die Identifizierung von Regionen von Interesse variiert zwischen den Zelltypen, wobei einige Zelllinien / Krebsarten unterschiedliche und unterschiedliche Grade der TMR-HMWFD- und / oder NHF-ATP-Aufnahme aufweisen. - Wählen Sie das 40-fache Objektiv aus und wechseln Sie mit dem Schalter am Mikroskop vom binokularen Modus in den Bildaufnahmemodus.
- Klicken Sie auf das Live Quality-Symbol im Imaging-Programm, um Bilder anzuzeigen und anschließend zu erfassen.
- Definieren Sie im OC-Bedienfeld auf der Symbolleiste Anmerkungen und Messungen die Belichtungsparameter für jeden Filterwürfel oder Fluoreszenzkanal.
HINWEIS: Wählen Sie die geeignete Belichtungszeit für jeden Kanal, da die Signalintensitäten unterschiedlich sind. Wählen Sie beispielsweise eine Belichtungszeit von 200 ms für DAPI, 2 s für HMWFD und 4 s für NHF-ATP aus. Sobald die Belichtungszeit pro Kanal bestimmt ist, verwenden Sie diese Einstellung für alle Bilder pro Kanal mit unterschiedlicher Behandlung oder Bedingungen. - Sobald die Belichtungseinstellungen für jeden Kanal festgelegt wurden, verwenden Sie die Symbolleiste für die Mehrkanalerfassung, um ein 3-Kanal-Bild mit den definierten Belichtungseinstellungen zu erfassen.
HINWEIS: Die Bildaufnahme über den Mehrkanal-ND-Aufnahmemodus ermöglicht die automatische Bilderfassung für jeden Kanal desselben Sichtfelds. Der Verschluss wird zwischen den Turmwechseln automatisch geschlossen. - Alternativ können Sie Mehrkanalbilder manuell aufnehmen, indem Sie zwischen filterwürfeln, die Belichtungszeit einstellen, den Verschluss zwischen der Bildaufnahme für jeden Kanal schließen/öffnen und jedes für einzelne Kanäle aufgenommene Bild überlagern.
HINWEIS: Der ND-Erfassungsmodus automatisiert diesen Prozess und stellt zusammengeführte Bilder bereit. - Speichern Sie das Bild als .nd2-Datei (Nikon Elements-Format speichert Metadaten). Speichern Sie TIF-Dateien, einschließlich des zusammengeführten Kanalbilds und einzelner Kanalbilder.
HINWEIS: TIF-Dateien können mit einer breiteren Auswahl an Softwareanwendungen verwendet werden. - Verwenden Sie die Funktion "Objektanzahl" auf der Werkzeugleiste "Analyse", um die Anzahl der NHF-ATP-, TMR-HMWFD- und/oder TMR-LMWFD-positiven Zellen in einer gespeicherten .nd2-Bilddatei zu zählen.
- Exportieren Sie die Daten über das Analyseprogramm in eine Tabelle.
- Erfassen Sie zwei bis 24 Stunden nach den oben genannten Schritten Bilder von Zellen und internalisierten HMWFD und / oder NHF-ATP mit einem Epifluoreszenz-Bildgebungssystem und einer Datenerfassungssoftware.
- Datenquantifizierung und -analyse
- Für jede untersuchte Bedingung werden 50 bis 100 Zellen zur Quantifizierung abgebildet. Mit der Datenanalysesoftware (Software, die im Epifluoreszenz-Bildgebungssystem oder einer anderen Software enthalten ist) zählen und berechnen Sie die mittlere Anzahl von fluoreszierenden Vesikeln pro Zelle.
- Verwenden Sie geeignete statistische Methoden, um die quantifizierten Ergebnisse zu analysieren.
3. ATP-Internalisierung in Tumoren, ex vivo (Abbildung 3)

Abbildung 3: In-vivo-Verfahren zur Untersuchung der ATP-Internalisierung. Schematische Darstellung des Protokolls zur Visualisierung der Internalisierung von extrazellulärem ATP in Tumor-Xenotransplantaten mittels Kryosektion und Fluoreszenzmikroskopie. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
- Vorbereitung von Zellkulturen für die Implantation
- Zucht von Krebszellen auf 80% Konfluenz bei 37 °C in einem 225 cm2 Kolben unter Verwendung von DMEM in Ergänzung mit FBS auf eine Endkonzentration von 10% (v/v) und Penicillin/Streptomycin bei 1% (v/v).
- Waschen Sie die Zellen zweimal mit 10 ml PBS. Vorwarmen Sie 0,25% Trypsin/EDTA auf 37 °C. 8 ml Trypsin/EDTA zugeben und 2 min bei 37 °C inkubieren.
- Sobald sich die Zellen vom Boden des Kolbens zu lösen beginnen, verwenden Sie eine 10 ml sterile serologische Pipette, um 8 ml DMEM / FBS hinzuzufügen. Aspirieren Sie zweimal, um adhärente Zellen zu entfernen. Verwenden Sie die Pipette, um die abgetrennten Zellen aus dem Kolben in ein 50 ml erisches Rohr zu übertragen.
- Fügen Sie 10 ml DMEM / FBS mit einer 10 ml Pipette hinzu und sammeln Sie alle verbleibenden schwimmenden Zellen in demselben konischen 50-ml-Rohr.
- Zentrifugieren Sie die Zellsuspension bei 600 × g, 4 °C für 4 min. Entfernen Sie den Überstand und resuspendieren Sie die Zellen in 1 ml eiskaltem PBS.
- Zählen Sie die Zelldichte mit einem Hämozytometer. Halten Sie die Zellsuspension während des Zählens auf Eis.
- Zentrifugieren Sie die Zellsuspension bei 600 × g, 4 °C für 4 min. Entfernen Sie den Überstand und suspendieren Sie die Zellen in eiskaltem PBS, so dass die Zelldichte 5 × 106 Zellen pro 100 μL PBS beträgt. Die Zellsuspension wird in ein 1,5 ml Mikrozentrifugenröhrchen überführt.
- Subkutane Injektion von Krebszellen zur Entwicklung von Xenotransplantattumoren
- Verwenden Sie eine latexfreie Spritze (1 ml) mit einer Präzisionsgleiternadel (27 G Nadel) für die Injektion von Krebszellen.
- Übertragen Sie die Zellsuspension (5 × 106 in 100 μL PBS) in ein 1,5 ml Mikrozentrifugenröhrchen. Ziehen Sie die Zellen in die Spritze.
- Wählen Sie eine Injektionsstelle an der Flanke einer immundefizienten (nackten) Maus und reinigen Sie die Haut sanft mit 75% Ethanol. Wischen Sie überschüssiges Ethanol mit einem empfindlichen Task-Wipe ab.
- Halten Sie für die subkutane Injektion die Nadel in einem Winkel von ca. 10° zur Haut. Führen Sie die Nadelspitze mit der abgeflachten Seite nach oben direkt unter die Haut ein, so dass nur 1-2 mm der Nadel außerhalb der Haut sichtbar sind. Geben Sie die Zellen langsam über ca. 10 s aus der Spritze.
- Nachdem Sie das gesamte Volumen injiziert haben, halten Sie die Nadel für 3-5 s an Ort und Stelle, ziehen Sie dann die Nadel zurück und üben Sie mit einem Finger für weitere 3-5 Sekunden sanften, aber festen Druck auf die Injektionsstelle aus, um ein Austreten des injizierten Inhalts zu verhindern.
- Überwachen und messen Sie das Tumorwachstum mit Messschiebern, bis die Tumore ein Volumen von 200-500 mmerreichen 3.
- Herstellung von HMWFD- und NHF-ATP-Lösungen für die Post-Tumor-Resektion
- 300 μL 16 mg/ml HMWFD in serumfreiem DMEM (Kulturmedium) auflösen, 30 min in einem 37 °C warmen Wasserbad inkubieren und wie oben beschrieben bei 12.000 × g 5 min zentrifugieren. Die Lösung wird in ein 1,5 ml Mikrozentrifugenröhrchen gegeben.
- Fügen Sie 40 μL NHF-ATP-Analogmaterial (1 mM) zu 160 μL serumfreiem DMEM hinzu, um eine 0,2 mM NHF-ATP-Lösung herzustellen.
- Vorbereitung von Versuchsbohrungen
HINWEIS: Dieses experimentelle Design wird die intrazelluläre Internalisierung von HMWFD + NHF-ATP untersuchen, was auf die Aufnahme durch Makropinosomen hinweist.- Bereiten Sie die Vertiefungen wie folgt vor: Vertiefung #1, Kontrolle: 200 μL serumfreies DMEM; Well #2, Kontrolle: 100 μL 16 mg/ml LMWFD + 100 μL serumfreies DMEM = 200 μL 8 mg/ml LMWFD; Well #3, Kontrolle: 100 μL 0,2 mM NHF-ATP + 100 μL serumfreies DMEM = 200 μL 0,1 mM NHF-ATP; Brunnen #4; Experimentell: 100 μL 16 mg/ml HMWFD + 100 μL 0,2 mM NHF-ATP = 200 μL 0,1 mM NHF-ATP und 8 mg/ml HMWFD.
- Vorbereitung von Tumorgewebe
- Euthanasie der Maus durch zervikale Luxation oder gemäß dem von der IACUC genehmigten Protokoll.
- Verwenden Sie ein Skalpell der Größe 10, um die isolierten Tumore mit einer Dicke von ~ 500-1.000 μm zu schneiden.
- Inkubieren Sie die Tumorscheiben in serumfreiem DMEM, ergänzt mit 100 μM NHF-ATP und/oder 8 mg/ml H/LMWFD in Mikrozentrifugenröhrchen für 40 min bei 37 °C mit 5%CO2-Fluss.
HINWEIS: Nach der Inkubation bewirkt der Stoffwechsel des Tumorgewebes, dass sich die Farbe des Mediums ändert. - Spülen Sie das Gewebe in 37 °C vorgewarmtem PBS (2 ml für jede Spülung in einer 24-Well-Platte).
- Übertragen Sie das Gewebe auf eine neue 24-Well-Platte mit vorgewärmtem frischem PBS, spülen Sie es ab und wiederholen Sie es viermal mit sanftem Schütteln.
- Kryo-Embedding (Vorbereitung von gefrorenen Gewebeblöcken)
- Bereiten Sie Identifikationsetiketten für jeden zu erntenden Tumor vor. Schneiden Sie ein 2 cm langes Stück Laborband und falten Sie es in zwei Hälften, kleben Sie die Seiten der Länge nach zusammen. Verwenden Sie einen Markierungsstift, um das Etikett zu beschriften, z. B. mit einer Maus-/Tumoridentifikationsnummer.
- Bereiten Sie Einbettformen vor, indem Sie Edelstahl-Tissueformen direkt auf Trockeneis legen.
HINWEIS: Trockeneis kann Erfrierungen, Verbrennungen und Ersticken verursachen. Tragen Sie isolierte Handschuhe beim Umgang mit Trockeneis. Verwenden Sie Trockeneis in einem gut belüfteten Bereich. Bewahren Sie Trockeneis nicht in einem dicht verschlossenen Behälter auf. Lagern Sie stattdessen in einem Behälter (z. B. einem Styroporkühler), in dem Gas entweichen kann. - Während die Form abkühlt, legen Sie einen kleinen Pool mit Gewebe-Gefriermedium in eine 10 mm große Gewebekulturplatte. Stellen Sie sicher, dass das Volumen ausreicht, um das Tumorgewebe, das geerntet wird, einzutauchen.
- Verwenden Sie einen perforierten Löffel, um das resezierte Tumorgewebe aufzusaugen und das Gewebe sofort in ein gefrierendes Medium zu legen, um sicherzustellen, dass das Gewebe untergetaucht ist. Rollen Sie das Gewebe mit dem perforierten Löffel vorsichtig im Gefriermedium und stellen Sie sicher, dass das Medium alle Gewebeoberflächen badet.
- Bewegen Sie das Gewebe vorsichtig in die Einbettform, die das Gefriermedium enthält. Platzieren Sie das entsprechende Etikettenetikett vertikal in das Gefriermedium/die Gefrierform, um es einzufrieren. Stellen Sie sicher, dass das geschriebene Etikett außerhalb des Mediums sichtbar ist.
- Wenn das Einfrieren abgeschlossen ist (das Gefriermedium wird undurchsichtig-weiß), entfernen Sie den Gewebeblock aus der Form, legen Sie ihn auf Trockeneis und wiederholen Sie ihn für jeden Tumor. Lagern Sie die Gewebeblöcke bei -80 °C für mehrere Monate vor der Kryosektion.
- Vorbereitung von Objektträgern von Gewebeproben
- Um die Wahrscheinlichkeit zu maximieren, internalisierungspositive Zellen zu finden und repräsentativere Geweberegionen zu haben, sammeln Sie serielle Kryosektionen bei -18 bis -20 ° C mit einem Kryostaten.
- Prechill-Kryostat-Werkzeuge (Klinge, Rasierklinge, Anti-Roll-Platte, Gewebefutterhalter, Pinsel) und gleichen die Tumorgewebeblöcke aus, indem Sie sie in eine Kryostatenkammer bei -18 bis -20 ° C legen. Stellen Sie den Winkel des Klingenhalters auf 5-10° ein. Schneiden Sie den Gewebeblock bei Bedarf vorsichtig mit einer Rasierklinge ab und befestigen Sie ihn mit Gewebegefriermedium als "Klebstoff" auf dem Futterhalter.
- Verriegeln Sie den Futterhalter in der vertikalen Position auf der Mikrotomeinheit, die mit jeder Umdrehung der Handkurbel auf den eingestellten Abstand (z. B. 10 μm) vorrückt. Positionieren Sie die Anti-Roll-Platte so, dass sie knapp über der Höhe der Klinge ruht. Um ein Einrollen des Gewebes vor dem Vorrücken des Mikrotoms zu verhindern, schieben Sie den Daumen vorsichtig über den unteren Rand des Gewebeblocks.
- Wenn das Mikrotom fortschreitet und der Gewebeabschnitt auf die Metallplatte fällt, verwenden Sie einen Pinsel, um den Gewebeabschnitt zu führen und das Gewebe bei Bedarf abzurollen.
- Bewegen Sie den Objektträger ohne Berührung über den Gewebeschnitt, so dass der Schnitt vom Objektträger angezogen wird.
HINWEIS: Kryostatenklingen (hochkarätig, Einwegklingen) sind extrem scharf und können schwere Verletzungen verursachen. Seien Sie vorsichtig bei der Handhabung von Klingen und der Bedienung des Kryostaten. Verwenden Sie einen Klingenschutz, falls verfügbar. Eine angemessene Schulung ist erforderlich. - Schneiden Sie den Tumor in Abschnitte von 10 μm Dicke. Übertragen Sie die geschnittenen Schnitte sofort auf einen positiv geladenen Glasmikroskop-Objektträger.
HINWEIS: Sammeln Sie bei seriellen Schnitten zunächst einen 10 μm dicken Schnitt in der oberen linken Ecke jedes der acht positiv geladenen Dias. Bewegen Sie den Kryostaten durch nachfolgende 100-200 μm Gewebe und verwerfen Sie das Gewebe. Übertragen Sie sofort alle geschnittenen Schnitte auf Glasmikroskopobjektträger. - Als nächstes sammeln Sie einen weiteren 10 μm dicken Abschnitt neben dem zuvor platzierten Gewebeschnitt für jeden der acht Objektträger. Wiederholen Sie diesen seriellen Sammelvorgang, bis jeder der acht Objektträger acht Gewebeschnitte im Abstand von jeweils 100-200 μm enthält. Halten Sie die Gewebeabschnitte im Dunkeln, um die Fluoreszenz zu erhalten.
HINWEIS: Gewebeschnitte auf Objektträgern können mehrere Monate in einer Objektträgerbox bei -80 °C aufbewahrt werden.
- Um die Wahrscheinlichkeit zu maximieren, internalisierungspositive Zellen zu finden und repräsentativere Geweberegionen zu haben, sammeln Sie serielle Kryosektionen bei -18 bis -20 ° C mit einem Kryostaten.
- Fixierung von Gewebeobjektträgern
- KRITISCHER SCHRITT: Fixieren Sie die Gewebeschnitte in 95% Ethanol bei -18 bis -20 °C bis für 5 min.
- Waschen Sie den festen Abschnitt für 5 minuten mit PBS bei Raumtemperatur und montieren Sie dann die festen Tumorabschnitte unter einem Glasdeckglas mit 10 μL eines wässrigen Montagemediums mit DAPI.
- Zwölf bis 24 h nach der Montage untersuchen Sie die fixierten Tumorabschnitte mittels Fluoreszenzmikroskopie und nehmen Bilder auf, wie oben für die kultivierten Zellen beschrieben.
- Fluoreszenzmikroskopie und Bildaufnahme
- Identifizieren Sie Interessante Regionen und erfassen Sie Bilder, wie in Abschnitt 2.6 beschrieben.
- Datenquantifizierung und -analyse
- Quantifizieren Sie die Zellen und wenden Sie geeignete statistische Analysen gemäß Abschnitt 2.7 an.
4. ATP-Internalisierung in Tumoren, in vivo
- Bereiten Sie Zellkulturen für die Implantation gemäß Abschnitt 3.1 vor.
- Subkutane Injektion von Krebszellen zur Entwicklung von Xenotransplantattumoren
- Erzeugen Sie xenotransplantierte Tumoren, wie in Abschnitt 3.2 beschrieben.
- ATP- und/oder Dextran-Injektion in Xenograft-Tumoren
- Herstellung der Behandlungslösungen von DMEM (Vehikel) oder 8 mg/ml HMWFD oder LMWFD, mit oder ohne NHF-ATP (100 μM) in DMEM, wie oben beschrieben.
- Verwenden Sie eine 1-ml-Spritze, um 50 μL einer Behandlungslösung zu sammeln, und injizieren Sie die Lösung direkt in jeden Xenotransplantattumor. Wiederholen Sie den Vorgang für vier biologische Replikate jeder Behandlung.
- Gewebegewinnung und Kryo-Einbettung
- Bereiten Sie Identifikationsetiketten für jeden zu erntenden Tumor vor. Schneiden Sie ein 2 cm langes Stück Laborband und falten Sie es in zwei Hälften, kleben Sie die Seiten der Länge nach zusammen. Verwenden Sie einen Markierungsstift, um das Etikett zu beschriften, z. B. mit einer Maus-/Tumoridentifikationsnummer.
- Ca. 5 min nach der Injektion wird die Maus durch zervikale Luxation oder gemäß dem von der IACUC genehmigten Protokoll eingeschläfert.
- Machen Sie mit einem Skalpell der Größe 10 einen Schnitt neben dem Tumor und ungefähr senkrecht zur Richtung der Nadelinjektion. Verwenden Sie eine Pinzette und eine chirurgische Schere, um das Tumorgewebe aus dem umgebenden Gewebe zu resezieren.
- Teilen Sie den Tumor in zwei bis vier 1 cm2 Stücke, abhängig von der Gesamtgröße des Tumors.
- Bereiten Sie die Einbettungsformen vor und betten Sie das Gewebe ein, wie oben in Abschnitt 3.6 beschrieben. Stellen Sie sicher, dass die Erntezeit von der intratumoralen Dextran-Injektion bis zur Kryo-Einbettung nicht mehr als 7-8 Minuten beträgt.
- Vorbereitung von Objektträgern von Gewebeproben
- Sammeln Sie serielle Tumorabschnitte, wie in Abschnitt 3.7 beschrieben.
- Fixierung von Gewebeobjektträgern
- Fixieren Sie das Gewebe, wie in Abschnitt 3.8 beschrieben.
- Fluoreszenzmikroskopie und Bildaufnahme
- Identifizieren Sie die Regionen von Interesse und erfassen Sie Bilder, wie in Abschnitt 2.6 beschrieben.
- Datenquantifizierung und -analyse
- Quantifizieren Sie die Zellen und wenden Sie geeignete statistische Analysen an, wie in Abschnitt 2.7 beschrieben.
Access restricted. Please log in or start a trial to view this content.
Ergebnisse
In-vitro-Studie
Die intrazelluläre Internalisierung von NHF-ATP wurde durch Kolokalisation von NHF-ATP mit HMWFD oder LMWFD nachgewiesen (Abbildung 4). Der Erfolg dieses Verfahrens beruht in erster Linie auf der Verwendung geeigneter Konzentrationen von NHF-ATP und Dextrans und auf der Bestimmung der geeigneten Art(en) von Dextrans (Poly-Lysin vs. neutral). Um beispielsweise die Makropinozytose zu untersuchen, wurde die HMWFD gewählt, da sie ...
Access restricted. Please log in or start a trial to view this content.
Diskussion
Es wurde eine Methode zur räumlichen, zeitlichen und quantitativen Analyse der zellulären Internalisierung von nicht hydrolysierbarem ATP entwickelt. Diese Methode ist breit anwendbar für den Einsatz in verschiedenen biologischen Systemen, einschließlich verschiedener tumorigener Modelle, für die wir technische Anweisungen und repräsentative Daten bereitstellen. Um interpretierbare Daten während in vivo ATP-Internalisierungsstudien (Abschnitt 4 des Protokolls) zu erhalten, ist es von entscheidender Bedeut...
Access restricted. Please log in or start a trial to view this content.
Offenlegungen
Die Autoren erklären keine gegensätzlichen Interessen.
Danksagungen
Die Kryosektion wurde vor Ort am Ohio University Histopathology Core durchgeführt. Diese Arbeit wurde teilweise durch Start-up-Fonds (Ohio University College of Arts & Sciences) an C Nielsen unterstützt; NIH gewähren R15 CA242177-01 und RSAC Award an X Chen.
Access restricted. Please log in or start a trial to view this content.
Materialien
| Name | Company | Catalog Number | Comments |
| A549 cells, human lung epithelial, carcinoma | National Cancer Institute | n/a | Less expensive source |
| Acetone | Fisher Scientific | S25904 | |
| Aluminum foil, Reynolds | Grainger | 6CHG6 | |
| Aqueous Mounting Medium, ProLong Gold Anti-fade Reagent | ThermoFisher | P36930 | |
| ATP analog | Jena Biosciences | NK-101 | |
| Autoclave, sterilizer | Grainger | 33ZZ40 | |
| Blades, cryostat, high profile | C. L. Sturkey, Inc. | DT554550 | |
| Calipers, vernier | Grainger | 4KU77 | |
| Cell medium, Ham's Nutrient Mixture F12, serum-free | Millipore Sigma | 51651C-1000ML | |
| Centrifuge, refrigerated with swinging bucket rotor | Eppendorf | 5810R | |
| Chloroform | Acros Organics | 423555000 | |
| Conical tube, 15 mL | VWR | 21008-216 | |
| Conical tube, 50 mL | VWR | 21008-242 | |
| Coverslips, glass, 12 mm | Corning | 2975-245 | |
| Cryostat, Leica CM1950 | Leica Biosystems | CM1950 | |
| Delicate task wipe, Kim Wipes | Kimberly-Clark | 34155 | |
| Dextran, Lysine fixable, High Molecular Weight (HMW) | Invitrogen | D1818 | MW = 70,000, Tetramethylrhodamine |
| Dextran, Neutral, High Molecular Weight (HMW) | Invitrogen | D1819 | |
| Dulbecco's Modified Eagle Medium (DMEM), serum-free | Fisher Scientific | 11885076 | |
| Dry ice | Local delivery | Custom order | |
| Epifluorescent imaging system, Nikon NiU and Nikon NIS Elements acquisition software | Nikon | Custom order | |
| Ethanol | Fisher Scientific | BP2818-4 | |
| Fetal bovine serum (FBS) | ThermoFisher | 16000044 | |
| Forceps, Dumont #7, curved | Fine Science Tools | 11274-20 | |
| Forceps, Dumont #5, straight | Fine Science Tools | 11254-20 | |
| Gloves (small, medium, large) | Microflex | N191, N192, N193 | |
| Gloves, MAPA Temp-Ice 700 Thermal (for handling dry ice) | Fisher Scientific | 19-046-563 | |
| Hemocytometer | Daigger | EF16034F EA | |
| Incubator, cell culture | Eppendorf | Galaxy 170 S | |
| Labelling tape | Fisher Scientific | 159015R | |
| Marking pen, Sharpie (ultra-fine) | Staples | 642736 | |
| Mice, immunodeficient (Nu/J) | Jackson Laboratory | 2019 | |
| Microcentrifuge, accuSpin Micro17 | Fisher Scientific | 13-100-675 | |
| Microcentrifgue tubes, Eppendorf tubes (1.5 mL) | Axygen | MCT-150-C | |
| Microscope slide box | Fisher Scientific | 50-751-4983 | |
| Needle, 27 gauge | Becton-Dickinson | 752 0071 | |
| Paintbrush | Grainger | 39AL12 | |
| Paper towels | Staples | 33550 | |
| Paraformaldehyde | Acros Organics | 416785000 | |
| Penicillin/Streptomycin | Gibco | 15140122 | |
| Perforated spoon, 15 mm diameter, 135 mm length | Roboz Surgical Instrument Co. | RS-6162 | |
| Phosphate buffered saline (PBS) | Fisher Scientific | BP3991 | |
| Pipet tips (10 μL) | Fisher Scientific | 02-707-438 | |
| Pipet tips (200 μL) | Fisher Scientific | 02-707-411 | |
| Pipet tips (1000 μL) | Fisher Scientific | 02-707-403 | |
| Pipets, serological (10 mL) | VWR | 89130-910 | |
| Pippetor, Gilson P2 | Daigger | EF9930A | |
| Pipettor Starter Kit, Gilson (2-10 μL, 20-200 μL, 200-1000 μL) | Daigger | EF9931A | |
| Platform shaker - orbital, benchtop | Cole-Parmer | EW-51710-23 | |
| Positively-charged microscope slides, Superfrost | Fisher Scientific | 12-550-15 | |
| Scalpel, size 10, Surgical Design, Inc. | Fisher Scientific | 22-079-707 | |
| Scissors, surgical - sharp, curved | Fine Science Tools | 14005-12 | |
| Software for image analysis, Nikon Elements | Nikon | Custom order | |
| Software for image analysis, ImageJ (FIJI) | National Institutes of Health | n/a | Download online (free) |
| Specimen disc 30 mm (chuck holder), cryostat accessory | Leica Biosystems | 14047740044 | |
| Staining tray, 245 mm BioAssay Dish | Corning | 431111 | |
| Syringe, 1 cc | Becton-Dickinson | 309623 | |
| Tape, laboratory, 19 mm width | Fisher Scientific | 15-901-5R | |
| Timer | Fisher Scientific | 14-649-17 | |
| Tissue culture dish, 100 x 15 mm diameter | Fisher Scientific | 08-757-100D | |
| Tissue culture flask, 225 cm2 | ThermoFisher | 159933 | |
| Tissue culture plate, 24-well | Becton-Dickinson | 353226 | |
| Tissue embedding mold, stainless steel | Tissue Tek | 4161 | |
| Tissue Freezing Medium, Optimal Cutting Temperature (OCT) | Fisher Scientific | 4585 | |
| Trypsin-EDTA (ethylenediaminetetraacetic acid), 0.25% | Gibco | 25200072 | |
| Water bath, Precision GP 2S | ThermoFisher | TSGP2S |
Referenzen
- Pavlova, N. N., Thompson, C. B. The emerging hallmarks of cancer metabolism. Cell Metabolism. 23 (1), 27-47 (2016).
- Pellegatti, P., et al. Increased level of extracellular ATP at tumor sites: in vivo imaging with plasma membrane luciferase. PLoS ONE. 3, 25992008(2008).
- Falzoni, S., Donvito, G., Di Virgilio, F. Detecting adenosine triphosphate in the pericellular space. Interface Focus. 3 (3), 20120101(2013).
- Michaud, M., et al. Autophagy-dependent anticancer immune responses induced by chemotherapeutic agents in mice. Science. 334, 1573-1577 (2011).
- Wilhelm, K., et al. Graft-versus-host disease is enhanced by extracellular ATP activating P2X7R. Nature Medicine. 16, 1434-1438 (2010).
- Vander Heiden, M. G., Cantley, L. C., Thompson, C. B. Understanding the Warburg effect: the metabolic requirements of cell proliferation. Science. 324, 1029-1033 (2009).
- Cairns, R. A., Harris, I. S., Mak, T. W. Regulation of cancer cell metabolism. Nature Reviews Cancer. 11, 85-95 (2011).
- Chen, X., Qian, Y., Wu, S. The Warburg effect: evolving interpretations of an established concept. Free Radical Biology & Medicine. 79, 253-263 (2015).
- Wang, X., et al. Extracellular ATP, as an energy and phosphorylating molecule, induces different types of drug resistances in cancer cells through ATP internalization and intracellular ATP level increase. Oncotarget. 8 (5), 87860-87877 (2017).
- Patel, A., et al. ATP as a biological hydrotrope. Science. 356, 753-756 (2017).
- Qian, Y., et al. Extracellular ATP is internalized by macropinocytosis and induces intracellular ATP increase and drug resistance in cancer cells. Cancer Letters. 351, 242-251 (2014).
- Qian, Y., Wang, X., Li, Y., Cao, Y., Chen, X. Extracellular ATP a new player in cancer metabolism: NSCLC cells internalize ATP in vitro and in vivo using multiple endocytic mechanisms. Molecular Cancer Research. 14, 1087-1096 (2016).
- Commisso, C., et al. Macropinocytosis of protein is an amino acid supply route in Ras-transformed cells. Nature. 497, 633-637 (2013).
- Li, L., et al. The effect of the size of fluorescent dextran on its endocytic pathway. Cell Biology International. 39, 531-539 (2015).
- Yanagawa, Y., Matsumoto, M., Togashi, H. Enhanced dendritic cell antigen uptake via alpha2 adrenoceptor-mediated PI3K activation following brief exposure to noradrenaline. Journal of Immunology. 185, 5762-5768 (2010).
- Hoppe, H. C., et al. Antimalarial quinolines and artemisinin inhibit endocytosis in Plasmodium falciparum. Antimicrobial Agents & Chemotherapy. 48, 2370-2378 (2004).
- Chaudry, I. H. Does ATP cross the cell plasma membrane. Yale Journal of Biology & Medicine. 55, 1-10 (1982).
- Pant, H. C., Terakawa, S., Yoshioka, T., Tasaki, I., Gainer, H. Evidence for the utilization of extracellular [gamma-32P]ATP for the phosphorylation of intracellular proteins in the squid giant axon. Biochimica et Biophysica Acta. 582, 107-114 (1979).
- Chaudry, I. H., Baue, A. E. Further evidence for ATP uptake by rat tissues. Biochimica et Biophysica Acta. 628, 336-342 (1980).
- Koppenol, W. H., Bounds, P. L., Dang, C. V. Otto Warburg's contributions to current concepts of cancer metabolism. Nature Reviews Cancer. 11, 325-337 (2011).
- Dang, C. V. Links between metabolism and cancer. Genes & Development. 26, 877-890 (2012).
- Israelsen, W. J., Vander Heiden, M. G. ATP consumption promotes cancer metabolism. Cell. 143, 669-671 (2010).
- Koster, J. C., Permutt, M. A., Nichols, C. G. Diabetes and insulin secretion: the ATP-sensitive K+ channel (K ATP) connection. Diabetes. 54, 3065-3072 (2005).
- Szendroedi, J., et al. Muscle mitochondrial ATP synthesis and glucose transport/phosphorylation in type 2 diabetes. PLoS Medicine. 4, 154(2007).
- Miyamoto, S., et al. Mass spectrometry imaging reveals elevated glomerular ATP/AMP in diabetes/obesity and identifies sphingomyelin as a possible mediator. EBioMedicine. 7, 121-134 (2016).
Access restricted. Please log in or start a trial to view this content.
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten