È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Microscopia a fluorescenza per l'internalizzazione dell'ATP mediata dalla macropinocitosi in cellule tumorali umane e topi xenotrapiantati da tumore
* Questi autori hanno contribuito in egual misura
In questo articolo
Riepilogo
Abbiamo sviluppato un metodo riproducibile per visualizzare l'internalizzazione dell'adenosina trifosfato fluorescente non idrolizzabile (ATP), un surrogato di ATP, ad alta risoluzione cellulare. Abbiamo convalidato il nostro metodo utilizzando saggi indipendenti in vitro e in vivo: linee cellulari tumorali umane e topi immunodeficienti xenografati con tessuto tumorale umano.
Abstract
L'adenosina trifosfato (ATP), compreso l'ATP extracellulare (eATP), ha dimostrato di svolgere ruoli significativi in vari aspetti della tumorigenesi, come la resistenza ai farmaci, la transizione epiteliale-mesenchimale (EMT) e le metastasi. L'eATP intratumorale è da10 3 a10 4 volte superiore in concentrazione rispetto ai tessuti normali. Mentre l'eATP funziona come un messaggero per attivare la segnalazione purinergica per l'induzione EMT, è anche internalizzato dalle cellule tumorali attraverso macropinocitosi sovraregolata, un tipo specifico di endocitosi, per svolgere un'ampia varietà di funzioni biologiche. Queste funzioni includono la fornitura di energia alle reazioni biochimiche che richiedono ATP, la donazione di gruppi fosfato durante la trasduzione del segnale e la facilitazione o l'accelerazione dell'espressione genica come cofattore trascrizionale. L'ATP è prontamente disponibile e il suo studio sul cancro e in altri campi aumenterà senza dubbio. Tuttavia, lo studio eATP rimane in una fase iniziale e le domande irrisolte rimangono senza risposta prima che le attività importanti e versatili svolte dall'eATP e dall'ATP intracellulare interiorizzato possano essere completamente svelate.
I contributi dei laboratori di questi autori a questi primi studi eATP includono l'imaging microscopico di ATP fluorescente non idrolizzabile, accoppiato con dextrans fluorescenti ad alto e basso peso molecolare, che fungono da traccianti di macropinocitosi ed endocitosi, nonché vari inibitori dell'endocitosi, per monitorare e caratterizzare il processo di internalizzazione dell'eATP. Questa modalità di imaging è stata applicata a linee cellulari tumorali e a topi immunodeficienti, xenotrapiantati con tumori tumorali umani, per studiare l'internalizzazione dell'eATP in vitro e in vivo. Questo articolo descrive questi protocolli in vitro e in vivo, con particolare attenzione alla modifica e alla messa a punto delle condizioni di analisi in modo che i saggi di internalizzazione eATP mediati da macropinocitosi / endocitosi possano essere eseguiti con successo in diversi sistemi.
Introduzione
L'assorbimento opportunistico di nutrienti extracellulari intratumorali (cioè) è stato recentemente nominato un segno distintivo chiave per il metabolismo del cancro1. Uno di questi importanti nutrienti è l'ATP, in quanto la concentrazione di ieATP è10 3 e10 4 volte superiore a quella che si trova nei tessuti normali, nell'intervallo da diverse centinaia di μM a bassi mM2,3,4,5. Come molecola chiave di energia e segnalazione, l'ATP svolge un ruolo centrale nel metabolismo cellulare nelle cellule cancerose e sane6,7,8. L'ATP extracellulare non è solo coinvolto nella crescita delle cellule tumorali, ma promuove anche la resistenza ai farmaci9. Funzioni precedentemente non riconosciute dell'ATP, come l'attività idrotropica, sono state recentemente identificate, implicando così il coinvolgimento dell'ATP in malattie come l'Alzheimer10. In effetti, sembra che la nostra comprensione dell'ATP e delle sue funzioni nelle cellule tumorali, nelle cellule sane e in altre cellule malate sia tutt'altro che completa. Tuttavia, a causa dell'instabilità dell'ATP e degli alti tassi di turnover nelle cellule, è tecnicamente difficile monitorare il movimento dell'ATP attraverso la membrana cellulare e nella cellula.
Per affrontare questo problema e soddisfare le esigenze di questa area di ricerca, è stato sviluppato un metodo in cui l'ATP fluorescente non idrolizzabile (NHF-ATP) (Figura 1) è stato utilizzato come surrogato per visualizzare l'internalizzazione dell'ATP e osservare la localizzazione spaziale intracellulare dell'ATP internalizzato, sia in vitro che in vivo11,12 . NHF-ATP ha dimostrato di sostituire l'ATP endogeno per studiare il movimento dell'ATP attraverso le membrane cellulari animali, sia nelle linee cellulari tumorali che nel tessuto tumorale umano xenotrapiantato su topi immunodeficienti11,12. Inoltre, la somministrazione di inibitori della macropinocitosi alle cellule ha bloccato l'internalizzazione dell'eATP, suggerendo che l'assorbimento intracellulare di eATP comporta un meccanismo macropinocitotico9,11,12. Questo protocollo consente il colabeling immunobased contro proteine cellulari specifiche e quindi l'identificazione di quale tipo di cellula interiorizza NHF-ATP. Utilizzando xenotrapianti tumorali in vivo e microscopia ad alta risoluzione, NHF-ATP può essere visualizzato spazialmente attraverso il campione di tessuto e anche all'interno di una singola cellula. Questi metodi consentono anche analisi quantitative, come la percentuale di assorbimento cellulare, il numero di vescicole macropinocitotiche e la cinetica di internalizzazione. Questo articolo descrive in dettaglio come NHF-ATP, lavorando da solo o insieme adextrans13, 14,15,16fluorescenti endocitosi-traccianti, può essere utilizzato in diversi contesti sperimentali per studiare l'internalizzazione e la localizzazione intracellulare dell'ATP, a seguito dell'internalizzazione nelle cellule.

Figura 1: Strutture di ATP fluorescente non idrolizzabile e tetrametilrodramina etichettate come destrano fluorescente ad alto peso molecolare. (A) Struttura di NHF-ATP. (B) Rappresentazione schematica dell'HMWFD. Abbreviazioni: ATP = adenosina trifosfato; NHF-ATP = ATP fluorescente non idrolizzabile; TMR = tetrametilrodramina; HMWFD = destrano fluorescente ad alto peso molecolare. Fare clic qui per visualizzare una versione più grande di questa figura.
Access restricted. Please log in or start a trial to view this content.
Protocollo
Tutte le procedure qui riportate sono state eseguite in conformità con la IACUC dell'Università dell'Ohio e con il NIH.
1. Selezione di ATP fluorescente non idrolizzabile (NHF-ATP) e dextrans
- Selezionare un NHF-ATP coniugato con fluoroforo (Figura 1A) e traccianti di endocitosi, dextrans fluorescenti ad alto e basso peso molecolare (TMR-HMWFD e TMR-LMWFD) (Figura 1B), in base alle lunghezze d'onda di emissione preferite (ad esempio, sistema di imaging dotato di filtri appropriati) e allo specifico processo di endocitosi da studiare.
2. Studi di localizzazione dell'ATP, in vitro (Figura 2)
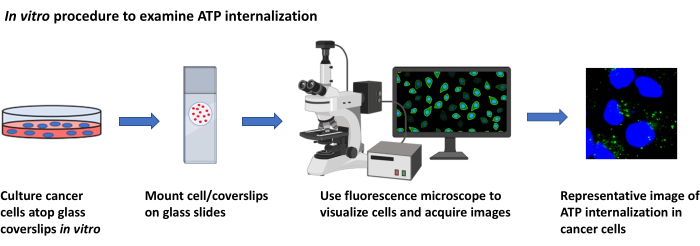
Figura 2: Procedura in vitro per esaminare l'internalizzazione dell'ATP. Rappresentazione schematica del protocollo per visualizzare l'internalizzazione dell'ATP extracellulare in cellule tumorali in coltura utilizzando la microscopia a fluorescenza. Fare clic qui per visualizzare una versione più grande di questa figura.
- Coltura cellulare e preparazione delle cellule
NOTA: Eseguire la coltura cellulare in condizioni sterili in una cappa di coltura tissutale.- Preparare il Modified Eagle Medium (DMEM) di Dulbecco, contenente il 10% (v/v) di siero bovino fetale (FBS) e l'1% (v/v) di penicillina/streptomicina (di seguito denominata DMEM/FBS), soluzione salina sterile tamponata con fosfato (PBS) e 0,25% di tripsina/acido etilendiamminotetraacetico (EDTA), in un bagno d'acqua a 37 °C.
- Coltivare cellule tumorali umane in DMEM/ FBS in un piatto di coltura tissutale da 100 mm. Mantenere le celle in un incubatore impostato a 37 °C con un'atmosfera di CO2 del 5%.
- Quando le cellule raggiungono la confluenza, passano le cellule rimuovendo prima il terreno di coltura. Quindi, risciacquare il piatto con 5 ml di PBS sterile, rimuovere il PBS e aggiungere 3 ml di tripsina allo 0,25%. Incubare a 37 °C in un'atmosfera di CO2 al 5% per 5 min.
- Recupera il piatto, quindi aggiungi 6 ml di DMEM / FBS per interrompere la tripsinizzazione. Trasferire le celle in sospensione in un tubo conico da 15 mL e centrifugare a 800 × g per 5 minuti per pellettizzare le celle.
- Dopo la centrifugazione, aspirare il surnatante e utilizzare 10 ml di DMEM/FBS per risospescere il pellet cellulare mediante pipettaggio.
- Contare la densità cellulare e la vitalità usando un emocitometro. Utilizzare DMEM/FBS per diluire la sospensione cellulare ad una densità di ~7,5 × 104 celle/ml.
- Preparazione di coverslips e cellule di semina
- Lavare i coperchi da 12 mm con etanolo al 70% e pulirli accuratamente con delicate salviette da lavoro. Sterilizzare i coverslip e un paio di pinze tramite autoclave.
- In una cappa di coltura di tessuto, utilizzare una pinza per posizionare una copertura in ciascun pozzetto di una piastra di coltura tissutale a 24 pozzetti.
NOTA: Successivamente, il coverslip, con le cellule, verrà montato direttamente su un vetrino per microscopio per l'imaging. - Erogare 300 μL della sospensione cellulare (cellule in DMEM/FBS), ad una densità di semina di ~2,5 × 104 cellule per pozzetto, nella piastra a 24 pozzetti contenente i coverslip sterilizzati. Incubare in condizioni sterili a 37 °C con un flusso di CO2 del 5%.
- Fame di cellule
- Ventiquattro ore dopo la semina, rimuovere il DMEM/FBS da ciascun pozzetto. Aggiungere immediatamente 300 μL di DMEM senza siero preriscaldato in ciascun pozzetto per affamare le cellule per 15-18 ore per indurre l'assorbimento di nutrienti extracellulari.
NOTA: Il periodo di fame di 15-18 ore è un parametro critico.
- Ventiquattro ore dopo la semina, rimuovere il DMEM/FBS da ciascun pozzetto. Aggiungere immediatamente 300 μL di DMEM senza siero preriscaldato in ciascun pozzetto per affamare le cellule per 15-18 ore per indurre l'assorbimento di nutrienti extracellulari.
- Preparazione di soluzioni NHF-ATP e HMWFD/LMWFD
- Utilizzare una bilancia analitica per pesare TMR-destrano fluorescente ad alto peso molecolare (70 kDa) (TMR-HMWFD, 1 mg/mL), un tracciante per la visualizzazione di macropinosomi, o NHF-ATP (10 μmol/L) in DMEM privo di siero in un tubo microcentrifuga da 1,5 mL. Posizionare i tubi, al riparo dalla luce, a bagnomaria a 37 °C per 15 min.
- Centrifugare a 12.000 × g per 5 minuti a temperatura ambiente. Trasferire con cura il surnatante trasparente in un nuovo tubo microcentrifuga da 1,5 ml, lasciando intatti eventuali pellet o detriti per rimuovere i cristalli indissolubili.
- Aggiungere le soluzioni del passaggio 2.4.1 alle cellule in ciascun pozzetto e incubare le cellule per 30 minuti a 37 °C.
NOTA: Se le soluzioni hmWFD e NHF-ATP devono essere miscelate per la co-incubazione con le cellule, preparare entrambe le soluzioni a 2 volte le concentrazioni finali. Le soluzioni saranno miscelate in seguito in un rapporto 1:1 per ottenere le concentrazioni di lavoro accurate finali. Evitare la luce in quanto i reagenti sono sensibili alla luce.
- Trattamento delle cellule e fissazione
- In una fresca piastra a 24 pozzetti, erogare 500 μL di PBS preriscaldato in ciascuno dei cinque pozzetti.
- Dopo l'incubazione cellulare, raccogliere con cura ogni coverslip usando una pinza. Risciacquare ogni coverslip immergendolo in 500 μL di PBS preriscaldato. Ripetere l'operazione cinque volte utilizzando i cinque pozzetti riempiti di PBS.
NOTA: Il lavaggio delicato delle cellule su coperchi è fondamentale per il successo di questo esperimento. - Dopo il lavaggio finale del PBS, picchiettare il coperchio su una delicata salvietta per assorbire il PBS extra e trasferire immediatamente il coperchio a freddo (4 °C) 3,7% di formaldeide, precaricato in una piastra a 24 pozzetti. Fissare le celle per 15 minuti a temperatura ambiente.
- Mentre le cellule vengono riparate, il microscopio pre-pulito scorre con il 70% di etanolo. Rimuovere i coperchi dai pozzetti e montarli sui vetrini, utilizzando 5 μL di mezzo di montaggio acquoso contenente la macchia nucleare 4′,6-diamidino-2-fenilindolo (DAPI), per coverslip. Tamponare delicatamente il PBS in eccesso con un tovagliolo di carta o una delicata salvietta.
- Microscopia a fluorescenza e acquisizione di immagini
- Da due a 24 ore dopo i passaggi precedenti, acquisire immagini di cellule e HMWFD e / o NHF-ATP interiorizzati utilizzando un sistema di imaging a epifluorescenza e un software di acquisizione dati.
NOTA: questa sottosezione descrive i passaggi per acquisire immagini utilizzando un microscopio Nikon NiU, dotato di funzionalità di imaging a epifluorescenza, e il software Nikon NIS Elements. Tuttavia, possono essere utilizzati altri sistemi di imaging e software di acquisizione comparabili. Seguire le istruzioni per l'uso fornite dal produttore.- Posizionare il vetrino sul palco di un microscopio a epifluorescenza verticale in modalità binoculare. Accedere al programma di imaging.
- Selezionare l'obiettivo 10x, regolare lo stage per definire la messa a fuoco ed eseguire la scansione della diapositiva da sinistra a destra in modo serpentino per identificare le regioni di interesse.
NOTA: l'identificazione delle regioni di interesse varierà tra i tipi di cellule, con alcune linee cellulari / tipi di cancro che mostrano gradi diversi e distinti di assorbimento di TMR-HMWFD e / o NHF-ATP. - Selezionare l'obiettivo 40x e passare dalla modalità binoculare alla modalità di acquisizione dell'immagine, utilizzando l'interruttore sul microscopio.
- Fare clic sull'icona Live Quality sul programma di imaging per visualizzare e successivamente acquisire immagini.
- Utilizzando il pannello OC sulla barra degli strumenti Annotazioni e misurazioni, definite i parametri di esposizione per ogni cubo di filtro o canale fluorescente.
NOTA: selezionare il tempo di esposizione appropriato per ciascun canale, poiché le intensità del segnale sono diverse. Ad esempio, selezionare un tempo di esposizione di 200 ms per DAPI, 2 s per HMWFD e 4 s per NHF-ATP. Una volta determinato il tempo di esposizione per canale, utilizzare questa impostazione per tutte le immagini, per canale, con trattamento o condizioni diverse. - Una volta impostate le impostazioni di esposizione per ciascun canale, utilizzare la barra degli strumenti di acquisizione multicanale per acquisire un'immagine a 3 canali con le impostazioni di esposizione definite.
NOTA: l'acquisizione di immagini tramite la modalità di acquisizione ND multicanale consente l'acquisizione automatica delle immagini per ciascun canale dello stesso campo visivo. L'otturatore si chiude automaticamente tra i cambi di torretta. - In alternativa, acquisire manualmente le immagini multicanale passando da un cubo di filtro all'altro, impostando il tempo di esposizione, chiudendo/aprendo l'otturatore tra l'acquisizione dell'immagine per ciascun canale e sovrapponendo ogni immagine scattata per i singoli canali.
NOTA: la modalità di acquisizione ND automatizza questo processo e fornisce immagini unite. - Salvare l'immagine come file .nd2 (il formato Nikon Elements salva i metadati). Salvare i file TIF, incluse l'immagine del canale unito e le singole immagini del canale.
NOTA: i file TIF possono essere utilizzati con una selezione più ampia di applicazioni software. - Utilizzare la funzionalità Conteggio oggetti sulla barra degli strumenti Analisi per contare il numero di celle NHF-ATP-, TMR-HMWFD- e/o TMR-LMWFD-positive in un file di immagine .nd2 salvato.
- Esporta i dati in un foglio di calcolo tramite il programma di analisi.
- Da due a 24 ore dopo i passaggi precedenti, acquisire immagini di cellule e HMWFD e / o NHF-ATP interiorizzati utilizzando un sistema di imaging a epifluorescenza e un software di acquisizione dati.
- Quantificazione e analisi dei dati
- Per ogni condizione analizzata, immagine da 50 a 100 celle per la quantificazione. Utilizzando il software di analisi dei dati (software incluso nel sistema di imaging a epifluorescenza o altro software), contare e calcolare il numero medio di vescicole fluorescenti per cella.
- Utilizzare metodi statistici appropriati per analizzare i risultati quantificati.
3. Internalizzazione dell'ATP nei tumori, ex vivo (Figura 3)

Figura 3: Procedura in vivo per esaminare l'internalizzazione dell'ATP. Rappresentazione schematica del protocollo per visualizzare l'internalizzazione dell'ATP extracellulare negli xenotrapianti tumorali mediante criosezione e microscopia a fluorescenza. Fare clic qui per visualizzare una versione più grande di questa figura.
- Preparazione di colture cellulari per l'impianto
- Far crescere le cellule tumorali all'80% di confluenza a 37 °C in un matraccio di225 cm 2, utilizzando DMEM integrato con FBS, ad una concentrazione finale del 10% (v/v) e penicillina/streptomicina all'1% (v/v).
- Lavare le cellule due volte con 10 ml di PBS. Preriscaldare 0,25% tripsina/EDTA a 37 °C. Aggiungere 8 mL di tripsina/EDTA e incubare a 37 °C per 2 min.
- Una volta che le cellule iniziano a staccarsi dal fondo del pallone, utilizzare una pipetta sierologica sterile da 10 ml per aggiungere 8 mL di DMEM/FBS. Aspirare due volte per rimuovere eventuali cellule aderenti. Utilizzare la pipetta per trasferire le celle staccate dal pallone in un tubo conico da 50 ml.
- Aggiungere 10 mL di DMEM/FBS utilizzando una pipetta da 10 mL e raccogliere tutte le celle flottanti rimanenti nello stesso tubo conico da 50 mL.
- Centrifugare la sospensione cellulare a 600 × g,4 °C per 4 min. Rimuovere il surnatante e risospese le cellule in 1 mL di PBS ghiacciato.
- Contare la densità cellulare usando un emocitometro. Mantenere la sospensione cellulare sul ghiaccio durante il conteggio.
- Centrifugare la sospensione cellulare a 600 × g,4 °C per 4 min. Rimuovere il surnatante e sospendere le cellule in PBS ghiacciato in modo tale che la densità cellulare diventi 5 × 106 cellule per 100 μL di PBS. Trasferire la sospensione cellulare in un tubo microcentrifuga da 1,5 mL.
- Iniezione sottocutanea di cellule tumorali per lo sviluppo del tumore allo xenotrapianto
- Utilizzare una siringa priva di lattice (1 mL) con un ago di scorrimento di precisione (ago da 27 G) per l'iniezione di cellule tumorali.
- Trasferire la sospensione cellulare (5 × 106 in 100 μL di PBS) in un tubo microcentrifuga da 1,5 ml. Aspirare le cellule nella siringa.
- Selezionare un sito di iniezione sul fianco di un topo immunodeficiente (nudo) e pulire delicatamente la pelle utilizzando il 75% di etanolo. Pulire l'etanolo in eccesso con una delicata salvietta.
- Per l'iniezione sottocutanea, tenere l'ago con un angolo di circa 10° rispetto alla pelle. Inserire la punta dell'ago, smussata verso l'alto, appena sotto la pelle, in modo che solo 1-2 mm dell'ago siano visibili all'esterno della pelle. Erogare le cellule dalla siringa lentamente per circa 10 s.
- Dopo aver iniettato l'intero volume, continuare a tenere l'ago in posizione per 3-5 s, quindi prelevare l'ago e utilizzare un dito per applicare una pressione delicata ma decisa sul sito di iniezione per altri 3-5 secondi per evitare perdite del contenuto iniettato.
- Monitorare e misurare la crescita tumorale utilizzando pinze vernier fino a quando i tumori raggiungono un volume di 200-500 mm3.
- Preparazione di soluzioni HMWFD e NHF-ATP da utilizzare dopo la resezione tumorale
- Sciogliere 300 μL di 16 mg/mL di HMWFD in DMEM (terreno di coltura) privo di siero, incubare a bagnomaria a 37 °C per 30 minuti e centrifugare a 12.000 × g per 5 minuti come descritto sopra. Trasferire la soluzione in un tubo microcentrifuga da 1,5 mL.
- Aggiungere 40 μL di stock analogico NHF-ATP (1 mM) a 160 μL di DMEM privo di siero per preparare una soluzione NHF-ATP da 0,2 mM.
- Preparazione di pozzi sperimentali
NOTA: Questo disegno sperimentale testerà l'internalizzazione intracellulare di HMWFD + NHF-ATP, indicativa dell'assorbimento da parte dei macropinosomi.- Preparare i pozzetti come segue: Pozzo #1, Controllo: 200 μL di DMEM senza siero; Bene #2, Controllo: 100 μL di 16 mg/mL LMWFD + 100 μL di DMEM siero-libero = 200 μL di 8 mg/mL LMWFD; Bene #3, Controllo: 100 μL di 0,2 mM NHF-ATP + 100 μL di DMEM senza siero = 200 μL di 0,1 mM NHF-ATP; Bene #4; Sperimentale: 100 μL di 16 mg/mL HMWFD + 100 μL di 0,2mM NHF-ATP = 200 μL di 0,1 mM NHF-ATP e 8 mg/mL HMWFD.
- Preparazione di tessuti tumorali
- Eutanasia del topo per lussazione cervicale o secondo il protocollo approvato dalla IACUC.
- Utilizzare un bisturi di taglia 10 per tagliare i tumori isolati ad uno spessore di ~ 500-1.000 μm.
- Incubare le fette tumorali in DMEM senza siero integrato con 100 μM NHF-ATP e/o 8 mg/mL H/LMWFD in tubi microcentrifuga per 40 minuti a 37 °C con flusso di CO2 al 5%.
NOTA: Dopo l'incubazione, il metabolismo del tessuto tumorale fa cambiare il colore del mezzo. - Risciacquare i fazzoletti in PBS preriscaldato a 37 °C (2 mL per ogni risciacquo in una piastra a 24 pozzetti).
- Trasferire il tessuto in una nuova piastra a 24 pozzetti con PBS fresco preriscaldato, risciacquare e ripetere quattro volte con un leggero scuotimento.
- Cryo-embedding (preparazione di blocchi di tessuto congelati)
- Preparare etichette di identificazione per ogni tumore da raccogliere. Tagliare un pezzo di nastro da laboratorio di 2 cm e piegare a metà, i lati adesivi insieme, longitudinalmente. Utilizzare una penna di marcatura per etichettare il tag, ad esempio con un numero di identificazione del mouse / tumore.
- Preparare stampi incorporanti posizionando stampi in tessuto di acciaio inossidabile direttamente sul ghiaccio secco.
NOTA: il ghiaccio secco può causare congelamento, ustioni e asfissia. Indossare guanti isolati quando si maneggia ghiaccio secco. Utilizzare ghiaccio secco in un'area ben ventilata. Non conservare il ghiaccio secco in un contenitore ben sigillato. Invece, conservare in un contenitore (come un dispositivo di raffreddamento in polistirolo) che consente al gas di fuoriuscire. - Mentre lo stampo si raffredda, posizionare un piccolo pool di mezzo di congelamento dei tessuti in una piastra di coltura tissutale da 10 mm. Assicurarsi che il volume sia sufficiente per immergere il tessuto tumorale che verrà raccolto.
- Utilizzare un cucchiaio perforato per raccogliere il tessuto tumorale resecato e posizionare immediatamente il tessuto in un mezzo di congelamento, assicurandosi che il tessuto sia sommerso. Usando il cucchiaio perforato, arrotolare delicatamente il tessuto nel mezzo di congelamento, assicurandosi che il mezzo stia bagnando tutte le superfici del tessuto.
- Spostare con attenzione il tessuto nello stampo di incorporamento contenente il mezzo di congelamento. Posizionare verticalmente l'etichetta dell'etichetta corrispondente nel mezzo di congelamento/ stampo per congelare in posizione. Assicurarsi che l'etichetta scritta sia visibile all'esterno del supporto.
- Quando il congelamento è completo (il mezzo di congelamento diventa bianco opaco), rimuovere il blocco di tessuto dallo stampo, posizionarlo su ghiaccio secco e ripetere per ogni tumore. Conservare i blocchi di tessuto a -80 °C per diversi mesi prima della procedura di criosezione.
- Preparazione di vetrini di campioni di tessuti
- Per massimizzare la possibilità di trovare cellule positive all'internalizzazione e avere regioni tissutali più rappresentative, raccogliere criosezioni seriali a -18--20 °C utilizzando un criostato.
- Gli strumenti criostatali Prechill (lama, lama di rasoio, piastra antirollio, porta mandrino in tessuto, pennello) ed equilibrano i blocchi di tessuto tumorale posizionandoli in una camera criostata a -18--20 °C. Impostare l'angolo del supporto della lama su 5-10°. Tagliare con cura il blocco di tessuto, se necessario, con una lama di rasoio e montarlo sul supporto del mandrino usando il mezzo di congelamento del tessuto come "colla".
- Bloccare il porta mandrino in posizione verticale sull'unità microtomatica, che avanza alla distanza impostata (ad esempio, 10 μm) ad ogni giro della manovella manuale. Posizionare la piastra antirollio in modo che si appoggi appena sopra l'altezza della lama. Per prevenire l'arricciamento dei tessuti prima di far avanzare il microtomo, far scorrere con attenzione il pollice sul bordo inferiore del blocco di tessuto.
- Mentre il microtomo avanza e la sezione del tessuto cade sulla piastra metallica, utilizzare un pennello per guidare la sezione del tessuto e srotolare il tessuto, se necessario.
- Passare il mouse sul vetrino del microscopio sopra la sezione del tessuto senza toccare in modo che la sezione sia attratta dalla diapositiva.
NOTA: le lame criostatali (di alto profilo, usa e getta) sono estremamente affilate e possono causare gravi lesioni. Prestare attenzione quando si maneggiano le lame e si aziona il criostato. Utilizzare una protezione per lame, se disponibile. È necessaria una formazione adeguata. - Tagliare il tumore in sezioni di 10 μm di spessore. Trasferire immediatamente le sezioni affettate su un vetrino per microscopio caricato positivamente.
NOTA: per le sezioni seriali, raccogliere prima una sezione spessa 10 μm nell'angolo superiore sinistro di ciascuna delle otto diapositive caricate positivamente. Avanzare il criostato attraverso i successivi 100-200 μm di tessuto e scartare il tessuto. Trasferire immediatamente tutte le sezioni affettate su vetrini per microscopio in vetro. - Quindi, raccogliere un'altra sezione di 10 μm di spessore, accanto alla sezione di tessuto precedentemente posizionata, per ciascuna delle otto diapositive. Ripetere questo processo di raccolta seriale fino a quando ciascuna delle otto diapositive contiene otto sezioni di tessuto, ciascuna a 100-200 μm di distanza. Mantenere le sezioni di tessuto al buio per preservare la fluorescenza.
NOTA: le sezioni di tessuto sui vetrini possono essere conservate in una scatola di diapositive a -80 °C per diversi mesi.
- Per massimizzare la possibilità di trovare cellule positive all'internalizzazione e avere regioni tissutali più rappresentative, raccogliere criosezioni seriali a -18--20 °C utilizzando un criostato.
- Fissazione di vetrini di tessuto
- PASSAGGIO CRITICO: Fissare le sezioni di tessuto in etanolo al 95% da -18 a -20 °C a 5 min.
- Lavare la sezione fissa per 5 minuti con PBS a temperatura ambiente, quindi montare le sezioni tumorali fisse sotto un coperchio di vetro utilizzando 10 μL di un mezzo di montaggio acquoso con DAPI.
- Da dodici a 24 ore dopo il montaggio, esaminare le sezioni tumorali fisse mediante microscopia a fluorescenza e acquisire immagini, come descritto per le cellule coltivate sopra.
- Microscopia a fluorescenza e acquisizione di immagini
- Identificare le regioni di interesse e acquisire immagini, come descritto nella sezione 2.6.
- Quantificazione e analisi dei dati
- Quantificare le cellule e applicare analisi statistiche appropriate, come nel paragrafo 2.7.
4. Internalizzazione dell'ATP nei tumori, in vivo
- Preparare colture cellulari per l'impianto come descritto al paragrafo 3.1.
- Iniezione sottocutanea di cellule tumorali per lo sviluppo del tumore allo xenotrapianto
- Generare tumori xenografati come descritto nel paragrafo 3.2.
- Iniezione di ATP e/o destrano nei tumori dello xenotrapianto
- Preparare le soluzioni di trattamento di DMEM (veicolo) o 8 mg/mL HMWFD o LMWFD, con o senza NHF-ATP (100 μM) in DMEM, come descritto sopra.
- Utilizzare una siringa da 1 mL per raccogliere 50 μL di una soluzione di trattamento e iniettare la soluzione direttamente in ciascun tumore allo xenotrapianto. Ripetere la procedura per quattro repliche biologiche di ciascun trattamento.
- Raccolta dei tessuti e crio-incorporamento
- Preparare etichette di identificazione per ogni tumore da raccogliere. Tagliare un pezzo di nastro da laboratorio di 2 cm e piegare a metà, i lati adesivi insieme, longitudinalmente. Utilizzare una penna di marcatura per etichettare il tag, ad esempio con un numero di identificazione del mouse / tumore.
- Circa 5 minuti dopo l'iniezione, eutanasia del topo per lussazione cervicale o secondo il protocollo approvato IACUC.
- Usando un bisturi di taglia 10, fare un'incisione adiacente al tumore e approssimativamente perpendicolare alla direzione dell'iniezione dell'ago. Utilizzare pinze e forbici chirurgiche per resecare il tessuto tumorale dal tessuto circostante.
- Dividere il tumore in due o quattro pezzi da 1 cm2, a seconda della dimensione totale del tumore.
- Preparare gli stampi di incorporamento e incorporare il tessuto, come descritto sopra al paragrafo 3.6. Assicurarsi che il tempo di raccolta, dall'iniezione intratumorale del destrano al crio-incorporamento, non sia superiore a 7-8 minuti.
- Preparazione di vetrini di campioni di tessuti
- Raccogliere sezioni tumorali seriali, come descritto nel paragrafo 3.7.
- Fissazione di vetrini di tessuto
- Fissare il tessuto, come descritto al paragrafo 3.8.
- Microscopia a fluorescenza e acquisizione di immagini
- Identificare le regioni di interesse e acquisire immagini, come descritto nella sezione 2.6.
- Quantificazione e analisi dei dati
- Quantificare le cellule e applicare analisi statistiche appropriate, come descritto nel paragrafo 2.7.
Access restricted. Please log in or start a trial to view this content.
Risultati
Studio in vitro
L'internalizzazione intracellulare di NHF-ATP è stata dimostrata dalla co-localizzazione di NHF-ATP con HMWFD o LMWFD (Figura 4). Il successo di questa procedura si basa principalmente sull'uso di concentrazioni appropriate di NHF-ATP e dextrans e sulla determinazione del tipo o dei tipi appropriati di dextrans (polilisina vs. neutro). Ad esempio, per indagare la macropinocitosi, è stato scelto HMWFD in quanto è internalizzat...
Access restricted. Please log in or start a trial to view this content.
Discussione
È stato sviluppato un metodo per l'analisi spaziale, temporale e quantitativa dell'internalizzazione cellulare dell'ATP non idrolizzabile. Questo metodo è ampiamente applicabile per l'uso in diversi sistemi biologici, compresi vari modelli tumorigenici, per i quali forniamo istruzioni tecniche e dati rappresentativi. Per acquisire dati interpretabili durante gli studi di internalizzazione dell'ATP in vivo (sezione 4 del protocollo), è fondamentale limitare il tempo sperimentale trascorso dall'iniezione intrat...
Access restricted. Please log in or start a trial to view this content.
Divulgazioni
Gli autori non dichiarano interessi concorrenti.
Riconoscimenti
La criosezione è stata eseguita in loco presso l'Ohio University Histopathology Core. Questo lavoro è stato sostenuto in parte da fondi di start-up (Ohio University College of Arts & Sciences) a C Nielsen; NIH concede R15 CA242177-01 e premio RSAC a X Chen.
Access restricted. Please log in or start a trial to view this content.
Materiali
| Name | Company | Catalog Number | Comments |
| A549 cells, human lung epithelial, carcinoma | National Cancer Institute | n/a | Less expensive source |
| Acetone | Fisher Scientific | S25904 | |
| Aluminum foil, Reynolds | Grainger | 6CHG6 | |
| Aqueous Mounting Medium, ProLong Gold Anti-fade Reagent | ThermoFisher | P36930 | |
| ATP analog | Jena Biosciences | NK-101 | |
| Autoclave, sterilizer | Grainger | 33ZZ40 | |
| Blades, cryostat, high profile | C. L. Sturkey, Inc. | DT554550 | |
| Calipers, vernier | Grainger | 4KU77 | |
| Cell medium, Ham's Nutrient Mixture F12, serum-free | Millipore Sigma | 51651C-1000ML | |
| Centrifuge, refrigerated with swinging bucket rotor | Eppendorf | 5810R | |
| Chloroform | Acros Organics | 423555000 | |
| Conical tube, 15 mL | VWR | 21008-216 | |
| Conical tube, 50 mL | VWR | 21008-242 | |
| Coverslips, glass, 12 mm | Corning | 2975-245 | |
| Cryostat, Leica CM1950 | Leica Biosystems | CM1950 | |
| Delicate task wipe, Kim Wipes | Kimberly-Clark | 34155 | |
| Dextran, Lysine fixable, High Molecular Weight (HMW) | Invitrogen | D1818 | MW = 70,000, Tetramethylrhodamine |
| Dextran, Neutral, High Molecular Weight (HMW) | Invitrogen | D1819 | |
| Dulbecco's Modified Eagle Medium (DMEM), serum-free | Fisher Scientific | 11885076 | |
| Dry ice | Local delivery | Custom order | |
| Epifluorescent imaging system, Nikon NiU and Nikon NIS Elements acquisition software | Nikon | Custom order | |
| Ethanol | Fisher Scientific | BP2818-4 | |
| Fetal bovine serum (FBS) | ThermoFisher | 16000044 | |
| Forceps, Dumont #7, curved | Fine Science Tools | 11274-20 | |
| Forceps, Dumont #5, straight | Fine Science Tools | 11254-20 | |
| Gloves (small, medium, large) | Microflex | N191, N192, N193 | |
| Gloves, MAPA Temp-Ice 700 Thermal (for handling dry ice) | Fisher Scientific | 19-046-563 | |
| Hemocytometer | Daigger | EF16034F EA | |
| Incubator, cell culture | Eppendorf | Galaxy 170 S | |
| Labelling tape | Fisher Scientific | 159015R | |
| Marking pen, Sharpie (ultra-fine) | Staples | 642736 | |
| Mice, immunodeficient (Nu/J) | Jackson Laboratory | 2019 | |
| Microcentrifuge, accuSpin Micro17 | Fisher Scientific | 13-100-675 | |
| Microcentrifgue tubes, Eppendorf tubes (1.5 mL) | Axygen | MCT-150-C | |
| Microscope slide box | Fisher Scientific | 50-751-4983 | |
| Needle, 27 gauge | Becton-Dickinson | 752 0071 | |
| Paintbrush | Grainger | 39AL12 | |
| Paper towels | Staples | 33550 | |
| Paraformaldehyde | Acros Organics | 416785000 | |
| Penicillin/Streptomycin | Gibco | 15140122 | |
| Perforated spoon, 15 mm diameter, 135 mm length | Roboz Surgical Instrument Co. | RS-6162 | |
| Phosphate buffered saline (PBS) | Fisher Scientific | BP3991 | |
| Pipet tips (10 μL) | Fisher Scientific | 02-707-438 | |
| Pipet tips (200 μL) | Fisher Scientific | 02-707-411 | |
| Pipet tips (1000 μL) | Fisher Scientific | 02-707-403 | |
| Pipets, serological (10 mL) | VWR | 89130-910 | |
| Pippetor, Gilson P2 | Daigger | EF9930A | |
| Pipettor Starter Kit, Gilson (2-10 μL, 20-200 μL, 200-1000 μL) | Daigger | EF9931A | |
| Platform shaker - orbital, benchtop | Cole-Parmer | EW-51710-23 | |
| Positively-charged microscope slides, Superfrost | Fisher Scientific | 12-550-15 | |
| Scalpel, size 10, Surgical Design, Inc. | Fisher Scientific | 22-079-707 | |
| Scissors, surgical - sharp, curved | Fine Science Tools | 14005-12 | |
| Software for image analysis, Nikon Elements | Nikon | Custom order | |
| Software for image analysis, ImageJ (FIJI) | National Institutes of Health | n/a | Download online (free) |
| Specimen disc 30 mm (chuck holder), cryostat accessory | Leica Biosystems | 14047740044 | |
| Staining tray, 245 mm BioAssay Dish | Corning | 431111 | |
| Syringe, 1 cc | Becton-Dickinson | 309623 | |
| Tape, laboratory, 19 mm width | Fisher Scientific | 15-901-5R | |
| Timer | Fisher Scientific | 14-649-17 | |
| Tissue culture dish, 100 x 15 mm diameter | Fisher Scientific | 08-757-100D | |
| Tissue culture flask, 225 cm2 | ThermoFisher | 159933 | |
| Tissue culture plate, 24-well | Becton-Dickinson | 353226 | |
| Tissue embedding mold, stainless steel | Tissue Tek | 4161 | |
| Tissue Freezing Medium, Optimal Cutting Temperature (OCT) | Fisher Scientific | 4585 | |
| Trypsin-EDTA (ethylenediaminetetraacetic acid), 0.25% | Gibco | 25200072 | |
| Water bath, Precision GP 2S | ThermoFisher | TSGP2S |
Riferimenti
- Pavlova, N. N., Thompson, C. B. The emerging hallmarks of cancer metabolism. Cell Metabolism. 23 (1), 27-47 (2016).
- Pellegatti, P., et al. Increased level of extracellular ATP at tumor sites: in vivo imaging with plasma membrane luciferase. PLoS ONE. 3, 25992008(2008).
- Falzoni, S., Donvito, G., Di Virgilio, F. Detecting adenosine triphosphate in the pericellular space. Interface Focus. 3 (3), 20120101(2013).
- Michaud, M., et al. Autophagy-dependent anticancer immune responses induced by chemotherapeutic agents in mice. Science. 334, 1573-1577 (2011).
- Wilhelm, K., et al. Graft-versus-host disease is enhanced by extracellular ATP activating P2X7R. Nature Medicine. 16, 1434-1438 (2010).
- Vander Heiden, M. G., Cantley, L. C., Thompson, C. B. Understanding the Warburg effect: the metabolic requirements of cell proliferation. Science. 324, 1029-1033 (2009).
- Cairns, R. A., Harris, I. S., Mak, T. W. Regulation of cancer cell metabolism. Nature Reviews Cancer. 11, 85-95 (2011).
- Chen, X., Qian, Y., Wu, S. The Warburg effect: evolving interpretations of an established concept. Free Radical Biology & Medicine. 79, 253-263 (2015).
- Wang, X., et al. Extracellular ATP, as an energy and phosphorylating molecule, induces different types of drug resistances in cancer cells through ATP internalization and intracellular ATP level increase. Oncotarget. 8 (5), 87860-87877 (2017).
- Patel, A., et al. ATP as a biological hydrotrope. Science. 356, 753-756 (2017).
- Qian, Y., et al. Extracellular ATP is internalized by macropinocytosis and induces intracellular ATP increase and drug resistance in cancer cells. Cancer Letters. 351, 242-251 (2014).
- Qian, Y., Wang, X., Li, Y., Cao, Y., Chen, X. Extracellular ATP a new player in cancer metabolism: NSCLC cells internalize ATP in vitro and in vivo using multiple endocytic mechanisms. Molecular Cancer Research. 14, 1087-1096 (2016).
- Commisso, C., et al. Macropinocytosis of protein is an amino acid supply route in Ras-transformed cells. Nature. 497, 633-637 (2013).
- Li, L., et al. The effect of the size of fluorescent dextran on its endocytic pathway. Cell Biology International. 39, 531-539 (2015).
- Yanagawa, Y., Matsumoto, M., Togashi, H. Enhanced dendritic cell antigen uptake via alpha2 adrenoceptor-mediated PI3K activation following brief exposure to noradrenaline. Journal of Immunology. 185, 5762-5768 (2010).
- Hoppe, H. C., et al. Antimalarial quinolines and artemisinin inhibit endocytosis in Plasmodium falciparum. Antimicrobial Agents & Chemotherapy. 48, 2370-2378 (2004).
- Chaudry, I. H. Does ATP cross the cell plasma membrane. Yale Journal of Biology & Medicine. 55, 1-10 (1982).
- Pant, H. C., Terakawa, S., Yoshioka, T., Tasaki, I., Gainer, H. Evidence for the utilization of extracellular [gamma-32P]ATP for the phosphorylation of intracellular proteins in the squid giant axon. Biochimica et Biophysica Acta. 582, 107-114 (1979).
- Chaudry, I. H., Baue, A. E. Further evidence for ATP uptake by rat tissues. Biochimica et Biophysica Acta. 628, 336-342 (1980).
- Koppenol, W. H., Bounds, P. L., Dang, C. V. Otto Warburg's contributions to current concepts of cancer metabolism. Nature Reviews Cancer. 11, 325-337 (2011).
- Dang, C. V. Links between metabolism and cancer. Genes & Development. 26, 877-890 (2012).
- Israelsen, W. J., Vander Heiden, M. G. ATP consumption promotes cancer metabolism. Cell. 143, 669-671 (2010).
- Koster, J. C., Permutt, M. A., Nichols, C. G. Diabetes and insulin secretion: the ATP-sensitive K+ channel (K ATP) connection. Diabetes. 54, 3065-3072 (2005).
- Szendroedi, J., et al. Muscle mitochondrial ATP synthesis and glucose transport/phosphorylation in type 2 diabetes. PLoS Medicine. 4, 154(2007).
- Miyamoto, S., et al. Mass spectrometry imaging reveals elevated glomerular ATP/AMP in diabetes/obesity and identifies sphingomyelin as a possible mediator. EBioMedicine. 7, 121-134 (2016).
Access restricted. Please log in or start a trial to view this content.
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon