Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Imágenes de autofluorescencia para evaluar la fisiología de las algas rojas
En este artículo
Resumen
El presente protocolo describe imágenes de autofluorescencia paso a paso y la evaluación de los cambios de ficobiliproteína en algas rojas basadas en análisis espectral. Este es un método no destructivo y sin etiquetas para evaluar la adaptación celular a hábitats extremos, cuando solo hay material escaso disponible y las células crecen lentamente, o no crecen en absoluto, en condiciones de laboratorio.
Resumen
Las algas rojas (Rhodophyta) contienen ficobiliproteínas y colonizan hábitats con poca luz, sin embargo, algunas (por ejemplo, algunas especies de Chroothece ) también pueden desarrollarse a pleno sol. La mayoría de las rodófitas son rojas, sin embargo, algunas pueden aparecer azuladas, dependiendo de la proporción de biliproteínas azules y rojas (ficocianina y ficoeritrina). Diferentes ficobiliproteínas pueden capturar la luz en diversas longitudes de onda y transmitirla a la clorofila a, lo que hace posible la fotosíntesis en condiciones de luz muy diferentes. Estos pigmentos responden a los cambios de hábitat en la luz, y su autofluorescencia puede ayudar a estudiar los procesos biológicos. Utilizando Chroothece mobilis como organismo modelo y el modo de escaneo lambda espectral en un microscopio confocal, se estudió la adaptación de pigmentos fotosintéticos a diferentes luces monocromáticas a nivel celular para adivinar las condiciones óptimas de crecimiento de la especie. Los resultados mostraron que, incluso cuando la cepa estudiada se aisló de una cueva, se adaptó a intensidades de luz tenue y media. El método presentado es especialmente útil para estudiar organismos fotosintéticos que no crecen o crecen muy lentamente en condiciones de laboratorio, que suele ser el caso de aquellos que viven en hábitats extremos.
Introducción
Las algas rojas, como el género Chroothece, pueden crecer en hábitats extremos, donde con frecuencia tienen que hacer frente a cambios ambientales marcados1. Las inundaciones y sequías son frecuentes en las regiones semiáridas donde se puede encontrar este género, y algunas especies han sido reportadas en arroyos, acantilados, cuevas o incluso aguas termales2. Sin embargo, la mayoría de las veces, las variables biológicas, como la competencia o el pastoreo, relegan a las especies a condiciones no óptimas para su crecimiento. Como estos organismos a menudo son difíciles de cultivar y no crecen o crecen muy lentamente en condiciones de laboratorio, una limitación importante es el tamaño de la muestra disponible. Por lo tanto, es muy importante seguir métodos no destructivos o métodos que impliquen una manipulación mínima de la muestra 3,4.
Las habilidades fisiológicas necesarias para sobrevivir en estos ambientes hostiles pueden ser monitoreadas siguiendo los cambios en sus sistemas fotosintéticos. Los mecanismos metabólicos, la eficiencia fotosintética y la sensibilidad a la luz o a las condiciones de cultivo pueden ser revelados por los perfiles de emisión de fluorescencia de pigmentos, debido a cambios precisos en su transferencia de energía o atrapamiento 5,6,7,8.
La autofluorescencia de compuestos celulares puede ser utilizada como marcador para el citodiagnóstico o como indicador natural del estado celular o metabolismo en respuesta a señales externas e internas a través de cambios en la emisión9. También se puede utilizar para discriminar taxonómicamente diferentes grupos de organismos fotosintéticos10. Dependiendo de la posición filogenética de los microorganismos fototróficos, se pueden encontrar diferentes características de fluorescencia in vivo. Por lo tanto, se ha intentado en varias ocasiones una identificación taxonómica basada en las características in vivo de la fluorescencia fototrófica (incluyendo espectros de absorción y emisión de fluorescencia)11,12. Debido a la diversidad de pigmentos accesorios entre los taxones de fitoplancton, las diferencias en las longitudes de onda en las que se estimula la fluorescencia de la clorofila a (Chl a), o las diferencias en los espectros de emisión, pueden usarse para inferir la taxonomía13. Los espectros de excitación y emisión de fluorescencia in vivo de estos especímenes dependen no sólo de los filos de algas, sino también de la adaptación del fotosistema14. La eficiencia de la transferencia de energía a Chl a, o la relación de Chl a a pigmentos accesorios, y el contenido de pigmento celular son sensibles a las condiciones de crecimiento5.
Las algas rojas, particularmente Chroothece, tienen varios pigmentos fluorescentes accesorios: ficobiliproteínas y carotenoides; Los primeros se concentran en ficobilisomas unidos a los tilacoides de los cloroplastos. Las ficobiliproteínas (ficocianina, ficoeritrina y aloficocianina) pueden capturar la luz en diferentes longitudes de onda y transmitirla a Chl a, lo que hace posible la fotosíntesis en condiciones de luz y cultivo muy diferentes15. Por ejemplo, las especies de Chroothece pueden crecer dentro de cuevas o casi emerger en arroyos calcáreos ligeramente salinos2.
Las luces monocromáticas afectan el crecimiento y la composición pigmentaria de los organismos fotosintéticos, y se han estudiado para prevenir o controlar el crecimiento de organismos fotosintéticos en cuevas. Mulec et al. demostraron que la iluminación enriquecida con rojo promueve el crecimiento de cianobacterias, algas y plantas16. Estudios previos también han reportado que la luz verde afecta la composición pigmentaria de las cianobacterias17, mientras que otros han revelado que la luz verde impide el crecimiento de la mayoría de los organismos fotosintéticos y algunas cianobacterias exhiben una reducción en los tilacoides y una intensidad de fluorescencia media más débil18.
Para comprender la capacidad de Chroothece como organismo modelo para superar condiciones difíciles, las células cultivadas han sido expuestas a intensidades de luz crecientes y luz monocromática (verde o roja)15, para ver cómo hace frente a las condiciones oscuras de las cuevas (donde predomina la luz roja). El protocolo aquí presentado reproduce el efecto de las variables antes mencionadas sobre las ficobiliproteínas de Chroothece a nivel celular utilizando su propia autofluorescencia.
Hoy en día, la fluorescencia se utiliza comúnmente como una herramienta para estudiar las respuestas fisiológicas de plantas vasculares, microalgas, macroalgas y cianobacterias13,14,16. La microscopía de fluorescencia confocal espectral es una excelente herramienta para estudios in vivo para evaluar la fisiología de las muestras fotosintéticas a nivel unicelular 10,17,18,19,20, evitando problemas asociados con la baja tasa de crecimiento en el laboratorio y las dificultades para obtener suficiente biomasa para los métodos de extracción y bioquímicos asociados8 . Una vez que las células se tratan bajo diferentes condiciones de cultivo durante 2 semanas, el perfil de exploración lambda se puede medir in vivo. Aunque hay varias publicaciones en las que se han utilizado diferentes longitudes de onda de excitación por imagen confocal 3,4,10,17, la mayoría de las ficobiliproteínas y Chl a se pueden detectar utilizando una línea de excitación de longitud de onda de 561 nm, y la emisión detectada varía de 570 a 760 nm de longitud de onda. Estos criterios se han basado en un análisis previamente realizado10 con pigmentos puros comerciales (Tabla 1) mediante imagen confocal y los resultados obtenidos en diferentes especies de algas20,21,22.
| Pigmentos | λflmáx. (nm) | λ exc (nm) | |||||||
| 351 | 364 | 458 | 476 | 488 | 514 | 543 | 633 | ||
| Chl a | 660.9-678.1 | 43,4 ± 1,8 | 11,2 ± 0,2 | 1,8 ± 0,05 | 2,0 ± 0,08 | 12,2 ± 0,7 | 6,0 ± 0,3 | 4.2 ± 0,16 | 80,7 ± 1,5 |
| R-PE | 569.2-583.3 | 5,9 ± 0,6 | 5,9 ± 0,16 | 11,1 ± 0,04 | 42,2 ± 0,3 | 100,0 ± 0 | 90,0 ± 0,3 | 99,2 ± 0,08 | - |
| 652.1-668.6 | - | - | 1,5 ± 0,01 | 3,7 ± 0,04 | 26,7 ± 0,5 | 8,7 ± 0,16 | 11,1 ± 0,16 | 11,3 ± 0,2 | |
| C-PC | 636.2-676.4 | 2.3 ± 0.04 | 1,0 ± 0,01 | 0,6 ± 0,004 | 0,7 ± 0,008 | 2,0 ± 0,08 | 2,0 ± 0,04 | 3.3 ± 0.16 | 33,6 ± 0,9 |
| APC-XL | 667.3-683.8 | 15,1 ± 1,5 | 9,6 ± 0,98 | 1,0 ± 0,04 | 1.2 ± 0.08 | 5,9 ± 0,7 | 4.1 ± 0.5 | 23,2 ± 3,5 | 91,4 ± 2,3 |
Tabla 1: La información de pigmento puro utilizada para ejecutar el análisis de escaneo lambda. Esta tabla muestra los picos de emisión y los hombros / máximos de banda de fluorescencia de diferentes fluorocromos / pigmentos por espectrofotometría de imagen confocal para todas las longitudes de onda de excitación, y el porcentaje de emisión de luz por pigmentos / fluorocromos. Los valores se calcularon mediante la fórmula: = MFI*100/255. Cada valor es la media ± SE (media ± error estándar de la media). Se utilizaron pigmentos puros para calibrar el microscopio láser de barrido confocal de la siguiente manera 1,2,10. La clorofila a se obtuvo de Spinacia oleracea, R-ficoeritrina (R-PE) de Porphyra tenera y C-ficocianina (C-PE) de Spirulina sp. Todas las especies fueron disueltas en agua destilada filtrada. La aloficocianina-XL (APC-XL) se obtuvo de Mastigocladus laminosus, que se disolvió en sulfato de amonio (60%) y fosfato de potasio (pH = 7) para alcanzar una concentración de 38 mM. Las exploraciones se realizaron con 400 μL de cada solución pigmentaria (concentración de 1 mg/ml) utilizando una cámara inferior de vidrio cubierta de 8 pocillos.
El estudio de una sola longitud de onda de excitación es una primera aproximación bastante útil. En este caso, sin embargo, es necesario dilucidar la contribución relativa de los diferentes complejos en la señal de fluorescencia, lo que se recomienda para realizar una relación de fluorescencia o análisis de espectro a varias longitudes de onda, entre otros métodos.
Protocolo
Para el presente estudio se utilizó la especie de alga Chroothece mobilis . La especie se obtuvo de la colección de cultivo de Microalgas Edáficas SE España, MAESE 20.29. En la figura 1 se muestra una descripción general del protocolo.
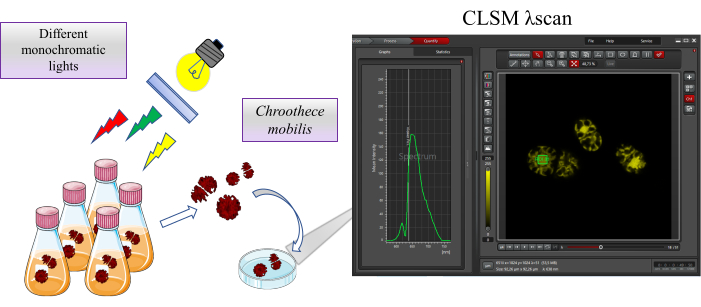
Figura 1: Resumen del estudio. Chroothece mobilis se incuba en condiciones de hábitat extremas, como diferentes luces monocromáticas, durante 2 semanas. El efecto sobre la fisiología de Chroothece se evalúa mediante la autofluorescencia de las proteínas contenidas en el ficobilisoma y los fotosistemas utilizando un microscopio de barrido láser confocal. Haga clic aquí para ver una versión más grande de esta figura.
1. Preparación de la muestra
- Preparar el inóculo de Chroothece mobilis a partir del cultivo en agar de la colección transfiriéndolo al medio líquido SWES (Tabla 2).
NOTA: SWES "Seewasser + Erddekokt + Salze" = medio de agua de mar23. - Mantener todos los cultivos durante 2 semanas con un fotoperiodo claro/oscuro de 16:8 a 20 °C en condiciones de baja intensidad de luz blanca (LL: 80 μM/m2/s) y sin agitación, hasta obtener la densidad celular deseada (ver paso 1.3).
NOTA: Las condiciones de luz de crecimiento son 80 μM/m2/s de intensidad lumínica (radiación fotosintética activa, PAR). Estas condiciones se utilizan como condiciones de poca luz (control). La composición del medio SWES se proporciona en la Tabla 2. - Realizar inoculaciones para los diferentes experimentos en la fase de cultivo exponencial con una densidad celular de 5 x 103 células/mL en una placa de 24 pocillos, utilizando 1 mL por pocillo.
NOTA: Realice el recuento de células con una cámara Neubauer24 y diluya el cultivo con SWES cuando sea necesario.
| SWES composición media | |
| Componente | Concentración |
| KNO3 | 1,98 mM |
| K2HPO4 | 115 μM |
| MgSO4 | 81 μM |
| ZnSO4, 7H2O | 17 nM |
| MnSO4, 7H2O | 45 Nm |
| H 3BO3, 4H2O | 3,1 mM |
| CO(NO3)2 | 17 mM |
| Na 2 MoO4, 6H2O | 21 nM |
| CuSO4, 2H2O | 0,1 nM |
| FeSO4, 5H2O | 13 μM |
| EDTA, 7H2O | 11 μM |
| Vit B12 | 5 μg |
| Extracto de suelo | 30 ml |
| Agua filtrada del río | 455 ml |
Tabla 2: Composición media de SWES.
2. Reproducción de condiciones extremas del hábitat de las algas: efecto de luz monocromática verde y roja
- Añadir 1 ml del cultivo celular del cultivo madre preparado con la densidad celular antes mencionada (paso 1.2) para inocular cada pocillo de una placa de 24 pocillos.
- Mantenga el cultivo celular cubierto durante 2 semanas para reproducir el efecto de luz monocromática.
- Utilice el filtro verde que permite que la luz verde pase de 470 a 570 nm con un pico en la longitud de onda de 506 nm (de acuerdo con las especificaciones del fabricante; consulte la Tabla de materiales) y exponerlo al cultivo25.
- Utilice el filtro rojo que permite la luz roja entre 590 y 720 nm y picos a 678 nm (según las especificaciones del fabricante; consulte la Tabla de materiales) para exponerlo al cultivo25.
NOTA: La condición de baja intensidad de luz blanca (LL: 80 μM/m2/s; ver Tabla de materiales) se utiliza como control de intensidad de luz para comparar los efectos obtenidos. Las unidades de intensidad de luz empleadas son micromoles por segundo y metro cuadrado (μmol m-2 s-1) o densidad de flujo de fotones fotosintéticos (PPFD).
3. Imágenes de autofluorescencia
NOTA: La configuración del software de imágenes (consulte Tabla de materiales) se ilustra en la Figura 2.
- Encienda todos los componentes del microscopio de barrido láser confocal invertido (CLSM; consulte Tabla de materiales), incluido el láser.
- Monte las células de cada pocillo experimental de la placa de 24 pocillos en medio de crecimiento SWES en un plato de fondo de vidrio de 35 mm (consulte la Tabla de materiales) para obtener imágenes.
- Elija el objetivo de inmersión en glicerol 63x/1.30 NA y coloque glicerol sobre la lente (consulte la Tabla de materiales).
- Coloque la placa del pocillo en la etapa del microscopio y asegúrese de que la muestra no se mueva durante la adquisición de imágenes.
- Centra la muestra en la trayectoria de la luz y enfócate en el centro de la celda seleccionando el plano con la mayor intensidad de fluorescencia.
- Abra el software de adquisición de imágenes y elija xyλ en la lista desplegable en el modode adquisición 26.
- Seleccione la línea de excitación del láser a 561 nm DPSS, 8 bits de rango dinámico y 1024 x 1024 píxeles.
NOTA: Asegúrese de que el microscopio confocal tenga un láser DPSS de 561 nm o un láser de luz blanca (WLL). - Recopile los espectros de emisión de fluorescencia en el ancho de banda de 10 nm y el tamaño de paso lambda de 4 nm dentro del rango de 570-760 nm.
- Establezca el agujero de alfiler en 1 unidad Airy y ejecute la adquisición de escaneo lambda.
- Repita este proceso tantas veces como sea posible en diferentes campos de visión para recopilar una cantidad aceptable de datos para el análisis estadístico (generalmente una desviación estándar inferior al 10%22).
- Repita el último paso bajo las diferentes condiciones (bajo luces rojas y verdes) y guarde los datos.
NOTA: Utilice la misma configuración de adquisición para las diferentes muestras y condiciones para comparar y realizar el análisis estadístico.

Figura 2: Configuración del software. Interfaz de usuario del software de creación de imágenes para configurar los parámetros de escaneo lambda. (A) De izquierda a derecha, para seleccionar el modo de adquisición xyλ de la lista desplegable, que corresponde al paso 3.6 en el protocolo, y para seleccionar el tipo de lente de inmersión derecha, correspondiente al paso 3.3 en el protocolo. Asegúrese de quitar cualquier filtro de la trayectoria de luz en el paso 3.9. (B) El panel para configurar los parámetros de escaneo lambda corresponde al paso 3.8 del protocolo. (C) Ejecute el escaneo lambda en el paso 3.10. Haga clic aquí para ver una versión más grande de esta figura.
4. Parámetros para evaluar la fisiología de Chroothece
- Una vez adquirido el escaneo lambda, haga clic en la ventana de cuantificación en la parte superior del software (como se muestra en la Figura 3) para evaluar los espectros de emisión de fluorescencia recopilados. Vaya a la ventana Abrir proyecto y seleccione un archivo xyλ (Figura 3).
- Seleccione Análisis de perfil de pila en el software de imágenes.
- Definir una región de interés (ROI) de 4μm2 en el centro de una célula para analizar la intensidad media de fluorescencia (MFI). Exporte los datos en formato CSV.
NOTA: Es importante evitar la presencia de píxeles negros (para garantizar que los píxeles seleccionados contengan valores positivos) y mantener siempre un ROI del mismo tamaño. - Repita este proceso con diferentes celdas bajo diferentes condiciones para producir suficientes datos para realizar análisis estadísticos.
- Abra los archivos CSV para seleccionar los diferentes picos de emisión de fluorescencia de las ficobiliproteínas y clorofilas de todos los ROI medidos.
- Seleccione los datos de fluorescencia de ficoeritrina-ficocianobilina (PE-PCB; 620 nm), C-ficocianina (CPC; 648 nm), aloficocianina (APC; 660 nm) y clorofila a (Chl a; 680 nm), respectivamente, en el archivo csv.
- Cree una nueva tabla con todos los valores máximos de fluorescencia obtenidos de cada pico de ficobiliproteína y clorofila y trace los datos en un gráfico.
- Realizar análisis estadísticos.
- Analizar si los datos obtenidos cumplen con la normalidad y la homocedasticidad27.
- Proceda al análisis de la prueba t27,28 si la primera condición es verdadera. Si no es así, ejecute una prueba U de Mann-Whitney29.
- Considere las diferencias significativas en valores de p <0,05.
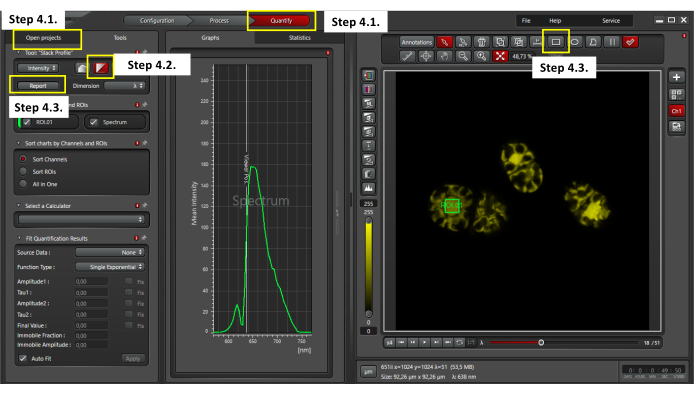
Figura 3: Evaluación de la autofluorescencia de Chroothece. Para realizar el análisis de autofluorescencia, asegúrese de seleccionar la ventana de cuantificación y un archivo xyλ en la ventana de proyectos abiertos (paso 4.1); seleccionar la visualización del perfil de pila (paso 4.2); seleccione un ROI de 4 μm2 en el centro de una celda y haga clic en el botón Informe para exportar los datos de escaneo lambda en formato CSV (paso 4.3). Haga clic aquí para ver una versión más grande de esta figura.
Resultados
La clorofila a generalmente absorbe longitudes de onda azules y rojas de la luz visible, mientras que las ficobiliproteínas usan longitudes de onda verde, amarilla y naranja7. La autofluorescencia de estos pigmentos hace posible el primer enfoque para estudiar las ficobiliproteínas y el comportamiento de la clorofila en condiciones experimentales y de campo.
Al comparar los datos obtenidos y trazar en diferentes gráficos, se pueden distinguir cambios significativos e...
Discusión
Algunas algas rojas unicelulares o coloniales, como Chroothece, crecen lentamente in vitro, pero contienen múltiples compuestos autofluorescentes que pueden analizarse mediante análisis espectral bajo un microscopio confocal, donde se pueden detectar diferencias en los picos de emisión de pigmentos. La microscopía de fluorescencia confocal espectral nos ha permitido realizar estudios in vivo para evaluar la adaptación o aclimatación de organismos fotosintéticos 8,10,17,18,19,20.
Divulgaciones
Los autores no tienen conflictos de intereses que declarar.
Agradecimientos
Esta investigación se ha realizado en el marco de los proyectos TIN2015-68454-R y 20961/PI/18, financiados por el Ministerio de Economía y Competitividad y la Fundación Séneca de la Región de Murcia. Irene Hernández Martínez y Francisco Javier Ibáñez López de la Sección de Apoyo Estadístico del Área Científica y de Investigación de la Universidad de Murcia (SAE), Área Científica y de Investigación (ACTI), Universidad de Murcia, (Figura 1 ) se dibujaron utilizando imágenes de Servier Medical Art. Servier Medical Art by Servier está bajo una licencia Creative Commons Attribution 3.0 Unported License (https://creativecommons.org/licenses/by/3.0/)
Materiales
| Name | Company | Catalog Number | Comments |
| µ-Dish 35 mm, high Glass Bottom | Ibidi | 81158 | - |
| 24 black well plate | Ibidi | 82406 | flat and clear bottom for high throughput microscopy |
| Algae Incubator | Panasonic | MLR-352-PE | |
| Confocal laser scanning microscope | Leica Microsystems | SP8 TCS | - |
| Flask | Fisher Scientific | 15380591 | Can be purchased in a local convenience store or online stores. |
| green filter | PNTA, LEE filters | - | Can be purchased in a local convenience store or online stores. |
| HC PL APO 63X/1.30 GLYC CORR CS2 | Leica Microsystems | 506353 | Glycerol immersion lens |
| Image acquisition software. LAS X | Leica Microsystems | SP8 TCS | - |
| Light source | Panasonic | FL40SSENW/37MLR-352-PE | |
| Quantum photoradiometer | DeltaOhm | DO 9721 | - |
| R software | R Core Team, 2020 | 4.0.2. | - |
| red filter | PNTA, LEE filters | - | Can be purchased in a local convenience store or online stores. |
| SWES medium | University of Murcia | - | - |
| Type G Immersion liquid | Leica Microsystems | 11513910 | Glycerol |
Referencias
- Vis, M. L., Necchi, O. . Freshwater Red Algae: Phylogeny, Taxonomy and Biogeography. , (2021).
- Aboal, M., et al. Diversity of Chroothece (Rhodophyta, Stylonematales) including two new species. European Journal of Phycology. 53 (2), 189-197 (2018).
- Millach, L., Obiol, A., Solé, A., Esteve, I. A novel method to analyse in vivo the physiological state and cell viability of phototrophic microorganisms by confocal laser scanning microscopy using a dual laser. Journal of Microscopy. 268 (1), 53-65 (2017).
- Millach, L., Villagrasa, E., Solé, A., Esteve, I. Combined confocal laser scanning microscopy techniques for a rapid assessment of the effect and cell viability of Scenedesmus sp. DE2009 under metal stress. Microscopy and Microanalysis. 25 (4), 998-1003 (2019).
- Poryvkina, L., Babichenko, S., Leeben, A. Analysis of phytoplankton pigments by excitation spectra of fluorescence. Proceedings of EARSeL-SIG-Workshop LIDAR. , (2000).
- Beutler, M., et al. A fluorometric method for the differentiation of algal populations in vivo and in situ. Photosynthesis Research. 72 (1), 39-53 (2002).
- Grigoryeva, N., Chistyakova, L. Fluorescence microscopic spectroscopy for investigation and monitoring of biological diversity and physiological state of cyanobacterial cultures. Cyanobacteria. , (2018).
- Grigoryeva, N. . Fluorescence Methods for Investigation of Living Cells and Microorganisms. , (2020).
- Roshchina, V. V. Vital autofluorescence: application to the study of plant living cells. International Journal of Spectroscopy. 2012, 5-18 (2012).
- Roldán, M., Thomas, F., Castel, S., Quesada, A., Hernández-Mariné, M. Noninvasive pigment identification in single cells from living phototrophic biofilms by confocal imaging spectrofluorometry. Applied and Environmental Microbiology. 70 (6), 3745-3750 (2004).
- Hense, B. A., Gais, P., Jütting, U., Scherb, H., Rodenacker, K. Use of fluorescence information for automated phytoplankton investigation by image analysis. Journal of Plankton Research. 30 (5), 587-606 (2008).
- Millie, D. F., Schofield, O. M., Kirkpatrick, G. J., Johnsen, G., Evens, T. J. Using absorbance and fluorescence spectra to discriminate microalgae. European Journal of Phycology. 37 (3), 313-322 (2002).
- Richardson, T. L., et al. Spectral fluorometric characterization of phytoplankton community composition using the Algae Online Analyser. Water Research. 44 (8), 2461-2472 (2010).
- Kieleck, C., Bousquet, B., Le Brun, G., Cariou, J., Lotrian, J. Laser induced fluorescence imaging: application to groups of macroalgae identification. Journal of Physics D: Applied Physics. 34 (16), 2561-2571 (2001).
- Coronado-Parra, T., Roldán, M., Aboal, M. Confocal microscopy in ecophysiological studies of algae: a door to understanding autofluorescence in red algae. Microscopy and Microanalysis. 28 (1), 218-226 (2022).
- Mulec, J., Kosi, G. Lampenflora algae and methods of growth control. Journal of Cave and Karst Studies. 71 (2), 109-115 (2009).
- Nelissen, B., De Baere, R., Wilmotte, A., De Wachter, R. Phylogenetic relationships of nonaxenic filamentous cyanobacterial strains based on 16S rRNA sequence analysis. Journal of Molecular Evolution. 42 (2), 194-200 (1996).
- Roldán, M., Oliva, F., Gónzalez Del Valle, M. A., Saiz-Jimenez, C., Hernández-Mariné, M. Does green light influence the fluorescence properties and structure of phototrophic biofilms. Applied and Environmental Microbiology. 72 (4), 3026-3031 (2006).
- Topinka, J. A., Bellows, W. K., Yentsch, C. S. Characterization of marine macroalgae by fluorescence signatures. International Journal of Remote Sensing. 11 (12), 2329-2335 (1990).
- Roldán, M., Ascaso, C., Wierzchos, J. Fluorescent fingerprints of endolithic phototrophic cyanobacteria living within halite rocks in the atacama desert. Applied and Environmental Microbiology. 80 (10), 2998-3006 (2014).
- Solé, A., Diestra, E., Esteve, I. Confocal laser scanning microscopy image analysis for cyanobacterial biomass determined at microscale level in different microbial mats. Microbial Ecology. 57 (4), 649-656 (2009).
- Ramírez, O., García, A., Rojas, R., Couve, A., Härtel, S. Confined displacement algorithm determines true and random colocalization in fluorescence microscopy. Journal of Microscopy. 239 (3), 173-183 (2010).
- Universität Gottingen-Georg-August. SAG Culture Collection of Algae. Universität Gottingen-Georg-August. , (2022).
- Zhang, M., et al. Improvement of cell counting method for Neubauer counting chamber. Journal of Clinical Laboratory Analysis. 34 (1), 23024 (2020).
- Wolf, E., Schüßler, A. Phycobiliprotein fluorescence of Nostoc punctiforme changes during the life cycle and chromatic adaptation: Characterization by spectral confocal laser scanning microscopy and spectral unmixing. Plant, Cell and Environment. 28 (4), 480-491 (2005).
- Zucker, R. M., Rigby, P., Clements, I., Salmon, W., Chua, M. Reliability of confocal microscopy spectral imaging systems: Use of multispectral beads. Cytometry Part A. 71 (3), 174-189 (2007).
- Field, A. M. . Discovering Statistics Using R. , (2013).
- Linnet, K. Limitations of the paired t-test for evaluation of method comparison data. Clinical Chemistry. 45 (2), 314-315 (1999).
- Rosner, B., Grove, D. Use of the Mann-Whitney U-test for clustered data. Statistics in Medicine. 18 (11), 1387-1400 (1999).
- Colin, L., et al. Imaging the living plant cell: from probes to quantification. The Plant Cell. 34 (1), 247-272 (2022).
- Borlinghaus, R. The white confical: continuous spectral tuning in excitation and emission. Optical Fluorescence Microscopy. , (2011).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados