È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Autofluorescenza per valutare la fisiologia delle alghe rosse
In questo articolo
Riepilogo
Il presente protocollo descrive l'imaging autofluorescenza passo-passo e la valutazione dei cambiamenti ficobiliproteici nelle alghe rosse sulla base dell'analisi spettrale. Questo è un metodo privo di etichette e non distruttivo per valutare l'adattamento cellulare ad habitat estremi, quando è disponibile solo materiale scarso e le cellule crescono lentamente, o per niente, in condizioni di laboratorio.
Abstract
Le alghe rosse (Rhodophyta) contengono ficobiliproteine e colonizzano gli habitat con luce fioca, tuttavia alcune (ad esempio, alcune specie di Chroothece ) possono anche svilupparsi in pieno sole. La maggior parte delle rodofite sono rosse, tuttavia alcune possono apparire bluastre, a seconda della proporzione di biliproteine blu e rosse (ficocianina e ficoeritrina). Diverse ficobiliproteine possono catturare la luce a diverse lunghezze d'onda e trasmetterla alla clorofilla a, il che rende possibile la fotosintesi in condizioni di luce molto diverse. Questi pigmenti rispondono ai cambiamenti dell'habitat nella luce e la loro autofluorescenza può aiutare a studiare i processi biologici. Utilizzando Chroothece mobilis come organismo modello e la modalità di scansione lambda spettrale in un microscopio confocale, è stato studiato l'adattamento dei pigmenti fotosintetici a diverse luci monocromatiche a livello cellulare per indovinare le condizioni di crescita ottimali della specie. I risultati hanno mostrato che, anche quando il ceppo studiato è stato isolato da una grotta, si è adattato sia a intensità luminose fioche che medie. Il metodo presentato è particolarmente utile per studiare gli organismi fotosintetici che non crescono o crescono molto lentamente in condizioni di laboratorio, come di solito accade per coloro che vivono in habitat estremi.
Introduzione
Le alghe rosse, come il genere Chroothece, possono crescere in habitat estremi, dove spesso devono far fronte a marcati cambiamenti ambientali1. Inondazioni e siccità sono frequenti nelle regioni semi-aride dove questo genere può essere trovato, e alcune specie sono state segnalate in torrenti, scogliere, grotte o persino acque termali2. Tuttavia, la maggior parte delle volte, le variabili biologiche, come la competizione o il pascolo, relegano le specie a condizioni non ottimali per la loro crescita. Poiché questi organismi sono spesso difficili da coltivare e non crescono o crescono molto lentamente in condizioni di laboratorio, una delle principali limitazioni è la dimensione del campione disponibile. Pertanto, è molto importante seguire metodi non distruttivi o metodi che comportano una manipolazione minima del campione 3,4.
Le abilità fisiologiche necessarie per sopravvivere in questi ambienti difficili possono essere monitorate seguendo i cambiamenti nei loro sistemi fotosintetici. I meccanismi metabolici, l'efficienza fotosintetica e la sensibilità alla luce o alle condizioni di coltura possono essere rivelati dai profili di emissione di fluorescenza dei pigmenti, a causa di accurati cambiamenti nel loro trasferimento di energia o intrappolamento 5,6,7,8.
L'autofluorescenza dei composti cellulari può essere utilizzata come marker per la citodiagnosi o come indicatore naturale dello stato cellulare o del metabolismo in risposta a segnali esterni e interni attraverso cambiamenti nell'emissione9. Può anche essere usato per discriminare tassonomicamente diversi gruppi di organismi fotosintetici10. A seconda della posizione filogenetica dei microrganismi fototrofi, si possono trovare diverse caratteristiche di fluorescenza in vivo. Pertanto, un'identificazione tassonomica basata sulle caratteristiche in vivo della fluorescenza fototrofica (compresi gli spettri di assorbimento e di emissione della fluorescenza) è stata tentata in diverse occasioni11,12. A causa della diversità nei pigmenti accessori tra i taxa di fitoplancton, le differenze nelle lunghezze d'onda alle quali viene stimolata la fluorescenza della clorofilla a (Chl a), o le differenze negli spettri di emissione, possono essere utilizzate per dedurre la tassonomia13. Gli spettri di eccitazione ed emissione di fluorescenza in vivo di questi campioni si basano non solo sui phyla delle alghe, ma anche sull'adattamento del fotosistema14. L'efficienza del trasferimento di energia a Chl a, o il rapporto tra Chl a e pigmenti accessori, e il contenuto di pigmenti cellulari sono sensibili alle condizioni di crescita5.
Le alghe rosse, in particolare Chroothece, hanno diversi pigmenti fluorescenti accessori: ficobiliproteine e carotenoidi; I primi si concentrano nei ficobilisomi attaccati ai tilacoidi dei cloroplasti. Le ficobiliproteine (ficocianina, ficoeritrina e alloficocianina) possono catturare la luce a diverse lunghezze d'onda e trasmetterla a Chl a, il che rende possibile la fotosintesi in condizioni di luce e coltura molto diverse15. Ad esempio, le specie di Chroothece possono crescere all'interno di grotte o quasi emergere in torrenti calcarei leggermente salini2.
Le luci monocromatiche influenzano la crescita e la composizione pigmentata degli organismi fotosintetici e sono state studiate per prevenire o controllare la crescita di organismi fotosintetici nelle caverne. Mulec et al. hanno dimostrato che l'illuminazione arricchita di rosso promuove la crescita di cianobatteri, alghe e piante16. Studi precedenti hanno anche riportato che la luce verde influisce sulla composizione del pigmento dei cianobatteri17, mentre altri hanno rivelato che la luce verde impedisce la crescita della maggior parte degli organismi fotosintetici e alcuni cianobatteri mostrano una riduzione dei tilacoidi e un'intensità media di fluorescenza più debole18.
Per comprendere la capacità di Chroothece come organismo modello di superare condizioni difficili, le cellule coltivate sono state esposte a crescenti intensità luminose e luce monocromatica (verde o rossa)15, per vedere come affronta le condizioni di scarsa illuminazione delle grotte (dove predomina la luce rossa). Il protocollo qui presentato riproduce l'effetto delle suddette variabili sulle ficobiliproteine di Chroothece a livello cellulare utilizzando la propria autofluorescenza.
Al giorno d'oggi, la fluorescenza è comunemente usata come strumento per studiare le risposte fisiologiche di piante vascolari, microalghe, macroalghe e cianobatteri13,14,16. La microscopia a fluorescenza confocale spettrale è uno strumento eccellente per gli studi in vivo per valutare la fisiologia dei campioni fotosintetici a livello di singola cellula 10,17,18,19,20, evitando i problemi associati al basso tasso di crescita in laboratorio e le difficoltà di ottenere abbastanza biomassa per i metodi di estrazione e biochimici associati8 . Una volta che le cellule vengono trattate in diverse condizioni di coltura per 2 settimane, il profilo di scansione lambda può essere misurato in vivo. Sebbene ci siano diverse pubblicazioni in cui sono state utilizzate diverse lunghezze d'onda di eccitazione mediante imaging confocale 3,4,10,17, la maggior parte delle ficobiliproteine e Chl a possono essere rilevate utilizzando una linea di eccitazione a lunghezza d'onda di 561 nm e l'emissione rilevata varia da 570 a 760 nm di lunghezza d'onda. Questi criteri sono stati basati su un'analisi precedentemente eseguita 10 con pigmenti puri commerciali (Tabella 1) mediante imaging confocale e sui risultati ottenuti in diverse specie di alghe20,21,22.
| Pigmenti | λflmax (nm) | λ exc (nm) | |||||||
| 351 | 364 | 458 | 476 | 488 | 514 | 543 | 633 | ||
| Chl a | 660.9-678.1 | 43,4 ± 1,8 | 11,2 ± 0,2 | 1,8 ± 0,05 | 2,0 ± 0,08 | 12,2 ± 0,7 | 6,0 ± 0,3 | 4,2 ± 0,16 | 80,7 ± 1,5 |
| R-PE | 569.2-583.3 | 5,9 ± 0,6 | 5,9 ± 0,16 | 11,1 ± 0,04 | 42,2 ± 0,3 | 100,0 ± 0 | 90,0 ± 0,3 | 99,2 ± 0,08 | - |
| 652.1-668.6 | - | - | 1,5 ± 0,01 | 3,7 ± 0,04 | 26,7 ± 0,5 | 8,7 ± 0,16 | 11,1 ± 0,16 | 11,3 ± 0,2 | |
| C-PC | 636.2-676.4 | 2,3 ± 0,04 | 1,0 ± 0,01 | 0,6 ± 0,004 | 0,7 ± 0,008 | 2,0 ± 0,08 | 2,0 ± 0,04 | 3,3 ± 0,16 | 33,6 ± 0,9 |
| APC-XL | 667.3-683.8 | 15,1 ± 1,5 | 9,6 ± 0,98 | 1,0 ± 0,04 | 1,2 ± 0,08 | 5,9 ± 0,7 | 4,1 ± 0,5 | 23,2 ± 3,5 | 91,4 ± 2,3 |
Tabella 1: Informazioni sui pigmenti puri utilizzate per eseguire l'analisi della scansione lambda. Questa tabella mostra i picchi di emissione e i massimi di banda di spalle/fluorescenza di diversi fluorocromi/pigmenti mediante spettrofotometria di imaging confocale per tutte le lunghezze d'onda di eccitazione e la percentuale di emissione luminosa da parte di pigmenti/fluorocromi. I valori sono stati calcolati con la formula: = MFI*100/255. Ogni valore è la media ± SE (media ± errore standard dalla media). Per calibrare il microscopio laser a scansione confocale sono stati utilizzati pigmenti puri come segue 1,2,10. La clorofilla a è stata ottenuta da Spinacia oleracea, R-phycoerythrin (R-PE) da Porphyra tenera e C-phycocyanin (C-PE) da Spirulina sp. Tutte le specie sono state sciolte in acqua distillata filtrata. L'allophycocyanin-XL (APC-XL) è stata ottenuta da Mastigocladus laminosus, che è stato sciolto in solfato di ammonio (60%) e fosfato di potassio (pH = 7) per raggiungere una concentrazione di 38 mM. Le scansioni sono state eseguite con 400 μL di ciascuna soluzione di pigmento (concentrazione di 1 mg/ml) utilizzando una camera inferiore di vetro coperta a 8 pozzetti.
Lo studio di una singola lunghezza d'onda di eccitazione è una prima approssimazione abbastanza utile. In questo caso, tuttavia, è necessario chiarire il contributo relativo dei diversi complessi nel segnale di fluorescenza, che si raccomanda di eseguire un rapporto di fluorescenza o un'analisi dello spettro a diverse lunghezze d'onda, tra gli altri metodi.
Protocollo
La specie algale Chroothece mobilis è stata utilizzata per il presente studio. La specie è stata ottenuta dalla collezione di colture Microalgae Edaphic SE Spain, MAESE 20.29. Una panoramica del protocollo è illustrata nella Figura 1.
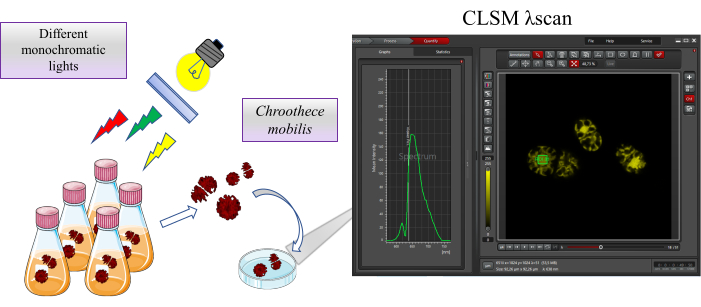
Figura 1: Panoramica dello studio. Chroothece mobilis viene incubato in condizioni di habitat estreme, come diverse luci monocromatiche, per 2 settimane. L'effetto sulla fisiologia di Chroothece viene valutato mediante autofluorescenza delle proteine contenute nei ficobilisomi e nei fotosistemi utilizzando un microscopio a scansione laser confocale. Fare clic qui per visualizzare una versione ingrandita di questa figura.
1. Preparazione del campione
- Preparare l'inoculo di Chroothece mobilis dalla coltura di agar della raccolta trasferendolo al mezzo liquido SWES (Tabella 2).
NOTA: SWES "Seewasser + Erddekokt + Salze" = mezzo di acqua di mare23. - Mantenere tutte le colture per 2 settimane con un fotoperiodo chiaro/scuro 16:8 a 20 °C in condizioni di bassa intensità della luce bianca (LL: 80 μM/m2/s) e senza agitare, fino ad ottenere la densità cellulare desiderata (vedere punto 1.3).
NOTA: Le condizioni di luce di crescita sono 80 μM/m2/s di intensità luminosa (radiazione fotosintetica attiva, PAR). Queste condizioni sono utilizzate come condizioni di scarsa illuminazione (controllo). La composizione del mezzo SWES è fornita nella tabella 2. - Effettuare inoculazioni per i diversi esperimenti nella fase di coltura esponenziale con una densità cellulare di 5 x 103 cellule/ml in una piastra a 24 pozzetti, utilizzando 1 mL per pozzetto.
NOTA: Eseguire il conteggio delle cellule con una camera Neubauer24 e diluire la coltura con SWES quando necessario.
| SWES composizione media | |
| Componente | Concentrazione |
| KNO3 | 1,98 mM |
| K2HPO4 | 115 μM |
| MgSO4 | 81 μM |
| ZnSO4, 7H2O | 17 nM |
| MnSO4, 7H2O | 45 Nm |
| H3 BO3, 4H2O | 3,1 mM |
| Co(N.3)2 | 17 mM |
| Na 2 MoO4, 6H2O | 21 nM |
| CuSO4, 2H2O | 0,1 nM |
| FeSO4, 5H2O | 13 μM |
| EDTA, 7H2O | 11 μM |
| Vit B12 | 5 μg |
| Estratto di terreno | 30 ml |
| Acqua fluviale filtrata | 455 ml |
Tabella 2: Composizione media SWES.
2. Riproduzione di condizioni estreme dell'habitat delle alghe: effetto di luce monocromatica verde e rossa
- Aggiungere 1 mL della coltura cellulare dalla coltura madre preparata alla suddetta densità cellulare (fase 1.2) per inoculare ciascun pozzetto di una piastra a 24 pozzetti.
- Tenere la coltura cellulare coperta per 2 settimane per riprodurre l'effetto di luce monocromatica.
- Utilizzare il filtro verde che consente alla luce verde di passare da 470 a 570 nm con un picco alla lunghezza d'onda di 506 nm (secondo le specifiche del produttore; vedere Tabella dei materiali) ed esporlo alla coltura25.
- Utilizzare il filtro rosso che consente alla luce rossa tra 590 e 720 nm e picchi a 678 nm (secondo le specifiche del produttore; vedi Tabella dei materiali) di esporlo alla coltura25.
NOTA: La condizione di bassa intensità della luce bianca (LL: 80 μM/m2/s; vedere la tabella dei materiali) viene utilizzata come controllo dell'intensità luminosa per confrontare gli effetti ottenuti. Le unità di intensità luminosa impiegate sono micromole al secondo e metro quadrato (μmol m-2 s-1) o densità di flusso di fotoni fotosintetici (PPFD).
3. Imaging ad autofluorescenza
NOTA: la configurazione del software di imaging (vedere la tabella dei materiali) è illustrata nella Figura 2.
- Accendere tutti i componenti del microscopio a scansione laser confocale invertita (CLSM; vedere Tabella dei materiali), compreso il laser.
- Montare le celle da ciascun pozzetto sperimentale della piastra a 24 pozzetti nel mezzo di crescita SWES su un piatto inferiore di vetro da 35 mm (vedi Tabella dei materiali) per l'imaging.
- Scegli l'obiettivo ad immersione con glicerolo 63x/1,30 NA e posizionalo sopra l'obiettivo (vedi Tabella dei materiali).
- Posizionare la piastra del pozzetto sul palco del microscopio e assicurarsi che il campione non si muova durante l'acquisizione dell'immagine.
- Centrare il campione nel percorso della luce e concentrarsi sul centro della cella selezionando il piano con la più alta intensità di fluorescenza.
- Apri il software di acquisizione immagini e scegli xyλ dall'elenco a discesa nella modalità di acquisizione26.
- Selezionare la linea di eccitazione del laser su 561 nm DPSS, 8 bit di gamma dinamica e 1024 x 1024 pixel.
NOTA: Assicurarsi che il microscopio confocale disponga di un laser DPSS da 561 nm o di un laser a luce bianca (WLL). - Raccogliere gli spettri di emissione di fluorescenza nella larghezza di banda di 10 nm e la dimensione del passo lambda di 4 nm nell'intervallo 570-760 nm.
- Impostare il foro stenopeico su 1 unità Airy ed eseguire l'acquisizione della scansione lambda.
- Ripetere questo processo il maggior numero di volte possibile in diversi campi visivi per raccogliere una quantità accettabile di dati per l'analisi statistica (di solito una deviazione standard inferiore al 10%22).
- Ripetere l'ultimo passaggio nelle diverse condizioni (sotto luci rosse e verdi) e salvare i dati.
NOTA: utilizzare le stesse impostazioni di acquisizione per i diversi campioni e condizioni per confrontare e condurre l'analisi statistica.

Figura 2: Configurazione del software. Interfaccia utente del software di imaging per impostare i parametri di scansione lambda. (A) Da sinistra a destra, selezionare la modalità di acquisizione xyλ dall'elenco a discesa, che corrisponde al punto 3.6 del protocollo, e selezionare il tipo di lente ad immersione destra, corrispondente al punto 3.3 del protocollo. Assicurarsi di rimuovere qualsiasi filtro dal percorso luminoso nel passaggio 3.9. (B) Il pannello per l'impostazione dei parametri di scansione lambda corrisponde al punto 3.8 del protocollo. (C) Eseguire la scansione lambda nel passaggio 3.10. Fare clic qui per visualizzare una versione ingrandita di questa figura.
4. Parametri per valutare la fisiologia di Chroothece
- Una volta acquisita la scansione lambda, fare clic sulla finestra di quantificazione nella parte superiore del software (come mostrato nella Figura 3) per valutare gli spettri di emissione di fluorescenza raccolti. Passare alla finestra Apri progetto e selezionare un file xyλ (Figura 3).
- Selezionare Analisi profilo stack nel software di imaging.
- Definire una regione di interesse (ROI) di 4 μm2 al centro di una cella per analizzare l'intensità media di fluorescenza (MFI). Esporta i dati in formato CSV.
NOTA: è importante evitare la presenza di pixel neri (per garantire che i pixel selezionati contengano valori positivi) e mantenere sempre un ROI delle stesse dimensioni. - Ripetere questo processo con celle diverse in condizioni diverse per produrre dati sufficienti per eseguire analisi statistiche.
- Apri i file CSV per selezionare i diversi picchi di emissione di fluorescenza dalle ficobiliproteine e dalle clorofille di tutti i ROI misurati.
- Selezionare i dati di fluorescenza di ficoeritrina-ficocianobilina (PE-PCB; 620 nm), C-ficocianina (CPC; 648 nm), alloficocianina (APC; 660 nm) e clorofilla a (Chl a; 680 nm), rispettivamente, nel file csv.
- Creare una nuova tabella con tutti i valori massimi di fluorescenza ottenuti da ciascun picco di ficobiliproteina e clorofilla e tracciare i dati su un grafico.
- Eseguire analisi statistiche.
- Analizzare se i dati ottenuti soddisfano la normalità e l'omoscedasticità27.
- Procedere all'analisi t-test27,28 se la prima condizione è vera. In caso contrario, eseguire un test Mann-Whitney U29.
- Considera le differenze significative ai valori p <0,05.
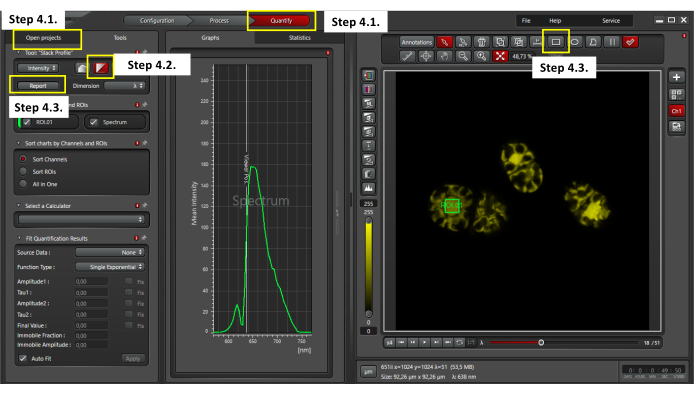
Figura 3: Valutazione dell'autofluorescenza di Chroothece. Per eseguire l'analisi di autofluorescenza, assicurarsi di selezionare la finestra di quantificazione e un file xyλ nella finestra dei progetti aperti (punto 4.1); Selezionare la visualizzazione del profilo dello stack (passaggio 4.2); selezionare un ROI di 4 μm2 al centro di una cella e fare clic sul pulsante Report per esportare i dati di scansione lambda in formato CSV (passaggio 4.3). Fare clic qui per visualizzare una versione ingrandita di questa figura.
Risultati
La clorofilla a generalmente assorbe le lunghezze d'onda blu e rosse della luce visibile, mentre le ficobiliproteine usano lunghezze d'onda verdi, gialle e arancioni7. L'autofluorescenza di questi pigmenti rende possibile il primo approccio per studiare le ficobiliproteine e il comportamento della clorofilla in condizioni sperimentali e di campo.
Confrontando i dati ottenuti e tracciando su grafici diversi, è possibile distinguere cambiamenti significativi nell'intensi...
Discussione
Alcune alghe rosse unicellulari o coloniali, come Chroothece, crescono lentamente in vitro, ma contengono più composti autofluorescenti che possono essere analizzati mediante analisi spettrale al microscopio confocale, dove possono essere rilevate differenze nei picchi di emissione di pigmento. La microscopia a fluorescenza confocale spettrale ha permesso di condurre studi in vivo per valutare l'adattamento o l'acclimatazione di organismi fotosintetici 8,10,17,18,19,20....
Divulgazioni
Gli autori non hanno conflitti di interesse da dichiarare.
Riconoscimenti
Questa ricerca è stata condotta nell'ambito dei progetti TIN2015-68454-R e 20961/PI/18, finanziati dal Ministero spagnolo dell'Economia e della Competitività e dalla Fondazione Séneca della Regione di Murcia. Irene Hernández Martínez e Francisco Javier Ibáñez López della Sezione di Supporto Statistico dell'Area Scientifica e di Ricerca dell'Università di Murcia (Sección de Apoyo Estadístico (SAE), Área Científica y de Investigación (ACTI), Universidad de Murcia, (Figura 1 sono stati disegnati utilizzando immagini di Servier Medical Art. Servier Medical Art by Servier è sotto licenza Creative Commons Attribuzione 3.0 Unported License (https://creativecommons.org/licenses/by/3.0/)
Materiali
| Name | Company | Catalog Number | Comments |
| µ-Dish 35 mm, high Glass Bottom | Ibidi | 81158 | - |
| 24 black well plate | Ibidi | 82406 | flat and clear bottom for high throughput microscopy |
| Algae Incubator | Panasonic | MLR-352-PE | |
| Confocal laser scanning microscope | Leica Microsystems | SP8 TCS | - |
| Flask | Fisher Scientific | 15380591 | Can be purchased in a local convenience store or online stores. |
| green filter | PNTA, LEE filters | - | Can be purchased in a local convenience store or online stores. |
| HC PL APO 63X/1.30 GLYC CORR CS2 | Leica Microsystems | 506353 | Glycerol immersion lens |
| Image acquisition software. LAS X | Leica Microsystems | SP8 TCS | - |
| Light source | Panasonic | FL40SSENW/37MLR-352-PE | |
| Quantum photoradiometer | DeltaOhm | DO 9721 | - |
| R software | R Core Team, 2020 | 4.0.2. | - |
| red filter | PNTA, LEE filters | - | Can be purchased in a local convenience store or online stores. |
| SWES medium | University of Murcia | - | - |
| Type G Immersion liquid | Leica Microsystems | 11513910 | Glycerol |
Riferimenti
- Vis, M. L., Necchi, O. . Freshwater Red Algae: Phylogeny, Taxonomy and Biogeography. , (2021).
- Aboal, M., et al. Diversity of Chroothece (Rhodophyta, Stylonematales) including two new species. European Journal of Phycology. 53 (2), 189-197 (2018).
- Millach, L., Obiol, A., Solé, A., Esteve, I. A novel method to analyse in vivo the physiological state and cell viability of phototrophic microorganisms by confocal laser scanning microscopy using a dual laser. Journal of Microscopy. 268 (1), 53-65 (2017).
- Millach, L., Villagrasa, E., Solé, A., Esteve, I. Combined confocal laser scanning microscopy techniques for a rapid assessment of the effect and cell viability of Scenedesmus sp. DE2009 under metal stress. Microscopy and Microanalysis. 25 (4), 998-1003 (2019).
- Poryvkina, L., Babichenko, S., Leeben, A. Analysis of phytoplankton pigments by excitation spectra of fluorescence. Proceedings of EARSeL-SIG-Workshop LIDAR. , (2000).
- Beutler, M., et al. A fluorometric method for the differentiation of algal populations in vivo and in situ. Photosynthesis Research. 72 (1), 39-53 (2002).
- Grigoryeva, N., Chistyakova, L. Fluorescence microscopic spectroscopy for investigation and monitoring of biological diversity and physiological state of cyanobacterial cultures. Cyanobacteria. , (2018).
- Grigoryeva, N. . Fluorescence Methods for Investigation of Living Cells and Microorganisms. , (2020).
- Roshchina, V. V. Vital autofluorescence: application to the study of plant living cells. International Journal of Spectroscopy. 2012, 5-18 (2012).
- Roldán, M., Thomas, F., Castel, S., Quesada, A., Hernández-Mariné, M. Noninvasive pigment identification in single cells from living phototrophic biofilms by confocal imaging spectrofluorometry. Applied and Environmental Microbiology. 70 (6), 3745-3750 (2004).
- Hense, B. A., Gais, P., Jütting, U., Scherb, H., Rodenacker, K. Use of fluorescence information for automated phytoplankton investigation by image analysis. Journal of Plankton Research. 30 (5), 587-606 (2008).
- Millie, D. F., Schofield, O. M., Kirkpatrick, G. J., Johnsen, G., Evens, T. J. Using absorbance and fluorescence spectra to discriminate microalgae. European Journal of Phycology. 37 (3), 313-322 (2002).
- Richardson, T. L., et al. Spectral fluorometric characterization of phytoplankton community composition using the Algae Online Analyser. Water Research. 44 (8), 2461-2472 (2010).
- Kieleck, C., Bousquet, B., Le Brun, G., Cariou, J., Lotrian, J. Laser induced fluorescence imaging: application to groups of macroalgae identification. Journal of Physics D: Applied Physics. 34 (16), 2561-2571 (2001).
- Coronado-Parra, T., Roldán, M., Aboal, M. Confocal microscopy in ecophysiological studies of algae: a door to understanding autofluorescence in red algae. Microscopy and Microanalysis. 28 (1), 218-226 (2022).
- Mulec, J., Kosi, G. Lampenflora algae and methods of growth control. Journal of Cave and Karst Studies. 71 (2), 109-115 (2009).
- Nelissen, B., De Baere, R., Wilmotte, A., De Wachter, R. Phylogenetic relationships of nonaxenic filamentous cyanobacterial strains based on 16S rRNA sequence analysis. Journal of Molecular Evolution. 42 (2), 194-200 (1996).
- Roldán, M., Oliva, F., Gónzalez Del Valle, M. A., Saiz-Jimenez, C., Hernández-Mariné, M. Does green light influence the fluorescence properties and structure of phototrophic biofilms. Applied and Environmental Microbiology. 72 (4), 3026-3031 (2006).
- Topinka, J. A., Bellows, W. K., Yentsch, C. S. Characterization of marine macroalgae by fluorescence signatures. International Journal of Remote Sensing. 11 (12), 2329-2335 (1990).
- Roldán, M., Ascaso, C., Wierzchos, J. Fluorescent fingerprints of endolithic phototrophic cyanobacteria living within halite rocks in the atacama desert. Applied and Environmental Microbiology. 80 (10), 2998-3006 (2014).
- Solé, A., Diestra, E., Esteve, I. Confocal laser scanning microscopy image analysis for cyanobacterial biomass determined at microscale level in different microbial mats. Microbial Ecology. 57 (4), 649-656 (2009).
- Ramírez, O., García, A., Rojas, R., Couve, A., Härtel, S. Confined displacement algorithm determines true and random colocalization in fluorescence microscopy. Journal of Microscopy. 239 (3), 173-183 (2010).
- Universität Gottingen-Georg-August. SAG Culture Collection of Algae. Universität Gottingen-Georg-August. , (2022).
- Zhang, M., et al. Improvement of cell counting method for Neubauer counting chamber. Journal of Clinical Laboratory Analysis. 34 (1), 23024 (2020).
- Wolf, E., Schüßler, A. Phycobiliprotein fluorescence of Nostoc punctiforme changes during the life cycle and chromatic adaptation: Characterization by spectral confocal laser scanning microscopy and spectral unmixing. Plant, Cell and Environment. 28 (4), 480-491 (2005).
- Zucker, R. M., Rigby, P., Clements, I., Salmon, W., Chua, M. Reliability of confocal microscopy spectral imaging systems: Use of multispectral beads. Cytometry Part A. 71 (3), 174-189 (2007).
- Field, A. M. . Discovering Statistics Using R. , (2013).
- Linnet, K. Limitations of the paired t-test for evaluation of method comparison data. Clinical Chemistry. 45 (2), 314-315 (1999).
- Rosner, B., Grove, D. Use of the Mann-Whitney U-test for clustered data. Statistics in Medicine. 18 (11), 1387-1400 (1999).
- Colin, L., et al. Imaging the living plant cell: from probes to quantification. The Plant Cell. 34 (1), 247-272 (2022).
- Borlinghaus, R. The white confical: continuous spectral tuning in excitation and emission. Optical Fluorescence Microscopy. , (2011).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon