Method Article
Aplicación del pseudovirus Ha-CoV-2 para la cuantificación rápida de variantes del SARS-CoV-2 y anticuerpos neutralizantes
En este artículo
Resumen
Este protocolo describe la aplicación de un nuevo pseudovirus híbrido alfavirus-SARS-CoV-2 (Ha-CoV-2) como plataforma para la cuantificación rápida de la infectividad de las variantes del SARS-CoV-2 y su sensibilidad a los anticuerpos neutralizantes.
Resumen
La pandemia de enfermedad por coronavirus 2019 (COVID-19) ha puesto de manifiesto la necesidad de realizar ensayos rápidos para medir con precisión la infectividad de las variantes emergentes del SARS-CoV-2 y la eficacia de los anticuerpos neutralizantes inducidos por la vacuna contra las variantes virales. Estos ensayos son esenciales para la vigilancia de la pandemia y la validación de vacunas y refuerzos específicos de variantes. Este manuscrito demuestra la aplicación de un nuevo pseudovirus híbrido alfavirus-SARS-CoV-2 (Ha-CoV-2) para la cuantificación rápida de la infectividad de la variante del SARS-CoV-2 y los anticuerpos neutralizantes inducidos por la vacuna contra las variantes virales. El Ha-CoV-2 es una partícula similar al virus SARS-CoV-2 que consta de proteínas estructurales virales (S, M, N y E) y un genoma de ARN de expresión rápida derivado de un alfavirus, el virus del bosque de Semliki (SFV). El Ha-CoV-2 también contiene genes de proteína fluorescente verde (GFP) y de luciferasa que permiten una rápida cuantificación de la infectividad viral. A modo de ejemplo, se cuantifica la infectividad de las variantes Delta del SARS-CoV-2 (B.1.617.2) y Ómicron (B.1.1.529), y también se mide su sensibilidad a un anticuerpo neutralizante (27VB). Estos ejemplos demuestran el gran potencial del Ha-CoV-2 como plataforma robusta para la cuantificación rápida de variantes del SARS-CoV-2 y su susceptibilidad a los anticuerpos neutralizantes.
Introducción
Hasta mayo de 2023, se habían registrado más de 766 millones de casos de COVID-191. A pesar de las campañas mundiales de vacunación, el SARS-CoV-2 circula continuamente e infecta a las personas, en gran parte debido a la aparición de nuevas variantes como Delta (B.1.617.2) y Ómicron (B.1.1.529) que impulsan nuevas olas de infección 2,3,4. Dado que el SARS-CoV-2 está en constante evolución, es importante desarrollar ensayos rápidos que puedan medir con precisión la infectividad de las variantes emergentes y la eficacia de los anticuerpos neutralizantes inducidos por la vacuna contra estas variantes. Estos ensayos son esenciales para la vigilancia de la pandemia y para determinar la eficacia de las vacunas y sus refuerzos específicos para cada variante.
Debido a la naturaleza altamente contagiosa del SARS-CoV-2, el Centro para el Control y la Prevención de Enfermedades (CDC, por sus siglas en inglés) exige que el estudio del SARS-CoV-2 y sus variantes se realice en instalaciones de nivel de bioseguridad (BSL) 3 5,6. Este requisito de BSL-3 limita el uso de virus vivos para cuantificar la infectividad de las variantes virales y sus anticuerpos neutralizantes en laboratorios clínicos y de investigación comunes. Además, los ensayos tradicionales de neutralización del SARS-CoV-2, como los ensayos basados en el efecto de la placa o del efecto citopático que utilizan virus vivos competentes para la replicación, requieren mucho tiempo y largos períodos de incubación7. Se han desarrollado varios pseudovirus del SARS-CoV-2 pseudotipados con proteína de la espícula (S) para cuantificar la eficacia de los anticuerpos neutralizantes 8,9,10,11,12. En el SARS-CoV-2, la proteína S es la principal proteína que media la entrada viral13 y es el principal antígeno utilizado en las vacunas contra el SARS-CoV-2 9,10,14,15,16. Los viriones pseudotipados de proteína S, como los del virus de la estomatitis vesicular (VSV-G) o lentivirus, se han utilizado para la cuantificación de anticuerpos neutralizantes 17,18,19. Sin embargo, el pseudovirus basado en lentivirus normalmente requiere de 2 a 3 días de infección para cuantificar las señales indicadoras. Los sistemas de pseudovirus basados en VSV a menudo contienen virus VSV residuales, lo que puede dar lugar a altas tasas de resultados falsos positivos y, por lo general, requiere 24 horas de infección20.
Un nuevo sistema de pseudovirus del SARS-CoV-2, el pseudovirus híbrido alfavirus-SARS-CoV-2 (Ha-CoV-2), ha sido desarrollado recientemente por Hetrick et al12. El Ha-CoV-2 proporciona una nueva herramienta para la cuantificación rápida de la infectividad del virus y la sensibilidad del virus a los anticuerpos neutralizantes en los laboratorios comunes de BSL-2. Estructuralmente, el Ha-CoV-2 se asemeja a la partícula de virión del SARS-CoV-2, que consta de proteínas estructurales del SARS-CoV-2, incluida la proteína S (S), la membrana (M), la nucleocápside (N) y la envoltura (E), y no hay proteína estructural de otros virus. Además, la partícula Ha-CoV-2 contiene un genoma de ARN de expresión rápida de un alfavirus para una rápida expresión reportera en las células. Se ha demostrado que el Ha-CoV-2 mide rápidamente la actividad neutralizante de los anticuerpos en los sueros de individuos vacunados y convalecientes12. Como demostraron Hetrick et al., cuando se comparó con el pseudovirus SARS-CoV-2 basado en lentivirus en un ensayo de curso temporal, el Ha-CoV-2 expresó el reportero de Luc tan pronto como 2-4 h después de la infección, mientras que el pseudovirus de lentivirus expresó Luc después de 24 h12. Además, la aplicación potencial de las variantes de Ha-CoV-2 para cuantificar anticuerpos neutralizantes se demuestra aún más mediante el uso de un anticuerpo neutralizante monoclonal estándar, el 27BV (véase la figura complementaria 1)12. En este trabajo se detalla el uso de la plataforma Ha-CoV-2 para la cuantificación rápida de la infectividad de las variantes del SARS-CoV-2, utilizando como ejemplos las variantes Delta (B.1.617.2) y Ómicron (B.1.1.529). Además, la aplicación potencial de las variantes de Ha-CoV-2 para cuantificar anticuerpos neutralizantes se demuestra aún más mediante el uso de un anticuerpo neutralizante monoclonal estándar, 27BV12.
Protocolo
1. Ensamblaje de virus y partículas virales
- Vectores: Compre comercialmente vectores de expresión para SARS-CoV-2 M, E o N, así como para SARS-CoV-2 S (tipo salvaje, Wt), Delta (B.1.617.2) y Ómicron (B.1.1.529).
NOTA: Las secuencias de proteínas de los vectores de expresión se proporcionan en el Archivo Suplementario 1. El proveedor de estos vectores de expresión también se puede encontrar en la Tabla de materiales. - Células y cultivo celular: Mantener HEK293T células en el medio de Eagle modificado (DMEM) de Dulbecco que contiene un 10% de suero fetal bovino (FBS) inactivado por calor, 50 unidades/ml de penicilina y 50 μg/ml de estreptomicina.
NOTA: Todo el trabajo de cultivo celular debe realizarse en una cabina de bioseguridad de flujo de aire laminar. La transfección de polietileno (PEI) suele requerir una incubación de 5-6 h. Los horarios de inicio deben considerarse cuidadosamente de antemano. - Ensamblaje de virus y cotransfección basada en PEI: Ensamblaje de partículas de Ha-CoV-2 mediante cotransfección de células HEK293T. Células semilla en una placa de Petri de cultivo celular de 10 cm (4-5 x 106 células por placa) en 10 mL de medio DMEM completo el día anterior a la cotransfección. Para este estudio, se utilizaron tres placas de Petri para sembrar células para el ensamblaje de Ha-CoV-2 de tipo salvaje, Delta y Ómicron, respectivamente.
- Incubar placas de Petri durante la noche en una incubadora deCO2 a 37 °C. Revise los platos a la mañana siguiente para asegurarse de que las células tengan un 80% de confluente. Retire el medio completo y reemplácelo con 9 ml de medio sin suero DMEM.
- Para cada plato, prepare una mezcla de cotransfección con 2,5 μg de cada uno de los vectores de expresión de proteínas estructurales del SARS-COV-2 (N, E, M), 10 μg de pAlphaPro-Luc-GFP-PreΨ (genoma de HaCoV2) y 2,5 μg del vector de expresión de la proteína S, ya sea la variante Delta u Ómicron S y 45 μL de reactivo de transfección basado en PEI. Deje que la mezcla de cotransfección forme complejos incubando durante 13 minutos (no incube durante más de 30 minutos).
- Después de la incubación, agregue la mezcla de cotransfección a cada placa de Petri lentamente, gota a gota. Colocar los platos dentro de una incubadora deCO2 a 37 °C durante 6 h. Después de 6 h, retire el DMEM sin suero y reemplácelo con un medio DMEM completo. Cosechar el virus a las 48-60 h después de la cotransfección.
- Recolección y almacenamiento de virus: Recolectar partículas a las 48 h después de la cotransfección. Separe las células pipeteando repetidamente sobre la superficie de la monocapa y recoja las células de cada placa, colóquelas en tubos de centrífuga de 15 ml y centrifugue a 400 x g durante 5 min. Recoger el sobrenadante y pasarlo por un filtro de 0,22 μM. Almacenar el pseudovirus Ha-CoV-2 a -80 °C.
2. Ensayo de infectividad viral
- Células y cultivo celular: Mantener células HEK293T(ACE2/TMPRSS2) en el medio de Eagle modificado de Dulbecco (DMEM) que contiene un 10% de FBS inactivado por calor, 50 unidades/ml de penicilina y 50 μg/ml de estreptomicina.
- Siembra de células HEK293T (ACE2/TMPRSS2): El día antes del ensayo de infectividad viral, siembre células HEK293T (ACE2/TMPRSS2) en una placa de 96 pocillos en 50 μL de medio DMEM completo. Para cada placa de 96 pocillos, siembre 2,5 x 104 celdas en cada pocillo, y se necesita un total de 2,5 x 106 celdas para una placa. Coloque la placa de 96 pocillos en una incubadora deCO2 a 37 °C durante la noche.
- Infección de HEK293T(ACE2/TMPRSS2): Usar partículas variantes de Ha-CoV-2 para infectar células HEK293T(ACE2/TMPRSS2). En la mañana de la infección, retire 50 μL de DMEM de la placa de 96 pocillos presembrada. Reemplace el medio con 50 μL de Ha-CoV-2 de tipo salvaje, Delta u Ómicron durante 18 h a 37 °C.
NOTA: El protocolo se puede detener aquí hasta que la infección haya alcanzado las 18 h de incubación y la placa esté lista para ser analizada mediante el ensayo de luciferasa. El éxito de la infección está determinado por el ensayo de luciferasa resultante del gen reportero de la luciferasa expresado en las células infectadas, por lo tanto, cuanta más señal se produce, más exitosa es la infección de la variante Ha-CoV-2. - Ensayo de luciferasa: Después de 18 h de incubación, añadir 7,5 μL de tampón de lisis celular directamente a cada pocillo y mezclar mediante agitación orbital durante 2 min. Lisar las células en tampón de lisis durante al menos 5 minutos a temperatura ambiente.
- Prepare la solución del ensayo de luciferasa Firefly mezclando la solución de D-luciferina con la solución del ensayo Luciferasa Firefly en una proporción de 1:50. Para obtener una placa completa de 96 pocillos, combine 3 ml de la solución de sustrato de luciferasa con 60 μl de la solución de D-luciferina con 2940 μl de la solución tampón de luciferasa Firefly.
- Añadir 25 μL de la solución de la prueba Luciferasa Firefly a los lisados celulares y mezclar la placa agitando orbitalmente durante 1 min. Analice la actividad de la luciferasa utilizando un lector comercial de microplacas de luciferasa.
3. Extracción de ARN de Ha-CoV-2 y PCR cuantitativa con transcriptasa inversa (RT-qPCR)
- Extracción de ARN viral: Extraiga el ARN viral de las partículas de Ha-CoV-2 tipo salvaje y de las variantes Delta y Ómicron de Ha-CoV-2 utilizando un kit comercial de extracción de ARN viral, siguiendo las instrucciones del fabricante. Almacene el ARN viral extraído a -80 °C o utilícelo inmediatamente para RT-qPCR.
- RT-qPCR: Realice RT-qPCR en ARN viral utilizando una mezcla maestra de un solo paso. Realice la reacción en una máquina de PCR comercial. Tenga en cuenta que el objetivo de la amplificación es el ARN genómico del Ha-CoV-2. Utilice el ADN vectorial Ha-CoV-2 como estándar para crear una curva estándar y calcular el número de copias de ARN de cada variante.
4. Ensayo de anticuerpos neutralizantes
- Siembra de células HEK293T (ACE2/TMPRSS2): El día anterior al ensayo, siembra células HEK293T (ACE2/TMPRSS2) en una placa de 96 pocillos en 50 μL de medio DMEM completo. Las células HEK293T(ACE2/TMPRSS2) se compran comercialmente.
- Para el recuento de células, se obtienen 20 μL de células de un matraz T75 que contenga células HEK293T(ACE2/TMPRSS2) y se mezcla con 20 μL de solución de azul de tripano. Agregue 20 μL de esta mezcla a la cámara de conteo celular y cuente el número de células por mL. Para sembrar una placa de 96 pocillos para la infección, use 2.5 x 104 celdas por pocillo, y se necesitarán 2.5 x 106 celdas en total. Coloque la placa de 96 pocillos en una incubadora deCO2 a 37 °C durante la noche.
- Ensayo de anticuerpos neutralizantes: En una placa estéril de polipropileno de 96 pocillos, prepare la mezcla estándar de anticuerpos neutralizantes 27BV y Ha-CoV-2. Añadir 8 μL de 27BV (45 mg/mL) a la placa y realizar diluciones seriadas del anticuerpo con 6 μL de DMEM sin suero.
NOTA: Asegúrese de intercambiar las puntas de pipeta entre las transferencias de pocillos y asegúrese de que el medio libre de anticuerpos y suero se mezclen bien para producir resultados precisos. - Al anticuerpo diluido en serie, añadir 54 μL de partícula Ha-CoV-2 y mezclar el virus y el anticuerpo. Preincubar partículas de Ha-CoV-2 con 27BV diluido en serie durante 1 h a 37 °C con 5% de CO2. Después de 1 h de incubación, aplique 50 μL de la mezcla de anticuerpos y Ha-CoV-2 a la placa de 96 pocillos que contiene las células HEK293T(ACE2/TMPRSS2) (2,5 x 104 células por pocillo) sembradas el día anterior.
- Para los controles, deje al menos tres pocillos que contengan solo las células HEK293T(ACE2/TMPRSS2). Agregue 50 μL de medio completo a estos pocillos para que sirvan como pocillos no infectados para la señal de fondo de las lecturas del ensayo de luciferasa.
NOTA: El protocolo se puede detener aquí hasta que la infección haya alcanzado las 18 h de incubación a 37 °C, y entonces la placa esté lista para ser analizada mediante ensayo de luciferasa. - Ensayo de luciferasa: Después de 18 h de incubación, añadir 7,5 μL de tampón de lisis directamente a cada pocillo y mezclar mediante agitación orbital durante 2 min. Lisar las células en tampón de lisis durante al menos 5 minutos a temperatura ambiente.
- Prepare la solución del ensayo de luciferasa Firefly mezclando la solución de D-luciferina con la solución del ensayo Luciferasa Firefly en una proporción de 1:50. Para obtener una placa completa de 96 pocillos, combine 3 ml de la solución de sustrato de luciferasa con 60 μl de la solución de D-luciferina con 2940 μl de la solución tampón de luciferasa de luciérnaga de luciérnaga.
- Añadir 25 μL de la solución de la prueba Luciferasa Firefly a los lisados celulares y mezclar la placa agitando orbitalmente durante 1 min. Analice la actividad de la luciferasa utilizando un lector comercial de microplacas de luciferasa.
5. Cuantificación y análisis estadístico
- Obtención de datos: Realizar ensayos de infección y luciferasa por triplicado como se indica (Figura 1). Cuantifique la expresión de luciferasa con lecturas de ensayos de luciferasa. La media es el valor promedio de las tres lecturas del ensayo de luciferasa. Las lecturas de la señal de fondo de los pozos no infectados se restan de este valor medio. La desviación estándar (DE) se determina a partir del valor promedio de las lecturas del ensayo de luciferasa.
- Análisis de datos: Grafique la actividad de neutralización de anticuerpos y calcule los valores de ID50 (dosis de inhibición del 50%) utilizando un software de gráficos comercial. El valor ID50 se define como las diluciones inhibitorias de anticuerpos en las que se logra una reducción del 50% en la infección de las células HEK293T(ACE2/TMPRSS2) (según las lecturas del ensayo de luciferasa).
Resultados
Las partículas de Ha-CoV-2 se ensamblaron utilizando cinco vectores de ADN diferentes que expresan el genoma de ARN de Ha-CoV-2 y las proteínas estructurales (M, N, E y S) del SARS-CoV-2 en células HEK293T. El vector de la proteína S varía en función de la variante S. La proteína S de la cepa original Ha-CoV-2 de Wuhan (Wild-type, Wt) se utilizó como control positivo, y se ensambló junto con la proteína S de cada una de las otras dos variantes: Delta (B.1.617.2) u Ómicron (B.1.1.529). Los mismos M, N, E se utilizaron en todas las variantes. El Ha-CoV-2(Wt) y las partículas variantes se recolectaron 48 h después de la cotransfección y luego se usaron para infectar células HEK293T(ACE2/TMPRSS2). La infectividad se midió por la expresión de luciferasa a las 18 h post-infección. En este sistema, los niveles más altos de expresión de la señal de luciferasa reflejan una mayor infección de las células por Ha-CoV-2. La señal de luciferasa se normalizó con copias genómicas de ARN mediante RT-qPCR para cada variante. Como se muestra en la Figura 1, la variante Ómicron del Ha-CoV-2 generó una señal de 4 a 10 veces mayor que la Ha-CoV-2(Wt) original, lo que sugiere una mayor infectividad.
Además, se cuantificó la capacidad de 27BV para neutralizar las variantes HaCoV-2(Wt), Delta y Ómicron. 27BV es un anticuerpo monoclonal de conejo que se desarrolló contra el dominio RBD de la proteína S1 del SARS-COV-2. Para los ensayos de neutralización, se realizaron diluciones seriadas de 27BV en una placa de 96 pocillos, preincubada con Ha-CoV-2 y luego se agregó a las células diana HEK293T(ACE2/TMPRSS2). Los resultados demostraron que el 27BV tuvo actividad neutralizante contra todas las variantes probadas (Figura 2). Curiosamente, el ID50 de 27VB para Ómicron fue aproximadamente 10 veces menos potente que el ID50 para Ha-CoV-2 (WT) y Ha-CoV-2 (Delta; Figura 2). Estos resultados demuestran que la plataforma Ha-COV-2 puede utilizarse como un método rápido para cuantificar anticuerpos neutralizantes inducidos por vacunas en variantes emergentes.
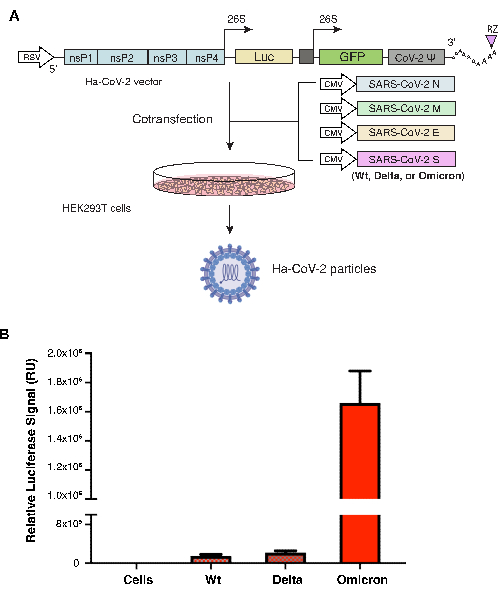
Figura 1. Montaje y cuantificación de variantes de Ha-CoV-2. (A) Ilustración del ensamblaje del Ha-CoV-2 y las partículas variantes. Los vectores que expresan el genoma reportero de Ha-CoV-2 y las proteínas estructurales (M, S, N y E) se cotransfectan en HEK293T células. Las partículas se recolectaron 48 h después de la cotransfección (la imagen de la partícula de virión y las células de HEK293T se crearon con Biorender.com). (B) Cuantificación de la infectividad de las variantes de Ha-CoV-2. La infectividad relativa de las dos variantes (Delta y Ómicron) se cuantifica y normaliza utilizando copias genómicas de ARN de variantes individuales de Ha-CoV-2 (Luc). El tipo salvaje es el Ha-COV-2 (Wt), que se utiliza como control para la comparación. Los ensayos de infección y luciferasa se realizaron 3x. RU, unidad relativa. Se muestran la media y la desviación estándar (DE). Haga clic aquí para ver una versión más grande de esta figura.
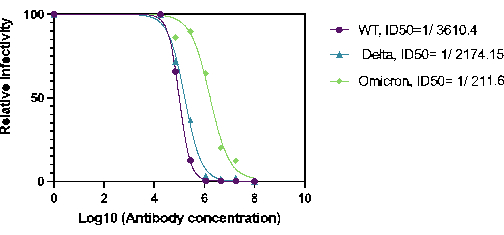
Figura 2. Cuantificación de la actividad de neutralización del 27BV frente a las variantes del Ha-CoV-2(Luc) Se analizó la actividad de neutralización del 27BV 18 h después de la infección de las células HEK293T(ACE2/TMPRSS2). El ID50 se calculó utilizando la tasa de infección relativa (actividad luciferasa) frente a la concentración de 27BV. Haga clic aquí para ver una versión más grande de esta figura.
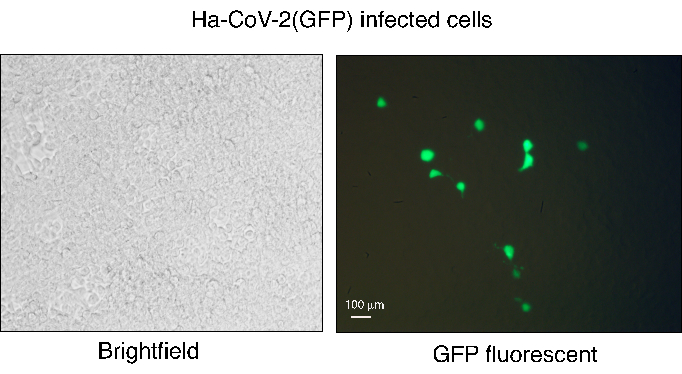
Figura 3. Infección por HEK293T(ACE2/TMPRSS2) con Ha-CoV-2(GFP). Las partículas de Ha-CoV-2 (GFP) se ensamblaron y luego se usaron para infectar células HEK293T (ACE2/TMPRSS2). La expresión de GFP se observó a las 48 h post-infección mediante microscopía fluorescente. El campo blanco de las células infectadas se muestra a la izquierda y las imágenes de GFP se muestran a la derecha. La barra blanca representa 100 μm. Haga clic aquí para ver una versión más grande de esta figura.
Figura complementaria 1. Resumen gráfico. La estructura y aplicación del pseudovirus Ha-CoV-2. Imagen creada con Biorender.com. Haga clic aquí para descargar este archivo.
Expediente Complementario 1. Secuencias de proteínas. Lista de las secuencias de las proteínas S, M, N y E del SARS-CoV-2. Las secuencias de la proteína S también incluyen las variantes del SARS-CoV-2 Ómicron (B.1.1.529) y Delta (B.1.617.2). Haga clic aquí para descargar este archivo.
Discusión
La plataforma Ha-CoV-2 proporciona un flujo de trabajo rápido, robusto y sencillo para cuantificar variantes virales y neutralizar anticuerpos. Sin embargo, hay algunos pasos críticos que requieren atención. La producción del pseudovirus Ha-CoV-2 debe realizarse utilizando células HEK293T con alta viabilidad. La eficiencia de la cotransfección se puede monitorizar 24 h después de la transfección utilizando el gen reportero GFP del genoma del Ha-CoV-2. El genoma del Ha-CoV-2 puede contener dos reporteros (GFP y Luc), y la GFP puede expresarse durante la cotransfección y después de la infección por Ha-CoV-2 de las células diana12. Las células GFP+ de la infección normalmente se encuentran en un porcentaje bajo (1% a 5%), pero cada célula infectada expresa fuertes señales de GFP (Figura 3). Este bajo porcentaje de GFP puede limitar el uso de GFP como una lectura robusta para cuantificar la neutralización de anticuerpos, en comparación con el reportero de Luc, que cuantifica toda la población de células infectadas.
Al realizar el ensayo de neutralización, es esencial cambiar las puntas de pipeta entre las transferencias de pocillos y asegurarse de que el anticuerpo y el medio libre de suero se mezclen bien para producir resultados precisos. Además, al realizar el protocolo de ensayo de luciferasa, las células deben lisarse completamente durante al menos 3 minutos para garantizar la lisis completa de las células y la liberación de la enzima luciferasa. Esto garantizará la precisión del ensayo. Además, una vez que la solución del ensayo Luciferasa Firefly se agrega a las placas ópticas de 96 pocillos de paredes blancas, la placa debe analizarse dentro de los 10 minutos, ya que la emisión de luz inicial es alta, pero disminuye con el tiempo a medida que se agota el ATP21.
A medida que más variantes del SARS-CoV-2 continúan evolucionando, existe una mayor necesidad de plataformas como Ha-CoV-2 para detectar rápidamente la infectividad de la variante y la sensibilidad de la variante a los anticuerpos neutralizantes inducidos por la vacuna. La plataforma Ha-CoV-2 ofrece una velocidad más rápida, una mayor relación señal-ruido y un protocolo simple en comparación con los ensayos de neutralización basados en pseudovirus existentes 8,9,10,11. La plataforma Ha-CoV-2 también ofrece la ventaja de que se puede utilizar en laboratorios BSL-2 y no requiere el uso de instalaciones BSL-3. Esto permite que la investigación del SARS-CoV-2 se lleve a cabo en laboratorios clínicos y de investigación comunes. Además, la plataforma Ha-CoV-2 produce resultados rápidos en comparación con otros sistemas. Por ejemplo, el estudio de los anticuerpos neutralizantes contra el virus infeccioso SARS-CoV-2 a menudo hace uso de la prueba de neutralización por reducción de placa (PRINT)22. A pesar de que PRINT produce resultados confiables, el conteo manual de las unidades formadoras de placas (UFP) es lento y requiere de 3 a 5 días para obtener resultados23,24. Otros sistemas de pseudotipos, como el lentivirus-pseudovirus, necesitan de 24 a 72 h para producir una señal reportera detectable12. En comparación, el ensayo de neutralización de Ha-CoV-2 puede generar resultados en 18 h. El Ha-CoV-2 proporciona una herramienta conveniente para la detección y cuantificación rápidas de variantes virales y anticuerpos neutralizantes para el monitoreo de pandemias.
El monitoreo de la infectividad del SARS-CoV-2 es esencial a medida que continúan surgiendo más variantes preocupantes (COV). El Ha-CoV-2 ofrece la ventaja de determinar rápidamente la infectividad de los COV. Estudios anteriores han utilizado modelos basados en inteligencia artificial (IA) para analizar cuantitativamente la infectividad de la subvariante Ómicron y las otras variantes del SARS-CoV-2, como la variante Delta25. Estos estudios han demostrado que la variante Ómicron es más contagiosa que el virus original y tiene más probabilidades de escapar a los anticuerpos neutralizantes25. En estos estudios, utilizando Ha-CoV-2, se observaron fenotipos similares. Además, en los ensayos de neutralización de anticuerpos, la variante Ómicron tiene diez veces menos probabilidades de ser neutralizada por 27BV que las cepas de Wuhan y Delta. Estos resultados también son consistentes con la mayor transmisibilidad reportada de la variante Ómicron, que tiene al menos 15 mutaciones en su dominio de unión al receptor (RBD), lo que probablemente aumente la afinidad de unión viral al receptor ACE2 para una mayor transmisibilidad y un mayor escape inmunológico26.
Divulgaciones
La Universidad George Mason ha presentado una patente y ha concedido una licencia a Virongy Biosciences Inc. para el desarrollo de productos. Y.W. es fundador y miembro del consejo asesor de Virongy Biosciences. B. H es actualmente el CEO y Director Científico de Virongy Biosciences. Los autores no tienen ningún otro conflicto de intereses.
Agradecimientos
Este trabajo fue apoyado por el fondo interno de investigación de la Universidad George Mason.
Materiales
| Name | Company | Catalog Number | Comments |
| 27VB1 20 µg SARS-CoV-2 Standard Neutralizing Antibody | Virongy Biosciences | 27VBI-01 | |
| 500 mL - US Origin FBS | Neuromics | FBS001 | |
| AB Mixing Plate: Olympus 96-Well PCR Plate, Non-Skirted UltraThin Wall, Natural, 25 Plates/Unit | Genesee Scientific | Cat# 24-300 | |
| Allegra 6R Centrifuge | Beckman Coulter | 2043-30-1158 | |
| DMEM (1x) | ThermoFisher | 11995-073 | |
| GenClone 25-209, TC Treated Flasks, 250ml, Vent Growth Area: 75.0cm2, 5 per Sleeve, 100 Flasks/Unit | Genesee Scientfic | 25-209 | |
| GlowMax Discover Microplate reader | Promega | GM3000 | |
| Ha-CoV-2 E Vector | Virongy Biosciences | pCoV2_E | |
| Ha-CoV-2 M Vector | Virongy Biosciences | pCoV2_M | |
| Ha-CoV-2 N Vector | Virongy Biosciences | pCoV2_N | |
| Ha-CoV-2 WT S Vector | Virongy Biosciences | pCoV2_WT S | |
| Hek293T cells | ATCC | CRL-3214 | |
| Illumination Firefly Luciferase Enhanced Assay Kit 1000 assays | Gold Bio | I-930-1000 | |
| Infection Plate: 96-Well Tissue Culture Plate, Greiner Bio-One (With Lid, μClear White Flat Round, Chimney) | VWR | Cat# 82050-758 | |
| pAlphaPro-Luc-GFP-PreΨ (Ha-CoV-2 Genome) Vector | In house | ||
| PEI-based Transfection Reagent | Virongy Biosciences | Transfectin | |
| Penicillin-Streptomycin-Glutamine (100X) | Invitrogen | 10378016 | |
| Polyethylenimine, branched | Millipore Sigma | 408727-100ML | |
| QuantStudio 7 Pro Real-Time PCR System | ThermoFisher | A43163 | |
| Ready to use (HEK293T)(ACE2/TMPRSS2) Cells | Virongy Biosciences | Ready-To-Use-Cells | |
| SARS-CoV-2 S Omicron (B.1.1.529) Vector | Virongy Biosciences | pCoV2-B.1.1.529 | |
| SARS-CoV-2 S Delta (B.1.617.2) Vector | Virongy Biosciences | pCoV2- B.1.617.2 | |
| Syringe Filters, PES, 0.22µm | Genesee Scientfic | 25-244 | |
| TaqMan Fast Virus 1-Step Master Mix | ThermoFisher | 4444432 | |
| Trypan Blue Solution, 0.4% | ThermoFisher | 15250061 |
Referencias
- World Health Organization. Weekly epidemiological update on COVID-19. 143, 1(2023).
- Dhama, K., et al. Global emerging Omicron variant of SARS-CoV-2: Impacts, challenges and strategies. Journal of Infection and Public Health. 16 (1), 4-14 (2023).
- Dhama, K., et al. Coronavirus Disease 2019-COVID-19. Clinical Microbiology Reviews. 33 (4), e00028-e00020 (2020).
- El-Shabasy, R. M., et al. Three waves changes, new variant strains, and vaccination effect against COVID-19 pandemic. International Journal of Biological Macromolecules. 204, 161-168 (2022).
- CDC. Interim laboratory biosafety guidelines for handling and processing specimens associated with coronavirus disease 2019 (COVID-19). , https://www.cdc.gov/coronavirus/2019-nCoV/lab/lab-biosafety-guidelines.html (2021).
- Kaufer, A., Theis, T., Lau, K., Gray, J., Rawlinson, W. Laboratory biosafety measures involving SARS-CoV-2 and the classification as a Risk Group 3 biological agent. Pathology. 52 (7), 790-795 (2020).
- Vanderheiden, A., et al. Development of a Rapid Focus Reduction Neutralization Test Assay for Measuring SARS-CoV-2 Neutralizing Antibodies. Current Protocols in Immunology. 131 (1), e116(2020).
- Dieterle, M. E., et al. A Replication-Competent Vesicular Stomatitis Virus for Studies of SARS-CoV-2 Spike-Mediated Cell Entry and Its Inhibition. Cell Host & Microbe. 28 (3), 486-496 (2020).
- Nie, J., et al. Quantification of SARS-CoV-2 neutralizing antibody by a pseudotyped virus-based assay. Nature Protocols. 15 (11), 3699-3715 (2020).
- Nie, J., et al. Establishment and validation of a pseudovirus neutralization assay for SARS-CoV-2. Emerging Microbes & Infections. 9 (1), 680-686 (2020).
- Yang, R., et al. Development and effectiveness of pseudotyped SARS-CoV-2 system as determined by neutralizing efficiency and entry inhibition test in vitro. Biosafety and Health. 2 (4), 226-231 (2020).
- Hetrick, B., et al. Development of a hybrid alphavirus-SARS-CoV-2 pseudovirion for rapid quantification of neutralization antibodies and antiviral drugs. Cell reports methods. 2 (3), 100181(2022).
- Jackson, C., Farzan, M., Chen, B., Choe, H. Mechanisms of SARS-CoV-2 entry into cells. Nature Reviews. Molecular Cell Biology. 23 (1), 3-20 (2022).
- Jackson, L. A., et al. An mRNA Vaccine against SARS-CoV-2 - Preliminary Report. The New England Journal of Medicine. 383 (20), 1920-1931 (2020).
- Polack, F. P., et al. Safety and Efficacy of the BNT162b2 mRNA Covid-19 Vaccine. The New England Journal of Medicine. 383 (27), 2603-2615 (2020).
- Li, F. Structure, function, and evolution of coronavirus spike proteins. Annual Review of Virology. 3 (1), 237-261 (2016).
- Donofrio, G. A Simplified SARS-CoV-2 Pseudovirus Neutralization Assay. Vaccines. 9 (4), 389(2021).
- Crawford, K. H. D., et al. Protocol and Reagents for Pseudotyping Lentiviral Particles with SARS-CoV-2 Spike Protein for Neutralization Assays. Viruses. 12 (5), 513(2020).
- Zettl, F., et al. Rapid Quantification of SARS-CoV-2-Neutralizing Antibodies Using Propagation-Defective Vesicular Stomatitis Virus Pseudotypes. Vaccines. 8 (3), 386(2020).
- Li, Q., Liu, Q., Huang, W., Li, X., Wang, Y. Current status on the development of pseudoviruses for enveloped viruses. Reviews in Medical Virology. 28 (1), e1963(2018).
- Lundin, A. Optimization of the firefly luciferase reaction for analytical purposes. Advances in Biochemical Engineering/Biotechnology. 145, 31-62 (2014).
- Deshpande, G. R., et al. Neutralizing antibody responses to SARS-CoV-2 in COVID-19 patients. The Indian Journal of Medical Research. 152 (1 & 2), 82-87 (2020).
- Chen, C., et al. Research progress in methods for detecting neutralizing antibodies against SARS-CoV-2. Analytical Biochemistry. 673, 115199(2023).
- Manischewitz, J., et al. Development of a novel vaccinia-neutralization assay based on reporter-gene expression. The Journal of Infectious Diseases. 188 (3), 440-448 (2003).
- Chen, J., Wang, R., Gilby, N., Wei, G. Omicron Variant (B.1.1.529): Infectivity, Vaccine Breakthrough, and Antibody Resistance. Journal of Chemical Information and Modeling. 62 (2), 412-422 (2022).
- Saxena, S. K., et al. Characterization of the novel SARS-CoV-2 Omicron (B.1.1.529) variant of concern and its global perspective. Journal of Medical Virology. 94 (4), 1738-1744 (2022).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoExplorar más artículos
This article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados