Method Article
Applicazione dello pseudovirus Ha-CoV-2 per la quantificazione rapida delle varianti di SARS-CoV-2 e degli anticorpi neutralizzanti
In questo articolo
Riepilogo
Questo protocollo descrive l'applicazione di un nuovo pseudovirus ibrido alfavirus-SARS-CoV-2 (Ha-CoV-2) come piattaforma per la quantificazione rapida dell'infettività delle varianti di SARS-CoV-2 e della loro sensibilità agli anticorpi neutralizzanti.
Abstract
La pandemia di coronavirus 2019 (COVID-19) ha evidenziato la necessità di test rapidi per misurare con precisione l'infettività delle varianti emergenti di SARS-CoV-2 e l'efficacia degli anticorpi neutralizzanti indotti dal vaccino contro le varianti virali. Questi test sono essenziali per la sorveglianza della pandemia e per la convalida dei vaccini e dei richiami specifici per le varianti. Questo manoscritto dimostra l'applicazione di un nuovo pseudovirus ibrido alfavirus-SARS-CoV-2 (Ha-CoV-2) per una rapida quantificazione dell'infettività della variante SARS-CoV-2 e degli anticorpi neutralizzanti indotti dal vaccino contro le varianti virali. Ha-CoV-2 è una particella simile al virus SARS-CoV-2 costituita da proteine strutturali virali (S, M, N ed E) e da un genoma a RNA a rapida espressione derivato da un alfavirus, il Semliki Forest Virus (SFV). Ha-CoV-2 contiene anche sia la proteina fluorescente verde (GFP) che i geni reporter della luciferasi che consentono una rapida quantificazione dell'infettività virale. Ad esempio, viene quantificata l'infettività delle varianti SARS-CoV-2 Delta (B.1.617.2) e Omicron (B.1.1.529) e viene misurata anche la loro sensibilità a un anticorpo neutralizzante (27VB). Questi esempi dimostrano il grande potenziale di Ha-CoV-2 come solida piattaforma per la quantificazione rapida delle varianti di SARS-CoV-2 e della loro suscettibilità agli anticorpi neutralizzanti.
Introduzione
A maggio 2023, ci sono stati più di 766 milioni di casi di COVID-191. Nonostante le campagne di vaccinazione in tutto il mondo, il SARS-CoV-2 circola continuamente e infetta le persone, in gran parte a causa dell'emergere di nuove varianti come Delta (B.1.617.2) e Omicron (B.1.1.529) che guidano nuove ondate di infezione 2,3,4. Dato che il SARS-CoV-2 è in continua evoluzione, è importante sviluppare test rapidi in grado di misurare con precisione l'infettività delle varianti emergenti e l'efficacia degli anticorpi neutralizzanti indotti dal vaccino contro queste varianti. Questi test sono essenziali per la sorveglianza della pandemia e per determinare l'efficacia dei vaccini e dei loro richiami specifici per le varianti.
A causa della natura altamente contagiosa del SARS-CoV-2, il Center for Disease Control and Prevention (CDC) richiede che lo studio del SARS-CoV-2 e delle sue varianti sia condotto in strutture con livello di biosicurezza (BSL) 3 5,6. Questo requisito BSL-3 limita l'uso di virus vivi per quantificare l'infettività delle varianti virali e dei loro anticorpi neutralizzanti nei comuni laboratori di ricerca e clinici. Inoltre, i tradizionali saggi di neutralizzazione del SARS-CoV-2, come i saggi basati sull'effetto della placca o del citopatico che utilizzano virus vivi competenti per la replicazione, richiedono molto tempo e lunghi periodi di incubazione7. Diversi pseudovirus SARS-CoV-2 pseudotipizzati con proteina spike (S) sono stati sviluppati per quantificare l'efficacia degli anticorpineutralizzanti 8,9,10,11,12. Nel SARS-CoV-2, la proteina S è la principale proteina che media l'ingresso virale13 ed è il principale antigene utilizzato nei vaccini SARS-CoV-2 9,10,14,15,16. I virioni pseudotipizzati della proteina S, come quelli del virus della stomatite vescicolare (VSV-G) o lentivirus, sono stati utilizzati per la quantificazione degli anticorpi neutralizzanti 17,18,19. Tuttavia, lo pseudovirus a base di lentivirus richiede normalmente da 2 a 3 giorni di infezione per quantificare i segnali reporter. I sistemi pseudovirali basati su VSV contengono spesso virus VSV residui, che possono portare a tassi elevati di risultati falsi positivi e in genere richiedono 24 ore di infezione20.
Un nuovo sistema di pseudovirus SARS-CoV-2, lo pseudovirus ibrido alfavirus-SARS-CoV-2 (Ha-CoV-2), è stato recentemente sviluppato da Hetrick et al12. Ha-CoV-2 fornisce un nuovo strumento per la quantificazione rapida dell'infettività del virus e della sensibilità del virus agli anticorpi neutralizzanti nei comuni laboratori BSL-2. Strutturalmente, Ha-CoV-2 assomiglia alla particella di virione SARS-CoV-2, costituita da proteine strutturali di SARS-CoV-2 tra cui la proteina S (S), la membrana (M), il nucleocapside (N) e l'involucro (E), e non esiste una proteina strutturale di altri virus. Inoltre, la particella Ha-CoV-2 contiene un genoma di RNA a rapida espressione da un alfavirus per una rapida espressione reporter nelle cellule. È stato dimostrato che l'Ha-CoV-2 misura rapidamente l'attività neutralizzante degli anticorpi nei sieri di individui vaccinati e convalescenti12. Come dimostrato da Hetrick et al., se confrontato con lo pseudovirus SARS-CoV-2 a base di lentivirus in un test del corso temporale, Ha-CoV-2 ha espresso il reporter di Luc già 2-4 ore dopo l'infezione, mentre lo pseudovirus lentivirale ha espresso Luc dopo 24 oree 12. Inoltre, la potenziale applicazione delle varianti di Ha-CoV-2 per la quantificazione degli anticorpi neutralizzanti è ulteriormente dimostrata dall'utilizzo di un anticorpo neutralizzante monoclonale standard, 27BV (vedere la figura supplementare 1)12. Questo lavoro descrive in dettaglio l'uso della piattaforma Ha-CoV-2 per la quantificazione rapida dell'infettività delle varianti di SARS-CoV-2, utilizzando come esempi le varianti Delta (B.1.617.2) e Omicron (B.1.1.529). Inoltre, la potenziale applicazione delle varianti di Ha-CoV-2 per la quantificazione degli anticorpi neutralizzanti è ulteriormente dimostrata dall'utilizzo di un anticorpo neutralizzante monoclonale standard, 27BV12.
Protocollo
1. Assemblaggio di virus e particelle virali
- Vettori: acquista commercialmente vettori di espressione per SARS-CoV-2 M, E o N, nonché SARS-CoV-2 S (Wild-type, Wt), Delta (B.1.617.2) e Omicron (B.1.1.529).
NOTA: Le sequenze proteiche dei vettori di espressione sono fornite nel file supplementare 1. Il fornitore di questi vettori di espressione può essere trovato anche nella Tabella dei materiali. - Cellule e colture cellulari: mantenere HEK293T cellule nel terreno di Eagle modificato (DMEM) di Dulbecco contenente il 10% di siero fetale bovino inattivato dal calore (FBS), 50 unità/mL di penicillina e 50 μg/mL di streptomicina.
NOTA: Tutto il lavoro di coltura cellulare deve essere eseguito in una cabina di biosicurezza a flusso d'aria laminare. La trasfezione della polietilenimmina (PEI) richiede tipicamente un'incubazione di 5-6 ore. Gli orari di inizio devono essere considerati attentamente in anticipo. - Assemblaggio di virus e cotrasfezione basata su PEI: assemblare particelle di Ha-CoV-2 mediante cotrasfezione di cellule HEK293T. Cellule seme in una capsula di Petri per coltura cellulare da 10 cm (4-5 x 10,6 cellule per capsula) in 10 mL di terreno DMEM completo il giorno prima della cotrasfezione. Per questo studio, tre piastre di Petri vengono utilizzate per seminare cellule per l'assemblaggio di Ha-CoV-2 wild-type, Delta e Omicron, rispettivamente.
- Incubare le piastre di Petri per una notte in un incubatore di CO2 a 37 °C. Controllare i piatti la mattina successiva per assicurarsi che le cellule siano confluenti all'80%. Rimuovere il terreno completo e sostituirlo con 9 mL di terreno privo di siero DMEM.
- Per ogni piatto, preparare una miscela di cotrasfezione con 2,5 μg di ciascuno dei vettori di espressione proteica strutturale SARS-CoV-2 (N, E, M), 10 μg di pAlphaPro-Luc-GFP-PreΨ (genoma HaCoV2) e 2,5 μg del vettore di espressione della proteina S, delle varianti Delta o Omicron S e 45 μL di reagente di trasfezione a base di PEI. Lasciare che la miscela di cotrasfezione formi complessi incubando per 13 minuti (non incubare per più di 30 minuti).
- Dopo l'incubazione, aggiungere la miscela di cotrasfezione a ciascuna capsula di Petri lentamente, goccia a goccia. Porre le piastre all'interno di un'incubatrice a CO2 a 37 °C per 6 ore. Dopo 6 ore, rimuovere il DMEM privo di siero e sostituirlo con un terreno DMEM completo. Raccogliere il virus a 48-60 ore dopo la cotrasfezione.
- Raccolta e conservazione del virus: Raccogliere le particelle a 48 ore dalla cotrasfezione. Staccare le cellule pipettando ripetutamente sulla superficie del monostrato e raccogliere le cellule da ciascuna capanna, inserirle in provette da centrifuga da 15 mL e centrifugare a 400 x g per 5 minuti. Raccogliere il surnatante e farlo passare attraverso un filtro da 0,22 μM. Conservare lo pseudovirus Ha-CoV-2 a -80 °C.
2. Test di infettività virale
- Cellule e colture cellulari: Mantenere cellule HEK293T(ACE2/TMPRSS2) nel terreno di Eagle modificato (DMEM) di Dulbecco contenente il 10% di FBS inattivato al calore, 50 unità/mL di penicillina e 50 μg/mL di streptomicina.
- Semina di cellule HEK293T (ACE2/TMPRSS2): il giorno prima del test di infettività virale, seminare cellule HEK293T(ACE2/TMPRSS2) in una piastra a 96 pozzetti in 50 μL di terreno DMEM completo. Per ogni piastra da 96 pozzetti, seminare 2,5 x 104 cellule in ciascun pozzetto e sono necessarie un totale di 2,5 x 106 cellule per una piastra. Posizionare la piastra a 96 pozzetti in un incubatore a CO2 a 37 °C per una notte.
- Infezione di HEK293T (ACE2/TMPRSS2): utilizzare le particelle delle varianti di Ha-CoV-2 per infettare le cellule HEK293T (ACE2/TMPRSS2). La mattina dell'infezione, rimuovere 50 μL di DMEM dalla piastra preseminata a 96 pozzetti. Sostituire il terreno con 50 μL di Ha-CoV-2 Wild-type, Delta o Omicron per 18 ore a 37 °C.
NOTA: Il protocollo può essere interrotto qui fino a quando l'infezione non ha raggiunto le 18 ore di incubazione e la piastra è pronta per essere analizzata tramite il test della luciferasi. Il successo dell'infezione è determinato dal test della luciferasi risultante dal gene reporter della luciferasi espresso nelle cellule infette, quindi più segnale viene prodotto, più successo ha l'infezione della variante Ha-CoV-2. - Test della luciferasi: Dopo 18 ore di incubazione, aggiungere 7,5 μL di tampone di lisi cellulare direttamente in ciascun pozzetto e mescolare agitando orbitalmente per 2 minuti. Lisare le cellule nel tampone di lisi per almeno 5 minuti a temperatura ambiente.
- Preparare la soluzione del saggio della luciferasi Firefly mescolando la soluzione di D-luciferina con la soluzione del saggio della luciferasi Firefly in un rapporto 1:50. Per un'intera piastra a 96 pozzetti, combinare 3 mL della soluzione di substrato di luciferasi con 60 μL della soluzione di D-luciferina con 2940 μL della soluzione tampone luciferasi Firefly.
- Aggiungere 25 μL della soluzione del saggio luciferasi Firefly ai lisati cellulari e mescolare la piastra agitando orbitalmente per 1 minuto. Analizzare l'attività della luciferasi utilizzando un lettore di micropiastre per luciferasi commerciale.
3. Estrazione di RNA di Ha-CoV-2 e PCR quantitativa con trascrittasi inversa (RT-qPCR)
- Estrazione dell'RNA virale: estrarre l'RNA virale dalle particelle Ha-CoV-2 Wild-type e dalle varianti Delta e Omicron di Ha-CoV-2 utilizzando un kit di estrazione dell'RNA virale commerciale, seguendo le istruzioni del produttore. Conservare l'RNA virale estratto a -80 °C o utilizzarlo immediatamente per la RT-qPCR.
- RT-qPCR: Eseguire la RT-qPCR sull'RNA virale utilizzando una miscela master in un'unica fase. Eseguire la reazione in una macchina PCR commerciale. Si noti che il bersaglio dell'amplificazione è l'RNA genomico di Ha-CoV-2. Utilizzare il DNA vettoriale Ha-CoV-2 come standard per creare una curva standard e calcolare il numero di copie di RNA di ciascuna variante.
4. Saggio degli anticorpi neutralizzanti
- Semina di cellule HEK293T(ACE2/TMPRSS2): il giorno prima del test, seminare cellule HEK293T(ACE2/TMPRSS2) in una piastra a 96 pozzetti in 50 μL di terreno DMEM completo. HEK293T celle (ACE2/TMPRSS2) vengono acquistate commercialmente.
- Per contare le cellule, prelevare 20 μL di cellule da un matraccio T75 contenente HEK293T(ACE2/TMPRSS2) e mescolare con 20 μL di soluzione blu di tripano. Aggiungere 20 μL di questa miscela alla camera di conteggio delle cellule e contare il numero di cellule per mL. Per seminare una piastra a 96 pozzetti per l'infezione, utilizzare 2,5 x 104 cellule per pozzetto e saranno necessarie 2,5 x 106 cellule in totale. Porre la piastra a 96 pozzetti in un incubatore CO2 a 37 °C per una notte.
- Dosaggio degli anticorpi neutralizzanti: in una piastra sterile in polipropilene a 96 pozzetti, preparare l'anticorpo neutralizzante standard 27BV e la miscela di Ha-CoV-2. Aggiungere 8 μL di 27BV (45 mg/mL) alla piastra ed eseguire diluizioni seriali dell'anticorpo con 6 μL di DMEM esente da siero.
NOTA: Assicurarsi di scambiare i puntali delle pipette tra un trasferimento di pozzetti e l'altro e assicurarsi che il mezzo privo di anticorpi e siero siano miscelati accuratamente per produrre risultati accurati. - All'anticorpo diluito in serie, aggiungere 54 μL di particella di Ha-CoV-2 e mescolare il virus e l'anticorpo. Pre-incubare le particelle di Ha-CoV-2 con 27BV diluito in serie per 1 ora a 37 °C al 5% di CO2 . Dopo l'incubazione di 1 ora, applicare 50 μL della miscela di anticorpi e Ha-CoV-2 sulla piastra a 96 pozzetti contenente le cellule HEK293T(ACE2/TMPRSS2) (2,5 x 104 cellule per pozzetto) seminate il giorno prima.
- Per i controlli, lasciare almeno tre pozzetti contenenti solo le celle HEK293T(ACE2/TMPRSS2). Aggiungere 50 μL di terreno completo a questi pozzetti per fungere da pozzetti non infetti per il segnale di fondo delle letture del saggio della luciferasi.
NOTA: Il protocollo può essere interrotto fino a quando l'infezione non ha raggiunto le 18 ore di incubazione a 37 °C, quindi la piastra è pronta per essere analizzata tramite il test della luciferasi. - Test della luciferasi: Dopo 18 ore di incubazione, aggiungere 7,5 μL di tampone di lisi direttamente a ciascun pozzetto e mescolare agitando orbitalmente per 2 minuti. Lisare le cellule nel tampone di lisi per almeno 5 minuti a temperatura ambiente.
- Preparare la soluzione del saggio della luciferasi Firefly mescolando la soluzione di D-luciferina con la soluzione del saggio della luciferasi Firefly in un rapporto 1:50. Per un'intera piastra a 96 pozzetti, combinare 3 mL della soluzione di substrato di luciferasi con 60 μL della soluzione di D-luciferina con 2940 μL della soluzione tampone luciferasi di lucciola.
- Aggiungere 25 μL della soluzione del saggio luciferasi Firefly ai lisati cellulari e mescolare la piastra agitando orbitalmente per 1 minuto. Analizzare l'attività della luciferasi utilizzando un lettore di micropiastre per luciferasi commerciale.
5. Quantificazione e analisi statistica
- Raccolta dati: eseguire i saggi di infezione e luciferasi in triplice copia come indicato (Figura 1). Quantificare l'espressione della luciferasi con le letture del saggio della luciferasi. La media è il valore medio delle tre letture del saggio della luciferasi. Le letture del segnale di fondo provenienti da pozzi non infetti vengono sottratte da questo valore medio. La deviazione standard (SD) è determinata dal valore medio delle letture del saggio della luciferasi.
- Analisi dei dati: tracciare l'attività di neutralizzazione degli anticorpi e calcolare i valori di ID50 (dosaggio di inibizione del 50%) utilizzando software grafici commerciali. Il valore ID50 è definito come la diluizione inibitoria degli anticorpi alla quale si ottiene una riduzione del 50% dell'infezione delle cellule HEK293T(ACE2/TMPRSS2) (sulla base delle letture del saggio della luciferasi).
Risultati
Le particelle di Ha-CoV-2 sono state assemblate utilizzando cinque diversi vettori di DNA che esprimono il genoma dell'RNA Ha-CoV-2 e le proteine strutturali (M, N, E e S) di SARS-CoV-2 nelle cellule HEK293T. Il vettore della proteina S varia a seconda della variante S. La proteina S del ceppo originale Ha-CoV-2 di Wuhan (Wild-type, Wt) è stata utilizzata come controllo positivo ed è stata assemblata insieme alla proteina S di ciascuna delle altre due varianti: la Delta (B.1.617.2) o la Omicron (B.1.1.529). Le stesse M, N, E sono state utilizzate in tutte le varianti. Le particelle Ha-CoV-2 (Wt) e varianti sono state raccolte 48 ore dopo la cotrasfezione e quindi utilizzate per infettare le cellule HEK293T (ACE2/TMPRSS2). L'infettività è stata misurata mediante l'espressione della luciferasi a 18 ore dall'infezione. In questo sistema, livelli di espressione più elevati del segnale della luciferasi riflettono una maggiore infezione delle cellule da parte di Ha-CoV-2. Il segnale della luciferasi è stato normalizzato con copie di RNA genomico mediante RT-qPCR per ciascuna variante. Come mostrato nella Figura 1, la variante Ha-CoV-2 Omicron ha generato un segnale da 4 a 10 volte superiore rispetto all'originale Ha-CoV-2 (Wt), suggerendo una maggiore infettività.
Inoltre, è stata quantificata la capacità di 27BV di neutralizzare le varianti HaCoV-2(Wt), Delta e Omicron. 27BV è un anticorpo monoclonale di coniglio sviluppato contro il dominio RBD della proteina SARS-COV-2 S1. Per i saggi di neutralizzazione, sono state eseguite diluizioni seriali di 27BV in una piastra a 96 pozzetti, pre-incubata con Ha-CoV-2 e quindi aggiunta a HEK293T cellule bersaglio (ACE2/TMPRSS2). I risultati hanno dimostrato che 27BV ha avuto attività neutralizzante contro tutte le varianti testate (Figura 2). È interessante notare che l'ID50 di 27VB per Omicron era circa 10 volte meno potente dell'ID50 per Ha-CoV-2 (WT) e Ha-CoV-2 (Delta; Figura 2). Questi risultati dimostrano che la piattaforma Ha-COV-2 può essere utilizzata come metodo rapido per quantificare gli anticorpi neutralizzanti indotti dal vaccino nelle varianti emergenti.
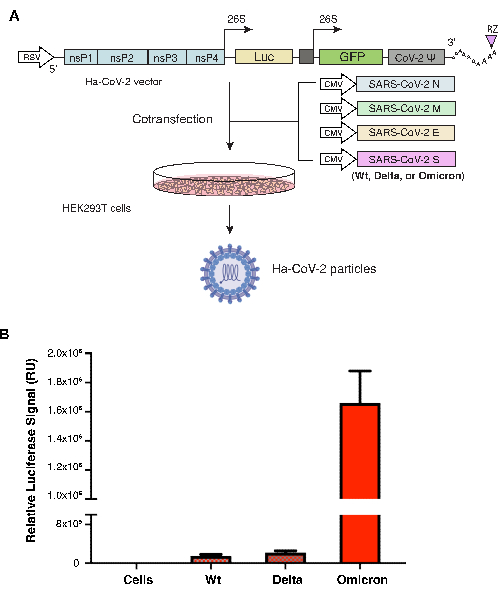
Figura 1. Assemblaggio e quantificazione di varianti di Ha-CoV-2. (A) Illustrazione dell'assemblaggio dell'Ha-CoV-2 e delle particelle varianti. I vettori che esprimono il genoma reporter di Ha-CoV-2 e le proteine strutturali (M, S, N ed E) sono cotrasfettati in cellule HEK293T. Le particelle sono state raccolte 48 ore dopo la cotrasfezione (l'imaging delle particelle di virione e delle cellule HEK293T sono state create con Biorender.com). (B) Quantificazione dell'infettività delle varianti di Ha-CoV-2. L'infettività relativa delle due varianti (Delta e Omicron) viene quantificata e normalizzata utilizzando copie di RNA genomico di singole varianti di Ha-CoV-2 (Luc). Il wild-type è l'Ha-COV-2 (Wt), che viene utilizzato come controllo per il confronto. I saggi di infezione e luciferasi sono stati eseguiti 3 volte. UR, unità relativa. Vengono mostrate la media e la deviazione standard (SD). Fare clic qui per visualizzare una versione più grande di questa figura.
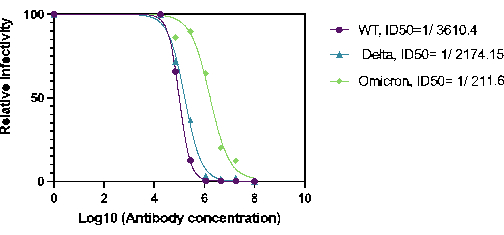
Figura 2. Quantificazione dell'attività di neutralizzazione di 27BV contro le varianti di Ha-CoV-2 (Luc) L'attività di neutralizzazione di 27BV è stata analizzata 18 ore dopo l'infezione delle cellule HEK293T (ACE2/TMPRSS2). L'ID50 è stato calcolato utilizzando il tasso di infezione relativo (attività della luciferasi) rispetto alla concentrazione di 27BV. Fare clic qui per visualizzare una versione più grande di questa figura.
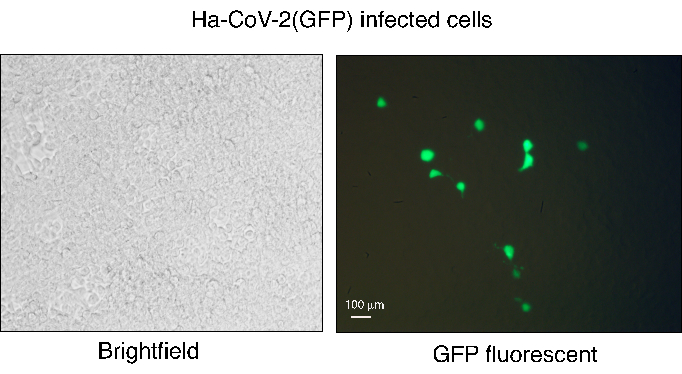
Figura 3. Infezione di HEK293T(ACE2/TMPRSS2) da Ha-CoV-2 (GFP). Le particelle di Ha-CoV-2 (GFP) sono state assemblate e poi utilizzate per infettare le cellule HEK293T (ACE2/TMPRSS2). L'espressione di GFP è stata osservata a 48 ore dopo l'infezione utilizzando microscopia a fluorescenza. Il campo bianco delle cellule infette è mostrato a sinistra e l'imaging GFP è mostrato a destra. La barra bianca rappresenta 100 μm. Fare clic qui per visualizzare una versione più grande di questa figura.
Figura supplementare 1. Abstract grafico. La struttura e l'applicazione dello pseudovirus Ha-CoV-2. Immagine creata con Biorender.com. Fare clic qui per scaricare il file.
Fascicolo supplementare 1. Sequenze proteiche. Elenco delle sequenze delle proteine SARS-CoV-2 S, M, N ed E. Le sequenze della proteina S includono anche le varianti di SARS-CoV-2 Omicron (B.1.1.529) e Delta (B.1.617.2). Fare clic qui per scaricare il file.
Discussione
La piattaforma Ha-CoV-2 fornisce un flusso di lavoro rapido, robusto e semplice per quantificare le varianti virali e neutralizzare gli anticorpi. Tuttavia, ci sono alcuni passaggi critici che richiedono attenzione. La produzione dello pseudovirus Ha-CoV-2 deve essere eseguita utilizzando cellule HEK293T ad alta vitalità. L'efficienza della cotrasfezione può essere monitorata 24 ore dopo la trasfezione utilizzando il gene reporter GFP del genoma Ha-CoV-2. Il genoma di Ha-CoV-2 può contenere due reporter (GFP e Luc) e la GFP può essere espressa durante la cotrasfezione e in seguito all'infezione da Ha-CoV-2 delle cellule bersaglio12. Le cellule GFP+ dell'infezione sono normalmente a bassa percentuale (dall'1% al 5%), ma ogni cellula infetta esprime forti segnali GFP (Figura 3). Questa bassa percentuale di GFP può limitare l'uso di GFP come lettura robusta per quantificare la neutralizzazione degli anticorpi, rispetto al reporter Luc, che quantifica l'intera popolazione di cellule infette.
Quando si esegue il test di neutralizzazione, è essenziale cambiare i puntali delle pipette tra un trasferimento di pozzetto e l'altro e assicurarsi che l'anticorpo e il terreno privo di siero siano miscelati accuratamente per produrre risultati accurati. Inoltre, quando si esegue il protocollo di analisi della luciferasi, le cellule devono essere completamente lisate per almeno 3 minuti per garantire la completa lisi delle cellule e il rilascio dell'enzima luciferasi. Ciò garantirà l'accuratezza del test. Inoltre, una volta che la soluzione del saggio luciferasi Firefly viene aggiunta alle piastre ottiche a 96 pozzetti a parete bianca, la piastra deve essere analizzata entro 10 minuti poiché l'emissione di luce iniziale è elevata ma diminuisce nel tempo man mano che l'ATP si esaurisce21.
Man mano che un numero sempre maggiore di varianti di SARS-CoV-2 continua a evolversi, c'è una crescente necessità di piattaforme come Ha-CoV-2 per lo screening rapido dell'infettività delle varianti e della sensibilità delle varianti agli anticorpi neutralizzanti indotti dal vaccino. La piattaforma Ha-CoV-2 offre una maggiore velocità, un rapporto segnale/rumore più elevato e un protocollo semplice rispettoai test di neutralizzazione basati su pseudovirus esistenti 8,9,10,11. La piattaforma Ha-CoV-2 offre anche il vantaggio di poter essere utilizzata nei laboratori BSL-2 e non richiede l'uso di strutture BSL-3. Ciò consente di perseguire la ricerca sul SARS-CoV-2 in laboratori di ricerca e clinici comuni. Inoltre, la piattaforma Ha-CoV-2 produce risultati rapidi rispetto ad altri sistemi. Ad esempio, lo studio degli anticorpi neutralizzanti contro il virus infettivo SARS-CoV-2 si avvale spesso del test di neutralizzazione della riduzione della placca (PRINT)22. Sebbene PRINT produca risultati affidabili, il conteggio manuale delle unità formanti placca (PFU) è lento e richiede 3-5 giorni per ottenere risultati23,24. Altri sistemi di pseudotipi, come il lentivirus-pseudovirus, hanno bisogno di 24-72 ore per produrre un segnale reporterrilevabile 12. In confronto, il test di neutralizzazione dell'Ha-CoV-2 può generare risultati entro 18 ore. L'Ha-CoV-2 fornisce un comodo strumento per lo screening e la quantificazione rapidi delle varianti virali e degli anticorpi neutralizzanti per il monitoraggio della pandemia.
Il monitoraggio dell'infettività del SARS-CoV-2 è essenziale poiché continuano a emergere più varianti di preoccupazione (COV). L'Ha-CoV-2 offre il vantaggio di determinare rapidamente l'infettività dei COV. Studi precedenti hanno utilizzato modelli basati sull'intelligenza artificiale (AI) per analizzare quantitativamente l'infettività della sottovariante Omicron e delle altre varianti di SARS-CoV-2, come la varianteDelta 25. Questi studi hanno dimostrato che la variante Omicron è più contagiosa del virus originale e ha maggiori probabilità di sfuggire agli anticorpi neutralizzanti25. In questi studi, utilizzando Ha-CoV-2, sono stati osservati fenotipi simili. Inoltre, nei test di neutralizzazione degli anticorpi, la variante Omicron ha dieci volte meno probabilità di essere neutralizzata da 27BV rispetto ai ceppi Wuhan e Delta. Questi risultati sono anche coerenti con la maggiore trasmissibilità della variante Omicron, che ha almeno 15 mutazioni sul suo dominio di legame del recettore (RBD), probabilmente migliorando l'affinità di legame virale al recettore ACE2 per una maggiore trasmissibilità e una maggiore fuga immunitaria26.
Divulgazioni
Un brevetto è stato depositato dalla George Mason University e concesso in licenza a Virongy Biosciences Inc. per lo sviluppo del prodotto. Y.W. è fondatore e membro del comitato consultivo di Virongy Biosciences. B. H è attualmente CEO e Chief Scientific Officer di Virongy Biosciences. Gli autori non hanno altri conflitti di interesse.
Riconoscimenti
Questo lavoro è stato sostenuto dal fondo di ricerca interno della George Mason University.
Materiali
| Name | Company | Catalog Number | Comments |
| 27VB1 20 µg SARS-CoV-2 Standard Neutralizing Antibody | Virongy Biosciences | 27VBI-01 | |
| 500 mL - US Origin FBS | Neuromics | FBS001 | |
| AB Mixing Plate: Olympus 96-Well PCR Plate, Non-Skirted UltraThin Wall, Natural, 25 Plates/Unit | Genesee Scientific | Cat# 24-300 | |
| Allegra 6R Centrifuge | Beckman Coulter | 2043-30-1158 | |
| DMEM (1x) | ThermoFisher | 11995-073 | |
| GenClone 25-209, TC Treated Flasks, 250ml, Vent Growth Area: 75.0cm2, 5 per Sleeve, 100 Flasks/Unit | Genesee Scientfic | 25-209 | |
| GlowMax Discover Microplate reader | Promega | GM3000 | |
| Ha-CoV-2 E Vector | Virongy Biosciences | pCoV2_E | |
| Ha-CoV-2 M Vector | Virongy Biosciences | pCoV2_M | |
| Ha-CoV-2 N Vector | Virongy Biosciences | pCoV2_N | |
| Ha-CoV-2 WT S Vector | Virongy Biosciences | pCoV2_WT S | |
| Hek293T cells | ATCC | CRL-3214 | |
| Illumination Firefly Luciferase Enhanced Assay Kit 1000 assays | Gold Bio | I-930-1000 | |
| Infection Plate: 96-Well Tissue Culture Plate, Greiner Bio-One (With Lid, μClear White Flat Round, Chimney) | VWR | Cat# 82050-758 | |
| pAlphaPro-Luc-GFP-PreΨ (Ha-CoV-2 Genome) Vector | In house | ||
| PEI-based Transfection Reagent | Virongy Biosciences | Transfectin | |
| Penicillin-Streptomycin-Glutamine (100X) | Invitrogen | 10378016 | |
| Polyethylenimine, branched | Millipore Sigma | 408727-100ML | |
| QuantStudio 7 Pro Real-Time PCR System | ThermoFisher | A43163 | |
| Ready to use (HEK293T)(ACE2/TMPRSS2) Cells | Virongy Biosciences | Ready-To-Use-Cells | |
| SARS-CoV-2 S Omicron (B.1.1.529) Vector | Virongy Biosciences | pCoV2-B.1.1.529 | |
| SARS-CoV-2 S Delta (B.1.617.2) Vector | Virongy Biosciences | pCoV2- B.1.617.2 | |
| Syringe Filters, PES, 0.22µm | Genesee Scientfic | 25-244 | |
| TaqMan Fast Virus 1-Step Master Mix | ThermoFisher | 4444432 | |
| Trypan Blue Solution, 0.4% | ThermoFisher | 15250061 |
Riferimenti
- World Health Organization. . Weekly epidemiological update on COVID-19. 143, 1 (2023).
- Dhama, K., et al. Global emerging Omicron variant of SARS-CoV-2: Impacts, challenges and strategies. Journal of Infection and Public Health. 16 (1), 4-14 (2023).
- Dhama, K., et al. Coronavirus Disease 2019-COVID-19. Clinical Microbiology Reviews. 33 (4), e00028-e00020 (2020).
- El-Shabasy, R. M., et al. Three waves changes, new variant strains, and vaccination effect against COVID-19 pandemic. International Journal of Biological Macromolecules. 204, 161-168 (2022).
- CDC. . Interim laboratory biosafety guidelines for handling and processing specimens associated with coronavirus disease 2019 (COVID-19). , (2021).
- Kaufer, A., Theis, T., Lau, K., Gray, J., Rawlinson, W. Laboratory biosafety measures involving SARS-CoV-2 and the classification as a Risk Group 3 biological agent. Pathology. 52 (7), 790-795 (2020).
- Vanderheiden, A., et al. Development of a Rapid Focus Reduction Neutralization Test Assay for Measuring SARS-CoV-2 Neutralizing Antibodies. Current Protocols in Immunology. 131 (1), e116 (2020).
- Dieterle, M. E., et al. A Replication-Competent Vesicular Stomatitis Virus for Studies of SARS-CoV-2 Spike-Mediated Cell Entry and Its Inhibition. Cell Host & Microbe. 28 (3), 486-496 (2020).
- Nie, J., et al. Quantification of SARS-CoV-2 neutralizing antibody by a pseudotyped virus-based assay. Nature Protocols. 15 (11), 3699-3715 (2020).
- Nie, J., et al. Establishment and validation of a pseudovirus neutralization assay for SARS-CoV-2. Emerging Microbes & Infections. 9 (1), 680-686 (2020).
- Yang, R., et al. Development and effectiveness of pseudotyped SARS-CoV-2 system as determined by neutralizing efficiency and entry inhibition test in vitro. Biosafety and Health. 2 (4), 226-231 (2020).
- Hetrick, B., et al. Development of a hybrid alphavirus-SARS-CoV-2 pseudovirion for rapid quantification of neutralization antibodies and antiviral drugs. Cell reports methods. 2 (3), 100181 (2022).
- Jackson, C., Farzan, M., Chen, B., Choe, H. Mechanisms of SARS-CoV-2 entry into cells. Nature Reviews. Molecular Cell Biology. 23 (1), 3-20 (2022).
- Jackson, L. A., et al. An mRNA Vaccine against SARS-CoV-2 - Preliminary Report. The New England Journal of Medicine. 383 (20), 1920-1931 (2020).
- Polack, F. P., et al. Safety and Efficacy of the BNT162b2 mRNA Covid-19 Vaccine. The New England Journal of Medicine. 383 (27), 2603-2615 (2020).
- Li, F. Structure, function, and evolution of coronavirus spike proteins. Annual Review of Virology. 3 (1), 237-261 (2016).
- Donofrio, G. A Simplified SARS-CoV-2 Pseudovirus Neutralization Assay. Vaccines. 9 (4), 389 (2021).
- Crawford, K. H. D., et al. Protocol and Reagents for Pseudotyping Lentiviral Particles with SARS-CoV-2 Spike Protein for Neutralization Assays. Viruses. 12 (5), 513 (2020).
- Zettl, F., et al. Rapid Quantification of SARS-CoV-2-Neutralizing Antibodies Using Propagation-Defective Vesicular Stomatitis Virus Pseudotypes. Vaccines. 8 (3), 386 (2020).
- Li, Q., Liu, Q., Huang, W., Li, X., Wang, Y. Current status on the development of pseudoviruses for enveloped viruses. Reviews in Medical Virology. 28 (1), e1963 (2018).
- Lundin, A. Optimization of the firefly luciferase reaction for analytical purposes. Advances in Biochemical Engineering/Biotechnology. 145, 31-62 (2014).
- Deshpande, G. R., et al. Neutralizing antibody responses to SARS-CoV-2 in COVID-19 patients. The Indian Journal of Medical Research. 152 (1 & 2), 82-87 (2020).
- Chen, C., et al. Research progress in methods for detecting neutralizing antibodies against SARS-CoV-2. Analytical Biochemistry. 673, 115199 (2023).
- Manischewitz, J., et al. Development of a novel vaccinia-neutralization assay based on reporter-gene expression. The Journal of Infectious Diseases. 188 (3), 440-448 (2003).
- Chen, J., Wang, R., Gilby, N., Wei, G. Omicron Variant (B.1.1.529): Infectivity, Vaccine Breakthrough, and Antibody Resistance. Journal of Chemical Information and Modeling. 62 (2), 412-422 (2022).
- Saxena, S. K., et al. Characterization of the novel SARS-CoV-2 Omicron (B.1.1.529) variant of concern and its global perspective. Journal of Medical Virology. 94 (4), 1738-1744 (2022).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon