Method Article
Desarrollo de un modelo de biofilm de colonias polimicrobianas para probar antimicrobianos en la fibrosis quística
En este artículo
Resumen
Esta metodología permite el establecimiento de un modelo de biofilm polimicrobiano en fibrosis quística para pruebas de sensibilidad antimicrobiana en laboratorios clínicos y de investigación. Este modelo proporciona resultados precisos y fiables en una amplia gama de salidas.
Resumen
Existe una variedad de modelos de biopelículas de bacterias para las pruebas de antibióticos. Sin embargo, muchos de ellos se limitan a una sola producción experimental, como las unidades formadoras de colonias o la actividad metabólica. Además, muchos modelos de biopelículas no reflejan las propiedades biológicas y fisicoquímicas del entorno humano. Este es un problema importante en muchas afecciones, pero más notablemente en la fibrosis quística (FQ). Una gran proporción de personas con FQ sufren infecciones crónicas e intermitentes, y in vitro, las pruebas de susceptibilidad a los antibióticos se correlacionan mal con los resultados del tratamiento del paciente. Algunos modelos de biopelícula incorporan medios relevantes para los pulmones de la FQ, incluidos los imitadores sintéticos de esputo, pero no tienen en cuenta la naturaleza polimicrobiana del entorno, que altera la arquitectura de la biopelícula, la fisiología y la forma en que los microbios responden al tratamiento. El modelo de biopelícula de colonias de interfaz sólido-aire descrito aquí es altamente adaptable e incorpora tanto medios relevantes para la FQ como un contexto polimicrobiano. Este modelo también se puede utilizar para el cribado de antimicrobianos de rendimiento medio y para estudiar su efecto sobre la dinámica polimicrobiana. Las mediciones de salida del modelo pueden ser unidades formadoras de colonias, actividad metabólica y análisis de microscopía confocal. El modelo se puede adaptar fácilmente a diferentes microorganismos, medios, temperaturas y condiciones variables de oxígeno y se puede utilizar para probar una amplia gama de tratamientos químicos, biológicos y físicos.
Introducción
La fibrosis quística (FQ) es una enfermedad genética que afecta a más de 11.000 personas en el Reino Unido y a 162.000 personas en todo el mundo 1,2. Aunque la FQ es una enfermedad multiorgánica, un síntoma clave que experimentan las personas con fibrosis quística (FQP) es la formación de moco anormalmente espeso y deshidratado dentro de sus víasrespiratorias. Esto, junto con la reducción del latido de los cilios, puede mejorar la colonización de los pulmones por una amplia gama de bacterias, hongos, virus y arqueas 4,5. A pesar de que la FQ pulmonar proporciona condiciones y presiones selectivas para limitar el crecimiento y la supervivencia de los microorganismos, bacterias como Pseudomonas aeruginosa están altamente adaptadas a estos entornos hostiles6. Esto permite tanto la colonización como la supervivencia, lo que resulta en la formación de infecciones crónicas persistentes7.
Muchos de los microbios que causan estas infecciones crónicas lo hacen utilizando un cambio fenotípico de un estilo de crecimiento planctónico inicial a un estilo de biopelícula, ya sea adherido a la superficie o en agregados7. Estas biopelículas se caracterizan por comunidades bacterianas muy compactas encerradas por una matriz de exopolisacáridos (EPS) que consta de una variedad de componentes, incluidos polisacáridos, proteínas, lípidos y ADN ambiental (eDNA)8. Esta matriz es una característica común entre los microorganismos; sin embargo, su composición puede diferir. Por ejemplo, los principales componentes polisacáridos de la matriz bacteriana de P. aeruginosa son Psl, Pel y alginato, a diferencia de los hongos Candida albicans, cuyos principales componentes de la matriz de polisacáridos son mananos y glucanos9. En las biopelículas monoespecie, esta matriz puede afectar en gran medida la tolerancia a los antibióticos en comparación con el planctónico, al reducir la penetración de los antibióticos en la biopelícula, disminuyendo su eficacia10. El estilo de crecimiento de la biopelícula también induce la formación de células persistentes con una actividad metabólica reducida en comparación con sus contrapartes planctónicas y, por lo tanto, una mayor susceptibilidad a los antibióticos11. Este estilo de crecimiento también se caracteriza por la regulación positiva de algunos mecanismos de resistencia a los antibióticos, como las bombas de eflujo y las necesarias para la transferencia horizontal de genes, lo que permite el intercambio de genes de resistencia 11,12,13. Además de estos, el entorno relacionado con la enfermedad del huésped influye en gran medida en la fisiología microbiana y la forma en que responden a los antibióticos. Esto incluye el aumento de la microbiosis dentro de la mucosidad espesa, así como la disponibilidad de fuentes de carbono no estándar, como los aminoácidos y el ADNe, ya sea derivado del huésped o de la degradación microbiana de los productos pulmonares 14,15,16.
Estas interacciones específicas se complican aún más por la naturaleza polimicrobiana de las biopelículas, que introduce una capa adicional de complejidad con interacciones complejas no solo entre bacterias, sino también entre bacterias y hongos. En comparación con las biopelículas bacterianas, se sabe menos sobre algunas de las interacciones entre las biopelículas bacterianas y fúngicas, a pesar de que C. albicans se aísla en más del 75% de las personas con FQ15. En general, las interacciones entre C. albicans y bacterias como P. aeruginosa son antagónicas, pero pueden dar lugar a infecciones más crónicas y graves18. La combinación de interacciones microbianas, tanto patógenas como comensales, junto con una serie de factores ambientales relacionados con la FQ puede, en última instancia, dar lugar a una mayor tolerancia a los antibióticos19,20. Muchos de estos factores no se consideran a menudo en las pruebas preclínicas de antibióticos, a pesar de que se atribuyen a una mayor tolerancia a los antibióticos en los modelos existentes21.
Estas condiciones también son difíciles de recapitular in vitro y, como resultado, muchos modelos carecen de la presencia de factores específicos inductores de tolerancia presentes en pwCF, como el aumento de la producción de betalactamasas en P. aeruginosa22, la inducción de pequeñas variantes de colonias en Staphylococcus aureus y la inhibición de la separación silábica en C. albicans; todos los cuales se ha demostrado que ocurren en el esputo de FQ23,24.
Existe, por lo tanto, una gran disparidad entre las condiciones utilizadas en los métodos actuales de pruebas de sensibilidad a los antibióticos, basados en cultivos planctónicos o en placas de agar en medios estandarizados como los ensayos de difusión en disco, Etest y microdilución en caldo CLSI, y las condiciones encontradas en el entorno del huésped25. Con frecuencia, esto no permite determinar con precisión la sensibilidad a los antibióticos26. Este problema se complica aún más por la falta de estandarización en las pruebas de biopelículas antimicrobianas, lo que dificulta la traducción precisa de la eficacia antimicrobiana del laboratorio a la clínica27,28.
El modelo polimicrobiano que hemos desarrollado aquí demuestra una mayor tolerancia de Pseudomonas aeruginosa a una serie de antimicrobianos, como el meropenem y la tobramicina. Esto ilustra la gran variación entre las pruebas antimicrobianas actuales que utilizan biopelículas de una sola especie y el modelo de biopelícula polimicrobiana desarrollado en este estudio para determinar las concentraciones inhibitorias mínimas. Este modelo también mantiene un rendimiento relativamente alto y un bajo costo deseable para las pruebas de antimicrobianos. El modelo también se puede utilizar para estudiar el impacto de la terapia antimicrobiana en la dinámica polimicrobiana y establecer si un tratamiento en particular puede conducir a que un patógeno en particular se convierta en dominante, lo que permite la predicción de complicaciones adicionales. Aunque este modelo permite la construcción de biopelículas complejas, su configuración no requiere equipos de laboratorio sofisticados y proporciona una plataforma para una amplia gama de resultados clínicos y de investigación.
Protocolo
1. Preparación de fibrosis quística sintética media-2 (SCFM2) y placas
- Prepare SCFM2 de acuerdo con la receta descrita en Palmer et al.16 en una botella de vidrio de borosilicato de 500 mL a una concentración de 2x con algunas modificaciones.
- Pese y prepare las existencias como se indica en la Tabla 1. Prepare las reservas de aminoácidos de acuerdo con la Tabla 2.
- Pesar 5 g de mucina (de estómago porcino (Tipo II)) y 600 mg de eDNA (de esperma de salmón) en 100 mL dedH2O en una botella de vidrio de borosilicato de 200 mL. Agitar a 4 °C durante la noche antes de la esterilización en autoclave.
- A un biberón esterilizado de 500 ml, agregue 0,54 g de glucosa, 6,06 g de NaCl y 2,228 g de KCl. Agregue 10 ml de cada una de las cepas preparadas en el paso 1.1.1. Ajuste el pH a 6.9 usando 1 M de NaOH o HCl.
- Añadir 10 mL dedH2O seguidos de 10 mL de 0,175 M de CaCl2 y 0,0606 M de MgCl2. Preparar y añadir 10 mL de ácido L-láctico 0,93 M. Inmediatamente antes de usar, añadir 10 mL de 0,36 mM de Fe(III)SO47H2O
- Filtro Esterilice el SCFM2 utilizando una unidad de filtro y una bomba de vacío, luego agregue la mucina esterilizada en autoclave y el eDNA.
NOTA: El SCFM2 preparado puede almacenarse a 4 °C durante un máximo de 1 mes.
- Prepare las placas de agar SCFM2 como se describe a continuación.
- Precaliente 25 mL de 2x SCFM2 en un tubo de 50 mL en un baño de agua a 55 °C antes de mezclar con 25 mL de agar técnico pre-fundido al 3% (p/v), lo que da como resultado una mezcla de 1x SCFM2 y 1,5% (p/v) de agar técnico.
- Con una pipeta serológica, transfiera 5 ml de la mezcla a cada pocillo de una placa de 6 pocillos.
- Prepare el mismo número de discos de policarbonato de 13 mm con un tamaño de poro de 0,2 μm colocándolos en el fondo de una placa de Petri y aplicando UV-C durante 600 s a una longitud de onda de 254 nm y una frecuencia de 60 Hz. Dé la vuelta a los discos y repita el procedimiento UV-C.
- Una vez que el agar técnico 1x SCFM2-1.5 % (p/v) se haya endurecido, use pinzas estériles para agregar un solo disco de policarbonato a la superficie de cada pocillo de la placa de 6 pocillos.
2. Preparación de bacterias para la infección
- Antes de configurar el modelo, prepare la/s placa/s de agar para las bacterias y cepas fúngicas requeridas de acuerdo con el agar estándar utilizado en el laboratorio para P. aeruginosa PAO1, S. aureus SH1000 y C. albicans SC5314 (por ejemplo, caldo de Lysogeny con agar 1,2% (p/v) para bacterias y agar dextrosa Sabouraud para C. albicans).
NOTA: Para establecer el modelo se han utilizado cepas de referencia específicas de laboratorio. Sin embargo, este modelo también se ha utilizado con otras cepas, incluidos los aislados clínicos, con algunas modificaciones, como el aumento del tiempo para el establecimiento de la biopelícula antes del tratamiento para las cepas de crecimiento más lento. - Utilice un circuito de inoculación de 1 μL para seleccionar una sola colonia de cada microorganismo y inocular 5 mL de medio líquido estándar de laboratorio (por ejemplo, caldo de lisogénia para P. aeruginosa, S. aureus y peptona dextrosa de levadura (YPD) para C. albicans). Incubar estos cultivos hasta que alcancen la fase de crecimiento exponencial a 200 rpm, 37 °C para las bacterias, y 200 rpm, 30 °C para C. albicans.
NOTA: La aireación variará según la incubadora agitadora y el radio de rotación
3. Configuración del modelo de interfaz de aire sólido
NOTA: En la Figura complementaria 1 se puede ver una representación esquemática general del modelo.
- Transfiera 1 mL de cada cultivo líquido a un tubo estéril separado de 1,5 mL y centrifugue durante 2 min a 8.000 x g para granular las bacterias/hongos. Realice este y todos los siguientes pasos de centrifugación a temperatura ambiente.
- Aspirar el sobrenadante con una pipeta y volver a suspender los pellets en 1 mL de PBS antes de la centrifugación, una vez más, durante 2 min a 8.000 x g.
- Retire el sobrenadante como antes y vuelva a suspender los gránulos en 1 mL de PBS. Diluir las células resuspendidas de P. aeruginosa y S. aureus 1:10 en PBS y medir la densidad óptica utilizando un espectrofotómetro a una longitud de onda de 600 nm.
- Ajustar las muestras lavadas a 1 x 108 unidades formadoras de colonias (UFC)/mL sobre la base de la validación previa de que una DO600 de 0,05 para P. aeruginosa y 0,1 para S. aureus es equivalente a 1 x 108 UFC/mL.
- En el caso de C. albicans, diluir las muestras lavadas 1:100 en PBS, añadir 10 μL de cada una a dos cámaras individuales de un hemocitómetro y utilizar las dimensiones fijas de la cámara y la dilución utilizada para calcular las UFC/mL. Utilizando el hemocitómetro Neubauer mejorado, el cálculo será el siguiente:
Número de células = recuento medio de células × 10.000 / (factor de dilución) - Las UFC calculadas representan UFC/ml de C. albicans en la muestra prediluida. Ajuste la muestra de C. albicans a 1 x 108 UFC/mL en PBS como con los cultivos bacterianos.
- Para preparar el inóculo para su adición a la biopelícula, ajuste el inóculo de P. aeruginosa a 1 x 104 UFC/mL (diluciones de 2 x 100 veces), C. albicans a 1 x 105 UFC/mL (dilución de 100 veces seguida de una dilución de 10 veces) y finalmente, S. aureus a 1 x 106 UFC/mL (dilución de 100 veces) usando PBS.
- En el caso de biopelículas monoespecie, añadir 10 μL del microbio deseado diluido en el centro del disco de policarbonato previamente colocado en las placas de seis pocillos de agar técnico SCFM2-1,5 % 1x e incubar estáticamente a 37 °C durante 24 h antes del tratamiento o la interrupción.
- Para biopelículas polimicrobianas, agregue 10 μL de cada S. aureus y C. albicans uno encima del otro en el mismo disco. Incubar el disco inoculado estáticamente durante 24 h a 37 °C.
NOTA: El orden en que se agregan los inóculos de S. aureus y C. albicans no afecta el desarrollo del modelo y genera los mismos resultados. - A continuación, transfiera los discos de policarbonato a placas de agar técnico 1x SCFM2-1,5% frescas con pinzas estériles y añada 10 μL de la dilución de 1 x 104 UFC/mL de P. aeruginosa sobre el biofilm preestablecido de S. aureus - C. albicans . Incubar durante otras 24 h a 37 °C.
NOTA: P. aeruginosa se añade 24 h después de S. aureus y C. albicans para reflejar el orden en que estos organismos colonizan frecuentemente el pulmón de la FQ en las personas con FQ. Esto también evita que P. aeruginosa supere a los otros dos microorganismos. Una vez que se ha agregado P. aeruginosa , el biofilm puede crecer durante más de 24 h; Sin embargo, el disco de policarbonato con la biopelícula debe transferirse a medios nuevos cada 24 horas. Antes de la interrupción, las biopelículas se pueden utilizar para la microscopía utilizando cepas que expresan proteínas fluorescentes, colorantes fluorescentes o anticuerpos específicos de la cepa/microbio. Sin embargo, vale la pena señalar que algunos tintes manchan el disco de policarbonato.
4. Disrupción de la biopelícula
- Añada perlas de cerámica con un diámetro de 2,8 mm a las placas y reticule las placas como se describe en el paso 1.3. Agregue 5 perlas de cerámica a un tubo homogeneizador estéril de 2 mL con pinzas esterilizadas con llama. A continuación, pipetee 1 mL de solución salina tamponada con fosfato (PBS) en el tubo de homogeneización de 2 mL.
- Tubos de vórtice durante al menos 10 s o hasta que se haya eliminado la biopelícula del disco de policarbonato, determinado por inspección visual, momento en el que se retira el disco de policarbonato.
- Coloque los tubos con las cuentas en el homogeneizador y bata 2 veces durante 10 s a 6 m/s con un intervalo de 10 segundos entre latidos.
NOTA: Esta combinación de perlas, tiempo de batido y uso de homogeneizadores da como resultado una interrupción suficiente de la biopelícula con una lisis mínima según lo determinado por las unidades de microscopía y formación de colonias. Esto puede variar dependiendo del batidor de cuentas específico utilizado. - Vierta la biopelícula interrumpida del tubo homogeneizador en una joya de 7 mL que contenga 4 mL de PBS, lo que da como resultado un volumen final de 5 mL.
- Asegúrese de que todo el líquido de los tubos del homogeneizador se haya transferido a la bisutería centrifugando los tubos a 8.000 x g durante 2 minutos y transfiriendo el líquido restante a la bisutería de 7 ml.
NOTA: A partir de estos 5 mL de biopelícula interrumpida, se pueden realizar una serie de ensayos, pero es clave recordar para cualquier cálculo la dilución de PBS de 1 mL de biopelícula en un volumen final de 5 mL.
5. Pruebas de actividad metabólica
- Agregue 200 μL de la biopelícula interrumpida del paso 4 anterior a una placa negra de 96 pocillos de fondo transparente. A continuación, añadir 10 μL de solución de resazurina al 0,02% (p/v).
- Incubar la biopelícula tratada con resazurina con un lector de placas y leer la actividad metabólica. Incubar la placa a 37 °C, aplicando una agitación orbital cada 30 minutos durante 10 s a 200 rpm antes de tomar cualquier lectura fluorescente.
- Mida la fluorescencia utilizando una excitación de 540 nm y una emisión de 590 nm. Repita este ciclo de 30 minutos durante 4 h con un lector de placas.
NOTA: Se utiliza el punto de tiempo de 4 h, ya que proporciona variaciones más claras entre las muestras tratadas y las no tratadas y entre las muestras con diferentes concentraciones de antibióticos.
6. Pruebas de sensibilidad a los antimicrobianos
- Para probar la tolerancia antimicrobiana de los antibióticos estándar, solubilice los antibióticos de elección en su solvente correspondiente y agréguelo al medio de agar técnico SCFM2-1.5% (p/v) 1x mientras aún está líquido antes de transferir 5 mL a cada pocillo de una placa de seis pocillos. Una vez colocado, transfiera la biopelícula cultivada en el disco de policarbonato a la parte superior del medio que contiene antibióticos con pinzas estériles y agregue una gota de 10 μL de la concentración de antibiótico correspondiente en la parte superior de la biopelícula.
NOTA: Para este estudio, se utilizó una dilución seriada de 2 pliegues logarítmicos entre 64 μg/mL y 0,125 μg/mL de meropenem y tobramicina para reflejar las concentraciones clínicamente relevantes. Es suficiente con administrar antibióticos u otros tratamientos antimicrobianos/antivirulentos únicamente sobre la biopelícula, ya que se han observado respuestas similares cuando se añaden tanto al medio como a la biopelícula. - Después de 24 h de incubación a 37 °C con el tratamiento, obtenga imágenes de las biopelículas y la interrupción como se describe en el paso 4.
- De los 5 mL de cultivo interrumpido, se toma una alícuota y se realiza una dilución en serie de 10 veces utilizando PBS. Colocar puntos de 10 μL por triplicado en agar para determinar las unidades formadoras de colonias utilizando el método de Miles y Misra30.
NOTA: El volumen agregado a las placas de agar afectará el límite de detección, los volúmenes más grandes reducirán el límite de detección pero requerirán más muestras y placas de agar. - En el caso de las biopelículas monoespecies, estrellen P. aeruginosa y S. aureus en placas de agar LB y C. albicans en agar SDA.
- En el caso de las biopelículas polimicrobianas, se colocan alícuotas en tres medios selectivos diferentes que contengan antibióticos para aislar cada una de las tres especies y permitir la determinación de las UFC. Esto incluye agar de aislamiento Pseudomonas y agar de sal de manitol que contiene nistatina a 10 μg/mL para aislar P. aeruginosa y S. aureus, respectivamente, y agar dextrosa Sabouraud con tetraciclina a 125 μg/mL para aislar C. albicans. Incubar las placas de S. aureus a 37 °C durante la noche y las placas de P. aeruginosa y C. albicans a 30 °C.
NOTA: Incubar las placas de P. aeruginosa a 30 °C para minimizar el crecimiento excesivo de la colonia y reducir el enjambre.
Resultados
La simplicidad del modelo de biopelícula de interfaz sólido-aire permite el cribado de un gran número de antimicrobianos en diferentes condiciones clínicamente relevantes a la vez. Este modelo permite evaluar la efectividad de los antibióticos mediante recuentos de UFC y ensayos metabólicos en biofilms monomicrobianos y polimicrobianos en una semana. Debido a la naturaleza del modelo, también permite una fácil manipulación de las condiciones ambientales, como cambiar la composición del medio y colocar las biopelículas bajo condiciones anaeróbicas y de oxígeno reducido. Utilizando este modelo, hemos mostrado cambios en la tolerancia a dos antibióticos comúnmente utilizados en pwCF entre P. aeruginosa cultivada en monoespecie y biofilms polimicrobianos (Figura 1). Junto a esto, hemos podido determinar cómo el tratamiento antimicrobiano puede afectar la dinámica poblacional dentro de las biopelículas polimicrobianas (Figura 2).
Para llevar a cabo estos experimentos, se preparó el inóculo inicial según lo especificado en el protocolo anterior, lo que condujo a la formación de monoespecies estables y biopelículas polimicrobianas con UFC/mL reproducibles y pequeñas desviaciones estándar determinadas por ANOVA de uno o dos factores. Este modelo también puso de manifiesto el crecimiento consistente y la recuperación de UFC, además de permitir la posibilidad de combinar estas técnicas clásicas de microbiología con otras técnicas no realizadas en este estudio sin necesidad de un procesamiento sofisticado. Estos incluyen imágenes como vivos/muertos, visualización de matrices y análisis molecular, que otros modelos tridimensionales no pueden ofrecer de una manera tan accesible (Figura 1)31. Cabe señalar que la desviación estándar esperada para UFC/ml aumenta cuando los datos se muestran como el porcentaje de supervivencia en comparación con las UFC/ml (Figura 1). Al comparar la efectividad del tratamiento con antibióticos entre monoespecies y biopelículas polimicrobianas, hubo un aumento significativo en todas las concentraciones de antibióticos requeridas para lograr el 50% de eliminación. Se necesitó un aumento de 2 logaritmos en la concentración de antibióticos para lograr este nivel de eliminación de meropenem y tobramicina (Figura 1). También hubo un aumento general en la supervivencia de P. aeruginosa en presencia de S. aureus y C. albicans en la biopelícula polimicrobiana cuando se trató con 64 μg/mL de tobramicina, mientras que lo contrario fue cierto para el meropenem.
El método utilizado para determinar la actividad metabólica mide la actividad global de todo el biofilm polimicrobiano sin poder distinguir la contribución individual de cada especie. Por esta razón, solo se muestran cambios metabólicos de una sola especie para demostrar el uso de ensayos de actividad metabólica para este modelo. En el caso de las biopelículas monoespecies, se observó una fuerte relación entre la supervivencia de P. aeruginosa y la actividad metabólica tanto para el meropenem como para la tobramicina (Figura 2). La utilización de la actividad metabólica y los recuentos de UFC de las mismas muestras permite la fácil identificación de los efectos bacteriostáticos y bactericidas. En el caso de las biopelículas polimicrobianas, la determinación de la actividad metabólica puede indicar cómo la selección de una especie puede inducir un aumento general de la actividad metabólica de las biopelículas y, por lo tanto, aumentar potencialmente el crecimiento microbiano de otras especies.
Aunque las UFC son el estándar de oro para las pruebas de sensibilidad a los antimicrobianos, en las biopelículas polimicrobianas también permiten evaluar la composición de especies dentro de la biopelícula. Al tratar P . aeruginosa en biopelículas polimicrobianas, no solo podemos mostrar el aumento de MBEC50 para este organismo, sino también establecer el efecto que esto tiene en las otras especies co-aisladas (Figura 3). Esto se observa, por ejemplo, con el meropenem (antipseudomonal), donde la reducción de P. aeruginosa va acompañada de un aumento de las UFC de S. aureus y C. albicans , lo que hace que S. aureus se convierta en el organismo más prevalente tras la exposición a ciertas concentraciones de antibióticos (Figura 3A). Esto pone de manifiesto la importancia de tener en cuenta la naturaleza polimicrobiana de la biopelícula debido al posible aumento del tamaño de la población de otras especies causantes de enfermedades al tratar un patógeno en particular.
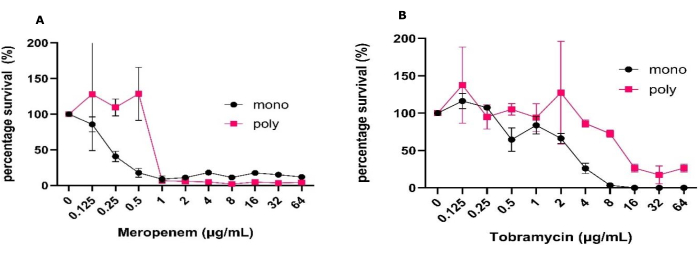
Figura 1: Variación en la tolerancia antimicrobiana de P. aeruginosa a meropenem y tobramicina entre biopelículas mono y polimicrobianas cultivadas en el modelo de interfaz sólido-aire. P. aeruginosa PAO1 cultivado en biopelículas mono o polimicrobianas con S. aureus y C. albicans se estableció utilizando los modelos de interfaz sólido-aire durante 24 h en SCFM2. Las biopelículas se trataron con un rango de concentraciones de (A) meropenem o (B) tobramicina de 0,125 μg/mL a 64 μg/mL junto con un control sin antibióticos. Se determinaron las UFC/mL de cada biopelícula. P. aeruginosa Las UFC se determinaron en el PIA y se convirtieron en porcentaje de supervivencia utilizando el control negativo como supervivencia del 100%. Las barras de error denotan una desviación estándar, y cada punto de datos se deriva de 3 repeticiones biológicas, cada una con tres repeticiones técnicas. Haga clic aquí para ver una versión más grande de esta figura.

Figura 2: Variación en la actividad metabólica de las biopelículas de P. aeruginosa en presencia de meropenem o tobramicina cultivadas en el modelo de interfaz sólido-aire. P. aeruginosa Las biopelículas de PAO1 se cultivaron en SCFM2 en el modelo de interfaz sólido-aire, y se añadieron un rango de concentraciones de (A) meropenem o (B) tobramicina de 0,125 μg/mL a 64 μg/mL junto con un control sin antibióticos. La placa se leyó fluorescentemente a una excitación de 540 nm y una emisión de 590 nm cada 30 min. El porcentaje de actividad metabólica se calculó determinando el porcentaje de esta actividad en cada muestra tratada con antibióticos en comparación con el control sin antibióticos. Las barras de error denotan una desviación estándar, y cada punto de datos se deriva de 3 repeticiones biológicas, cada una con tres repeticiones técnicas. Haga clic aquí para ver una versión más grande de esta figura.
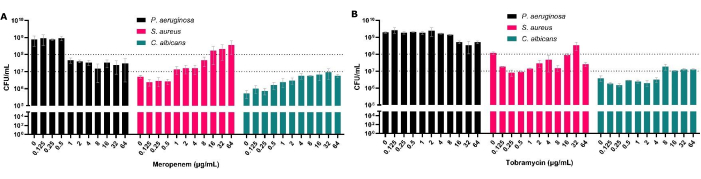
Figura 3: Impacto en los recuentos totales de UFC de P. aeruginosa, S. aureus y C. albicans recuperados del modelo polimicrobiano de interfaz sólido-aire después del tratamiento antimicrobiano. S. aureus y C. albicans se cultivaron en SCFM2 en el modelo de interfaz sólido-aire. Se añadió P. aeruginosa y se añadió un rango de concentraciones de 0,125 μg/mL a 64 μg/mL de (A) meropenem o (B) tobramicina a las biopelículas junto con un control sin antibióticos. Se determinaron las UFC/mL de cada biopelícula. Las barras de error denotan desviación estándar, y cada punto de datos se derivó de 3 repeticiones biológicas, cada una con tres repeticiones técnicas. Haga clic aquí para ver una versión más grande de esta figura.
Figura complementaria 1: Establecimiento y flujo de trabajo del modelo de interfaz sólido-aire. Representación gráfica del establecimiento del modelo de interfaz sólido-aire, la interrupción de la biopelícula y las salidas. Haga clic aquí para descargar este archivo.
Discusión
El modelo de biopelícula descrito aquí nos permite imitar algunos aspectos del entorno de infección pulmonar por FQ mediante la incorporación de una variedad de interacciones polimicrobianas comunes de una manera más accesible que los métodos de prueba de antimicrobianos in vitro que se utilizan actualmente, como el reactor CDC y el modelo de biopelícula de Lubbock31. Esto es importante cuando una serie de factores, incluidas las propiedades físicas de la biopelícula, la disponibilidad de nutrientes y las interacciones moleculares entre las especies dentro de la biopelícula, pueden afectar tanto la actividad del agente antimicrobiano como la respuesta de los microbios al antimicrobiano21. Al incorporar estos factores, hemos podido desarrollar un modelo altamente versátil que puede permitir la predicción más precisa de los resultados del tratamiento antimicrobiano que los modelos actuales que no consideran los factores antes mencionados.
Se eligieron P. aeruginosa, S. aureus y C. albicans como Gram-positivos, Gram-negativos y hongos representativos comúnmente aislados en CF32. Sin embargo, también hemos podido demostrar que este modelo se puede utilizar para una serie de otros microbios relevantes para la FQ, como Burkholderia cenocepacia, Burkholderia multivorans, Aspergillus fumigatus, y actualmente se está adaptando para su uso con bacterias anaeróbicas como Prevotella melaninogenica. Esto demuestra la versatilidad del modelo, ya que se podría incorporar una amplia gama de microbios, lo que pone de manifiesto la adaptabilidad de este modelo para las pruebas de antimicrobianos contra una amplia gama de patógenos microbianos. El modelo también se puede adaptar fácilmente a una variedad de condiciones ambientales para pruebas de sensibilidad antimicrobiana más personalizadas, incluida la sustitución de SCFM2 por esputo del paciente.
La consideración de modelos de biopelículas polimicrobianas para el ensayo de nuevos antimicrobianos también es primordial, ya que pueden cubrir un vacío existente en la cartera de desarrollo de antimicrobianos antes de su uso en estudios con animales o humanos33,34. Junto a esto, el modelo mantiene un rendimiento relativamente alto y permite una gama de resultados relevantes tanto para la industria como para la investigación.
Aunque solo hemos ejemplificado las UFC y la actividad metabólica en este artículo de métodos, también hemos demostrado la utilidad del modelo en el análisis del exometaboloma de la biopelícula utilizando espectrometría de masas de análisis de superficie de extracción líquida (LESA). Esto permite sondear algunos de los mecanismos moleculares que subyacen a la tolerancia antimicrobiana35. Además de estos otros resultados, también hemos demostrado el uso de este modelo para probar nuevos sistemas de administración de antimicrobianos. Entre ellos se encuentran los conjugados polímero-ciprofloxacino para potenciar la penetración de este antibiótico en el interior del biofilm a la vez que aumenta su actividad y reduce el desarrollo de resistencias36.
Pasos críticos y consideraciones
Es importante mantener la esterilidad, especialmente cuando se producen SCFM2 y medios, ya que la contaminación puede dar lugar a la introducción de especies no naturales, lo que puede interferir con los resultados. Diferentes cepas del mismo microbio y diferentes microbios pueden requerir diferentes condiciones ambientales para su crecimiento o pueden tener diferentes características de crecimiento. Por lo tanto, el modelo debe optimizarse antes de realizar cualquier prueba de sensibilidad a los antibióticos. Recomendamos llevar a cabo un pequeño estudio piloto de 24 h y 48 h para monoespecies y biofilm polimicrobiano para evaluar el crecimiento en condiciones de modelo estándar. Hemos encontrado que diferentes cepas de P. aeruginosa y ciertas concentraciones de antibióticos pueden inducir el enjambre del disco de policarbonato. Esto se puede superar fácilmente mediante el uso de discos de mayor tamaño que están disponibles comercialmente. La disrupción de la biopelícula también puede requerir optimización dependiendo del batidor de cuentas y los microbios utilizados. Optimizamos el método de interrupción mediante la determinación de las UFC/mL de una muestra conocida antes y después del batido de las perlas. También utilizamos la microscopía para visualizar las muestras de batido de cuentas para visualizar qué tan bien se rompió la biopelícula.
Una de las limitaciones de este modelo de biopelícula que imita la CF es la unión a una superficie sólida que contiene policarbonato, que no se encuentra en el pulmón de la FQ, en lugar de ser agregados que flotan libremente dentro del esputo de la CF37 . Sin embargo, el modelo de agar sólido presentado aquí excluye la necesidad de lavar las biopelículas para eliminar los microbios planctónicos y garantiza que los resultados solo incluyan microbios derivados de la biopelícula en lugar de un cultivo de biopelícula planctónica mixta. También hemos encontrado que se puede reducir la penetración de algunos antibióticos en las áreas más profundas de este modelo de biopelícula, y puede ser necesario el uso de DNasa para alterar la reología de la matriz extracelular y disminuir las interacciones electrostáticas con el eDNA.
Creemos que el modelo es muy adecuado para las pruebas antimicrobianas debido a su versatilidad, adaptabilidad e inclusión de factores ambientales que muchos modelos existentes no tienen en cuenta. Su uso en la investigación fundamental y en las pruebas preclínicas de sensibilidad a los antimicrobianos puede proporcionar un enfoque más relevante desde el punto de vista clínico para la AST de muestras clínicas y el desarrollo de nuevas terapias.
Divulgaciones
No se declara ningún conflicto de intereses o contraposición de intereses financieros para este trabajo.
Agradecimientos
Este trabajo ha sido financiado por el Centro Nacional de Innovación en Biofilms (NBIC), que es un Centro de Innovación y Conocimiento financiado por el Consejo de Investigación en Biotecnología y Ciencias Biológicas, Innovate UK y Hartree Centre [Premios BB/R012415/1 y BB/X002950/1] y por el UK CF Trust y el Centro de Investigación Estratégica de la Fundación CF de EE. UU.: 'Un marco preclínico basado en la evidencia para el desarrollo de terapias antimicrobianas en la fibrosis quística' (PIPE-CF) [Premio SRC022].
Materiales
| Name | Company | Catalog Number | Comments |
| 1 µL inoculation loops | |||
| 13 mm 0.2 µm pore size polycarbonate discs | Isopore | GTTP01300 | Larger discs are also available |
| 2 mL reinforced tubes | Thermofisher | 15545809 | |
| 2.5 mL ceramic beads | Qiagen | 13114-325 | |
| 500 mL borosilicate glass Duran bottle | Sigma Aldrich | Z305197 | larger bottles available in larger volumes are desired |
| 6-well culture plates | Greiner | 657165 | |
| 7 mL Bijou | Thermofisher | 129B | |
| 96-well plates | Thermofisher | 167008 | for serial dilutions in CFU assay |
| Agar plates for preparing plates of P. aerugnisa, S. aureus, and C. albicans | LB miller for P. aeruginosa and S. aureus and Sabouraud dextrose agar for C. albicans | ||
| Bead beater - suitable for 2 mL tubes | Fisherbrand | 15515799 | Thermofisher bead mill 24 |
| bench top centrifuge | must be capable of at least 8000 x g | ||
| Black clear bottom 96 well plates | Costar | 3603 | |
| Bunsen Burner | |||
| Containers for disposing of contaminated equipment and material according to the institutes health and safety regulations. | |||
| deionised water | |||
| eDNA | Sigma Aldrich | 31149 | |
| Filter unit | Fisherbrand | FB12566504 | Interchangeable depending on the vacuum pump used but must have a pore size of 0.2 µm |
| Haemocytometer and cover slip | Hawksley | HC001 | Haemocytometers may differ in size and volume. Double check and adjust CFU calculations accordingly |
| LB broth | oxoid | 1.46813 | |
| Mannitol salt agar | Oxoid | CM0085B | |
| meropenem | abcr | Ab243429 | |
| Mucin from porcine stomach Type II | Sigma Aldrich | M2378 | |
| Nystatin | Millipore | 1003352658 | |
| petri dishes | SLS | SLS2000 | |
| Phosphate buffered saline | |||
| Pseudomonas isolation agar | Millipore | 17208 | |
| Resazurin sodium salt | Sigma Aldrich | 199303 | |
| Sabouraud dextrose agar | Oxoid | CM0041 | |
| selection of forceps | fine tipped and tissue forceps with teeth for transferring ceramic beads | ||
| serological pipette | |||
| shaking and static incubators | must be temperature controlled | ||
| Sparks microtitre plate reader | Tecan | For Resazurin assay the microtitre plate reader must have the appropriate filters or be a monochromator for detecting flourescence. | |
| spectrophotometer | |||
| Technical agar (Agar Technical No.2 ) | Oxoid | LP0012B | |
| tetracycline | Sigma Aldrich | T7660 | |
| UV crosslinker | Spectroline | 11-992-89 | |
| vacuum pump/ flask | Fisherbrand | FB12566504 | |
| water bath | must be capable of maintaining 55 °C | ||
| YPD broth | Millipore | Y1375 | Can be bought pre-made or made using the base ingredients |
Referencias
- . 2021 Cf foundation patient registry highlights report Available from: https://cysticfibrosis.msu.edu/index.php/welcome/news-and-events/39-2021-cf-foundation-patient-registry-highlights-report (2022)
- Guo, J., Garratt, A., Hill, A. Worldwide rates of diagnosis and effective treatment for cystic fibrosis. J Cyst Fibros. 21 (3), 456-462 (2022).
- Mandal, V., Ghosh, N. N., Mitra, P. K., Mandal, S., Mandal, V. Production and characterization of a broad-spectrum antimicrobial 5-butyl-2-pyridine carboxylic acid from Aspergillus fumigatus nhf-01. Sci Rep. 12 (1), 6006 (2022).
- Françoise, A., Héry-Arnaud, G. The microbiome in cystic fibrosis pulmonary disease. Genes. 11 (5), 536 (2020).
- Grasemann, H., Ratjen, F. Cystic fibrosis. New Engl J Med. 389 (18), 1693-1707 (2023).
- Camus, L., Vandenesch, F., Moreau, K. From genotype to phenotype: Adaptations of pseudomonas aeruginosa to the cystic fibrosis environment. Microb Genom. 7 (3), mgen000513 (2021).
- Rossi, E., et al. Pseudomonas aeruginosa adaptation and evolution in patients with cystic fibrosis. Nat Rev Microbiol. 19 (5), 331-342 (2021).
- Flemming, H. -. C., Wingender, J. The biofilm matrix. Nat Rev Microbiol. 8 (9), 623-633 (2010).
- Grainha, T., Jorge, P., Alves, D., Lopes, S. P., Pereira, M. O. Unraveling pseudomonas aeruginosa and candida albicans communication in coinfection scenarios: Insights through network analysis. Front Cell Infect Microbiol. 10, 550505 (2020).
- Flemming, H. C., et al. Biofilms: An emergent form of bacterial life. Nat Rev Microbiol. 14 (9), 563-575 (2016).
- Høiby, N., Bjarnsholt, T., Givskov, M., Molin, S., Ciofu, O. Antibiotic resistance of bacterial biofilms. Int J Antimicrob Agents. 35 (4), 322-332 (2010).
- Lorusso, A. B., Carrara, J. A., Barroso, C. D. N., Tuon, F. F., Faoro, H. Role of efflux pumps on antimicrobial resistance in Pseudomonas aeruginosa. Int J Mol Sci. 23 (24), 15779 (2022).
- Michaelis, C., Grohmann, E. Horizontal gene transfer of antibiotic resistance genes in biofilms. Antibiotics .(Basel). 12 (2), 328 (2023).
- Worlitzsch, D., et al. Effects of reduced mucus oxygen concentration in airway pseudomonas infections of cystic fibrosis patients. J Clin Invest. 109 (3), 317-325 (2002).
- König, J., Schreiber, R., Voelcker, T., Mall, M., Kunzelmann, K. The cystic fibrosis transmembrane conductance regulator (CFTR) inhibits ENaC through an increase in the intracellular Cl- concentration. EMBO Reps. 2 (11), 1047-1051 (2001).
- Palmer, K. L., Mashburn, L. M., Singh, P. K., Whiteley, M. Cystic fibrosis sputum supports growth and cues key aspects of Pseudomonas aeruginosa physiology. J Bacteriol. 187 (15), 5267-5277 (2005).
- Valenza, G., et al. Prevalence and antimicrobial susceptibility of microorganisms isolated from sputa of patients with cystic fibrosis. J Cyst Fibros. 7 (2), 123-127 (2008).
- Alam, F., Catlow, D., Di Maio, A., Blair, J. M. A., Hall, R. A. Candida albicans enhances meropenem tolerance of Pseudomonas aeruginosa in a dual-species biofilm. J Antimicrob Chemother. 75 (4), 925-935 (2020).
- Burmølle, M., Ren, D., Bjarnsholt, T., Sørensen, S. J. Interactions in multispecies biofilms: Do they actually matter. Trends Microbiol. 22 (2), 84-91 (2014).
- Lories, B., Belpaire, T. E. R., Smeets, B., Steenackers, H. P. Competition quenching strategies reduce antibiotic tolerance in polymicrobial biofilms. NPJ Biofilms Microbiomes. 10 (1), 23 (2024).
- Nabb, D. L., et al. Polymicrobial interactions induce multidrug tolerance in Staphylococcus aureus through energy depletion. Front Microbiol. 10, 2803 (2019).
- Cornforth, D. M., et al. Pseudomonas aeruginosa transcriptome during human infection. Proc Natl Acad Sci U S A. 115 (22), E5125-E5134 (2018).
- Wolter, D. J., et al. Staphylococcus aureus small-colony variants are independently associated with worse lung disease in children with cystic fibrosis. Clin Infect Dis. 57 (3), 384-391 (2013).
- Takagi, J., et al. Mucin o-glycans are natural inhibitors of Candida albicans pathogenicity. Nat Chem Biol. 18 (7), 762-773 (2022).
- Cinical and Laboratory Standard Institute. . Methods for dilution susceptibility tests for bacteria that grow aerobically. 35 (2), (2015).
- Roberts, A. E., Kragh, K. N., Bjarnsholt, T., Diggle, S. P. The limitations of in vitro experimentation in understanding biofilms and chronic infection. J Mol Biol. 427 (23), 3646-3661 (2015).
- Macia, M. D., Rojo-Molinero, E., Oliver, A. Antimicrobial susceptibility testing in biofilm-growing bacteria. Clin Microbiol Infect. 20 (10), 981-990 (2014).
- Coenye, T., Goeres, D., Van Bambeke, F., Bjarnsholt, T. Should standardized susceptibility testing for microbial biofilms be introduced in clinical practice. Clin Microbiol Infect. 24 (6), 570-572 (2018).
- Turner, K. H., Wessel, A. K., Palmer, G. C., Murray, J. L., Whiteley, M. Essential genome of Pseudomonas aeruginosa in cystic fibrosis sputum. Proc Natl Acad Sci U S A. 112 (13), 4110-4115 (2015).
- Miles, A. A., Misra, S. S., Irwin, J. O. The estimation of the bactericidal power of the blood. J Hyg. 38 (6), 732-749 (1938).
- Robertson, S. N., Romero, M., Fenn, S., Kohler Riedi, P. L., Cámara, M. Development, characterization, and evaluation of a simple polymicrobial colony biofilm model for testing of antimicrobial wound dressings. J Appl Microbiol. 135 (3), lxae042 (2024).
- Rumpf, C., Lange, J., Schwartbeck, B., Kahl, B. C. Staphylococcus aureus and cystic fibrosis-a close relationship. What can we learn from sequencing studies. Pathogens. 10 (9), 1177 (2021).
- Tay, W. H., Chong, K. K. L., Kline, K. A. Polymicrobial-host interactions during infection. J Mol Biol. 428 (17), 3355-3371 (2016).
- Orazi, G., O'toole, G. A. "It takes a village": Mechanisms underlying antimicrobial recalcitrance of polymicrobial biofilms. J Bacteriol. 202 (1), e00530-e00519 (2019).
- Arjes, H. A., et al. Three-dimensional biofilm colony growth supports a mutualism involving matrix and nutrient sharing. eLife. 10, e64145 (2021).
- Kasza, K., et al. Ciprofloxacin poly(β-amino ester) conjugates enhance antibiofilm activity and slow the development of resistance. ACS Appl Mater Interf. 16 (5), 5412-5425 (2024).
- Martin, I., Waters, V., Grasemann, H. Approaches to targeting bacterial biofilms in cystic fibrosis airways. Int J Mol Sci. 22 (4), 2155 (2021).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoExplorar más artículos
This article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados