Method Article
Sviluppo di un modello di biofilm di colonia polimicrobica per testare gli antimicrobici nella fibrosi cistica
In questo articolo
Riepilogo
Questa metodologia consente la creazione di un modello di biofilm polimicrobico nella fibrosi cistica per i test di sensibilità antimicrobica nei laboratori di ricerca e clinici. Questo modello fornisce risultati accurati e affidabili su una gamma di uscite.
Abstract
Esiste una serie di modelli di biofilm batterico per la sperimentazione di antibiotici. Tuttavia, molti di questi sono limitati a un singolo output sperimentale, come le unità formanti colonie o l'attività metabolica. Inoltre, molti modelli di biofilm non riflettono le proprietà biologiche e fisico-chimiche dell'ambiente umano ospite. Questo è un problema importante in molte condizioni, ma in modo più evidente nella fibrosi cistica (FC). Un'ampia percentuale di persone con FC soffre di infezioni croniche e intermittenti e, in vitro, i test di sensibilità agli antibiotici sono scarsamente correlati con gli esiti del trattamento da parte del paziente. Alcuni modelli di biofilm incorporano terreni rilevanti per il polmone FC, inclusi imitatori sintetici dell'espettorato, ma non considerano la natura polimicrobica dell'ambiente, che altera l'architettura del biofilm, la fisiologia e il modo in cui i microbi rispondono al trattamento. Il modello di biofilm di colonia di interfaccia solido-aria qui descritto è altamente adattabile e incorpora sia terreni rilevanti per la FC che un contesto polimicrobico. Questo modello può essere utilizzato anche per lo screening a medio rendimento di antimicrobici e per studiare il loro effetto sulla dinamica polimicrobica. Le misure di output del modello possono essere unità formanti colonie, attività metabolica e analisi al microscopio confocale. Il modello può essere facilmente adattato a diversi microrganismi, fluidi, temperature e condizioni variabili di ossigeno e può essere utilizzato per testare un'ampia gamma di trattamenti chimici, biologici e fisici.
Introduzione
La fibrosi cistica (FC) è una condizione genetica che colpisce oltre 11.000 persone nel Regno Unito e 162.000 persone in tutto il mondo 1,2. Sebbene la FC sia una malattia multiorgano, un sintomo chiave sperimentato dalle persone con fibrosi cistica (pwCF) è la formazione di muco anormalmente denso e disidratato all'interno del tratto respiratorio3. Questo, insieme alla riduzione del battito delle ciglia, può migliorare la colonizzazione dei polmoni da parte di un'ampia gamma di batteri, funghi, virus e archei 4,5. Nonostante il polmone FC fornisca condizioni e pressioni selettive per limitare la crescita e la sopravvivenza dei microrganismi, batteri come Pseudomonas aeruginosa sono altamente adattati a questi ambienti difficili6. Ciò consente sia la colonizzazione che la sopravvivenza, con conseguente formazione di infezioni croniche persistenti7.
Molti dei microbi che causano queste infezioni croniche lo fanno utilizzando un passaggio fenotipico da uno stile di crescita planctonico iniziale a uno stile di crescita a biofilm, attaccato alla superficie o in aggregati7. Questi biofilm sono caratterizzati da comunità batteriche strettamente impacchettate racchiuse da una matrice esopolisaccaridica (EPS) costituita da una varietà di componenti, tra cui polisaccaridi, proteine, lipidi e DNA ambientale (eDNA)8. Questa matrice è una caratteristica comune tra i microrganismi; Tuttavia, la sua composizione può differire. Ad esempio, i principali componenti polisaccaridici della matrice batterica di P. aeruginosa sono Psl, Pel e alginato, a differenza dei funghi Candida albicans, i cui principali componenti della matrice polisaccaridica sono mannani e glucani9. Nei biofilm monospecie, questa matrice può influenzare notevolmente la tolleranza agli antibiotici rispetto al planctonico, riducendo la penetrazione degli antibiotici nel biofilm, diminuendone l'efficacia10. Lo stile di crescita del biofilm induce anche la formazione di cellule persistenti con ridotta attività metabolica rispetto alle loro controparti planctoniche e, quindi, una suscettibilità ulteriormente diminuita agli antibiotici11. Questo stile di crescita è anche caratterizzato dalla sovraregolazione di alcuni meccanismi di resistenza agli antibiotici, come le pompe di efflusso e quelle necessarie per il trasferimento genico orizzontale, che consentono lo scambio di geni di resistenza 11,12,13. Oltre a questi, l'ambiente dell'ospite correlato alla malattia influenza molto la fisiologia microbica e il modo in cui rispondono agli antibiotici. Ciò include l'aumento della micro-aerobiosi all'interno del muco denso e la disponibilità di fonti di carbonio non standard come amminoacidi ed eDNA, derivati dall'ospite o dalla degradazione microbica dei prodotti polmonari 14,15,16.
Queste interazioni specifiche sono ulteriormente complicate dalla natura polimicrobica dei biofilm, che introduce un ulteriore livello di complessità con interazioni complesse non solo tra batteri ma anche tra batteri e funghi. Rispetto ai biofilm batterici, c'è meno conoscenza per quanto riguarda alcune delle interazioni tra biofilm batterici e fungini, nonostante C. albicans sia isolato da oltre il 75% delle persone con CF15. In generale, le interazioni tra C. albicans e batteri come P. aeruginosa sono antagoniste, ma possono provocare infezioni più croniche e gravi18. La combinazione di interazioni microbiche, sia patogene che commensali, insieme a una serie di fattori ambientali correlati alla FC può in ultima analisi portare a un aumento della tolleranza agli antibiotici19,20. Molti di questi fattori non sono spesso considerati nei test antibiotici preclinici, nonostante siano attribuiti a una maggiore tolleranza agli antibiotici nei modelli esistenti21.
Queste condizioni sono anche difficili da ricapitolare in vitro e, di conseguenza, molti modelli mancano della presenza di specifici fattori che inducono tolleranza presenti in pwCF, come quelli che aumentano la produzione di beta-lattamasi in P. aeruginosa22, l'induzione di varianti di piccole colonie in Staphylococcus aureus e l'inibizione della sillabazione in C. albicans; è stato dimostrato che si verificano tutti nell'espettorato FC 23,24.
Esiste, quindi, una grande disparità tra le condizioni utilizzate negli attuali metodi di test di sensibilità agli antibiotici, basati su colture planctoniche o su piastra di agar in terreni standardizzati come Mueller-Hinton con diffusione su disco, Etest e saggi di microdiluizione del brodo CLSI, e le condizioni riscontrate nell'ambiente ospite25. Questo spesso non riesce a determinare con precisione la sensibilità agli antibiotici26. Questo problema è ulteriormente complicato dalla mancanza di standardizzazione nei test del biofilm antimicrobico, che rende difficile tradurre con precisione l'efficacia antimicrobica dal laboratorio alla clinica27,28.
Il modello polimicrobico che abbiamo sviluppato qui dimostra una maggiore tolleranza di Pseudomonas aeruginosa a una gamma di antimicrobici, tra cui meropenem e tobramicina. Ciò illustra l'ampia variazione tra gli attuali test antimicrobici che utilizzano biofilm monospecie e il modello di biofilm polimicrobico sviluppato in questo studio per determinare le concentrazioni minime di inibitorie. Questo modello mantiene anche una produttività relativamente elevata e un basso costo desiderabile per i test antimicrobici. Il modello può anche essere utilizzato per studiare l'impatto della terapia antimicrobica sulla dinamica polimicrobica e stabilire se un particolare trattamento può portare a un particolare patogeno a diventare dominante, consentendo la previsione di ulteriori complicanze. Sebbene questo modello consenta la formazione di biofilm complessi, la sua configurazione non richiede sofisticate apparecchiature di laboratorio e fornisce una piattaforma per un'ampia gamma di risultati clinici e di ricerca.
Protocollo
1. Preparazione di piastre e piastre per fibrosi cistica sintetica media-2 (SCFM2)
- Preparare SCFM2 secondo la ricetta descritta in Palmer et al.16 in un flacone di vetro borosilicato da 500 ml a concentrazione 2x con alcune modifiche.
- Pesare e preparare le scorte come indicato nella Tabella 1. Preparare le scorte di aminoacidi secondo la Tabella 2.
- Pesare 5 g di mucina (da stomaco suino (tipo II)) e 600 mg di eDNA (da sperma di salmone) in 100 mL di dH2O in un flacone di vetro borosilicato da 200 mL. Mescolare a 4 °C per una notte prima della sterilizzazione in autoclave.
- A un flacone sterilizzato da 500 ml, aggiungere 0,54 g di glucosio, 6,06 g di NaCl e 2,228 g di KCl. Aggiungere 10 ml di ciascuno dei brodi preparati al punto 1.1.1. Regolare il pH a 6,9 utilizzando 1 M NaOH o HCl.
- Aggiungere 10 mL di dH2O seguiti da 10 mL di 0,175 M CaCl2 e 0,0606 M di MgCl2. Preparare e aggiungere 10 ml di acido L-lattico 0,93 M. Immediatamente prima dell'uso, aggiungere 10 mL di 0,36 mM di Fe(III)SO47H2O
- Sterilizzare l'SCFM2 utilizzando un'unità filtro e una pompa a vuoto, quindi aggiungere la mucina autoclavata e l'eDNA.
NOTA: L'SCFM2 preparato può essere conservato a 4 °C per un massimo di 1 mese.
- Preparare le piastre per agar SCFM2 come descritto di seguito.
- Preriscaldare 25 mL di 2x SCFM2 in una provetta da 50 mL in un bagno d'acqua a 55 °C prima di miscelare con 25 mL di agar tecnico pre-fuso al 3% (p/v), ottenendo una miscela di 1x SCFM2 e 1,5% (p/v) di agar tecnico.
- Utilizzando una pipetta sierologica, trasferire 5 mL della miscela in ciascun pozzetto di una piastra a 6 pozzetti.
- Preparare lo stesso numero di dischi in policarbonato da 13 mm con una dimensione dei pori di 0,2 μm posizionandoli sul fondo di una piastra di Petri e applicando UV-C per 600 s a una lunghezza d'onda di 254 nm e una frequenza di 60 Hz. Capovolgere i dischi e ripetere la procedura UV-C.
- Una volta che l'agar tecnico 1x SCFM2-1,5 % (p/v) si è solidificato, utilizzare una pinza sterile per aggiungere un singolo disco di policarbonato alla superficie di ciascun pozzetto della piastra a 6 pozzetti.
2. Preparazione dei batteri per l'infezione
- Prima di impostare il modello, preparare la/e piastra/e di agar per i batteri e i ceppi fungini necessari secondo l'agar standard utilizzato in laboratorio per P. aeruginosa PAO1, S. aureus SH1000 e C. albicans SC5314 (ad esempio, brodo di lisogenesi con agar all'1,2% (p/v) per i batteri e agar destrosio Sabouraud per C. albicans).
NOTA: Per stabilire il modello, sono stati utilizzati specifici ceppi di riferimento di laboratorio. Tuttavia, questo modello è stato utilizzato anche con altri ceppi, compresi gli isolati clinici, con alcune modifiche, come l'aumento del tempo per la formazione del biofilm prima del trattamento per i ceppi a crescita più lenta. - Utilizzare un ciclo di inoculazione da 1 μL per prelevare una singola colonia di ciascun microrganismo per inoculare 5 ml di terreno liquido di laboratorio standard (ad esempio, brodo di lisogenia per P. aeruginosa, S. aureus e destrosio peptone di lievito (YPD) per C. albicans). Incubare queste colture fino a raggiungere la fase di crescita esponenziale a 200 giri/min, 37 °C per i batteri, e 200 giri/min, 30 °C per C. albicans.
NOTA: L'aerazione varia a seconda dell'incubatrice vibrante e del raggio di rotazione
3. Impostazione del modello di interfaccia ad aria solida
NOTA: Una rappresentazione schematica generale del modello può essere vista nella Figura 1 supplementare.
- Trasferire 1 mL di ciascuna coltura liquida in una provetta sterile separata da 1,5 mL e centrifugare per 2 minuti a 8.000 x g per pellettare i batteri/funghi. Eseguire questa e tutte le seguenti fasi di centrifugazione a temperatura ambiente.
- Aspirare il surnatante utilizzando una pipetta e risospendere i pellet in 1 mL di PBS prima della centrifugazione, ancora una volta, per 2 minuti a 8.000 x g.
- Rimuovere il surnatante come prima e risospendere i pellet in 1 mL di PBS. Diluire le cellule risospese di P. aeruginosa e S. aureus 1:10 in PBS e misurare la densità ottica utilizzando uno spettrofotometro a una lunghezza d'onda di 600 nm.
- Regolare i campioni lavati a 1 x 108 unità formanti colonie (CFU)/mL sulla base di una precedente convalida che un OD600 di 0,05 per P. aeruginosa e 0,1 per S. aureus equivale a 1 x 108 CFU/mL.
- Per C. albicans, diluire i campioni lavati 1:100 in PBS, aggiungere 10 μl di ciascuno a due camere individuali di un emocitometro e utilizzare le dimensioni fisse della camera e la diluizione utilizzata per calcolare CFU/mL. Utilizzando l'emocitometro Neubauer migliorato, il calcolo sarà il seguente:
Numero di cellule = numero medio di cellule × 10.000 / (fattore di diluizione) - Le CFU calcolate rappresentano C . albicans CFU/mL nel campione prediluito. Regolare il campione di C. albicans a 1 x 108 CFU/mL in PBS come con le colture batteriche.
- Per preparare l'inoculo per l'aggiunta al biofilm, regolare l'inoculo di P. aeruginosa a 1 x 104 CFU/mL (2 diluizioni da 100 volte), C. albicans a 1 x 105 CFU/mL (diluizione di 100 volte seguita da una diluizione di 10 volte) e infine, S. aureus a 1 x 106 CFU/mL (diluizione di 100 volte) utilizzando PBS.
- Per i biofilm monospecie, aggiungere 10 μl del microbo desiderato diluito al centro del disco in policarbonato precedentemente posizionato sulle piastre a sei pozzetti di agar tecnico 1x SCFM2-1,5 % e incubare staticamente a 37 °C per 24 ore prima del trattamento o dell'interruzione.
- Per i biofilm polimicrobici, aggiungere 10 μl di S. aureus e C. albicans uno sopra l'altro sullo stesso disco. Incubare staticamente il disco inoculato per 24 ore a 37 °C.
NOTA: L'ordine in cui vengono aggiunti gli inoculi di S. aureus e C. albicans non influisce sullo sviluppo del modello e genera gli stessi risultati. - Successivamente, trasferire i dischi in policarbonato su piastre di agar tecniche 1x SCFM2-1,5% fresche con pinza sterile e aggiungere 10 μL della diluizione 1 x 104 CFU/mL di P. aeruginosa sopra il biofilm prestabilito S. aureus - C. albicans . Incubare per altre 24 ore a 37 °C.
NOTA: P. aeruginosa viene aggiunto 24 ore dopo S. aureus e C. albicans per riflettere l'ordine in cui questi organismi colonizzano frequentemente il polmone FC nelle persone con FC. Ciò impedisce anche a P. aeruginosa di crescere oltre gli altri due microrganismi. Una volta aggiunto P . aeruginosa , il biofilm può essere coltivato per più di 24 ore; Tuttavia, il disco in policarbonato con il biofilm deve essere trasferito su un terreno fresco ogni 24 ore. Prima dell'interruzione, i biofilm possono essere utilizzati per la microscopia utilizzando ceppi che esprimono proteine fluorescenti, coloranti fluorescenti o anticorpi specifici per ceppo/microbi. Tuttavia, vale la pena notare che alcuni coloranti macchiano il disco di policarbonato.
4. Distruzione del biofilm
- Aggiungere perle di ceramica con un diametro di 2,8 mm alle piastre e reticolare le piastre con i raggi UV come descritto al punto 1.3. Aggiungere 5 perle di ceramica a una provetta sterile da 2 mL utilizzando una pinza sterilizzata a fiamma. Quindi, pipettare 1 mL di soluzione salina tamponata con fosfato (PBS) nella provetta di omogeneizzazione da 2 mL.
- Tubi vortex per almeno 10 s o fino a quando il biofilm non è stato rimosso dal disco di policarbonato, determinato mediante ispezione visiva, a quel punto rimuovere il disco di policarbonato.
- Posizionare i tubi con le perline nell'omogeneizzatore e sbattere per 2 volte per 10 s a 6 m/s con un intervallo di 10 s tra i battiti.
NOTA: Questa combinazione di perline, tempo di battitura e uso dell'omogeneizzatore si traduce in una sufficiente rottura del biofilm con una lisi minima, come determinato dalla microscopia e dalle unità formanti colonie. Questo può variare a seconda dello specifico battitore di perline utilizzato. - Versare il biofilm disgregato dalla provetta dell'omogeneizzatore in un bijou da 7 mL contenente 4 mL di PBS, ottenendo un volume finale di 5 mL.
- Assicurarsi che tutto il liquido dalle provette dell'omogeneizzatore sia stato trasferito al bijou centrifugando le provette a 8.000 x g per 2 minuti e trasferendo il liquido rimanente nel bijou da 7 mL.
NOTA: Da questi 5 mL di biofilm interrotto, è possibile condurre una serie di saggi, ma è fondamentale ricordare per qualsiasi calcolo la diluizione di 1 mL di biofilm PBS in un volume finale di 5 mL.
5. Test dell'attività metabolica
- Aggiungere 200 μl del biofilm disgregato dalla fase 4 di cui sopra a una piastra nera a 96 pozzetti a fondo chiaro. Quindi, aggiungere 10 μl di soluzione di resazurina allo 0,02% (p/v).
- Incubare il biofilm trattato con resazurina con un lettore di piastre e leggere l'attività metabolica. Incubare la piastra a 37 °C, agitando orbitalmente ogni 30 minuti per 10 s a 200 giri/min prima di effettuare qualsiasi lettura fluorescente.
- Misurare la fluorescenza utilizzando un'eccitazione di 540 nm e un'emissione di 590 nm. Ripetere questo ciclo di 30 minuti per 4 ore utilizzando un lettore di piastre.
NOTA: Viene utilizzato il punto temporale di 4 ore in quanto fornisce variazioni più distinte tra campioni trattati e non trattati e tra campioni con concentrazioni di antibiotici variabili.
6. Test di sensibilità antimicrobica
- Per testare la tolleranza antimicrobica degli antibiotici standard, solubilizzare gli antibiotici di scelta nel loro solvente pertinente e aggiungerlo al terreno di agar tecnico 1x SCFM2-1,5% (p/v) mentre è ancora liquido prima di trasferire 5 mL in ciascun pozzetto di una piastra a sei pozzetti. Una volta indurito, trasferire il biofilm cresciuto sul disco di policarbonato sulla parte superiore del terreno contenente antibiotici utilizzando una pinza sterile e aggiungere una goccia di 10 μl della corrispondente concentrazione di antibiotico sulla parte superiore del biofilm.
NOTA: Per questo studio, è stata utilizzata una diluizione seriale di 2 log fold tra 64 μg/mL e 0,125 μg/mL di meropenem e tobramicina per riflettere le concentrazioni clinicamente rilevanti. È sufficiente somministrare antibiotici o altri trattamenti antimicrobici/anti-virulenza esclusivamente sopra il biofilm, poiché sono state osservate risposte simili quando vengono aggiunti sia al terreno che al biofilm. - Dopo 24 ore di incubazione a 37 °C con il trattamento, visualizzare i biofilm e interrompere come descritto nella fase 4.
- Dai 5 mL di coltura interrotta, prendere un'aliquota ed eseguire una diluizione seriale di 10 volte utilizzando PBS. Piastra di 10 μl di macchie in triplicato su agar per determinare le unità formanti colonie utilizzando il metodo Miles e Misra30.
NOTA: Il volume aggiunto alle piastre di agar influirà sul limite di rilevamento, volumi maggiori ridurranno il limite di rilevamento ma richiederanno più campioni e piastre di agar. - Per i biofilm monospecie, striare P. aeruginosa e S. aureus su piastre di agar LB e C. albicans su agar SDA.
- Per i biofilm polimicrobici, posizionare le aliquote su tre diversi terreni selettivi contenenti antibiotici per isolare ciascuna delle tre specie e consentire la determinazione delle CFU. Ciò include l'agar di isolamento Pseudomonas e l'agar sale di mannitolo contenenti nistatina a 10 μg/mL per isolare rispettivamente P. aeruginosa e S. aureus, e l'agar destrosio Sabouraud con tetraciclina a 125 μg/mL per isolare C. albicans. Incubare le piastre di S. aureus a 37 °C per una notte e le piastre di P. aeruginosa e C. albicans a 30 °C.
NOTA: Incubare le piastre di P. aeruginosa a 30 °C per ridurre al minimo la crescita eccessiva delle colonie e ridurre la sciamatura.
Risultati
La semplicità del modello di biofilm con interfaccia solido-aria consente lo screening di un gran numero di antimicrobici in diverse condizioni clinicamente rilevanti contemporaneamente. Questo modello consente di valutare l'efficacia degli antibiotici utilizzando la conta delle CFU e i saggi metabolici sia in biofilm mono che polimicrobici da effettuare in una settimana. A causa della natura del modello, consente anche una facile manipolazione delle condizioni ambientali, come la modifica della composizione del supporto e il posizionamento dei biofilm in condizioni di ossigeno ridotto e anaerobiche. Utilizzando questo modello, abbiamo mostrato cambiamenti nella tolleranza a due antibiotici comunemente usati nella pwCF tra P. aeruginosa coltivata in biofilm monospecie e polimicrobici (Figura 1). Oltre a ciò, siamo stati in grado di determinare in che modo il trattamento antimicrobico può influire sulle dinamiche della popolazione all'interno dei biofilm polimicrobici (Figura 2).
Per eseguire questi esperimenti, l'inoculo iniziale è stato preparato come specificato nel protocollo di cui sopra, portando alla formazione di biofilm stabili monospecie e polimicrobici con CFU/mL riproducibili e piccole deviazioni standard determinate da ANOVA a una o due vie. Questo modello ha anche evidenziato una crescita costante e un recupero di UFC, oltre a consentire la possibilità di combinare queste tecniche di microbiologia classica con altre tecniche non eseguite in questo studio senza la necessità di elaborazioni sofisticate. Questi includono l'imaging come vivo/morto, la visualizzazione matriciale e l'analisi molecolare, che altri modelli tridimensionali non sono in grado di offrire in modo così accessibile (Figura 1)31. Va notato che la deviazione standard attesa per CFU/mL aumenta quando i dati vengono visualizzati come percentuale di sopravvivenza rispetto a CFU/mL (Figura 1). Confrontando l'efficacia del trattamento antibiotico tra monospecie e biofilm polimicrobici, c'è stato un aumento significativo di tutte le concentrazioni di antibiotici necessarie per raggiungere il 50% di uccisione. È stato necessario un aumento di 2 log della concentrazione di antibiotici per raggiungere questo livello di uccisione per meropenem e tobramicina (Figura 1). C'è stato anche un aumento complessivo della sopravvivenza di P. aeruginosa in presenza di S. aureus e C. albicans nel biofilm polimicrobico quando trattato con 64 μg/mL di tobramicina, mentre il contrario era vero per il meropenem.
Il metodo utilizzato per determinare l'attività metabolica misura l'attività complessiva dell'intero biofilm polimicrobico senza essere in grado di distinguere il contributo individuale di ciascuna specie. Per questo motivo, solo i cambiamenti metabolici delle monospecie hanno dimostrato l'uso di saggi di attività metabolica per questo modello. Per i biofilm monospecie c'era una forte relazione tra la sopravvivenza di P. aeruginosa e l'attività metabolica sia per meropenem che per tobramicina (Figura 2). L'utilizzo dell'attività metabolica e della conta delle CFU dagli stessi campioni consente di identificare facilmente gli effetti batteriostatici e battericidi. Per i biofilm polimicrobici, la determinazione dell'attività metabolica può indicare come il targeting di una specie possa indurre un aumento complessivo dell'attività metabolica del biofilm e, quindi, potenzialmente aumentare la crescita microbica di altre specie.
Sebbene le CFU siano il gold standard per i test di sensibilità antimicrobica, nei biofilm polimicrobici consentono anche la valutazione della composizione delle specie all'interno del biofilm. Trattando P. aeruginosa in biofilm polimicrobici, non solo siamo in grado di mostrare l'aumento di MBEC50 per questo organismo, ma anche di stabilire l'effetto che questo ha sulle altre specie co-isolate (Figura 3). Questo è, ad esempio, visto con il meropenem (antipseudomonale), dove la riduzione di P. aeruginosa è accompagnata da un aumento di S. aureus e C. albicans CFU, con il risultato che S. aureus diventa l'organismo più diffuso dopo l'esposizione a determinate concentrazioni di antibiotici (Figura 3A). Ciò evidenzia l'importanza di considerare la natura polimicrobica del biofilm a causa del potenziale aumento delle dimensioni della popolazione di altre specie patogene nel trattamento di un particolare patogeno.
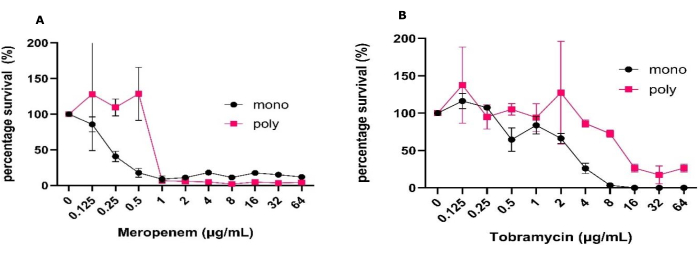
Figura 1: Variazione della tolleranza antimicrobica di P. aeruginosa a meropenem e tobramicina tra biofilm mono e polimicrobici cresciuti nel modello di interfaccia solido-aria. P. aeruginosa PAO1 coltivato in biofilm mono o polimicrobici con S. aureus e C. albicans è stato stabilito utilizzando i modelli di interfaccia solido-aria per 24 ore su SCFM2. I biofilm sono stati trattati con un intervallo di concentrazioni di (A) meropenem o (B) tobramicina da 0,125 μg/mL a 64 μg/mL insieme a un controllo senza antibiotici. È stato determinato il CFU/mL di ciascun biofilm. P. aeruginosa Le CFU sono state determinate in PIA e convertite in percentuale di sopravvivenza utilizzando il controllo negativo come sopravvivenza del 100%. Le barre di errore denotano la deviazione standard e ogni punto dati è derivato da 3 ripetizioni biologiche, ciascuna con tre ripetizioni tecniche. Clicca qui per visualizzare una versione più grande di questa figura.

Figura 2: Variazione dell'attività metabolica dei biofilm di P. aeruginosa in presenza di meropenem o tobramicina cresciuti nel modello di interfaccia solido-aria. P. aeruginosa I biofilm di PAO1 sono stati coltivati su SCFM2 nel modello di interfaccia solido-aria e sono state aggiunte un intervallo di concentrazioni di (A) meropenem o (B) tobramicina da 0,125 μg/mL a 64 μg/mL insieme a un controllo senza antibiotici. La lastra è stata letta in fluorescenza ad un'eccitazione di 540 nm e un'emissione di 590 nm ogni 30 minuti. La percentuale di attività metabolica è stata calcolata determinando la percentuale di questa attività su ciascun campione trattato con antibiotici rispetto al controllo senza antibiotici. Le barre di errore denotano la deviazione standard e ogni punto dati è derivato da 3 ripetizioni biologiche, ciascuna con tre ripetizioni tecniche. Clicca qui per visualizzare una versione più grande di questa figura.
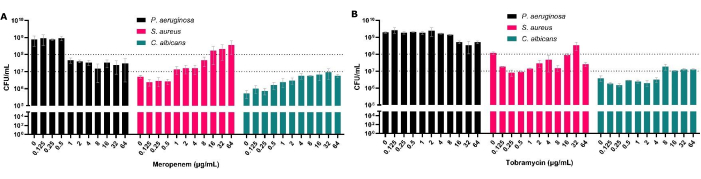
Figura 3: Impatto sulla conta totale delle CFU di P. aeruginosa, S. aureus e C. albicans recuperate dal modello di interfaccia polimicrobica solido-aria dopo trattamento antimicrobico. S. aureus e C. albicans sono stati coltivati su SCFM2 sul modello di interfaccia solid-air. È stato aggiunto P. aeruginosa e un intervallo compreso tra 0,125 μg/mL e 64 μg/mL di concentrazioni di (A) meropenem o (B) tobramicina sono state aggiunte ai biofilm insieme a un controllo senza antibiotici. È stato determinato il CFU/mL di ciascun biofilm. Le barre di errore denotano la deviazione standard e ogni punto dati è stato derivato da 3 ripetizioni biologiche, ciascuna con tre ripetizioni tecniche. Clicca qui per visualizzare una versione più grande di questa figura.
Figura supplementare 1: Definizione e flusso di lavoro del modello di interfaccia solido-aria. Rappresentazione grafica della creazione del modello di interfaccia solido-aria, della disgregazione del biofilm e dei risultati. Clicca qui per scaricare questo file.
Discussione
Il modello di biofilm qui descritto ci consente di imitare alcuni aspetti dell'ambiente di infezione polmonare da FC incorporando una serie di interazioni polimicrobiche comuni in un modo più accessibile rispetto ai metodi di test antimicrobici in vitro attualmente utilizzati come il reattore CDC e il modello di biofilm di Lubbock31. Ciò è importante quando una serie di fattori, tra cui le proprietà fisiche del biofilm, la disponibilità di nutrienti e le interazioni molecolari tra le specie all'interno del biofilm, possono influenzare sia l'attività dell'agente antimicrobico che la risposta dei microbi all'antimicrobico21. Incorporando questi fattori, siamo stati in grado di sviluppare un modello altamente versatile che può consentire una previsione più accurata dei risultati del trattamento antimicrobico rispetto ai modelli attuali che non considerano i fattori di cui sopra.
P. aeruginosa, S. aureus e C. albicans sono stati scelti come funghi rappresentativi Gram-positivi, Gram-negativi e comunemente isolati nella CF32. Tuttavia, siamo stati anche in grado di dimostrare che questo modello può essere utilizzato per una serie di altri microbi rilevanti per la fibrosi cistica, tra cui Burkholderia cenocepacia, Burkholderia multivorans, Aspergillus fumigatus, ed è attualmente in fase di adattamento per l'uso con batteri anaerobi come Prevotella melaninogenica. Ciò dimostra la versatilità del modello in quanto è stato possibile incorporare un'ampia gamma di microbi, evidenziando l'adattabilità di questo modello per i test antimicrobici contro un'ampia gamma di agenti patogeni microbici. Il modello può anche essere facilmente adattato a una serie di condizioni ambientali per test di sensibilità antimicrobica più personalizzati, inclusa la sostituzione di SCFM2 con l'espettorato del paziente.
Anche la considerazione di modelli di biofilm polimicrobico per la sperimentazione di nuovi antimicrobici è fondamentale, in quanto possono colmare una lacuna esistente nella pipeline di sviluppo degli antimicrobici prima del loro utilizzo in studi su animali o sull'uomo33,34. Accanto a ciò, il modello mantiene una produttività relativamente elevata e consente una serie di risultati rilevanti sia per l'industria che per la ricerca.
Sebbene in questo articolo abbiamo solo esemplificato le CFU e l'attività metabolica, abbiamo anche dimostrato l'utilità del modello nell'analisi dell'esometaboloma del biofilm utilizzando la spettrometria di massa per l'analisi superficiale di estrazione di liquidi (LESA). Ciò consente di sondare alcuni dei meccanismi molecolari alla base della tolleranza antimicrobica35. Oltre a questi altri risultati, abbiamo anche dimostrato l'uso di questo modello per testare nuovi sistemi di somministrazione di antimicrobici. Questi includono coniugati polimero-ciprofloxacina per migliorare la penetrazione di questo antibiotico all'interno del biofilm, aumentandone l'attività e riducendo lo sviluppo di resistenza36.
Passaggi critici e considerazioni
È importante mantenere la sterilità, soprattutto quando si producono SCFM2 e terreni, poiché la contaminazione può provocare l'introduzione di specie non presenti in natura, che possono interferire con i risultati. Ceppi diversi dello stesso microbo e microbi diversi possono richiedere condizioni ambientali diverse per la crescita o possono avere caratteristiche di crescita diverse. Pertanto, il modello deve essere ottimizzato prima di eseguire qualsiasi test di sensibilità agli antibiotici. Si consiglia di effettuare un piccolo studio pilota di 24 e 48 ore per le specie mono e il biofilm polimicrobico per valutare la crescita in condizioni di modello standard. Abbiamo scoperto che diversi ceppi di P. aeruginosa e alcune concentrazioni di antibiotici possono indurre la sciamatura dal disco di policarbonato. Questo problema può essere facilmente superato utilizzando dischi di dimensioni maggiori disponibili in commercio. La disgregazione del biofilm può anche richiedere un'ottimizzazione a seconda del battitore di microsfere e dei microbi utilizzati. Abbiamo ottimizzato il metodo di disgregazione determinando il CFU/mL di un campione noto prima e dopo la battitura del microschino. Abbiamo anche usato la microscopia per visualizzare i campioni di bead beat per visualizzare quanto bene il biofilm è stato interrotto.
Uno dei limiti di questo modello di biofilm che imita la FC è l'attaccamento a una superficie solida contenente policarbonato, che non si trova nel polmone FC, piuttosto che essere aggregati liberi all'interno dell'espettorato CF37 . Tuttavia, il modello di agar solido qui presentato preclude la necessità di lavare i biofilm per rimuovere i microbi planctonici e garantisce che i risultati includano solo i microbi derivati dal biofilm rispetto a una coltura di biofilm planctonica mista. Abbiamo anche scoperto che la penetrazione di alcuni antibiotici nelle aree più profonde di questo modello di biofilm può essere ridotta e l'uso di DNasi può essere necessario per alterare la reologia della matrice extracellulare e diminuire le interazioni elettrostatiche con l'eDNA.
Riteniamo che il modello sia altamente adatto per i test antimicrobici grazie alla sua versatilità, adattabilità e inclusione di fattori ambientali che molti modelli esistenti non riescono a prendere in considerazione. Il suo utilizzo nella ricerca di base e nei test preclinici di sensibilità antimicrobica può fornire un approccio clinicamente più rilevante per l'AST di campioni clinici e lo sviluppo di nuove terapie.
Divulgazioni
Per questo lavoro non viene dichiarato alcun conflitto di interessi o interesse finanziario concorrente.
Riconoscimenti
Questo lavoro è stato finanziato dal National Biofilms Innovation Centre (NBIC), un centro di innovazione e conoscenza finanziato dal Biotechnology and Biological Sciences Research Council, Innovate UK e Hartree Centre [Awards BB/R012415/1 e BB/X002950/1] e dal CF Trust del Regno Unito e dal CF Foundation Strategic Research Centre del Regno Unito: "Un quadro preclinico basato sull'evidenza per lo sviluppo di terapie antimicrobiche nella fibrosi cistica" (PIPE-CF) [Premio SRC022].
Materiali
| Name | Company | Catalog Number | Comments |
| 1 µL inoculation loops | |||
| 13 mm 0.2 µm pore size polycarbonate discs | Isopore | GTTP01300 | Larger discs are also available |
| 2 mL reinforced tubes | Thermofisher | 15545809 | |
| 2.5 mL ceramic beads | Qiagen | 13114-325 | |
| 500 mL borosilicate glass Duran bottle | Sigma Aldrich | Z305197 | larger bottles available in larger volumes are desired |
| 6-well culture plates | Greiner | 657165 | |
| 7 mL Bijou | Thermofisher | 129B | |
| 96-well plates | Thermofisher | 167008 | for serial dilutions in CFU assay |
| Agar plates for preparing plates of P. aerugnisa, S. aureus, and C. albicans | LB miller for P. aeruginosa and S. aureus and Sabouraud dextrose agar for C. albicans | ||
| Bead beater - suitable for 2 mL tubes | Fisherbrand | 15515799 | Thermofisher bead mill 24 |
| bench top centrifuge | must be capable of at least 8000 x g | ||
| Black clear bottom 96 well plates | Costar | 3603 | |
| Bunsen Burner | |||
| Containers for disposing of contaminated equipment and material according to the institutes health and safety regulations. | |||
| deionised water | |||
| eDNA | Sigma Aldrich | 31149 | |
| Filter unit | Fisherbrand | FB12566504 | Interchangeable depending on the vacuum pump used but must have a pore size of 0.2 µm |
| Haemocytometer and cover slip | Hawksley | HC001 | Haemocytometers may differ in size and volume. Double check and adjust CFU calculations accordingly |
| LB broth | oxoid | 1.46813 | |
| Mannitol salt agar | Oxoid | CM0085B | |
| meropenem | abcr | Ab243429 | |
| Mucin from porcine stomach Type II | Sigma Aldrich | M2378 | |
| Nystatin | Millipore | 1003352658 | |
| petri dishes | SLS | SLS2000 | |
| Phosphate buffered saline | |||
| Pseudomonas isolation agar | Millipore | 17208 | |
| Resazurin sodium salt | Sigma Aldrich | 199303 | |
| Sabouraud dextrose agar | Oxoid | CM0041 | |
| selection of forceps | fine tipped and tissue forceps with teeth for transferring ceramic beads | ||
| serological pipette | |||
| shaking and static incubators | must be temperature controlled | ||
| Sparks microtitre plate reader | Tecan | For Resazurin assay the microtitre plate reader must have the appropriate filters or be a monochromator for detecting flourescence. | |
| spectrophotometer | |||
| Technical agar (Agar Technical No.2 ) | Oxoid | LP0012B | |
| tetracycline | Sigma Aldrich | T7660 | |
| UV crosslinker | Spectroline | 11-992-89 | |
| vacuum pump/ flask | Fisherbrand | FB12566504 | |
| water bath | must be capable of maintaining 55 °C | ||
| YPD broth | Millipore | Y1375 | Can be bought pre-made or made using the base ingredients |
Riferimenti
- . 2021 Cf foundation patient registry highlights report Available from: https://cysticfibrosis.msu.edu/index.php/welcome/news-and-events/39-2021-cf-foundation-patient-registry-highlights-report (2022)
- Guo, J., Garratt, A., Hill, A. Worldwide rates of diagnosis and effective treatment for cystic fibrosis. J Cyst Fibros. 21 (3), 456-462 (2022).
- Mandal, V., Ghosh, N. N., Mitra, P. K., Mandal, S., Mandal, V. Production and characterization of a broad-spectrum antimicrobial 5-butyl-2-pyridine carboxylic acid from Aspergillus fumigatus nhf-01. Sci Rep. 12 (1), 6006 (2022).
- Françoise, A., Héry-Arnaud, G. The microbiome in cystic fibrosis pulmonary disease. Genes. 11 (5), 536 (2020).
- Grasemann, H., Ratjen, F. Cystic fibrosis. New Engl J Med. 389 (18), 1693-1707 (2023).
- Camus, L., Vandenesch, F., Moreau, K. From genotype to phenotype: Adaptations of pseudomonas aeruginosa to the cystic fibrosis environment. Microb Genom. 7 (3), mgen000513 (2021).
- Rossi, E., et al. Pseudomonas aeruginosa adaptation and evolution in patients with cystic fibrosis. Nat Rev Microbiol. 19 (5), 331-342 (2021).
- Flemming, H. -. C., Wingender, J. The biofilm matrix. Nat Rev Microbiol. 8 (9), 623-633 (2010).
- Grainha, T., Jorge, P., Alves, D., Lopes, S. P., Pereira, M. O. Unraveling pseudomonas aeruginosa and candida albicans communication in coinfection scenarios: Insights through network analysis. Front Cell Infect Microbiol. 10, 550505 (2020).
- Flemming, H. C., et al. Biofilms: An emergent form of bacterial life. Nat Rev Microbiol. 14 (9), 563-575 (2016).
- Høiby, N., Bjarnsholt, T., Givskov, M., Molin, S., Ciofu, O. Antibiotic resistance of bacterial biofilms. Int J Antimicrob Agents. 35 (4), 322-332 (2010).
- Lorusso, A. B., Carrara, J. A., Barroso, C. D. N., Tuon, F. F., Faoro, H. Role of efflux pumps on antimicrobial resistance in Pseudomonas aeruginosa. Int J Mol Sci. 23 (24), 15779 (2022).
- Michaelis, C., Grohmann, E. Horizontal gene transfer of antibiotic resistance genes in biofilms. Antibiotics .(Basel). 12 (2), 328 (2023).
- Worlitzsch, D., et al. Effects of reduced mucus oxygen concentration in airway pseudomonas infections of cystic fibrosis patients. J Clin Invest. 109 (3), 317-325 (2002).
- König, J., Schreiber, R., Voelcker, T., Mall, M., Kunzelmann, K. The cystic fibrosis transmembrane conductance regulator (CFTR) inhibits ENaC through an increase in the intracellular Cl- concentration. EMBO Reps. 2 (11), 1047-1051 (2001).
- Palmer, K. L., Mashburn, L. M., Singh, P. K., Whiteley, M. Cystic fibrosis sputum supports growth and cues key aspects of Pseudomonas aeruginosa physiology. J Bacteriol. 187 (15), 5267-5277 (2005).
- Valenza, G., et al. Prevalence and antimicrobial susceptibility of microorganisms isolated from sputa of patients with cystic fibrosis. J Cyst Fibros. 7 (2), 123-127 (2008).
- Alam, F., Catlow, D., Di Maio, A., Blair, J. M. A., Hall, R. A. Candida albicans enhances meropenem tolerance of Pseudomonas aeruginosa in a dual-species biofilm. J Antimicrob Chemother. 75 (4), 925-935 (2020).
- Burmølle, M., Ren, D., Bjarnsholt, T., Sørensen, S. J. Interactions in multispecies biofilms: Do they actually matter. Trends Microbiol. 22 (2), 84-91 (2014).
- Lories, B., Belpaire, T. E. R., Smeets, B., Steenackers, H. P. Competition quenching strategies reduce antibiotic tolerance in polymicrobial biofilms. NPJ Biofilms Microbiomes. 10 (1), 23 (2024).
- Nabb, D. L., et al. Polymicrobial interactions induce multidrug tolerance in Staphylococcus aureus through energy depletion. Front Microbiol. 10, 2803 (2019).
- Cornforth, D. M., et al. Pseudomonas aeruginosa transcriptome during human infection. Proc Natl Acad Sci U S A. 115 (22), E5125-E5134 (2018).
- Wolter, D. J., et al. Staphylococcus aureus small-colony variants are independently associated with worse lung disease in children with cystic fibrosis. Clin Infect Dis. 57 (3), 384-391 (2013).
- Takagi, J., et al. Mucin o-glycans are natural inhibitors of Candida albicans pathogenicity. Nat Chem Biol. 18 (7), 762-773 (2022).
- Cinical and Laboratory Standard Institute. . Methods for dilution susceptibility tests for bacteria that grow aerobically. 35 (2), (2015).
- Roberts, A. E., Kragh, K. N., Bjarnsholt, T., Diggle, S. P. The limitations of in vitro experimentation in understanding biofilms and chronic infection. J Mol Biol. 427 (23), 3646-3661 (2015).
- Macia, M. D., Rojo-Molinero, E., Oliver, A. Antimicrobial susceptibility testing in biofilm-growing bacteria. Clin Microbiol Infect. 20 (10), 981-990 (2014).
- Coenye, T., Goeres, D., Van Bambeke, F., Bjarnsholt, T. Should standardized susceptibility testing for microbial biofilms be introduced in clinical practice. Clin Microbiol Infect. 24 (6), 570-572 (2018).
- Turner, K. H., Wessel, A. K., Palmer, G. C., Murray, J. L., Whiteley, M. Essential genome of Pseudomonas aeruginosa in cystic fibrosis sputum. Proc Natl Acad Sci U S A. 112 (13), 4110-4115 (2015).
- Miles, A. A., Misra, S. S., Irwin, J. O. The estimation of the bactericidal power of the blood. J Hyg. 38 (6), 732-749 (1938).
- Robertson, S. N., Romero, M., Fenn, S., Kohler Riedi, P. L., Cámara, M. Development, characterization, and evaluation of a simple polymicrobial colony biofilm model for testing of antimicrobial wound dressings. J Appl Microbiol. 135 (3), lxae042 (2024).
- Rumpf, C., Lange, J., Schwartbeck, B., Kahl, B. C. Staphylococcus aureus and cystic fibrosis-a close relationship. What can we learn from sequencing studies. Pathogens. 10 (9), 1177 (2021).
- Tay, W. H., Chong, K. K. L., Kline, K. A. Polymicrobial-host interactions during infection. J Mol Biol. 428 (17), 3355-3371 (2016).
- Orazi, G., O'toole, G. A. "It takes a village": Mechanisms underlying antimicrobial recalcitrance of polymicrobial biofilms. J Bacteriol. 202 (1), e00530-e00519 (2019).
- Arjes, H. A., et al. Three-dimensional biofilm colony growth supports a mutualism involving matrix and nutrient sharing. eLife. 10, e64145 (2021).
- Kasza, K., et al. Ciprofloxacin poly(β-amino ester) conjugates enhance antibiofilm activity and slow the development of resistance. ACS Appl Mater Interf. 16 (5), 5412-5425 (2024).
- Martin, I., Waters, V., Grasemann, H. Approaches to targeting bacterial biofilms in cystic fibrosis airways. Int J Mol Sci. 22 (4), 2155 (2021).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon