Method Article
Profilage de la méthyltransférase et autres S-Adénosyl- L-Homocystéine protéines liant par spectrométrie de masse de captation composé (CDSM)
Dans cet article
Résumé
Composés de capture sont trifonctionnel petites molécules afin de réduire la complexité du protéome par fonctionnels réversibles petite molécule de protéine d'interaction suivie par la photo-réticulation et de purification. Ici, nous utilisons un composé de capture avec des S-Adénosyl- L-Homocystéine-contraignante que la fonction de sélectivité pour isoler méthyltransférases d'un Escherichia coli Cellule entière lysat et les identifier par la SP.
Résumé
Il ya une variété d'approches pour réduire la complexité du protéome sur la base de petites molécules fonctionnelles interactions protéine-telles que la chromatographie d'affinité ou d'une protéine de profilage Activity Based 2. Composés de capture trifonctionnelle (CC, figure 1A) 3 sont à la base d'une approche générique, dans lequel l'équilibre initial axé sur l'interaction entre une sonde petite molécule (la fonction de sélectivité, ici S-adénosyl-L-homocystéine, HSA, figure 1A) et les protéines cibles est irréversiblement fixée sur la photo-réticulation entre une fonction de réactivité photo activables indépendante (ici un phénylazide) du CC et la surface des protéines cibles. La fonction de tri (ici biotine) sert à isoler le CC - conjugués de protéines à partir de mélanges complexes biologiques à l'aide d'une phase solide (ici des billes de streptavidine magnétique). Deux configurations de ces expériences sont possibles: "off-talon" 4 ou le décrit présentement «sur-talon" de configuration (figure 1B). La fonction de sélectivité peut être pratiquement n'importe quelle petite molécule d'intérêt (substrats, inhibiteurs, des molécules de médicaments).
S-adénosyl-L-méthionine (SAM, figure 1A) est probablement, la deuxième à l'ATP, le cofacteur le plus largement utilisé dans la nature 5, 6. Il est utilisé comme donneur de groupe méthyle majeur dans tous les organismes vivants avec la réaction chimique catalysée par SAM-dépendante méthyltransférases (MTases), qui méthylate 7 de l'ADN, l'ARN 8, 9 protéines, ou des petites molécules 10. Étant donné le rôle crucial des réactions de méthylation dans divers scénarios physiologiques (la régulation des gènes, l'épigénétique, le métabolisme), le profilage des MTases peut être appelé à devenir d'une importance similaire dans la protéomique fonctionnelle le profilage de kinases. Des outils analytiques pour leur profilage, cependant, n'ont pas été disponibles. Nous avons récemment introduit un CC avec HSA en tant que groupe de sélectivité pour combler ce fossé technologique (figure 1A).
HSA, le produit de la SAM après le transfert de méthyle, est un inhibiteur général des produits connus MTase 11. Pour cette raison et parce que le naturel cofacteur SAM est utilisé par d'autres enzymes de transférer d'autres parties du cofacteur ou initier des réactions radicalaires ainsi qu'en raison de son instabilité chimique 12, SEP est une fonction de sélectivité idéale pour un CC de cibler MTases. Nous rapportons ici l'utilité de la SAH-CC et CCMS par MTases profilage et d'autres protéines liant l'HSA de la souche DH5a d'Escherichia coli (E. coli), l'un des mieux caractérisés les procaryotes, qui a servi de modèle préféré organisme dans d'innombrables études biochimiques, biologiques et biotechnologiques. Photo-activé réticulation améliore le rendement et la sensibilité de l'expérience, et la spécificité peuvent être facilement testés dans des expériences de compétition en utilisant un excès du libre HSA.
Protocole
1) Préparation de E. lysat cellulaire coli DH5a
- Inoculer une culture 2mL (milieu LB dans un tube à essais) avec les E. coli souche DH5a directement à partir d'un stock de glycérol et incuber à 37 ° C et 250 rpm pendant 8 h. Utiliser les médias LB autoclave (10 g / l de Bacto-tryptone, extrait de levure 5 g / L, 10 g / L de NaCl, pH 7,5).
- Inoculer 250 milieux LB mL dans une fiole de 1 L shaker avec chicanes avec la culture de 2 ml et incuber pendant la nuit à 37 ° C et 166 rpm dans un incubateur avec agitation.
- Inoculer quatre 5 L flacons shaker avec chicanes contenant chacun 2,5 L milieux LB avec la culture de 250 ml (50 ml pour chaque flacon) et incuber les cultures à 37 ° C et 166 rpm jusqu'à une DO 600 de 0,8 est atteint.
- Récolter les cellules par centrifugation à 4 ° C g, 3000x pour 20 min. Effectuez la manipulation ultérieure à 0-4 ° C ou sur glace.
- Re-suspendre le matériel récolté dans la cellule eau Milli-Q, se combinent dans un seau à centrifuger et centrifuger pendant 30 min supplémentaires à 6000x g et 4 ° C.
- Stocker le matériel cellulaire résultant 20 g à -20 ° C ou -78 ° C.
- Re-suspendre les cellules dans 100 ml de glace ouverture de la cellule tampon froid (6,7 mM MES, 6,7 mM de NaOAc, 6,7 mM d'HEPES, 1 mM EDTA, 10 mM de β-mercaptoéthanol, 200 mM NaCl, pH 7,5, 10% (p / v) glycérol; 0,2 mM PMSF) et soniquer trois reprises pendant 1 min dans quatre portions de 25 ml sur la glace en utilisant un sonificateur (par exemple SONOPULS HD 2070 d'Bandelin electronic GmbH & Co. KG, amplitude maximale, sortie en continu).
- Centrifuger le lysat pendant la nuit à 2370x g et 2 ° C.
- Concentrer le surnageant à 14 ml par ultrafiltration (par exemple en utilisant l'icône concentrateurs, 7 mL/9K, de Pierce) à 2370x g et 2 ° C.
- Appauvrissent le concentré visqueux de petites molécules comme SAM ou HSA par filtration sur gel à 2 ° C (par exemple les colonnes Zeba Spin dessaler, 10 ml, de Pierce, quatre colonnes; stockage tampon retiré à g 1000x pendant 2 min; quatre fois avec 5 ml d'équilibration tampon froid de glace ouverture de la cellule et le tampon retiré à g 1000x pendant 2 min, respectivement, 3,5 ml solution à diluer appliquée à chaque colonne; 45 min à centrifugation g 24x, puis deux fois 2 min à 1000x g)
- Supplément du 13 lysat résultant ml avec 13 ml de glycérol de la glace froide et Roche mini tablettes cocktail inhibiteur de protéase, sans EDTA. Mélanger et dissoudre les comprimés.
- Stocker le lysat à -20 ° C
- Déterminer la concentration en protéines totales par le dosage de Bradford (21 mg / ml dans le cas présent). Réactif de Bradford: 100 mg Coomassie Brilliant Blue G-250 dans 50 ml d'éthanol à 95%, ajouter 100 ml d'acide phosphorique à 85% (p / v), diluer à 1 L lorsque le colorant est totalement dissous, et le filtre Whatman n ° 1 de papier juste avant l'utilisation.
Dosage de capture 2) (A), contrôle de la concurrence (C), pulldown (PD), le contrôle de la concurrence du pulldown (DPC), et dosage de capture combinés ainsi pulldown (A PD +)
- Pour les expériences de capture, le caproKit SAH (caprotec bioanalytique GmbH) a été utilisé, ce qui inclut le SEP-CC, sans HSA en tant que concurrent, la streptavidine billes magnétiques recouvertes avec 1 pm de diamètre (Dynabeads MyOne Streptavidine C1, Invitrogen Dynal), mémoire tampon de capture 5X (5X CB, contenant 100 mM d'HEPES, 250 mM d'acétate de potassium, 50 mM acétate de magnésium et de glycérol à 50%), et un tampon de lavage 5X (5X BM, contenant 250 mM de Tris HCl, pH 7,5, EDTA 5 mM, 5 M de NaCl, 42,5 octyl-β uM-D-glucopyranoside).
- Pour plusieurs expériences en parallèle, il est recommandé de préparer un mélange maître de l'eau, la capture de tampon et E. lysat coli et de réaliser des réactions au sein de différents tubes d'un 200 bandes à tubes uL-PCR (recommandé 0,2 mL Thermo-bande, Thermo Scientific, AB-1114). Ici, les quantités pour un tube de réaction sont donnés. Résultats pour cinq expériences différentes seront présentées, qui sont dosage de capture (A), contrôle de la concurrence (C), pulldown (PD), contrôle de la concurrence du pulldown (DPC), et dosage de capture combinés à la pulldown (A PD +).
- Pour chaque réaction, préparer 1,5 ml 1X BM en ajoutant 0,3 ml 5X BM à 1,2 mL d'eau Milli-Q.
- Préparer SAH-CC chargés recouvertes de streptavidine billes magnétiques (caproBeads) dans 200 ul barrettes de tubes PCR. Par conséquent, mélanger 25 uL de 100 uM SAH-CC avec 50 ul de 10 mg / ml de streptavidine billes magnétiques recouvertes de chaque aliquote, agiter vigoureusement les suspensions résultant à la température ambiante pendant 2 min pour permettre la liaison du fragment de biotine du SEP- CC de la streptavidine sur la surface de bille magnétique, et de recueillir les perles de l'aide d'un aimant puissant (par exemple dans les bouchons des barrettes de tubes PCR en utilisant les caproMagTM dispositif magnétique, caprotec bioanalytique GmbH). Jeter le surnageant, remettre en suspension les caproBeads résultant dans 200 ul BM, magnétiquement recueillir les caproBeads (dans les bouchons des barrettes de tubes PCR), et jetez-la BM supernating. Fermer les tubes afin d'éviter le séchage des billes.
- Préparer des aliquotes de E. coli DH5a toutelysat cellulaire dans des tubes PCR nouvelle à 0-4 ° C en utilisant un mélange maître (voir 2.2). Pour une réaction, de compléter un volume d'eau Milli-Q pour un volume de mélange réactionnel final de 100 pl avec 20 uL 5X CB. Mélangez, ajoutez 0,26 mg E. lysat coli, et mélanger délicatement par inversion. Uniquement pour les C et DPC, ajouter 20 uL solution à 10 mM concurrent HSA et mélanger délicatement par inversion (ajouter eau Milli-Q à la place de la solution de HSA à A PD, PD et A +). Dessinez un échantillon de 1 uL de A pour une analyse approfondie (voir ci-dessous).
- Suspendre le caproBeads dans le lysat respectifs et incuber pendant 3 h à 4 ° C garder les billes en suspension par rotation pour permettre la liaison réversible de protéines se liant à l'HSA la fonction de sélectivité de l'HSA HSA-CC.
- Placez les suspensions A, C et A + PD dans la caproBoxTM (appareil pour irradier des échantillons biochimiques avec la lumière UV et en même temps de refroidissement, caprotec bioanalytique GmbH) et irradient d'une durée totale de 30 min dans des tubes fermés entre 0-4 ° C pour former une réticulation covalente entre la fonction de la réactivité du SEP-CC pour les protéines HSA contraignant. Par conséquent, supprimer les suspensions après des intervalles d'irradiation de 2,5 min, respectivement, de la caproBoxTM, refroidir dans l'eau glacée pour ~ 15 s (en particulier les paupières), mélanger plusieurs fois par inversion, très peu de temps (~ 2 s) centrifugeuse pour enlever la suspension restante dans les couvercles, et le lieu de nouveau dans le caproBoxTM pour l'intervalle de l'irradiation prochaine.
- Ajouter 20 uL 10 mM HSA solution à un, ou 20 uL eau Milli-Q à C, PD, DPC, et A + PD et incuber la suspension pendant 10 min à 4 ° C à supplanter, en A, protéines liant HSA non réticulé au SEP-CC. Gardez les billes en suspension par rotation manuelle ou par intermittence la remise en suspension.
- Recueillir les caproBeads des suspensions en utilisant un aimant puissant (par exemple le caproMagTM), jeter le surnageant et laver les billes de six fois - par la remise en suspension et de la collecte - avec 200 pi WB 1X et une fois avec 200 ul eau Milli-Q.
- Les billes peuvent être stockées dans eau Milli-Q pour plusieurs semaines à 4 ° C. Ces autres protocoles existent pour le traitement ultérieur des protéines capturées et leur identification (voir la discussion).
- Laver les billes trois fois avec 200 uL d'acétonitrile 60% (ACN) et la libération des protéines capturées à partir des billes de 10 minutes d'incubation à température ambiante sous agitation vigoureuse avec 200 ul de 60% d'acide trifluoroacétique ACN/0.2% (TFA) (préparation fraîchement) . Utilisez LC-MS-réactifs de qualité et de l'eau.
- Magnétiquement recueillir les perles, séparées et évaporer le surnageant à sec à l'aide d'un évaporateur centrifuge (concentrateur par exemple l'ADN à partir MiVac Genevac, Inc, Royaume-Uni). Jeter des perles.
3) SDS-PAGE des protéines capturées
- Pour SDS-PAGE, dissoudre les protéines capturées libérés des billes magnétiques (évaporé ACN / TFA solutions de l'étape 2.12) dans 20 uL de tampon d'échantillon SDS (Tris HCl 50 mM, 320 mM β-mercaptoéthanol, 2,5% SDS, 0,05% bleu de bromophénol , 10% glycérol, pH 6,8). Mélanger l'échantillon 1 ul tirées de A (voir étape 2.5) avec 19 uL de tampon d'échantillon SDS; utiliser 5 uL de cette solution pour analyse SDS-PAGE (0,25% du dosage). Chauffer les échantillons dans l'échantillon de tampon SDS 10 min à 95 ° C et laissez laisser refroidir à température ambiante.
- Analyser par SDS-PAGE (configuration générique: MCO ® Propage 4-20% Tris / glycine préfabriqués gels; MCO OmniPage mini système d'électrophorèse sur gel; tampon SDS cours d'exécution: 25 mM Tris base, 200 mM de glycine, 0,1% SDS, pH 8,3 ; le temps d'exécution 90 min à une tension constante de 180 V, sous refroidissement de la glace de la mémoire tampon courir SDS).
- Coloration à l'argent du gel en utilisant une méthode compatible avec MS argent tache (par exemple ProteoSilver Argent Kit Stain de Sigma). Un résultat représentatif est montré dans la figure 2.
4) En-gel Digest trypsique de protéines et de peptides à partir d'extractions bandes de gel
- Laver le gel coloré à l'argent au moins trois fois avec 100 ml d'eau Milli-Q pendant 10 min après la solution d'arrêt coloration à l'argent a été retiré.
- Découpez les bandes de gel (par exemple en utilisant un scalpel propre et transférer dans un tube Eppendorf de 0,5 mL) et conserver à -20 ° C ou directement processus. Laver les bandes de gel pendant 15 min, respectivement, avec 100 uL d'eau, 100 ul d'éthanol à 50%, 100 uL d'eau, 100 ul d'éthanol à 50%, et pendant 5 min avec de l'éthanol pur. Répétez cette procédure de lavage une fois de plus.
- Réhydrater la bande de gel avec 10 uL dans le gel de solution de digestion (12,5 ng / uL séquençage trypsine de qualité dans 50 mM de bicarbonate d'ammonium, de préparer en ajoutant 7,5 uL 0,5 ug / uL solution de trypsine à 1 mM HCl à 292,5 uL 50 mM de bicarbonate d'ammonium ) pendant 45 min à 4 ° C. Enlever le surnageant et le remplacer par 20 uL 50 mM de bicarbonate d'ammonium (sans trypsine) suivie d'une incubation à 37 ° C pendant la nuit tout en secouant.
- Recueillir le surnageant. Pour l'extraction des peptides, incuber la bande de gel avec 20 uL d'acide formique 5% (FA) pendant 15 min tout en agitant, ajouter20 uL d'ACN et incubé pendant 15 min tout en secouant. Combinez le surnageant avec le surnageant précédent et répétez la procédure d'extraction de peptides à nouveau.
- Evaporer les trois combinés surnageants à sec, se dissoudre dans 10 uL FA 5% tout en agitant et en appliquant des ultrasons (bain à ultrasons, par exemple à partir Sonorex Bandelin, Allemagne) et de procéder à dessalage (étape 5.2 et plus).
5) Recueil tryptique des protéines capturées et préparation des peptides pour LC-MS/MS
- Dissoudre les protéines capturées libérés des billes magnétiques (évaporé ACN / TFA solutions de l'étape 2.12) dans 10 ul 50 mM de bicarbonate d'ammonium en utilisant un bain à ultrasons et des vortex, ajoutez 1 uL 0,5 ug / uL dans la trypsine HCl 1 mM et incuber la solution à 37 ° C pendant la nuit.
- Dessaler la solution contenant les peptides tryptiques de protéines capturées à l'aide du matériel C18 (par exemple 20-10 StageTips ul, 20 uL astuce, Proxeon Biosystems A / S, Odense, au Danemark, la procédure du fabricant: pré-condition avec 10 uL de 50% de méthanol / 5 FA%, s'équilibrer avec 10 uL FA 5%, la charge avec des peptides de protéines capturées, laver avec 10 FA 5% ul, éluer avec deux fois 10 uL 50% méthanol / FA 5%).
- Evaporer les peptides dessalés dans 50% de méthanol / FA 5% à la sécheresse, se dissoudre dans 5,5 FA 0,1% uL tout en agitant et en appliquant des ultrasons (échographie de bain) et d'analyser l'échantillon par nanoLC-MS/MS.
6) Analyse nanoLC-MS/MS
- Transférer l'échantillon dans la plaque de l'échantillon et la plaque de placer dans la chromatographie liquide nano-débit (nanoLC) système (par exemple facile NLC système de chromatographie liquide; Proxeon Biosystems A / S, Danemark).
- Utilisez FA 0,1% dans l'eau comme phase mobile A et 0,1% en FA ACN comme phase mobile B. Seule l'utilisation de solvants de grade LC-MS.
- Charge 5 pi de la solution de peptide directement sur une pré-colonne (par exemple la biosphère nanodébit C18, 5 um, 120 A, 20 x 0,1 mm; NanoSeparations, Pays-Bas) couplé à une colonne d'analyse (par exemple la biosphère nanodébit C18, 5 um, 120 Å , 100 x 0,075 mm; NanoSeparations, Pays-Bas) en utilisant FA 5% ACN/0.1%.
- Pendant LC, les peptides éluer lors d'un gradient de 80 min linéaire de 5% FA ACN/0.1% à 40% FA ACN/0.1%, suivi par un supplément de 2 min à 100% ACN/0.1% FA et reste à 100% ACN/0.1% FA pour un autre 8 min avec un débit contrôlé de 400 Nl / min.
- Effectuer spectrométrie de masse (MS) l'analyse des peptides élues sur un spectromètre de masse de haute précision l'état de l'art (par exemple LTQ Orbitrap XL spectromètre de masse; Thermo FisherScientific, l'Allemagne, équipée d'une source d'ions pour l'ionisation nanoélectrospray électronébulisation (ESI); Proxeon Biosystems Un / S, Danemark).
- Effectuer l'analyse par spectrométrie de masse en mode dépendant des données pour basculer automatiquement entre les Orbitrap-MS (mode profil) et LTQ-MS/MS (mode centroïde) d'acquisition.
- Contrôle du cycle spectromètre de masse en réglant le temps d'injection contrôle automatique de gain.
- Acquérir enquête balayage complet MS spectres (à partir de m / Z 300 à 2000) dans le Orbitrap avec une résolution R = 60 000 à m / z 400 (après l'accumulation d'une valeur cible de 500 000 accusations dans le piège à ions linéaire).
- Régler l'instrument à isoler de façon séquentielle les ions les plus intenses (jusqu'à cinq, selon l'intensité du signal) pour la fragmentation dans le piège à ions linéaire utilisant dissociation induite par collision (CID) à une valeur cible de 10 000 chefs d'accusation. Les ions fragments résultant sont enregistrés dans le LTQ.
- Pour des mesures précises de masse en mode MS, l'utilisation de la seule charge polydiméthylcyclosiloxane fond ion (Si (CH 3) 2 O) 6 H + (m / z 445,120025) générées pendant le processus d'électronébulisation de l'air ambiant comme la masse de verrouillage pour interne en temps réel recalibrage.
- Dynamiquement excluent les ions de masse cible déjà sélectionnés pour CID pour la durée de 60 s.
- Set de dépistage état de charge et de rejet des ions pour CID avec charge non affectée.
- D'autres paramètres de masse spectrométrique sont comme suit: régler la tension de pulvérisation à 1,6 kV, température de consigne de transfert chauffée capillaire à 200 ° C, et l'énergie de collision normalisée est de 35% pour MS 2. Le signal minimale requise pour MS 2 est de 500 chefs d'accusation. Appliquer une activation q = 0,25 et un temps d'activation de 30 ms pour MS 2 acquisitions.
- Pour nettoyer le système LC, effectuer un seul passage vierge entre deux mesures consécutives CCMS.
7) L'identification des peptides et des protéines via la recherche de séquences base de données automatisée
- Utiliser un algorithme d'identification de protéines pour analyser les données MS / MS (dans le cas présent stockées dans des fichiers bruts), par exemple, mis en œuvre dans SEQUEST BioworksBrowser 3.3.1 SP1 (FisherScientific Thermo, Allemagne) et X! Tandem (L'Organisation mondiale de la machine Proteome, version 2007.01.01.1) mis en œuvre dans le Scaffold 3 du logiciel (version Scaffold_3_00_03, Proteome Software Inc, USA).
- Effectuer base de données automatisée de recherche contre le UniProtKB plus récente / Swiss-Prot www.expasy.org communiqué base de données de l'organisme étudié (base de données utilisée pour la présente étude: Escherichia coli, souche K12, version 57-11).
- Utilisez les paramètres suivants pour base de données automatisée de recherche au sein SEQUEST: tolérance de 5 ppm précurseur, une tolérance de uma fragment ionique, la trypsine et la spécificité complète permettant jusqu'à deux clivages manqués. Permettez que la phosphorylation des modifications variable à sérine, la thréonine, et la tyrosine; oxydation des méthionines; désamidation au asparagines et la glutamine; acétylation à la lysine et la sérine; formylation à la lysine et la méthylation au arginine, la lysine, la sérine, la thréonine, et l'asparagine. Ne pas utiliser modifications fixes dans la recherche de base de données.
- Charge CRS ou DTA et les fichiers générés par SEQUEST dans l'échafaudage 3, qui effectue une évaluation des probabilités de missions de peptides et protéines en combinant les identifications et les recherches SEQUEST X! Tandem base de données. Échafaudage est utile pour comparer facilement et en visualisant les listes de protéines à partir de plusieurs échantillons (en l'espèce A, C, PD, DPC, et A + PD).
- Définissez les paramètres au sein de l'échafaudage 3 logiciels à considérer que les peptides avec une probabilité ≥ 95% comme indiqué par l'algorithme Prophète Peptide (ref.13). Régler les probabilités d'identification de protéines pour des missions de peptides multiples ≥ 95% selon l'algorithme Prophète Protéines 13. Pour les identifications des protéines peptide seul, arbitrairement fixée probabilité protéines à ≥ 50% et inspecter manuellement le peptide correspondant spectres MS / MS. Les protéines qui composent les peptides similaires et ne pouvaient pas être différenciés en fonction de MS / MS seule analyse sont regroupées par le logiciel pour satisfaire aux principes de parcimonie. Le taux de découverte estimé fausses identifications du peptide peut être déterminée en utilisant l'approche de protéines inversée base de données et doit être <1%.
- Les résultats représentatifs d'expériences CDSM sont donnés dans les tableaux 1, 2 et S1 Tableau complémentaire (l'esprit que la base de données des protéines n'est pas mise à jour pour certaines protéines, par exemple PrmB (aka YfcB) ou RsmH (aka MraW)), ainsi comme dans la Figure 3.
8) Les résultats représentatifs
Tableau 1:
| Protéines | ORF | MW / kDa | Descriptif | Substrat | Une | C | PD | DPC | Un PD + |
| Dcm | b1961 | 53,5 | ADN cytosine MTase | ADN (M5C) | 1 | 0 | 0 | 0 | 1 |
| RlmI | b0967 | 44,4 | ARNr 23S m5C1962 MTase | ARNr (M5C) | 17 | 0 | 17 | 0 | 20 |
| RlmL | b0948 | 78,9 | ARNr 23S m2G2445 MTase | ARNr (M2G) | 12 | 0 | 0 | 0 | 10 |
| TRMB | b2960 | 27,3 | ARNt (guanine-N (7) -)-MTase | ARNt (m7G) | 11 | 0 | 0 | 0 | 13 |
| CMOA | b1870 | 27,8 | ARNt (cmo5U34)-MTase | ARNt (mcmo5U) | 7 | 0 | 0 | 0 | 4 |
| RsmG | b3740 | 23,4 | ARNr 16S m7G MTase | ARNr (m7G) | 6 | 0 | 1 | 0 | 5 |
| RsmH | b0082 | 34,9 | ARNr 16S m4C1402 MTase | ARNr (M4C) | 5 | 0 | 0 | 0 | 7 |
| RsmD | b3465 | 21,7 | ARNr 16S m2G966 MTase | ARNr (M2G) | 2 | 0 | 0 | 0 | 2 |
| RSMB | b3289 | 48,3 | ARNr 16S m5C967 MTase | ARNr (M5C) | 1 | 0 | 0 | 0 | 0 |
| MnmC | b2324 | 74,4 | Protéine bifonctionnelle comprend ARNt (MNM (5) s (2) U34)-MTase | ARNt (mnm5s2U) | 1 | 0 | 0 | 0 | 0 |
| PrmB | b2330 | 35,0 | 50S du ribosome protéines L3 Gln150 MTase | protéines (Gin) | 13 | 0 | 0 | 0 | 15 |
| Cher | b1884 | 32,8 | MTase protéines chimiotactisme | protéines (Glu) | 0 | 0 | 0 | 0 | 1 |
| Cfa | b1661 | 44,9 | Cyclopropane-gras-acyl-synthase phospholipides | petite molécule | 15 | 0 | 0 | 0 | 14 |
| Tam | b1519 | 29,0 | Trans-2-aconitate MTase | petite molécule | 2 | 0 | 0 | 0 | 3 |
| CYSG | b3368 | 50,0 | Synthase Siroheme comprend uroporphyrinogène-III-C MTase | petite molécule | 1 | 0 | 0 | 0 | 2 |
| ATTM | b0921 | 29,8 | Protéines ATTM | (? A) | 7 | 1 | 0 | 0 | 8 |
| MtnN | b0159 | 24,4 | 5'-méthylthioadénosine / SAH nucleosidase | b petite molécule | 36 | 0 | 0 | 0 | 39 |
| GlnA | b3870 | 51,9 | La glutamine synthétase | petite molécule C | 90 | 0 | 0 | 0 | 97 |
| RplK | b3983 | 14,9 | Protéines ribosomales 50S L11 | de protéines MTase d PRMA | 2 | 0 | 0 | 0 | 2 |
une pas (complètement) caractérisé
b Pas de méthylation, mais le clivage de la liaison glycosidique du SAH
c Pas de méthylation, mais contraignant de HSA dans le site fixation de l'ATP, comme indiqué par des expériences CCMS avec l'ATP en tant que concurrent (données non présentées)
Substrat d de la 50S ribosomal protéines L11 PRMA MTase; reproductibles identification spécifique par le CCMS (données non présentées)
Tableau 1: MTases et d'autres protéines sélectionnées identifiés par des expériences CCMS. Les chiffres donnés indiquent le nombre de peptides non pondéré spectrale par protéine. Les échantillons sont des duplicata de celles analysées par SDS-PAGE/silver tache dans la Figure 2. MTases beaucoup plus et d'autres protéines de liaison HSA sont identifiés dans le dosage de CCMS (A) par rapport à la liste déroulante (PD) et la spécificité SAH est montré par l'absence presque complète de ces protéines dans le contrôle de la concurrence (C).
Tableau 2:
| Une | C | PD | DPC | Un PD + | |
| Une | 111 (64) | ||||
| C | 65 (41) | 107 (46) | |||
| PD | 25 (15) | 23 (13) | 61 (17) | ||
| DPC | 23 (13) | 22 (12) | 20 (14) | 47 (14) | |
| Un PD + | 87 (61) | 64 (41) | 23 (14) | 22 (12) | 124 (67) |
Tableau 2: Nombre total de protéines identifiées dans le CCMS fonctionne et les chevauchements entre les protéines s'exécute. Le nombre de protéines identifiées avec au moins deux peptides sont indiqués entre parenthèses. La grande reproductibilité de la méthode peut être déduit de la superposition élevée en protéines (principalement des protéines non spécifiques) entre les expériences comparables (A vs C, et surtout A vs A + PD, mais aussi PD vs DPC), en particulier avec les protéines robuste identifié avec au moins 2 peptides. Voir aussi la figure 3 pour les diagrammes de Venn et S1 tableau supplémentaire pour obtenir une liste de toutes les protéines identifiées.
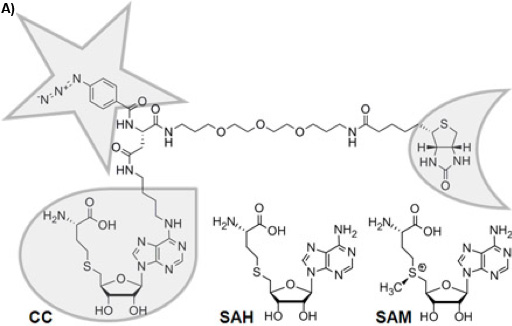
Figure 1A: la structure chimique du composé de capture trifonctionnel (CC). La fonction de sélectivité est encadrée avec une gouttelette, la fonction de réactivité avec une étoile, et la fonction de tri avec une demi-lune. La chimiquement stable S-adénosyl-L-homocystéine (SAH) est le produit cofacteur de la S-adénosyl-L-méthionine (SAM) après le transfert du groupe méthyle par SAM-dépendante MTases, pour lequel HSA agit comme un inhibiteur de produit.
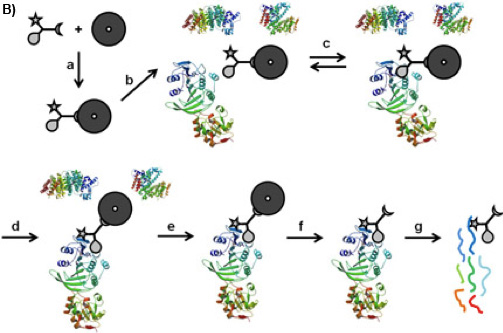
Figure 1B: CCMS "sur-talon "workflow. Le CC est lié sur les billes magnétiques par sa fonction de tri (a), le caproBeads ainsi formées sont incubés avec le mélange complexe de protéines (b), où un équilibre réversible contraignant (c) est établi entre la fonction de sélectivité de le CC et les protéines cibles. Après irradiation UV (d), la fonction de la réactivité constitue un covalente réticuler. Après le lavage des billes magnétiques portant les protéines capturées (e), le clivage de la réticulé CC complexes protéine-des billes magnétiques (f) , et de digestion trypsique (g), les protéines capturées peuvent être identifiés par analyse MS des peptides tryptiques.
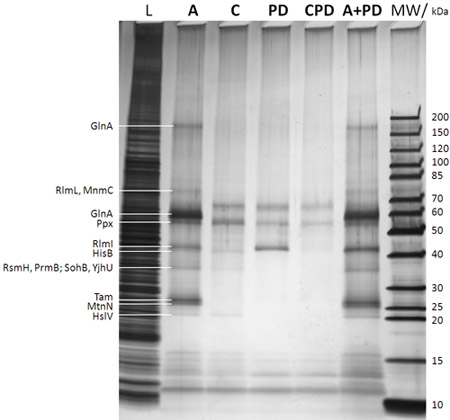
Figure 2: Analyse SDS-PAGE/silver tache de protéines capturées (après l'étape f à la figure 1B). La description est donnée voies sur le dessus du gel (MW: marqueur de poids moléculaire avec les pondérations correspondantes moléculaire des bandes de marqueur donné à la même droite; L: 0,25% de l'échantillon tiré à partir du lysat de E. coli DH5a cellules entières avant d'ajouter le caproBeads à l'étape b dans la figure 1B; A: dosage avec addition d'un excès de SAH libre après l'irradiation d étape UV dans la figure 1B, C: contrôle du dosage, y compris un excès de SAH gratuitement en tant que concurrent au cours des étapes c et d dans la figure 1B (indispensable pour déterminer les protéines non spécifiquement capturés); PD: signification déroulant pas d irradiation UV étape dans la figure 1B et aucun ajout de libre SAH; CPD: le contrôle de pulldown utilisant HSA en tant que concurrent; Un PD +: test combiné, plus pulldown sens pas plus des HSA gratuitement pendant le workflow). Les protéines identifiées par MS à partir découpe des bandes de gel protéique après en gel digestion trypsique sont donnés à l'leftt même. Il est évident que la photo-réticulation améliore le rendement et la sensibilité de l'expérience, et la spécificité peuvent être facilement testés dans des expériences de compétition en utilisant un excès du libre HSA. Voir tableau 1 pour MTases et d'autres protéines sélectionnées identifiés par des expériences CDSM des échantillons en double de ceux indiqués dans le chiffre actuel.

Figure 3: Les diagrammes de Venn qui expliquent le chevauchement des protéines identifiées dans le dosage du CCMS (A), contrôle de la concurrence (C), et pulldown (PD). Gauche: Nombre de MTases et nucleosidase HSA, seulement, se référant au tableau 1. Droite: Nombre de toutes les protéines identifiées se référant au tableau 2 et S1 Tableau complémentaire. Le nombre de protéines identifiées avec au moins deux peptides sont indiqués entre parenthèses.
Discussion
Les précautions suivantes et les commentaires peuvent être utiles quand on suit le protocole décrit: a) Un avantage majeur du CC réside dans la formation d'une liaison covalente entre le CC et le MTase, car cela permet subséquente des conditions de lavage rigoureux. La réticulation covalente est réalisée par un photoréaction déclenchée par la lumière UV (310 nm max.). Plafonnier normal contient seulement une petite fraction des rayons UV, toutefois, de protéger le SEP-CC de plus l'exposition au rétroprojecteur ou la lumière, même lever du soleil, à l'irradiation contrôlée et refroidi dans le caproBox. b) Les échantillons biologiques à partir desquels l'MTases doivent être isolés peuvent contenir des protéines susceptibles de dénaturation, par conséquent, il est obligatoire de conserver les échantillons frais et d'éviter de faire mousser à tout moment. c) Le caproBox refroidit les échantillons à 0-4 ° C, les lampes émettant de la lumière UV, cependant, émettent également de la chaleur. Par conséquent, il est nécessaire de centrifuger brièvement les flacons avant l'irradiation, donc des protéines en respectant les plafonds ou les murs flacon ne peuvent pas former des graines de précipitation. d) Si la remise en suspension des billes magnétiques (par exemple dans les solutions de lavage) n'est pas possible à la main, peu applicables à ultrasons en plaçant les échantillons dans un bain à ultrasons. e) fraîchement préparer la solution d'ACN 0,2% TFA/60%. Nous avons constaté que les protéines capturées contraire ne peut être clivée de la perles. f) L'analyse finale des protéines capturées est réalisée par LC-MS/MS. La spectrométrie de masse est une méthode hautement sensible. Il est nécessaire d'utiliser des réactifs exclusivement LC-MS de qualité dans les étapes finales (étape 2.11 et plus). Eviter la contamination des expériences par des sources de protéines externes, la kératine, par exemple en provenance de la poussière ou de l'expérimentateur. Particulièrement au cours des étapes de digestion finale, il est recommandé de faire attention à un espace de travail propre, de porter des gants et une blouse de laboratoire et, éventuellement, un filet à cheveux ou idéalement exécuter les étapes finales sous un banc propre. Préparer le tampon de 50 mM de bicarbonate d'ammonium utilisé pour digestion trypsique en LC MS eau de qualité, filtrer à travers filtre de 0,22 um, aliquote, conserver à -20 ° C, et utiliser chaque aliquote qu'une seule fois pour éviter les contaminations. La solution de trypsine (séquençage de grade, Roche, préparer un g 0,5 / ul solution en ajoutant HCl 1 mM d'lyophilisé trypsine) peuvent être conservés à 4 ° C pendant plusieurs semaines. g) Pour obtenir des spectres de masse fiable, il est essentiel d'avoir un spray stable dans l'analyse ESI-MS/MS. h) Pour les systèmes LC-MS/MS autre que celui utilisé dans la présente étude, les paramètres de mesure et d'algorithmes d'identification de peptides doivent être ajustés individuellement.
Les modifications suivantes sont possibles en ce qui concerne le protocole décrit: a) Comme alternative à la libération des protéines à partir des billes par du TFA 60% ACN/0.2% (étape 2.11), les protéines peuvent être directement tryptically digérés dans un suspension de billes (mêmes volumes que à l'étape 5.1) ou, pour SDS-PAGE, les protéines peuvent être libérés par la suspension et le chauffage des perles recueillies après l'étape de 2,9 à 95 ° C pendant 10 min dans du tampon échantillon de SDS (à la fois, la suspension totale ou seulement le surnageant peut être chargé dans la poche de gel). Nous avons constaté que les billes de libérer lentement de petites quantités de polymère dans la solution aqueuse digérer tryptique cours sur billes digérer tryptique même après plusieurs étapes de lavage aqueux. La contamination de polymères interfère avec MS peptide d'identification et peuvent être lavés les perles avec 80% d'ACN (au moins trois fois). Après les étapes de lavage de 80% d'ACN, les billes doivent être lavés une fois avec de l'eau avant de le digérer tryptique-billes. b) Western blot à l'aide de streptavidine-peroxydase et le substrat horseraddish ECL peut également être utilisé pour visualiser réticulation succès du CC biotine contenant les protéines. Par conséquent, soit le gel obtenu après l'étape 3.2 peut être effacé ou, parce que la sensibilité est environ 10 fois plus élevé que coloration à l'argent de gels, de 10 échantillons ul après l'étape 2.7 peut également être analysée par western blot. L'esprit que dans ce dernier cas protéines biotinylées endogeneously seront également détectés en dehors des protéines biotinylées artificiellement par le SEP-CC. c) Nous avons constaté que cela dépend du système spécial (lysat, adressée protéines cibles, la sélectivité et la fonction qu'il réactivité de CC), si le "hors-talon" de configuration, où la réaction de réticulation a lieu entre CC libres et des protéines en solution à 4 ou l'présentement décrit "sur-talon" de configuration (figure 1B) fonctionne mieux.
En général, la méthode doit également être compatible avec n'importe quel état de la protéine d'isotopes stables ou l'art technologie d'étiquetage peptide, ou une évaluation de l'échantillon capture par électrophorèse en gel 2D. Dans le "off-talon" de configuration, il est également possible de capturer des protéines dans des cellules entières (résultats non publiés). Par ailleurs, le médicament ou le site cofacteur liaison d'une protéine peut être décrite par la détermination de la position de réticulation de près par le CC dans la séquence protéique par MS séquençage peptidique. Le mode de fixation d'une petite molécule d'une protéine peut être explorée en utilisant différentes cpositions de fixation CHIMIQUE à la fonction de sélectivité et de différentes longueurs de liaison. Comme indiqué dans la présente étude, les partenaires de la protéine lie également des protéines traitées par la fonction de sélectivité (RplK comme substrat du PRMA) ou inconnus petites interactions protéine molécule (HSA au glnA) peuvent être identifiés. Résumé, la caractéristique supplémentaire de la CCS, la réactivité de photo-réticulation, permet à l'isolement et l'identification de protéines de faible abondance ou de familles de protéines fonctionnelles à partir de mélanges complexes de protéines avec une sensibilité élevée et fournit aux scientifiques un outil supplémentaire pour l'étude de petites molécules - les interactions entre protéines .
Déclarations de divulgation
Remerciements
Ce travail a été soutenu par l'Organisation Human Frontier Science Program (HFSP Award 2007, RGP0058/2007-C). Nous tenons à remercier le professeur Richard Roberts pour amorcer le projet et pour des discussions fructueuses.
matériels
| Name | Company | Catalog Number | Comments |
| SAH caproKit™ | caprotec bioanalytics GmbH | 1-1010-050 (50 reactions) 1-1010-010 (10 reactions) | Includes the SAH-CC, SAH competitor, streptavidin coated magnetic beads, capture buffer, and wash buffer |
| caproBox™ | caprotec bioanalytics GmbH | 1-5010-003 (110 V) 1-5010-004 (230 V) | For reproducible photo-activation while cooling the samples |
| caproMag™ | caprotec bioanalytics GmbH | 1-5100-001 | For easy handling of magnetic particles without pipetting |
Références
- Katayama, H., Oda, Y. Chemical proteomics for drug discovery based on compound-immobilized affinity chromatography. J. Chromatogr. B Analyt. Technol. Biomed. Life Sci. 855, 21-27 (2007).
- Barglow, K. T., Cravatt, B. F. Activity-based protein profiling for the functional annotation of enzymes. Nat. Methods. 4, 822-827 (2007).
- Koster, H., Little, D. P., Luan, P., Muller, R., Siddiqi, S. M., Marappan, S., Yip, P. Capture compound mass spectrometry: a technology for the investigation of small molecule protein interactions. Assay Drug Dev. Technol. 5, 381-390 (2007).
- Dalhoff, C., Hueben, M., Lenz, T., Poot, P., Nordhoff, E., Koster, H., Weinhold, E. Synthesis of S-Adenosyl-L-homocysteine Capture Compounds for Selective Photoinduced Isolation of Methyltransferases. ChemBioChem. 11, 256-265 (2010).
- Lu, S. C. S-Adenosylmethionine. Int. J. Biochem. Cell. Biol. 32, 391-3952 (2000).
- Cantoni, G. L. Biological methylation: selected aspects. Annu. Rev. Biochem. 44, 435-451 (1975).
- Jeltsch, A. Beyond Watson and Crick: DNA methylation and molecular enzymology of DNA methyltransferases. ChemBioChem. 3, 274-293 (2002).
- Chow, C. S., Lamichhane, T. N., Mahto, S. K. Expanding the nucleotide repertoire of the ribosome with post-transcriptional modifications. ACS Chem. Biol. 2, 610-619 (2007).
- Grillo, M. A., Colombatto, S. S-Adenosylmethionine and protein methylation. Amino Acids. 28, 357-362 (2005).
- Fujioka, M. Mammalian small molecule methyltransferases: their structural and functional features. Int. J. Biochem. 24, 1917-1924 (1992).
- Borchardt, R. T., Salvatore, F., Borek, E., Zappia, V., Williams-Ashman, H. G., Schlenk, F. . The Biochemistry of S-Adenosylmethionine. , 151-171 (1977).
- Hoffman, J. L. Chromatographic analysis of the chiral and covalent instability of S-adenosyl-l-methionine. Biochemistry. 25, 4444-4449 (1986).
- Keller, A., Nesvizhskii, A. I., Kolker, E., Aebersold, R. Empirical statistical model to estimate the accuracy of peptide identifications made by MS/MS and database search. Anal. Chem. 74, 5383-5392 (2002).
- Nesvizhskii, A. I., Keller, A., Kolker, E., Aebersold, R. A statistical model for identifying proteins by tandem mass spectrometry. Anal. Chem. 75, 4646-4658 (2003).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon