Method Article
FISH pour le diagnostic préimplantatoire génétique
Dans cet article
Résumé
Cet article décrit la sélection des sondes appropriées pour les mono-cellules de poisson, les techniques de propagation pour les noyaux des blastomères, et l'hybridation in situ et la notation du signal, appliquées au diagnostic génétique pré-implantatoire (DPI) dans un contexte clinique.
Résumé
Diagnostic pré-implantatoire génétique (DPI) est une alternative établie pour le diagnostic prénatal, et implique la sélection pré-implantatoire des embryons à partir d'une cohorte générées par la technologie de reproduction assistée (TRA). Cette sélection peut être nécessaire en raison de maladie familiale monogénique (par exemple la mucoviscidose), ou parce que l'un des partenaires réalise un réarrangement chromosomique (par exemple une translocation réciproque dans les deux sens). Le DPI est disponible pour les couples qui ont déjà eu des enfants affectés et / ou dans le cas des réarrangements chromosomiques, fausses couches à répétition, ou l'infertilité. Les ovocytes aspirés stimulation ovarienne sont fécondés par la suite à l'immersion dans le sperme in vitro (FIV) ou par injection intracytoplasmique d'un spermatozoïde individuel (ICSI). Pré-implantatoire des embryons au stade clivage sont biopsiées, habituellement par le prélèvement d'une cellule unique sur 3 jours après la fécondation, et la cellule de biopsie est testé pour établir le statut génétique de l'embryon. L'hybridation fluorescente in situ (FISH) sur les noyaux de cellules fixes biopsiés avec la cible spécifique des sondes d'ADN est la technique de choix pour détecter les déséquilibres chromosomiques associées à des réarrangements chromosomiques, et de sélectionner les embryons féminins dans les familles avec des maladies liées à l'X pour lesquels il n'existe aucune mutation spécifique de test. FISH a également été utilisé pour les embryons de l'écran de l'aneuploïdie chromosomique spontanée (aussi connu comme PGS ou PGD-AS), afin de tenter d'améliorer l'efficacité de la procréation assistée, mais la valeur prédictive de ce test en utilisant la diffusion et la technique de FISH décrite ici est susceptible d'être beaucoup trop faible dans les mains de la plupart des gens, et il n'est pas recommandé pour une utilisation clinique de routine. Nous décrivons le choix des sondes appropriées pour les mono-cellules de poisson, les techniques de propagation pour les noyaux des blastomères, et l'hybridation in situ et la notation du signal, appliqué aux DPI dans un cadre clinique.
Protocole
1. Lyse et la diffusion de noyau d'une blastomère biopsiés
- Tampon de lyse cellulaire des cellules propagation (0,2% de Tween 20 dans 0,01 M HCl, pH 2,0) doit être préparée 24 heures à l'avance et stocké à -20 ° C. Préparer 100 ml et 20 ml de filtre dans un conteneur de 30 ml stériles universelle en utilisant une seringue stérile de 20 ml et un filtre seringue. Distribuer des aliquotes de 1 mL dans 10 à 15 1,7 ml microtubes stériles, fermer et étiqueter les tubes avant de le congeler. Il est recommandé d'avoir deux lots différents est l'utilisation, le nouveau lot et le lot précédent, qui a été testé et peut être utilisé si le nouveau lot est insatisfaisante.
- Décongeler le tampon de lyse à température ambiante 30 minutes avant la biopsie. Pour des raisons pratiques, la température de travail se situe entre la température ambiante et la température de la main.
- Score d'un petit cercle (environ 5 mm de diamètre) sur la partie inférieure d'une lame d'amine-couché (par exemple Genetix) en utilisant un stylo de diamants et de pré-label de la diapositive avec le numéro de dossier, numéro de la diapositive unique, et la date de biopsie. Utilisez une lame séparée pour chaque blastomère dans l'ordre numérique, et l'étiquette avec le numéro de l'embryon. Les diapositives doivent être étiquetés avec un crayon dur comme 4H, et "effacé" avec un gant de latex pour enlever toute la poussière de graphite.
- Placez un petit volume de tampon de lyse dans le cercle.
- Transférer le blastomère biopsié dans le tampon de lyse. Si nécessaire, ajouter un tampon de lyse dans le cercle jusqu'à ce que la cellule commence à se lyser, la cellule doit lyser complètement et le cytoplasme se disperser avant le séchage de la mémoire tampon.
- Observez le noyau afin de s'assurer qu'il reste dans le cercle et ne sont pas perdues; si la cellule n'a pas de noyau ou a noyaux multiples, la biopsie d'une autre cellule.
- Laisser la lame sécher à l'air à température ambiante.
2. Hybridation in situ d'un seul noyau de blastomères
- Préparer une série d'éthanol (70, 90 et 100%), composé dans l'eau distillée stérile.
- Allumez-le et mettre le bloc chaud (par exemple Hybaid Omnislide ou Vysis Hybrite) à 75 ° C.
- Sondes de dégivrage, vortex et centrifuger. Les réactifs doivent être témoin et vérifiées pour être correcte avant de faire le mélange de la sonde. Volumes de pipette comme spécifié par le fabricant pour rattraper le mélange de sonde dans un tube à 0,65 ml microtubes stériles et le vortex et centrifuger avant l'utilisation. Le volume total du mélange de la sonde devrait être suffisante pour permettre à 2 l par le noyau pour être testé et arrondi au plus proche des 10 uL pour permettre une marge de sécurité.
- Pré-laver les lames dans des bocaux Coplin en utilisant un tampon phosphate salin (PBS) (pH 7,0: 0,14 M de NaCl, 3 mM de KCl, 10 mM Na 2 HPO 4, 2 mM KH 2 PO 4) pendant 5 min à température ambiante.
- Rincer les lames à deux reprises dans de l'eau distillée stérile.
- Déshydrater les lames avec la série d'éthanol (70, 90 et 100%) pendant 2 min chacun à la température ambiante et l'air sec. Assurez diapositives sont entièrement immergés et si toute la poussière de graphite flotte à la surface, éponger avec un tissu propre.
- Enregistrer la position du noyau dans le cercle en visualisant avec un microscope à contraste de phase.
- Déshydrater à l'éthanol à 100% pendant 2 min à température ambiante et l'air sec.
- Appliquer 2 pl de mélange de sonde, et couvrir avec une lamelle de 9 x 9 mm (un quart de 18 x 18 mm lamelle n ° 1).
- Scellez les bords de la lamelle de caoutchouc ciment (gomme de vache par exemple; Proofing vache).
- Codenature les diapositives sur un bloc chaude à 75 ° C pendant 5 min, puis l'hybridation des diapositives nuit (16-20 h) dans une chambre humidifiée à 37 ° C. Mêle sonde constitués entièrement de sondes centromère (ie pour liée au sexe des cas) donnera un résultat satisfaisant après 60 min d'hybridation.
- Préparez un bain d'eau avec des jarres Coplin suffisant et la chaleur à 71 ° C.
- Préparer un 0.4x citrate saline standard (SSC) de solution de lavage sévères (pH 7,0 à 71 ° C, 0,06 M de NaCl, 6 mM C 6 H 5 Na 3 O 7 .2 H 2 O) et de chaleur dans l'eau du bain.
- Déposer 50 ml de lavage rigoureuses par Coplin jar nécessaires et vérifier que la température est de 71 ° C immédiatement avant l'utilisation à l'aide d'un thermomètre propre.
- Retirer délicatement le ciment de caoutchouc de chaque diapositive et rincer la lamelle à l'aide 4x SSC/0.05% de Tween 20 (pH 7,0) à température ambiante.
- Laver les lames dans le lavage SSC 0.4x strictes à 71 ° C pendant 5 min. Lavez pas plus de 6 diapositives par jarre de Coplin.
- Laver les lames dans Tween 20% SSC/0.05 4x à température ambiante pendant 2 min.
- Si le mélange sonde contient la sonde indirectement marqué (s), égoutter les diapositives de l'excès de liquide et d'appliquer 20 uL d'anticorps par fluorescence conjugué dans un carré de 20 x 20 mm de Parafilm.
- Incuber dans une chambre humidifiée à 37 ° C pendant 15 min.
- Retirez le Parafilm et laver une fois dans Tween 20% SSC/0.05 4x à température ambiante pendant 2 min.
- Laver deux fois pendant 2 min dans du PBS unela température ambiante t et égoutter les lames.
- Appliquer 6 uL d'DAPI / Vectashield (160 ng de 4 ',6-diamidino-2-phénylindole dihydrochlorure dans 1 ml de milieu de montage Vectashield, Vector Laboratories) à un 22 x 22 mm non. 0 lamelle et inverser la glisser sur la lamelle.
- Blot et sceller les bords de la lamelle avec du vernis à ongles transparent.
3. Analyse au microscope à fluorescence convenablement équipé avec des filtres appropriés pour les sondes utilisées.
- Score signaux par visualisation directe en utilisant un microscope à fluorescence et unique filtres passe-bande pour chaque fluorochrome dans le dosage. Chaque noyau devrait être marqué par deux analystes. Une ligne directrice générale devrait conduire à marquer d'un signal unique lorsque deux signaux sont espacées de moins d'un domaine (le signal de largeur) à part, cependant, le jugement fondé sur l'expérience doit être exercé à interpréter les signaux de taille variable, l'intensité et de séparation.
- Utiliser un logiciel d'imagerie (par exemple Isis, métasystèmes, Altlussheim, Allemagne; CytoVision, Genetix) pour capturer une image du noyau pour la confirmation du diagnostic visuel, et pour l'archivage des images dans le cadre d'un plan d'assurance qualité du laboratoire.
4. Les résultats représentatifs:
Métaphase et noyaux interphasiques de cultures de lymphocytes du sang périphérique devraient être examinés pour confirmer que les sondes sélectionnées sont spécifiques pour les chromosomes de translocation, instructive pour les points d'arrêt (les sondes subtelomere devrait s'hybrider seulement pour les segments translocation et la sonde centromère (s) à l'centric segment (s)) et les signaux dans les noyaux interphasiques doit être brillante et discrète. Notation du nombre de signaux pour chaque sonde dans 100 noyaux interphasiques de chaque partenaire est recommandé d'évaluer l'efficacité de l'essai. Dans ce cas, deux signaux ont été marqués en 95% -99% de noyaux pour chaque sonde. La figure 1 montre une métaphase et noyau interphasique d'une préparation à une translocation réciproque entre les bras courts des chromosomes 5 et 9.
Signaux dans les noyaux interphasiques à partir de blastomères d'embryons doit être brillant et discret et marqué à l'aide séparées filtres passe-bande pour les couleurs utilisées. La figure 2 montre un noyau de blastomères avec un motif de signal normal (2 copies pour tous les loci testés) compatible avec un complément chromosomique normal ou équilibré pour le chromosome 5 et 9, et un noyau avec un modèle de signal anormal compatible avec un produit de la translocation déséquilibrée qui a monosomie (une copie) pour le segment transloqué du chromosome 5 et trisomie (3 copies) pour le segment transloqué du chromosome 9.
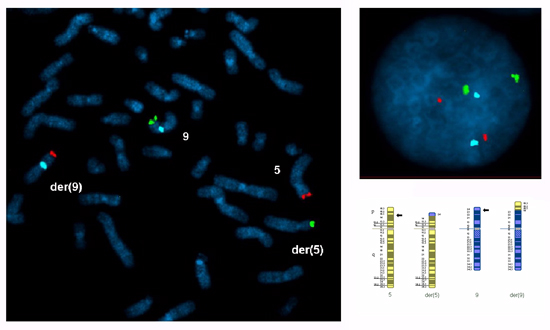
Figure 1 Un écart métaphase et un noyau interphasique préparés à partir de cultures de lymphocytes du sang périphérique à partir d'un porteur d'une translocation réciproque entre les bras courts des chromosomes 5 et 9 avec points d'arrêt à 5p14.3 et 9p24.1:. 46, XY, t (5 ; 9) (p14.3;. p24.1) ish t (5; 9) (5ptel48-, 9ptel30 +; 9ptel30-, 5ptel48 +, 9cen +). Sondes FISH ont été sélectionnés pour les deux segments translocation (5p14.3 → 5pter, rouge Cytocell subtelomere5ptel48 TexasRed; 9p24.1 → 9pter, vert Cytocell subtelomere 9ptel30 FITC) et centrée sur le segment du chromosome 9 (9p24.1 → 9qter, bleu Abbott CEP 9 alpha-satellites SpectrumAqua).
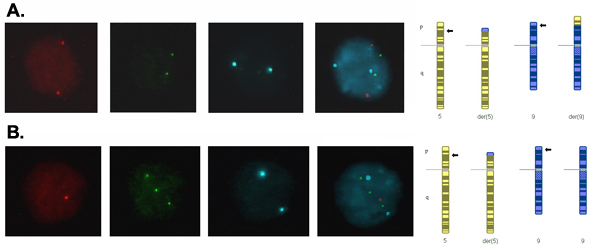
Signaux Figure 2. Blastomère en interphase noyaux de jours-3 embryons capturées à l'aide d'un filtre différent pour chaque fluorochrome et ont fusionné pour former une image composite. (A) Deux signaux pour chaque sonde indiquant deux copies de chaque locus, ce qui est cohérent avec un complément normal ou équilibré pour les chromosomes de translocation. (B) Un signal rouge, vert et trois signaux de deux signaux bleu indiquant une copie du segment transloqué du chromosome 5, trois copies du segment transloqué du chromosome 9 et deux copies du segment centrique du chromosome 9, ce qui est cohérent avec les voisins -1 ségrégation de la translocation résultant en un produit déséquilibrée avec monosomie pour 5p14.3 → 5pter et une trisomie pour 9p24.1 → 9pter.
Discussion
L'application de l'hybridation fluorescente in situ (FISH) à une cellule d'embryon unique (blastomère) présente des défis particuliers à la fois dans les pratiques et dans l'interprétation du modèle de signal. La cellule de biopsie doit être réparti dans une zone pré-définie sur la lame afin de faciliter ses poissons de localisation suivants; un soin extrême doit être pris en veillant à ce que la cellule est lysée, que le cytoplasme a été dispersée, et que le noyau est visible et intacte, et, comme le diagnostic repose sur les résultats de cette cellule unique, la notation et l'interprétation stricte des lignes directrices devraient être appliquées. Toutefois, dans des mains expérimentées, le poisson est une technique robuste pour diagnostic pré-implantatoire génétique (DPI) en pratique clinique. Le principe du DPI par FISH est que les sondes spécifiques de la cible d'ADN marqués avec des fluorochromes différents ou haptènes peuvent être utilisés pour détecter le nombre de copies de loci spécifiques, et ainsi de détecter les déséquilibres chromosomiques associées à de ségrégation méiotique des réarrangements chromosomiques (1), y compris Robertsonienne translocations, translocations réciproques, inversions, et des réarrangements complexes (2). Le poisson peut aussi être utilisé pour sélectionner des embryons féminins dans les familles avec X-linked maladie, pour laquelle il n'existe pas de test de mutation spécifique (3, 4). De façon plus controversée, le poisson a également été utilisé pour dépister l'aneuploïdie chromosomique sporadiques afin d'essayer d'améliorer l'efficacité de la reproduction assistée (5, 6); toutefois, la valeur prédictive de ce test à l'aide FSIH est susceptible d'être trop bas dans la plupart des gens les mains et il n'est pas recommandé pour une utilisation clinique de routine (7). Cet article se concentre sur les aspects techniques et les limites de la FISH appliquée aux cliniques unicellulaires diagnostic.
Les méthodes de propagation et la fixation des blastomères simples comprennent méthanol / acide acétique (5, 8), Tween / HCl (9), et une combinaison de Tween / HCl et méthanol / acide acétique (10). Les variations incluent le traitement hypotonique des cellules avant l'épandage et / ou la pepsine et le traitement paraformaldéhyde après fixation. La méthode doit être correctement validé pour le laboratoire (14). La méthode Tween / HCl est décrite en détail dans cet article. La méthode Tween / HCl est techniquement simple et hautement reproductibles dans différents laboratoires. Cette méthode peut être utilisée pour préparer des noyaux simples pour le diagnostic FISH de la détermination du sexe et des réarrangements chromosomiques avec une précision acceptable de diagnostic (7).
Mêle sonde peut combiner directement et indirectement étiqueté sondes marquées, et des sondes de différents fabricants. Sondes pour connaître les régions du chromosome polymorphe (11, 12), ou ceux qui sont connus de contre-s'hybrider de façon significative avec d'autres chromosomes (13) doit être évitée, mais peut être utilisé s'il est démontré d'être précis et adapté à un DPI par des essais préalables sur les deux partenaires de reproduction . Fluorochromes disponibles / haptènes et des stratégies pour les sondes discriminante sont les suivantes: le rouge Texas (TR), l'isothiocyanate de fluorescéine (FITC), SpectrumGreen (Vysis), SpectrumOrange (Vysis), SpectrumAqua, sondes biotinylées détectés avec TR-avidine, FITC-avidine, ou Cy-5 streptavidine (visualisés à l'aide d'un filtre Farred), un mélange de sondes rouges et vertes pour produire un signal jaune, un second tour de l'hybridation et une troisième couleur créé par la capture séquentielle des SpectrumOrange utilisant un TexasRed et un filtre SpectrumGold).
Un ensemble de sondes contenant trois sondes, spécifiques pour les régions de l'centromère des chromosomes X et Y et un autosome, est recommandé pour la détermination du sexe (14); la sonde autosomique est utilisé pour établir la ploïdie et ainsi faire la différence entre X trisomie (2n, 47 , XXX) et triploïdie (3n, 69, XXX), et entre tétrasomie X (48, XXXX) et tétraploïdie (4n, 92, XXXX). Une sonde typique située à appliquer dans ce contexte est le mélange AneuVysion Abbott contenant l'alpha-satellites X, Y, et 18; cet ensemble de sonde a été démontré que le polymorphisme ont un taux très faible, et donc de pré-traitement du travail de reproduction des partenaires Il n'est pas nécessaire (14).
Le mélange de sonde pour tout réarrangement spécifique doit: Idéalement contiennent des sondes au moins suffisant pour détecter tous les produits attendus de la transposition à un déséquilibre chromosomique. Si cela n'est pas possible, la sonde se mélange où les produits détectés déséquilibrés ont été évalués comme non viables dans une grossesse reconnaissables et sont susceptibles d'avoir une très faible prévalence peut être acceptable (14). Être testés sur des cultures de lymphocytes métaphases des deux partenaires de la reproduction. Au moins dix propage métaphase devraient être examinés pour la spécificité de la sonde, polymorphismes et hybridation croisée, et, pour le transporteur réarrangement chromosomique, afin de s'assurer que les sondes hybrident comme prévu aux différents segments du réarrangement. En outre, au moins 100 noyaux interphasiques de ces préparations doit être marqué d'évaluer la spécificité du signal, la luminosité et discret (14).
Commercial ensembles PGS sonde sont disponibles (par exemple, Abbott MultiVysion PB ou TCP), en ciblant les chromosomes les plus fréquemment trouvés à aneuploïdes dans les produits de la conception, et comprenant une seule sonde par chromosome ciblé. Typiquement le noyau peut avoir un seconde hybridation avec des sondes pour les chromosomes supplémentaires fournissant un test pour les chromosomes 13, 15, 16, 18, 21, 22, et XY (14). La valeur prédictive de ce test en utilisant la diffusion et la technique de FISH décrite ici est susceptible d'être beaucoup trop faible dans les mains de la plupart des gens et il n'est pas recommandé pour une utilisation clinique de routine (7, 15).
Des techniques telles que l'hybridation génomique comparative (CGH) appliquée à des cellules individuelles sont en mesure de tester pour le nombre de copies de chaque chromosome (Wilton et al. 2001), et l'utilisation de polymorphismes nucléotidiques simples (SNP) des tableaux et l'analyse quantitative (Wells et al . 2008) ou "karyomapping« génotypage (Handyside et al. 2009) sont prometteurs techniques alternatives pour détecter l'aneuploïdie chromosomique. Toutefois, la limite de résolution et de précision pour le déséquilibre du segment restent incertaines et il est probable que les poissons continuent d'être la technique de choix pour les réarrangements chromosomiques impliquant de petits segments.
Déclarations de divulgation
matériels
| Material Name | Type | Company | Catalogue Number | Comment |
|---|---|---|---|---|
| Name | Company | Catalog Number | Comments | |
| Hydrochloric Acid, 2.5L | VWR | 101254H | ||
| Sodium Chloride, 5Kg | VWR | 443827W | ||
| Potassium Chloride, 250g | VWR | 26764.232 | ||
| di-Sodium Hydrogen Orthophosphate Anhydrous, 500g | VWR | 102494C | ||
| Potassium Dihydrogen Orthophosphate, 500g | VWR | 10203 4B | ||
| Sodium Hydroxide Pellets, 500g | VWR | 28245.265 | ||
| Tween20, 500mL | VWR | 663684B | ||
| Ethanol, 2.5L | VWR | 8.18760.2500 | Duty Paid | |
| Tri-Sodium Citrate, 5Kg | VWR | 27833.363 | ||
| Cow Gum, 250mL | Cow Proofings Ltd. | No longer available | Old stocks can still be found in art shops | |
| DAPI/Antifade ES, 0.125μg/mL, 500ul | Cytocell | DES500L | 150μl vial rec’d with each Cytocell TEL Probe | |
| Nail Varnish, 12mL | Boots | 10088316001 | ||
| Amine-coated Slides, 25 | Genetix | K2615 | ||
| DAPI, 0.1mL | Sigma-Aldrich | D-9542 | ||
| Vectashield, 10mL | Vector Laboratories | H1000 | ||
| Texas-Red-avidin, 0.001mL | Vector Laboratories | A-2016 | ||
| FITC-avidin, 0.001mL | Vector Laboratories | A-2011 | ||
| Cy-5 Streptavidin, 0.001mL | GE Healthcare | PA45001 | ||
| Microcentrifuge Tubes, 1.75mL, box of 500 | Thistle Scientific | AX-MCT-175-C | ||
| Microcentrifuge Tubes, 0.6mL, box of 1000 | Thistle Scientific | AX-MCT-060-C-CS | ||
| 30ml Universal Containers, box of 400 | Sterilin | 128B | Available from NHS Supply Chain, Cat No. KCP053 | |
| Hot Block | ||||
| Spirit Burner Spirit Burner Socket Spirit Burner Wick | VWR VWR VWR | 451-0107 451-3112 451-3111 | ||
| Plugged Glass Pipettes | Fisher Scientific | PMK-300-052R | ||
| Dissecting Microscope | Wild Heerbrugg | |||
| Tissues, 100boxes | NHS SUPPLY CHAIN | MJT058 | ||
| Metal Culture Trays | ||||
| Plastic Melamine Trays | VWR | 682-009 | ||
| Oven | ||||
| Mouth Pipette Mouthpiece | Scientific Laboratory Supplies | HAE3700 | ||
| Tubing | ||||
| Syringe Filter, 0.2μm pore size, 25mm diameter | Fisher Scientific | FDM-340-010U | ||
| Diamond Pen | VWR | 818-0021 | ||
| Hot Block for Slides | Thermo Hybaid | HBOSBB | ||
| Hybridization Chamber | ||||
| Tissue Roll | NHS SUPPLY CHAIN | MJT004 | ||
| Schieferdecker Jar | VWR | 631-9313 | ||
| Coplin Jar | VWR | 631-9331 | ||
| Forceps, Fine | VWR | 232-2123 | ||
| Forceps, Blunt | VWR | 232-2113 | ||
| 1000mL Beaker | VWR | 213-1642 | ||
| Phase Contrast Microscope | Nikon | E200 | ||
| Fibre-free Blotting Cards, box of 100 | Fisher Scientific (Raymond Lamb) | SDE1 | ||
| Blue-Frosted Microscope Slides, box of 100 | Scientific Laboratory Supplies | MIC3022 | ||
| Coverslips, 18x18mm, No. 1, box of 200 | Scientific Laboratory Supplies | MIC3110 | ||
| Coverslips, 22x22mm, No. 0, box of 200 | Scientific Laboratory Supplies | MIC3104 | ||
| Coverslips, 22x50mm, No. 0, box of 100 | Scientific Laboratory Supplies | MIC3206 | ||
| 1mL Syringe, box of 100 | NHS Supply Chain | FWC045 | ||
| 1mL Pipette Tip, 10racks of 1000 | Thistle Scientific | AX-T-1000-C-L-R | ||
| Incubator | LEEC | |||
| Waterbath | Grant | W22 | ||
| Alcohol Thermometer | Various sources available | |||
| pH Meter | Jenway | 3305 | ||
| pH Electrode | VWR | 662-1759 | BNC Plug to fit Jenway pH meter 3305 | |
| Heated Stirrer | Bibby | HC502 | ||
| 1mL Pasteur Pipettes, box of 500 | Scientific Laboratory Supplies | PIP4202 | ||
| Parafilm, 50mm x 75m | VWR | 291-1214 |
Références
- Scriven, P. N., Handyside, A. H., Ogilvie, C. M. Chromosome translocations: segregation modes and strategies for preimplantation genetic diagnosis. Prenat. Diagn. 18, 1437-1449 (1998).
- Mackie Ogilvie, C., Scriven, P. N., Harper, J. C. Preimplantation genetic diagnosis for chromosome rearrangements. Preimplantation Genetic Diagnosis. , 194-201 (2009).
- Harper, J. C., Coonen, E., Ramaekers, F. C. S., Delhanty, J. D. A., Handyside, A. H., Winston, R. M. L., Hopman, A. H. N. Identification of the sex of human preimplantation embryos in two hours using an improved spreading method and fluorescent in situ hybridisation using directly labelled probes. Hum. Reprod. 9, 721-724 (1994).
- Mackie Ogilvie, C., Scriven, P. N., Harper, J. C. Preimplantation genetic diagnosis for sex-linked diseases and sex-selection for non-medical reasons. Preimplantation Genetic Diagnosis. , 230-235 (2009).
- Munné, S., Lee, A., Rosenwaks, Z., Grifo, J., Cohen, J. Diagnosis of major chromosome aneuploidies in human preimplantation embryos. Hum. Reprod. 8, 2185-2192 (1993).
- Mackie Ogilvie, C., Scriven, P. N., Harper, J. C. Preimplantation genetic diagnosis for infertility (preimplantation genetic screening). Preimplantation Genetic Diagnosis. , 203-229 (2009).
- Scriven, P. N., Bossuyt, P. M. Diagnostic accuracy: theoretical models for preimplantation genetic testing of a single nucleus using the fluorescence in situ hybridization technique. Hum Reprod. Cytogenetics. 5, 394-400 (2010).
- Coonen, E., Dumoulin, J. C., Ramaekers, F. C., Hopman, A. H. Optimal preparation of preimplantation embryo interphase nuclei for analysis by fluorescence in-situ hybridization. Hum. Reprod. 9, 533-537 (1994).
- Dozortsev, D. I., McGinnis, K. T. An improved fixation technique for fluorescence in situ hybridization for preimplantation genetic diagnosis. Fertil. Steril. 76, 186-188 (2001).
- Hsu, L. Y., Benn, P. A., Tannenbaum, H. L., Perlis, T., Carlson, A. D. Chromosomal polymorphisms of 1, 9, 16, and Y in 4 major ethnic groups: a large prenatal study. Am. J. Med. Genet. 26, 95-101 (1987).
- Shim, S. H., Pan, A., Huang, X. L., Tonk, V. S., Varma, S. K., Milunsky, J. M., Wyandt, H. E. FISH variants with D15Z1. J. Assoc. Genet. Technol. 29, 146-151 (2003).
- Knight, S. J., Flint, J. Perfect endings: a review of subtelomeric probes and their use in clinical diagnosis. J. Med. Genet. 37, 401-409 (2000).
- Thornhill, A. R., de Die-Smulders, C. E., Geraedts, J. P., Harper, J. C., Harton, G. L., Lavery, S. A., Moutou, C., Robinson, M. D., Schmutzler, A. G., Scriven, P. N., Sermon, K. D., Wilton, L. ESHRE PGD Consortium (PGS) ESHRE PGD Consortium 'Best practice guidelines for clinical preimplantation genetic diagnosis (PGD) and preimplantation genetic screening (PGS)'. Hum. Reprod. 20, 35-48 (2005).
- Munné, S., Wells, D., Cohen, J. Technology requirements for preimplantation genetic diagnosis to improve assisted reproduction outcomes. Fertil. Steril. 94, 408-430 (2010).
- Wilton, L., Williamson, R., McBain, J., Edgar, D., Voullaire, L. Birth of a healthy infant after preimplantation confirmation of euploidy by comparative genomic hybridization. N. Engl. J. Med. 345, 1537-1541 (2001).
- Wells, D., Alfarawati, S., Fragouli, E. Use of comprehensive chromosomal screening for embryo assessment: microarrays and CGH. Mol. Hum. Reprod. 14, 703-710 (2008).
- Handyside, A. H., Harton, G. L., Mariani, B., Thornhill, A. R., Affara, N., Shaw, M. A., Griffin, D. K. Karyomapping, a universal method for genome wide analysis of genetic disease based on mapping crossovers between parental haplotypes. J. Med. Genet. 47, 651-658 (2010).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon