Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Séquençage de la microflore bactérienne dans le sang périphérique: notre expérience avec des patients infectés par le VIH
Dans cet article
Résumé
Notre expérience montrera comment effectuer une analyse par séquençage des espèces bactériennes translocation dans le sang périphérique des patients VIH positifs.
Résumé
Le tractus gastro-intestinal sain est physiologiquement colonisé par une grande variété de microbes commensaux qui influencent le développement de l'1,2 humorales et cellulaires du système immunitaire muqueux.
Microbiote est protégé par le système immunitaire par une barrière forte muqueuses. Infections et antibiotiques sont connus pour altérer la barrière normale tractus gastro-intestinal et la composition des bactéries résidentes, ce qui peut entraîner des possibles anomalies immunitaires 3.
VIH provoque une brèche dans la barrière gastro-intestinaux avec défaillance progressive de l'immunité des muqueuses et des fuites dans la circulation systémique des bioproduits bactériennes, telles que des lipopolysaccharides et des fragments d'ADN bactérien, qui contribuent à l'activation immunitaire systémique 4-7. Translocation microbienne est impliqué dans le VIH / SIDA immunopathogenèse et réponse au traitement 4,8.
Nous avons cherché à caractériser la composition des bactéries translocation dans le sang périphérique des patients infectés par le VIH. Pour poursuivre notre but, nous avons créé une réaction PCR pour le gène 16S panbacteric ribosomial suivie d'une analyse par séquençage.
Brièvement, le sang total à la fois de sujets infectés par le VIH et sain est utilisé. Étant donné que les individus sains normaux présents homéostasie intestinale aucune translocation de la microflore est attendu chez ces patients. Après la collecte de sang total par ponction veineuse et la séparation du plasma, l'ADN est extrait du plasma et utilisés pour effectuer un large éventail de réaction PCR pour le gène 16S panbacteric ribosomial 9. Après purification des analyses de produits PCR, le clonage et le séquençage sont effectuées.
Protocole
Manipulation des échantillons sanguins infectés au VIH nécessite quelques recommandations importantes.
Tous les échantillons de sang doivent être transportés dans robustes conteneurs étanches. Des précautions doivent être prises lors de la collecte du spécimen pour éviter toute contamination de l'extérieur du conteneur et de tout document accompagnant le spécimen.
Toutes les personnes de traitement du sang infectés doivent porter des gants. Les gants doivent être changés et lavés les mains après l'achèvement du traitement des échantillons.
Traitement des échantillons de sang infectés par le VIH doit être effectuée sous une hotte de classe II du cabinet Biohazard.
Les aides mécaniques de pipetage doit être utilisé.
L'utilisation de seringues ou autres objets tranchants (y compris par exemple les pipettes en verre ou tubes capillaires) doit être limitée aux situations dans lesquelles il n'ya pas d'alternative.
Surfaces de laboratoire doivent être décontaminés avec un désinfectant chimique approprié après un déversement de sang et quand les activités de travail sont terminés.
Les matériaux contaminés utilisés doivent être décontaminés avant réutilisation ou doivent être éliminés correctement via la route des déchets cliniques.
Chaque incident d'exposition professionnelle à du sang potentiellement infectieux ou des liquides (ie, ceux nécessitant des précautions universelles) doivent être traités comme une urgence médicale comme les interventions doivent être initiées rapidement pour être efficace.
Le protocole nécessite 5 jours pour son achèvement. Le calendrier est détaillé dans la Figure 1.
Les espèces bactériennes sont identifiés en utilisant les méthodes décrites dans la Figure 2.
1. Prélèvement d'échantillons
- 9 ml de sang total est prélevé dans des tubes contenant de l'EDTA.
- Les tubes sont centrifugés à 2000 rpm pendant 10 minutes à température ambiante pour obtenir un plasma.
- Le plasma est recueilli dans une solution stérile de 2 ml Eppendorf tube.
2. Extraction d'ADN d'échantillons de plasma
- Suffisamment désinfecter le capot, des pipettes et le matériel nécessaire pour l'expérience en vue de garantir la stérilité.
- Placer tous les matériaux sous les lumières UV pendant au moins 30 minutes.
- Essuyez les gants avec un désinfectant.
- Tremper un essuie quelques dans l'éthanol et les placer sous le capot. Chaque fois qu'un pourboire est jeté essuyer la pipette sur les serviettes de papier humide.
L'ADN est extrait en utilisant un kit commercial en suivant les instructions du fabricant (Easy-DNA Kit, Invitrogen, Carlsbad CA, USA).
- 350 uL de plasma sont placés dans un tube stérile de 2 ml Eppendorf.
- 350 uL d'eau ultrapure filtrée sont utilisées comme contrôle négatif.
- Ajouter 10 uL de lysozyme (1mg/ml) pour les échantillons.
- Incuber pendant 30 minutes à 37 ° C.
- Ajouter 500 ul de solution de lyse et mélanger délicatement pour les échantillons.
- Incuber pendant 7 minutes à 65 ° C.
- Ajouter 900 ul de chloroforme pour les échantillons.
- Vortex vigoureusement jusqu'à les échantillons sont uniformément visqueux.
- Ajouter 200 pi de solution de précipitations et vortexer vigoureusement.
- Centrifuger les échantillons à 10500 rpm pendant 10 minutes à température ambiante pour séparer les phases et la forme de l'interface.
- Transférer la phase aqueuse supérieure dans un tube à centrifuger frais contenant 1 ml d'éthanol à 100%.
- Centrifuger les échantillons à 10500 rpm pendant 10 minutes à 4 ° C.
- Enlever l'éthanol.
- Ajouter 1 ml d'éthanol à 70%.
- Centrifuger les échantillons à vitesse maximale pendant 10 minutes à 4 ° C.
- Enlever l'éthanol. L'éthanol résiduelle doit être éliminée avec un pipettator.
- Reprendre le culot dans 50 ul d'eau ultrapure.
- Lire la concentration d'ADN avec un spectrophotomètre.
3. ARNr 16S par PCR
L'amplification par PCR est effectuée comme décrit précédemment 9.
- Amplifier l'ADN dans un mélange réactionnel de 100 uL composé de 10 pi de tampon PCR 10X, 5 pi de 25 mM de MgCl2, 5 pi de 2 mM dNTP total, 1 pl de 50 uM apprêt RW01 (AACTGGAGGAAGGTGGGGAT), 1 pl de 50 uM apprêt DG74 (AGGAGGTGATCCAACCGCA), 34 uL d'H20 et 0,5 uL de Taq polymérase (AmpliTaq Gold, Applied Biosystem, Foster City, CA, USA).
- Transférer le mélange PCR dans les filtres afin d'éviter toute contamination possible par Taq polymérase bactérienne.
- Filtres à centrifuger (Microcon, Millipore, Billerica, MA, USA) pendant 30 minutes à 500 FCR à 4 ° C.
- Aliquoter le mélange PCR selon le nombre d'échantillons qui doivent être amplifiés (positifs et négatifs contrôles PCR, les échantillons et de l'eau extraite).
- Utiliser les conditions suivantes thermocycleur: 94 ° C pendant 10 minutes, 40 fois 1 minute à chaque fois à 94 ° C, 55 ° C et 72 ° C, et 10 min à 72 ° C.
- Visualisez le produit de PCR sur un gel agarose 2%. Utiliser 100 pb échelle d'ADN. Taille du produit PCR est d'environ 360 pb (figure 3).
4. Purification de produits PCR
Seuls les échantillons positifs par PCR doit être purifiée. La purification est réalisée à l'aideun fabricant de kit commercial suivant les instructions du (PCR Purelink MICROKIT, Invitrogen, Carlsbad CA, USA).
- Ajouter 4 volumes de tampon de liaison contenant de l'isopropanol pour 1 volume de produit de PCR.
- Transfert de produits PCR avec Binding Buffer à une colonne.
- Centrifuger pendant 1 minute à 10000 rcf à température ambiante.
- Laver la colonne avec le tampon de lavage contenant de l'éthanol.
- Centrifuger pendant 1 minute à 10000 rcf à température ambiante.
- Centrifuger à vitesse maximale pendant 1 minute à température ambiante pour sécher la membrane de silice et d'enlever tout le tampon de lavage résiduelle avec de l'éthanol.
- Ajouter 10 uL d'eau ultrapure.
- Incuber pendant 1 minute à température ambiante.
- Centrifuger à vitesse maximale pendant 2 minutes à température ambiante pour recueillir l'ADN purifié.
5. Le clonage
Lysogénie Broth (LB) la préparation des plaques
- Dissoudre 25 gr de mélange de LB dans environ 800 ml d'eau.
- Amener le volume final à 1 L.
- Ajouter 15 gr de Bactoagar.
- Autoclave pendant 20 minutes.
- Après autoclavage, agiter énergiquement par rotation de la solution dans le flacon pour mélanger agar fondu.
- Refroidir la solution à 50 ° C.
- Ajouter l'ampicilline (50 ug / ml) et agiter jusqu'à ce qu'il soit dissout.
- Verser dans les boîtes à une profondeur d'environ 3mm.
- Laisser les plaques à température ambiante.
- Fais X-Gal (40mg/ml) et IPTG (100 mM) sur chaque plaque LB et incuber à 37 ° C jusqu'à utilisation.
Transformation des cellules compétentes
La transformation est réalisée à l'aide des instructions d'un fabricant kit commercial suivante (TOPO TA cloning kit, Invitrogen, Carlsbad CA, USA).
- Préparer le mélange de réaction de clonage (1-4 uL de PCR produit frais, 1 ml de solution de sel, 1 pl de vecteur et de l'eau si nécessaire).
- Mélanger délicatement la réaction et incuber pendant 5-10 minutes à température ambiante.
- Placez la réaction sur la glace.
- Ajouter 2 ul de la réaction de clonage dans un flacon de cellules compétentes et mélanger doucement.
- Incuber dans la glace pendant 30 minutes.
- Choc thermique des cellules pendant 30 secondes à 42 ° C.
- Transférer immédiatement le tube dans la glace.
- Ajouter 250 ul de milieu.
- Boucher le tube hermétiquement et secouer le tube horizontal à 37 ° C pendant 1 heure.
- Fais 50 ul de chaque transformation sur une plaque préchauffée sélective.
- Incuber une nuit à 37 ° C.
Après incubation, les colonies blanches et bleues se développent sur les plaques. Colonies blanches sont positifs pour l'insertion des produits de PCR et des colonies bleues sont négatifs pour l'insertion des produits de PCR.
6. L'analyse par séquençage
Réaction de séquençage
- Un mélange est faite en utilisant ces réactifs: 2,5 ul d'ADN, une primaire ul (5pmol / ul) 1,1 BigDye ul (Applied Biosystem, Foster City, CA, USA).
- Conditions Thermocycleur: 96 ° C pendant 1 minutes, 25 fois pour 96 ° C pendant 10 sec, 55 ° C pendant 15 sec et 60 ° C pendant 4 min.
Purification sur colonne
- Pour chaque réaction, préparer une colonne MicroSpin (Qiagen, Milan, Italie).
- Inverser la colonne et de vortex pour mélanger la résine.
- Aligner le fond de colonne, desserrer le couvercle ¼ de tour, et la colonne dans un microtube.
- Centrifugeuse 3200 rpm pendant 1 minute.
- Transfert colonne à un microtube propre.
- Soigneusement pipette la réaction de séquençage complet PCR dans le centre de la colonne.
- Centrifugeuse 3200 rpm pendant 1 minute.
- ADN purifié sera eluite dans le tube (20 pi d'eau ultra-pure).
L'analyse par séquençage
- Charger l'échantillon purifié (20 pi) dans 96 multipuits assiette.
- Charger la plaque dans le séquenceur. Séquenceurs d'ADN automatisé générer un chromatogramme en quatre couleurs montrant les résultats de l'exécution de séquençage.
- Entrée de la séquence nucléotidique comme une requête contre les banques de séquences publiques. La recherche est effectuée sur les bases de données NCBI et les serveurs, avec Basic Alignment Tool Recherche locale (BLAST).
- Considérons seulement les bactéries avec des homologies 98-100%.
7. Les résultats représentatifs
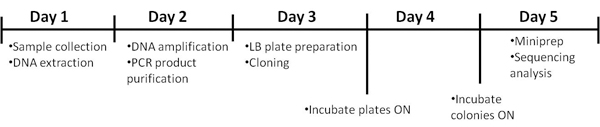
Figure 1. Chronologie de la procédure.
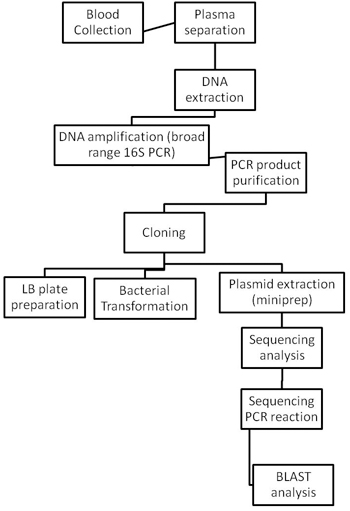
Figure 2. Diagrammes de flux de la procédure d'identification bactérienne entière.
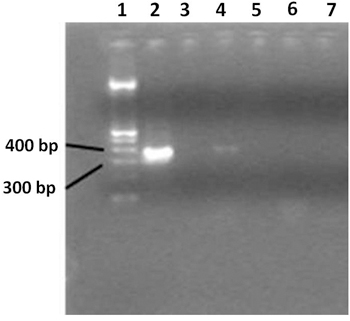
Figure 3. Gel à 2% d'agarose montrant large gamme de 16S ARNr produits de PCR. Voie 1 contient une échelle d'ADN de 100 pb, piste 2 contient le contrôle PCR positive, piste 3 montre le contrôle négatif de PCR. Lane 4 montre des échantillons provenant d'un patient séropositif, et la piste 5 contient de l'eau. Voie 6 shOWS échantillons à partir d'un individu sain et une réaction négative par PCR; piste 7 contient de l'eau. Seuls les patients VIH positifs affiche une amplification PCR positive. Eau ultrapure utilisée comme contrôle négatif pendant l'étape d'extraction, indique qu'aucune contamination s'est produite.
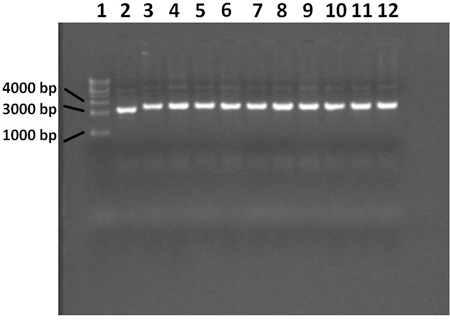
Figure 4. Gel à 0,7% d'agarose montrant plasmidique extrait avec la procédure miniprep. Voie 1 contient une échelle 1 ADN Kilobase, piste 2 contient la colonie de contrôle bleu, les voies 3 à 12 contiennent des colonies blanches. Le plasmide de la colonie bleue ne contient pas la notice du produit PCR. Tous les 10 colonies blanches contiennent plasmide avec l'insert correct.

Figure 5. Indique l'exemple d'analyse par séquençage des bactéries dans le plasma d'un VIH-positifs individuels. Nos résultats montrent que la translocation microbienne dans l'infection à VIH implique une flore polimicrobic, qui n'est pas vu dans le VIH-négatifs sujets, suggérant l'échec substantiel de l'immunité intestinale dans le contrôle de la translocation bactérienne.
Access restricted. Please log in or start a trial to view this content.
Discussion
Par la présente nous montrent un protocole PCR / séquençage de caractériser la translocation de bactéries dans le sang périphérique d'individus infectés par le VIH.
Résultats les plus fiables sont obtenus en utilisant le plasma recueilli dans des tubes contenant de l'EDTA-. Après prélèvement de sang, le plasma doit être séparé par centrifugation dans les 2 / 3 heures, pour éviter l'hémolyse et la dégradation de fragment d'ADN. Les échantillons sont ...
Access restricted. Please log in or start a trial to view this content.
Déclarations de divulgation
Remerciements
Nous sommes reconnaissants à Scimone Gianni pour l'assistance excellente de faire des vidéos.
Access restricted. Please log in or start a trial to view this content.
matériels
| Name | Company | Catalog Number | Comments |
| Nom de réactif | Société | Numéro de catalogue | Commentaires (optionnel) |
|---|---|---|---|
| Easty-DNA Kit | Invitrogen | K 180001 | |
| Chloroforme | Sigma-Aldrich | C2432 | |
| Éthanol | Sigma-Aldrich | E7203 | |
| UltraPure Waer | Invitrogen | 10977049 | |
| Le lysozyme | Fluka | 62970 | 10 ug / ml dans l'eau distillée |
| AmpliTaq Gold | Applied Biosystem | 26478701 | |
| Microcon 100 | Millipore | 42413 | |
| Des amorces de PCR | Invitrogen | ||
| Agarose | Eppendorf | C1343 | |
| Échelle d'ADN | Invitrogen | 15628019 | |
| Purelink PCR kit micro | Invitrogen | K310050 | |
| LB agar, la poudre | Invitrogen | 22700025 | |
| Bactoagar | Invitrogen | ||
| Ampicilline | Invitrogen | 11593027 | 10 mg / mL dans l'eau distillée |
| X-gal | Invitrogen | 15520034 | 40mg/mL dans le DMF |
| IPTG | Invitrogen | 15529019 | 100 mM dans de l'eau distillée |
| TOPO TA Cloning kit | Invitrogen | K450002 | |
| Purelink rapide kit Plasmid Miniprep | Invitrogen | K210010 | |
| Big Dye | Applied Biosystem | 4337455 | |
| DyeEx 2,0 tour kit | Qiagen | 63204 |
Références
- Hooper, L. V., Macpherson, A. J. Immune adaptations that maintain homeostasis with the intestinal microbiota. Nat Rev Immunol. 10, 159-169 (2010).
- Macpherson, A. J., Harris, N. L. Interactions between commensal intestinal bacteria and the immune system. Nat Rev Immunol. 4, 478-485 (2004).
- Kanauchi, O., Mitsuyama, K., Araki, Y., Andoh, A. Modification of intestinal flora in the treatment of inflammatory bowel disease. Curr Pharm Des. 9, 333-346 (2003).
- Brenchley, J. M. Microbial translocation is a cause of systemic immune activation in chronic HIV infection. Nat Med. 12, 1365-1371 (2006).
- Brenchley, J. M., Price, D. A., Douek, D. C. HIV disease: fallout from a mucosal catastrophe. Nat Immunol. 7, 235-239 (2006).
- Jiang, W. Plasma levels of bacterial DNA correlate with immune activation and the magnitude of immune restoration in persons with antiretroviral-treated HIV infection. J Infect Dis. 199, 1177-1185 (2009).
- Marchetti, G. Microbial translocation is associated with sustained failure in CD4+ T-cell reconstitution in HIV-infected patients on long-term highly active antiretroviral therapy. AIDS. 22, 2035-2038 (2008).
- Marchetti, G. Role of Microbial Translocation and Immune Hyperactivation in Disease Progression of HIV+ Patients with Preserved CD4 Count in the Absence of ART. The 17th Conference on Reteroviruses and Opportunistic Infections (CROI), San Francisco, CA, USA, , (2010).
- Greisen, K., Loeffelholz, M., Purohit, A., Leong, D. PCR primers and probes for the 16S rRNA gene of most species of pathogenic bacteria, including bacteria found in cerebrospinal fluid. J Clin Microbiol. 32, 335-351 (1994).
Access restricted. Please log in or start a trial to view this content.
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon